히스톤
Histone생물학에서 히스톤은 진핵세포 핵에서 발견되는 리신과 아르기닌 잔기에 풍부한 매우 염기성 단백질이다.그것들은 DNA가 감겨 [1][2]뉴클레오솜이라고 불리는 구조적 단위를 만드는 스풀 역할을 한다.뉴클레오솜은 30나노미터 섬유로 싸여 촘촘히 채워진 크로마틴을 형성한다.히스톤은 DNA가 엉키는 것을 방지하고 DNA 손상으로부터 보호합니다.또한 히스톤은 유전자 조절과 DNA 복제에 중요한 역할을 한다.히스톤이 없다면 염색체에서 풀린 DNA는 매우 길 것이다.예를 들어, 각각의 인간 세포는 완전히 뻗으면 약 1.8미터의 DNA를 가지고 있지만, 히스톤에 감길 때, 이 길이는 30nm 직경의 염색질 섬유 [3]중 약 90마이크로미터(0.09mm)로 감소한다.
히스톤에는 H1/H5(링커 히스톤), H2, H3, H4(핵심 히스톤)로 지정된 5개의 패밀리가 있습니다.뉴클레오솜 코어는 2개의 H2A-H2B 이합체와 H3-H4 4중합체로 구성되어 있습니다.히스톤 주위에 DNA를 단단히 감싸는 것은 양의 전하를 띤 히스톤과 음의 전하를 띤 인산염 골격 사이의 정전적 흡인력의 결과이다.
히스톤은 유전자 전사를 조절하기 위해 효소의 작용을 통해 화학적으로 변형될 수 있다.가장 일반적인 변형은 아르기닌 또는 리신 잔기의 메틸화 또는 리신의 아세틸화이다.메틸화는 전사인자와 같은 다른 단백질이 뉴클레오솜과 상호작용하는 방법에 영향을 줄 수 있다.리신아세틸화는 리신에 대한 양전하를 제거함으로써 히스톤과 DNA 사이의 정전 흡인을 약화시켜 DNA의 부분적인 풀림을 유발하여 유전자 발현에 보다 접근하기 쉽게 한다.
클래스 및 변종
히스톤에는 H1/H5, H2A, H2B, H3 및 [2][4][5][6]H4의 5가지 주요 패밀리가 있습니다.히스톤 H2A, H2B, H3, H4는 코어 히스톤, 히스톤 H1/H5는 링커 히스톤으로 알려져 있다.
코어 히스톤은 모두 이합체로 존재하며, 모두 히스톤 접힘 도메인을 가지고 있다는 점에서 유사합니다: 두 개의 루프로 연결된 세 개의 알파 나선입니다.특히 헤드테일(악수 [7]모티브라고도 함) 방식으로 구별되는 이합체 간의 상호작용을 가능하게 하는 것이 이 나선 구조입니다.그 결과 생성된 4개의 서로 다른 이합체가 합쳐져 지름 약 63 앵스트롬(DNA) 같은 입자)의 8중 핵소체 핵을 형성합니다.약 146쌍의 염기쌍(bp)의 DNA가 이 핵심 입자를 왼손 초나선 방향으로 1.65회 감싸 100앙스트롬 정도의 [8]입자를 형성합니다.링커 히스톤 H1은 DNA의 입구 및 출구 부위에 뉴클레오솜을 결합시켜 DNA를 제자리에[9] 고정시키고 고차 구조를 형성할 수 있게 한다.가장 기본적인 구성은 10 nm 파이버 또는 스트링 구성의 비즈입니다.이것은 각각의 뉴클레오솜 쌍(링커 DNA라고도 함)을 분리하는 약 50개의 염기쌍의 DNA로 뉴클레오솜 주위에 DNA를 감싼다.고차 구조에는 30 nm 섬유(불규칙한 지그재그를 형성)와 100 nm 섬유(이것들은 일반 세포에서 볼 수 있는 구조)가 포함됩니다.유사분열과 감수분열 동안, 응축 염색체는 뉴클레오솜과 다른 조절 단백질 사이의 상호작용을 통해 조립된다.
히스톤은 세포주기의 S상 동안 발현되는 표준 복제의존성 히스톤과 전체 세포주기 동안 발현되는 복제의존성 히스톤 변이체로 세분된다.동물에서 표준 히스톤을 코드하는 유전자는 전형적으로 염색체를 따라 군집되어 있고, 인트론이 없으며, 폴리A 꼬리 대신 3' 말단의 줄기 루프 구조를 사용한다.히스톤 변이체를 코드하는 유전자는 보통 군집화되지 않고 인트론을 가지며 mRNA는 폴리A 꼬리로 조절된다.복잡한 다세포 유기체는 일반적으로 다양한 기능을 제공하는 더 많은 수의 히스톤 변형을 가지고 있다.최근 다양한 히스톤 변종의 역할에 대한 데이터가 축적되고 있으며 변종 간의 기능적 연계와 유기체 [10]발달의 섬세한 조절이 강조되고 있다.다른 유기체의 히스톤 변종, 그 분류 및 변종별 특징은 "HistoneDB 2.0 - Variants" 데이터베이스에서 찾을 수 있다.
다음은 인간 히스톤 단백질의 목록입니다.
| 슈퍼패밀리 | 가족 | 서브패밀리 | 회원들 |
|---|---|---|---|
| 링커 | H1 | H1F | H1F0, H1FNT, H1FOO, H1FX |
| H1H1 | HIST1H1A, HIST1H1B, HIST1H1C, HIST1H1D, HIST1H1E, HIST1H1T | ||
| 핵심 | H2A | H2AF | H2AFB1, H2AFB2, H2AFB3, H2AFJ, H2AFV, H2AFX, H2AFY, H2AFY2, H2AFZ |
| H2A1 | HIST1H2AA, HIST1H2AB, HIST1H2AC, HIST1H2AD, HIST1H2AE, HIST1H2AG, HIST1H2AJ, HIST1H2AK, HIST1H2AL, HIST1HAM2 | ||
| H2A2 | HIST2H2AA3, HIST2H2AC | ||
| H2B | H2BF | H2BFM, H2BFS, H2BFWT | |
| H2B1 | HIST1H2BA, HIST1H2BB, HIST1H2BC, HIST1H2BD, HIST1H2BE, HIST1H2BG, HIST1H2BH, HIST1H2BG, HIST1H2BJ | ||
| H2B2 | HIST2H2BE | ||
| H3 | H3A1 | HIST1H3A, HIST1H3B, HIST1H3C, HIST1H3D, HIST1H3E, HIST1H3F, HIST1H3G, HIST1H3I, HIST1H3J | |
| H3A2 | HIST2H3C | ||
| H3A3 | HIST3H3 | ||
| H4 | H41 | HIST1H4A, HIST1H4B, HIST1H4C, HIST1H4D, HIST1H4E, HIST1H4F, HIST1H4G, HIST1H4H, HIST1H4J, HIST1H4H4H | |
| H44 | HIST4H4 |
구조.
뉴클레오솜 코어는 2개의 H2A-H2B 이합체와 1개의 H3-H4 사분자로 구성되어 있으며, 3차 구조(C2 대칭, 한 고분자는 다른 [8]고분자의 거울상)에 의해 거의 대칭인 두 개의 반쪽을 형성한다.H2A-H2B 이합체 및 H3-H4 사량체도 의사대칭성을 나타냅니다.4개의 '핵심' 히스톤(H2A, H2B, H3, H4)은 비교적 구조가 유사하며 진화를 통해 보존성이 높으며, 모두 '나선 회전 나선' 모티브(특정 DNA 서열을 인식하는 DNA 결합 단백질 모티브)를 특징으로 한다.그들은 또한 아미노산 구조의 한쪽 끝에 긴 '꼬리'의 특징을 공유합니다 - 이것은 번역 후 수정의 위치입니다 (아래 [11]참조).
고고 히스톤은 H3-H4와 같은 이합체 구조를 가지고 있을 뿐이다.이러한 이합체 구조는 뉴클레오솜 스풀과 [12]유사한 방식으로 DNA가 감기는 높은 초나선("하이퍼뉴클레오솜")에 쌓일 수 있습니다.몇몇 고대 히스톤만이 [13]꼬리를 가지고 있다.
진핵세포가 DNA를 감아올리는 스풀 사이의 거리는 59 ~ 70 [14]ö 범위로 결정되었다.
히스톤은 DNA와 5가지 상호작용을 한다.
- 염기성 아미노산(특히 리신 및 아르기닌)의 측쇄와 DNA의 인산염 옥시겐 사이의 염교 및 수소 결합
- H2B, H3, H4에서 알파-헬릭스를 형성하는 나선 다이폴은 DNA에서 음전하를 띤 인산기와의 상호작용 지점에서 순 양전하를 축적시킨다.
- DNA 골격과 히스톤 단백질의 주요 사슬의 아미드 그룹 사이의 수소 결합
- DNA에서 히스톤과 디옥시리보스당 사이의 비극성 상호작용
- DNA 분자의 두 개의 작은 홈에 H3 및 H2B N 말단 꼬리의 비특이적인 작은 홈 삽입
히스톤의 매우 기본적인 성질은 DNA-히스톤 상호작용을 촉진하는 것 외에도 히스톤의 수용성에 기여합니다.
히스톤은 주로 N 말단 꼬리에서 효소에 의해 번역 후 변형을 겪지만 구상 [15][16]영역에서도 변형을 겪는다.이러한 변형에는 메틸화, 시트룰화, 아세틸화, 인산화, SMOylation, 유비퀴티네이션 및 ADP-리보실화가 포함된다.이것은 그들의 유전자 조절 기능에 영향을 미친다.
일반적으로 활성 유전자는 결합 히스톤이 적은 반면,[17] 비활성 유전자는 중간 단계 동안 히스톤과 높은 연관성을 가지고 있습니다.또한 히스톤의 구조는 어떠한 유해한 돌연변이도 심각하게 부적응할 수 있기 때문에 진화적으로 보존된 것으로 보인다.모든 히스톤은 많은 리신 및 아르기닌 잔류물과 함께 매우 양전하를 띤 N 말단을 가진다.
진화 및 종 분포
핵심 히스톤은 진핵세포의 핵과 대부분의 고세포 세포에서 발견되지만 [13]박테리아에서는 발견되지 않는다.디노플라겔라테스라고 알려진 단세포 조류는 이전에는 히스톤이 [18]완전히 없는 유일한 진핵생물이라고 생각되었지만, 이후의 연구는 그들의 DNA가 여전히 히스톤 [19]유전자를 암호화하고 있다는 것을 보여주었다.코어 히스톤과는 달리, 리신이 풍부한 링커 히스톤(H1) 단백질의 상동체는 핵단백질 HC1/[20]HC2로 알려진 박테리아에서 발견됩니다.
코어 히스톤 단백질은 확장된 AAA+ ATPase 도메인의 나선 부분, C-도메인 및 Clp/Hsp100 단백질의 N-말단 기질 인식 도메인과 진화적으로 관련이 있다고 제안되었다.위상의 차이에도 불구하고, 이 세 개의 접힘은 상동 나선-스트랜드-나선(HSH) [11]모티브를 공유한다.또한 그들은 짧고 염기성 [21]단백질인 리보솜 단백질(RPS6/RPS15)로부터 진화했을지도 모른다고 제안되었다.
고고 히스톤은 진핵생물 [13]히스톤의 진화적 전조물질과 매우 유사할 수 있다.히스톤 단백질은 [2]: 939 핵의 생물학에서 중요한 역할을 강조하면서 진핵생물에서 가장 많이 보존된 단백질 중 하나이다.대조적으로 성숙한 정자 세포는 주로 그들의 게놈 DNA를 포장하기 위해 프로타민을 사용하는데, 이것은 그들이 훨씬 더 높은 포장 [22]비율을 달성할 수 있게 해주기 때문일 것이다.
일부 주요 클래스에는 몇 가지 변형된 형태가 있습니다.그들은 주요 히스톤의 특정 등급과 아미노산 배열 상동성과 핵심 구조 유사성을 공유하지만 또한 주요 히스톤과 구별되는 그들만의 특징을 가지고 있다.이러한 작은 히스톤들은 보통 염색질 대사의 특정한 기능을 수행한다.예를 들어 히스톤 H3와 같은 CENPA는 염색체의 동원체 영역에만 관련된다.히스톤 H2A 변종 H2A.Z는 능동적으로 전사된 유전자의 촉진제와 관련되며, 또한 침묵 헤테로크로마틴의 [23]확산 방지에도 관여한다.또한 H2A.Z는 게놈 [24]안정성을 위해 염색질에서 역할을 한다.또 다른 H2A 바리안트 H2AX는 이중 가닥 절단 주변 영역에서 S139에서 인산화되어 DNA [25]수복 중인 영역을 나타낸다.히스톤 H3.3은 활발하게 전사된 [26]유전자의 신체와 관련이 있다.
기능
DNA 가닥의 압축
히스톤은 DNA가 감기는 스풀 역할을 한다.이것은 세포핵 안에 있는 진핵생물의 큰 게놈에 맞는 압축이 가능하게 한다: 압축된 분자는 포장되지 않은 분자보다 40,000배 짧다.
염색질 조절
히스톤은 DNA 및 핵단백질과의 상호작용을 변화시키는 번역 후 변형을 겪는다.H3와 H4 히스톤은 뉴클레오솜에서 튀어나온 긴 꼬리를 가지고 있으며, 여러 곳에서 공유적으로 변형될 수 있습니다.꼬리의 변형은 메틸화, 아세틸화, 인산화, 유비퀴티화, SMOylation, 시트룰화 및 ADP-리보실화를 포함한다.히스톤 H2A와 H2B의 코어도 수정할 수 있다.수정의 조합은 소위 "히스톤 코드"[27][28]라고 불리는 코드를 구성하는 것으로 생각됩니다.히스톤 변형은 유전자 조절, DNA 복구, 염색체 응축, 정자 [29]형성과 같은 다양한 생물학적 과정에서 작용한다.
히스톤 수정의 일반적인 명칭은 다음과 같습니다.
- 히스톤 이름(예: H3)
- 단일 문자 아미노산 약어(예: Lysine의 경우 K) 및 단백질 내 아미노산 위치
- 개조의 종류(Me:메틸, P:인산, Ac:아세틸, Ub:유비퀴틴)
- 수정 횟수(나만이 잔류물당 두 개 이상의 복사본에서 발생하는 것으로 알려져 있습니다. 1, 2, 3은 모노메틸화, 디메틸화 또는 트리메틸화입니다.)
따라서 H3K4me1은 H3 단백질의 시작(즉 N-말단)부터의 네 번째 잔기(리신)의 모노메틸화를 나타낸다.
| 종류 수정. | 히스톤 | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| H3K4 | H3K9 | H3K14 | H3K27 | H3K79 | H3K36 | H4K20 | H2BK5 | H2BK20 | |
| 모노메틸화 | 액티베이션[30] | 액티베이션[31] | 액티베이션[31] | 액티베이션[31][32] | 액티베이션[31] | 액티베이션[31] | |||
| 디메틸화 | 억제.[33] | 억제[33]. | 액티베이션[32] | ||||||
| 트리메틸화 | 액티베이션[34] | 억제.[31] | 억제.[31] | 활성화,[32] 억제[31]. | 액티베이션 | 억제[33]. | |||
| 아세틸화 | 액티베이션[35] | 액티베이션[34] | 액티베이션[34] | 액티베이션[36] | 액티베이션 | ||||
수정
히스톤 변형에 대한 방대한 카탈로그가 설명되었지만, 대부분의 경우 아직 기능적인 이해가 부족하다.종합적으로, 히스톤 수정은 히스톤 코드의 기초가 될 수 있다고 생각되며, 히스톤 수정의 조합은 특정한 의미를 갖는다.그러나 대부분의 기능적 데이터는 상세 연구에 생화학적으로 적응할 수 있는 개별 두드러진 히스톤 수정에 관한 것이다.
화학
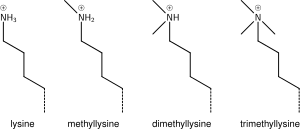
리신메틸화
리신에 1개, 2개 또는 많은 메틸기를 추가하는 것은 히스톤의 화학에 거의 영향을 미치지 않는다; 메틸화는 리신의 전하를 그대로 두고 최소한의 원자를 추가하여 입체 상호작용에 대부분 영향을 미치지 않는다.그러나 튜더, 크로모 또는 PHD 도메인을 포함하는 단백질은 정교한 감도로 리신 메틸화를 인식하고 일부 리신(예:H4K20) 모노메틸화, 디메틸화 및 트리메틸화는 서로 다른 의미를 갖는 것으로 보인다.이 때문에, 리신 메틸화는 매우 유익한 마크인 경향이 있고 알려진 히스톤 수정 기능을 지배한다.
글루타민 혈소토닐화
최근에는 세로토닌 그룹의 H3 위치 5 글루타민 첨가가 뉴런과 같은 세로토닌 작동 세포에서 일어나는 것으로 나타났다.이것은 세로토닌 작동성 세포의 분화의 일부이다.이 변환 후의 변경은 H3K4me3의 변경과 함께 이루어집니다.혈소토닐화는 TATA [37]박스에 대한 일반적인 전사 인자 TFIID의 결합을 강화한다.
아르기닌메틸화
위에서 설명한 리신 메틸화 화학은 아르기닌 메틸화에도 적용되며, 일부 단백질 도메인(예: 튜더 도메인)은 메틸 리신 대신 메틸 아르기닌에 특이적일 수 있다.아르기닌은 모노메틸화 또는 디메틸화인 것으로 알려져 있으며, 메틸화는 대칭 또는 비대칭으로 잠재적으로 다른 의미를 가질 수 있다.
아르기닌 시트룰화
펩티딜아르기닌탈미나아제(PADs)라 불리는 효소는 아르기닌의 이미닌기를 가수분해하여 케토기를 부착하여 아미노산 잔기에 대한 양의 전하를 하나 줄인다.이 과정은 변형된 히스톤을 DNA에 덜 단단하게 결합시켜 염색질을 더 [38]쉽게 접근하게 함으로써 유전자 발현 활성화에 관여해왔다.또한 PAD는 히스톤에서 아르기닌 잔기의 모노메틸화를 제거 또는 억제하여 아르기닌 메틸화가 전사 [39]활성에 미치는 긍정적 영향을 길항시킴으로써 반대 효과를 낼 수 있다.
리신 아세틸화
아세틸기 첨가는 양전하를 중화시키기 때문에 리신에 주요한 화학적 영향을 미친다.이것은 히스톤과 음전하 DNA 골격 사이의 정전적 흡인력을 감소시켜 염색질 구조를 느슨하게 한다; 높은 아세틸화 히스톤은 더 접근하기 쉬운 염색질을 형성하고 활성 전사와 연관되는 경향이 있다.리신 아세틸화는 히스톤 아세틸전달효소가 하나 이상의 리신에 작용하는 경향이 있다는 점에서 메틸화보다 의미가 덜 정확한 것으로 보인다. 이는 아마도 염색질 구조에 유의한 영향을 미치기 위해 여러 개의 리신을 바꿀 필요성을 반영한다.변경에는 H3K27ac이 포함됩니다.
세린/트레오닌/티로신인산화
음전하를 띤 인산기를 추가하면 단백질 구조의 큰 변화를 초래할 수 있으며, 이는 단백질 기능을 제어하는데 있어 인산화 역할을 잘 한다.히스톤 인산화에는 어떤 구조적 의미가 있는지는 명확하지 않지만, 히스톤 인산화에는 번역 후 변형으로서의 분명한 기능이 있으며, BRCT와 같은 결합 도메인이 특징지어졌다.
전사에 미치는 영향
가장 잘 연구된 히스톤 변형은 전사의 제어와 관련이 있다.
활발하게 전사된 유전자
액티브한 전사에는, 특히 다음의 2개의 히스톤의 변경이 관련지어져 있습니다.
- H3 리신4의 트리메틸화(H3K4me3)
- 이 트리메틸화는 활성[40][41][42] 유전자의 프로모터에서 발생하며 COMS [43][44][45]복합체에 의해 수행됩니다.효모에서 포유동물로의 이 복합체 및 히스톤 변형이 보존됨에도 불구하고, 이 변형이 어떤 역할을 하는지 완전히 명확하지는 않다.단, 활성촉진제의 우수한 표시이며 유전자 프로모터에서 히스톤 수식의 수준은 유전자의 전사 활성과 광범위하게 관련되어 있다.이 표식의 형성은 다소 복잡한 방식으로 전사에 관련된다: 유전자의 전사 초기에, RNA 중합효소 II는 RNA 중합효소 II C 말단 도메인(CTD)의 인산화 상태의 변화로 특징지어지는 시작에서 용리화'로 전환된다.CTD를 인산화시키는 동일한 효소는 Rad6 복합체를 [46][47]인산화하여 H2B K123([48]포유동물에서는 K120)에 유비퀴틴 마크를 추가합니다.H2BK123Ub는 전사된 영역 전체에서 발생하지만, 이 마크는 [49][50]COMPASS가 프로모터에서 H3K4를 트리메틸화하려면 필요합니다.
- H3 리신 36의 트리메틸화(H3K36me3)
- 이 트리메틸화는 활성 유전자의 몸에서 발생하며 메틸전달효소 세트2에 [51]의해 축적된다.이 단백질은 신장하는 RNA 중합효소 II와 관련되며, H3K36Me3는 활발하게 전사된 [52]유전자를 나타낸다.H3K36Me3는 Rpd3 히스톤 탈아세틸화효소 복합체에 의해 인식되며, 이는 주변 히스톤으로부터 아세틸 변형을 제거하여 크로마틴 압축을 증가시키고 가짜 [53][54][55]전사를 억제한다.증가된 크로마틴 압축은 전사 인자가 DNA에 접근하는 것을 방지하고 유전자 체내에서 새로운 전사 이벤트가 시작될 가능성을 감소시킨다.따라서 이 프로세스는 문자 변환이 중단되지 않도록 하는 데 도움이 됩니다.
억제 유전자
세 가지 히스톤 변형은 특히 억제된 유전자와 관련이 있다.
- H3 리신 27의 트리메틸화(H3K27me3)
- 이 히스톤 변형은 폴리콤 복합체 PRC2에 [56]의해 퇴적된다.그것은 유전자 [57]억제의 명확한 지표이며, 억제 기능을 발휘하기 위해 다른 단백질에 의해 결합될 가능성이 있다.또 다른 폴리콤 복합체 PRC1은 H3K27me3와[57] 결합할 수 있으며 염색질 압축을 [58][59]돕는 히스톤 수식 H2AK119Ub를 첨가한다.이 데이터를 바탕으로 PRC1은 PRC2의 활동을 통해 모집되는 것으로 보이지만 최근 연구에 따르면 PRC1은 [60][61]PRC2가 없을 때 동일한 사이트에 모집된다.
- H3 리신9의 Di 및 트리메틸화(H3K9me2/3)
- H3K9me2/3는 헤테로크로마틴에 대해 잘 특징지어지는 마커이므로 유전자 억제와 강하게 관련된다.헤테로크로마틴의 형성은 효모인 시조당류 폼브에서 가장 잘 연구되었으며, 여기서 센트로머 [62]반복에서 생성된 이중 가닥 RNA에 RNA 유도 전사 사일링(RITS) 복합체를 모집함으로써 시작된다.RITS는 H3K9me2/[63]3를 축적하는 Clr4 히스톤 메틸전달효소를 모집한다.이 과정은 히스톤 메틸화라고 불린다.H3K9Me2/3는 Swi6(헤테로크로마틴 단백질 1 또는 HP1, 또 다른 고전적 헤테로크로마틴 마커)[64][65]의 신병을 위한 결합부위로서 기능하며, 이는 히스톤탈아세틸라아제 및 히스톤메틸전달효소 [66]등의 히스톤 수식제를 포함한 추가적인 억제활성을 다시 획득한다.
- H4 리신 20의 트리메틸화(H4K20me3)
- 이 수정은 기능적 중요성이 불분명하지만 헤테로크로마틴과 [67][68]밀접하게 관련되어 있다.이 마크는 적어도 헤테로크로마틴 단백질 [67]1에 의해 획득되는 부분인 Suv4-20h 메틸전달효소에 의해 배치된다.
2가 프로모터
배아줄기세포(및 다른 줄기세포)의 히스톤 변형을 분석한 결과 H3K4Me3와 H3K27Me3를 모두 가진 많은 유전자 촉진제가 발견되었는데, 다시 말해 이러한 촉진제는 활성화와 억제 표시를 동시에 나타낸다.이 독특한 변형 조합은 전사가 가능한 유전자를 나타낸다; 그것들은 줄기세포에서는 필요하지 않지만, 몇몇 계통으로 분화된 후에 빠르게 필요하다.일단 세포가 분화되기 시작하면, 이 2가 프로모터는 선택된 [69]혈통에 따라 활성화되거나 억압적인 상태로 해결된다.
기타 기능
DNA손상
DNA 손상 부위를 표시하는 것은 히스톤 변형을 위한 중요한 기능이다.그것은 또한 태양의 자외선에 의해 DNA가 파괴되는 것을 막아준다.
- 세린 139에서 H2AX의 인산화(hH2AX)
- 인산화 H2AX(gamma H2AX라고도 함)는 DNA 이중 가닥 [70]파괴에 대한 표지자이며, DNA [25][71]손상에 대한 반응의 일부를 형성한다.H2AX는 DNA 이중 가닥 파괴를 검출한 후 초기에 인산화되어 손상 [70][72][73]양쪽에 많은 킬로베이스가 확장되는 도메인을 형성한다.Gamma H2AX는 단백질 MDC1의 결합 부위로 작용하며, MDC1은 주요 DNA 복구[74] 단백질(이 복잡한 주제는[75] 에서 잘 검토된다)을 모집하며, 따라서 감마 H2AX는 게놈 안정성을 보장하는 조직의 중요한 부분을 형성한다.
- H3 리신 56(H3K56Ac)의 아세틸화
- H3K56Acx는 게놈 [76][77]안정성을 위해 필요합니다.H3K56은 p300/Rtt109 [78][79][80]복합체에 의해 아세틸화되지만 DNA 손상 부위 주변에서 빠르게 탈아세틸화된다.또한 H3K56 아세틸화는 정지된 레플리케이션 포크를 안정시켜 위험한 [81][82]레플리케이션 포크의 붕괴를 방지하기 위해 필요합니다.일반적인 포유류는 미생물보다 히스톤 변형을 훨씬 더 많이 사용하지만, DNA 복제에서 H3K56Ac의 주요 역할은 곰팡이에만 존재하며, 이것이 항생제 [83]개발의 표적이 되었다.
DNA복구
- H3 리신 36의 트리메틸화(H3K36me3)
H3K36me3는 DNA 불일치 복구 [84]경로의 MSH2-MSH6(hMutSα) 복합체를 모집하는 능력을 가지고 있다.일관되게 인간 게놈의 H3K36me3 수치가 높은 부위는 불일치 복구 [85]활동으로 인해 체세포 돌연변이가 적게 축적된다.
염색체 응축
- 세린10에서의 H3 인산화(phospho-H3S10)
- 유사분열 키나제 오로라 B는 세린 10에서 히스톤 H3를 인산화하여 유사분열 염색체 [86][87]축합을 매개하는 일련의 변화를 유발한다.따라서 응축 염색체는 이 표시에 대해 매우 강하게 염색되지만, H3S10 인산화도 유사분열 외부의 특정 염색체 부위, 예를 들어 G2 동안 세포의 단실 헤테로크로마틴에서 나타난다.H3S10 인산화 또한 고도로 전사된 [88]부위의 R-루프 형성에 의해 야기된 DNA 손상과 관련이 있다.
- 세린 10/14에서의 인산화 H2B(phospho-H2BS10/14)
- 세린 10(효모) 또는 세린 14(모말)에서 H2B의 인산화도 염색질 응축과 관련이 있지만,[89][90] 아포토시스 동안 염색체 응축을 매개하는 매우 다른 목적을 위해서이다.이 마크는 단순히 아포토시스(apoptosis)에서 늦게 작용하는 방관자가 아니며, 이 잔류물의 돌연변이를 운반하는 효모는 과산화수소 유도 아포토시스 세포사에 내성이 있기 때문이다.
중독
뇌의 특정 부위에 있는 히스톤 꼬리의 후생유전학적 변형이 [91][92][93]중독에서 가장 중요하다.일단 특정한 후생유전학적 변화가 일어나면,[91] 그것들은 중독의 지속성을 설명할 수 있는 오래 지속되는 "분자 흉터"로 보입니다.
담배를 피우는 사람들은 [94]보통 니코틴에 중독되어 있다.생쥐의 니코틴 치료 7일 후, 뇌의 핵에 있는 FosB 프로모터에서 히스톤 H3와 히스톤 H4의 아세틸화가 증가하여 FosB [95]발현을 61% 증가시켰다.이는 또한 스플라이스 변형 Delta FosB의 발현을 증가시킵니다.뇌의 핵에서 Delta FosB는 [96][97]중독의 발달에서 "유지된 분자 스위치"와 "마스터 컨트롤 단백질"로 기능합니다.
미국 인구의 약 7%가 알코올에 중독되어 있다.최대 5일 동안 알코올에 노출된 랫드에서 뇌 편도체 내 프로노시셉틴 프로모터에서 히스톤3 리신9 아세틸화가 증가하였다.이 아세틸화는 프로노시셉틴의 활성 표시이다.노시셉틴/노시셉틴 오피오이드 수용체 시스템은 [98]알코올의 강화 또는 조절 효과에 관여합니다.
필로폰 중독은 미국 [99]인구의 약 0.2%에서 발생한다.만성적인 필로폰 사용은 c-fos 및 C-C 케모카인 수용체 2(ccr2) 유전자의 프로모터에 위치한 히스톤 3의 위치 4에서 리신의 메틸화를 유발하며,[101] 이들 유전자를 nuclear accumbens(NAC)[100]에서 활성화시키는 것으로 잘 알려져 있다.이 유전자의 돌연변이 불활성화는 중독을 손상시키기 때문에 ccr2 유전자는 중독에서도 [100]중요하다.
합성
염색질 구조 복제의 첫 번째 단계는 히스톤 단백질의 합성이다: H1, H2A, H2B, H3, H4.이 단백질들은 세포주기의 S단계 동안 합성된다.히스톤 합성의 증가에 기여하는 다른 메커니즘들이 있다.
효모균
효모는 각각의 히스톤 유전자의 한두 개의 복사본을 가지고 있는데, 그것들은 군집화되지 않고 오히려 염색체 전체에 흩어져 있다.히스톤 유전자 전사는 히스톤 프로모터 영역에 결합하는 전사 인자와 같은 여러 유전자 조절 단백질에 의해 제어된다.발아 효모에서 히스톤 유전자 발현 활성화 후보 유전자는 SBF이다.SBF는 억제제 Whi5에서 분리될 때 G1 후기 단계에서 활성화되는 전사 인자입니다.이것은 Whi5가 G1/[102]S Cdk인 Cdc8에 의해 인산화되었을 때 발생한다.S상 이외의 히스톤 유전자 발현 억제는 히스톤 유전자의 궤적에서 비활성 크로마틴 구조를 형성하는 Hir 단백질에 의존하여 전사 활성제를 [103][104]차단한다.
메타조아
메타존에서 히스톤 합성 속도의 증가는 mRNA의 성숙한 형태로의 처리의 증가와 mRNA 분해의 감소에 기인한다. 이는 히스톤 단백질의 번역을 위한 활성 mRNA의 증가를 초래한다.mRNA 활성화 메커니즘은 mRNA 가닥의 3' 말단 부분을 제거하는 것으로 밝혀졌으며, 스템-루프 결합 단백질(SLBP)[105]과의 연관성에 의존합니다.또한 SLBP는 3'HExo [106]뉴클레아제에 의한 분해를 차단하여 S상 동안 히스톤 mRNA를 안정화시킨다.SLBP 수치는 세포주기 단백질에 의해 제어되며, 세포가 S상으로 진입하면 SLBP가 축적되고 세포가 S상을 벗어나면 SLBP가 분해된다.SLBP는 S상 [107]말기에 사이클린 의존성 키나제(아마도 사이클린 A/cdk2)에 의한 2개의 트레오닌 잔기에서 인산화로 분해된다.메타조아들은 또한 염색체 위에 군집된 여러 개의 히스톤 유전자의 복사본을 가지고 있는데, 이는 게놈 전체의 염색체 구조 포착 분석(4C-Seq)[108]에 의해 결정되는 카잘 체라고 불리는 구조에서 국지화된다.
세포주기 제어와 합성 사이의 연계
히스톤 전사의 핵단백질 공동활성화제로도 알려진 핵단백질 아탁시아-텔랑지옥타시아(NPAT)는 인간 세포의 1번과 6번 염색체에서 히스톤 유전자 전사를 활성화하는 전사인자다.또한 NPAT는 사이클린 E-Cdk2의 기질이며, 이는 G1상과 S상 사이의 이행에 필요하다.NPAT는 초기 S 단계에서 G1/[109]S-Cdk 사이클린 E-Cdk2에 의해 인산화 된 후에만 히스톤 유전자 발현을 활성화한다.이것은 세포 주기 제어와 히스톤 합성 사이의 중요한 조절 관계를 보여준다.
역사
히스톤은 1884년 알브레히트 코셀에 [110]의해 발견되었다."히스톤"이라는 단어는 19세기 후반에서 유래되었으며, 독일어 "히스톤"에서 유래한 것으로, 아마도 고대 그리스어 "히스톤" 또는 "룸"에서 유래했을 것이다.
1960년대 초, 히스톤의 종류가 알려지기 전, 그리고 히스톤이 분류학적으로 다양한 유기체에 걸쳐 고도로 보존되는 것으로 알려지기 전에, 제임스 F. 보너와 그의 협력자들은 고등 [111]유기체의 핵에 있는 DNA와 밀접하게 연관되어 있는 것으로 알려진 이 단백질들에 대한 연구를 시작했다.보너와 그의 박사후 동료 루치 C. Huang은 분리된 염색질은 시험관에서 RNA 전사를 지원하지 않는다는 것을 보여주었지만, 만약 히스톤이 염색질에서 추출되었다면, 남은 [112]DNA에서 RNA가 전사될 수 있었다.그들의 논문은 인용의 [113]고전이 되었다.폴 티소와 제임스 보너는 1964년에 히스톤 화학과 생물학에 관한 세계 회의를 소집했는데, 그 회의에서 히스톤의 종류에 대한 합의가 없었고 다른 [114][111]유기체로부터 분리되었을 때 그들이 어떻게 비교될지 아무도 모른다는 것이 분명해졌다.그리고 나서 보너와 그의 협력자들은 정제된 개별 히스톤의 각 유형을 분리하는 방법을 개발했고, 동일한 히스톤의 아미노산 조성을 다른 유기체로부터 비교했으며,[115] UCLA의 Emil Smith와 공동으로 동일한 히스톤의 아미노산 배열을 비교했다.예를 들어, 그들은 히스톤 IV 염기서열이 완두콩과 송아지 [115]흉선 사이에서 매우 잘 보존된다는 것을 발견했다.그러나 개별 히스톤의 생화학적 특성에 대한 그들의 연구는 히스톤이 어떻게 서로 또는 그들이 단단히 [114]결합되어 있는 DNA와 상호 작용하는지 밝히지 않았다.
또한 1960년대에 빈센트 올프리와 알프레드 미르스키는 히스톤의 분석을 바탕으로 히스톤의 아세틸화와 메틸화가 전사 제어 메커니즘을 제공할 수 있다고 제안했지만, 이후 연구자들이 그러한 조절이 어떻게 유전자 특이적일 수 있는지를 보여줄 수 있는 상세한 분석의 종류를 이용할 수 없었다.1990년대 초까지 [116]히스톤은 진핵핵 DNA의 불활성 패킹 물질로 대부분 무시되었다. 이는 박테리아의 경우와 마찬가지로 거의 벌거벗은 DNA 템플릿의 단백질-DNA 및 단백질-단백질 상호작용에 의해 전사가 활성화된다고 믿었던 Mark Ptashne 등의 모델에 부분적으로 기반을 둔 견해이다.
1980년대에 야흘리 로치와 로저 콘버그는[117] 핵심 프로모터의 뉴클레오솜이 체외 전사의 시작을 방해한다는 것을 보여주었고, 마이클 그룬스타인은[118] 히스톤이 체내 전사를 억제한다는 것을 증명하여 일반적인 유전자 억제제로서의 뉴클레오솜의 개념을 이끌어냈다.억제로부터의 완화는 히스톤 수식 및 크로마틴-리모델링 복합체의 작용을 모두 포함하는 것으로 여겨진다.빈센트 올프리와 알프레드 미르스키는 일찍이 후생유전학의 분자적 발현으로 간주되는 전사 [119]활성화에서 히스톤 수정의 역할을 제안했다.Michael Grunstein과[120] David[121] Allis는 효모의 전사를 위한 히스톤 아세틸화의 중요성과 히스톤 아세틸전달효소로서의 전사활성제 Gcn5의 활성에서 이 제안에 대한 지지를 발견했다.
H5 히스톤의 발견은 1970년대로 [122]거슬러 올라가며, 현재는 히스톤 [2][4][5][6]H1의 등유형으로 여겨지고 있다.
「 」를 참조해 주세요.
레퍼런스
- ^ Youngson RM (2006). Collins Dictionary of Human Biology. Glasgow: HarperCollins. ISBN 978-0-00-722134-9.
- ^ a b c d Cox M, Nelson DR, Lehninger AL (2005). Lehninger Principles of Biochemistry. San Francisco: W.H. Freeman. ISBN 978-0-7167-4339-2.
- ^ Redon C, Pilch D, Rogakou E, Sedelnikova O, Newrock K, Bonner W (April 2002). "Histone H2A variants H2AX and H2AZ". Current Opinion in Genetics & Development. 12 (2): 162–9. doi:10.1016/S0959-437X(02)00282-4. PMID 11893489.
- ^ a b "Histone Variants Database 2.0". National Center for Biotechnology Information. Retrieved 13 January 2017.
- ^ a b Bhasin M, Reinherz EL, Reche PA (2006). "Recognition and classification of histones using support vector machine" (PDF). Journal of Computational Biology. 13 (1): 102–12. doi:10.1089/cmb.2006.13.102. PMID 16472024.
- ^ a b Hartl DL, Freifelder D, Snyder LA (1988). Basic Genetics. Boston: Jones and Bartlett Publishers. ISBN 978-0-86720-090-4.
- ^ Mariño-Ramírez L, Kann MG, Shoemaker BA, Landsman D (October 2005). "Histone structure and nucleosome stability". Expert Review of Proteomics. 2 (5): 719–29. doi:10.1586/14789450.2.5.719. PMC 1831843. PMID 16209651.
- ^ a b Luger K, Mäder AW, Richmond RK, Sargent DF, Richmond TJ (September 1997). "Crystal structure of the nucleosome core particle at 2.8 A resolution". Nature. 389 (6648): 251–60. Bibcode:1997Natur.389..251L. doi:10.1038/38444. PMID 9305837. S2CID 4328827. PDB: 1AOI
- ^ Farkas D (1996). DNA simplified: the hitchhiker's guide to DNA. Washington, D.C: AACC Press. ISBN 978-0-915274-84-0.
- ^ Jang CW, Shibata Y, Starmer J, Yee D, Magnuson T (July 2015). "Histone H3.3 maintains genome integrity during mammalian development". Genes & Development. 29 (13): 1377–92. doi:10.1101/gad.264150.115. PMC 4511213. PMID 26159997.
- ^ a b Alva V, Ammelburg M, Söding J, Lupas AN (March 2007). "On the origin of the histone fold". BMC Structural Biology. 7: 17. doi:10.1186/1472-6807-7-17. PMC 1847821. PMID 17391511.
- ^ Mattiroli F, Bhattacharyya S, Dyer PN, White AE, Sandman K, Burkhart BW, et al. (August 2017). "Structure of histone-based chromatin in Archaea". Science. 357 (6351): 609–612. Bibcode:2017Sci...357..609M. doi:10.1126/science.aaj1849. PMC 5747315. PMID 28798133.
- ^ a b c Henneman B, van Emmerik C, van Ingen H, Dame RT (September 2018). "Structure and function of archaeal histones". PLOS Genetics. 14 (9): e1007582. Bibcode:2018BpJ...114..446H. doi:10.1371/journal.pgen.1007582. PMC 6136690. PMID 30212449.
- ^ Ward R, Bowman A, El-Mkami H, Owen-Hughes T, Norman DG (February 2009). "Long distance PELDOR measurements on the histone core particle". Journal of the American Chemical Society. 131 (4): 1348–9. doi:10.1021/ja807918f. PMC 3501648. PMID 19138067.
- ^ Mersfelder EL, Parthun MR (19 May 2006). "The tale beyond the tail: histone core domain modifications and the regulation of chromatin structure". Nucleic Acids Research. 34 (9): 2653–62. doi:10.1093/nar/gkl338. PMC 1464108. PMID 16714444.
- ^ Tropberger P, Schneider R (June 2013). "Scratching the (lateral) surface of chromatin regulation by histone modifications". Nature Structural & Molecular Biology. 20 (6): 657–61. doi:10.1038/nsmb.2581. PMID 23739170. S2CID 2956823.
- ^ Allison LA (2012). Fundamental Molecular Biology (Second ed.). United States of America: John Wiley & Sons. p. 102. ISBN 9781118059814.
- ^ Rizzo PJ (August 2003). "Those amazing dinoflagellate chromosomes". Cell Research. 13 (4): 215–7. doi:10.1038/sj.cr.7290166. PMID 12974611.
- ^ Talbert PB, Henikoff S (December 2012). "Chromatin: packaging without nucleosomes". Current Biology. 22 (24): R1040-3. doi:10.1016/j.cub.2012.10.052. PMID 23257187.
- ^ Kasinsky HE, Lewis JD, Dacks JB, Ausió J (January 2001). "Origin of H1 linker histones". FASEB Journal. 15 (1): 34–42. doi:10.1096/fj.00-0237rev. PMID 11149891. S2CID 10089116.
- ^ Bozorgmehr JH (February 2020). "The origin of chromosomal histones in a 30S ribosomal protein". Gene. 726: 144155. doi:10.1016/j.gene.2019.144155. PMID 31629821.
- ^ Clarke HJ (1992). "Nuclear and chromatin composition of mammalian gametes and early embryos". Biochemistry and Cell Biology. 70 (10–11): 856–66. doi:10.1139/o92-134. PMID 1297351.
- ^ Guillemette B, Bataille AR, Gévry N, Adam M, Blanchette M, Robert F, Gaudreau L (December 2005). "Variant histone H2A.Z is globally localized to the promoters of inactive yeast genes and regulates nucleosome positioning". PLOS Biology. 3 (12): e384. doi:10.1371/journal.pbio.0030384. PMC 1275524. PMID 16248679.
- ^ Billon P, Côté J (October 2011). "Precise deposition of histone H2A.Z in chromatin for genome expression and maintenance". Biochim Biophys Acta. 1819 (3–4): 290–302. doi:10.1016/j.bbagrm.2011.10.004. PMID 22027408.
- ^ a b Paull TT, Rogakou EP, Yamazaki V, Kirchgessner CU, Gellert M, Bonner WM (2000). "A critical role for histone H2AX in recruitment of repair factors to nuclear foci after DNA damage". Current Biology. 10 (15): 886–95. doi:10.1016/S0960-9822(00)00610-2. PMID 10959836.
- ^ Ahmad K, Henikoff S (June 2002). "The histone variant H3.3 marks active chromatin by replication-independent nucleosome assembly". Molecular Cell. 9 (6): 1191–200. doi:10.1016/S1097-2765(02)00542-7. PMID 12086617.
- ^ Strahl BD, Allis CD (January 2000). "The language of covalent histone modifications". Nature. 403 (6765): 41–5. Bibcode:2000Natur.403...41S. doi:10.1038/47412. PMID 10638745. S2CID 4418993.
- ^ Jenuwein T, Allis CD (August 2001). "Translating the histone code" (PDF). Science. 293 (5532): 1074–80. CiteSeerX 10.1.1.453.900. doi:10.1126/science.1063127. PMID 11498575. S2CID 1883924.
- ^ Song N, Liu J, An S, Nishino T, Hishikawa Y, Koji T (August 2011). "Immunohistochemical Analysis of Histone H3 Modifications in Germ Cells during Mouse Spermatogenesis". Acta Histochemica et Cytochemica. 44 (4): 183–90. doi:10.1267/ahc.11027. PMC 3168764. PMID 21927517.
- ^ Benevolenskaya EV (August 2007). "Histone H3K4 demethylases are essential in development and differentiation". Biochemistry and Cell Biology. 85 (4): 435–43. doi:10.1139/o07-057. PMID 17713579.
- ^ a b c d e f g h Barski A, Cuddapah S, Cui K, Roh TY, Schones DE, Wang Z, et al. (May 2007). "High-resolution profiling of histone methylations in the human genome". Cell. 129 (4): 823–37. doi:10.1016/j.cell.2007.05.009. PMID 17512414.
- ^ a b c Steger DJ, Lefterova MI, Ying L, Stonestrom AJ, Schupp M, Zhuo D, et al. (April 2008). "DOT1L/KMT4 recruitment and H3K79 methylation are ubiquitously coupled with gene transcription in mammalian cells". Molecular and Cellular Biology. 28 (8): 2825–39. doi:10.1128/MCB.02076-07. PMC 2293113. PMID 18285465.
- ^ a b c Rosenfeld JA, Wang Z, Schones DE, Zhao K, DeSalle R, Zhang MQ (March 2009). "Determination of enriched histone modifications in non-genic portions of the human genome". BMC Genomics. 10: 143. doi:10.1186/1471-2164-10-143. PMC 2667539. PMID 19335899.
- ^ a b c Koch CM, Andrews RM, Flicek P, Dillon SC, Karaöz U, Clelland GK, et al. (June 2007). "The landscape of histone modifications across 1% of the human genome in five human cell lines". Genome Research. 17 (6): 691–707. doi:10.1101/gr.5704207. PMC 1891331. PMID 17567990.
- ^ Guillemette B, Drogaris P, Lin HH, Armstrong H, Hiragami-Hamada K, Imhof A, et al. (March 2011). "H3 lysine 4 is acetylated at active gene promoters and is regulated by H3 lysine 4 methylation". PLOS Genetics. 7 (3): e1001354. doi:10.1371/journal.pgen.1001354. PMC 3069113. PMID 21483810.
- ^ Creyghton MP, Cheng AW, Welstead GG, Kooistra T, Carey BW, Steine EJ, et al. (December 2010). "Histone H3K27ac separates active from poised enhancers and predicts developmental state". Proceedings of the National Academy of Sciences of the United States of America. 107 (50): 21931–6. doi:10.1073/pnas.1016071107. PMC 3003124. PMID 21106759.
- ^ Farrelly LA, Thompson RE, Zhao S, Lepack AE, Lyu Y, Bhanu NV, et al. (March 2019). "Histone serotonylation is a permissive modification that enhances TFIID binding to H3K4me3". Nature. 567 (7749): 535–539. Bibcode:2019Natur.567..535F. doi:10.1038/s41586-019-1024-7. PMC 6557285. PMID 30867594.
- ^ Christophorou MA, Castelo-Branco G, Halley-Stott RP, Oliveira CS, Loos R, Radzisheuskaya A, et al. (March 2014). "Citrullination regulates pluripotency and histone H1 binding to chromatin". Nature. 507 (7490): 104–8. Bibcode:2014Natur.507..104C. doi:10.1038/nature12942. PMC 4843970. PMID 24463520.
- ^ Cuthbert GL, Daujat S, Snowden AW, Erdjument-Bromage H, Hagiwara T, Yamada M, et al. (September 2004). "Histone deimination antagonizes arginine methylation". Cell. 118 (5): 545–53. doi:10.1016/j.cell.2004.08.020. PMID 15339660.
- ^ Krogan NJ, Dover J, Wood A, Schneider J, Heidt J, Boateng MA, et al. (March 2003). "The Paf1 complex is required for histone H3 methylation by COMPASS and Dot1p: linking transcriptional elongation to histone methylation". Molecular Cell. 11 (3): 721–9. doi:10.1016/S1097-2765(03)00091-1. PMID 12667454.
- ^ Ng HH, Robert F, Young RA, Struhl K (March 2003). "Targeted recruitment of Set1 histone methylase by elongating Pol II provides a localized mark and memory of recent transcriptional activity". Molecular Cell. 11 (3): 709–19. doi:10.1016/S1097-2765(03)00092-3. PMID 12667453.
- ^ Bernstein BE, Kamal M, Lindblad-Toh K, Bekiranov S, Bailey DK, Huebert DJ, et al. (January 2005). "Genomic maps and comparative analysis of histone modifications in human and mouse". Cell. 120 (2): 169–81. doi:10.1016/j.cell.2005.01.001. PMID 15680324.
- ^ Krogan NJ, Dover J, Khorrami S, Greenblatt JF, Schneider J, Johnston M, Shilatifard A (March 2002). "COMPASS, a histone H3 (Lysine 4) methyltransferase required for telomeric silencing of gene expression". The Journal of Biological Chemistry. 277 (13): 10753–5. doi:10.1074/jbc.C200023200. PMID 11805083.
- ^ Roguev A, Schaft D, Shevchenko A, Pijnappel WW, Wilm M, Aasland R, Stewart AF (December 2001). "The Saccharomyces cerevisiae Set1 complex includes an Ash2 homologue and methylates histone 3 lysine 4". The EMBO Journal. 20 (24): 7137–48. doi:10.1093/emboj/20.24.7137. PMC 125774. PMID 11742990.
- ^ Nagy PL, Griesenbeck J, Kornberg RD, Cleary ML (January 2002). "A trithorax-group complex purified from Saccharomyces cerevisiae is required for methylation of histone H3". Proceedings of the National Academy of Sciences of the United States of America. 99 (1): 90–4. Bibcode:2002PNAS...99...90N. doi:10.1073/pnas.221596698. PMC 117519. PMID 11752412.
- ^ Wood A, Schneider J, Dover J, Johnston M, Shilatifard A (November 2005). "The Bur1/Bur2 complex is required for histone H2B monoubiquitination by Rad6/Bre1 and histone methylation by COMPASS". Molecular Cell. 20 (4): 589–99. doi:10.1016/j.molcel.2005.09.010. PMID 16307922.
- ^ Sarcevic B, Mawson A, Baker RT, Sutherland RL (April 2002). "Regulation of the ubiquitin-conjugating enzyme hHR6A by CDK-mediated phosphorylation". The EMBO Journal. 21 (8): 2009–18. doi:10.1093/emboj/21.8.2009. PMC 125963. PMID 11953320.
- ^ Robzyk K, Recht J, Osley MA (January 2000). "Rad6-dependent ubiquitination of histone H2B in yeast". Science. 287 (5452): 501–4. Bibcode:2000Sci...287..501R. doi:10.1126/science.287.5452.501. PMID 10642555.
- ^ Sun ZW, Allis CD (July 2002). "Ubiquitination of histone H2B regulates H3 methylation and gene silencing in yeast". Nature. 418 (6893): 104–8. Bibcode:2002Natur.418..104S. doi:10.1038/nature00883. PMID 12077605. S2CID 4338471.
- ^ Dover J, Schneider J, Tawiah-Boateng MA, Wood A, Dean K, Johnston M, Shilatifard A (August 2002). "Methylation of histone H3 by COMPASS requires ubiquitination of histone H2B by Rad6". The Journal of Biological Chemistry. 277 (32): 28368–71. doi:10.1074/jbc.C200348200. PMID 12070136.
- ^ Strahl BD, Grant PA, Briggs SD, Sun ZW, Bone JR, Caldwell JA, et al. (March 2002). "Set2 is a nucleosomal histone H3-selective methyltransferase that mediates transcriptional repression". Molecular and Cellular Biology. 22 (5): 1298–306. doi:10.1128/MCB.22.5.1298-1306.2002. PMC 134702. PMID 11839797.
- ^ Li J, Moazed D, Gygi SP (December 2002). "Association of the histone methyltransferase Set2 with RNA polymerase II plays a role in transcription elongation". The Journal of Biological Chemistry. 277 (51): 49383–8. doi:10.1074/jbc.M209294200. PMID 12381723.
- ^ Carrozza MJ, Li B, Florens L, Suganuma T, Swanson SK, Lee KK, et al. (November 2005). "Histone H3 methylation by Set2 directs deacetylation of coding regions by Rpd3S to suppress spurious intragenic transcription". Cell. 123 (4): 581–92. doi:10.1016/j.cell.2005.10.023. PMID 16286007.
- ^ Keogh MC, Kurdistani SK, Morris SA, Ahn SH, Podolny V, Collins SR, et al. (November 2005). "Cotranscriptional set2 methylation of histone H3 lysine 36 recruits a repressive Rpd3 complex". Cell. 123 (4): 593–605. doi:10.1016/j.cell.2005.10.025. PMID 16286008.
- ^ Joshi AA, Struhl K (December 2005). "Eaf3 chromodomain interaction with methylated H3-K36 links histone deacetylation to Pol II elongation". Molecular Cell. 20 (6): 971–8. doi:10.1016/j.molcel.2005.11.021. PMID 16364921.
- ^ Kuzmichev A, Nishioka K, Erdjument-Bromage H, Tempst P, Reinberg D (November 2002). "Histone methyltransferase activity associated with a human multiprotein complex containing the Enhancer of Zeste protein". Genes & Development. 16 (22): 2893–905. doi:10.1101/gad.1035902. PMC 187479. PMID 12435631.
- ^ a b Cao R, Wang L, Wang H, Xia L, Erdjument-Bromage H, Tempst P, et al. (November 2002). "Role of histone H3 lysine 27 methylation in Polycomb-group silencing". Science. 298 (5595): 1039–43. Bibcode:2002Sci...298.1039C. doi:10.1126/science.1076997. PMID 12351676. S2CID 6265267.
- ^ de Napoles M, Mermoud JE, Wakao R, Tang YA, Endoh M, Appanah R, et al. (November 2004). "Polycomb group proteins Ring1A/B link ubiquitylation of histone H2A to heritable gene silencing and X inactivation". Developmental Cell. 7 (5): 663–76. doi:10.1016/j.devcel.2004.10.005. PMID 15525528.
- ^ Wang H, Wang L, Erdjument-Bromage H, Vidal M, Tempst P, Jones RS, Zhang Y (October 2004). "Role of histone H2A ubiquitination in Polycomb silencing". Nature. 431 (7010): 873–8. Bibcode:2004Natur.431..873W. doi:10.1038/nature02985. hdl:10261/73732. PMID 15386022. S2CID 4344378.
- ^ Tavares L, Dimitrova E, Oxley D, Webster J, Poot R, Demmers J, et al. (February 2012). "RYBP-PRC1 complexes mediate H2A ubiquitylation at polycomb target sites independently of PRC2 and H3K27me3". Cell. 148 (4): 664–78. doi:10.1016/j.cell.2011.12.029. PMC 3281992. PMID 22325148.
- ^ Gao Z, Zhang J, Bonasio R, Strino F, Sawai A, Parisi F, et al. (February 2012). "PCGF homologs, CBX proteins, and RYBP define functionally distinct PRC1 family complexes". Molecular Cell. 45 (3): 344–56. doi:10.1016/j.molcel.2012.01.002. PMC 3293217. PMID 22325352.
- ^ Verdel A, Jia S, Gerber S, Sugiyama T, Gygi S, Grewal SI, Moazed D (January 2004). "RNAi-mediated targeting of heterochromatin by the RITS complex". Science. 303 (5658): 672–6. Bibcode:2004Sci...303..672V. doi:10.1126/science.1093686. PMC 3244756. PMID 14704433.
- ^ Rea S, Eisenhaber F, O'Carroll D, Strahl BD, Sun ZW, Schmid M, et al. (August 2000). "Regulation of chromatin structure by site-specific histone H3 methyltransferases". Nature. 406 (6796): 593–9. Bibcode:2000Natur.406..593R. doi:10.1038/35020506. PMID 10949293. S2CID 205008015.
- ^ Bannister AJ, Zegerman P, Partridge JF, Miska EA, Thomas JO, Allshire RC, Kouzarides T (March 2001). "Selective recognition of methylated lysine 9 on histone H3 by the HP1 chromo domain". Nature. 410 (6824): 120–4. Bibcode:2001Natur.410..120B. doi:10.1038/35065138. PMID 11242054. S2CID 4334447.
- ^ Lachner M, O'Carroll D, Rea S, Mechtler K, Jenuwein T (March 2001). "Methylation of histone H3 lysine 9 creates a binding site for HP1 proteins". Nature. 410 (6824): 116–20. Bibcode:2001Natur.410..116L. doi:10.1038/35065132. PMID 11242053. S2CID 4331863.
- ^ Bajpai G, Jain I, Inamdar MM, Das D, Padinhateeri R (January 2017). "Binding of DNA-bending non-histone proteins destabilizes regular 30-nm chromatin structure". PLOS Computational Biology. 13 (1): e1005365. Bibcode:2017PLSCB..13E5365B. doi:10.1371/journal.pcbi.1005365. PMC 5305278. PMID 28135276.
- ^ a b Schotta G, Lachner M, Sarma K, Ebert A, Sengupta R, Reuter G, et al. (June 2004). "A silencing pathway to induce H3-K9 and H4-K20 trimethylation at constitutive heterochromatin". Genes & Development. 18 (11): 1251–62. doi:10.1101/gad.300704. PMC 420351. PMID 15145825.
- ^ Kourmouli N, Jeppesen P, Mahadevhaiah S, Burgoyne P, Wu R, Gilbert DM, et al. (May 2004). "Heterochromatin and tri-methylated lysine 20 of histone H4 in animals". Journal of Cell Science. 117 (Pt 12): 2491–501. doi:10.1242/jcs.01238. PMID 15128874.
- ^ Bernstein BE, Mikkelsen TS, Xie X, Kamal M, Huebert DJ, Cuff J, et al. (April 2006). "A bivalent chromatin structure marks key developmental genes in embryonic stem cells". Cell. 125 (2): 315–26. doi:10.1016/j.cell.2006.02.041. PMID 16630819.
- ^ a b Rogakou EP, Pilch DR, Orr AH, Ivanova VS, Bonner WM (March 1998). "DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139". The Journal of Biological Chemistry. 273 (10): 5858–68. doi:10.1074/jbc.273.10.5858. PMID 9488723.
- ^ Celeste A, Petersen S, Romanienko PJ, Fernandez-Capetillo O, Chen HT, Sedelnikova OA, et al. (May 2002). "Genomic instability in mice lacking histone H2AX". Science. 296 (5569): 922–7. Bibcode:2002Sci...296..922C. doi:10.1126/science.1069398. PMC 4721576. PMID 11934988.
- ^ Shroff R, Arbel-Eden A, Pilch D, Ira G, Bonner WM, Petrini JH, et al. (October 2004). "Distribution and dynamics of chromatin modification induced by a defined DNA double-strand break". Current Biology. 14 (19): 1703–11. doi:10.1016/j.cub.2004.09.047. PMC 4493763. PMID 15458641.
- ^ Rogakou EP, Boon C, Redon C, Bonner WM (September 1999). "Megabase chromatin domains involved in DNA double-strand breaks in vivo". The Journal of Cell Biology. 146 (5): 905–16. doi:10.1083/jcb.146.5.905. PMC 2169482. PMID 10477747.
- ^ Stewart GS, Wang B, Bignell CR, Taylor AM, Elledge SJ (February 2003). "MDC1 is a mediator of the mammalian DNA damage checkpoint". Nature. 421 (6926): 961–6. Bibcode:2003Natur.421..961S. doi:10.1038/nature01446. PMID 12607005. S2CID 4410773.
- ^ Bekker-Jensen S, Mailand N (December 2010). "Assembly and function of DNA double-strand break repair foci in mammalian cells". DNA Repair. 9 (12): 1219–28. doi:10.1016/j.dnarep.2010.09.010. PMID 21035408.
- ^ Ozdemir A, Spicuglia S, Lasonder E, Vermeulen M, Campsteijn C, Stunnenberg HG, Logie C (July 2005). "Characterization of lysine 56 of histone H3 as an acetylation site in Saccharomyces cerevisiae". The Journal of Biological Chemistry. 280 (28): 25949–52. doi:10.1074/jbc.C500181200. PMID 15888442.
- ^ Masumoto H, Hawke D, Kobayashi R, Verreault A (July 2005). "A role for cell-cycle-regulated histone H3 lysine 56 acetylation in the DNA damage response". Nature. 436 (7048): 294–8. Bibcode:2005Natur.436..294M. doi:10.1038/nature03714. PMID 16015338. S2CID 4414433.
- ^ Driscoll R, Hudson A, Jackson SP (February 2007). "Yeast Rtt109 promotes genome stability by acetylating histone H3 on lysine 56". Science. 315 (5812): 649–52. Bibcode:2007Sci...315..649D. doi:10.1126/science.1135862. PMC 3334813. PMID 17272722.
- ^ Han J, Zhou H, Horazdovsky B, Zhang K, Xu RM, Zhang Z (February 2007). "Rtt109 acetylates histone H3 lysine 56 and functions in DNA replication". Science. 315 (5812): 653–5. Bibcode:2007Sci...315..653H. doi:10.1126/science.1133234. PMID 17272723. S2CID 19056605.
- ^ Das C, Lucia MS, Hansen KC, Tyler JK (May 2009). "CBP/p300-mediated acetylation of histone H3 on lysine 56". Nature. 459 (7243): 113–7. Bibcode:2009Natur.459..113D. doi:10.1038/nature07861. PMC 2756583. PMID 19270680.
- ^ Han J, Zhou H, Li Z, Xu RM, Zhang Z (September 2007). "Acetylation of lysine 56 of histone H3 catalyzed by RTT109 and regulated by ASF1 is required for replisome integrity". The Journal of Biological Chemistry. 282 (39): 28587–96. doi:10.1074/jbc.M702496200. PMID 17690098.
- ^ Wurtele H, Kaiser GS, Bacal J, St-Hilaire E, Lee EH, Tsao S, et al. (January 2012). "Histone H3 lysine 56 acetylation and the response to DNA replication fork damage". Molecular and Cellular Biology. 32 (1): 154–72. doi:10.1128/MCB.05415-11. PMC 3255698. PMID 22025679.
- ^ Wurtele H, Tsao S, Lépine G, Mullick A, Tremblay J, Drogaris P, et al. (July 2010). "Modulation of histone H3 lysine 56 acetylation as an antifungal therapeutic strategy". Nature Medicine. 16 (7): 774–80. doi:10.1038/nm.2175. PMC 4108442. PMID 20601951.
- ^ Li F, Mao G, Tong D, Huang J, Gu L, Yang W, Li GM (April 2013). "The histone mark H3K36me3 regulates human DNA mismatch repair through its interaction with MutSα". Cell. 153 (3): 590–600. doi:10.1016/j.cell.2013.03.025. PMC 3641580. PMID 23622243.
- ^ Supek F, Lehner B (July 2017). "Clustered Mutation Signatures Reveal that Error-Prone DNA Repair Targets Mutations to Active Genes". Cell. 170 (3): 534–547.e23. doi:10.1016/j.cell.2017.07.003. hdl:10230/35343. PMID 28753428.
- ^ Wilkins BJ, Rall NA, Ostwal Y, Kruitwagen T, Hiragami-Hamada K, Winkler M, et al. (January 2014). "A cascade of histone modifications induces chromatin condensation in mitosis". Science. 343 (6166): 77–80. Bibcode:2014Sci...343...77W. doi:10.1126/science.1244508. hdl:11858/00-001M-0000-0015-11C0-5. PMID 24385627. S2CID 7698266.
- ^ Johansen KM, Johansen J (2006). "Regulation of chromatin structure by histone H3S10 phosphorylation". Chromosome Research. 14 (4): 393–404. doi:10.1007/s10577-006-1063-4. PMID 16821135. S2CID 8556959.
- ^ Castellano-Pozo M, Santos-Pereira JM, Rondón AG, Barroso S, Andújar E, Pérez-Alegre M, et al. (November 2013). "R loops are linked to histone H3 S10 phosphorylation and chromatin condensation". Molecular Cell. 52 (4): 583–90. doi:10.1016/j.molcel.2013.10.006. PMID 24211264.
- ^ Cheung WL, Ajiro K, Samejima K, Kloc M, Cheung P, Mizzen CA, et al. (May 2003). "Apoptotic phosphorylation of histone H2B is mediated by mammalian sterile twenty kinase". Cell. 113 (4): 507–17. doi:10.1016/s0092-8674(03)00355-6. PMID 12757711.
- ^ Ahn SH, Cheung WL, Hsu JY, Diaz RL, Smith MM, Allis CD (January 2005). "Sterile 20 kinase phosphorylates histone H2B at serine 10 during hydrogen peroxide-induced apoptosis in S. cerevisiae". Cell. 120 (1): 25–36. doi:10.1016/j.cell.2004.11.016. PMID 15652479.
- ^ a b Robison AJ, Nestler EJ (October 2011). "Transcriptional and epigenetic mechanisms of addiction". Nature Reviews. Neuroscience. 12 (11): 623–37. doi:10.1038/nrn3111. PMC 3272277. PMID 21989194.
- ^ Hitchcock LN, Lattal KM (2014). "Histone-mediated epigenetics in addiction". Epigenetics and Neuroplasticity—Evidence and Debate. Prog Mol Biol Transl Sci. Progress in Molecular Biology and Translational Science. Vol. 128. pp. 51–87. doi:10.1016/B978-0-12-800977-2.00003-6. ISBN 9780128009772. PMC 5914502. PMID 25410541.
- ^ McQuown SC, Wood MA (April 2010). "Epigenetic regulation in substance use disorders". Current Psychiatry Reports. 12 (2): 145–53. doi:10.1007/s11920-010-0099-5. PMC 2847696. PMID 20425300.
- ^ "Is nicotine addictive?".
- ^ Levine A, Huang Y, Drisaldi B, Griffin EA, Pollak DD, Xu S, et al. (November 2011). "Molecular mechanism for a gateway drug: epigenetic changes initiated by nicotine prime gene expression by cocaine". Science Translational Medicine. 3 (107): 107ra109. doi:10.1126/scitranslmed.3003062. PMC 4042673. PMID 22049069.
- ^ Ruffle JK (November 2014). "Molecular neurobiology of addiction: what's all the (Δ)FosB about?". The American Journal of Drug and Alcohol Abuse. 40 (6): 428–37. doi:10.3109/00952990.2014.933840. PMID 25083822. S2CID 19157711.
- ^ Nestler EJ, Barrot M, Self DW (September 2001). "DeltaFosB: a sustained molecular switch for addiction". Proceedings of the National Academy of Sciences of the United States of America. 98 (20): 11042–6. Bibcode:2001PNAS...9811042N. doi:10.1073/pnas.191352698. PMC 58680. PMID 11572966.
- ^ D'Addario C, Caputi FF, Ekström TJ, Di Benedetto M, Maccarrone M, Romualdi P, Candeletti S (February 2013). "Ethanol induces epigenetic modulation of prodynorphin and pronociceptin gene expression in the rat amygdala complex". Journal of Molecular Neuroscience. 49 (2): 312–9. doi:10.1007/s12031-012-9829-y. PMID 22684622. S2CID 14013417.
- ^ "What is the scope of methamphetamine abuse in the United States?".
- ^ a b Godino A, Jayanthi S, Cadet JL (2015). "Epigenetic landscape of amphetamine and methamphetamine addiction in rodents". Epigenetics. 10 (7): 574–80. doi:10.1080/15592294.2015.1055441. PMC 4622560. PMID 26023847.
- ^ Cruz FC, Javier Rubio F, Hope BT (December 2015). "Using c-fos to study neuronal ensembles in corticostriatal circuitry of addiction". Brain Research. 1628 (Pt A): 157–73. doi:10.1016/j.brainres.2014.11.005. PMC 4427550. PMID 25446457.
- ^ de Bruin RA, McDonald WH, Kalashnikova TI, Yates J, Wittenberg C (June 2004). "Cln3 activates G1-specific transcription via phosphorylation of the SBF bound repressor Whi5". Cell. 117 (7): 887–98. doi:10.1016/j.cell.2004.05.025. PMID 15210110.
- ^ Xu H, Kim UJ, Schuster T, Grunstein M (November 1992). "Identification of a new set of cell cycle-regulatory genes that regulate S-phase transcription of histone genes in Saccharomyces cerevisiae". Molecular and Cellular Biology. 12 (11): 5249–59. doi:10.1128/mcb.12.11.5249. PMC 360458. PMID 1406694.
- ^ Dimova D, Nackerdien Z, Furgeson S, Eguchi S, Osley MA (July 1999). "A role for transcriptional repressors in targeting the yeast Swi/Snf complex". Molecular Cell. 4 (1): 75–83. doi:10.1016/S1097-2765(00)80189-6. PMID 10445029.
- ^ Dominski Z, Erkmann JA, Yang X, Sànchez R, Marzluff WF (January 2002). "A novel zinc finger protein is associated with U7 snRNP and interacts with the stem-loop binding protein in the histone pre-mRNP to stimulate 3'-end processing". Genes & Development. 16 (1): 58–71. doi:10.1101/gad.932302. PMC 155312. PMID 11782445.
- ^ Dominski Z, Yang XC, Kaygun H, Dadlez M, Marzluff WF (August 2003). "A 3' exonuclease that specifically interacts with the 3' end of histone mRNA". Molecular Cell. 12 (2): 295–305. doi:10.1016/S1097-2765(03)00278-8. PMID 14536070.
- ^ Zheng L, Dominski Z, Yang XC, Elms P, Raska CS, Borchers CH, Marzluff WF (March 2003). "Phosphorylation of stem-loop binding protein (SLBP) on two threonines triggers degradation of SLBP, the sole cell cycle-regulated factor required for regulation of histone mRNA processing, at the end of S phase". Molecular and Cellular Biology. 23 (5): 1590–601. doi:10.1128/MCB.23.5.1590-1601.2003. PMC 151715. PMID 12588979.
- ^ Wang Q, Sawyer IA, Sung MH, Sturgill D, Shevtsov SP, Pegoraro G, et al. (March 2016). "Cajal bodies are linked to genome conformation". Nature Communications. 7: 10966. Bibcode:2016NatCo...710966W. doi:10.1038/ncomms10966. PMC 4802181. PMID 26997247.
- ^ Zhao J, Kennedy BK, Lawrence BD, Barbie DA, Matera AG, Fletcher JA, Harlow E (September 2000). "NPAT links cyclin E-Cdk2 to the regulation of replication-dependent histone gene transcription". Genes & Development. 14 (18): 2283–97. doi:10.1101/GAD.827700. PMC 316937. PMID 10995386.
- ^ Luck JM (1965). "Histone Chemistry: the Pioneers". In Bonner J, Ts'o P (eds.). The Nucleohistones. San Francisco, London, and Amsterdam: Holden-Day, Inc.
- ^ a b Bonner J (1994). "Chapters from my life". Annual Review of Plant Physiology and Plant Molecular Biology. 45: 1–23. doi:10.1146/annurev.pp.45.060194.000245.
- ^ Huang RC, Bonner J (July 1962). "Histone, a suppressor of chromosomal RNA synthesis". Proceedings of the National Academy of Sciences of the United States of America. 48 (7): 1216–22. Bibcode:1962PNAS...48.1216H. doi:10.1073/pnas.48.7.1216. PMC 220935. PMID 14036409.
- ^ "Huang R C & Bonner J. Histone, a suppressor of chromosomal RNA synthesis. Proc. Nat. Acad. Sci. US 48:1216-22, 1962" (PDF). Citation Classics (12): 79. 20 March 1978.
- ^ a b 제임스 보너와 폴 티소(1965년)뉴클레오히스톤스.홀든데이 주식회사, 샌프란시스코, 런던, 암스테르담
- ^ a b DeLange RJ, Fambrough DM, Smith EL, Bonner J (October 1969). "Calf and pea histone IV. 3. Complete amino acid sequence of pea seedling histone IV; comparison with the homologous calf thymus histone". The Journal of Biological Chemistry. 244 (20): 5669–79. doi:10.1016/S0021-9258(18)63612-9. PMID 5388597.
- ^ Allfrey VG, Faulkner R, Mirsky AE (May 1964). "Acetylation and methylation of histones and their possible role in the regulation of RNA synthesis". Proceedings of the National Academy of Sciences of the United States of America. 51 (5): 786–94. Bibcode:1964PNAS...51..786A. doi:10.1073/pnas.51.5.786. PMC 300163. PMID 14172992.
- ^ Lorch Y, LaPointe JW, Kornberg RD (April 1987). "Nucleosomes inhibit the initiation of transcription but allow chain elongation with the displacement of histones". Cell. 49 (2): 203–10. doi:10.1016/0092-8674(87)90561-7. PMID 3568125. S2CID 21270171.
- ^ Kayne PS, Kim UJ, Han M, Mullen JR, Yoshizaki F, Grunstein M (October 1988). "Extremely conserved histone H4 N terminus is dispensable for growth but essential for repressing the silent mating loci in yeast". Cell. 55 (1): 27–39. doi:10.1016/0092-8674(88)90006-2. PMID 3048701. S2CID 7994350.
- ^ Pogo BG, Allfrey VG, Mirsky AE (April 1966). "RNA synthesis and histone acetylation during the course of gene activation in lymphocytes". Proceedings of the National Academy of Sciences of the United States of America. 55 (4): 805–12. Bibcode:1966PNAS...55..805P. doi:10.1073/pnas.55.4.805. PMC 224233. PMID 5219687.
- ^ Durrin LK, Mann RK, Kayne PS, Grunstein M (June 1991). "Yeast histone H4 N-terminal sequence is required for promoter activation in vivo". Cell. 65 (6): 1023–31. doi:10.1016/0092-8674(91)90554-c. PMID 2044150. S2CID 28169631.
- ^ Brownell JE, Zhou J, Ranalli T, Kobayashi R, Edmondson DG, Roth SY, Allis CD (March 1996). "Tetrahymena histone acetyltransferase A: a homolog to yeast Gcn5p linking histone acetylation to gene activation". Cell. 84 (6): 843–51. doi:10.1016/s0092-8674(00)81063-6. PMID 8601308.
- ^ Aviles FJ, Chapman GE, Kneale GG, Crane-Robinson C, Bradbury EM (August 1978). "The conformation of histone H5. Isolation and characterisation of the globular segment". European Journal of Biochemistry. 88 (2): 363–71. doi:10.1111/j.1432-1033.1978.tb12457.x. PMID 689022.