BRCA1
BRCA1유방암 제1형 감수성 단백질은 BRCA1(/ (brékˈʌw)n/) [5]유전자에 의해 인간에서 코드되는 단백질이다.척추동물의 유전체는 다른 척추동물 종에서 흔히 볼 수 있는 반면 무척추동물의 게놈은 더 먼 친척의 [6]유전자를 부호화할 수 있다.BRCA1은 인간 종양 억제 유전자[7][8](관리 유전자라고도 함)로 DNA를 [9]수리하는 역할을 한다.
BRCA1과 BRCA2는 관련이 없는 [10]단백질이지만, 둘 다 보통 유방과 다른 조직의 세포에서 발현되며, 손상된 DNA를 복구하거나 DNA가 복구될 수 없다면 세포를 파괴하는 것을 돕는다.이들은 염색체 손상 복구에 관여하며 DNA 이중 가닥 절단의 [11][12]오류 없는 복구에 중요한 역할을 한다.BRCA1이나 BRCA2 자체가 BRCA 돌연변이에 의해 손상되면 손상된 DNA가 제대로 복구되지 않아 유방암에 [13][14]걸릴 위험이 높아진다.BRCA1과 BRCA2는 "유방암 감수성 유전자"와 "유방암 감수성 단백질"로 설명되었다.지배적인 대립 유전자는 정상적이고 종양 억제 기능을 가지고 있는 반면, 이러한 유전자의 높은 투과성 돌연변이는 유방암의 [15]증가 위험과 관련이 있는 종양 억제 기능의 상실을 야기합니다.
BRCA1은 다른 종양 억제제, DNA 손상 센서 및 신호 변환기와 결합하여 BRCA1-관련 게놈 감시 복합체(BASC)[16]로 알려진 대규모 다중 서브유닛 단백질 복합체를 형성합니다.BRCA1 단백질은 RNA 중합효소 II 및 C 말단 도메인을 통해 히스톤 탈아세틸화효소 복합체와도 상호작용한다.따라서, 이 단백질은 전사에 역할을 하며, 이중 가닥 DNA의[14] DNA 복구는 유비쿼티네이션, 전사 조절 및 다른 [17]기능을 파괴한다.
BRCA1과 BRCA2에 돌연변이가 있는 환자의 암 발생 가능성을 검사하는 방법은 Miriad [18][19]Genetics가 소유하거나 통제하는 특허에 의해 다루어졌다.1994년에 진단 시험 독점적으로 Myriad하는을 제공하 Myriad의 사업 모델을 시작 1200직원과달러 500M 연간 매출액은 2012년에, 상장 회사;[20]그것은 또한 높은 물가로 인해 논란과 이것이 이번에는으로 이어졌다 다른 진단 실험실,에서 두번째 의견을을 가져오는 것을 이끌었다.l그리고 마크 분자병리학회 대 미리어드 유전학 소송.[21]
검출
유방암 감수성과 관련된 DNA 복구 효소를 코드하는 유전자의 존재에 대한 첫 번째 증거는 1990년 [22]UC 버클리 소재 메리-클레어 킹 연구소에 의해 제공되었다.4년 후,[23] 그것을 찾기 위한 국제적인 경쟁 끝에, 이 유전자는 유타 대학, 국립환경보건과학연구소, 그리고 미리어드 [18][24]유전학 연구자들에 의해 1994년에 복제되었다.
유전자 위치
인간 BRCA1 유전자는 염기쌍 41,196,312에서 염기쌍 41,277,500(구축 GRCh37/hg19)(맵)[25]까지 영역 2 밴드 1에서 염색체 17의 긴 팔(q)에 위치한다.BRCA1 정형어는 완전한 게놈 데이터를 이용할 [6]수 있는 대부분의 척추동물에서 확인되었다.
단백질 구조
BRCA1 단백질에는 다음 [26]도메인이 포함됩니다.
이 단백질은 핵 국재 신호와 핵 수출 신호 [27]모티브도 포함하고 있다.
인간 BRCA1 단백질은 Znf C3HC4-RING 도메인, BRCA1 세린 도메인 및 2개의 BRCT 도메인 등 4개의 주요 단백질 도메인으로 구성된다.이러한 도메인은 BRCA1 단백질의 약 27%를 인코딩합니다.BRCA1에는 [28]6개의 알려진 이소형식이 있으며, 이소형식 1과 2는 [citation needed]각각 1863개의 아미노산으로 구성됩니다.
BRCA1은 BRCA2와 관련이 없다. 즉, 호몰로그나 [10]패럴로그가 아니다.

아연 약지 도메인
진핵생물 펩타이드에서 발견되는 Zn 핑거인 RING 모티브는 40~60개의 아미노산 길이로 보존된 8개의 금속 결합 잔류물, 두 개의 아연 [30]원자를 조정하는 시스테인 또는 히스티딘 잔류물 2개 쿼텟으로 구성된다.이 모티브는 짧은 반평행 베타 시트, 두 개의 아연 결합 루프 및 작은 영역의 중앙 알파 나선을 포함한다.이 RING 도메인은 또한 RING 모티브를 포함하는 BARD1을 포함한 관련 단백질과 상호작용하여 헤테로디머를 형성한다.BRCA1 RING 모티브는 BRCA1 단백질의 잔류물 8~22와 81~96에 의해 형성된 알파 헬리크로 측면 형성된다.또한 잔류물 36–48과 101–116에서 형성된 두 개의 알파-헬리크로 측면의 RING 핑거로 구성된 BARD1의 상동 영역과 상호작용한다.이들 4개의 나선은 결합되어 헤테로다이머화 계면을 형성하고 BRCA1-BAR1 헤테로다이머 복합체를 안정화한다.측면 영역의 인접 잔류물과 소수성 상호작용 간의 상호작용에 의해 추가적인 안정화가 달성된다.BARD1/BRCA1 상호작용은 BRCA1의 종양유전성 아미노산 치환에 의해 파괴되며, 이는 이들 단백질 사이의 안정적인 복합체 형성이 BRCA1 종양 억제의 [30]필수적인 측면일 수 있음을 암시한다.
고리 도메인은 단백질 유비퀴티네이션을 촉매하는 유비퀴틴 E3 연결효소의 중요한 요소입니다.유비퀴틴은 모든 조직에서 발견되는 작은 조절 단백질로 단백질을 세포 내의 구획으로 유도한다.특히 BRCA1 폴리펩타이드, Lys-48 연결 폴리유비퀴틴 사슬은 휴지 세포핵 전체에 분산되어 있지만 DNA 복제 개시 시에는 BRCA2 및 BARD1을 포함한 억제된 그룹으로 모인다.BARD1은 유비퀴타이션을 [31]위한 단백질 타깃의 인식 및 결합에 관여하는 것으로 생각된다.단백질에 달라붙어 파괴의 꼬리표를 붙인다.유비퀴티네이션은 BRCA1 융합 단백질을 통해 발생하며 아연 킬레이트화에 의해 [30]폐지된다.융합 단백질의 효소 활성은 고리 [citation needed]영역의 적절한 접힘에 따라 달라집니다.
Serine 클러스터 도메인
BRCA1 세린 클러스터 도메인(SCD)은 아미노산 1280–1524에 걸쳐 있다.도메인의 일부는 exon 11 ~13에 있습니다.높은 돌연변이율은 엑손 11-13에서 발생한다.보고된 BRCA1의 인산화 부위는 시험관내 및 생체내 ATM/ATR 키나제에 의해 인산화된다.ATM/ATR은 DNA 손상에 의해 활성화된 키나아제입니다.세린 잔기의 돌연변이는 DNA 손상 부위 및 DNA 손상 반응 [29]기능에 대한 BRCA1의 국재화에 영향을 미칠 수 있다.
BRCT 도메인
BRCA1 단백질의 이중 반복 BRCT 도메인은 길이가 약 70Ω, [32]폭이 30~35Ω인 가늘고 긴 구조이다.BRCT의 85~95개의 아미노산 도메인은 단일 모듈 또는 [33]두 개의 도메인을 포함하는 다중 탠덤 반복으로 확인할 수 있다.이 두 가지 가능성 모두 다양한 형태의 [32]단일 단백질에서 발생할 수 있습니다.BRCA1 단백질의 C 말단 BRCT 영역은 DNA의 복구, 전사 조절 및 종양 억제 기능을 [34]위해 필수적이다.BRCA1에서 이중 탠덤 반복 BRCT 도메인은 3차원 구조에서 머리부터 꼬리까지 배치되어 계면에 소수성 용매 접근 가능한 표면적 1600Ω을 묻는다.이 모든 것이 인터페이스를 구성하는 꽉 막힌 노브인홀 구조의 원인이 됩니다.이러한 상동 도메인은 DNA 손상에 대한 세포 반응을 제어하기 위해 상호작용합니다.이 두 단백질의 계면에서의 미스센스 돌연변이는 세포주기를 교란시켜 암에 [citation needed]걸릴 위험이 더 크다.
기능 및 메커니즘
BRCA1은 DNA의 이중 가닥 파손을 복구하는 복합체의 일부이다.DNA 이중나선의 가닥은 손상되면서 지속적으로 끊어지고 있다.한 가닥만 끊어질 때도 있고 두 가닥이 동시에 끊어질 때도 있습니다.DNA 가교제는 염색체/DNA 손상의 중요한 원천이다.이중사슬 절단은 가교 제거 후 중간체로서 발생하며, 실제로 BRCA1의 바이알렐 돌연변이는 DNA 가교제에 대한 과민증과 관련된 유전병인 [35]판코니 빈혈의 원인이 되는 것으로 확인되었다.BRCA1은 두 가닥이 끊어졌을 때 DNA를 복구하는 단백질 복합체의 일부이다.이 경우 복구 메커니즘이 올바른 DNA 염기서열을 대체하는 방법을 아는 것은 어렵고 복구를 시도하는 방법은 여러 가지가 있습니다.BRCA1이 참여하는 이중 가닥 복구 메커니즘은 호몰로지 방향 복구이며, 여기서 복구 단백질은 온전한 [36]자매 염색체로부터 동일한 염기서열을 복제한다.
많은 유형의 정상 세포의 핵에서, BRCA1 단백질은 DNA 이중 가닥 [37]절단 수복 중에 RAD51과 상호작용합니다.이러한 파손은 자연 방사선이나 다른 피폭에 의해 발생할 수 있지만, 염색체가 유전 물질을 교환할 때(예: 감수 분열 중 "교차") 발생한다.BRCA1과 유사한 기능을 가진 BRCA2 단백질도 RAD51 단백질과 상호작용한다.DNA 손상 복구에 영향을 줌으로써, 이 세 가지 단백질은 인간 [citation needed]게놈의 안정성을 유지하는 역할을 한다.
BRCA1은 불일치 복구라고 불리는 또 다른 유형의 DNA 복구에도 관여합니다.BRCA1은 DNA 미스매치 복구 단백질 MSH2와 [38]상호작용한다.MSH2, MSH6, PARP 및 단사슬 복구에 관여하는 다른 단백질은 BRCA1 결손 유선종양에서 [39]상승하는 것으로 보고되고 있다.
발로신 함유 단백질이라고 불리는 단백질은 손상된 DNA 부위에 BRCA1을 모집하는 역할을 한다.전리방사선 후 VCP를 DNA 병변으로 끌어들여 유비퀴틴 리가아제 RNF8과 협력하여 효율적인 DSB [40]수복을 위한 시그널링 복합체 조립을 조정한다.BRCA1은 [41]VCP와 상호작용합니다.BRCA1은 또한 c-Myc 및 게놈 [42]안정성을 유지하기 위해 중요한 다른 단백질과도 상호작용합니다.
BRCA1은 분지 DNA 구조에 대해 높은 친화력을 가지고 DNA에 직접 결합한다.DNA에 결합하는 이러한 능력은 Mre11만의 [43]핵산가수분해효소 활성뿐만 아니라 MRN 복합체의 핵산가수분해효소 활성도 억제하는 능력에 기여한다.이는 BRCA1이 비호몰로지 엔드 결합(NHEJ)[44]에 의한 저충실도 DNA 수복을 촉진하는 역할을 설명할 수 있다.BRCA1은 또한 DNA 이중사슬 단절 복구 포치에서 γ-H2AX(세린-139에서 인산화되는 히스톤 H2AX)와 함께 열원소화되며, [17][45]이는 복구 인자를 모집하는 데 역할을 할 수 있음을 나타낸다.
포름알데히드와 아세트알데히드는 [46]흔히 경로를 포함하는 BRCA1에 의해 매개되는 DNA 교차 연결의 일반적인 환경 소스입니다.
이 DNA 복구 기능은 필수적이다. 두 BRCA1 대립 유전자에 기능 돌연변이가 있는 생쥐는 생존할 수 없으며, 2015년 현재 두 대립 유전자에 기능 돌연변이가 있는 성인은 두 명뿐이며, 선천적 또는 발달적 문제가 있으며, 둘 다 암에 걸렸다.하나는 BRCA1 돌연변이 중 하나가 [47]하이포메이션이었기 때문에 성인기까지 생존한 것으로 추정되었다.
문자 변환
BRCA1은 Hela 추출물에서 인간 RNA 중합효소 II 홀로엔자임과 함께 정제되는 것으로 나타나 홀로엔자임의 [48]성분임을 시사했다.그러나 이후 연구는 이 가정을 반박하고 대신 HeLa 세포에서 BRCA1을 포함한 주요 복합체는 SWI/[49]SNF를 포함하는 2메가달톤 복합체이며, SWI/SNF는 크로마틴 리모델링 복합체라는 것을 보여주었다.염색질에 대한 BRCA1의 인위적 테더링은 [45]이 역할에 SWI/SNF 상호작용 도메인이 필요하지 않았지만 헤테로크로마틴을 축합하는 것으로 나타났다.BRCA1은 NELF [45]복합체의 NELF-B(COBRA1) 서브유닛과 상호작용한다.
돌연변이 및 암 위험
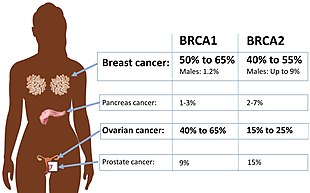
BRCA1 유전자의 특정 변이는 유전성 유방암-이온성 암 증후군의 일부로서 유방암 위험을 증가시킨다.연구자들은 BRCA1 유전자에서 수백 개의 돌연변이를 확인했는데, 그 중 다수는 암의 위험 증가와 관련이 있다.비정상적인 BRCA1 또는 BRCA2 유전자를 가진 여성은 90세까지 유방암에 걸릴 위험이 최대 80%에 이른다. 난소암 발병 위험은 BRCA1 돌연변이를 가진 여성의 경우 약 55%, BRCA2 [51]돌연변이를 가진 여성의 경우 약 25%이다.
이러한 돌연변이는 하나 또는 소수의 DNA 염기쌍(DNA의 구성 요소)의 변화일 수 있으며 PCR 및 DNA 염기서열 [citation needed]분석으로 식별될 수 있다.
경우에 따라서는 DNA의 큰 부분이 재배치됩니다.큰 재배치라고도 불리는 이러한 큰 부분들은 유전자의 하나 또는 여러 엑손의 결실 또는 복제일 수 있다.돌연변이 검출(시퀀싱)을 위한 고전적인 방법으로는 이러한 유형의 [52]돌연변이를 밝힐 수 없다.기존의 정량적 PCR,[53] Multiplex Lection-Dependent Probe Amplification(MLPA;[54] 다중화 프로브 증폭) 및 Quantitative Multiplex PCR of Short Fluorects(QMPSF;[55] 짧은 형광 조각의 정량적 다중화 PCR) 등 다른 방법이 제안되었습니다.새로운 방법: 다모양 전기영동에 의한 헤테로듀플렉스 분석(HDA) 또는 비교 유전체 혼성(array-CGH)[56]에 기초한 전용 올리고뉴클레오티드 어레이도 최근 제안되었다.
일부 결과에 따르면 일부 암에서 보고된 BRCA1 프로모터의 과메틸화는 BRCA1 [57]발현 불활성화 메커니즘으로 간주될 수 있다.
변이된 BRCA1 유전자는 보통 제대로 기능하지 않는 단백질을 만든다.연구자들은 결함이 있는 BRCA1 단백질이 다른 유전자의 돌연변이를 일으키는 DNA 손상을 고치는 데 도움을 줄 수 없다고 믿고 있다.이러한 돌연변이는 축적될 수 있고 세포들이 통제할 수 없이 자라고 분열하여 종양을 형성하게 할 수 있다.따라서, BRCA1 불활성화 돌연변이는 [citation needed]암에 대한 소인으로 이어진다.
BRCA1 mRNA 3' UTR은 miRNA, Mir-17 마이크로RNA와 결합할 수 있다.Mir-30 마이크로RNA와 함께 이 miRNA의 변이가 유방암에 [58]걸리기 쉽다고 제안되었다.
유방암 외에도 BRCA1 유전자의 돌연변이는 난소암과 전립선암의 위험도 높인다.또한 팔로피아 튜브 내의 전암성 병변(이형성증)은 BRCA1 유전자 돌연변이와 관련이 있다.BRCA1과 BRCA2를 포함하는 모델 경로에서 병원성 돌연변이는 백혈병과 림프종의 [14]하위 집합에 대한 위험을 크게 증가시킨다.
결함이 있는 BRCA1 또는 BRCA2 유전자를 물려받은 여성들은 유방암과 난소암에 걸릴 위험이 매우 높다.유방암이나 난소암에 걸릴 위험이 매우 높고 암에만 특이해 많은 돌연변이 보균자들이 예방 수술을 받기를 선택한다.그렇게 눈에 띄는 조직의 특이성을 설명하는 데는 많은 추측이 있었다.BRCA 1/2 유전성 암이 발생하는 주요 결정요인은 만성 염증을 일으키는 물질인 암 병원체의 조직 특이성과 관련이 있다.대상 조직은 병원체에 대한 수용체를 가질 수 있으며 염증 과정 또는 발암 물질에 선택적으로 노출될 수 있습니다.종양억제유전자의 선천적인 유전체결핍은 정상반응을 저해하고 장기표적에서 질병에 대한 감수성을 악화시킨다.이 이론은 또한 BRCA1 또는 BRCA2를 초과하는 여러 종양 억제제에 대한 데이터에도 적합하다.이 모델의 주요 장점은 예방 [59]수술 외에 몇 가지 옵션이 있을 수 있다는 것입니다.
유방암과 난소암에서 BRCA1의 낮은 발현
BRCA1 발현은 대부분의 고악도 유관성 [60]유방암에서 감소되거나 검출되지 않는다.BRCA1 활성의 상실은 생식계 돌연변이 또는 유전자 발현의 하향 조절에 의해 특정 표적 조직에서 종양 형성으로 이어진다는 것은 오랫동안 알려져 왔다.특히 BRCA1 발현 감소는 산발성 및 유전성 유방종양의 [61]진행에 모두 기여한다.BRCA1의 감소된 발현은 상동 재조합의 [62]잠재적 무오류 경로에 의한 DNA 손상, 특히 이중 가닥 파괴의 복구에 중요한 역할을 하기 때문에 종양 유발이다.BRCA1 단백질이 부족한 세포는 보다 오류가 발생하기 쉬운 대체 메커니즘에 의해 DNA 손상을 복구하는 경향이 있기 때문에, 이 단백질의 감소 또는 침묵은 유방암으로 [62]진행될 수 있는 돌연변이와 총염색체 재배열을 생성한다.
마찬가지로, EOC가 난소암의 가장 흔한 유형으로 난소암의 약 90%를 차지하는 산발성 상피 난소암([63]EOCs)의 과반수(55%)에서 BRC1 발현이 낮다.EOCs의 약 3분의 2를 구성하는 하위 범주인 장액성 난소암종에서 낮은 BRCA1 발현이 50%[64] 이상에서 발생한다.Bowtell은[65] BRCA1 결핍으로 인한 상동 재조합 수리가 부족하다는 것을 나타내는 문헌을 검토했다.특히 이 결핍은 고농도의 장액성 난소암의 진화를 조각하고 치료에 대한 반응을 지시하는 일련의 분자적 사건을 일으킨다.특히 BRCA1 결핍은 BRCA1 돌연변이 또는 BRCA1 발현 결핍을 유발하는 다른 사건으로 인해 종양 발생의 원인이 될 수 있다는 점에 주목했다.
유방암과 난소암의 BRCA1 돌연변이
유방암에 걸린 모든 여성의 약 3%-8%만이 BRCA1 또는 BRCA2에 [66]돌연변이를 가지고 있다.마찬가지로, BRCA1 돌연변이는 난소암의 약 18%에서만 볼 수 있다(배아세포 돌연변이 13%, 체세포 [67]돌연변이 5%).
따라서 이러한 암의 대부분에서 BRCA1 발현이 낮은 반면, BRCA1 돌연변이는 발현 감소의 주요 원인이 아니다.유방암 종양에서 자주 발견되는 특정 잠복 바이러스는 BRCA1 유전자의 발현을 감소시켜 유방암의 [68]발생을 유발할 수 있다.
유방암과 난소암의 BRCA1 프로모터 과메틸화
BRCA1 프로모터 과메틸화는 선택되지 않은 1차 유방암의 [69]13%에서만 나타났다.마찬가지로, BRC1 프로모터 과메틸화는 EOC [63]사례의 5% ~ 15%에서만 존재했다.
따라서 이러한 암에서 BRCA1 발현이 낮은 반면, BRCA1 프로모터 메틸화는 발현 감소의 사소한 원인일 뿐이다.
유방암에서 BRCA1의 MicroRNA 억제
과도하게 발현되면 특정 DNA 복구 단백질의 발현을 직접적으로 감소시키는 많은 특정 마이크로RNA가 있다(MicroRNA 섹션 DNA 복구 및 암 참조). 유방암의 경우, 마이크로RNA-182(miR-182)가 특히 BRCA1을 [70]대상으로 한다.유방암은 수용체 상태 또는 조직학에 따라 분류할 수 있으며, 삼중 음성 유방암(유방암의 15%–25%), HER2+(유방암의 15%–30%), ER+/PR+(유방암의 약 70%), 침습성 소엽암(침습성 유방암의 약 5%–10%)이 있다.4종류의 유방암 모두 정상 유방조직에 비해 [71]miR-182가 평균 약 100배 증가한 것으로 나타났다.유방암 세포주에서는 BRCA1 단백질 [70]수치와 miR-182 발현과의 역상관관계가 있다.따라서 고악도관성 유방암에서 BRCA1의 감소 또는 부재는 과다 발현된 miR-182에 기인할 수 있다.
miR-182 외에 miR-146a 및 miR-146b-5p 한 쌍의 거의 동일한 마이크로RNA도 BRCA1 발현을 억제한다.이 두 개의 마이크로RNA는 삼중 음성 종양에서 과잉 발현되어 BRCA1 [72]불활성화를 초래한다.따라서 miR-146a 및/또는 miR-146b-5p도 이러한 3중 음성 유방암에서 BRCA1의 발현 감소에 기여할 수 있다.
난소암에서 BRCA1의 마이크로RNA 억제
장액세관내암(고급장액난소암(HG-SOC)의 전구병변)과 HG-SOC 자체에서 약 70%의 사례에서 [73]miR-182가 과압된다.miR-182가 과다 발현된 세포에서 BRCA1은 이온화 방사선에 피폭된 후에도 낮은 수준을 유지했다(일반적으로 BRCA1 [73]발현을 증가시킨다).따라서 HG-SOC에서의 BRCA1의 감소 또는 부재는 과잉 발현 miR-182에 의한 것일 수 있습니다.
난소암세포에서 BRCA1의 발현을 감소시키는 것으로 알려진 또 다른 마이크로RNA는 miR-9이다.[63]2기 IIIC 또는 4기 장액 난소암(HG-SOG) 환자의 종양 58개 중 miR-9 발현과 BRCA1 [63]발현 사이에 역상관관계가 발견되었으므로 miR-9 증가가 이들 난소암에서 BRCA1 발현 감소에도 기여할 수 있다.
BRCA1 발현 결핍은 종양유전성일 가능성이 있습니다
DNA 손상은 [74]암의 주요 근본 원인인 것으로 보이며, DNA 수복의 결핍은 많은 형태의 [75]암의 근저에 있는 것으로 보인다.DNA 복구가 부족하면 DNA 손상이 축적되는 경향이 있다.이러한 과도한 DNA 손상은 오류가 발생하기 쉬운 전이 합성으로 인해 DNA 복제 중 돌연변이 오류를 증가시킬 수 있습니다.과도한 DNA 손상은 또한 DNA [76][77]복구 중 오류로 인한 후생유전학적 변화를 증가시킬 수 있다.그러한 돌연변이와 후생유전적 변화는 암을 유발할 수 있다.유방암과 난소암에서 빈번한 마이크로RNA 유도 BRCA1 결핍은 이러한 암의 진행에 기여할 수 있다.
생식계 돌연변이와 창시자 효과
현재까지 확인된 모든 배아줄 BRCA1 돌연변이는 유전되었고, 이는 특정 돌연변이가 잘 정의된 모집단 그룹에 공통적이고 이론적으로 공통의 조상으로 거슬러 올라갈 수 있는 큰 "창시자" 효과의 가능성을 시사한다.BRCA1에 대한 돌연변이 검사의 복잡성을 고려할 때, 이러한 일반적인 돌연변이는 특정 모집단에서 돌연변이 검사에 필요한 방법을 단순화할 수 있다.높은 빈도로 발생하는 돌연변이를 분석하면 임상 발현에 대한 [78]연구도 가능하다.창시자 효과의 징후는 아슈케나지 유대인들 사이에서 볼 수 있다.BRCA1의 세 가지 돌연변이는 유전 BRCA1 관련 유방 및/또는 난소암을 가진 아슈케나지 유대인 환자의 대다수를 설명하는 것으로 보고되었다: BRCA1 [79][80]유전자의 185delAG, 188del11 및 5382insC.실제로 유대인 여성이 BRCA1 185delAG, BRCA1 5382insC 설립자 돌연변이를 갖고 있지 않으면 다른 BRCA1 돌연변이가 [81]발견될 가능성은 매우 낮은 것으로 나타났다.BRCA1의 설립자 돌연변이의 추가 예는 표 1(주로 에서 도출[78])에 제시되어 있다.
| 모집단 또는 부분군 | BRCA1 [82]변환 | 레퍼런스 |
|---|---|---|
| 흑인 | 943ins10, M1775R | [83] |
| 아프리카인 | E881X, 1374delC | [84][85] |
| 아슈케나지 유대인 | 185delAG, 188del11, 5382inc | [79][80] |
| 오스트리아인 | 2795delA, C61G, 5382insC, Q1806 스톱 | [86] |
| 벨기에인 | 2804delAA, IVS5+3A>g | [87][88] |
| 네덜란드 | Exon 2 삭제, Exon 13 삭제, 2804delAA | [87][89][90] |
| 핀 | 3745delT, IVS11-2A>g | [91][92] |
| 프랑스 | 3600del11, G1710X | [93] |
| 프랑스계 캐나다인 | C4446T | [94] |
| 독일인들. | 5382inC, 4184del4 | [95][96] |
| 그리스인 | 5382inC | [97] |
| 헝가리인 | 300T>G, 5382inC, 185delAG | [98] |
| 이탈리아인 | 5083del19 | [99] |
| 일본인입니다 | L63X, Q934X | [100] |
| 북미 원주민 | 1510insG, 1506A>g | [101] |
| 북아일랜드어 | 2800delAA | [102] |
| 노르웨이인 | 816delGT, 1135insA, 1675delA, 3347delAG | [103][104] |
| 파키스탄인 | 2080insA, 3889delAG, 4184del4, 4284delAG, IVS14-1A>g | [105] |
| 폴란드의 | 300T>G, 5382insC, C61G, 4153dela | [106][107] |
| 러시아인 | 5382inC, 4153dela | [108] |
| 스코틀랜드어 | 2800delAA | [102][109] |
| 스페인어 | R71G | [110][111] |
| 스웨덴어 | Q563X, 3171ins5, 1201del11, 2594delC | [83][112] |
여성의 생식력
여성이 나이가 들면서 생식능력이 떨어져 폐경이 된다.이 감소는 난포의 수 감소와 관련이 있다.난소에는 약 100만 개의 난모세포가 존재하지만, 이러한 난소의 약 500개(약 0.05%)만 존재한다.난소 예비력의 감소는 나이가 [113]들수록 지속적으로 증가하는 것으로 보이며, 약 52세까지 예비력이 거의 완전히 소진된다.나이가 들면서 난소 예비력과 출산력이 떨어지면서 임신부전과 감수분열 오류도 함께 증가해 염색체 [114]이상개념이 나타난다.
생식선 BRCA1 돌연변이를 가진 여성들은 정상적으로 노화된 [115]여성들에 비해 난모세포의 비축량이 감소하고 출산능력이 저하된 것으로 보인다.게다가 유전적인 BRCA1 돌연변이를 가진 여성은 조기 [116]폐경을 겪는다.BRCA1은 핵심 DNA 복구 단백질이기 때문에, 이러한 연구결과는 난모세포에서 자연적으로 발생하는 DNA 손상이 BRCA1 결함이 있는 여성에서 덜 효율적으로 복구되고, 이러한 복구 비효율성이 조기 생식 [115]실패로 이어진다는 것을 시사한다.
상술한 바와 같이 BRCA1 단백질은 상동 재조합 수복에 중요한 역할을 한다.이것은 DNA 이중가닥 절단을 정확하게 복구할 수 있는 알려진 유일한 세포 과정이다.원시 모낭에서 [117]인간과 생쥐의 나이가 들수록 DNA 이중 가닥이 끊어진다.원시 모낭은 감수 분열의 중간 단계인 난모세포를 포함한다.감수분열은 진핵생물에서 생식세포가 형성되는 일반적인 과정으로, 생식선 [citation needed]DNA에서 DNA 손상, 특히 이중사슬 파손을 제거하기 위한 적응일 수 있다(감수분열 참조).감수분열 시에는 특히 BRCA1을 이용한 상동재조합수리가 촉진된다.4개가 중요한 유전자 DNAdouble-strand 휴식의 상동recombinational 수리에 필요한 표현 나이와 함께 인간과 mice,[117]의 이론이 DNAdouble-strand 방학 수리 난모 세포 저장소의 유지에 필요한 것의 난모 세포가 하락(1기 유방 암, MRE11, RAD51와 현금 자동 지급기)발견되었다 effic의 하락.ien나이와 함께 수복되는 cy는 난소 노화에 역할을 한다.
암화학요법
비소세포 폐암은 전 세계적으로 암 사망의 주요 원인이다.진단 시 NSCLC 환자 중 거의 70%가 국소적으로 진행되거나 전이성 질환을 앓고 있습니다.NSCLC를 가진 사람은 종종 DNA에서 가닥간 교합을 일으키는 치료용 백금 화합물(예: 시스플라틴, 카르보플라틴 또는 옥살플라틴)로 치료된다.NSCLC를 가진 사람 중, 1차 종양에서 BRCA1의 낮은 발현은 백금 함유 화학 치료 [118][119]후 생존 개선과 관련이 있었다.이러한 상관관계는 암의 낮은 BRCA1과 그에 따른 낮은 수준의 DNA 복구가 DNA 가교제에 의한 치료에 대한 암의 취약성을 야기한다는 것을 의미한다.높은 BRCA1은 백금 약물에 의해 도입된 DNA의 손상을 제거하는 경로에서 작용함으로써 암세포를 보호할 수 있다.따라서 BRCA1 발현 수준은 폐암 [118][119]관리에 있어 화학요법을 맞춤화하는 데 잠재적으로 중요한 도구이다.
BRCA1 발현 수준은 난소암 치료와도 관련이 있다.백금제 치료를 받은 산발성 난소암 환자는 BRCA1 발현이 높은 환자(33개월에 비해 46개월)[120]에 비해 BRCA1 발현이 낮으면 중앙 생존 시간이 더 길었다.
특허, 집행, 소송 및 논란
이 고립된 1기 유방 암 유전자 암 돌연변이 위 홍보뿐만 아니라 방법 유방 암에 걸릴 가능성을 진단하기에 대한 특허 출원 유타 대학, 국립 환경 보건 과학원(국립 환경 보건 과학원)과 Myriad Genetics1994년, 1년에 걸쳐 Myriad과의 협력에[18]에 의해 제기됐습니다. inveEndo Recherche, Inc., HSC Research & Development Limited Partnership 및 펜실베니아 대학의 스티그네이터는 BRCA2 유전자를 분리 및 배열하고 주요 돌연변이를 확인했으며,[19] 1995년 미리아드와 다른 기관에 의해 미국에서 첫 번째 BRCA2 특허가 출원되었다.Miriard는 이러한 특허의 배타적 사용권자로서 미국에서 임상 진단 [21]연구소에 대해 특허를 시행하고 있습니다.이것은 1994년 사업 모델 Myriad하는 것은 창업 1200직원과달러 500M 연간 매출액은 2012년에, 상장 회사;[20]그것은 또한 높은 물가로 인해 논란과 이것이 이번에는 획기적인 협회 분자 병리학적 대 내로 다른 진단 실험실,에서 두번째 의견을 받기 위해서 이끌었다.리광고제네틱스 소송.[21][121]특허는 2014년에 만료되기 시작했다.
Genetic Medicine 저널에 2010년에 게재된 기사에 따르면, "미국 밖의 특허 이야기는 더 복잡하다...예를 들어, 특허는 획득되었지만 캐나다의 지방 보건 시스템에 의해 무시되고 있습니다.호주와 영국에서는 미리어드의 면허소지자가 보건 시스템의 사용을 허용했지만 2008년 8월에 계획 변경을 발표했다.일부 특허는 이의신청 절차를 검토 중이지만, 미리어드의 유일한 유럽 전체 특허에서 단 하나의 돌연변이만 특허를 받았다.사실상 미리어드의 강력한 특허 지위가 단독 제공자 [122][123]지위를 부여한 것은 미국뿐입니다.Miriard Genetics의 CEO인 Peter Meldrum은 Miriard가 유럽에서 "[124]이러한 [특허] 강제성을 불필요하게 만들 수 있는 다른 경쟁 우위"를 가지고 있다고 인정했습니다.
다른 유전자와 마찬가지로 BRCA1의 변이를 찾는 것은 어렵지 않다.실제 가치는 특정 변종의 임상적 결과가 무엇인지 이해하는 데서 나온다.Miriard는 이러한 유전자형-표현형 상관관계를 가진 대규모 독점 데이터베이스를 보유하고 있습니다.이에 대응하여 병렬 오픈 소스 데이터베이스가 개발되고 있습니다.
BRCA1 및 BRCA2 특허를 둘러싼 법적 결정은 일반적으로 [125]유전자 검사 분야에 영향을 미칠 것이다.2013년 6월 '분자병리학회 v. 미리아드 유전학(Association for Molecular Pathology v. Mariard Genetics, No. 12-398)'의 기사는 "자연발생 DNA 세그먼트는 자연의 산물이며 단지 분리되었다는 이유만으로 특허를 받을 자격이 없다"는 미국 대법원의 만장일치 판결을 인용, 미리아드의 BRCA1 및 BRCA2 유전자 특허를 무효화했다.그러나 법원은 또한 자연에서 발견되지 않는 것을 만들기 위해 유전자를 조작하는 것은 여전히 특허 [126]보호를 받을 자격이 있다고 판결했다.호주 연방법원은 2013년 [127]2월 BRCA1 유전자에 대한 호주 미리어드 제네틱스 특허의 유효성을 주장하며 정반대의 결론을 내렸다.연방법원도 2014년 [128]9월 항소를 기각했다.Yvonne D'Arcy는 호주 고등법원에서 미국 소재 생명공학 회사인 Miriad Genetics를 상대로 한 소송에서 승소했다.2015년 10월 7일 만장일치로, 고등법원은 BRCA1 단백질을 코드화한 분리된 핵산이 유방암과 난소암에 걸리기 쉽다는 것을 나타내는 기준과 특정한 차이가 있는 것은 '특허 발명'이 아니라고 판결했다."[129]
상호 작용
BRCA1은 다음과 같은 단백질과 상호작용하는 것으로 나타났다.
- ABL1[130]
- AKT1[131][132]
- AR[133]
- ATR[134][135][136][137]
- 현금 자동 입출금기[16][134][135][136][137][138][139]
- ATF1[140]
- 바흐1[141]
- 바드1[30][38][42][141]
- BRCA2[142][143][144][145]
- 브릭스3[142]
- BRE[142]
- BRIP1[34][146][147][148][149][150]
- 씨준[151]
- CHEK2[152][153]
- CLSPN[154]
- 코브라1[155]
- 삐걱거리다[156][157][158][159][160]
- CSNK2B[161]
- CSTF2[162][163]
- CDK2[164][165][166]
- DHX9[167][168]
- ELK4[169]
- EP300[157][159]
- ESR1[159][170][171][172]
- 팬카[173]
- FANCD2[174][144]
- FHL2[175][176]
- H2AFX[177][178][179]
- 준비[151]
- 준디[151]
- LMO4[180][181]
- 맵3K3[182]
- MED1[147]
- MED17[183][147][184]
- MED21[185]
- MED24[147]
- MRE11A[16][183][186][187]
- MSH2[16][38]
- MSH3[38][146]
- MSH6[16][38]
- 마이크[42][188][189][190]
- NBN[16][183][186]
- NMI[188]
- NPM1[191]
- NCOA2[146][192]
- NUFIP1[193]
- P53[142][158][194][195][196]
- PALB2[197]
- POLR2A[183][185][198][199]
- PPP1CA[200]
- Rad50[16][183][186]
- RAD51[38][142][143][201]
- RBBP4[202]
- RBBP7[202][203][204]
- RBBP8[205][146][206][207][208][209][210]
- 리라[156]
- RB1[202][211][212]
- RBL1[211]
- RBL2[211]
- RPL31[204]
- SMARCA4[213][214]
- SMARCB1[213]
- STAT1[215]
- UBE2D1[177][216][217][218][178][142][191][174][219][220]
- USF2[221]
- VCP[222]
- XIST[223][224]
- ZNF350[225]
레퍼런스
- ^ a b c GRCh38: 앙상블 릴리즈 89: ENSG000012048 - 앙상블, 2017년 5월
- ^ a b c GRCm38: 앙상블 릴리즈 89: ENSMUSG000017146 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ Hamel PJ (2007-05-29). "BRCA1 and BRCA2: No Longer the Only Troublesome Genes Out There". HealthCentral. Retrieved 2010-07-02.
- ^ a b "BRCA1 gene tree". Ensembl.
- ^ Duncan JA, Reeves JR, Cooke TG (October 1998). "BRCA1 and BRCA2 proteins: roles in health and disease". Molecular Pathology. 51 (5): 237–47. doi:10.1136/mp.51.5.237. PMC 395646. PMID 10193517.
- ^ Yoshida K, Miki Y (November 2004). "Role of BRCA1 and BRCA2 as regulators of DNA repair, transcription, and cell cycle in response to DNA damage". Cancer Science. 95 (11): 866–71. doi:10.1111/j.1349-7006.2004.tb02195.x. PMID 15546503. S2CID 24297965.
- ^ Check W (2006-09-01). "BRCA: What we know now". College of American Pathologists. Retrieved 2010-08-23.
- ^ a b Irminger-Finger I, Ratajska M, Pilyugin M (2016). "New concepts on BARD1: Regulator of BRCA pathways and beyond". The International Journal of Biochemistry & Cell Biology. 72: 1–17. doi:10.1016/j.biocel.2015.12.008. PMID 26738429.
- ^ Friedenson B (August 2007). "The BRCA1/2 pathway prevents hematologic cancers in addition to breast and ovarian cancers". BMC Cancer. 7: 152–162. doi:10.1186/1471-2407-7-152. PMC 1959234. PMID 17683622.
- ^ Friedenson B (2008-06-08). "Breast cancer genes protect against some leukemias and lymphomas" (video). SciVee.
- ^ "Breast and Ovarian Cancer Genetic Screening". Palo Alto Medical Foundation. Archived from the original on 4 October 2008. Retrieved 2008-10-11.
- ^ a b c Friedenson B (2007). "The BRCA1/2 pathway prevents hematologic cancers in addition to breast and ovarian cancers". BMC Cancer. 7: 152. doi:10.1186/1471-2407-7-152. PMC 1959234. PMID 17683622.
- ^ O'Donovan PJ, Livingston DM (April 2010). "BRCA1 and BRCA2: breast/ovarian cancer susceptibility gene products and participants in DNA double-strand break repair". Carcinogenesis. 31 (6): 961–7. doi:10.1093/carcin/bgq069. PMID 20400477.
- ^ a b c d e f g Wang Y, Cortez D, Yazdi P, Neff N, Elledge SJ, Qin J (April 2000). "BASC, a super complex of BRCA1-associated proteins involved in the recognition and repair of aberrant DNA structures". Genes Dev. 14 (8): 927–39. doi:10.1101/gad.14.8.927. PMC 316544. PMID 10783165.
- ^ a b Starita LM, Parvin JD (2003). "The multiple nuclear functions of BRCA1: transcription, ubiquitination and DNA repair". Current Opinion in Cell Biology. 15 (3): 345–350. doi:10.1016/S0955-0674(03)00042-5. PMID 12787778.
- ^ a b c 미국 특허 5747282, Skolnick HS, Goldgar DE, Miki Y, Swenson J, Kamb A, Harsman KD, Shattuck-Eidens DM, Tavtigian SV, Wiseman RW, Futreal PA, "7Q 연계 유방 및 난소암 감수성 유전자" 1998년 발표보건 및 인적 서비스 장관 및 유타 대학 연구 재단으로 대표되는 미국
- ^ a b 미국 특허 5837492, Tavtigian SV, Kamb A, Simard J, Couch F, Rommens JM, Weber BL, 1998-11-17 발행, Mirious Genetics, Inc., Endo Recherche, HSC.에 할당된 "Chromosome 13-linked 유방암 감수성 유전자.
- ^ a b Mirad Investor 페이지—2012년 10월에 액세스한 웨이백 머신에서 2012-10-18년 아카이브된 "Myriad 요약" 참조
- ^ a b c Schwartz J (2009-05-12). "Cancer Patients Challenge the Patenting of a Gene". The New York Times. Health.
- ^ Hall JM, Lee MK, Newman B, Morrow JE, Anderson LA, Huey B, King MC (December 1990). "Linkage of early-onset familial breast cancer to chromosome 17q21". Science. 250 (4988): 1684–9. Bibcode:1990Sci...250.1684H. doi:10.1126/science.2270482. PMID 2270482.
- ^ 영향력이 큰 과학: BRCA 유전자 추적 (Part 1) – Cancer Research UK 과학 블로그, 2012
- ^ Miki Y, Swensen J, Shattuck-Eidens D, Futreal PA, Harshman K, Tavtigian S, Liu Q, Cochran C, Bennett LM, Ding W (October 1994). "A strong candidate for the breast and ovarian cancer susceptibility gene BRCA1". Science. 266 (5182): 66–71. Bibcode:1994Sci...266...66M. doi:10.1126/science.7545954. PMID 7545954.
- ^ 국립생명공학정보센터, 미국 국립의학도서관 EntrezGene의 BRCA1 유방암에 대한 참조정보, 조기발병(Homo sapiens)
- ^ Paterson JW (February 1998). "BRCA1: a review of structure and putative functions". Dis. Markers. 13 (4): 261–74. doi:10.1155/1998/298530. PMID 9553742.
- ^ Henderson BR (September 2005). "Regulation of BRCA1, BRCA2 and BARD1 intracellular trafficking". BioEssays. 27 (9): 884–93. doi:10.1002/bies.20277. PMID 16108063. S2CID 10138907.
- ^ UniProt의 "유방암 타입 1 감수성 단백질"에 대한 범용 단백질 자원 가입 번호 P38398.
- ^ a b Clark SL, Rodriguez AM, Snyder RR, Hankins GD, Boehning D (April 2012). "Structure-Function Of The Tumor Suppressor BRCA1". Comput Struct Biotechnol J. 1 (1): e201204005. doi:10.5936/csbj.201204005. PMC 3380633. PMID 22737296.
- ^ a b c d Brzovic PS, Rajagopal P, Hoyt DW, King MC, Klevit RE (October 2001). "Structure of a BRCA1-BARD1 heterodimeric RING-RING complex". Nature Structural & Molecular Biology. 8 (10): 833–7. doi:10.1038/nsb1001-833. PMID 11573085. S2CID 37617901.
- ^ Baer R (October 2001). "With the ends in sight: images from the BRCA1 tumor suppressor". Nature Structural & Molecular Biology. 8 (10): 822–4. doi:10.1038/nsb1001-822. PMID 11573079. S2CID 20552445.
- ^ a b Williams RS, Green R, Glover JN (October 2001). "Crystal structure of the BRCT repeat region from the breast cancer-associated protein BRCA1". Nature Structural & Molecular Biology. 8 (10): 838–42. doi:10.1038/nsb1001-838. PMID 11573086. S2CID 19275284.
- ^ Huyton T, Bates PA, Zhang X, Sternberg MJ, Freemont PS (August 2000). "The BRCA1 C-terminal domain: structure and function". Mutat. Res. 460 (3–4): 319–32. doi:10.1016/S0921-8777(00)00034-3. PMID 10946236.
- ^ a b Joo WS, Jeffrey PD, Cantor SB, Finnin MS, Livingston DM, Pavletich NP (March 2002). "Structure of the 53BP1 BRCT region bound to p53 and its comparison to the Brca1 BRCT structure". Genes Dev. 16 (5): 583–93. doi:10.1101/gad.959202. PMC 155350. PMID 11877378.
- ^ Sawyer SL, Tian L, Kahkonen M, Schwartzentruber J, Kircher M, Majewski J, Dyment DA, Innes AM, Boycott KM, Moreau LA, Moilanen JS, Greenberg RA (2014). "Biallelic Mutations in BRCA1 Cause a New Fanconi Anemia Subtype". Cancer Discov. 5 (2): 135–42. doi:10.1158/2159-8290.CD-14-1156. PMC 4320660. PMID 25472942.
- ^ "Kimball's Biologh Pages". Archived from the original on 2018-02-12. Retrieved 2010-02-25.
- ^ Boulton SJ (November 2006). "Cellular functions of the BRCA tumour-suppressor proteins". Biochem. Soc. Trans. 34 (Pt 5): 633–45. doi:10.1042/BST0340633. PMID 17052168.
- ^ a b c d e f Wang Q, Zhang H, Guerrette S, Chen J, Mazurek A, Wilson T, Slupianek A, Skorski T, Fishel R, Greene MI (August 2001). "Adenosine nucleotide modulates the physical interaction between hMSH2 and BRCA1". Oncogene. 20 (34): 4640–9. doi:10.1038/sj.onc.1204625. PMID 11498787.
- ^ Warmoes M, Jaspers JE, Pham TV, Piersma SR, Oudgenoeg G, Massink MP, Waisfisz Q, Rottenberg S, Boven E, Jonkers J, Jimenez CR (July 2012). "Proteomics of mouse BRCA1-deficient mammary tumors identifies DNA repair proteins with potential diagnostic and prognostic value in human breast cancer". Mol. Cell. Proteomics. 11 (7): M111.013334-1-M111.013334-19. doi:10.1074/mcp.M111.013334. PMC 3394939. PMID 22366898.
- ^ Meerang M, Ritz D, Paliwal S, Garajova Z, Bosshard M, Mailand N, Janscak P, Hübscher U, Meyer H, Ramadan K (November 2011). "The ubiquitin-selective segregase VCP/p97 orchestrates the response to DNA double-strand breaks". Nat. Cell Biol. 13 (11): 1376–82. doi:10.1038/ncb2367. PMID 22020440. S2CID 22109822.
- ^ Zhang H, Wang Q, Kajino K, Greene MI (2000). "VCP, a weak ATPase involved in multiple cellular events, interacts physically with BRCA1 in the nucleus of living cells". DNA Cell Biol. 19 (5): 253–263. doi:10.1089/10445490050021168. PMID 10855792.
- ^ a b c Wang Q, Zhang H, Kajino K, Greene MI (October 1998). "BRCA1 binds c-Myc and inhibits its transcriptional and transforming activity in cells". Oncogene. 17 (15): 1939–48. doi:10.1038/sj.onc.1202403. PMID 9788437.
- ^ Paull TT, Cortez D, Bowers B, Elledge SJ, Gellert M (2001). "Direct DNA binding by Brca1". Proceedings of the National Academy of Sciences. 98 (11): 6086–6091. doi:10.1073/pnas.111125998. PMC 33426. PMID 11353843.
- ^ Durant ST, Nickoloff JA (2005). "Good timing in the cell cycle for precise DNA repair by BRCA1". Cell Cycle. 4 (9): 1216–22. doi:10.4161/cc.4.9.2027. PMID 16103751.
- ^ a b c Ye Q, Hu YF, Zhong H, Nye AC, Belmont AS, Li R (2001). "BRCA1-induced large-scale chromatin unfolding and allele-specific effects of cancer-predisposing mutations". The Journal of Cell Biology. 155 (6): 911–922. doi:10.1083/jcb.200108049. PMC 2150890. PMID 11739404.
- ^ Ridpath JR, Nakamura A, Tano K, Luke AM, Sonoda E, Arakawa H, Buerstedde JM, Gillespie DA, Sale JE, Yamazoe M, Bishop DK, Takata M, Takeda S, Watanabe M, Swenberg JA, Nakamura J (December 2007). "Cells deficient in the FANC/BRCA pathway are hypersensitive to plasma levels of formaldehyde". Cancer Res. 67 (23): 11117–22. doi:10.1158/0008-5472.CAN-07-3028. PMID 18056434.
- ^ Prakash R, Zhang Y, Feng W, Jasin M (April 2015). "Homologous recombination and human health: the roles of BRCA1, BRCA2, and associated proteins". Cold Spring Harbor Perspectives in Biology. 7 (4): a016600. doi:10.1101/cshperspect.a016600. PMC 4382744. PMID 25833843.
- ^ Scully R, Anderson SF, Chao DM, Wei W, Ye L, Young RA, Livingston DM, Parvin JD (1997). "BRCA1 is a component of the RNA polymerase II holoenzyme". Proceedings of the National Academy of Sciences. 94 (11): 5605–10. Bibcode:1997PNAS...94.5605S. doi:10.1073/pnas.94.11.5605. PMC 20825. PMID 9159119.
- ^ Bochar DA, Wang L, Beniya H, Kinev A, Xue Y, Lane WS, Wang W, Kashanchi F, Shiekhattar R (2000). "BRCA1 Is Associated with a Human SWI/SNF-Related Complex Linking Chromatin Remodeling to Breast Cancer". Cell. 102 (2): 257–265. doi:10.1016/S0092-8674(00)00030-1. PMID 10943845.
- ^ Petrucelli N, Daly MB, Pal T (December 2016) [September 1998]. "BRCA1- and BRCA2-Associated Hereditary Breast and Ovarian Cancer". In Adam MP, Ardinger HH, Pagon RA, Wallace SE, Bean LJ, Mirzaa G, Amemiya A (eds.). GeneReviews. University of Washington, Seattle. PMID 20301425.
- ^ "Genetics". Breastcancer.org. 2012-09-17.
- ^ Mazoyer S (May 2005). "Genomic rearrangements in the BRCA1 and BRCA2 genes". Hum. Mutat. 25 (5): 415–22. doi:10.1002/humu.20169. PMID 15832305. S2CID 32023181.
- ^ Barrois M, Bièche I, Mazoyer S, Champème MH, Bressac-de Paillerets B, Lidereau R (February 2004). "Real-time PCR-based gene dosage assay for detecting BRCA1 rearrangements in breast-ovarian cancer families". Clin. Genet. 65 (2): 131–6. doi:10.1111/j.0009-9163.2004.00200.x. PMID 14984472. S2CID 11583160.
- ^ Hogervorst FB, Nederlof PM, Gille JJ, McElgunn CJ, Grippeling M, Pruntel R, Regnerus R, van Welsem T, van Spaendonk R, Menko FH, Kluijt I, Dommering C, Verhoef S, Schouten JP, van't Veer LJ, Pals G (April 2003). "Large genomic deletions and duplications in the BRCA1 gene identified by a novel quantitative method". Cancer Res. 63 (7): 1449–53. PMID 12670888.
- ^ Casilli F, Di Rocco ZC, Gad S, Tournier I, Stoppa-Lyonnet D, Frebourg T, Tosi M (September 2002). "Rapid detection of novel BRCA1 rearrangements in high-risk breast-ovarian cancer families using multiplex PCR of short fluorescent fragments". Hum. Mutat. 20 (3): 218–26. doi:10.1002/humu.10108. PMID 12203994. S2CID 24737909.
- ^ Rouleau E, Lefol C, Tozlu S, Andrieu C, Guy C, Copigny F, Nogues C, Bieche I, Lidereau R (September 2007). "High-resolution oligonucleotide array-CGH applied to the detection and characterization of large rearrangements in the hereditary breast cancer gene BRCA1". Clin. Genet. 72 (3): 199–207. doi:10.1111/j.1399-0004.2007.00849.x. PMID 17718857. S2CID 2393567.
- ^ Tapia T, Smalley SV, Kohen P, Muñoz A, Solis LM, Corvalan A, Faundez P, Devoto L, Camus M, Alvarez M, Carvallo P (2008). "Promoter hypermethylation of BRCA1 correlates with absence of expression in hereditary breast cancer tumors". Epigenetics. 3 (3): 157–63. doi:10.4161/epi.3.3.6387. PMID 18567944. S2CID 35616414.
- ^ Shen J, Ambrosone CB, Zhao H (March 2009). "Novel genetic variants in microRNA genes and familial breast cancer". Int. J. Cancer. 124 (5): 1178–82. doi:10.1002/ijc.24008. PMID 19048628.
- ^ Levin B, Lech D, Friedenson B (2012). "Evidence that BRCA1- or BRCA2-associated cancers are not inevitable". Mol Med. 18 (9): 1327–37. doi:10.2119/molmed.2012.00280. PMC 3521784. PMID 22972572.
- ^ Wilson CA, Ramos L, Villaseñor MR, Anders KH, Press MF, Clarke K, Karlan B, Chen JJ, Scully R, Livingston D, Zuch RH, Kanter MH, Cohen S, Calzone FJ, Slamon DJ (1999). "Localization of human BRCA1 and its loss in high-grade, non-inherited breast carcinomas". Nat. Genet. 21 (2): 236–40. doi:10.1038/6029. PMID 9988281. S2CID 7988460.
- ^ Mueller CR, Roskelley CD (2003). "Regulation of BRCA1 expression and its relationship to sporadic breast cancer". Breast Cancer Res. 5 (1): 45–52. doi:10.1186/bcr557. PMC 154136. PMID 12559046.
- ^ a b Jacinto FV, Esteller M (2007). "Mutator pathways unleashed by epigenetic silencing in human cancer". Mutagenesis. 22 (4): 247–53. doi:10.1093/mutage/gem009. PMID 17412712.
- ^ a b c d Sun C, Li N, Yang Z, Zhou B, He Y, Weng D, Fang Y, Wu P, Chen P, Yang X, Ma D, Zhou J, Chen G (2013). "miR-9 regulation of BRCA1 and ovarian cancer sensitivity to cisplatin and PARP inhibition". J. Natl. Cancer Inst. 105 (22): 1750–8. doi:10.1093/jnci/djt302. PMID 24168967.
- ^ McMillen BD, Aponte MM, Liu Z, Helenowski IB, Scholtens DM, Buttin BM, Wei JJ (2012). "Expression analysis of MIR182 and its associated target genes in advanced ovarian carcinoma". Mod. Pathol. 25 (12): 1644–53. doi:10.1038/modpathol.2012.118. PMID 22790015.
- ^ Bowtell DD (2010). "The genesis and evolution of high-grade serous ovarian cancer". Nat. Rev. Cancer. 10 (11): 803–8. doi:10.1038/nrc2946. PMID 20944665. S2CID 22688947.
- ^ Brody LC, Biesecker BB (1998). "Breast cancer susceptibility genes. BRCA1 and BRCA2". Medicine (Baltimore). 77 (3): 208–26. doi:10.1097/00005792-199805000-00006. PMID 9653432.
- ^ Pennington KP, Walsh T, Harrell MI, Lee MK, Pennil CC, Rendi MH, Thornton A, Norquist BM, Casadei S, Nord AS, Agnew KJ, Pritchard CC, Scroggins S, Garcia RL, King MC, Swisher EM (2014). "Germline and somatic mutations in homologous recombination genes predict platinum response and survival in ovarian, fallopian tube, and peritoneal carcinomas". Clin. Cancer Res. 20 (3): 764–75. doi:10.1158/1078-0432.CCR-13-2287. PMC 3944197. PMID 24240112.
- ^ Polansky H, Schwab H (August 2019). "How latent viruses cause breast cancer: An explanation based on the microcompetition model". Bosnian Journal of Basic Medical Sciences. 19 (3): 221–226. doi:10.17305/bjbms.2018.3950. PMC 6716096. PMID 30579323.
- ^ Esteller M, Silva JM, Dominguez G, Bonilla F, Matias-Guiu X, Lerma E, Bussaglia E, Prat J, Harkes IC, Repasky EA, Gabrielson E, Schutte M, Baylin SB, Herman JG (2000). "Promoter hypermethylation and BRCA1 inactivation in sporadic breast and ovarian tumors". J. Natl. Cancer Inst. 92 (7): 564–9. doi:10.1093/jnci/92.7.564. PMID 10749912.
- ^ a b Moskwa P, Buffa FM, Pan Y, Panchakshari R, Gottipati P, Muschel RJ, Beech J, Kulshrestha R, Abdelmohsen K, Weinstock DM, Gorospe M, Harris AL, Helleday T, Chowdhury D (2011). "miR-182-mediated downregulation of BRCA1 impacts DNA repair and sensitivity to PARP inhibitors". Mol. Cell. 41 (2): 210–20. doi:10.1016/j.molcel.2010.12.005. PMC 3249932. PMID 21195000.
- ^ Krishnan K, Steptoe AL, Martin HC, Wani S, Nones K, Waddell N, Mariasegaram M, Simpson PT, Lakhani SR, Gabrielli B, Vlassov A, Cloonan N, Grimmond SM (2013). "MicroRNA-182-5p targets a network of genes involved in DNA repair". RNA. 19 (2): 230–42. doi:10.1261/rna.034926.112. PMC 3543090. PMID 23249749.
- ^ Garcia AI, Buisson M, Bertrand P, Rimokh R, Rouleau E, Lopez BS, Lidereau R, Mikaélian I, Mazoyer S (2011). "Down-regulation of BRCA1 expression by miR-146a and miR-146b-5p in triple negative sporadic breast cancers". EMBO Mol Med. 3 (5): 279–90. doi:10.1002/emmm.201100136. PMC 3377076. PMID 21472990.
- ^ a b Liu Z, Liu J, Segura MF, Shao C, Lee P, Gong Y, Hernando E, Wei JJ (2012). "MiR-182 overexpression in tumourigenesis of high-grade serous ovarian carcinoma". J. Pathol. 228 (2): 204–15. doi:10.1002/path.4000. PMID 22322863. S2CID 206325689.
- ^ Kastan MB (2008). "DNA damage responses: mechanisms and roles in human disease: 2007 G.H.A. Clowes Memorial Award Lecture". Mol. Cancer Res. 6 (4): 517–24. doi:10.1158/1541-7786.MCR-08-0020. PMID 18403632.
- ^ Harper JW, Elledge SJ (2007). "The DNA damage response: ten years after". Mol. Cell. 28 (5): 739–45. doi:10.1016/j.molcel.2007.11.015. PMID 18082599.
- ^ O'Hagan HM, Mohammad HP, Baylin SB (2008). "Double strand breaks can initiate gene silencing and SIRT1-dependent onset of DNA methylation in an exogenous promoter CpG island". PLOS Genetics. 4 (8): e1000155. doi:10.1371/journal.pgen.1000155. PMC 2491723. PMID 18704159.
- ^ Cuozzo C, Porcellini A, Angrisano T, Morano A, Lee B, Di Pardo A, Messina S, Iuliano R, Fusco A, Santillo MR, Muller MT, Chiariotti L, Gottesman ME, Avvedimento EV (Jul 2007). "DNA damage, homology-directed repair, and DNA methylation". PLOS Genetics. 3 (7): e110. doi:10.1371/journal.pgen.0030110. PMC 1913100. PMID 17616978.
- ^ a b Lacroix M, Leclercq G (2005). "The "portrait" of hereditary breast cancer". Breast Cancer Research and Treatment. 89 (3): 297–304. doi:10.1007/s10549-004-2172-4. PMID 15754129. S2CID 23327569.
- ^ a b Struewing JP, Abeliovich D, Peretz T, Avishai N, Kaback MM, Collins FS, Brody LC (October 1995). "The carrier frequency of the BRCA1 185delAG mutation is approximately 1 percent in Ashkenazi Jewish individuals". Nat. Genet. 11 (2): 198–200. doi:10.1038/ng1095-198. PMID 7550349. S2CID 21387351.
- ^ a b Tonin P, Serova O, Lenoir G, Lynch H, Durocher F, Simard J, Morgan K, Narod S (1995). "BRCA1 mutations in Ashkenazi Jewish women". American Journal of Human Genetics. 57 (1): 189. PMC 1801236. PMID 7611288.
- ^ Narod SA, Foulkes WD (2004). "BRCA1 and BRCA2: 1994 and beyond". Nature Reviews Cancer. 4 (9): 665–676. doi:10.1038/nrc1431. PMID 15343273. S2CID 30686068.
- ^ den Dunnen JT, Antonarakis SE (2000). "Mutation nomenclature extensions and suggestions to describe complex mutations: a discussion". Human Mutation. 15 (1): 7–12. doi:10.1002/(SICI)1098-1004(200001)15:1<7::AID-HUMU4>3.0.CO;2-N. PMID 10612815.
- ^ a b Neuhausen SL (2000). "Founder populations and their uses for breast cancer genetics". Cancer Research. 2 (2): 77–81. doi:10.1186/bcr36. PMC 139426. PMID 11250694.
- ^ Reeves MD, Yawitch TM, van der Merwe NC, van den Berg HJ, Dreyer G, van Rensburg EJ (July 2004). "BRCA1 mutations in South African breast and/or ovarian cancer families: evidence of a novel founder mutation in Afrikaner families". Int. J. Cancer. 110 (5): 677–82. doi:10.1002/ijc.20186. PMID 15146556. S2CID 22970255.
- ^ Francies FZ, Wainstein T, De Leeneer K, Cairns A, Murdoch M, Nietz S, Cubasch H, Poppe B, Van Maerken T, Crombez B, Coene I, Kerr R, Slabbert JP, Vral A, Krause A, Baeyens A (Nov 2015). "BRCA1, BRCA2 and PALB2 mutations and CHEK2 c.1100delC in different South African ethnic groups diagnosed with premenopausal and/or triple negative breast cancer". BMC Cancer. 15: 912. doi:10.1186/s12885-015-1913-6. PMC 4647511. PMID 26577449.
- ^ Wagner TM, Möslinger RA, Muhr D, Langbauer G, Hirtenlehner K, Concin H, Doeller W, Haid A, Lang AH, Mayer P, Ropp E, Kubista E, Amirimani B, Helbich T, Becherer A, Scheiner O, Breiteneder H, Borg A, Devilee P, Oefner P, Zielinski C (1998). "BRCA1-related breast cancer in Austrian breast and ovarian cancer families: specific BRCA1 mutations and pathological characteristics". International Journal of Cancer. 77 (3): 354–360. doi:10.1002/(SICI)1097-0215(19980729)77:3<354::AID-IJC8>3.0.CO;2-N. PMID 9663595.
- ^ a b Peelen T, van Vliet M, Petrij-Bosch A, Mieremet R, Szabo C, van den Ouweland AM, Hogervorst F, Brohet R, Ligtenberg MJ, Teugels E, van der Luijt R, van der Hout AH, Gille JJ, Pals G, Jedema I, Olmer R, van Leeuwen I, Newman B, Plandsoen M, van der Est M, Brink G, Hageman S, Arts PJ, Bakker MM, Devilee P (1997). "A high proportion of novel mutations in BRCA1 with strong founder effects among Dutch and Belgian hereditary breast and ovarian cancer families". American Journal of Human Genetics. 60 (5): 1041–1049. PMC 1712432. PMID 9150151.
- ^ Claes K, Machackova E, De Vos M, Poppe B, De Paepe A, Messiaen L (1999). "Mutation analysis of the BRCA1 and BRCA2 genes in the Belgian patient population and identification of a Belgian founder mutation BRCA1 IVS5 + 3A > G". Disease Markers. 15 (1–3): 69–73. doi:10.1155/1999/241046. PMC 3851655. PMID 10595255.
- ^ Petrij-Bosch A, Peelen T, van Vliet M, van Eijk R, Olmer R, Drüsedau M, Hogervorst FB, Hageman S, Arts PJ, Ligtenberg MJ, Meijers-Heijboer H, Klijn JG, Vasen HF, Cornelisse CJ, van 't Veer LJ, Bakker E, van Ommen GJ, Devilee P (1997). "BRCA1 genomic deletions are major founder mutations in Dutch breast cancer patients" (PDF). Nature Genetics. 17 (3): 341–345. doi:10.1038/ng1197-341. hdl:1765/54808. PMID 9354803. S2CID 13028232.
- ^ Verhoog LC, van den Ouweland AM, Berns E, van Veghel-Plandsoen MM, van Staveren IL, Wagner A, Bartels CC, Tilanus-Linthorst MM, Devilee P, Seynaeve C, Halley DJ, Niermeijer MF, Klijn JG, Meijers-Heijboer H (2001). "Large regional differences in the frequency of distinct BRCA1/BRCA2 mutations in 517 Dutch breast and/or ovarian cancer families". European Journal of Cancer. 37 (16): 2082–2090. doi:10.1016/S0959-8049(01)00244-1. PMID 11597388.
- ^ Huusko P, Pääkkönen K, Launonen V, Pöyhönen M, Blanco G, Kauppila A, Puistola U, Kiviniemi H, Kujala M, Leisti J, Winqvist R (1998). "Evidence of founder mutations in Finnish BRCA1 and BRCA2 families". American Journal of Human Genetics. 62 (6): 1544–1548. doi:10.1086/301880. PMC 1377159. PMID 9585608.
- ^ Pääkkönen K, Sauramo S, Sarantaus L, Vahteristo P, Hartikainen A, Vehmanen P, Ignatius J, Ollikainen V, Kääriäinen H, Vauramo E, Nevanlinna H, Krahe R, Holli K, Kere J (2001). "Involvement of BRCA1 and BRCA2 in breast cancer in a western Finnish sub-population". Genetic Epidemiology. 20 (2): 239–246. doi:10.1002/1098-2272(200102)20:2<239::AID-GEPI6>3.0.CO;2-Y. PMID 11180449. S2CID 41804152.
- ^ Muller D, Bonaiti-Pellié C, Abecassis J, Stoppa-Lyonnet D, Fricker JP (2004). "BRCA1 testing in breast and/or ovarian cancer families from northeastern France identifies two common mutations with a founder effect". Familial Cancer. 3 (1): 15–20. doi:10.1023/B:FAME.0000026819.44213.df. PMID 15131401. S2CID 24615109.
- ^ Tonin PN, Mes-Masson AM, Narod SA, Ghadirian P, Provencher D (1999). "Founder BRCA1 and BRCA2 mutations in French Canadian ovarian cancer cases unselected for family history". Clinical Genetics. 55 (5): 318–324. doi:10.1034/j.1399-0004.1999.550504.x. PMID 10422801. S2CID 23931343.
- ^ Backe J, Hofferbert S, Skawran B, Dörk T, Stuhrmann M, Karstens JH, Untch M, Meindl A, Burgemeister R, Chang-Claude J, Weber BH (1999). "Frequency of BRCA1 mutation 5382insC in German breast cancer patients". Gynecologic Oncology. 72 (3): 402–406. doi:10.1006/gyno.1998.5270. PMID 10053113.
- ^ "Mutation data of the BRCA1 gene". KMDB/MutationView (Keio Mutation Databases). Keio University.
- ^ Ladopoulou A, Kroupis C, Konstantopoulou I, Ioannidou-Mouzaka L, Schofield AC, Pantazidis A, Armaou S, Tsiagas I, Lianidou E, Efstathiou E, Tsionou C, Panopoulos C, Mihalatos M, Nasioulas G, Skarlos D, Haites NE, Fountzilas G, Pandis N, Yannoukakos D (2002). "Germ line BRCA1 and BRCA2 mutations in Greek breast/ovarian cancer families: 5382insC is the most frequent mutation observed". Cancer Letters. 185 (1): 61–70. doi:10.1016/S0304-3835(01)00845-X. PMID 12142080.
- ^ Van Der Looij M, Szabo C, Besznyak I, Liszka G, Csokay B, Pulay T, Toth J, Devilee P, King MC, Olah E (2000). "Prevalence of founder BRCA1 and BRCA2 mutations among breast and ovarian cancer patients in Hungary". International Journal of Cancer. 86 (5): 737–740. doi:10.1002/(SICI)1097-0215(20000601)86:5<737::AID-IJC21>3.0.CO;2-1. PMID 10797299. S2CID 25394976.
- ^ Baudi F, Quaresima B, Grandinetti C, Cuda G, Faniello C, Tassone P, Barbieri V, Bisegna R, Ricevuto E, Conforti S, Viel A, Marchetti P, Ficorella C, Radice P, Costanzo F, Venuta S (2001). "Evidence of a founder mutation of BRCA1 in a highly homogeneous population from southern Italy with breast/ovarian cancer". Human Mutation. 18 (2): 163–164. doi:10.1002/humu.1167. PMID 11462242. S2CID 2995.
- ^ Sekine M, Nagata H, Tsuji S, Hirai Y, Fujimoto S, Hatae M, Kobayashi I, Fujii T, Nagata I, Ushijima K, Obata K, Suzuki M, Yoshinaga M, Umesaki N, Satoh S, Enomoto T, Motoyama S, Tanaka K (2001). "Mutational analysis of BRCA1 and BRCA2 and clinicopathologic analysis of ovarian cancer in 82 ovarian cancer families: two common founder mutations of BRCA1 in Japanese population". Clinical Cancer Research. 7 (10): 3144–3150. PMID 11595708.
- ^ Liede A, Jack E, Hegele RA, Narod SA (2002). "A BRCA1 mutation in Native North American families". Human Mutation. 19 (4): 460. doi:10.1002/humu.9027. PMID 11933205. S2CID 37710898.
- ^ a b The Scottish/Northern Irish BRCA1/BRCA2 Consortium (2003). "BRCA1 and BRCA2 mutations in Scotland and Northern Ireland". British Journal of Cancer. 88 (8): 1256–1262. doi:10.1038/sj.bjc.6600840. PMC 2747571. PMID 12698193.
- ^ Borg A, Dørum A, Heimdal K, Maehle L, Hovig E, Møller P (1999). "BRCA1 1675delA and 1135insA account for one third of Norwegian familial breast-ovarian cancer and are associated with later disease onset than less frequent mutations". Disease Markers. 15 (1–3): 79–84. doi:10.1155/1999/278269. PMC 3851406. PMID 10595257.
- ^ Heimdal K, Maehle L, Apold J, Pedersen JC, Møller P (2003). "The Norwegian founder mutations in BRCA1: high penetrance confirmed in an incident cancer series and differences observed in the risk of ovarian cancer". European Journal of Cancer. 39 (15): 2205–2213. doi:10.1016/S0959-8049(03)00548-3. PMID 14522380.
- ^ Liede A, Malik IA, Aziz Z, Rios Pd Pde L, Kwan E, Narod SA (2002). "Contribution of BRCA1 and BRCA2 Mutations to Breast and Ovarian Cancer in Pakistan". American Journal of Human Genetics. 71 (3): 595–606. doi:10.1086/342506. PMC 379195. PMID 12181777.
- ^ Górski B, Byrski T, Huzarski T, Jakubowska A, Menkiszak J, Gronwald J, Pluzańska A, Bebenek M, Fischer-Maliszewska L, Grzybowska E, Narod SA, Lubiński J (2000). "Founder mutations in the BRCA1 gene in Polish families with breast-ovarian cancer". American Journal of Human Genetics. 66 (6): 1963–1968. doi:10.1086/302922. PMC 1378051. PMID 10788334.
- ^ Perkowska M, BroZek I, Wysocka B, Haraldsson K, Sandberg T, Johansson U, Sellberg G, Borg A, Limon J (May 2003). "BRCA1 and BRCA2 mutation analysis in breast-ovarian cancer families from northeastern Poland". Hum. Mutat. 21 (5): 553–4. doi:10.1002/humu.9139. PMID 12673801. S2CID 7001156.
- ^ Gayther SA, Harrington P, Russell P, Kharkevich G, Garkavtseva RF, Ponder BA (May 1997). "Frequently occurring germ-line mutations of the BRCA1 gene in ovarian cancer families from Russia". Am. J. Hum. Genet. 60 (5): 1239–42. PMC 1712436. PMID 9150173.
- ^ Liede A, Cohen B, Black DM, Davidson RH, Renwick A, Hoodfar E, Olopade OI, Micek M, Anderson V, De Mey R, Fordyce A, Warner E, Dann JL, King MC, Weber B, Narod SA, Steel CM (February 2000). "Evidence of a founder BRCA1 mutation in Scotland". Br. J. Cancer. 82 (3): 705–11. doi:10.1054/bjoc.1999.0984. PMC 2363321. PMID 10682686.
- ^ Vega A, Campos B, Bressac-De-Paillerets B, Bond PM, Janin N, Douglas FS, Domènech M, Baena M, Pericay C, Alonso C, Carracedo A, Baiget M, Diez O (June 2001). "The R71G BRCA1 is a founder Spanish mutation and leads to aberrant splicing of the transcript". Hum. Mutat. 17 (6): 520–1. doi:10.1002/humu.1136. PMID 11385711. S2CID 39462456.
- ^ Campos B, Díez O, Odefrey F, Domènech M, Moncoutier V, Martínez-Ferrandis JI, Osorio A, Balmaña J, Barroso A, Armengod ME, Benítez J, Alonso C, Stoppa-Lyonnet D, Goldgar D, Baiget M (April 2003). "Haplotype analysis of the BRCA2 9254delATCAT recurrent mutation in breast/ovarian cancer families from Spain". Hum. Mutat. 21 (4): 452. doi:10.1002/humu.9133. PMID 12655574. S2CID 34333797.
- ^ Bergman A, Einbeigi Z, Olofsson U, Taib Z, Wallgren A, Karlsson P, Wahlström J, Martinsson T, Nordling M (October 2001). "The western Swedish BRCA1 founder mutation 3171ins5; a 3.7 cM conserved haplotype of today is a reminiscence of a 1500-year-old mutation". Eur. J. Hum. Genet. 9 (10): 787–93. doi:10.1038/sj.ejhg.5200704. PMID 11781691.
- ^ Hansen KR, Knowlton NS, Thyer AC, Charleston JS, Soules MR, Klein NA (March 2008). "A new model of reproductive aging: the decline in ovarian non-growing follicle number from birth to menopause". Hum. Reprod. 23 (3): 699–708. doi:10.1093/humrep/dem408. PMID 18192670.
- ^ Hassold T, Hunt P (December 2009). "Maternal age and chromosomally abnormal pregnancies: what we know and what we wish we knew". Current Opinion in Pediatrics. 21 (6): 703–8. doi:10.1097/MOP.0b013e328332c6ab. PMC 2894811. PMID 19881348.
- ^ a b Oktay K, Kim JY, Barad D, Babayev SN (January 2010). "Association of BRCA1 mutations with occult primary ovarian insufficiency: a possible explanation for the link between infertility and breast/ovarian cancer risks". J. Clin. Oncol. 28 (2): 240–4. doi:10.1200/JCO.2009.24.2057. PMC 3040011. PMID 19996028.
- ^ Rzepka-Górska I, Tarnowski B, Chudecka-Głaz A, Górski B, Zielińska D, Tołoczko-Grabarek A (November 2006). "Premature menopause in patients with BRCA1 gene mutation". Breast Cancer Res. Treat. 100 (1): 59–63. doi:10.1007/s10549-006-9220-1. PMID 16773440. S2CID 19572648.
- ^ a b Titus S, Li F, Stobezki R, Akula K, Unsal E, Jeong K, Dickler M, Robson M, Moy F, Goswami S, Oktay K (February 2013). "Impairment of BRCA1-related DNA double-strand break repair leads to ovarian aging in mice and humans". Sci Transl Med. 5 (172): 172ra21. doi:10.1126/scitranslmed.3004925. PMC 5130338. PMID 23408054.
- ^ a b Taron M, Rosell R, Felip E, Mendez P, Souglakos J, Ronco MS, Queralt C, Majo J, Sanchez JM, Sanchez JJ, Maestre J (October 2004). "BRCA1 mRNA expression levels as an indicator of chemoresistance in lung cancer". Hum. Mol. Genet. 13 (20): 2443–9. doi:10.1093/hmg/ddh260. PMID 15317748.
- ^ a b Papadaki C, Sfakianaki M, Ioannidis G, Lagoudaki E, Trypaki M, Tryfonidis K, Mavroudis D, Stathopoulos E, Georgoulias V, Souglakos J (April 2012). "ERCC1 and BRAC1 mRNA expression levels in the primary tumor could predict the effectiveness of the second-line cisplatin-based chemotherapy in pretreated patients with metastatic non-small cell lung cancer". J Thorac Oncol. 7 (4): 663–71. doi:10.1097/JTO.0b013e318244bdd4. PMID 22425915.
- ^ Weberpals J, Garbuio K, O'Brien A, Clark-Knowles K, Doucette S, Antoniouk O, Goss G, Dimitroulakos J (February 2009). "The DNA repair proteins BRCA1 and ERCC1 as predictive markers in sporadic ovarian cancer". Int. J. Cancer. 124 (4): 806–15. doi:10.1002/ijc.23987. PMID 19035454. S2CID 13357407.
- ^ "ACLU sues over patents on breast cancer genes". CNN. Archived from the original on 15 May 2009. Retrieved 2009-05-14.
- ^ Robert Cook-Deegan, MD et al (2010) 유전자 특허 및 라이선스 관행이 유전암에 대한 유전학적 테스트 접근에 미치는 영향: 유방암 및 난소암과 대장암 비교: 유방암, 난소 및 대장암 검사 특허 및 허가 유전자 의학.12(4 Supl):S15~S38.
- ^ Benowitz S (January 2003). "European groups oppose Myriad's latest patent on BRCA1". J. Natl. Cancer Inst. 95 (1): 8–9. doi:10.1093/jnci/95.1.8. PMID 12509391.
- ^ Conley J, Vorhous D, Cook-Deegan J (2011-03-01). "How Will Myriad Respond to the Next Generation of BRCA Testing?". Robinson, Bradshaw, and Hinson. Retrieved 2012-12-09.
- ^ "Genetics and Patenting". Human Genome Project Information. U.S. Department of Energy Genome Programs. 2010-07-07.
- ^ Liptak A (June 13, 2013). "Supreme Court Rules Human Genes May Not Be Patented". The New York Times. Retrieved June 13, 2013.
- ^ Corderoy A (February 15, 2013). "Landmark patent ruling over breast cancer gene BRCA1". Sydney Morning Herald. Retrieved June 14, 2013.
- ^ "Australian federal court rules isolated genetic material can be patented". The Guardian. 5 September 2014. Retrieved 14 September 2014.
- ^ "Patient wins high court challenge against company's cancer gene patent". The Guardian. 7 October 2015. Retrieved 6 October 2015.
- ^ Foray N, Marot D, Randrianarison V, Venezia ND, Picard D, Perricaudet M, Favaudon V, Jeggo P (June 2002). "Constitutive association of BRCA1 and c-Abl and its ATM-dependent disruption after irradiation". Mol. Cell. Biol. 22 (12): 4020–32. doi:10.1128/MCB.22.12.4020-4032.2002. PMC 133860. PMID 12024016.
- ^ Altiok S, Batt D, Altiok N, Papautsky A, Downward J, Roberts TM, Avraham H (November 1999). "Heregulin induces phosphorylation of BRCA1 through phosphatidylinositol 3-Kinase/AKT in breast cancer cells". J. Biol. Chem. 274 (45): 32274–8. doi:10.1074/jbc.274.45.32274. PMID 10542266.
- ^ Xiang T, Ohashi A, Huang Y, Pandita TK, Ludwig T, Powell SN, Yang Q (December 2008). "Negative Regulation of AKT Activation by BRCA1". Cancer Res. 68 (24): 10040–4. doi:10.1158/0008-5472.CAN-08-3009. PMC 2605656. PMID 19074868.
- ^ Yeh S, Hu YC, Rahman M, Lin HK, Hsu CL, Ting HJ, Kang HY, Chang C (October 2000). "Increase of androgen-induced cell death and androgen receptor transactivation by BRCA1 in prostate cancer cells". Proc. Natl. Acad. Sci. U.S.A. 97 (21): 11256–61. Bibcode:2000PNAS...9711256Y. doi:10.1073/pnas.190353897. PMC 17187. PMID 11016951.
- ^ a b Kim ST, Lim DS, Canman CE, Kastan MB (December 1999). "Substrate specificities and identification of putative substrates of ATM kinase family members". J. Biol. Chem. 274 (53): 37538–43. doi:10.1074/jbc.274.53.37538. PMID 10608806.
- ^ a b Tibbetts RS, Cortez D, Brumbaugh KM, Scully R, Livingston D, Elledge SJ, Abraham RT (December 2000). "Functional interactions between BRCA1 and the checkpoint kinase ATR during genotoxic stress". Genes Dev. 14 (23): 2989–3002. doi:10.1101/gad.851000. PMC 317107. PMID 11114888.
- ^ a b Chen J (September 2000). "Ataxia telangiectasia-related protein is involved in the phosphorylation of BRCA1 following deoxyribonucleic acid damage". Cancer Res. 60 (18): 5037–9. PMID 11016625.
- ^ a b Gatei M, Zhou BB, Hobson K, Scott S, Young D, Khanna KK (May 2001). "Ataxia telangiectasia mutated (ATM) kinase and ATM and Rad3 related kinase mediate phosphorylation of Brca1 at distinct and overlapping sites. In vivo assessment using phospho-specific antibodies". J. Biol. Chem. 276 (20): 17276–80. doi:10.1074/jbc.M011681200. PMID 11278964.
- ^ Gatei M, Scott SP, Filippovitch I, Soronika N, Lavin MF, Weber B, Khanna KK (June 2000). "Role for ATM in DNA damage-induced phosphorylation of BRCA1". Cancer Res. 60 (12): 3299–304. PMID 10866324.
- ^ Cortez D, Wang Y, Qin J, Elledge SJ (November 1999). "Requirement of ATM-dependent phosphorylation of brca1 in the DNA damage response to double-strand breaks". Science. 286 (5442): 1162–6. doi:10.1126/science.286.5442.1162. PMID 10550055.
- ^ Houvras Y, Benezra M, Zhang H, Manfredi JJ, Weber BL, Licht JD (November 2000). "BRCA1 physically and functionally interacts with ATF1". J. Biol. Chem. 275 (46): 36230–7. doi:10.1074/jbc.M002539200. PMID 10945975.
- ^ a b Cantor SB, Bell DW, Ganesan S, Kass EM, Drapkin R, Grossman S, Wahrer DC, Sgroi DC, Lane WS, Haber DA, Livingston DM (April 2001). "BACH1, a novel helicase-like protein, interacts directly with BRCA1 and contributes to its DNA repair function". Cell. 105 (1): 149–60. doi:10.1016/S0092-8674(01)00304-X. PMID 11301010.
- ^ a b c d e f Dong Y, Hakimi MA, Chen X, Kumaraswamy E, Cooch NS, Godwin AK, Shiekhattar R (November 2003). "Regulation of BRCC, a holoenzyme complex containing BRCA1 and BRCA2, by a signalosome-like subunit and its role in DNA repair". Mol. Cell. 12 (5): 1087–99. doi:10.1016/S1097-2765(03)00424-6. PMID 14636569.
- ^ a b Chen J, Silver DP, Walpita D, Cantor SB, Gazdar AF, Tomlinson G, Couch FJ, Weber BL, Ashley T, Livingston DM, Scully R (September 1998). "Stable interaction between the products of the BRCA1 and BRCA2 tumor suppressor genes in mitotic and meiotic cells". Mol. Cell. 2 (3): 317–28. doi:10.1016/S1097-2765(00)80276-2. PMID 9774970.
- ^ a b Reuter TY, Medhurst AL, Waisfisz Q, Zhi Y, Herterich S, Hoehn H, Gross HJ, Joenje H, Hoatlin ME, Mathew CG, Huber PA (October 2003). "Yeast two-hybrid screens imply involvement of Fanconi anemia proteins in transcription regulation, cell signaling, oxidative metabolism, and cellular transport". Exp. Cell Res. 289 (2): 211–21. doi:10.1016/S0014-4827(03)00261-1. PMID 14499622.
- ^ Sarkisian CJ, Master SR, Huber LJ, Ha SI, Chodosh LA (October 2001). "Analysis of murine Brca2 reveals conservation of protein–protein interactions but differences in nuclear localization signals". J. Biol. Chem. 276 (40): 37640–8. doi:10.1074/jbc.M106281200. PMID 11477095.
- ^ a b c d Rodriguez M, Yu X, Chen J, Songyang Z (December 2003). "Phosphopeptide binding specificities of BRCA1 COOH-terminal (BRCT) domains". J. Biol. Chem. 278 (52): 52914–8. doi:10.1074/jbc.C300407200. PMID 14578343.
- ^ a b c d Wada O, Oishi H, Takada I, Yanagisawa J, Yano T, Kato S (August 2004). "BRCA1 function mediates a TRAP/DRIP complex through direct interaction with TRAP220". Oncogene. 23 (35): 6000–5. doi:10.1038/sj.onc.1207786. PMID 15208681.
- ^ Botuyan MV, Nominé Y, Yu X, Juranic N, Macura S, Chen J, Mer G (July 2004). "Structural basis of BACH1 phosphopeptide recognition by BRCA1 tandem BRCT domains". Structure. 12 (7): 1137–46. doi:10.1016/j.str.2004.06.002. PMC 3652423. PMID 15242590.
- ^ Yu X, Chini CC, He M, Mer G, Chen J (October 2003). "The BRCT domain is a phospho-protein binding domain". Science. 302 (5645): 639–42. Bibcode:2003Sci...302..639Y. doi:10.1126/science.1088753. PMID 14576433. S2CID 29407635.
- ^ Clapperton JA, Manke IA, Lowery DM, Ho T, Haire LF, Yaffe MB, Smerdon SJ (June 2004). "Structure and mechanism of BRCA1 BRCT domain recognition of phosphorylated BACH1 with implications for cancer". Nature Structural & Molecular Biology. 11 (6): 512–8. doi:10.1038/nsmb775. PMID 15133502. S2CID 7354915.
- ^ a b c Hu YF, Li R (June 2002). "JunB potentiates function of BRCA1 activation domain 1 (AD1) through a coiled-coil-mediated interaction". Genes Dev. 16 (12): 1509–17. doi:10.1101/gad.995502. PMC 186344. PMID 12080089.
- ^ Lee JS, Collins KM, Brown AL, Lee CH, Chung JH (March 2000). "hCds1-mediated phosphorylation of BRCA1 regulates the DNA damage response". Nature. 404 (6774): 201–4. Bibcode:2000Natur.404..201L. doi:10.1038/35004614. PMID 10724175. S2CID 4345911.
- ^ Chabalier-Taste C, Racca C, Dozier C, Larminat F (December 2008). "BRCA1 is regulated by Chk2 in response to spindle damage". Biochim. Biophys. Acta. 1783 (12): 2223–33. doi:10.1016/j.bbamcr.2008.08.006. PMID 18804494.
- ^ Lin SY, Li K, Stewart GS, Elledge SJ (April 2004). "Human Claspin works with BRCA1 to both positively and negatively regulate cell proliferation". Proc. Natl. Acad. Sci. U.S.A. 101 (17): 6484–9. Bibcode:2004PNAS..101.6484L. doi:10.1073/pnas.0401847101. PMC 404071. PMID 15096610.
- ^ Ye Q, Hu YF, Zhong H, Nye AC, Belmont AS, Li R (December 2001). "BRCA1-induced large-scale chromatin unfolding and allele-specific effects of cancer-predisposing mutations". J. Cell Biol. 155 (6): 911–21. doi:10.1083/jcb.200108049. PMC 2150890. PMID 11739404.
- ^ a b Benezra M, Chevallier N, Morrison DJ, MacLachlan TK, El-Deiry WS, Licht JD (July 2003). "BRCA1 augments transcription by the NF-kappaB transcription factor by binding to the Rel domain of the p65/RelA subunit". J. Biol. Chem. 278 (29): 26333–41. doi:10.1074/jbc.M303076200. PMID 12700228.
- ^ a b Pao GM, Janknecht R, Ruffner H, Hunter T, Verma IM (February 2000). "CBP/p300 interact with and function as transcriptional coactivators of BRCA1". Proc. Natl. Acad. Sci. U.S.A. 97 (3): 1020–5. Bibcode:2000PNAS...97.1020P. doi:10.1073/pnas.97.3.1020. PMC 15508. PMID 10655477.
- ^ a b Chai YL, Cui J, Shao N, Shyam E, Reddy P, Rao VN (January 1999). "The second BRCT domain of BRCA1 proteins interacts with p53 and stimulates transcription from the p21WAF1/CIP1 promoter". Oncogene. 18 (1): 263–8. doi:10.1038/sj.onc.1202323. PMID 9926942.
- ^ a b c Fan S, Ma YX, Wang C, Yuan RQ, Meng Q, Wang JA, Erdos M, Goldberg ID, Webb P, Kushner PJ, Pestell RG, Rosen EM (January 2002). "p300 Modulates the BRCA1 inhibition of estrogen receptor activity". Cancer Res. 62 (1): 141–51. PMID 11782371.
- ^ Neish AS, Anderson SF, Schlegel BP, Wei W, Parvin JD (February 1998). "Factors associated with the mammalian RNA polymerase II holoenzyme". Nucleic Acids Res. 26 (3): 847–53. doi:10.1093/nar/26.3.847. PMC 147327. PMID 9443979.
- ^ O'Brien KA, Lemke SJ, Cocke KS, Rao RN, Beckmann RP (July 1999). "Casein kinase 2 binds to and phosphorylates BRCA1". Biochem. Biophys. Res. Commun. 260 (3): 658–64. doi:10.1006/bbrc.1999.0892. PMID 10403822.
- ^ Kleiman FE, Manley JL (March 2001). "The BARD1-CstF-50 interaction links mRNA 3' end formation to DNA damage and tumor suppression". Cell. 104 (5): 743–53. doi:10.1016/S0092-8674(01)00270-7. PMID 11257228.
- ^ Kleiman FE, Manley JL (September 1999). "Functional interaction of BRCA1-associated BARD1 with polyadenylation factor CstF-50". Science. 285 (5433): 1576–9. doi:10.1126/science.285.5433.1576. PMID 10477523.
- ^ Wang H, Shao N, Ding QM, Cui J, Reddy ES, Rao VN (Jul 1997). "BRCA1 proteins are transported to the nucleus in the absence of serum and splice variants BRCA1a, BRCA1b are tyrosine phosphoproteins that associate with E2F, cyclins and cyclin dependent kinases". Oncogene. 15 (2): 143–57. doi:10.1038/sj.onc.1201252. PMID 9244350.
- ^ Chen Y, Farmer AA, Chen CF, Jones DC, Chen PL, Lee WH (July 1996). "BRCA1 is a 220-kDa nuclear phosphoprotein that is expressed and phosphorylated in a cell cycle-dependent manner". Cancer Res. 56 (14): 3168–72. PMID 8764100.
- ^ Ruffner H, Jiang W, Craig AG, Hunter T, Verma IM (July 1999). "BRCA1 is phosphorylated at serine 1497 in vivo at a cyclin-dependent kinase 2 phosphorylation site". Mol. Cell. Biol. 19 (7): 4843–54. doi:10.1128/MCB.19.7.4843. PMC 84283. PMID 10373534.
- ^ Schlegel BP, Starita LM, Parvin JD (February 2003). "Overexpression of a protein fragment of RNA helicase A causes inhibition of endogenous BRCA1 function and defects in ploidy and cytokinesis in mammary epithelial cells". Oncogene. 22 (7): 983–91. doi:10.1038/sj.onc.1206195. PMID 12592385.
- ^ Anderson SF, Schlegel BP, Nakajima T, Wolpin ES, Parvin JD (July 1998). "BRCA1 protein is linked to the RNA polymerase II holoenzyme complex via RNA helicase A". Nat. Genet. 19 (3): 254–6. doi:10.1038/930. PMID 9662397. S2CID 10953768.
- ^ Chai Y, Chipitsyna G, Cui J, Liao B, Liu S, Aysola K, Yezdani M, Reddy ES, Rao VN (March 2001). "c-Fos oncogene regulator Elk-1 interacts with BRCA1 splice variants BRCA1a/1b and enhances BRCA1a/1b-mediated growth suppression in breast cancer cells". Oncogene. 20 (11): 1357–67. doi:10.1038/sj.onc.1204256. PMID 11313879.
- ^ Zheng L, Annab LA, Afshari CA, Lee WH, Boyer TG (August 2001). "BRCA1 mediates ligand-independent transcriptional repression of the estrogen receptor". Proc. Natl. Acad. Sci. U.S.A. 98 (17): 9587–92. Bibcode:2001PNAS...98.9587Z. doi:10.1073/pnas.171174298. PMC 55496. PMID 11493692.
- ^ Fan S, Ma YX, Wang C, Yuan RQ, Meng Q, Wang JA, Erdos M, Goldberg ID, Webb P, Kushner PJ, Pestell RG, Rosen EM (January 2001). "Role of direct interaction in BRCA1 inhibition of estrogen receptor activity". Oncogene. 20 (1): 77–87. doi:10.1038/sj.onc.1204073. PMID 11244506.
- ^ Kawai H, Li H, Chun P, Avraham S, Avraham HK (October 2002). "Direct interaction between BRCA1 and the estrogen receptor regulates vascular endothelial growth factor (VEGF) transcription and secretion in breast cancer cells". Oncogene. 21 (50): 7730–9. doi:10.1038/sj.onc.1205971. PMID 12400015.
- ^ Folias A, Matkovic M, Bruun D, Reid S, Hejna J, Grompe M, D'Andrea A, Moses R (October 2002). "BRCA1 interacts directly with the Fanconi anemia protein FANCA". Hum. Mol. Genet. 11 (21): 2591–7. doi:10.1093/hmg/11.21.2591. PMID 12354784.
- ^ a b Vandenberg CJ, Gergely F, Ong CY, Pace P, Mallery DL, Hiom K, Patel KJ (July 2003). "BRCA1-independent ubiquitination of FANCD2". Mol. Cell. 12 (1): 247–54. doi:10.1016/S1097-2765(03)00281-8. PMID 12887909.
- ^ Yan J, Zhu J, Zhong H, Lu Q, Huang C, Ye Q (October 2003). "BRCA1 interacts with FHL2 and enhances FHL2 transactivation function". FEBS Lett. 553 (1–2): 183–9. doi:10.1016/S0014-5793(03)00978-5. PMID 14550570. S2CID 31566004.
- ^ Yan JH, Ye QN, Zhu JH, Zhong HJ, Zheng HY, Huang CF (December 2003). "[Isolation and characterization of a BRCA1-interacting protein]". Yi Chuan Xue Bao (in Chinese). 30 (12): 1161–6. PMID 14986435.
- ^ a b Mallery DL, Vandenberg CJ, Hiom K (December 2002). "Activation of the E3 ligase function of the BRCA1/BARD1 complex by polyubiquitin chains". EMBO J. 21 (24): 6755–62. doi:10.1093/emboj/cdf691. PMC 139111. PMID 12485996.
- ^ a b Chen A, Kleiman FE, Manley JL, Ouchi T, Pan ZQ (June 2002). "Autoubiquitination of the BRCA1*BARD1 RING ubiquitin ligase". J. Biol. Chem. 277 (24): 22085–92. doi:10.1074/jbc.M201252200. PMID 11927591.
- ^ Paull TT, Rogakou EP, Yamazaki V, Kirchgessner CU, Gellert M, Bonner WM (2000). "A critical role for histone H2AX in recruitment of repair factors to nuclear foci after DNA damage". Curr. Biol. 10 (15): 886–95. doi:10.1016/S0960-9822(00)00610-2. PMID 10959836.
- ^ Sutherland KD, Visvader JE, Choong DY, Sum EY, Lindeman GJ, Campbell IG (October 2003). "Mutational analysis of the LMO4 gene, encoding a BRCA1-interacting protein, in breast carcinomas". Int. J. Cancer. 107 (1): 155–8. doi:10.1002/ijc.11343. PMID 12925972. S2CID 20908722.
- ^ Sum EY, Peng B, Yu X, Chen J, Byrne J, Lindeman GJ, Visvader JE (March 2002). "The LIM domain protein LMO4 interacts with the cofactor CtIP and the tumor suppressor BRCA1 and inhibits BRCA1 activity". J. Biol. Chem. 277 (10): 7849–56. doi:10.1074/jbc.M110603200. PMID 11751867.
- ^ Gilmore PM, McCabe N, Quinn JE, Kennedy RD, Gorski JJ, Andrews HN, McWilliams S, Carty M, Mullan PB, Duprex WP, Liu ET, Johnston PG, Harkin DP (June 2004). "BRCA1 interacts with and is required for paclitaxel-induced activation of mitogen-activated protein kinase kinase kinase 3". Cancer Res. 64 (12): 4148–54. doi:10.1158/0008-5472.CAN-03-4080. PMID 15205325.
- ^ a b c d e Chiba N, Parvin JD (October 2001). "Redistribution of BRCA1 among four different protein complexes following replication blockage". J. Biol. Chem. 276 (42): 38549–54. doi:10.1074/jbc.M105227200. PMID 11504724.
- ^ Chiba N, Parvin JD (August 2002). "The BRCA1 and BARD1 association with the RNA polymerase II holoenzyme". Cancer Res. 62 (15): 4222–8. PMID 12154023.
- ^ a b Scully R, Anderson SF, Chao DM, Wei W, Ye L, Young RA, Livingston DM, Parvin JD (May 1997). "BRCA1 is a component of the RNA polymerase II holoenzyme". Proc. Natl. Acad. Sci. U.S.A. 94 (11): 5605–10. Bibcode:1997PNAS...94.5605S. doi:10.1073/pnas.94.11.5605. PMC 20825. PMID 9159119.
- ^ a b c Zhong Q, Chen CF, Li S, Chen Y, Wang CC, Xiao J, Chen PL, Sharp ZD, Lee WH (July 1999). "Association of BRCA1 with the hRad50-hMre11-p95 complex and the DNA damage response". Science. 285 (5428): 747–50. doi:10.1126/science.285.5428.747. PMID 10426999.
- ^ Paull TT, Cortez D, Bowers B, Elledge SJ, Gellert M (May 2001). "Direct DNA binding by Brca1". Proc. Natl. Acad. Sci. U.S.A. 98 (11): 6086–91. doi:10.1073/pnas.111125998. PMC 33426. PMID 11353843.
- ^ a b Li H, Lee TH, Avraham H (June 2002). "A novel tricomplex of BRCA1, Nmi, and c-Myc inhibits c-Myc-induced human telomerase reverse transcriptase gene (hTERT) promoter activity in breast cancer". J. Biol. Chem. 277 (23): 20965–73. doi:10.1074/jbc.M112231200. PMID 11916966.
- ^ Xiong J, Fan S, Meng Q, Schramm L, Wang C, Bouzahza B, Zhou J, Zafonte B, Goldberg ID, Haddad BR, Pestell RG, Rosen EM (December 2003). "BRCA1 inhibition of telomerase activity in cultured cells". Mol. Cell. Biol. 23 (23): 8668–90. doi:10.1128/MCB.23.23.8668-8690.2003. PMC 262673. PMID 14612409.
- ^ Zhou C, Liu J (March 2003). "Inhibition of human telomerase reverse transcriptase gene expression by BRCA1 in human ovarian cancer cells". Biochem. Biophys. Res. Commun. 303 (1): 130–6. doi:10.1016/S0006-291X(03)00318-8. PMID 12646176.
- ^ a b Sato K, Hayami R, Wu W, Nishikawa T, Nishikawa H, Okuda Y, Ogata H, Fukuda M, Ohta T (July 2004). "Nucleophosmin/B23 is a candidate substrate for the BRCA1-BARD1 ubiquitin ligase". J. Biol. Chem. 279 (30): 30919–22. doi:10.1074/jbc.C400169200. PMID 15184379.
- ^ Park JJ, Irvine RA, Buchanan G, Koh SS, Park JM, Tilley WD, Stallcup MR, Press MF, Coetzee GA (November 2000). "Breast cancer susceptibility gene 1 (BRCAI) is a coactivator of the androgen receptor". Cancer Res. 60 (21): 5946–9. PMID 11085509.
- ^ Cabart P, Chew HK, Murphy S (July 2004). "BRCA1 cooperates with NUFIP and P-TEFb to activate transcription by RNA polymerase II". Oncogene. 23 (31): 5316–29. doi:10.1038/sj.onc.1207684. PMID 15107825.
- ^ Abramovitch S, Werner H (2003). "Functional and physical interactions between BRCA1 and p53 in transcriptional regulation of the IGF-IR gene". Horm. Metab. Res. 35 (11–12): 758–62. doi:10.1055/s-2004-814154. PMID 14710355.
- ^ Ouchi T, Monteiro AN, August A, Aaronson SA, Hanafusa H (March 1998). "BRCA1 regulates p53-dependent gene expression". Proc. Natl. Acad. Sci. U.S.A. 95 (5): 2302–6. Bibcode:1998PNAS...95.2302O. doi:10.1073/pnas.95.5.2302. PMC 19327. PMID 9482880.
- ^ Zhang H, Somasundaram K, Peng Y, Tian H, Zhang H, Bi D, Weber BL, El-Deiry WS (April 1998). "BRCA1 physically associates with p53 and stimulates its transcriptional activity". Oncogene. 16 (13): 1713–21. doi:10.1038/sj.onc.1201932. PMID 9582019.
- ^ Sy SM, Huen MS, Chen J (April 2009). "PALB2 is an integral component of the BRCA complex required for homologous recombination repair". Proc. Natl. Acad. Sci. U.S.A. 106 (17): 7155–60. Bibcode:2009PNAS..106.7155S. doi:10.1073/pnas.0811159106. PMC 2678481. PMID 19369211.
- ^ Krum SA, Miranda GA, Lin C, Lane TF (December 2003). "BRCA1 associates with processive RNA polymerase II". J. Biol. Chem. 278 (52): 52012–20. doi:10.1074/jbc.M308418200. PMID 14506230.
- ^ Krum SA, Womack JE, Lane TF (September 2003). "Bovine BRCA1 shows classic responses to genotoxic stress but low in vitro transcriptional activation activity". Oncogene. 22 (38): 6032–44. doi:10.1038/sj.onc.1206515. PMID 12955082.
- ^ Liu Y, Virshup DM, White RL, Hsu LC (November 2002). "Regulation of BRCA1 phosphorylation by interaction with protein phosphatase 1alpha". Cancer Res. 62 (22): 6357–61. PMID 12438214.
- ^ Scully R, Chen J, Plug A, Xiao Y, Weaver D, Feunteun J, Ashley T, Livingston DM (January 1997). "Association of BRCA1 with Rad51 in mitotic and meiotic cells". Cell. 88 (2): 265–75. doi:10.1016/S0092-8674(00)81847-4. PMID 9008167.
- ^ a b c Yarden RI, Brody LC (April 1999). "BRCA1 interacts with components of the histone deacetylase complex". Proc. Natl. Acad. Sci. U.S.A. 96 (9): 4983–8. Bibcode:1999PNAS...96.4983Y. doi:10.1073/pnas.96.9.4983. PMC 21803. PMID 10220405.
- ^ Chen GC, Guan LS, Yu JH, Li GC, Choi Kim HR, Wang ZY (June 2001). "Rb-associated protein 46 (RbAp46) inhibits transcriptional transactivation mediated by BRCA1". Biochem. Biophys. Res. Commun. 284 (2): 507–14. doi:10.1006/bbrc.2001.5003. PMID 11394910.
- ^ a b Yarden RI, Brody LC (2001). "Identification of proteins that interact with BRCA1 by Far-Western library screening". J. Cell. Biochem. 83 (4): 521–31. doi:10.1002/jcb.1257. PMID 11746496. S2CID 29703139.
- ^ Yu X, Wu LC, Bowcock AM, Aronheim A, Baer R (September 1998). "The C-terminal (BRCT) domains of BRCA1 interact in vivo with CtIP, a protein implicated in the CtBP pathway of transcriptional repression". J. Biol. Chem. 273 (39): 25388–92. doi:10.1074/jbc.273.39.25388. PMID 9738006.
- ^ Li S, Chen PL, Subramanian T, Chinnadurai G, Tomlinson G, Osborne CK, Sharp ZD, Lee WH (April 1999). "Binding of CtIP to the BRCT repeats of BRCA1 involved in the transcription regulation of p21 is disrupted upon DNA damage". J. Biol. Chem. 274 (16): 11334–8. doi:10.1074/jbc.274.16.11334. PMID 10196224.
- ^ Wong AK, Ormonde PA, Pero R, Chen Y, Lian L, Salada G, Berry S, Lawrence Q, Dayananth P, Ha P, Tavtigian SV, Teng DH, Bartel PL (November 1998). "Characterization of a carboxy-terminal BRCA1 interacting protein". Oncogene. 17 (18): 2279–85. doi:10.1038/sj.onc.1202150. PMID 9811458.
- ^ Li S, Ting NS, Zheng L, Chen PL, Ziv Y, Shiloh Y, Lee EY, Lee WH (July 2000). "Functional link of BRCA1 and ataxia telangiectasia gene product in DNA damage response". Nature. 406 (6792): 210–5. Bibcode:2000Natur.406..210L. doi:10.1038/35018134. PMID 10910365. S2CID 3266654.
- ^ Wu-Baer F, Baer R (November 2001). "Effect of DNA damage on a BRCA1 complex". Nature. 414 (6859): 36. doi:10.1038/35102118. PMID 11689934. S2CID 4329675.
- ^ Yu X, Baer R (June 2000). "Nuclear localization and cell cycle-specific expression of CtIP, a protein that associates with the BRCA1 tumor suppressor". J. Biol. Chem. 275 (24): 18541–9. doi:10.1074/jbc.M909494199. PMID 10764811.
- ^ a b c Fan S, Yuan R, Ma YX, Xiong J, Meng Q, Erdos M, Zhao JN, Goldberg ID, Pestell RG, Rosen EM (August 2001). "Disruption of BRCA1 LXCXE motif alters BRCA1 functional activity and regulation of RB family but not RB protein binding". Oncogene. 20 (35): 4827–41. doi:10.1038/sj.onc.1204666. PMID 11521194.
- ^ Aprelikova ON, Fang BS, Meissner EG, Cotter S, Campbell M, Kuthiala A, Bessho M, Jensen RA, Liu ET (October 1999). "BRCA1-associated growth arrest is RB-dependent". Proc. Natl. Acad. Sci. U.S.A. 96 (21): 11866–71. Bibcode:1999PNAS...9611866A. doi:10.1073/pnas.96.21.11866. PMC 18378. PMID 10518542.
- ^ a b Bochar DA, Wang L, Beniya H, Kinev A, Xue Y, Lane WS, Wang W, Kashanchi F, Shiekhattar R (July 2000). "BRCA1 is associated with a human SWI/SNF-related complex: linking chromatin remodeling to breast cancer". Cell. 102 (2): 257–65. doi:10.1016/S0092-8674(00)00030-1. PMID 10943845.
- ^ Hill DA, de la Serna IL, Veal TM, Imbalzano AN (April 2004). "BRCA1 interacts with dominant negative SWI/SNF enzymes without affecting homologous recombination or radiation-induced gene activation of p21 or Mdm2". J. Cell. Biochem. 91 (5): 987–98. doi:10.1002/jcb.20003. PMID 15034933. S2CID 40668596.
- ^ Ouchi T, Lee SW, Ouchi M, Aaronson SA, Horvath CM (May 2000). "Collaboration of signal transducer and activator of transcription 1 (STAT1) and BRCA1 in differential regulation of IFN-gamma target genes". Proc. Natl. Acad. Sci. U.S.A. 97 (10): 5208–13. Bibcode:2000PNAS...97.5208O. doi:10.1073/pnas.080469697. PMC 25807. PMID 10792030.
- ^ Brzovic PS, Keeffe JR, Nishikawa H, Miyamoto K, Fox D, Fukuda M, Ohta T, Klevit R (May 2003). "Binding and recognition in the assembly of an active BRCA1/BARD1 ubiquitin-ligase complex". Proc. Natl. Acad. Sci. U.S.A. 100 (10): 5646–51. Bibcode:2003PNAS..100.5646B. doi:10.1073/pnas.0836054100. PMC 156255. PMID 12732733.
- ^ Nishikawa H, Ooka S, Sato K, Arima K, Okamoto J, Klevit RE, Fukuda M, Ohta T (February 2004). "Mass spectrometric and mutational analyses reveal Lys-6-linked polyubiquitin chains catalyzed by BRCA1-BARD1 ubiquitin ligase". J. Biol. Chem. 279 (6): 3916–24. doi:10.1074/jbc.M308540200. PMID 14638690.
- ^ Kentsis A, Gordon RE, Borden KL (November 2002). "Control of biochemical reactions through supramolecular RING domain self-assembly". Proc. Natl. Acad. Sci. U.S.A. 99 (24): 15404–9. Bibcode:2002PNAS...9915404K. doi:10.1073/pnas.202608799. PMC 137729. PMID 12438698.
- ^ Wu-Baer F, Lagrazon K, Yuan W, Baer R (September 2003). "The BRCA1/BARD1 heterodimer assembles polyubiquitin chains through an unconventional linkage involving lysine residue K6 of ubiquitin". J. Biol. Chem. 278 (37): 34743–6. doi:10.1074/jbc.C300249200. PMID 12890688.
- ^ Hashizume R, Fukuda M, Maeda I, Nishikawa H, Oyake D, Yabuki Y, Ogata H, Ohta T (May 2001). "The RING heterodimer BRCA1-BARD1 is a ubiquitin ligase inactivated by a breast cancer-derived mutation". J. Biol. Chem. 276 (18): 14537–40. doi:10.1074/jbc.C000881200. PMID 11278247.
- ^ Cable PL, Wilson CA, Calzone FJ, Rauscher FJ, Scully R, Livingston DM, Li L, Blackwell CB, Futreal PA, Afshari CA (October 2003). "Novel consensus DNA-binding sequence for BRCA1 protein complexes". Mol. Carcinog. 38 (2): 85–96. doi:10.1002/mc.10148. PMID 14502648. S2CID 24956554.
- ^ Zhang H, Wang Q, Kajino K, Greene MI (May 2000). "VCP, a weak ATPase involved in multiple cellular events, interacts physically with BRCA1 in the nucleus of living cells". DNA Cell Biol. 19 (5): 253–63. doi:10.1089/10445490050021168. PMID 10855792.
- ^ Ganesan S, Silver DP, Drapkin R, Greenberg R, Feunteun J, Livingston DM (January 2004). "Association of BRCA1 with the inactive X chromosome and XIST RNA". Philos. Trans. R. Soc. Lond. B Biol. Sci. 359 (1441): 123–8. doi:10.1098/rstb.2003.1371. PMC 1693294. PMID 15065664.
- ^ Ganesan S, Silver DP, Greenberg RA, Avni D, Drapkin R, Miron A, Mok SC, Randrianarison V, Brodie S, Salstrom J, Rasmussen TP, Klimke A, Marrese C, Marahrens Y, Deng CX, Feunteun J, Livingston DM (November 2002). "BRCA1 supports XIST RNA concentration on the inactive X chromosome". Cell. 111 (3): 393–405. doi:10.1016/S0092-8674(02)01052-8. PMID 12419249.
- ^ Zheng L, Pan H, Li S, Flesken-Nikitin A, Chen PL, Boyer TG, Lee WH (October 2000). "Sequence-specific transcriptional corepressor function for BRCA1 through a novel zinc finger protein, ZBRK1". Mol. Cell. 6 (4): 757–68. doi:10.1016/S1097-2765(00)00075-7. PMID 11090615.