단백질 1차 구조
Protein primary structure단백질 1차 구조는 펩타이드 또는 [1]단백질에 있는 아미노산의 선형 배열이다.관례상 단백질의 1차 구조는 아미노 말단(N)에서 카르복실 말단(C)으로 보고된다.단백질 생합성은 세포의 리보솜에 의해 가장 일반적으로 수행된다.펩타이드는 또한 실험실에서 합성될 수 있다.단백질 1차 구조는 직접 배열되거나 DNA 배열로부터 추론될 수 있다.
형성
생물학적
아미노산은 펩타이드 결합을 통해 중합되어 긴 골격을 형성하며, 이를 따라 다른 아미노산 측쇄가 돌출되어 있다.생물학적 시스템에서 단백질은 세포의 리보솜에 의해 번역되는 동안 생성됩니다.일부 유기체는 종종 표준 20 이외의 아미노산을 사용하는 비리보솜 펩타이드 합성에 의해 짧은 펩타이드를 만들 수 있으며, 환화, 변형 및 가교될 수 있다.
화학의
펩타이드는 다양한 실험실 방법을 통해 화학적으로 합성될 수 있다.화학적 방법은 일반적으로 생물학적 단백질 합성(N 말단에서 시작)과 반대 순서로 펩타이드를 합성한다.
표기법
단백질 배열은 일반적으로 아미노 말단에서 시작하여 카르복실 말단까지 아미노산을 나열하는 일련의 문자로 표기됩니다.3글자 코드 또는 1글자 코드 중 하나를 사용하여 20개의 자연발생 아미노산, 혼합물 또는 애매한 아미노산(핵산 [1][2][3]표기법과 유사)을 나타낼 수 있습니다.
펩타이드는 직접 배열되거나 DNA 배열로부터 추론될 수 있다.이제 알려진 단백질 서열을 대조하는 대규모 배열 데이터베이스가 존재합니다.
| 아미노산 | 3글자[4] | 1글자[4] |
|---|---|---|
| 알라닌 | 알라 | A |
| 아르기닌 | 아르그 | R |
| 아스파라긴 | ASN | N |
| 아스파라긴산 | ASP | D |
| 시스테인 | 씨스 | C |
| 글루탐산 | 글루 | E |
| 글루타민 | 글린 | Q |
| 글리신 | 글리 | G |
| 히스티딘 | 그의 | H |
| 이소류신 | 일레 | I |
| 류신 | 류 | L |
| 리신 | 리스 | K |
| 메티오닌 | 만났다 | M |
| 페닐알라닌 | 페 | F |
| 프롤린 | 프로 | P |
| 세린 | 서 | S |
| 트레오닌 | 트레오닌 | T |
| 트립 토판 | Trp | W |
| 티로신 | 티르 | Y |
| 발린. | 발 | V |
| 기호. | 묘사 | Residues를 대표했다 |
|---|---|---|
| X | 어떤 아미노산, 알려지지 않은. | 모든. |
| B | Aspartate 또는 아스파라긴 | D, N |
| Z | 글루타 메이트 또는 글루타민 | E, Q |
| J | 류신 또는 이소류신 | 나는, L |
| Φ | 소수성 | V, I, L, F, W, M |
| Ω | 방향제 | F, W, Y, H |
| Ψ | 지방족 | V, I, L, M |
| π | 작은. | P, G, A, S |
| ζ | 친수성 | S, T, H, N, Q, E, D, K, R, Y |
| + | 정전기 충전 | K, R, H |
| - | 음전하 | D, E |
수정.
일반적으로, 폴리펩타이드는 분지가 없는 고분자이기 때문에, 그들의 1차 구조는 종종 등뼈를 따르는 아미노산의 배열에 의해 지정될 수 있다.그러나 단백질은 가장 일반적으로 디술피드 결합에 의해 가교될 수 있으며, 1차 구조도 예를 들어 단백질의 디술피드 결합에 관여하는 시스테인을 명시하는 가교 원자를 명시해야 한다.다른 교차 링크에는 데스모신이 포함됩니다.
이성질화
폴리펩타이드 사슬의 키랄 중심은 라세미화를 겪을 수 있다.염기서열을 바꾸지는 않지만, 염기서열의 화학적 특성에 영향을 미칩니다.특히 단백질에서 일반적으로 발견되는 L-아미노산은 에서 자발적으로 이성질화되어 D-아미노산을 형성할 수 있으며, 대부분의 단백질 분해효소에서는 분해할 수 없다.또한 프롤린은 펩타이드 결합에서 안정된 트랜스 이성질체를 형성할 수 있다.
번역 후 수정
마지막으로 단백질은 여기에 간략히 요약된 다양한 번역 후 변형을 겪을 수 있다.
폴리펩타이드의 N 말단 아미노기는 예를 들어 다음과 같이 공유적으로 변형될 수 있다.
- 아세틸화
- N 말단 아미노기의 양전하는 아세틸기로 바꿈으로써 제거될 수 있다(N 말단 차단).
- 포르밀화
- 보통 번역 후에 발견되는 N 말단 메티오닌은 포밀기에 의해 차단된 N 말단을 가지고 있다.이 포르밀기(그리고 때로는 글리 또는 Ser가 이어지는 경우 메티오닌 잔기 자체)는 효소 변형 효소에 의해 제거된다.
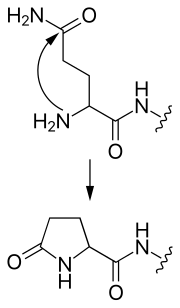
- 파이로글루탐산염
- N 말단 글루타민은 자신을 공격하여 고리형 파이로글루탐산기를 형성할 수 있다.
- 미리스토일화
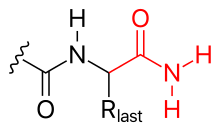
폴리펩타이드의 C 말단 카르본산염기도 다음과 같이 변형될 수 있다.
- 아미노메이션(그림 참조)
- 또한 C 말단은 아민에 의해 차단될 수 있습니다(따라서 음전하를 중화시킬 수 있습니다.
- 글리코실포스파티딜이노시톨(GPI) 부착체
- 글리코실 포스파티딜이노시톨(GPI)은 단백질을 세포막에 고정시키는 큰 소수성 인지질 보철기이다.그것은 에탄올아민, 잡탕, 그리고 마지막으로 포스파티딜이노시톨 지질 부분에 연결되는 아미드 결합을 통해 폴리펩타이드 C-말단에 부착됩니다.
마지막으로 펩타이드 측쇄는 예를 들어 다음과 같이 공유적으로 변형될 수 있다.
- 인산화
- 분열과는 별도로, 인산화 작용은 아마도 단백질의 가장 중요한 화학적 변형일 것이다.세린, 트레오닌 및 티로신 잔기의 측쇄 하이드록실기에 인산기를 부착하여 해당 부위에 음전하를 부가하여 부자연스러운 아미노산을 생성할 수 있다.이러한 반응은 키나아제에 의해 촉매되고, 역반응은 포스파타아제에 의해 촉매된다.인산화 티로신은 종종 단백질이 서로 결합할 수 있는 "핸들"로 사용되는 반면, Ser/Thr의 인산화물은 종종 도입된 음전하 때문에 입체구조 변화를 유도한다.Ser/Thr의 인산화 효과는 Ser/Th 잔기를 글루탐산염으로 변이시켜 시뮬레이션할 수 있다.
- 매우 일반적이고 매우 이질적인 화학 수정의 집합의 포괄적인 이름입니다.당분은 Ser/Thr의 측쇄수산화기 또는 Asn의 측쇄아미드기에 부착할 수 있다.이러한 첨부 파일은 용해성의 향상에서 복잡한 인식까지 다양한 기능을 제공할 수 있습니다.모든 글리코실화는 튜니카마이신과 같은 특정 억제제에 의해 차단될 수 있다.
- 탈아미드화(아미드 생성)
- 이 변형에서 아스파라긴 또는 아스파르트산 측쇄는 다음 펩타이드 결합을 공격하여 대칭적인 숙시니미드 중간체를 형성한다.중간체의 가수분해는 아스파르트산 또는 β-아미노산, 이소(Asp)를 생성한다.아스파라긴의 경우, 어느 하나의 생성물이 아미드기를 잃게 되고, 따라서 "탈아미드화"된다.
- 프롤린 잔류물은 리신과 마찬가지로 두 원자 중 하나에서 수산기산염일 수 있다.히드록시프롤린은 콜라겐의 중요한 성분으로, 콜라겐이 손실되면 불안정해집니다.히드록실화 반응은 아스코르브산(비타민 C)을 필요로 하는 효소에 의해 촉매되며, 결핍은 괴혈병과 같은 많은 결합 조직 질환으로 이어진다.
- 몇몇 단백질 잔류물은 메틸화 될 수 있으며, 특히 리신과 아르기닌의 양성기가 대표적이다.아르기닌 잔기는 핵산 인산염 골격과 상호작용하며, 일반적으로 단백질에서 염기 잔류물, 특히 구아닌과 수소 결합을 형성한다.DNA 콤플렉스리신 잔기는 단독, 이중, 심지어 3중 메틸화 될 수 있다.그러나 메틸화는 사이드 체인의 양전하를 변화시키지 않습니다.
- 리신 아미노기의 아세틸화는 화학적으로 N말단의 아세틸화와 유사하다.그러나 기능적으로 리신 잔기의 아세틸화는 핵산에 대한 단백질의 결합을 조절하기 위해 사용된다.리신에서 양전하가 제거되면 (음전하) 핵산의 정전 흡인력이 약해집니다.
- 황화
- 티로신은 에서 황산염될 수 있다.다소 특이하게, 이 변형이 내소형성 망막이 아닌 골지 기구에서 일어난다.인산화 티로신과 유사하게, 황산 티로신은 세포 표면의 케모카인 수용체 등에서 특정 인식을 위해 사용된다.인산화와 마찬가지로 황산화도 이전에 중성이었던 부위에 음전하를 더한다.
- 및 팔미토일화-C ( )- ( C ) - 3( \ \ { - C ( =O ) - \ (_ {2 ) { } - _ {3
- 소수성 이소프렌(예: 파르네실, 게라닐 및 게라닐제라닐기) 및 팔미토일기는 시스테인 잔기의 S(\ 에 첨가하여 단백질을 세포막에 고정할 수 있다.GPI 및 미리토일 앵커와 달리 이들 그룹은 반드시 흰자리에서 추가되는 것은 아닙니다.
- 카르복실화
- 글루탐산염 측쇄에 여분의 카르본산기(및 이중 음전하)를 첨가하여 Gla 잔기를 생성하는 비교적 드문 변성.이것은 칼슘과 같은 "경질" 금속 이온에 대한 결합을 강화하기 위해 사용됩니다.
- ADP 리보실화
- 큰 ADP-리보실 그룹은 단백질 내의 여러 유형의 곁사슬에 이종 효과와 함께 전달될 수 있다.이 수정은 Vibrio cholerae, Corynebacterium diphtheriae 및 Bordetella 백일해와 같은 이질적인 박테리아의 강력한 독소를 대상으로 합니다.
- 다양한 전장 접힌 단백질은 C-termini에서 다른 단백질의 리신의 측쇄 암모늄 그룹에 부착될 수 있습니다.유비퀴틴은 이것들 중 가장 흔하며, 보통 유비퀴틴 태그가 있는 단백질이 분해되어야 한다는 신호를 보냅니다.
위에 열거된 폴리펩타이드 변형의 대부분은 번역 후, 즉 단백질이 리보솜에서 합성된 후, 전형적으로 진핵세포의 아세포 기관인 내질망막에서 발생한다.
많은 다른 화학 반응들(예: 시아닐화)은 생물학 시스템에서 발견되지 않지만 화학자들에 의해 단백질에 적용되어 왔다.
절단 및 결찰
위에 열거된 것 외에 1차 구조의 가장 중요한 변형은 펩타이드 분해(화학적 가수분해 또는 단백질 분해 효소에 의한)이다.단백질은 종종 비활성 전구체 형태로 합성된다. 전형적으로, N 말단 또는 C 말단 세그먼트는 단백질의 활성 부위를 차단하여 그 기능을 억제한다.단백질은 억제성 펩타이드를 분리함으로써 활성화된다.
어떤 단백질들은 심지어 스스로를 쪼개는 힘도 가지고 있다.일반적으로 세린의 수산기(희소하지만, 트레오닌) 또는 시스테인 잔기의 티올기는 앞의 펩타이드 결합의 카르보닐 탄소를 공격하여 사중 결합 중간체[히드록시옥사졸리딘(Ser/Thr) 또는 히드록시티아졸리딘(Cys) 중간체로 분류됨]를 형성한다.아미드 형태는 일반적으로 자유 에너지에 의해 선호되기 때문에(아마도 펩타이드 그룹의 강한 공진 안정화에 의해) 이 중간체는 공격 그룹을 배제하면서 아미드 형태로 되돌아가는 경향이 있다.그러나 추가적인 분자 상호작용은 아미드 형태를 덜 안정되게 만들 수 있습니다. 아미노기는 대신 배출되어 펩타이드 결합 대신 에스테르 결합(Ser/Thr) 또는 티오에스테르 결합(Cys)이 생성됩니다.이 화학 반응을 N-O 아실 이동이라고 합니다.
에스테르/티오에스테르 결합은 여러 가지 방법으로 해결할 수 있습니다.
- 단순 가수분해는 폴리펩타이드 사슬을 분열시켜 치환된 아미노기가 새로운 N 말단이 됩니다.이것은 글리코실라스파라긴아제의 성숙에서 나타난다.
- β 제거 반응도 사슬을 분할하지만 새로운 N 말단에서 피루보일기를 생성한다.이 피루보일기는 일부 효소, 특히 피루보일기의 전자 인출력을 이용하는 S-아데노실메티오닌 탈카르복실화효소(SAMDC)에서 공유 결합 촉매 보조 인자로 사용될 수 있다.
- 분지형 폴리펩타이드를 생성하는 분자내 에스테르 교환.내부에서는 새로운 에스테르 결합이 곧 C 말단 아스파라긴의 분자 내 공격에 의해 파괴된다.
- 헤지호그 단백질 자동 처리에서 볼 수 있듯이 분자간 에스테르 변환은 한 폴리펩타이드에서 다른 폴리펩타이드로 전체 세그먼트를 이동할 수 있습니다.
시퀀스 압축
아미노산 배열의 압축은 비교적 어려운 작업이다.기존의 특수 아미노산 배열 압축기는 주로 데이터의 특성 때문에 DNA 배열 압축기에 비해 낮은 편이다.예를 들어, 역방향 정보 손실(아미노산에서 DNA 배열로) 때문에 역방향 모델링이 더 어렵습니다.현재 높은 압축을 제공하는 무손실 데이터 압축기는 AC2입니다.[5]AC2는 Neural Networks를 사용하여 다양한 컨텍스트 모델을 혼합하고 산술 인코딩을 사용하여 데이터를 인코딩합니다.
역사
단백질이 α-아미노산의 선형 사슬이라는 제안은 1902년 칼스바드에서 열린 제74회 독일 과학자와 의사 협회의 같은 회의에서 두 과학자에 의해 거의 동시에 이루어졌다.Franz Hofmeister는 단백질의 비우레 반응을 관찰한 것을 바탕으로 아침에 이 제안을 했다.몇 시간 후 호프마이스터는 펩타이드 결합 모델을 뒷받침하는 풍부한 화학적 세부사항을 축적한 에밀 피셔에 의해 따라왔다.완전성을 위해, 단백질에 아미드 결합이 포함되어 있다는 제안은 1882년 프랑스 화학자 E에 의해 이미 이루어졌다.그리모.[6]
이러한 데이터와 나중에 단백질 분해로 소화된 단백질이 올리고펩타이드만을 산출했다는 증거에도 불구하고, 단백질이 아미노산의 선형적이고 가지 없는 중합체라는 생각은 즉시 받아들여지지 않았다.윌리엄 애스트버리와 같은 존경받는 과학자들은 공유 결합이 그렇게 긴 분자들을 함께 유지할 수 있을 만큼 충분히 강하다는 것을 의심했다; 그들은 열 교반으로 인해 그렇게 긴 분자들이 흔들릴 것을 우려했다.헤르만 슈타우딩거도 1920년대에 고무가 [6]고분자로 이루어져 있다고 주장하면서 비슷한 편견에 직면했다.
따라서, 몇 가지 대안 가설이 생겨났다.콜로이드 단백질 가설은 단백질이 더 작은 분자의 콜로이드 집합체라고 말했다.이 가설은 1920년대에 단백질이 잘 정의되고 재현 가능한 분자량을 가지고 있다는 것을 보여주는 테오도르 스베드베르크의 초원심화 측정과 단백질이 단일 분자임을 나타내는 아르네 티젤리우스의 전기영동 측정으로 반증되었다.도로시 윈치가 제시한 두 번째 가설인 사이클론 가설은 선형 폴리펩타이드가 골격 아미드 그룹을 가교하는 화학적 사이클론 재배치 C=O + HN (\ C(OH)-N을 거치면서 2차원 직물을 형성한다는 것을 제안했다.단백질의 다른 1차 구조는 1942년 에밀 압데르할덴의 디케토피페라진 모델과 트로엔고르의 피롤/피페리딘 모델과 같은 다양한 연구자들에 의해 제안되었다.많은 신빙성을 부여한 적은 없지만, 프레드릭 생어가 인슐린을 성공적으로[when?] 배열하고 Max Perutz와 John[when?] Kendrew에 의한 미오글로빈과 헤모글로빈의 결정학적 측정에 의해 이러한 대체 모델은 마침내 반증되었다.
다른 분자의 1차 구조
어떤 선형 사슬 헤테로폴리머도 단백질에 대한 용어의 사용에 유추하여 "1차 구조"를 가지고 있다고 말할 수 있지만, 단백질에 관한 극히 일반적인 용도와 비교하면 이 용도는 드물다.또한 광범위한 2차 구조를 가진 RNA에서, 염기의 선형 사슬은 일반적으로 DNA에서와 같이 "서열"로 언급된다.다당류와 같은 다른 생물학적 고분자도 사용이 표준적이지 않지만 1차 구조를 가진 것으로 간주될 수 있다.
2차 및 3차 구조와의 관계
생물학적 고분자의 1차 구조는 대부분 3차원 형태(teriary structure)를 결정한다.단백질 배열을 사용하여 2차 구조의 세그먼트 또는 막 통과 영역과 같은 국소적 특징을 예측할 수 있습니다.그러나 단백질 접힘의 복잡성은 현재 배열만으로 단백질의 3차 구조를 예측하는 것을 금지하고 있다.유사한 호몰로지 배열의 구조를 아는 것(예를 들어 동일한 단백질 계열의 구성원)은 호몰로지 모델링에 의한 3차 구조의 매우 정확한 예측을 가능하게 한다.만약 전장 단백질 배열을 이용할 수 있다면, 등전점과 같은 일반적인 생물물리학적 특성을 추정할 수 있다.
시퀀스 패밀리는 종종 시퀀스 클러스터링에 의해 결정되며, 구조 게놈 프로젝트는 가능한 비장 시퀀스의 시퀀스 공간을 커버하는 일련의 대표 구조를 생성하는 것을 목표로 한다.
「 」를 참조해 주세요.
주 및 참고 자료
- ^ a b SANGER F (1952). "The arrangement of amino acids in proteins". In M.L. Anson; Kenneth Bailey; John T. Edsall (eds.). Advances in Protein Chemistry. Vol. 7. pp. 1–67. doi:10.1016/S0065-3233(08)60017-0. ISBN 9780120342075. PMID 14933251.
- ^ Aasland, Rein; Abrams, Charles; Ampe, Christophe; Ball, Linda J.; Bedford, Mark T.; Cesareni, Gianni; Gimona, Mario; Hurley, James H.; Jarchau, Thomas (2002-02-20). "Normalization of nomenclature for peptide motifs as ligands of modular protein domains". FEBS Letters. 513 (1): 141–144. doi:10.1016/S0014-5793(01)03295-1. ISSN 1873-3468.
- ^ Aasland R, Abrams C, Ampe C, Ball LJ, Bedford MT, Cesareni G, Gimona M, Hurley JH, Jarchau T, Lehto VP, Lemmon MA, Linding R, Mayer BJ, Nagai M, Sudol M, Walter U, Winder SJ (1968-07-01). "A One-Letter Notation for Amino Acid Sequences*". European Journal of Biochemistry. 5 (2): 151–153. doi:10.1111/j.1432-1033.1968.tb00350.x. ISSN 1432-1033. PMID 11911894.
- ^ a b Hausman, Robert E.; Cooper, Geoffrey M. (2004). The cell: a molecular approach. Washington, D.C: ASM Press. p. 51. ISBN 978-0-87893-214-6.
- ^ Silva M, Pratas D, Pinho AJ (April 2021). "AC2: An Efficient Protein Sequence Compression Tool Using Artificial Neural Networks and Cache-Hash Models". Entropy. 23: 530. doi:10.3390/e23050530. PMC 8146440.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ a b Fruton JS (May 1979). "Early theories of protein structure". Ann. N. Y. Acad. Sci. 325: xiv, 1–18. doi:10.1111/j.1749-6632.1979.tb14125.x. PMID 378063.

 자발적으로 이성질화되어 D-아미노산을 형성할 수 있으며, 대부분의
자발적으로 이성질화되어 D-아미노산을 형성할 수 있으며, 대부분의 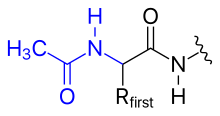



 황산염될 수 있다.다소 특이하게, 이 변형이
황산염될 수 있다.다소 특이하게, 이 변형이  첨가하여 단백질을
첨가하여 단백질을 


