BRAF(제네)
BRAF (gene)BRAF는 B-Rraf라는 단백질을 암호화하는 인간 유전자다. 이 유전자는 프로토온코인 B-Rraf, v-Rraf murine sarcin virgin virgin homological B로도 불리며, 단백질은 세린/스레오닌-단백질 키나아제 B-Rraf로 더 공식적으로 알려져 있다.[5][6]
B-Rraf 단백질은 세포 성장을 지시하는 세포 내부의 신호를 보내는 것에 관여한다. 2002년에는 일부 인간암에서 변이된 것으로 나타났다.[7]
어떤 다른 유전적 BRAF 돌연변이는 선천적 결함을 유발한다.
BRAF 돌연변이가 몰고 온 암을 치료하는 약이 개발됐다. 이 중 두 가지 약인 베무라페니브와[8] 다브라페니브는 FDA로부터 말기 흑색종 치료를 승인받았다. 베무라페니브는 파편 기반 약물 발견에서 나온 최초의 승인된 약물이었다.[9]
함수

B-Rraf는 Raf kinase 계열의 성장 신호 전달 단백질 키나제 소속이다. 이 단백질은 세포분열, 분화, 분비에 영향을 미치는 MAP키나아제/ERK 신호 경로를 조절하는 역할을 한다.[10]
구조
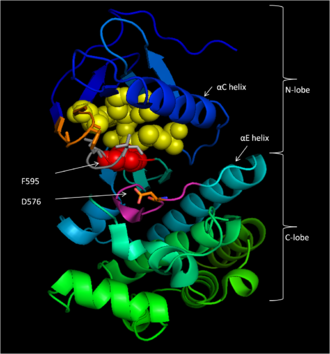
B-Rraf는 766-아미노산, 규제 신호 전달 세린/트레오닌 특이 단백질 키나아제다. 대체로 말하면 3단축 도메인이 Raf 인산화 효소 가족의 특징:보전 지역 1(CR1), Ras-GTP-binding[11]의 자율적 도메인,(CR2),, 지역 3(CR3), 단백질 substr에 대한 공통 배열 phosphorylates 촉매 단백질 인산화 효소 도메인 conserved 지역 2는serine-rich 경첩 영역 conserved으로 구성되어 있다.에서es.[12] 그것의 능동적 순응에서, B-Rraf는 그것의 키나아제 영역의 수소 결합과 정전기적 상호작용을 통해 조광기를 형성한다.[13]
CR1
보존 지역 1은 B-Rraf의 키나아제 도메인(CR3)을 자동 금지하여 B-Rraf 신호가 구성보다는 규제되도록 한다.[12] 잔류물 155–227은[14] Ras-GTP의 이펙터 도메인에 바인딩되어 CR1을 해제하고 키나제 억제를 중지하는 RBD(Ras-binding Domain)를 구성한다. 잔류물 234–280은 포볼 에스테르/DAG 결합 아연 핑거 모티브로 구성되며, Ras-binding 후 B-Rraf 막 도킹에 참여한다.[14][15]
CR2
보존 지역 2(CR2)는 CR1과 CR3를 연결하고 힌지 역할을 하는 유연한 링커를 제공한다.[citation needed]
CR3
잔류물 457–717인 보존 지역 3(CR3)은 B-Rraf의 효소 키나아제 도메인을 구성한다.[14] 주로 보존된 이[16] 구조물은 짧은 힌지 영역으로 연결된 바이로발이다.[17] 더 작은 N-로브(리소스 457–530)는 ATP 결합을 주로 담당하는 반면, 더 큰 C-로브(리소스 535–717)는 기질 단백질을 결합한다.[16] 활성 부위는 두 로브 사이의 갈라진 틈이며, 촉매 Asp576 잔여물은 이 갈라진 틈의 내부를 향하면서 C-로브에 위치한다.[14][16]
하위 영역
P-루프
B-Rraf(리소스 464–471)의 P-루프는 효소 ATP 바인딩 중 ATP의 비전달 인산염 그룹을 안정화시킨다. 구체적으로는 S467, F468, G469 백본은 수소 본드를 ATP의 β-인산염에 아미드화하여 분자를 고정시킨다. 행크스와 헌터가 분석한 PKA의 호몰로지(homology)를 B-Rraf kinase 도메인으로 분석해 B-Rap 기능 모티브를 파악했다.[16]
뉴클레오티드 바인딩 포켓
V471, C532, W531, T529, L514, A481은 ATP 바인딩 시 반 데르발스 명소를 통해 ATP의 아데닌이 고정되는 소수성 포켓을 형성한다.[16][18]
촉매 루프
잔류물 574–581은 ATP의 osph-인산염의 B-Rraf의 단백질 기질에의 이전을 지원하는 책임이 있는 키나제 영역의 한 부분을 구성한다. 특히 D576은 기질세린이나 트레오닌 잔류물에 있는 핵포질 히드록실 산소를 활성화하는 양성자 수용체 역할을 해 염기투석(base-catalism)에 의해 인산염 전달반응이 일어날 수 있다.[16]
DFG 모티브
D594, F595 및 G596은 B-Rraf의 기능이 비활성 상태와 활성 상태 모두에서 중심인 모티브를 구성한다. 비활성 상태에서는 F595가 뉴클레오티드 바인딩 포켓을 차지하여 ATP의 진입을 금지하고 효소 카탈루션 가능성을 감소시킨다.[13][18][19] 활성 상태에서 D594는 ATP의 β-와 γ-인산염 그룹을 안정화하는 이분 마그네슘 계통을 첼로 처리하여 γ-인산염의 전달 방향을 정한다.[16]
활성화 루프
잔류물 596–600은 키나제의 비활성 순응에서 P-루프와 강한 친수성 교호작용을 형성하여 활성화 루프가 인산염화될 때까지 키나제를 비활성 상태로 잠가 음전하의 존재와 이러한 교호작용을 불안정하게 한다. 이것은 키나제의 활성 상태로의 이동을 촉발한다. 구체적으로 활성화 루프의 L597과 V600은 P-루프의 G466, F468 및 V471과 상호 작용하여 키나제 도메인을 인산화될 때까지 비활성 상태로 유지한다.[17]
효소
B-Rraf는 세린/트레오닌 특이 단백질 키나아제다. 이와 같이 ATP에 의한 표적 단백질에 대한 컨센서스 시퀀스로 세린과 트레오닌 잔류물의 인산화 촉매를 촉진하여 ADP와 인산화 단백질을 생산물로 산출한다.[16] 규제가 심한 신호전달키나아제인 만큼 B-Rraf는 효소로 활동하기 전에 먼저 Ras-GTP를 묶어야 한다.[15] B-Rraf가 활성화되면 보존 단백질 키나아제 촉매핵심인산화효소(Core phosphyll)는 바이몰리 핵분자 치환을 통해 ATP의 γ-인산소 그룹에 활성 기질 세린 또는 트레오닌 히드록실 산소 원자의 핵포질 공격을 촉진하여 단백질 기판을 기판화시킨다.[16][20][21][22]
활성화
CR1 자동 표시 해제
인간 Raf 키나제의 키나제(CR3) 영역은 두 가지 메커니즘에 의해 억제된다: 자체 규제 Ras-GTP 바인딩 CR1 영역에 의한 자동 억제와 CR2 힌지 영역에 있는 키 세린 및 티로신 잔여물(C-Rraf의 경우 S338 및 Y341)의 변환 후 인산화 결여. B-Rraf 활성화 동안 단백질의 자동 억제 CR1 도메인은 먼저 Ras-GTP의 이펙터 도메인을 CR1 Ras-binding 도메인(RBD)에 바인딩하여 인간 Raf kinase 계열의 다른 구성원들처럼 키나제 CR3 도메인을 해제한다. CR1-Ras 상호작용은 나중에 CR1의 CRD(cysteine-rich subdomain)와 Ras 및 막인산인산화물의 결합을 통해 강화된다.[12] CR1을 완전히 방출하기 전에 히드록실 함유 CR2 잔류물에 인광 도금해야 하는 A-Rraf와 달리 B-Rraf는 CR2 S445에 구성 인광 도금된다.[23] 이를 통해 음전하 인산세린은 규제 영역이 결합되지 않은 후 스테릭과 정전기적 상호작용을 통해 CR1을 즉시 격퇴할 수 있어 CR3 키나제 영역이 기질 단백질과 상호작용을 할 수 있다.
CR3 도메인 활성화
자동 억제 CR1 규제 도메인이 공개된 후 B-Rraf의 CR3 키나제 도메인은 단백질 인산화 촉매를 촉진하기 전에 ATP 바인딩 활성 컨포머로 변경해야 한다. 비활성 순응에서 DFG 모티브의 F595는 소수성 아데닌 결합 주머니를 차단하고 활성화 루프 잔류물은 P-루프와 소수성 상호작용을 형성하여 ATP가 결합 부지에 접근하지 못하게 한다. 활성화 루프가 인산화되면 P-루프의 소수성 환경에서 인산염의 음전하가 불안정해진다. 결과적으로 활성화 루프는 순응을 변경하여 키나세 도메인의 C-로브에 걸쳐 확장된다. 이 과정에서 β6 스트랜드와의 안정화 β-시트 상호작용을 형성한다. 한편 인산화 잔여물은 K507에 접근하여 안정화 염교를 형성하여 활성화 루프를 제자리에 고정시킨다. DFG 모티브는 활성화 루프에 따른 순응을 변화시켜 F595가 아데닌 뉴클레오티드 결합 부위에서 벗어나 αC와 αE 헬리코스에 접하는 소수성 포켓으로 이동하게 한다. DFG와 인산화 시 활성화 루프 움직임이 함께 ATP 바인딩 사이트를 연다. 다른 모든 기질 결합 및 촉매 도메인은 이미 제자리에 있으므로, 활성화 루프의 인산화만으로도 달리 준비된 활성 사이트의 뚜껑을 본질적으로 제거하는 연쇄 반응을 통해 B-Rraf의 키나아제 도메인을 활성화한다.[17]
카탈루션
ADP를 이탈 그룹으로 하는 세린과 트레오닌 잔류물의 양분자 치환으로 단백질 인산화를 효과적으로 촉진하려면 B-Rraf는 먼저 ATP를 결합한 다음 ATP의 γ-인산염이 전달됨에 따라 전환 상태를 안정화해야 한다.[16]
ATP 바인딩
B-Rraf는 아데닌 뉴클레오티드를 비극 포켓(노란색, 그림 1)에 고정시키고 수소결합과 인산염 그룹과의 정전기적 상호작용을 통해 분자의 방향을 정하여 ATP를 결합한다. 위에서 설명한 P-루프 및 DFG 모티브 인산염 바인딩 외에도 K483과 E501은 비전달 인산염 그룹을 안정화시키는 데 핵심적인 역할을 한다. K483의 1차 아민에 대한 양전하를 통해 ATP가 결합할 때 ATP α- 및 β-인산염 그룹의 음전하를 안정시킬 수 있다. ATP가 없을 때 E501 카복실 그룹의 음전하가 이 전하의 균형을 맞춘다.[16][17]
인산화
ATP가 B-Rraf kinase 영역에 결합되면 촉매 루프의 D576은 기질 히드록실 그룹을 활성화하여 인산화 반응을 키네틱하게 구동하도록 핵소독성을 증가시키고 다른 촉매 루프 잔류물은 전환 상태를 안정시킨다.(그림 2) N581은 ATP와 관련된 분자 마그네슘 양이온을 킬레이트화하여 최적의 치환을 위해 분자의 방향을 정하는 것을 돕는다. K578은 ATP의 when-인산염 그룹에 대한 음전하를 중화시켜 인산염을 공격할 때 활성화된 세르/thr 기질 잔류물이 전자 전자-전자 반발력을 그만큼 경험하지 않도록 한다. 인산염 그룹이 전달된 후, ADP와 새로운 인산염이 분비된다.[16]
억제제
구성성 활성 B-Rraf 돌연변이는 세포의 성장을 과도하게 지시하여 암을 유발하기 때문에(임상 중요도 참조), 키나제 영역의 비활성 및 활성 순응 모두에 대해 암 치료 후보로서 B-Rraf의 억제제가 개발되었다.[17][18][19]
소라페니브

Bay43-9006(Sorafenib, Nexavar)은 1차 간암과 신장암 치료를 위해 FDA가 승인한 V600E 돌연변이 B-Rraf 및 C-Rraf 억제제다. Bay43-9006은 효소를 비활성 형태로 잠가 B-Rraf kinase 도메인을 비활성화한다. 억제제는 키나제 영역에 대한 고선호도를 통해 ATP 바인딩 포켓을 차단함으로써 이를 달성한다. 그런 다음 키 활성화 루프와 DFG 모티브 잔여물을 결합하여 활성화 루프와 DFG 모티브가 활성 순응으로 이동하지 않도록 한다. 마지막으로, 트라이플루오로메틸 페닐 모이티(trifluoromethyl penyl moiety)가 DFG 모티브와 활성화 루프 활성 순응 사이트를 강직하게 차단하여 키나제 도메인이 순응을 이동하는 것이 불가능하게 한다.[17]
W531, F583 및 F595와 상호 작용하는 키나제 N-로브의 소수성 뉴클레오티드 바인딩 포켓에 있는 BAY43-9006 앵커의 원위 피리딜 링. 촉매 루프 F583 및 DFG 모티브 F595와 소수성 상호작용은 이러한 구조의 비활성 준수를 안정화시켜 효소 활성화 가능성을 감소시킨다. 중심 페닐 링과 K483, L514 및 T529의 추가적인 소수성 상호작용은 억제제에 대한 키나제 영역의 친화력을 증가시킨다. F595와 중앙 링의 소수성 상호작용은 DFG 순응 스위치의 에너지 선호도를 더욱 떨어뜨린다. 마지막으로, 키나제 영역과 Bay43-9006의 극성 상호작용은 억제제에 대한 효소 친화력을 증가시키고 비활성 순응에서 DFG 잔류물을 안정화하는 이 추세를 지속한다. E501과 C532 수소는 각각 억제제의 요소와 피리딜 그룹을 결합하는 반면 요소 카보닐은 D594의 백본 아미드 질소로부터 수소 결합을 받아들여 DFG 모티브를 제자리에 잠근다.[17]
3fluoromethyl penyl moiety는 키나제 영역이 프로테이의 능동적 순응에서 DFG 모티브와 활성화 루프가 위치 이동 시 상주하는 αC와 αE 나선 사이의 친수성 포켓을 강체적으로 차단하여 비활성 순응의 열역학적 호감도를 억제한다.n.[17]
베무라페니브
PLX4032(Vemurafenib)는 후기 흑색종 치료를 위해 FDA가 승인한 V600 돌연변이 B-Rraf 억제제다.[13] 키나제 도메인의 비활성 형태를 억제하는 BAY43-9006과 달리 베무라페니브는 ATP 바인딩 사이트에 단단히 고정하면서 [18][19]키나제의 활성 "DFG-in" 형태를 억제한다. 베무라페니브는 키나아제의 활성 형태만 억제함으로써 비규제 B-Rap, 보통 암을 유발하는 세포로 세포의 증식을 선택적으로 억제한다.
베무라페니브는 약동학상의 이유로 첨가된 페닐 링에서 전구체인 PLX4720과 다를 뿐이므로 PLX4720의 작용 방식은 베무라페니브와 동등하다.[19] PLX4720은 질소 원자가 탄소로 대체된 두 곳의 부지를 점유하는 천연 아데닌과 7-아제인 자전거인 앵커 영역이 다를 뿐이어서 부분적으로 ATP 결합 부지에 대한 친화력이 좋다. 이를 통해 C532에 대한 N7 수소 본딩과 Q530에 대한 N1 수소 본딩과 같은 강력한 분자간 상호작용이 보존될 수 있다. ATP 바인딩 소수성 포켓(C532, W531, T529, L514, A481)에 잘 맞으면 바인딩 친화력도 높아진다. 물에 대한 케톤 링커 수소 본딩과 두 번째 소수성 포켓(A481, V482, K483, V471, I527, T529, L514, F583)의 디플루오로-페닐 적합성은 전반적으로 결합 친화력이 매우 높은 데 기여한다. 능동형 Raf에 대한 선택적 결합은 αC 나선형의 이동에 의해 생성된 Raf 선택형 포켓에 결합하는 단자 프로필 그룹에 의해 이루어진다. 활성 상태에서 D594의 백본 펩타이드 NH와 수소 결합하여 안정화된 pH 민감 감응성 디프로톤화 술폰아미드 그룹에 의해 키나아제의 활성 순응 선택성이 더욱 증가한다. 비활성 상태에서는 억제제의 술폰아미드 그룹이 대신 그 잔여물의 백본 카보닐과 상호작용을 하여 거부반응을 일으킨다. 따라서 베무라페니브는 B-Rraf's kinase 도메인의 활성 상태에 우선적으로 결합한다.[18][19]
임상적 유의성
BRAF 유전자의 돌연변이는 두 가지 방법으로 질병을 일으킬 수 있다. 첫째, 돌연변이는 유전될 수 있고 선천적인 결함을 유발할 수 있다. 둘째, 돌연변이는 나중에 나타나 종양 유전자로서 암을 유발할 수 있다.
이 유전자의 유전적 돌연변이는 심장결함, 정신지체, 그리고 독특한 얼굴 생김새로 특징지어지는 병인 심근경색증후군을 유발한다.[24]
이 유전자의 돌연변이는 비호지킨림프종, 대장암, 악성 흑색종, 유두갑상선암, 비소세포폐암, 폐의 아데노카시노카르시종, 교모세포종, 플랑모피성혈소판트로사이토마를 포함한 뇌종양뿐만 아니라 에르드하임-체스터병과 같은 염증성 질환에서도 발견되었다.[10]
BRAF 유전자의 V600E 돌연변이는 수많은 연구에서 털이 많은 세포 백혈병과 연관되어 왔고 불필요한 MLH1 염기서열을 겪는 환자의 수를 줄이기 위해 린치 증후군 검사에 사용할 것을 제안했다.[25][26]
돌연변이
인간 암과 관련된 BRAF 유전자의 30개 이상의 돌연변이가 확인되었다. BRAF 돌연변이의 빈도는 멜라노마와 네비의 80% 이상에서 폐암의 경우 1~3%, 대장암의 경우 5% 등 다른 종양의 경우 0~18% 미만으로 매우 다양하다.[27] 그 사례의 90%에서 티민은 뉴클레오티드 1799에서 아데닌으로 대체된다. 이는 발린(V)이 인간 암에서 발견된 활성화 부분에서 600번 코돈(현재의 V600E)에서 글루탐산염(E)으로 대체되는 결과를 초래한다.[28] 이 돌연변이는 유두 갑상선암, 대장암, 흑색종, 비소세포 폐암에서 광범위하게 관찰되었다.[29][30][31][32][33][34][35] BRAF-V600E 돌연변이는 Langerhans 세포 조직증 환자의 57%에 존재한다.[36] V600E 돌연변이는 털이 많은 세포 백혈병 환자의 100%에서 발생할 가능성이 있는 운전자 돌연변이다.[37] BRAF V600E 돌연변이가 양성이지만 국소적으로 침투하는 오도독성 신엽종에서 고주파수가 검출됐다.[38] V600E 돌연변이는 단일 운전자 돌연변이(유전자 '흡연건')로서 유두정맥류 발병의 특정 사례에 연계될 수도 있다.[39]
Other mutations which have been found are R461I, I462S, G463E, G463V, G465A, G465E, G465V, G468A, G468E, N580S, E585K, D593V, F594L, G595R, L596V, T598I, V599D, V599E, V599K, V599R, V600K, A727V, etc. and most of these mutations are clustered to two regions: the glycine-rich P loop of the N lobe and the activation segment and flanking regions.[17] 이러한 돌연변이는 활성화 세그먼트를 비활성 상태에서 활성 상태로 변경하는데, 예를 들어 앞의 인용 논문에서 Val599의 편협한 사이드 체인이 Ph467의 페닐 링과 상호작용을 한다고 보고되었다. 중간 크기의 소수성 Val 사이드 체인을 인간 암(Glu, Asp, Lys 또는 Arg)에서 발견된 것처럼 더 크고 충전된 잔류물로 교체하면 DFG 모티브를 비활성 순응으로 유지하는 상호작용이 불안정해져 활성화 세그먼트가 활성 위치로 전환될 것으로 예상된다. 돌연변이의 유형에 따라 MEK에 대한 키나아제 활동도 달라질 수 있다. 대부분의 돌연변이들은 MEK를 향한 강화된 B-Rraf kinase 활동을 자극한다. 그러나 소수의 돌연변이들은 MEK에 대한 활동이 줄어들기는 하지만 야생형 C-RAF를 활성화하는 순응을 채택하기 때문에 다른 메커니즘을 통해 행동하며, 이는 ERK에 신호를 보낸다.
BRAF-V600E
- BRAF V600E는 단백질 억제제에 대한 민감도의 결정 요인이다. PLX4720에 의한 BRAF-V600E 봉쇄로 BRAF-mutant 대장암 세포의 카필조미브에 대한 민감도가 역전되었기 때문에 프로테아솜 억제제에 대한 취약성은 지속적인 BRAF 신호에 의존한다. 프로테아솜 억제는 BRAF V600E 뮤탄트 대장암에서 중요한 표적 전략을 나타낼 수 있다.[40]
BRAF 억제제
위에서 언급했듯이, 일부 제약회사들은 BRAF가 잘 이해되고 높은 수율의 목표물이기 때문에 항암제 사용을 위한 돌연변이 B-raf 단백질의 특정한 억제제를 개발하고 있다.[18][41] 베무라페니브(RG7204 또는 PLX4032)는 2011년 8월 미국 식품의약국(FDA)으로부터 3단계 임상 데이터를 바탕으로 전이성 흑색종 치료를 위한 젤보라프(Zelboraf)로 면허를 받았다. 기존 최고 화학요법 치료제인 다카바진 7-12%에 비해 53%의 치료 응답률과 함께 생존율도 개선된 것으로 나타났다.[42] 임상시험에서 B-Rraf는 전이성 흑색종 환자의 생존 가능성을 높였다. 이 약의 높은 효능에도 불구하고 종양의 20%는 여전히 치료에 대한 내성이 생긴다. 생쥐의 경우 종양의 20%가 56일 후에 내성이 생긴다.[43] 이러한 저항의 메커니즘은 여전히 논쟁의 여지가 있지만, 일부 가설에는 고농도의 베무라페니브와[43] 성장 신호의 업스트림 업스트림 조절을 보상하기 위한 B-Rraf의 과도한 억제가 포함된다.[44]
보다 일반적인 B-Rraf 억제제로는 GDC-0879, PLX-4720, 소라페니브, 다브라페니브, LGX818 등이 있다.
상호작용
BRAF(gene)는 다음과 상호작용하는 것으로 나타났다.
참조
- ^ a b c GRCh38: 앙상블 릴리스 89: ENSG00000157764 - 앙상블, 2017년 5월
- ^ a b c GRCm38: 앙상블 릴리스 89: ENSMUSG00000002413 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ Sithanandam G, Kolch W, Duh FM, Rapp UR (December 1990). "Complete coding sequence of a human B-raf cDNA and detection of B-raf protein kinase with isozyme specific antibodies". Oncogene. 5 (12): 1775–80. PMID 2284096.
- ^ Sithanandam G, Druck T, Cannizzaro LA, Leuzzi G, Huebner K, Rapp UR (April 1992). "B-raf and a B-raf pseudogene are located on 7q in man". Oncogene. 7 (4): 795–9. PMID 1565476.
- ^ Davies H, Bignell GR, Cox C, Stephens P, Edkins S, Clegg S, et al. (June 2002). "Mutations of the BRAF gene in human cancer" (PDF). Nature. 417 (6892): 949–54. Bibcode:2002Natur.417..949D. doi:10.1038/nature00766. PMID 12068308. S2CID 3071547.
- ^ "FDA Approves Zelboraf (Vemurafenib) and Companion Diagnostic for BRAF Mutation-Positive Metastatic Melanoma, a Deadly Form of Skin Cancer" (Press release). Genentech. Retrieved 2011-08-17.
- ^ Erlanson DA, Fesik SW, Hubbard RE, Jahnke W, Jhoti H (September 2016). "Twenty years on: the impact of fragments on drug discovery". Nature Reviews. Drug Discovery. 15 (9): 605–619. doi:10.1038/nrd.2016.109. PMID 27417849. S2CID 19634793.
- ^ a b "Entrez Gene: BRAF".
- ^ Daum G, Eisenmann-Tappe I, Fries HW, Troppmair J, Rapp UR (November 1994). "The ins and outs of Raf kinases". Trends in Biochemical Sciences. 19 (11): 474–80. doi:10.1016/0968-0004(94)90133-3. PMID 7855890.
- ^ a b c Cutler RE, Stephens RM, Saracino MR, Morrison DK (August 1998). "Autoregulation of the Raf-1 serine/threonine kinase". Proceedings of the National Academy of Sciences of the United States of America. 95 (16): 9214–9. Bibcode:1998PNAS...95.9214C. doi:10.1073/pnas.95.16.9214. PMC 21318. PMID 9689060.
- ^ a b c Bollag G, Tsai J, Zhang J, Zhang C, Ibrahim P, Nolop K, Hirth P (November 2012). "Vemurafenib: the first drug approved for BRAF-mutant cancer". Nature Reviews. Drug Discovery. 11 (11): 873–86. doi:10.1038/nrd3847. PMID 23060265. S2CID 9337155.
- ^ a b c d "Serine/threonine protein kinase B-rAF". Retrieved 4 Mar 2013.
- ^ a b Morrison DK, Cutler RE (April 1997). "The complexity of Raf-1 regulation". Current Opinion in Cell Biology. 9 (2): 174–9. doi:10.1016/S0955-0674(97)80060-9. PMID 9069260.
- ^ a b c d e f g h i j k l Hanks SK, Hunter T (May 1995). "Protein kinases 6. The eukaryotic protein kinase superfamily: kinase (catalytic) domain structure and classification". FASEB Journal. 9 (8): 576–96. doi:10.1096/fasebj.9.8.7768349. PMID 7768349. S2CID 21377422.
- ^ a b c d e f g h i Wan PT, Garnett MJ, Roe SM, Lee S, Niculescu-Duvaz D, Good VM, et al. (March 2004). Cancer Genome Project. "Mechanism of activation of the RAF-ERK signaling pathway by oncogenic mutations of B-RAF". Cell. 116 (6): 855–67. doi:10.1016/S0092-8674(04)00215-6. PMID 15035987. S2CID 126161.
- ^ a b c d e f Tsai J, Lee JT, Wang W, Zhang J, Cho H, Mamo S, et al. (February 2008). "Discovery of a selective inhibitor of oncogenic B-Raf kinase with potent antimelanoma activity". Proceedings of the National Academy of Sciences of the United States of America. 105 (8): 3041–6. Bibcode:2008PNAS..105.3041T. doi:10.1073/pnas.0711741105. PMC 2268581. PMID 18287029.
- ^ a b c d e Bollag G, Hirth P, Tsai J, Zhang J, Ibrahim PN, Cho H, et al. (September 2010). "Clinical efficacy of a RAF inhibitor needs broad target blockade in BRAF-mutant melanoma". Nature. 467 (7315): 596–9. Bibcode:2010Natur.467..596B. doi:10.1038/nature09454. PMC 2948082. PMID 20823850.
- ^ Hanks SK, Quinn AM, Hunter T (July 1988). "The protein kinase family: conserved features and deduced phylogeny of the catalytic domains". Science. 241 (4861): 42–52. Bibcode:1988Sci...241...42H. doi:10.1126/science.3291115. PMID 3291115.
- ^ Hanks SK (June 1991). "Eukaryotic protein kinases". Curr. Opin. Struct. Biol. 1 (3): 369–383. doi:10.1016/0959-440X(91)90035-R.
- ^ Hanks SK, Quinn AM (1991). "[2] Protein kinase catalytic domain sequence database: Identification of conserved features of primary structure and classification of family members". Protein kinase catalytic domain sequence database: identification of conserved features of primary structure and classification of family members. Methods in Enzymology. Vol. 200. pp. 38–62. doi:10.1016/0076-6879(91)00126-H. ISBN 9780121821012. PMID 1956325.
- ^ Mason CS, Springer CJ, Cooper RG, Superti-Furga G, Marshall CJ, Marais R (April 1999). "Serine and tyrosine phosphorylations cooperate in Raf-1, but not B-Raf activation". The EMBO Journal. 18 (8): 2137–48. doi:10.1093/emboj/18.8.2137. PMC 1171298. PMID 10205168.
- ^ Roberts A, Allanson J, Jadico SK, Kavamura MI, Noonan J, Opitz JM, et al. (November 2006). "The cardiofaciocutaneous syndrome". Journal of Medical Genetics. 43 (11): 833–42. doi:10.1136/jmg.2006.042796. PMC 2563180. PMID 16825433.
- ^ Ewalt M, Nandula S, Phillips A, Alobeid B, Murty VV, Mansukhani MM, Bhagat G (December 2012). "Real-time PCR-based analysis of BRAF V600E mutation in low and intermediate grade lymphomas confirms frequent occurrence in hairy cell leukaemia". Hematological Oncology. 30 (4): 190–3. doi:10.1002/hon.1023. PMID 22246856. S2CID 204843221.
- ^ Palomaki GE, McClain MR, Melillo S, Hampel HL, Thibodeau SN (January 2009). "EGAPP supplementary evidence review: DNA testing strategies aimed at reducing morbidity and mortality from Lynch syndrome". Genetics in Medicine. 11 (1): 42–65. doi:10.1097/GIM.0b013e31818fa2db. PMC 2743613. PMID 19125127.
- ^ Namba H, Nakashima M, Hayashi T, Hayashida N, Maeda S, Rogounovitch TI, et al. (September 2003). "Clinical implication of hot spot BRAF mutation, V599E, in papillary thyroid cancers". The Journal of Clinical Endocrinology and Metabolism. 88 (9): 4393–7. doi:10.1210/jc.2003-030305. PMID 12970315.
- ^ Tan YH, Liu Y, Eu KW, Ang PW, Li WQ, Salto-Tellez M, et al. (April 2008). "Detection of BRAF V600E mutation by pyrosequencing". Pathology. 40 (3): 295–8. doi:10.1080/00313020801911512. PMID 18428050. S2CID 32051681.
- ^ Li WQ, Kawakami K, Ruszkiewicz A, Bennett G, Moore J, Iacopetta B (January 2006). "BRAF mutations are associated with distinctive clinical, pathological and molecular features of colorectal cancer independently of microsatellite instability status". Molecular Cancer. 5 (1): 2. doi:10.1186/1476-4598-5-2. PMC 1360090. PMID 16403224.
- ^ Benlloch S, Payá A, Alenda C, Bessa X, Andreu M, Jover R, et al. (November 2006). "Detection of BRAF V600E mutation in colorectal cancer: comparison of automatic sequencing and real-time chemistry methodology". The Journal of Molecular Diagnostics. 8 (5): 540–3. doi:10.2353/jmoldx.2006.060070. PMC 1876165. PMID 17065421.
- ^ Deng G, Bell I, Crawley S, Gum J, Terdiman JP, Allen BA, et al. (January 2004). "BRAF mutation is frequently present in sporadic colorectal cancer with methylated hMLH1, but not in hereditary nonpolyposis colorectal cancer". Clinical Cancer Research. 10 (1 Pt 1): 191–5. doi:10.1158/1078-0432.CCR-1118-3. PMID 14734469.
- ^ Gear H, Williams H, Kemp EG, Roberts F (August 2004). "BRAF mutations in conjunctival melanoma". Investigative Ophthalmology & Visual Science. 45 (8): 2484–8. doi:10.1167/iovs.04-0093. PMID 15277467.
- ^ Maldonado JL, Fridlyand J, Patel H, Jain AN, Busam K, Kageshita T, et al. (December 2003). "Determinants of BRAF mutations in primary melanomas". Journal of the National Cancer Institute. 95 (24): 1878–90. doi:10.1093/jnci/djg123. PMID 14679157.
- ^ Puxeddu E, Moretti S, Elisei R, Romei C, Pascucci R, Martinelli M, et al. (May 2004). "BRAF(V599E) mutation is the leading genetic event in adult sporadic papillary thyroid carcinomas". The Journal of Clinical Endocrinology and Metabolism. 89 (5): 2414–20. doi:10.1210/jc.2003-031425. PMID 15126572.
- ^ Elisei R, Ugolini C, Viola D, Lupi C, Biagini A, Giannini R, et al. (October 2008). "BRAF(V600E) mutation and outcome of patients with papillary thyroid carcinoma: a 15-year median follow-up study". The Journal of Clinical Endocrinology and Metabolism. 93 (10): 3943–9. doi:10.1210/jc.2008-0607. PMID 18682506.
- ^ Badalian-Very G, Vergilio JA, Degar BA, Rodriguez-Galindo C, Rollins BJ (January 2012). "Recent advances in the understanding of Langerhans cell histiocytosis". British Journal of Haematology. 156 (2): 163–72. doi:10.1111/j.1365-2141.2011.08915.x. PMID 22017623. S2CID 34922416.
- ^ Tiacci E, Trifonov V, Schiavoni G, Holmes A, Kern W, Martelli MP, et al. (June 2011). "BRAF mutations in hairy-cell leukemia". The New England Journal of Medicine. 364 (24): 2305–15. doi:10.1056/NEJMoa1014209. PMC 3689585. PMID 21663470. Lay summary – Science Update blog: Cancer Research UK.
{{cite journal}}: Cite는 사용되지 않는 매개 변수를 사용한다.lay-url=(도움말) - ^ Kurppa KJ, Catón J, Morgan PR, Ristimäki A, Ruhin B, Kellokoski J, et al. (April 2014). "High frequency of BRAF V600E mutations in ameloblastoma". The Journal of Pathology. 232 (5): 492–8. doi:10.1002/path.4317. PMC 4255689. PMID 24374844.
- ^ Brastianos PK, Taylor-Weiner A, Manley PE, Jones RT, Dias-Santagata D, Thorner AR, et al. (February 2014). "Exome sequencing identifies BRAF mutations in papillary craniopharyngiomas". Nature Genetics. 46 (2): 161–5. doi:10.1038/ng.2868. PMC 3982316. PMID 24413733. Lay summary – Broad Institute.
{{cite journal}}: Cite는 사용되지 않는 매개 변수를 사용한다.lay-url=(도움말) - ^ Zecchin D, Boscaro V, Medico E, Barault L, Martini M, Arena S, et al. (December 2013). "BRAF V600E is a determinant of sensitivity to proteasome inhibitors" (PDF). Molecular Cancer Therapeutics. 12 (12): 2950–61. doi:10.1158/1535-7163.MCT-13-0243. PMID 24107445. S2CID 17012966.
- ^ King AJ, Patrick DR, Batorsky RS, Ho ML, Do HT, Zhang SY, et al. (December 2006). "Demonstration of a genetic therapeutic index for tumors expressing oncogenic BRAF by the kinase inhibitor SB-590885". Cancer Research. 66 (23): 11100–5. doi:10.1158/0008-5472.CAN-06-2554. PMID 17145850.
- ^ Chapman PB, Hauschild A, Robert C, Haanen JB, Ascierto P, Larkin J, et al. (June 2011). BRIM-3 Study Group. "Improved survival with vemurafenib in melanoma with BRAF V600E mutation". The New England Journal of Medicine. 364 (26): 2507–16. doi:10.1056/NEJMoa1103782. PMC 3549296. PMID 21639808.
- ^ a b Das Thakur M, Salangsang F, Landman AS, Sellers WR, Pryer NK, Levesque MP, et al. (February 2013). "Modelling vemurafenib resistance in melanoma reveals a strategy to forestall drug resistance". Nature. 494 (7436): 251–5. Bibcode:2013Natur.494..251D. doi:10.1038/nature11814. PMC 3930354. PMID 23302800.
- ^ Nazarian R, Shi H, Wang Q, Kong X, Koya RC, Lee H, et al. (December 2010). "Melanomas acquire resistance to B-RAF(V600E) inhibition by RTK or N-RAS upregulation". Nature. 468 (7326): 973–7. Bibcode:2010Natur.468..973N. doi:10.1038/nature09626. PMC 3143360. PMID 21107323.
- ^ Guan KL, Figueroa C, Brtva TR, Zhu T, Taylor J, Barber TD, Vojtek AB (September 2000). "Negative regulation of the serine/threonine kinase B-Raf by Akt". The Journal of Biological Chemistry. 275 (35): 27354–9. doi:10.1074/jbc.M004371200. PMID 10869359.
- ^ Weber CK, Slupsky JR, Kalmes HA, Rapp UR (May 2001). "Active Ras induces heterodimerization of cRaf and BRaf". Cancer Research. 61 (9): 3595–8. PMID 11325826.
- ^ Stang S, Bottorff D, Stone JC (June 1997). "Interaction of activated Ras with Raf-1 alone may be sufficient for transformation of rat2 cells". Molecular and Cellular Biology. 17 (6): 3047–55. doi:10.1128/MCB.17.6.3047. PMC 232157. PMID 9154803.
- ^ Reuter CW, Catling AD, Jelinek T, Weber MJ (March 1995). "Biochemical analysis of MEK activation in NIH3T3 fibroblasts. Identification of B-Raf and other activators". The Journal of Biological Chemistry. 270 (13): 7644–55. doi:10.1074/jbc.270.13.7644. PMID 7706312.
- ^ Ewing RM, Chu P, Elisma F, Li H, Taylor P, Climie S, et al. (2007). "Large-scale mapping of human protein-protein interactions by mass spectrometry". Molecular Systems Biology. 3 (1): 89. doi:10.1038/msb4100134. PMC 1847948. PMID 17353931.
- ^ Qiu W, Zhuang S, von Lintig FC, Boss GR, Pilz RB (October 2000). "Cell type-specific regulation of B-Raf kinase by cAMP and 14-3-3 proteins". The Journal of Biological Chemistry. 275 (41): 31921–9. doi:10.1074/jbc.M003327200. PMID 10931830.
추가 읽기
- Garnett MJ, Marais R (October 2004). "Guilty as charged: B-RAF is a human oncogene". Cancer Cell. 6 (4): 313–9. doi:10.1016/j.ccr.2004.09.022. PMID 15488754.
- Quiros RM, Ding HG, Gattuso P, Prinz RA, Xu X (June 2005). "Evidence that one subset of anaplastic thyroid carcinomas are derived from papillary carcinomas due to BRAF and p53 mutations". Cancer. 103 (11): 2261–8. doi:10.1002/cncr.21073. PMID 15880523. S2CID 29665029.
- Karbowniczek M, Henske EP (November 2005). "The role of tuberin in cellular differentiation: are B-Raf and MAPK involved?". Annals of the New York Academy of Sciences. 1059 (1): 168–73. Bibcode:2005NYASA1059..168K. doi:10.1196/annals.1339.045. PMID 16382052. S2CID 39146204.
- Ciampi R, Nikiforov YE (March 2007). "RET/PTC rearrangements and BRAF mutations in thyroid tumorigenesis". Endocrinology. 148 (3): 936–41. doi:10.1210/en.2006-0921. PMID 16946010.
- Espinosa AV, Porchia L, Ringel MD (January 2007). "Targeting BRAF in thyroid cancer". British Journal of Cancer. 96 (1): 16–20. doi:10.1038/sj.bjc.6603520. PMC 2360215. PMID 17179987.
- Allanson JE, Roberts AE (8 August 2019). "Noonan Syndrome". In Pagon RA, Bird TD, Dolan CR, et al. (eds.). GeneReviews [Internet]. Seattle WA: University of Washington, Seattle.
- Rauen KA (3 March 2016) [18 January 2007]. "Cardiofaciocutaneous Syndrome". In Pagon RA, Bird TD, Dolan CR (eds.). GeneReviews [Internet]. Seattle WA: University of Washington, Seattle.
- Gelb BD, Tartaglia M (14 May 2015) [30 November 2007]. "LEOPARD Syndrome". In Pagon RA, Bird TD, Dolan CR (eds.). GeneReviews [Internet]. Seattle WA: University of Washington, Seattle.
외부 링크
- "BRAF gene". NCI Dictionary of Cancer Terms. Retrieved 2007-11-25.
- BRAF에서 결함 찾기 — 암을 유발하는 BRAF 돌연변이의 발견에 대한 영국 암 연구 블로그 게시물(내부 비디오)
- UCSC 게놈 브라우저의 인간 BRAF 게놈 위치 및 BRAF 유전자 세부 정보 페이지.
![]() 이 글에는 미국 국립 암 연구소(National Cancer Institute) 문서의 공용 도메인 자료가 포함되어 있다. "Dictionary of Cancer Terms".이 기사는 공공영역에 있는 미국 국립 의학 도서관의 텍스트를 통합하고 있다.
이 글에는 미국 국립 암 연구소(National Cancer Institute) 문서의 공용 도메인 자료가 포함되어 있다. "Dictionary of Cancer Terms".이 기사는 공공영역에 있는 미국 국립 의학 도서관의 텍스트를 통합하고 있다.