알파나선
Alpha helix
알파나선(α-나선)은 단백질의 2차 구조에서 일반적인 모티브이며, 모든 골격 N-H 그룹이 아미노산의 골격 C=O 그룹에 결합하고 단백질 염기서열을 따라 4개의 잔류물이 초기에 위치하는 오른손 방향 배열이다.
알파 나선은 고전적인 폴링-코리-브랜슨 알파-헬릭스라고도 불린다.3.6-나선이라는13 이름도 이러한 유형의 나선에 사용되며, 이는 수소 결합에 의해 형성된 고리에 13개의 원자가 관여하는 나선 회전당 평균 잔류물 수를 나타낸다.
단백질의 국소 구조 유형 중 α-나선은 가장 극단적이고 배열에서 가장 예측 가능하며 가장 널리 퍼져 있다.
검출
1930년대 초, 윌리엄 애스트베리는 상당한 스트레칭 시 습한 양털이나 머리카락 섬유의 X선 섬유 회절에 급격한 변화가 있음을 보여주었다.데이터에 따르면 늘어나지 않은 섬유는 5.1옹스트롬(0.51나노미터) 이상의 특성 반복을 갖는 코일 분자 구조를 가지고 있었다.
Astbury는 처음에 섬유에 대한 링크 체인 구조를 제안했다.그는 나중에 다른 연구원들(특히 미국의 화학자 모리스 허긴스)과 함께 다음과 같은 제안을 했다.
- 늘어나지 않은 단백질 분자는 나선형(그는 그것을 알파형이라고 불렀다)을 형성했다.
- 스트레칭은 나선을 풀어 확장 상태를 형성하게 했다(그는 이것을 β 형태라고 불렀다).
세부 사항은 부정확했지만, Astbury의 이러한 형태의 모델은 본질적으로 정확했고, 2차 구조의 현대 요소인 α-나선 및 β-strand(Astbury의 명명법은 유지되었다)에 대응한다. 이들은 1951년 Linus Pauling, Robert Corey 및 Herman Branson에 의해 개발되었으며, 오른쪽과 왼쪽을 모두 보여주었다.비록 1960년에 미오글로빈의[1] 결정 구조가 오른손의 형태가 일반적인 형태라는 것을 보여주었지만, ed 나선형이다.Hans Neurath는 Astbury의 모형은 [2]원자의 충돌을 수반하기 때문에 상세하게 정확할 수 없다는 것을 처음으로 보여주었다.Neurath의 논문과 Astbury의 데이터는 H. S.[3] Taylor, Maurice Huggins[4], Bragg와 공동[5] 연구자들이 현대의 α-나선과 다소 유사한 케라틴 모델을 제안하도록 영감을 주었습니다.
현대 α-나선 모델링의 두 가지 주요 발전은 아미노산과 펩타이드의 결정 구조 결정과 평면 펩타이드 결합에 대한 폴링의 예측 덕분에 정확한 결합 기하학, 그리고 나선 회전당 정수 잔기의 가정에 대한 포기였다.가장 중요한 순간은 폴링이 감기에 걸려 잠자리에 들었던 1948년 초봄이었다.그는 지루해서 종이 조각에 대략 정확한 치수의 폴리펩타이드 사슬을 그리고 그것을 나선으로 접어서 평면 펩타이드 결합을 유지하도록 주의했다.몇 번의 시도 후, 그는 물리적으로 그럴듯한 수소 결합을 가진 모형을 제작했다.폴링은 그 후 코리, 브랜슨과 함께 자신의 모델을 [6]출판하기 전에 확인했습니다.1954년 폴링은 "화학 결합의 본질에 대한 연구와 단백질과 같은 복잡한 물질의 [7]구조에 대한 설명에 대한 적용"으로 그의 첫 번째 노벨상을 받았다.
구조.
지오메트리 및 수소 결합
α-나선의 아미노산은 각 아미노산 잔기가 나선의 100° 회전(즉, 나선은 회전당 3.6개의 잔기를 가지며)과 나선 축을 따라 1.5Ω(0.15nm)의 변환에 해당하는 우측 나선 구조로 배열된다.Dunitz는[8] 이 주제에 대한 Pauling의 첫 번째 기사에서 어떻게 실제 구조의 에난티오머인 왼손잡이 나선을 보여주는지를 설명합니다.왼손잡이 나선의 짧은 조각은 때때로 많은 양의 아키랄 글리신 아미노산과 함께 발생하지만, 다른 정상적인 생물학적 L-아미노산에는 좋지 않습니다.알파나선의 피치(나선의 연속 회전 사이의 수직 거리)는 5.4Ω(0.54nm)로 1.5와 3.6의 곱이다.가장 중요한 것은 아미노산의 N-H기가 아미노산의 4잔기 C=O기와 수소 결합을 형성한다는 것이다. 이 반복적인 i + 4 → i 수소 결합은 α-아미노산의 가장 두드러진 특징이다.공식 국제 명명법은[9][10] α-헬리스를 정의하는 두 가지 방법, 즉 반복적인 β, β 비틀림 각도(아래 참조)와 피치와 수소 결합의 조합 패턴에 관한 규칙 6.3을 규정한다.α-헬리스는 여러 계산 방법을 사용하여 단백질 구조에서 식별될 수 있으며, 그 중 하나는 DSSP(Define Secondary Structure of Protein)[11]이다.
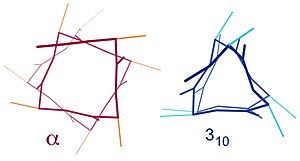
유사한 구조로는 3나선10(i + 3 → i 수소 결합)과 γ-나선(i + 5 → i 수소 결합)이 있다.α-나선은 3.6나선으로13 설명할 수 있는데, 이는 i + 4 간격은 H 결합 고리에 더 많은 원자를 추가하며, 평균적으로10 3.6아미노산이 α-나선의 한 고리에 관여하기 때문이다.첨자는 수소 [12]결합에 의해 형성된 폐쇄 루프 내의 원자 수(수소를 포함한다)를 나타냅니다.

α-헬리스크의 잔류물은 우측의 영상과 같이 (-60°, -45°) 주변의 골격(θ, θ) 이면각을 채택한다.보다 일반적인 용어로 말하면, 한 잔기의 θ이면각과 다음 잔기의 θ이면각이 약 -105°가 되도록 이면각을 채용한다.그 결과, 일반적으로 (사면 -1)의 라마찬드란 다이어그램상의 대각선상에 (-90°, -15°)부터 (-70°, -35°)까지의 α-헬리컬 이면각이 놓인다.비교를 위해 3나선의10 경우 이면각의 합계는 약 -75°이고 γ나선의 경우 약 -130°이다.트랜스 이성질체를 가진 폴리펩타이드 나선의 잔기당 회전각 δ의 일반적인 공식은 다음과[14][15] 같다.
- 3 cos δ = 1 ~42 cos ψ + ψ/2
α-나선은 촘촘히 채워져 있으며, 나선 안에는 빈 공간이 거의 없습니다.아미노산 측쇄는 나선의 바깥쪽에 있으며, 상록수의 가지처럼 대략적으로 "아래쪽으로" (즉, N-말단 쪽으로) 가리키고 있다(크리스마스 트리 효과).이 방향성은 때때로 단백질 [16]골격의 방향을 결정하기 위해 예비, 저해상도 전자 밀도 지도에서 사용됩니다.
안정성.
단백질에서 관찰되는 나선은 길이가 4에서 40개 이상일 수 있지만, 전형적인 나선은 약 10개의 아미노산(약 3회전)을 포함하고 있다.일반적으로 짧은 폴리펩타이드는 폴리펩타이드 사슬의 접힘과 관련된 엔트로피 비용이 충분한 양의 안정화 상호작용에 의해 보상되지 않기 때문에 용액 중에 α-나선 구조를 많이 나타내지 않는다.일반적으로 α-헬리체의 골격 수소 결합은 β-시트에서 발견되는 수소 결합보다 약간 약한 것으로 간주되며, 주변 물 분자에 의해 쉽게 공격된다.단, 플라즈마막과 같은 소수성 환경이나 트리플루오로에탄올(TFE)과 같은 공동용제의 존재 또는 기체상의 [17]용매로부터 격리된 경우에는 올리고펩타이드는 안정된 α-헬리컬 구조를 쉽게 채택한다.또한 나선주름을 입체구조적으로 안정시키기 위해 교차링크를 펩타이드에 포함할 수 있다.교차 링크는 전개된 상태를 엔트로피적으로 불안정하게 하고 완전한 헬리컬 [18]상태와 경쟁하는 엔탈피적으로 안정된 "디코이" 접힘을 제거함으로써 헬리컬 상태를 안정화시킨다.α-헬리케인은 [19]자연 단백질 및 인공 [20]설계 단백질에서 β-스트랜드보다 안정적이고 돌연변이에 강하며 지정이 가능한 것으로 나타났다.
시각화
올리고펩타이드 시퀀스의 알파-헬리컬 2차 구조를 시각화하는 가장 일반적인 3가지 방법은 (1) 헬리컬 [21]휠, (2) 원샹 다이어그램,[22] (3) 헬리컬 [23]네트이다.이들 각각은 다양한 소프트웨어 패키지 및 웹 서버로 시각화할 수 있습니다.적은 수의 다이어그램을 생성하려면 헬리퀘스트를[24] 헬리컬 휠에 사용하고 NetWheels를[25] 헬리컬 휠 및 헬리컬 네트에 사용할 수 있습니다.많은[26][27] 수의 다이어그램을 프로그래밍 방식으로 생성하기 위해 헬릭스비스를 사용하여 R 및 Python 프로그래밍 언어로 나선형 휠과 원샹 다이어그램을 그릴 수 있습니다.
실험적 결정
α-나선은 수소 결합과 골격 구조에 의해 정의되기 때문에 α-나선 구조의 가장 상세한 실험 증거는 오른쪽 예시와 같은 원자 분해능 X선 결정학에서 나온다.모든 골격 카르보닐 옥시겐이 아래쪽(C 말단 방향)을 가리키지만 약간 벌어지며 H 결합은 나선 축과 거의 평행하다.또한 NMR 스펙트럼 분석의 단백질 구조는 인접 나선 선회 시 원자 간 핵 오버하우저 효과(NOE) 커플링의 특성을 관찰하여 나선형을 잘 보여준다.경우에 따라 개별 수소 결합은 NMR에서 작은 스칼라 결합으로 직접 관찰될 수 있다.
일반적인 나선 구조를 할당하기 위한 몇 가지 저해상도 방법이 있다.NMR 화학 변화(특히 Cα, Cβ, C c)와 잔류 쌍극자 커플링은 종종 나선형의 특성이다.나선형의 원 UV(170~250 nm) 원형 이색성 스펙트럼도 특이하며, 약 208 및 222 nm에서 뚜렷한 이중 최소치를 나타낸다.α-나선 스펙트럼은 무작위 코일의 스펙트럼과 유사하기 때문에 적외선 분광법은 거의 사용되지 않는다(예를 들어 수소-중수소 교환에 의해 식별될 수 있다).마지막으로, 잔류물에 대한 할당은 여전히 활발한 연구 영역이지만, 저온 전자 현미경은 이제 단백질 내의 개별 α-헬리스를 식별할 수 있다.
아미노산의 긴 호모폴리머는 용해되면 종종 나선형을 형성합니다.이렇게 긴 고립된 나선형은 유전체 완화, 흐름 복굴절률 및 확산 상수 측정과 같은 다른 방법으로도 검출할 수 있다.보다 엄격한 관점에서 이러한 방법은 나선의 특징적인 프롤레이트(긴 시가 같은) 유체역학적 형태 또는 큰 쌍극자 모멘트만 감지합니다.
아미노산 성향
다른 아미노산 배열은 α-나선 구조를 형성하기 위한 성질이 다르다.메티오닌, 알라닌, 류신, 글루탐산염 및 리신 언차지드(아미노산 1글자 코드에서의 MALK)는 모두 특히 높은 나선 형성 성향을 가지고 있는 반면, 프롤린과 글리신은 낮은 나선 형성 [28]성향을 가지고 있다.프롤린은 아미드 수소 결합(아미드 수소가 없음)을 기증할 수 없기 때문에 나선을 부수거나 꼬이게 되며, 또한 옆사슬이 나선의 안쪽인 나선의 [12]축에서 약 30°의 굽힘을 강요한다.그러나 프롤린은 종종 나선의 첫 번째 잔류물로 나타나며, 구조적인 강성으로 인해 추정됩니다.다른 극단에서 글리신은 또한 나선 구조를 파괴하는 경향이 있는데, 그 이유는 글리신의 높은 배향 유연성이 상대적으로 구속된 α-나선 구조를 채택하는 데 장내적으로 비용이 많이 들기 때문이다.
표준 아미노산 알파 나선성 표
유리 에너지 변화의 추정 차이 δ(δG), 임의로 0으로 설정된 알라닌에 대해 α-나선 구성에서 잔기당 kcal/mol로 추정됩니다.수치가 높을수록(자유 에너지 변화가 더 많음) 선호도가 낮아집니다.인접 잔류물의 정체성에 따라 이러한 평균 수치에서 상당한 편차가 발생할 수 있다.
잔류물당[29] 자유 에너지 변화 차이
아미노산 3-
편지1-
편지헬리컬 패널티 kcal/표준 kJ/mol 알라닌 알라 A 0.00 0.00 아르기닌 아르그 R 0.21 0.88 아스파라긴 ASN N 0.65 2.72 아스파라긴산 ASP D 0.69 2.89 시스테인 씨스 C 0.68 2.85 글루탐산 글루 E 0.40 1.67 글루타민 글린 Q 0.39 1.63 글리신 글리 G 1.00 4.18 히스티딘 그의 H 0.61 2.55 이소류신 일레 I 0.41 1.72 류신 류 L 0.21 0.88 리신 리스 K 0.26 1.09 메티오닌 만났다 M 0.24 1.00 페닐알라닌 페 F 0.54 2.26 프롤린 프로 P 3.16 13.22 세린 서 S 0.50 2.09 트레오닌 스루 T 0.66 2.76 트립토판 Trp W 0.49 2.05 티로신 티르 Y 0.53 2.22 발린. 발 V 0.61 2.55
쌍극자 모멘트
나선축을 [30]따라 가리키는 펩타이드 결합의 카르보닐기로부터의 개별 마이크로디폴의 응집효과에 의한 전체적인 쌍극자 모멘트를 가진다.이 매크로 다이폴의 효과는 논란의 여지가 있다.α-헬리케인은 종종 음전하를 띤 그룹, 때로는 글루탐산 또는 아스파르트산 등의 아미노산 측쇄 또는 때로는 인산 이온에 의해 결합되는 N 말단에서 발생한다.일부에서는 나선 매크로디폴이 이러한 그룹과 정전적으로 상호작용한다고 간주한다.다른 사람들은 이것이 오해의 소지가 있다고 느끼며, α-리듬의 N 말단에서 자유 NH 그룹의 수소 결합 전위는 수소 결합에 의해 충족될 수 있다고 말하는 것이 더 현실적이다. 이는 또한 C=O···[31][32]H-N과 같은 국소 마이크로 다이폴 간의 상호작용 세트로 간주될 수 있다.
코일 코일
코일코일α나선은 2개 이상의 나선이 "슈퍼코일" 구조로 서로를 감싸는 매우 안정적인 형태이다.코일형 코일은 헵타드 반복으로 알려진 매우 특징적인 배열 모티브를 포함하며, 모티브는 배열에 따라 7개의 잔류물(DNA 염기쌍이 아닌 아미노산 잔류물)마다 반복됩니다.첫 번째 및 특히 네 번째 잔류물(a 및 d 위치로 알려져 있음)은 거의 항상 소수성이고, 네 번째 잔류물은 전형적으로 류신이다. 이는 코일 코일 형태의 일종인 류신 지퍼라고 불리는 구조적 모티브의 이름을 가져왔다.이러한 소수성 잔류물은 나선 다발의 내부에 함께 채워집니다.일반적으로 제5 및 제7의 잔류물(e 및 g 위치)은 서로 반대되는 전하를 가지며, 정전 상호작용에 의해 안정화 된 염교(salt bridge)를 형성한다.케라틴이나 미오신 또는 키네신의 "줄기"와 같은 섬유질 단백질은 종종 여러 이량화 단백질처럼 코일 구조를 채택합니다.코일 코일 한 쌍 즉, 4개의 나선 다발은 단백질에서 매우 일반적인 구조적 모티브입니다.예를 들어, 그것은 인간의 성장 호르몬과 여러 종류의 시토크롬에서 발생한다.박테리아의 플라스미드 복제를 촉진하는 Rop 단백질은 하나의 폴리펩타이드로 코일형 코일을 형성하고 두 개의 단량체가 모여 4나선 다발을 형성하는 흥미로운 경우이다.
얼굴 정리
특정 나선을 구성하는 아미노산은 구성 아미노산의 방향을 나타내는 나선형 바퀴에 표시할 수 있습니다(이러한 다이어그램은 류신 지퍼에 대한 기사 참조).종종 구상 단백질에서, 코일 코일 및 류신 지퍼와 같은 특수 구조에서, α-나선은 두 개의 "면"을 나타낼 것입니다. 하나는 단백질의 내부로 향하는 소수성 아미노산을 포함하고, 하나는 소수성 코어에 있고, 다른 하나는 용매-e로 향하는 극성 아미노산을 포함합니다.단백질의 xposed 표면.
결합 방향의 변화는 또한 표면적으로 조직화된 올리고펩티드에 대해서도 일어난다.이 패턴은 항균 펩타이드에서 특히 흔하며, 이것이 그들의 기능과 어떻게 관련되어 있는지를 설명하기 위해 많은 모델이 고안되었다.이들 중 많은 것에 공통적으로 항균 펩타이드의 소수성 면이 [33][34]막심부의 지방사슬과 연관된 후 혈장막에 모공을 형성한다는 것이다.
대규모 어셈블리

X선 결정학에 의해 구조가 해결된 첫 번째 두 단백질인 미오글로빈과 헤모글로빈은 약 70%의 α-나선으로 이루어진 매우 유사한 주름을 가지고 있으며, 나머지는 나선을 연결하는 비반복 영역, 즉 "루프"이다.단백질의 지배적 접힘에 따라 단백질을 분류할 때, 단백질 구조 분류 데이터베이스는 특히 all-α 단백질에 대한 큰 범주를 유지한다.
헤모글로빈은 기능성 산소 결합 분자가 4개의 서브 유닛으로 구성된 훨씬 더 큰 규모의 4차 구조를 가지고 있습니다.
기능적 역할

DNA결합
α-헬리체는 나선-나선-나선 모티브, 류신 지퍼 모티브 및 아연 핑거 모티브를 포함한 DNA 결합 모티브에서 특히 중요하다.이는 α-나선의 지름이 B-형 DNA의 주요 홈의 폭과 거의 동일한 평균 세트를 포함하여 약 12Ω(1.2nm)이라는 편리한 구조적 사실과 또한 나선형의 코일 코일(또는 류신 지퍼) 이합체가 접촉하는 종류의 상호작용 표면을 쉽게 위치시킬 수 있기 때문이다.이중 나선형 [35]DNA에서 공통적으로 나타나는 대칭 반복입니다.두 측면의 예로는 전사인자 Max(왼쪽 이미지 참조)가 있으며, 이는 나선 코일형 코일을 사용하여 이량화하며, DNA 메이저 홈의 2회 연속적인 회전으로 상호작용을 위한 다른 나선 쌍을 위치시킨다.
막 스패닝
또한 α-헬리케인은 생물학적 막(막 통과 단백질)을 가로지르는 가장 일반적인 [36]단백질 구조 요소이며, 나선 구조가 내부적으로 모든 골격 수소 결합을 만족시킬 수 있고, 측쇄가 소수성일 경우 극성 그룹이 막에 노출되지 않기 때문에 추정됩니다.단백질은 때로는 단일 막 스판 나선에 의해 고정되기도 하고, 때로는 쌍에 의해 고정되기도 하며, 가장 고전적으로 로돕신(오른쪽 그림 참조) 및 기타 G 단백질 결합 수용체(GPCRs)와 같은 고리 모양으로 위아래로 배열된 7개의 나선으로 구성된다.α-Helical transmbrane 도메인 쌍 사이의 구조적 안정성은 예를 들어 글리신-xx-글리신(또는 small-xx-small)[37] 모티브와 같은 보존된 막간 패킹 모티브에 의존한다.
기계적 특성
많은 알파 나선형 필라멘트 및 조직에 나타나는 특징적인 하중 조건인 축방향 인장 변형 하의 α-나선은 강직-연강 탄젠트 [38]계수의 특징적인 3상 거동을 초래한다.1단계는 나선이 균등하게 늘어나는 작은 변형 상태에 해당하고, 2단계는 H-결합 그룹의 파열에 의해 알파-나선 회전이 중단된다.단계 III는 일반적으로 큰 변형 공유 결합 스트레칭과 관련이 있습니다.
동적 기능
단백질의 알파헬리체는 라만 분광법에[39] 의해 관찰되고 준연속 [40][41]모델을 통해 분석되는 저주파 아코디언 유사 운동을 가질 수 있다.3차 상호작용에 의해 안정화되지 않은 나선은 동적 거동을 나타내며,[42] 이는 주로 끝부분에서 나선이 벗겨지는 것에 기인할 수 있다.
나선-코일 전이
아미노산의 호모폴리머(폴리리신과 같은)는 고온에서 "녹아 없어지는" 저온에서 α-나선 구조를 채택할 수 있습니다.이러한 나선-코일 전이는 한때 단백질 변성과 유사한 것으로 생각되었다.이 전환의 통계 역학은 두 가지 매개변수로 특징지어지는 우아한 전달 매트릭스 방법을 사용하여 모델링할 수 있다. 즉, 나선 시작 성향과 나선 확장 성향이다.
인 아트

적어도 다섯 명의 예술가들이 그들의 작품에서 알파-나선을 분명히 언급했다: 회화의 줄리 뉴돌, 조각의 줄리안 보스-안드레에, 밧세바 그로스만, 바이런 루빈, 그리고 마이크 타이카.
미술 부전공으로 미생물학 학위를 가진 샌프란시스코 지역 화가 줄리 [43]뉴돌은 1990년부터 현미경 이미지와 분자에서 영감을 얻은 그림을 전문으로 해왔습니다.그녀의 그림 "알파 나선의 출현"(2003)은 알파 나선형으로 배열된 인간 형상을 특징으로 한다.화가에 따르면, "꽃은 각각의 아미노산이 세상에 내밀고 있는 다양한 종류의 곁사슬을 반영한다."[43]이 같은 은유 또한 과학자 쪽에서 반향된다: "β 시트는 단단하게 반복되는 규칙성을 보여주지 않고 우아하고 꼬불꼬불한 곡선으로 흐른다. 그리고 심지어 알파-나선은 꽃 줄기처럼 더 규칙적이다. 그들의 분기하는 노드는 환경, 발달의 역사, 그리고 각각의 부분의 진화의 영향을 보여준다.독특한 기능"[12]이라고 말합니다.
Julian Voss-Andreae는 실험 물리학과 조각학 학위를 가진 독일 태생의 조각가입니다.2001년 이후 Voss-Andreae는 단백질 구조에 기초한 "단백질 조각"[44]을 만들어 냈고, 알파-나선은 그가 선호하는 물체 중 하나이다.보스-안드레에는 대나무와 나무 전체를 포함한 다양한 재료로 알파 나선형 조각품을 만들었습니다.2004년 알파나선의 발견자인 리누스 폴링을 추모하기 위해 만들어진 Voss-Andreae 기념비는 알파나선의 구조에 정렬된 대형 강철 빔으로 만들어졌다.10피트(3m) 높이의 밝은 빨간색 조형물은 오리건 주 포틀랜드에 있는 폴링의 어린 시절 집 앞에 서 있다.
α-헬리체의 리본 다이어그램은 인슐린, 헤모글로빈 및 DNA 중합효소 [45]등 예술가 Bathsheba Grossman이 만든 단백질 구조의 레이저 식각 결정 조각에서 중요한 요소입니다.Byron Rubin은 전직 단백질 결정학자이며 현재는 단백질, 핵산, 약물 분자의 금속을 전문으로 조각하고 있습니다.이들 중 다수는 서브틸리신, 인간 성장 호르몬, 그리고 포스포리파아제 [46]A2와 같은 알파-헬리스를 특징으로 합니다.
마이크 타이카는 데이비드 베이커와 함께 일하는 워싱턴 대학의 컴퓨터 생화학자입니다.타이카는 2010년부터 유비퀴틴과 칼륨 채널 4중합체를 [47]포함한 구리와 강철로 단백질 분자를 조각해 왔다.
「 」를 참조해 주세요.
레퍼런스
- ^ Kendrew JC, Dickerson RE, Strandberg BE, Hart RG, Davies DR, Phillips DC, Shore VC (February 1960). "Structure of myoglobin: A three-dimensional Fourier synthesis at 2 Å resolution". Nature. 185 (4711): 422–7. Bibcode:1960Natur.185..422K. doi:10.1038/185422a0. PMID 18990802. S2CID 4167651.
- ^ Neurath H (1940). "Intramolecular folding of polypeptide chains in relation to protein structure". Journal of Physical Chemistry. 44 (3): 296–305. doi:10.1021/j150399a003.
- ^ Taylor HS (1942). "Large molecules through atomic spectacles". Proceedings of the American Philosophical Society. 85 (1): 1–12. JSTOR 985121.
- ^ Huggins M (1943). "The structure of fibrous proteins". Chemical Reviews. 32 (2): 195–218. doi:10.1021/cr60102a002.
- ^ Bragg WL, Kendrew JC, Perutz MF (1950). "Polypeptide chain configurations in crystalline proteins". Proceedings of the Royal Society of London. Series A. Mathematical and Physical Sciences. 203 (1074): 321–?. Bibcode:1950RSPSA.203..321B. doi:10.1098/rspa.1950.0142. S2CID 93804323.
- ^ Pauling L, Corey RB, Branson HR (April 1951). "The structure of proteins; two hydrogen-bonded helical configurations of the polypeptide chain". Proceedings of the National Academy of Sciences of the United States of America. 37 (4): 205–11. Bibcode:1951PNAS...37..205P. doi:10.1073/pnas.37.4.205. PMC 1063337. PMID 14816373.
- ^ "The Nobel Prize in Chemistry 1954".
- ^ Dunitz J (2001). "Pauling's Left-Handed α-Helix". Angewandte Chemie International Edition. 40 (22): 4167–4173. doi:10.1002/1521-3773(20011119)40:22<4167::AID-ANIE4167>3.0.CO;2-Q. PMID 29712120.
- ^ IUPAC-IUB Commission on Biochemical Nomenclature (1970). "Abbreviations and symbols for the description of the conformation of polypeptide chains". Journal of Biological Chemistry. 245 (24): 6489–6497. doi:10.1016/S0021-9258(18)62561-X.
- ^ "Polypeptide Conformations 1 and 2". www.sbcs.qmul.ac.uk. Retrieved 5 November 2018.
- ^ Kabsch W, Sander C (December 1983). "Dictionary of protein secondary structure: pattern recognition of hydrogen-bonded and geometrical features". Biopolymers. 22 (12): 2577–637. doi:10.1002/bip.360221211. PMID 6667333. S2CID 29185760.
- ^ a b c Richardson JS (1981). "The anatomy and taxonomy of protein structure". Advances in Protein Chemistry. 34: 167–339. doi:10.1016/S0065-3233(08)60520-3. ISBN 9780120342341. PMID 7020376.
- ^ Lovell SC, Davis IW, Arendall WB, de Bakker PI, Word JM, Prisant MG, Richardson JS, Richardson DC (February 2003). "Structure validation by Calpha geometry: phi,psi and Cbeta deviation". Proteins. 50 (3): 437–50. doi:10.1002/prot.10286. PMID 12557186. S2CID 8358424.
- ^ Dickerson RE, Geis I (1969), Structure and Action of Proteins, Harper, New York
- ^ Zorko, Matjaž (2010). "Structural Organization of Proteins". In Langel, Ülo; Cravatt, Benjamin F.; Gräslund, Astrid; von Heijne, Gunnar; Land, Tiit; Niessen, Sherry; Zorko, Matjaž (eds.). Introduction to Peptides and Proteins. Boca Raton: CRC Press. pp. 36–57. ISBN 9781439882047.
- ^ Terwilliger TC (March 2010). "Rapid model building of alpha-helices in electron-density maps". Acta Crystallographica Section D. 66 (Pt 3): 268–75. doi:10.1107/S0907444910000314. PMC 2827347. PMID 20179338.
- ^ Hudgins RR, Jarrold MF (1999). "Helix Formation in Unsolvated Alanine-Based Peptides: Helical Monomers and Helical Dimers". Journal of the American Chemical Society. 121 (14): 3494–3501. doi:10.1021/ja983996a.
- ^ Kutchukian PS, Yang JS, Verdine GL, Shakhnovich EI (April 2009). "All-atom model for stabilization of alpha-helical structure in peptides by hydrocarbon staples". Journal of the American Chemical Society. 131 (13): 4622–7. doi:10.1021/ja805037p. PMC 2735086. PMID 19334772.
- ^ Abrusan G, Marsh JA (2016). "Alpha helices are more robust to mutations than beta strands". PLOS Computational Biology. 12 (12): e1005242. Bibcode:2016PLSCB..12E5242A. doi:10.1371/journal.pcbi.1005242. PMC 5147804. PMID 27935949.
- ^ Rocklin GJ, et al. (2017). "Global analysis of protein folding using massively parallel design, synthesis, and testing". Science. 357 (6347): 168–175. Bibcode:2017Sci...357..168R. doi:10.1126/science.aan0693. PMC 5568797. PMID 28706065.
- ^ Schiffer M, Edmundson AB (1967). "Use of helical wheels to represent the structures of proteins and to identify segments with helical potential". Biophysical Journal. 7 (2): 121–135. doi:10.1016/S0006-3495(67)86579-2.
- ^ Chou KC, Zhang CT, Maggiora GM (1997). "Disposition of amphiphilic helices in heteropolar environments". Proteins: Structure, Function, and Genetics. 28: 99–108.
- ^ Dunnill P (1968). "The Use of Helical Net-Diagrams to Represent Protein Structures". Biophysical Journal. 8 (7): 865–875. doi:10.1016/S0006-3495(68)86525-7. PMC 1367563. PMID 5699810.
- ^ Gautier R, Douguet D, Antonny B, Drin G (2008). "HELIQUEST: a web server to screen sequences with specific alpha-helical properties". Bioinformatics. 24 (18): 2101–2102. doi:10.1093/bioinformatics/btn392.
- ^ Mol AR, Castro MS, Fontes W (2018). "NetWheels: A web application to create high quality peptide helical wheel and net projections". bioRxiv. doi:10.1101/416347.
- ^ Wadhwa RR, Subramanian V, Stevens-Truss R (2018). "Visualizing alpha-helical peptides in R with helixvis". Journal of Open Source Software. 3 (31): 1008. doi:10.21105/joss.01008.
- ^ Subramanian V, Wadhwa RR, Stevens-Truss R (2020). "Helixvis: Visualize alpha-helical peptides in Python". chemRxiv.
- ^ Pace CN, Scholtz JM (July 1998). "A helix propensity scale based on experimental studies of peptides and proteins". Biophysical Journal. 75 (1): 422–7. Bibcode:1998BpJ....75..422N. doi:10.1016/S0006-3495(98)77529-0. PMC 1299714. PMID 9649402.
- ^ Pace, C. Nick; Scholtz, J. Martin (1998). "A Helix Propensity Scale Based on Experimental Studies of Peptides and Proteins". Biophysical Journal. Vol. 75. pp. 422–427. Bibcode:1998BpJ....75..422N. doi:10.1016/s0006-3495(98)77529-0.
- ^ Hol WG, van Duijnen PT, Berendsen HJ (1978). "The alpha helix dipole and the properties of proteins". Nature. 273 (5662): 443–446. Bibcode:1978Natur.273..443H. doi:10.1038/273443a0. PMID 661956. S2CID 4147335.
- ^ He JJ, Quiocho FA (October 1993). "Dominant role of local dipoles in stabilizing uncompensated charges on a sulfate sequestered in a periplasmic active transport protein". Protein Science. 2 (10): 1643–7. doi:10.1002/pro.5560021010. PMC 2142251. PMID 8251939.
- ^ Milner-White EJ (November 1997). "The partial charge of the nitrogen atom in peptide bonds". Protein Science. 6 (11): 2477–82. doi:10.1002/pro.5560061125. PMC 2143592. PMID 9385654.
- ^ Kohn, Eric M.; Shirley, David J.; Arotsky, Lubov; Picciano, Angela M.; Ridgway, Zachary; Urban, Michael W.; Carone, Benjamin R.; Caputo, Gregory A. (2018-02-04). "Role of Cationic Side Chains in the Antimicrobial Activity of C18G". Molecules. 23 (2): 329. doi:10.3390/molecules23020329. PMC 6017431. PMID 29401708.
- ^ Toke, Orsolya (2005). "Antimicrobial peptides: new candidates in the fight against bacterial infections". Biopolymers. 80 (6): 717–735. doi:10.1002/bip.20286. ISSN 0006-3525. PMID 15880793.
- ^ Branden & Tooze, 10장
- ^ Branden & Tooze, 12장
- ^ Nash A, Notman R, Dixon AM (2015). "De novo design of transmembrane helix–helix interactions and measurement of stability in a biological membrane". Biochimica et Biophysica Acta (BBA) - Biomembranes. 1848 (5): 1248–57. doi:10.1016/j.bbamem.2015.02.020. PMID 25732028.
- ^ Ackbarow T, Chen X, Keten S, Buehler MJ (October 2007). "Hierarchies, multiple energy barriers, and robustness govern the fracture mechanics of alpha-helical and beta-sheet protein domains". Proceedings of the National Academy of Sciences of the United States of America. 104 (42): 16410–5. Bibcode:2007PNAS..10416410A. doi:10.1073/pnas.0705759104. PMC 2034213. PMID 17925444.
- ^ Painter PC, Mosher LE, Rhoads C (July 1982). "Low-frequency modes in the Raman spectra of proteins". Biopolymers. 21 (7): 1469–72. doi:10.1002/bip.360210715. PMID 7115900.
- ^ Chou KC (December 1983). "Identification of low-frequency modes in protein molecules". The Biochemical Journal. 215 (3): 465–9. doi:10.1042/bj2150465. PMC 1152424. PMID 6362659.
- ^ Chou KC (May 1984). "Biological functions of low-frequency vibrations (phonons). III. Helical structures and microenvironment". Biophysical Journal. 45 (5): 881–9. Bibcode:1984BpJ....45..881C. doi:10.1016/S0006-3495(84)84234-4. PMC 1434967. PMID 6428481.
- ^ Fierz B, Reiner A, Kiefhaber T (January 2009). "Local conformational dynamics in alpha-helices measured by fast triplet transfer". Proceedings of the National Academy of Sciences of the United States of America. 106 (4): 1057–62. Bibcode:2009PNAS..106.1057F. doi:10.1073/pnas.0808581106. PMC 2633579. PMID 19131517.
- ^ a b "Julie Newdoll Scientifically Inspired Art, Music, Board Games". www.brushwithscience.com. Retrieved 2016-04-06.
- ^ Voss-Andreae J (2005). "Protein Sculptures: Life's Building Blocks Inspire Art". Leonardo. 38: 41–45. doi:10.1162/leon.2005.38.1.41. S2CID 57558522.
- ^ Grossman, Bathsheba. "About the Artist". Bathsheba Sculpture. Retrieved 2016-04-06.
- ^ "About". molecularsculpture.com. Retrieved 2016-04-06.
- ^ Tyka, Mike. "About". www.miketyka.com. Retrieved 2016-04-06.
추가 정보
- 를 클릭합니다Tooze J, Brändén C (1999). Introduction to protein structure. New York: Garland Pub. ISBN 0-8153-2304-2..
- Eisenberg D (September 2003). "The discovery of the alpha-helix and beta-sheet, the principal structural features of proteins". Proceedings of the National Academy of Sciences of the United States of America. 100 (20): 11207–10. Bibcode:2003PNAS..10011207E. doi:10.1073/pnas.2034522100. PMC 208735. PMID 12966187.
- Astbury WT, Woods HJ (1931). "The Molecular Weights of Proteins". Nature. 127 (3209): 663–665. Bibcode:1931Natur.127..663A. doi:10.1038/127663b0. S2CID 4133226.
- Astbury WT, Street A (1931). "X-ray studies of the structures of hair, wool and related fibres. I. General". Trans. R. Soc. Lond. A230: 75–101. Bibcode:1932RSPTA.230...75A. doi:10.1098/rsta.1932.0003.
- Astbury WT (1933). "Some Problems in the X-ray Analysis of the Structure of Animal Hairs and Other Protein Fibers". Trans. Faraday Soc. 29 (140): 193–211. doi:10.1039/tf9332900193.
- Astbury WT, Woods HJ (1934). "X-ray studies of the structures of hair, wool and related fibres. II. The molecular structure and elastic properties of hair keratin". Philosophical Transactions of the Royal Society of London, Series A. 232 (707–720): 333–394. Bibcode:1934RSPTA.232..333A. doi:10.1098/rsta.1934.0010.
- Astbury WT, Sisson WA (1935). "X-ray studies of the structures of hair, wool and related fibres. III. The configuration of the keratin molecule and its orientation in the biological cell". Proceedings of the Royal Society. A150 (871): 533–551. Bibcode:1935RSPSA.150..533A. doi:10.1098/rspa.1935.0121.
- Sugeta H, Miyazawa T (1967). "General Method for Calculating Helical Parameters of Polymer Chains from Bond Lengths, Bond Angles, and Internal-Rotation Angles". Biopolymers. 5 (7): 673–679. doi:10.1002/bip.1967.360050708. S2CID 97785907.
- Wada A (1976). "The alpha-helix as an electric macro-dipole". Advances in Biophysics: 1–63. PMID 797240.
- Chothia C, Levitt M, Richardson D (October 1977). "Structure of proteins: packing of alpha-helices and pleated sheets". Proceedings of the National Academy of Sciences of the United States of America. 74 (10): 4130–4. Bibcode:1977PNAS...74.4130C. doi:10.1073/pnas.74.10.4130. PMC 431889. PMID 270659.
- Chothia C, Levitt M, Richardson D (January 1981). "Helix to helix packing in proteins". Journal of Molecular Biology. 145 (1): 215–50. doi:10.1016/0022-2836(81)90341-7. PMID 7265198.
- Hol WG (1985). "The role of the alpha-helix dipole in protein function and structure". Progress in Biophysics and Molecular Biology. 45 (3): 149–95. doi:10.1016/0079-6107(85)90001-X. PMID 3892583.
- Barlow DJ, Thornton JM (June 1988). "Helix geometry in proteins". Journal of Molecular Biology. 201 (3): 601–19. doi:10.1016/0022-2836(88)90641-9. PMID 3418712.
- Murzin AG, Finkelstein AV (December 1988). "General architecture of the alpha-helical globule". Journal of Molecular Biology. 204 (3): 749–69. doi:10.1016/0022-2836(88)90366-X. PMID 3225849.