의약품 설계
Drug design종종 합리적인 약물 설계 또는 단순히 합리적인 설계로 언급되는 약물 설계는 생물학적 표적에 [1]대한 지식을 바탕으로 새로운 약을 찾는 창의적인 과정이다.그 약은 가장 일반적으로 단백질과 같은 생체 분자의 기능을 활성화하거나 억제하는 유기적인 작은 분자이며, 이는 결국 환자에게 치료상의 이점을 가져다 준다.가장 기본적인 의미에서, 약물 설계는 그들이 상호작용하는 생체 분자 표적에 상호 보완적인 모양과 전하로 결합하는 분자의 설계를 포함합니다.약물 설계는 자주 하지만 반드시 컴퓨터 모델링 [2]기술에 의존하지는 않습니다.이러한 유형의 모델링을 컴퓨터 지원 의약품 설계라고 부르기도 한다.마지막으로, 생체분자 타겟의 3차원 구조에 대한 지식에 의존하는 약물 설계는 구조 기반 약물 [2]설계로 알려져 있다.소분자 외에 펩타이드[3][4], 특히 치료용 항체를 포함한 바이오 의약품은 점점 더 중요한 약물 종류이며 이들 단백질 기반 치료제의 친화력, 선택성 및 안정성을 개선하기 위한 계산 방법도 개발되었다.[5]
"약물 설계"라는 문구는 어느 정도 잘못된 명칭이다.보다 정확한 용어는 리간드 설계(즉,[6] 목표물에 단단히 결합하는 분자의 설계)이다.결합 친화성 예측을 위한 설계 기법은 상당히 성공적이지만, 리간드가 안전하고 효과적인 약물이 되기 전에 먼저 최적화되어야 하는 생물 가용성, 대사 반감기, 부작용 등 다른 많은 특성이 있다.이러한 다른 특성은 합리적인 설계 기법으로 예측하기 어려운 경우가 많습니다.그럼에도 불구하고, 높은 감소율 때문에, 특히 약물 개발의 임상 단계 동안, 물리 화학적 특성이 개발 중 합병증을 줄이고 따라서 승인된 시판 약물로 이어질 것으로 예상되는 후보 약물을 선택하는 데 더 많은 관심이 의약품 설계 과정 초기에 집중되고 있다.g.[7] 또한, 계산 방법을 보완한 시험관내 실험은 보다 유리한 ADME(흡수, 분포, 대사 및 배설)와 독성학적 [8]프로파일을 가진 화합물을 선택하기 위해 초기 약물 발견에 점점 더 많이 사용되고 있다.
약물 대상
생체분자 표적(가장 일반적으로 단백질 또는 핵산)은 특정 질병 상태 또는 병리, 미생물 병원체의 감염성 또는 생존과 관련된 특정 대사 경로 또는 신호 경로와 관련된 핵심 분자이다.잠재적 약물 표적은 반드시 질병을 유발하는 것은 아니지만, 정의상 질병을 [9]수정해야 한다.어떤 경우에, 작은 분자는 특정 질병 수정 경로에서 표적 기능을 강화하거나 억제하도록 설계될 것이다.작은 분자(예: 수용체 작용제, 길항제, 역작용제 또는 조절제, 효소 활성제 또는 억제제, 이온 채널 개방제 또는 차단제)[10]는 [11]표적 결합 부위를 보완하는 방식으로 설계될 것이다.작은 분자(약물)는 표적외 분자와의 약물 상호작용이 바람직하지 않은 [12]부작용을 초래할 수 있기 때문에 다른 중요한 "표적외" 분자(종종 항타겟이라고 함)에 영향을 미치지 않도록 설계될 수 있다.결합 부위의 유사성으로 인해 시퀀스 호몰로지를 통해 식별된 밀접하게 관련된 표적은 교차 반응성의 가능성이 가장 높고 따라서 부작용 잠재력이 가장 높다.
가장 일반적으로, 약물은 화학 합성을 통해 생성된 유기 소분자이지만, 생물학적 과정을 통해 생성된 생체 고분자 기반 약물은 점점 더 [13]흔해지고 있다.또한 mRNA 기반의 유전자 사일런싱 기술은 치료상의 [14]응용을 가질 수 있다.
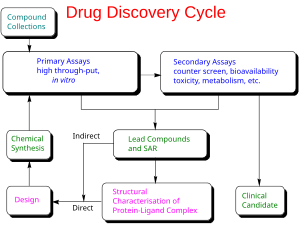
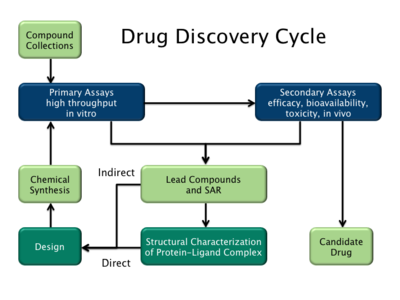
합리적인 약물 발견
배양 세포나 동물에 대한 화학 물질의 시행착오 테스트에 의존하고 치료에 명백한 효과를 맞추는 전통적인 약물 발견 방법과는 대조적으로, 합리적인 약물 설계(역 약리학이라고도 함)는 특정 생물학적 ta의 변조가 있다는 가설에서 시작된다.rget은 치료적 가치가 있을 수 있다.생체분자를 약물 타겟으로 선택하기 위해서는 두 가지 필수 정보가 필요하다.첫 번째는 목표물의 변조가 질병을 수정한다는 증거입니다.이 지식은 예를 들어 생물학적 표적의 돌연변이와 특정 질병 [15]상태 사이의 연관성을 보여주는 질병 연계 연구로부터 얻을 수 있다.두 번째는 타겟이 "약물"이라는 것입니다.이것은 그것이 작은 분자에 결합할 수 있고 그 활동이 작은 [16]분자에 의해 조절될 수 있다는 것을 의미한다.
적합한 타겟이 확인되면 타깃은 정상적으로 복제되고 생성되며 정제됩니다.정제된 단백질은 선별 분석을 확립하기 위해 사용됩니다.또, 타겟의 3차원 구조를 결정해도 좋다.
표적에 결합하는 작은 분자에 대한 연구는 잠재적 약물 화합물의 라이브러리를 선별함으로써 시작된다.이 작업은 스크리닝 검사("습기 스크린")를 사용하여 수행할 수 있습니다.또, 타겟의 구조를 이용할 수 있는 경우는, 후보 약물의 가상 화면을 실시할 수 있다.이상적으로 후보 약물 화합물은 "약물과 유사"해야 한다. 즉, 경구 생체 가용성, 적절한 화학적 및 대사 안정성 및 최소한의 독성 [17]효과로 이어질 것으로 예상되는 특성을 가져야 한다.리핀스키의 규칙 5와 같은 약물 유사성 및 친유성 [18]효율과 같은 다양한 점수 매기기 방법을 추정하기 위해 여러 가지 방법을 사용할 수 있다.약물의 신진대사를 예측하는 몇 가지 방법도 과학 [19]문헌에서 제안되었다.
설계 과정 동안 동시에 최적화되어야 하는 많은 수의 약물 특성 때문에, 때때로 다목적 최적화 기법이 사용된다.[20]마지막으로, 활동 예측을 위한 현재의 방법의 한계 때문에, 약물 설계는 여전히 세렌디피티와[21] 한정적 [22]합리성에 매우 많이 의존한다.
컴퓨터 지원 의약품 설계
약물 설계에서 가장 기본적인 목표는 주어진 분자가 목표물에 결합할 것인지, 만약 그렇다면 얼마나 강하게 결합할 것인지를 예측하는 것이다.분자역학 또는 분자역학은 작은 분자와 그 생물학적 목표물 사이의 분자간 상호작용의 강도를 추정하는데 가장 자주 사용된다.이 방법들은 또한 작은 분자의 배열을 예측하고 작은 분자가 [3][4]결합할 때 발생할 수 있는 목표물의 배향 변화를 모델링하기 위해 사용된다.반경험적, ab initio 양자화학법 또는 밀도함수이론은 종종 분자역학 계산에 최적화된 파라미터를 제공하고 결합 [23]친화성에 영향을 미치는 약물 후보의 전자특성(정전기전위, 분극성 등)의 추정치를 제공하기 위해 사용된다.
결합 친화성의 반정량적 예측을 제공하기 위해 분자역학법을 사용할 수도 있다.또한 결합 친화성 추정치를 제공하기 위해 지식 기반 스코어링 함수를 사용할 수 있다.이 방법들은 작은 분자와 [24][25]표적 사이의 계산적으로 도출된 상호작용 에너지에 실험 친화성을 적합시킴으로써 예측 결합 친화성 방정식을 도출하기 위해 선형 회귀, 기계 학습, 신경망 또는 기타 통계 기법을 사용한다.
이상적으로 계산 방법은 화합물이 합성되기 전에 친화력을 예측할 수 있으며, 따라서 이론적으로는 하나의 화합물만 합성하면 되며, 엄청난 시간과 비용을 절약할 수 있다.현실은 현재의 계산 방법이 불완전하고 기껏해야 질적으로 정확한 친화력 추정치만 제공한다는 것이다.실제로 최적의 약이 발견되기까지는 설계, 합성 및 테스트를 여러 번 반복해야 합니다.계산 방법은 필요한 반복 횟수를 줄임으로써 발견을 가속화하고 종종 새로운 [26][27]구조를 제공해 왔다.
컴퓨터의 도움을 받아 약물 설계를 다음과 같은 약물 발견 단계에서 사용할 수 있다.
- 가상 스크리닝을 사용한 히트 식별(표준 또는 리간드 기반 설계)
- 선호도 및 선택성의 히트 투 리드 최적화(구조 기반 설계, QSAR 등)
- 친화성을 유지하면서 다른 약품의 최적화를 유도하는
최근 스코어링 함수에 의해 산출된 결합 친화성의 예측부족을 극복하기 위해 단백질-리간드 상호작용 및 화합물 3D 구조정보를 분석에 이용한다.구조 기반 의약품 설계의 경우, 농축도를 개선하고 잠재적 후보를 효과적으로 채굴하기 위해 단백질-리간드 상호작용에 초점을 맞춘 몇 가지 사후 선별 분석이 개발되었다.
- 컨센서스[28][29] 스코어링
- 여러 채점 기능 투표로 후보 선정
- 단백질-리간드 구조 정보와 점수 기준 사이의 관계를 잃을 수 있다.
- 클러스터[30][31] 분석
- 단백질 리간드 3D 정보에 따라 후보를 표현하고 클러스터화합니다.
- 단백질-리간드 상호작용의 의미 있는 표현이 필요하다.
종류들
약물 설계에는 크게 두 가지 유형이 있습니다.첫 번째는 리간드 기반 약물 설계, 두 번째는 구조 기반 약물 [2]설계라고 한다.
리간드 베이스의
리간드 기반 약물 설계(또는 간접 약물 설계)는 생물학적 대상과 결합하는 다른 분자에 대한 지식에 의존합니다.이러한 다른 분자들은 [32]목표물에 결합하기 위해 분자가 가져야 하는 최소한의 필수 구조적 특성을 정의하는 약초 모델을 도출하기 위해 사용될 수 있다.즉, 생물학적 타깃의 모델은 무엇이 결합하는지에 대한 지식에 기초하여 구축될 수 있으며, 이 모델은 타깃과 상호작용하는 새로운 분자 실체를 설계하기 위해 사용될 수 있다.또는 분자의 계산된 특성과 실험적으로 결정된 생물학적 활성 사이의 상관관계를 도출할 수 있다.이러한 QSAR 관계를 사용하여 [33]새로운 아날로그의 액티비티를 예측할 수 있습니다.
구조 기반
구조 기반 의약품 설계(또는 직접 의약품 설계)는 X선 결정학 또는 NMR 스펙트럼 [34]분석과 같은 방법을 통해 얻은 생물학적 표적의 3차원 구조에 대한 지식에 의존한다.타깃의 실험구조를 이용할 수 없는 경우에는 관련된 단백질의 실험구조에 기초하여 타깃의 호몰로지 모델을 작성할 수 있다.생물학적 표적의 구조를 사용하여 표적에 높은 친화력과 선택성으로 결합할 것으로 예상되는 후보 약물은 대화형 그래픽과 약제화자의 직관을 사용하여 설계할 수 있다.또는 신약 [35]후보를 제안하기 위해 다양한 자동화된 계산 절차를 사용할 수 있다.
구조 기반 의약품 설계를 위한 현재의 방법은 크게 세 가지 [36]범주로 나눌 수 있다.첫 번째 방법은 빠른 근사 도킹 프로그램을 사용하여 수용체의 결합 포켓에 적합한 것을 찾기 위해 작은 분자의 3D 구조의 큰 데이터베이스를 검색하여 주어진 수용체에 대한 새로운 리간드를 식별하는 것입니다.이 방법은 가상 스크리닝이라고 불립니다.두 번째 범주는 새로운 리간드의 de novo 설계이다.본 발명의 방법에서는 작은 조각을 단계적으로 조립함으로써 결합 포켓의 제약 내에 리간드 분자를 축적한다.이 조각들은 개별 원자이거나 분자 조각일 수 있다.이러한 방법의 주요 장점은 데이터베이스에 포함되지 않은 새로운 구조를 [37][38][39]제안할 수 있다는 것이다.세 번째 방법은 결합 [36]캐비티 내에서 제안된 아날로그를 평가함으로써 알려진 리간드를 최적화하는 것이다.
바인딩 사이트 식별
바인딩 사이트 식별은 구조 기반 [16][40]설계의 첫 번째 단계입니다.결합배위자의 존재 하에서 타깃의 구조 또는 충분히 유사한 호몰로지가 결정되면 결합부위의 위치가 사소한 구조에서 리간드를 관찰할 수 있어야 한다.단, 관심 있는 알로스테릭 결합 부위가 비어 있을 수 있습니다.또한 아포단백질(배위자가 없는 단백질) 구조만이 이용 가능하며, 높은 친화성으로 리간드를 결합할 가능성이 있는 비점유 부위의 신뢰성 있는 식별은 중요하지 않을 수 있다.간단히 말해서 결합 부위 식별은 일반적으로 리간드 [16][40]결합을 촉진하는 적절한 "핫 스폿"(소수성 표면, 수소 결합 부위 등)을 가진 약제 크기 분자를 수용할 수 있는 단백질의 오목 표면 식별에 의존한다.
스코어링 기능
구조 기반 의약품 설계는 분자 인식 원리를 적용하여 단백질의 구조를 새로운 배위자를 설계하기 위한 기초로서 사용하려고 한다.표적과의 선택적 고친화성 결합은 부작용이 적은 보다 효과적인 약물로 이어지기 때문에 일반적으로 바람직하다.따라서 잠재적인 새로운 리간드를 설계하거나 얻기 위한 가장 중요한 원칙 중 하나는 특정 리간드의 표적(및 알려진 안티타겟)에 대한 결합 친화성을 예측하고 예측된 친화성을 [41]선택 기준으로 사용하는 것이다.
수용체에 대한 리간드의 결합 에너지를 설명하기 위한 일반적인 목적의 초기 경험적 스코어링 함수는 Böhm에 [42][43]의해 개발되었다.이 경험적 점수 매기기 함수는 다음과 같은 형태를 취했다.
여기서:
- δG0 – 결합 시 리간드의 변환 및 회전 엔트로피의 전체적인 손실에 해당하는 경험적으로 도출된 오프셋.
- δGhb – 수소 결합의 기여
- δGionic – 이온 상호작용에 의한 기여
- δGlip – A가 리간드와 수용체 사이의 친유성 접촉 표면적인 친유성 상호작용의lipo 기여
- δGrot – 결합 시 리간드 결합 내 회전 가능 동결에 따른 엔트로피 패널티
보다 일반적인 열역학적 "마스터" 방정식은 다음과 같습니다.[44]
여기서:
- 용해 – 용매에서 배위자를 제거하기 위한 장내 패널티
- 움직임 – 리간드가 수용체에 결합할 때 자유도를 감소시키는 엔트로피 패널티
- 구성 – 배위자를 "활성" 구성으로 만드는 데 필요한 구성 변형률 에너지
- 상호 작용 – 리셉터와 리간드를 "변환"하기 위한 장내 이득
기본적인 생각은 전체적인 결합 자유 에너지가 결합 과정에 중요한 것으로 알려진 독립 성분으로 분해될 수 있다는 것입니다.각 성분은 리간드와 그 표적 수용체 사이의 결합 과정 중 특정 종류의 자유 에너지 변화를 반영한다.마스터 방정식은 이 성분들의 선형 조합입니다.깁스 자유 에너지 방정식에 따르면 해리 평형 상수 K와d 자유 에너지 성분 간의 관계가 구축되었다.
마스터 방정식의 각 성분을 추정하기 위해 다양한 계산 방법이 사용됩니다.예를 들어 리간드 결합 시 극 표면적의 변화를 이용하여 용해에너지를 추정할 수 있다.리간드 결합에 동결된 회전 가능한 결합의 수는 운동 항에 비례합니다.구성 또는 변형 에너지는 분자역학 계산을 사용하여 추정할 수 있습니다.마지막으로 비극성 표면의 변화, 평균력의 통계적으로 도출된 전위, 형성된 수소결합 수 등의 방법을 사용하여 상호작용 에너지를 추정할 수 있다.실제로 주 방정식의 성분은 다중 선형 회귀 분석을 사용하여 실험 데이터에 적합합니다.이것은 보다 정확하지는 않지만 보다 일반적인 "글로벌" 모델을 생산하기 위해 많은 종류의 리간드와 수용체를 포함한 다양한 훈련 세트 또는 보다 정확하지만 덜 일반적인 "로컬" [45]모델을 생산하기 위해 보다 제한된 일련의 리간드와 수용체를 사용하여 수행될 수 있다.
예
합리적인 의약품 설계의 특정 예는 X선 결정학 및 NMR 분광학 등의 기술에서 얻은 생체분자에 대한 3차원 정보의 사용을 포함한다.특히 컴퓨터 지원 약물 설계는 잠재적 배위자에 결합된 표적 단백질의 고해상도 구조가 있을 때 훨씬 다루기 쉬워진다.약물 발견에 대한 이러한 접근방식을 구조 기반 약물 설계라고 부르기도 한다.승인된 의약품으로 이어지는 구조 기반 의약품 설계의 적용에 대한 첫 번째 분명한 예는 [46][47]1995년에 승인된 탄산 무수분해효소 억제제 도르졸라미드이다.
합리적인 약물 설계에서 또 다른 중요한 사례 연구는 필라델피아 염색체 양성 백혈병(만성 골수성 백혈병 및 때때로 급성 림프구 백혈병)에 특징적인 bcr-abl 융합 단백질을 위해 특별히 설계된 티로신 키나아제 억제제인 이마티닙이다.이마티닙은 대부분의 화학요법 약물이 암세포와 다른 [48]조직을 구별하지 않고 빠르게 분열하는 세포를 목표로 하기 때문에 이전의 암 치료약과는 상당히 다르다.
기타 예는 다음과 같습니다.
도입 사례
비판
합리적인 약물 설계의 고도로 경직되고 집중적인 특성이 약물 [50]발견의 세렌디피티를 억제한다는 주장이 제기되어 왔다.
「 」를 참조해 주세요.
레퍼런스
- ^ Madsen U, Krogsgaard-Larsen P, Liljefors T (2002). Textbook of Drug Design and Discovery. Washington, D.C.: Taylor & Francis. ISBN 978-0-415-28288-8.
- ^ a b c Reynolds CH, Merz KM, Ringe D, eds. (2010). Drug Design: Structure- and Ligand-Based Approaches (1 ed.). Cambridge, UK: Cambridge University Press. ISBN 978-0521887236.
- ^ a b Fosgerau K, Hoffmann T (January 2015). "Peptide therapeutics: current status and future directions". Drug Discovery Today. 20 (1): 122–128. doi:10.1016/j.drudis.2014.10.003. PMID 25450771.
- ^ a b Ciemny M, Kurcinski M, Kamel K, Kolinski A, Alam N, Schueler-Furman O, Kmiecik S (August 2018). "Protein-peptide docking: opportunities and challenges". Drug Discovery Today. 23 (8): 1530–1537. doi:10.1016/j.drudis.2018.05.006. PMID 29733895.
- ^ Shirai H, Prades C, Vita R, Marcatili P, Popovic B, Xu J, et al. (November 2014). "Antibody informatics for drug discovery". Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1844 (11): 2002–2015. doi:10.1016/j.bbapap.2014.07.006. PMID 25110827.
- ^ Tollenaere JP (Apr 1996). "The role of structure-based ligand design and molecular modelling in drug discovery". Pharmacy World & Science. 18 (2): 56–62. doi:10.1007/BF00579706. PMID 8739258. S2CID 21550508.
- ^ Waring MJ, Arrowsmith J, Leach AR, Leeson PD, Mandrell S, Owen RM, Pairaudeau G, Pennie WD, Pickett SD, Wang J, Wallace O, Weir A (2015). "An analysis of the attrition of drug candidates from four major pharmaceutical companies". Nature Reviews Drug Discovery. 14 (7): 475–86. doi:10.1038/nrd4609. PMID 26091267. S2CID 25292436.
- ^ Yu H, Adedoyin A (Sep 2003). "ADME-Tox in drug discovery: integration of experimental and computational technologies". Drug Discovery Today. 8 (18): 852–61. doi:10.1016/S1359-6446(03)02828-9. PMID 12963322.
- ^ Dixon SJ, Stockwell BR (Dec 2009). "Identifying druggable disease-modifying gene products". Current Opinion in Chemical Biology. 13 (5–6): 549–55. doi:10.1016/j.cbpa.2009.08.003. PMC 2787993. PMID 19740696.
- ^ Imming P, Sinning C, Meyer A (Oct 2006). "Drugs, their targets and the nature and number of drug targets". Nature Reviews. Drug Discovery. 5 (10): 821–34. doi:10.1038/nrd2132. PMID 17016423. S2CID 8872470.
- ^ Anderson AC (Sep 2003). "The process of structure-based drug design". Chemistry & Biology. 10 (9): 787–97. doi:10.1016/j.chembiol.2003.09.002. PMID 14522049.
- ^ Recanatini M, Bottegoni G, Cavalli A (Dec 2004). "In silico antitarget screening". Drug Discovery Today: Technologies. 1 (3): 209–15. doi:10.1016/j.ddtec.2004.10.004. PMID 24981487.
- ^ Wu-Pong S, Rojanasakul Y (2008). Biopharmaceutical drug design and development (2nd ed.). Totowa, NJ Humana Press: Humana Press. ISBN 978-1-59745-532-9.
- ^ Scomparin A, Polyak D, Krivitsky A, Satchi-Fainaro R (Apr 2015). "Achieving successful delivery of oligonucleotides - From physico-chemical characterization to in vivo evaluation". Biotechnology Advances. 33 (6): 1294–309. doi:10.1016/j.biotechadv.2015.04.008. PMID 25916823.
- ^ Ganellin CR, Jefferis R, Roberts SM (2013). "The small molecule drug discovery process — from target selection to candidate selection". Introduction to Biological and Small Molecule Drug Research and Development: theory and case studies. Elsevier. ISBN 9780123971760.
- ^ a b c Yuan Y, Pei J, Lai L (Dec 2013). "Binding site detection and druggability prediction of protein targets for structure-based drug design". Current Pharmaceutical Design. 19 (12): 2326–33. doi:10.2174/1381612811319120019. PMID 23082974.
- ^ Rishton GM (Jan 2003). "Nonleadlikeness and leadlikeness in biochemical screening". Drug Discovery Today. 8 (2): 86–96. doi:10.1016/s1359644602025722. PMID 12565011.
- ^ Hopkins AL (2011). "Chapter 25: Pharmacological space". In Wermuth CG (ed.). The Practice of Medicinal Chemistry (3 ed.). Academic Press. pp. 521–527. ISBN 978-0-12-374194-3.
- ^ Kirchmair J (2014). Drug Metabolism Prediction. Wiley's Methods and Principles in Medicinal Chemistry. Vol. 63. Wiley-VCH. ISBN 978-3-527-67301-8.
- ^ Nicolaou CA, Brown N (Sep 2013). "Multi-objective optimization methods in drug design". Drug Discovery Today: Technologies. 10 (3): 427–35. doi:10.1016/j.ddtec.2013.02.001. PMID 24050140.
- ^ Ban TA (2006). "The role of serendipity in drug discovery". Dialogues in Clinical Neuroscience. 8 (3): 335–44. doi:10.31887/DCNS.2006.8.3/tban. PMC 3181823. PMID 17117615.
- ^ Ethiraj SK, Levinthal D (Sep 2004). "Bounded Rationality and the Search for Organizational Architecture: An Evolutionary Perspective on the Design of Organizations and Their Evolvability". Administrative Science Quarterly. Sage Publications, Inc. on behalf of the Johnson Graduate School of Management, Cornell University. 49 (3): 404–437. doi:10.2307/4131441. JSTOR 4131441. S2CID 142910916. SSRN 604123.
- ^ Lewis RA (2011). "Chapter 4: The Development of Molecular Modelling Programs: The Use and Limitations of Physical Models". In Gramatica P, Livingstone DJ, Davis AM (eds.). Drug Design Strategies: Quantitative Approaches. RSC Drug Discovery. Royal Society of Chemistry. pp. 88–107. doi:10.1039/9781849733410-00088. ISBN 978-1849731669.
- ^ Rajamani R, Good AC (May 2007). "Ranking poses in structure-based lead discovery and optimization: current trends in scoring function development". Current Opinion in Drug Discovery & Development. 10 (3): 308–15. PMID 17554857.
- ^ de Azevedo WF, Dias R (Dec 2008). "Computational methods for calculation of ligand-binding affinity". Current Drug Targets. 9 (12): 1031–9. doi:10.2174/138945008786949405. PMID 19128212.
- ^ Singh J, Chuaqui CE, Boriack-Sjodin PA, Lee WC, Pontz T, Corbley MJ, Cheung HK, Arduini RM, Mead JN, Newman MN, Papadatos JL, Bowes S, Josiah S, Ling LE (Dec 2003). "Successful shape-based virtual screening: the discovery of a potent inhibitor of the type I TGFbeta receptor kinase (TbetaRI)". Bioorganic & Medicinal Chemistry Letters. 13 (24): 4355–9. doi:10.1016/j.bmcl.2003.09.028. PMID 14643325.
- ^ Becker OM, Dhanoa DS, Marantz Y, Chen D, Shacham S, Cheruku S, Heifetz A, Mohanty P, Fichman M, Sharadendu A, Nudelman R, Kauffman M, Noiman S (Jun 2006). "An integrated in silico 3D model-driven discovery of a novel, potent, and selective amidosulfonamide 5-HT1A agonist (PRX-00023) for the treatment of anxiety and depression". Journal of Medicinal Chemistry. 49 (11): 3116–35. doi:10.1021/jm0508641. PMID 16722631.
- ^ Liang S, Meroueh SO, Wang G, Qiu C, Zhou Y (May 2009). "Consensus scoring for enriching near-native structures from protein-protein docking decoys". Proteins. 75 (2): 397–403. doi:10.1002/prot.22252. PMC 2656599. PMID 18831053.
- ^ Oda A, Tsuchida K, Takakura T, Yamaotsu N, Hirono S (2006). "Comparison of consensus scoring strategies for evaluating computational models of protein-ligand complexes". Journal of Chemical Information and Modeling. 46 (1): 380–91. doi:10.1021/ci050283k. PMID 16426072.
- ^ Deng Z, Chuaqui C, Singh J (Jan 2004). "Structural interaction fingerprint (SIFt): a novel method for analyzing three-dimensional protein-ligand binding interactions". Journal of Medicinal Chemistry. 47 (2): 337–44. doi:10.1021/jm030331x. PMID 14711306.
- ^ Amari S, Aizawa M, Zhang J, Fukuzawa K, Mochizuki Y, Iwasawa Y, Nakata K, Chuman H, Nakano T (2006). "VISCANA: visualized cluster analysis of protein-ligand interaction based on the ab initio fragment molecular orbital method for virtual ligand screening". Journal of Chemical Information and Modeling. 46 (1): 221–30. doi:10.1021/ci050262q. PMID 16426058.
- ^ Guner OF (2000). Pharmacophore Perception, Development, and use in Drug Design. La Jolla, Calif: International University Line. ISBN 978-0-9636817-6-8.
- ^ Tropsha A (2010). "QSAR in Drug Discovery". In Reynolds CH, Merz KM, Ringe D (eds.). Drug Design: Structure- and Ligand-Based Approaches (1st ed.). Cambridge, UK: Cambridge University Press. pp. 151–164. ISBN 978-0521887236.
- ^ Leach AR, Harren J (2007). Structure-based Drug Discovery. Berlin: Springer. ISBN 978-1-4020-4406-9.
- ^ Mauser H, Guba W (May 2008). "Recent developments in de novo design and scaffold hopping". Current Opinion in Drug Discovery & Development. 11 (3): 365–74. PMID 18428090.
- ^ a b Klebe G (2000). "Recent developments in structure-based drug design". Journal of Molecular Medicine. 78 (5): 269–81. doi:10.1007/s001090000084. PMID 10954199. S2CID 21314020.
- ^ Wang R, Gao Y, Lai L (2000). "LigBuilder: A Multi-Purpose Program for Structure-Based Drug Design". Journal of Molecular Modeling. 6 (7–8): 498–516. doi:10.1007/s0089400060498. S2CID 59482623.
- ^ Schneider G, Fechner U (Aug 2005). "Computer-based de novo design of drug-like molecules". Nature Reviews. Drug Discovery. 4 (8): 649–63. doi:10.1038/nrd1799. PMID 16056391. S2CID 2549851.
- ^ Jorgensen WL (Mar 2004). "The many roles of computation in drug discovery". Science. 303 (5665): 1813–8. Bibcode:2004Sci...303.1813J. doi:10.1126/science.1096361. PMID 15031495. S2CID 1307935.
- ^ a b Leis S, Schneider S, Zacharias M (2010). "In silico prediction of binding sites on proteins". Current Medicinal Chemistry. 17 (15): 1550–62. doi:10.2174/092986710790979944. PMID 20166931.
- ^ Warren GL, Warren SD (2011). "Chapter 16: Scoring Drug-Receptor Interactions". In Gramatica P, Livingstone DJ, Davis AM (eds.). Drug Design Strategies: Quantitative Approaches. RSC Drug Discovery. Royal Society of Chemistry. pp. 440–457. doi:10.1039/9781849733410-00440. ISBN 978-1849731669.
- ^ Böhm HJ (Jun 1994). "The development of a simple empirical scoring function to estimate the binding constant for a protein-ligand complex of known three-dimensional structure". Journal of Computer-Aided Molecular Design. 8 (3): 243–56. Bibcode:1994JCAMD...8..243B. doi:10.1007/BF00126743. PMID 7964925. S2CID 2491616.
- ^ Liu J, Wang R (23 March 2015). "Classification of Current Scoring Functions". Journal of Chemical Information and Modeling. 55 (3): 475–482. doi:10.1021/ci500731a. PMID 25647463.
- ^ Ajay, Murcko MA (1995). "Computational methods to predict binding free energy in ligand-receptor complexes". J. Med. Chem. 38 (26): 4953–67. doi:10.1021/jm00026a001. PMID 8544170.
- ^ Gramatica P (2011). "Chapter 17: Modeling Chemicals in the Environment". In Gramatica P, Livingstone DJ, Davis AM (eds.). Drug Design Strategies: Quantitative Approaches. RSC Drug Discovery. Royal Society of Chemistry. p. 466. doi:10.1039/9781849733410-00458. ISBN 978-1849731669.
- ^ Greer J, Erickson JW, Baldwin JJ, Varney MD (Apr 1994). "Application of the three-dimensional structures of protein target molecules in structure-based drug design". Journal of Medicinal Chemistry. 37 (8): 1035–54. doi:10.1021/jm00034a001. PMID 8164249.
- ^ Timmerman H, Gubernator K, Böhm HJ, Mannhold R, Kubinyi H (1998). Structure-based Ligand Design (Methods and Principles in Medicinal Chemistry). Weinheim: Wiley-VCH. ISBN 978-3-527-29343-8.
- ^ Capdeville R, Buchdunger E, Zimmermann J, Matter A (Jul 2002). "Glivec (STI571, imatinib), a rationally developed, targeted anticancer drug". Nature Reviews. Drug Discovery. 1 (7): 493–502. doi:10.1038/nrd839. PMID 12120256. S2CID 2728341.
- ^ "AutoDock's role in Developing the First Clinically-Approved HIV Integrase Inhibitor". Press Release. The Scripps Research Institute. 2007-12-17.
- ^ Klein DF (Mar 2008). "The loss of serendipity in psychopharmacology". JAMA. 299 (9): 1063–5. doi:10.1001/jama.299.9.1063. PMID 18319418.


![{\displaystyle {\begin{array}{lll}\Delta G_{\text{bind}}=-RT\ln K_{\text{d}}\\[1.3ex]K_{\text{d}}={\dfrac {[{\text{Ligand}}][{\text{Receptor}}]}{[{\text{Complex}}]}}\\[1.3ex]\Delta G_{\text{bind}}=\Delta G_{\text{desolvation}}+\Delta G_{\text{motion}}+\Delta G_{\text{configuration}}+\Delta G_{\text{interaction}}\end{array}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/ba49ddd9dec7415d129787213744ca1afcd2d021)


