가상 선별
Virtual screening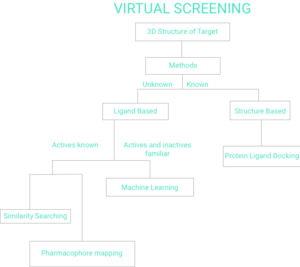
가상 선별(VS)은 약물 표적(일반적으로 단백질 수용체 또는 효소)에 가장 결합할 가능성이 높은 구조를 식별하기 위해 작은 분자의 라이브러리를 검색하기 위해 약물 발견에 사용되는 연산 기법이다.[2][3]null
가상 선별은 컴퓨터 프로그램을 사용하여 "대형 복합 라이브러리를 자동으로 평가"하는 것으로 정의되었다.[4]이 정의에서 알 수 있듯이 VS는 주로 10개60 이상의 상상할 수 있는[5] 화합물의 거대한 화학 공간을 어떻게 합성, 구매 및 테스트가 가능한 관리 가능한 숫자로 필터링할 수 있는지에 초점을 맞춘 숫자 게임이었다.전체 화학적 우주를 탐색하는 것이 이론적으로 흥미로운 문제일 수 있지만, 보다 실용적인 VS 시나리오는 표적 결합 라이브러리 설계 및 최적화와 사내 복합 저장소 또는 공급업체 제공물에서 이용할 수 있는 화합물의 라이브러리를 강화하는데 초점을 맞추고 있다.수법의 정확도가 높아지면서 가상 심사는 마약 발견 과정의 필수 요소가 됐다.[6][1]가상 선별은 선별을 위해 사내 데이터베이스 화합물을 선택하고, 외부에서 구입할 수 있는 화합물을 선택하며, 다음에 합성할 화합물을 선택하는 데 사용할 수 있다.null
방법들
선별 기법에는 리간드 기반과 구조 기반이라는 두 가지 범주가 있다.[7]이 페이지의 나머지 부분에는 가상 선별의 그림 1 흐름도가 반영된다.null
리간드 기반 방법
수용체에 결합하는 구조적으로 다양한 리간드의 집합이 주어진다면, 그러한 리간드의 집합에 포함된 집합적인 정보를 이용하여 수용체의 모델을 구축할 수 있다.다른 계산 기법들은 특정한 분자 수용체나 세포 라인에 대한 작용 방식을 암시할 수 있는 다른 리간드의 구조, 전자, 분자 모양 및 물리화학 유사성을 탐구한다.[8]후보 리간드는 이후 약리학적 모델과 비교하여 그것이 그것과 호환되는지 그리고 따라서 결합될 가능성이 있는지를 판단할 수 있다.[9]활성 리간드를 찾기 위해 데이터베이스를 스캔하기 위해 다양한 2D 화학 유사성 분석 방법이[10] 사용되어 왔다.리간드에 기반한 가상 선별에 사용되는 또 다른 일반적인 접근법은 알려진 활성제와 유사한 모양의 분자를 검색하는 것이다. 그러한 분자는 대상의 결합 부위에 적합하게 되어 대상을 묶을 가능성이 있기 때문이다.문헌에는 이 종류의 기법을 전진적으로 응용하는 것이 많이 있다.[11][12][13]이러한 3D 방법의 약리학적 확장 또한 웹서버로서 자유롭게 이용할 수 있다.[14][15]또한 형태 기반 가상 심사는 상당한 인기를 얻었다.[16]null
구조 기반 방법
구조 기반 가상 선별 접근방식은 조사된 활성 리간드의 분자 대상인 수용체 구조를 고려하는 다양한 계산 기법을 포함한다.이러한 기법으로는 분자 도킹, 구조 기반 약리학적 예측, 분자 역학 시뮬레이션 등이 있다.[17][18][8]분자 도킹은 가장 많이 사용되는 구조 기반 기법으로 각 리간드가 고분자 수용체 결합 부위에 맞닿아 적합성을 추정하는 점수 기능을 적용해 친화력이 가장 높은 리간드를 선택하는 데 도움을 준다.[19][20][21]현재 예비 가상 선별을 지향하는 웹서버가 있다.[22][23]null
혼성법
기존 VLS 접근법의 한계를 극복하기 위해 구조와 리간드 유사성에 의존하는 하이브리드 방식도 개발되었다.이 방법론은 진화 기반 리간드 바인딩 정보를 활용하여 소분자 바인더를[24][25] 예측하고 글로벌 구조 유사성과 포켓 유사성 모두를 사용할 수 있다.[24]글로벌 구조 유사성 기반 접근방식은 PDB 홀로템플릿 라이브러리에서 단백질과의 구조적 유사성을 찾기 위해 실험 구조 또는 예측 단백질 모델을 모두 사용한다.상당한 구조적 유사성을 감지하면 선택된 홀로 PDB 템플릿에서 추출한 리간드와 유사한 소분자 화면에는 2D 지문 기반 타니모토 계수 메트릭이 적용된다.[26][27]이 방법의 예측은 실험적으로 평가되었고 활성 소분자를 식별하는데 좋은 농축을 보여준다.null
위에서 지정한 방법은 전지구적 구조 유사성에 따라 달라지며 관심 단백질에서 특정 리간드-벤딩 부위를 선택할 수 없다.또한 이 방법은 리간드에 대한 2D 유사성 평가에 의존하기 때문에 실질적으로 다르지만 기하학적 형상 유사성을 보이는 소분자의 입체화학 유사성을 인식할 수 없다.이러한 우려를 해소하기 위해 홀로 단백질 템플릿에서 특정 바인딩 포켓을 타깃으로 할 수 있는 새로운 포켓 중심 접근 방식인 PoLi를 개발하여 실험적으로 평가하였다.null
컴퓨팅 인프라
많은 가상 선별 프로그램의 작동을 위한 전제 조건인 원자 간의 쌍방향 상호작용 계산은 ( ) 로 척도되며 N은 시스템 내 원자의 수입니다.이차적 스케일링으로 계산 비용이 빠르게 증가한다.null
리간드 기반 접근법
리간드 기반 방법은 일반적으로 단일 구조 비교 연산을 위해 1초의 단위가 필요하다.때로는 CPU 한 개면 몇 시간 안에 큰 검사를 수행할 수 있다.그러나, 대규모 화합물 데이터베이스를 신속하게 처리하기 위해 여러 가지 비교가 병행될 수 있다.null
구조 기반 접근법
작업의 크기는 Linux 시스템 클러스터와 같은 병렬 컴퓨팅 인프라가 필요하며, Sun Grid Engine이나 Torque PBS와 같은 작업을 처리하기 위한 배치 대기열 프로세서를 실행해야 한다.null
대형 복합 라이브러리의 입력을 처리하는 수단이 필요하다.이를 위해서는 병렬 클러스터로 쿼리할 수 있는 복합 데이터베이스의 형태가 필요하며, 다양한 컴퓨팅 노드와 병렬로 화합물을 전달한다.상업용 데이터베이스 엔진은 너무 무거울 수 있고, 버클리 DB와 같은 고속 인덱싱 엔진이 더 나은 선택일 수 있다.또한 클러스터 노드의 상승 시간이 유용한 작업의 양을 쉽게 초과할 수 있기 때문에 작업당 하나의 비교를 실행하는 것은 효율적이지 않을 수 있다.이 문제를 해결하려면 각 클러스터 작업에서 화합물 배치를 처리하여 결과를 일종의 로그 파일로 집계해야 한다.로그 파일을 채굴하고 높은 점수를 얻은 후보를 추출하는 보조 프로세스는 전체 실험이 실행된 후에 실행될 수 있다.null
정확도
가상 선별의 목적은 관심 대상인 고분자 대상과 결합하는 새로운 화학 구조의 분자를 식별하는 것이다.따라서 가상 화면의 성공은 총 조회수보다는 흥미로운 새로운 비계를 찾는 관점에서 정의된다.따라서 가상 선별 정확도에 대한 해석을 주의하여 고려해야 한다.흥미로운 비계의 낮은 적중률은 이미 알려진 비계의 높은 적중률보다 분명히 선호된다.null
문헌에서 가상 선별 연구의 대부분의 테스트는 회고적이다.이러한 연구에서 VS 기법의 성능은 훨씬 더 높은 비율의 가정된 부정행위나 미끼가 들어 있는 도서관에서 관심 대상(활성 분자 또는 단지 활성 분자)에 대한 친화력을 가지고 이전에 알려진 작은 일련의 분자를 회수하는 능력에 의해 측정된다.해당 활성 분자의[28] 성질을 매칭하여 데코이를 선택하는 방법에는 몇 가지가 뚜렷이 있으며, 최근에는 더 최근에는 특성 비일관적으로 데코이를 선택하기도 한다.[29]훈련용이든 시험용이든 미끼선택의 실제 영향도 논의됐다.[29][30]null
이와는 대조적으로 가상 선별의 전진적 적용에서는 결과 적중률이 실험 확인(예50: IC 측정)을 받는다.소급 벤치마킹은 미래 성과에 대한 좋은 예측 변수가 아니며, 따라서 오직 선행 연구만이 특정 대상에 대한 기법의 적합성에 대한 결정적인 증거를 구성한다는 데 의견이 일치한다.[31][32][33][34][35]null
약물 발견에 대한 적용
가상 선별은 약용 화학의 시작으로서 히트 분자를 식별하는 데 있어 매우 유용한 응용 프로그램이다.가상 선별 접근법이 약용 화학 산업 내에서 더욱 중요하고 실질적인 기술이 되기 시작하면서, 그 접근법은 급속도로 증가해왔다.[36]null
리간드 기반 방법
구조는 알 수 없지만 리간드가 수용체에 어떻게 결합할지를 예측하려고 한다.약리학적 특징을 사용하여 각 리간드는 기증자와 수용자를 식별했다.동일한 형상이 중첩되지만, 단 하나의 정확한 해결책이 있을 가능성은 낮다.[1]null
약리학적 모형
이 기법은 참조 화합물과 달리 설명자 및 계수가 동일하지만 활성 화합물이 다른 것을 사용하여 검색 결과를 병합할 때 사용된다.이 기법은 다양한 행위와 관련하여 가장 정확한 수행과 함께 하나의 기준 구조를 사용하는 것보다 더 효율적이기 때문에 유익하다.[1]null
약리학적 특성은 생물학적 반응을 촉진하기 위해 생물학적 표적 구조와 최적의 초분자적 상호작용 또는 상호작용을 하는 데 필요한 강직성과 전자적 특징의 앙상블이다.행위자 집합으로 대표자를 선택하라. 대부분의 방법은 유사한 바인딩을 찾을 것이다.[37]여러 개의 단단한 분자를 갖는 것이 선호되고 리간드를 다양화해야 하며, 다시 말해 결합 단계에서는 발생하지 않는 다른 특징을 갖도록 해야 한다.[1]null
형상 기반 가상 선별
형태 기반 분자 유사성 접근법은 중요하고 인기 있는 가상 선별 기법으로 확립되었다.현재 고도로 최적화된 스크리닝 플랫폼 ROCS(Rapid Overlay of Chemical Structures)는 형상 기반, 리간드 중심 가상 스크리닝의 사실상의 산업 표준으로 간주되고 있다.[38][39][40]가우스 함수를 사용하여 작은 유기 분자의 분자량을 정의한다.쿼리 순응의 선택은 덜 중요하므로 리간드 기반 모델링에 이상적인 형태 기반 선별:쿼리에 대한 생체적합성 순응의 가용성이 스크리닝의 제한 요인이 아니기 때문에, 스크리닝 성능에 결정적인 것은 더 많은 쿼리 컴파운드의 선택이다.[16]null
정량적 구조 활동 관계
정량적 구조 활동 관계(QSAR) 모델은 알려진 활성 및 알려진 비활성 화합물 집합에서 추출한 정보에 기초한 예측 모델로 구성된다.[41]데이터를 정성적으로 처리하고 구조 등급 및 둘 이상의 바인딩 모드와 함께 사용할 수 있는 SAR(구조 활동 관계)모델은 납 발견을 위해 화합물의 우선순위를 정한다.[1]null
머신러닝 알고리즘
머신러닝 알고리즘은 가상 선별 접근법에 널리 사용되어 왔다.감독된 학습 기법은 알려진 활성 및 알려진 비활성 화합물로 구성된 교육 및 테스트 데이터 세트를 사용한다.재귀 파티셔닝, 지원 벡터 머신, k-nearly neighbors, 신경망 등 가상 선별 전략에 성공하면서 서로 다른 ML 알고리즘이 적용됐다.[42][43][44]이 모델들은 화합물이 활성 상태일 확률을 찾은 다음 그 확률을 기초로 각 화합물의 순위를 매긴다.[1]null
기계학습의 하부구조해석
대규모 데이터셋에 사용되는 첫 번째 머신러닝 모델은 1973년에 만들어진 서브시스템 분석이다.각 단편적 하부 구조는 특정 유형의 활동을 지속적으로 기여한다.[1]하부구조는 의약품 설계에서 구조물을 분석하는 데 있어 대규모 차원성의 난이도를 극복하는 방식이다.다층건물이나 주탑과 유사성이 있는 구조물에 대해서는 효율적인 하부구조 분석이 사용된다.기하학은 시작과 클라이맥스를 향해 주어진 구조물에 대한 경계 이음부의 번호를 매기기 위해 사용된다.특수한 정적 응축 및 대체 루틴의 방법이 개발되었을 때, 이 방법은 이전의 하부구조 분석 모델보다 더 생산적인 것으로 입증된다.[45]null
재귀 파티셔닝
재귀 파티셔닝은 질적 데이터를 사용하여 의사결정 트리를 만드는 방법이다.적절한 분할이 없을 때까지 각 단계를 반복하면서 잘못된 분류의 낮은 오류로 클래스를 분할하는 방법을 이해하는 것.그러나 재귀 파티셔닝은 같은 속도로 잠재적으로 미세한 모델을 만들 수 있는 예측 능력이 떨어질 수 있다.[1]null
구조 기반 방법 알려진 단백질 리간드 도킹
리간드는 우선순위를 지정하면서 개별 리간드에 가장 가능성이 높은 원인을 파악하기 위해 도킹 검색 알고리즘과 채점 기능을 사용하여 단백질 내 활성 사이트에 바인딩할 수 있다.[1][46]null
참고 항목
참조
- ^ a b c d e f g h i j Gillet V (2013). "Ligand-Based and Structure-Based Virtual Screening" (PDF). The University of Sheffield.
- ^ Rester U (July 2008). "From virtuality to reality - Virtual screening in lead discovery and lead optimization: a medicinal chemistry perspective". Current Opinion in Drug Discovery & Development. 11 (4): 559–68. PMID 18600572.
- ^ Rollinger JM, Stuppner H, Langer T (2008). "Virtual screening for the discovery of bioactive natural products". Natural Compounds as Drugs Volume I. Progress in Drug Research. Fortschritte der Arzneimittelforschung. Progrès des Recherches Pharmaceutiques. Progress in Drug Research. Vol. 65. pp. 211, 213–49. doi:10.1007/978-3-7643-8117-2_6. ISBN 978-3-7643-8098-4. PMC 7124045. PMID 18084917.
- ^ Walters WP, Stahl MT, Murcko MA (1998). "Virtual screening – an overview". Drug Discov. Today. 3 (4): 160–178. doi:10.1016/S1359-6446(97)01163-X.
- ^ Bohacek RS, McMartin C, Guida WC (1996). "The art and practice of structure-based drug design: a molecular modeling perspective". Med. Res. Rev. 16 (1): 3–50. doi:10.1002/(SICI)1098-1128(199601)16:1<3::AID-MED1>3.0.CO;2-6. PMID 8788213.
- ^ McGregor MJ, Luo Z, Jiang X (June 11, 2007). "Chapter 3: Virtual screening in drug discovery". In Huang Z (ed.). Drug Discovery Research. New Frontiers in the Post-Genomic Era. Wiley-VCH: Weinheim, Germany. pp. 63–88. ISBN 978-0-471-67200-5.
- ^ McInnes C (October 2007). "Virtual screening strategies in drug discovery". Current Opinion in Chemical Biology. 11 (5): 494–502. doi:10.1016/j.cbpa.2007.08.033. PMID 17936059.
- ^ a b Santana K, do Nascimento LD, Lima e Lima A, Damasceno V, Nahum C, Braga RC, Lameira J (2021-04-29). "Applications of Virtual Screening in Bioprospecting: Facts, Shifts, and Perspectives to Explore the Chemo-Structural Diversity of Natural Products". Frontiers in Chemistry. 9: 662688. Bibcode:2021FrCh....9..155S. doi:10.3389/fchem.2021.662688. ISSN 2296-2646. PMC 8117418. PMID 33996755.
- ^ Sun H (2008). "Pharmacophore-based virtual screening". Current Medicinal Chemistry. 15 (10): 1018–24. doi:10.2174/092986708784049630. PMID 18393859.
- ^ Willet P, Barnard JM, Downs GM (1998). "Chemical similarity searching". Journal of Chemical Information and Computer Sciences. 38 (6): 983–996. CiteSeerX 10.1.1.453.1788. doi:10.1021/ci9800211.
- ^ Rush TS, Grant JA, Mosyak L, Nicholls A (March 2005). "A shape-based 3-D scaffold hopping method and its application to a bacterial protein-protein interaction". Journal of Medicinal Chemistry. 48 (5): 1489–95. CiteSeerX 10.1.1.455.4728. doi:10.1021/jm040163o. PMID 15743191.
- ^ Ballester PJ, Westwood I, Laurieri N, Sim E, Richards WG (February 2010). "Prospective virtual screening with Ultrafast Shape Recognition: the identification of novel inhibitors of arylamine N-acetyltransferases". Journal of the Royal Society, Interface. 7 (43): 335–42. doi:10.1098/rsif.2009.0170. PMC 2842611. PMID 19586957.
- ^ Kumar A, Zhang KY (2018). "Advances in the Development of Shape Similarity Methods and Their Application in Drug Discovery". Frontiers in Chemistry. 6: 315. Bibcode:2018FrCh....6..315K. doi:10.3389/fchem.2018.00315. PMC 6068280. PMID 30090808.
- ^ Li H, Leung KS, Wong MH, Ballester PJ (July 2016). "USR-VS: a web server for large-scale prospective virtual screening using ultrafast shape recognition techniques". Nucleic Acids Research. 44 (W1): W436–41. doi:10.1093/nar/gkw320. PMC 4987897. PMID 27106057.
- ^ Sperandio O, Petitjean M, Tuffery P (July 2009). "wwLigCSRre: a 3D ligand-based server for hit identification and optimization". Nucleic Acids Research. 37 (Web Server issue): W504–9. doi:10.1093/nar/gkp324. PMC 2703967. PMID 19429687.
- ^ a b Kirchmair J, Distinto S, Markt P, Schuster D, Spitzer GM, Liedl KR, Wolber G (2009). "How To Optimize Shape-Based Virtual Screening: Choosing the Right Query and Including Chemical Information". Journal of Chemical Information and Modeling. 49: 678–692. doi:10.1021/ci8004226.
- ^ Toledo Warshaviak D, Golan G, Borrelli KW, Zhu K, Kalid O (July 2014). "Structure-based virtual screening approach for discovery of covalently bound ligands". Journal of Chemical Information and Modeling. 54 (7): 1941–50. doi:10.1021/ci500175r. PMID 24932913.
- ^ Maia EH, Assis LC, de Oliveira TA, da Silva AM, Taranto AG (2020-04-28). "Structure-Based Virtual Screening: From Classical to Artificial Intelligence". Frontiers in Chemistry. 8: 343. Bibcode:2020FrCh....8..343M. doi:10.3389/fchem.2020.00343. PMC 7200080. PMID 32411671.
- ^ Kroemer RT (August 2007). "Structure-based drug design: docking and scoring". Current Protein & Peptide Science. 8 (4): 312–28. CiteSeerX 10.1.1.225.959. doi:10.2174/138920307781369382. PMID 17696866.
- ^ Cavasotto CN, Orry AJ (2007). "Ligand docking and structure-based virtual screening in drug discovery". Current Topics in Medicinal Chemistry. 7 (10): 1006–14. doi:10.2174/156802607780906753. PMID 17508934.
- ^ Kooistra AJ, Vischer HF, McNaught-Flores D, Leurs R, de Esch IJ, de Graaf C (June 2016). "Function-specific virtual screening for GPCR ligands using a combined scoring method". Scientific Reports. 6: 28288. Bibcode:2016NatSR...628288K. doi:10.1038/srep28288. PMC 4919634. PMID 27339552.
- ^ Irwin JJ, Shoichet BK, Mysinger MM, Huang N, Colizzi F, Wassam P, Cao Y (September 2009). "Automated docking screens: a feasibility study". Journal of Medicinal Chemistry. 52 (18): 5712–20. doi:10.1021/jm9006966. PMC 2745826. PMID 19719084.
- ^ Li H, Leung KS, Ballester PJ, Wong MH (2014-01-24). "istar: a web platform for large-scale protein-ligand docking". PLOS ONE. 9 (1): e85678. Bibcode:2014PLoSO...985678L. doi:10.1371/journal.pone.0085678. PMC 3901662. PMID 24475049.
- ^ a b Zhou H, Skolnick J (January 2013). "FINDSITE(comb): a threading/structure-based, proteomic-scale virtual ligand screening approach". Journal of Chemical Information and Modeling. 53 (1): 230–40. doi:10.1021/ci300510n. PMC 3557555. PMID 23240691.
- ^ Roy A, Skolnick J (February 2015). "LIGSIFT: an open-source tool for ligand structural alignment and virtual screening". Bioinformatics. 31 (4): 539–44. doi:10.1093/bioinformatics/btu692. PMC 4325547. PMID 25336501.
- ^ Gaulton A, Bellis LJ, Bento AP, Chambers J, Davies M, Hersey A, Light Y, McGlinchey S, Michalovich D, Al-Lazikani B, Overington JP (January 2012). "ChEMBL: a large-scale bioactivity database for drug discovery". Nucleic Acids Research. 40 (Database issue): D1100–7. doi:10.1093/nar/gkr777. PMC 3245175. PMID 21948594.
- ^ Wishart DS, Knox C, Guo AC, Shrivastava S, Hassanali M, Stothard P, Chang Z, Woolsey J (January 2006). "DrugBank: a comprehensive resource for in silico drug discovery and exploration". Nucleic Acids Research. 34 (Database issue): D668–72. doi:10.1093/nar/gkj067. PMC 1347430. PMID 16381955.
- ^ Réau M, Langenfeld F, Zagury JF, Lagarde N, Montes M (2018). "Decoys Selection in Benchmarking Datasets: Overview and Perspectives". Frontiers in Pharmacology. 9: 11. doi:10.3389/fphar.2018.00011. PMC 5787549. PMID 29416509.
- ^ a b Ballester PJ (December 2019). "Selecting machine-learning scoring functions for structure-based virtual screening". Drug Discovery Today: Technologies. 32–33: 81–87. doi:10.1016/j.ddtec.2020.09.001. PMID 33386098. S2CID 224968364.
- ^ Li H, Sze KH, Lu G, Ballester PJ (2021). "Machine-learning scoring functions for structure-based virtual screening". WIREs Computational Molecular Science. 11 (1): e1478. doi:10.1002/wcms.1478. ISSN 1759-0884. S2CID 219089637.
- ^ Wallach I, Heifets A (2018). "Most Ligand-based classification benchmarks reward memorization rather than generalization". Journal of Chemical Information and Modeling. 58 (5): 916–932. arXiv:1706.06619. doi:10.1021/acs.jcim.7b00403. PMID 29698607. S2CID 195345933.
- ^ Irwin JJ (2008). "Community benchmarks for virtual screening". Journal of Computer-Aided Molecular Design. 22 (3–4): 193–9. Bibcode:2008JCAMD..22..193I. doi:10.1007/s10822-008-9189-4. PMID 18273555. S2CID 26260725.
- ^ Good AC, Oprea TI (2008). "Optimization of CAMD techniques 3. Virtual screening enrichment studies: a help or hindrance in tool selection?". Journal of Computer-Aided Molecular Design. 22 (3–4): 169–78. Bibcode:2008JCAMD..22..169G. doi:10.1007/s10822-007-9167-2. PMID 18188508. S2CID 7738182.
- ^ Schneider G (April 2010). "Virtual screening: an endless staircase?". Nature Reviews. Drug Discovery. 9 (4): 273–6. doi:10.1038/nrd3139. PMID 20357802. S2CID 205477076.
- ^ Ballester PJ (January 2011). "Ultrafast shape recognition: method and applications". Future Medicinal Chemistry. 3 (1): 65–78. doi:10.4155/fmc.10.280. PMID 21428826.
- ^ Lavecchia A, Di Giovanni C (2013). "Virtual screening strategies in drug discovery: a critical review". Current Medicinal Chemistry. 20 (23): 2839–60. doi:10.2174/09298673113209990001. PMID 23651302.
- ^ Spitzer GM, Heiss M, Mangold M, Markt P, Kirchmair J, Wolber G, Liedl KR (2010). "One concept, three implementations of 3D pharmacophore-based virtual screening: distinct coverage of chemical search space". Journal of Chemical Information and Modeling. 50 (7): 1241–1247. doi:10.1021/ci100136b.
- ^ Grant JA, Gallard MA, Pickup BT (1996). "A fast method of molecular shape comparison: a simple application of a Gaussian description of molecular shape". Journal of Computational Chemistry. 17: 1653–1666. doi:10.1002/(SICI)1096-987X(19961115)17:14<1653::AID-JCC7>3.0.CO;2-K.
- ^ Nicholls A, Grant JA (2005). "Molecular shape and electrostatics in the encoding of relevant chemical information". Journal of Computer-Aided Molecular Design. 19: 661–686. doi:10.1007/s10822-005-9019-x.
- ^ Rush TS, Grant JA, Mosyak L, Nicholls A (2005). "A shape-based 3-D scaffold hopping method and its application to a bacterial protein-protein interaction". Journal of Medicinal Chemistry. 48: 1489–1495. doi:10.1021/jm040163o.
- ^ Neves BJ, Braga RC, Melo-Filho CC, Moreira-Filho JT, Muratov EN, Andrade CH (2018-11-13). "QSAR-Based Virtual Screening: Advances and Applications in Drug Discovery". Frontiers in Pharmacology. 9: 1275. doi:10.3389/fphar.2018.01275. PMC 6262347. PMID 30524275.
- ^ Alsenan S, Al-Turaiki I, Hafez A (December 2020). "A Recurrent Neural Network model to predict blood-brain barrier permeability". Computational Biology and Chemistry. 89: 107377. doi:10.1016/j.compbiolchem.2020.107377. PMID 33010784.
- ^ Dimitri GM, Lió P (June 2017). "DrugClust: A machine learning approach for drugs side effects prediction". Computational Biology and Chemistry. 68: 204–210. doi:10.1016/j.compbiolchem.2017.03.008. PMID 28391063.
- ^ Shoombuatong W, Schaduangrat N, Pratiwi R, Nantasenamat C (June 2019). "THPep: A machine learning-based approach for predicting tumor homing peptides". Computational Biology and Chemistry. 80: 441–451. doi:10.1016/j.compbiolchem.2019.05.008. PMID 31151025.
- ^ Gurujee CS, Deshpande VL (February 1978). "An improved method of substructure analysis". Computers & Structures. 8 (1): 147–152. doi:10.1016/0045-7949(78)90171-2.
- ^ Pradeepkiran JA, Reddy PH (March 2019). "Structure Based Design and Molecular Docking Studies for Phosphorylated Tau Inhibitors in Alzheimer's Disease". Cells. 8 (3): 260. doi:10.3390/cells8030260. PMC 6468864. PMID 30893872.
추가 읽기
- Melagraki G, Afantitis A, Sarimveis H, Koutentis PA, Markopoulos J, Igglessi-Markopoulou O (May 2007). "Optimization of biaryl piperidine and 4-amino-2-biarylurea MCH1 receptor antagonists using QSAR modeling, classification techniques and virtual screening". Journal of Computer-Aided Molecular Design. 21 (5): 251–67. Bibcode:2007JCAMD..21..251M. doi:10.1007/s10822-007-9112-4. PMID 17377847. S2CID 19563229.
- Afantitis A, Melagraki G, Sarimveis H, Koutentis PA, Markopoulos J, Igglessi-Markopoulou O (February 2006). "Investigation of substituent effect of 1-(3,3-diphenylpropyl)-piperidinyl phenylacetamides on CCR5 binding affinity using QSAR and virtual screening techniques". Journal of Computer-Aided Molecular Design. 20 (2): 83–95. Bibcode:2006JCAMD..20...83A. CiteSeerX 10.1.1.716.8148. doi:10.1007/s10822-006-9038-2. PMID 16783600. S2CID 21523436.
- Eckert H, Bajorath J (March 2007). "Molecular similarity analysis in virtual screening: foundations, limitations and novel approaches". Drug Discovery Today. 12 (5–6): 225–33. doi:10.1016/j.drudis.2007.01.011. PMID 17331887.
- Willett P (December 2006). "Similarity-based virtual screening using 2D fingerprints" (PDF). Drug Discovery Today (Submitted manuscript). 11 (23–24): 1046–53. doi:10.1016/j.drudis.2006.10.005. PMID 17129822.
- Fara DC, Oprea TI, Prossnitz ER, Bologa CG, Edwards BS, Sklar LA (2006). "Integration of virtual and physical screening". Drug Discovery Today: Technologies. 3 (4): 377–385. doi:10.1016/j.ddtec.2006.11.003. PMC 7105924.
- Muegge I, Oloffa S (2006). "Advances in virtual screening". Drug Discovery Today: Technologies. 3 (4): 405–411. doi:10.1016/j.ddtec.2006.12.002. PMC 7105922.
- Schneider G (April 2010). "Virtual screening: an endless staircase?". Nature Reviews. Drug Discovery. 9 (4): 273–6. doi:10.1038/nrd3139. PMID 20357802. S2CID 205477076.
외부 링크
- VLS3D – 2000개 이상의 데이터베이스 목록, 실리코 툴의 온라인 및 독립 실행형



