효소
Enzyme| 시리즈의 일부 |
| 생화학 |
|---|
 |
효소(/ɛnzmmz/)는 생물학적 촉매(생물 촉매)로 작용하는 단백질이다.촉매는 화학 반응을 가속화한다.효소가 작용하는 분자는 기질이라고 불리며, 효소는 기질을 산물로 알려진 다른 분자로 변환합니다.세포 내의 거의 모든 대사 과정은 생명을 [1]: 8.1 유지하기에 충분히 빠른 속도로 일어나기 위해 효소 촉매 작용을 필요로 한다.대사 경로는 개별 단계를 촉매하기 위해 효소에 의존합니다.효소에 대한 연구는 효소학이라고 불리며, 의사 효소 분석 분야는 진화 과정에서 일부 효소가 종종 아미노산 배열과 특이한 '의사 촉매'[2][3] 특성에 반영되는 생물학적 촉매 작용을 수행하는 능력을 잃었다는 것을 인정합니다.
효소는 5,000가지 이상의 [4]생화학 반응을 촉매하는 것으로 알려져 있다.다른 생물 촉매는 리보자임이라고 불리는 촉매 RNA 분자이다.효소의 특이성은 그들의 독특한 3차원 구조에서 나온다.
모든 촉매와 마찬가지로 효소는 활성화 에너지를 낮춤으로써 반응 속도를 증가시킨다.어떤 효소는 기질에서 생산물로의 전환을 수백만 배 더 빠르게 만들 수 있다.극단적인 예로는 오로티딘 5'-인산 탈탄산화효소가 있는데, 이것은 그렇지 않으면 [5][6]밀리초 안에 수백만 년이 걸릴 반응을 가능하게 한다.화학적으로 효소는 촉매와 같으며 화학 반응에서 소비되지 않으며 반응의 평형을 변화시키지도 않습니다.효소는 훨씬 더 특이적이기 때문에 대부분의 다른 촉매들과 다르다.효소 활성은 다른 분자에 의해 영향을 받을 수 있습니다: 억제제는 효소 활성을 감소시키는 분자이고 활성제는 활성을 증가시키는 분자입니다.많은 치료약과 독은 효소 억제제이다.효소의 활성은 최적 온도와 pH를 벗어나 현저하게 감소하며, 많은 효소는 과도한 열에 노출되면 (영구적으로) 변성되어 구조와 촉매 특성을 잃는다.
일부 효소는 예를 들어 항생제 합성에 상업적으로 사용된다.일부 가정용 제품들은 화학 반응을 빠르게 하기 위해 효소를 사용한다: 생물학적 세척 분말의 효소는 단백질, 전분 또는 옷에 묻은 지방 얼룩을 분해하고, 고기 연화제의 효소는 단백질을 더 작은 분자로 분해하여 고기를 씹기 쉽게 만든다.
어원과 역사
17세기 후반과 18세기 초까지, 위[7] 분비물에 의한 육류의 소화, 식물 추출물과 침에 의한 전분의 당으로의 전환은 알려졌지만, 이러한 현상이 일어나는 메커니즘은 [8]확인되지 않았다.
프랑스의 화학자 안셀메 파옌은 1833년에 [9]디아스타아제라는 효소를 최초로 발견했다.몇 십 년 후, 효모에 의한 설탕의 알코올 발효를 연구했을 때, 루이 파스퇴르는 이 발효가 살아있는 유기체 내에서만 기능하는 것으로 생각되는 효모 세포 안에 포함된 생명력에 의해 발생했다고 결론지었다.그는 "알코올 발효는 [10]효모세포의 생명과 조직과 관련된 행동이지 세포의 죽음이나 부패와 관련이 없다"고 썼다.
1877년, 독일의 생리학자 빌헬름 퀴네(1837–1900)는 이 [11]과정을 설명하기 위해 그리스어 νυομον, "leavened" 또는 "in exym"에서 유래한 효소라는 용어를 처음 사용했다.효소라는 단어는 나중에 펩신과 같은 무생물 물질을 지칭하는 데 사용되었고 발효라는 단어는 [12]생물에 의해 생성된 화학 활동을 지칭하는 데 사용되었다.
에두아르트 부흐너는 1897년에 효모 추출물 연구에 대한 첫 논문을 제출했다.베를린 대학의 일련의 실험에서,[13] 그는 설탕이 혼합물에 살아있는 효모 세포가 없을 때에도 효모 추출물에 의해 발효된다는 것을 발견했다.그는 수크로스의 발효를 가져온 효소를 "지마아제"[14]라고 명명했다.1907년, 그는 "무세포 발효의 발견"으로 노벨 화학상을 받았다.부치너의 예를 따라, 효소는 보통 그들이 수행하는 반응에 따라 이름이 붙여진다: 접미사 -ase는 기질의 이름(예를 들어, 락타아제는 유당을 분해하는 효소) 또는 반응 유형(예를 들어, DNA 중합효소는 DNA [15]중합체를 형성한다)과 결합된다.
효소의 생화학적 정체는 1900년대 초반에도 여전히 알려지지 않았다.많은 과학자들은 효소활동이 단백질과 관련이 있다고 관찰했지만, 다른 과학자들은 단백질은 진정한 효소의 운반체일 뿐이고 단백질 자체는 촉매작용을 [16]할 수 없다고 주장했다.1926년 제임스 B. 섬너는 우레아제 효소가 순수한 단백질이라는 것을 보여주었고 그것을 결정화했다; 그는 1937년에 카탈라아제 효소에 대해서도 똑같이 했다.순수 단백질이 효소가 될 수 있다는 결론은 소화 효소인 펩신, 트립신, 키모트립신에 대해 연구한 존 하워드 노스럽과 웬델 메레디스 스탠리에 의해 확실히 입증되었다.이 세 명의 과학자들은 1946년 노벨 [17]화학상을 받았다.
효소가 결정화될 수 있다는 발견은 결국 그들의 구조를 X선 결정학으로 해결할 수 있게 해주었다.이것은 눈물, 침, 그리고 일부 박테리아의 코팅을 소화시키는 달걀 흰자에서 발견되는 효소인 리조자임을 위해 처음 행해졌다; 그 구조는 데이비드 칠튼 필립스가 이끄는 그룹에 의해 해결되었고 [18]1965년에 발표되었다.리소자임의 이 고해상도 구조는 구조 생물학 분야의 시작을 의미하고 효소가 [19]원자 수준에서 어떻게 작용하는지를 이해하기 위한 노력을 나타냅니다.
분류 및 명명법
효소는 아미노산 배열 유사성(따라서 진화적 관계) 또는 효소 활성의 두 가지 주요 기준으로 분류될 수 있습니다.
효소 활성.효소의 이름은 종종 효소의 기질 또는 효소가 촉매하는 화학 반응에서 유래하며, 효소는 [1]: 8.1.3 -ase로 끝난다.예를 들어 락타아제, 알코올탈수소효소, DNA중합효소 등이 있다.동일한 화학 반응을 촉매하는 다른 효소들은 아이소자임이라고 불린다.[1]: 10.3
국제생화학분자생물학연합은 효소에 대한 명명법, EC 번호('효소 위원회')를 개발했다.각 효소는 효소 활성의 위계를 나타내는 4개의 숫자에 이어 "EC"에 의해 설명된다(매우 일반적인 것부터 매우 특이적인 것까지).즉, 첫 번째 숫자는 그 메커니즘에 따라 효소를 광범위하게 분류하고 다른 숫자는 점점 [20]더 특이성을 더한다.
최상위 분류는 다음과 같습니다.
- EC 1, 산화환원효소: 산화/환원반응을 촉매한다.
- EC 2, Transferase: 관능기(예: 메틸기 또는 인산기)를 전달합니다.
- EC 3, 가수분해효소: 다양한 결합의 가수분해를 촉매합니다.
- EC4, 리아제: 가수분해 및 산화 이외의 방법으로 다양한 결합을 분해함
- EC5, 이성질화효소: 단일 분자 내에서 이성질화 변화를 촉매한다.
- EC 6, 연결효소: 공유 결합을 가진 두 분자를 결합합니다.
- EC 7, 전장효소: 이온 또는 분자의 막간 이동 또는 막내 분리를 촉매합니다.
이러한 섹션은 기판, 제품 및 화학 메커니즘과 같은 다른 특징에 따라 세분됩니다.효소는 4가지 수치적 명칭으로 완전히 규정된다.예를 들어 헥소키나아제(EC 2.7.1)는 알코올기(EC 2.7.1)[21]를 포함하는 분자인 헥소당에 인산기(EC 2.7)를 첨가하는 전이효소(EC 2)이다.
시퀀스 유사성EC 범주는 시퀀스 유사성을 반영하지 않습니다.예를 들어, 정확히 동일한 반응을 촉매하는 동일한 EC 번호의 두 연결 효소는 완전히 다른 서열을 가질 수 있습니다.다른 단백질과 마찬가지로 효소는 기능과는 무관하게 배열 유사성에 의해 수많은 과로 분류되어 왔다.이 가족들은 Pfam과 [22]같은 수십 개의 다른 단백질 및 단백질 패밀리 데이터베이스에 기록되었습니다.
구조.
효소는 일반적으로 단독으로 또는 더 큰 복합체로 작용하는 구상 단백질입니다.아미노산의 배열은 [23]효소의 촉매 활성을 결정하는 구조를 지정합니다.구조가 기능을 결정하지만 구조만으로는 [24]아직 새로운 효소 활성을 예측할 수 없다.효소 구조는 가열되거나 화학적 변성제에 노출되면 펼쳐지고(변성) 이러한 구조의 교란은 일반적으로 활동 [25]상실을 야기합니다.효소 변성은 보통 종의 정상 수준 이상의 온도와 관련이 있다. 그 결과, 온천과 같은 화산 환경에 사는 박테리아에서 나오는 효소는 산업 사용자들에 의해 높은 온도에서 기능하는 능력으로 귀중하게 여겨져 효소 촉매 반응이 매우 빠른 속도로 작동될 수 있다.
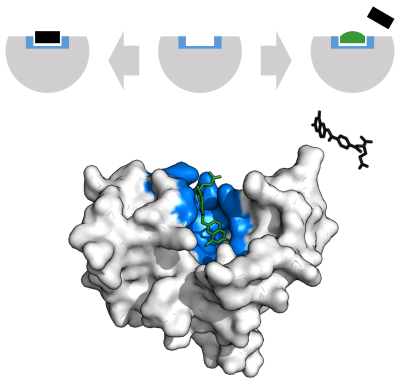
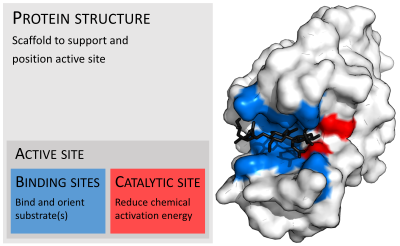
효소는 보통 그들의 기질보다 훨씬 크다.크기는 4-옥살로크로토네이트 호토머라아제의 [26]단량체에 대해 62개의 아미노산 잔류물부터 동물성 지방산 [27]합성효소 내 2,500개 이상의 잔류물까지 다양하다.촉매 [28]작용에는 구조의 극히 일부(약 2-4개의 아미노산)만 직접적으로 관여한다.이 촉매 부위는 잔류물이 기판의 방향을 결정하는 하나 이상의 결합 부위 옆에 있습니다.촉매 부위와 결합 부위가 함께 효소의 활성 부위를 구성합니다.효소 구조의 나머지 대부분은 활성 [29]부위의 정확한 방향과 역학을 유지하는 역할을 합니다.
일부 효소에서는 아미노산이 촉매 작용에 직접 관여하지 않으며, 대신 촉매 보조 [29]인자와 결합하고 방향을 지정하는 부위가 포함되어 있습니다.효소 구조는 또한 작은 분자의 결합이 [30]활성을 증가시키거나 감소시키는 구조 변화를 일으키는 알로스테릭 부위를 포함할 수 있다.
리보자임이라고 불리는 소수의 RNA 기반 생물학적 촉매가 존재하며, 리보자임은 다시 단백질과 단독으로 또는 복합적으로 작용할 수 있다.이들 중 가장 일반적인 것은 단백질과 촉매 RNA [1]: 2.2 성분의 복합체인 리보솜이다.
메커니즘
기판 결합
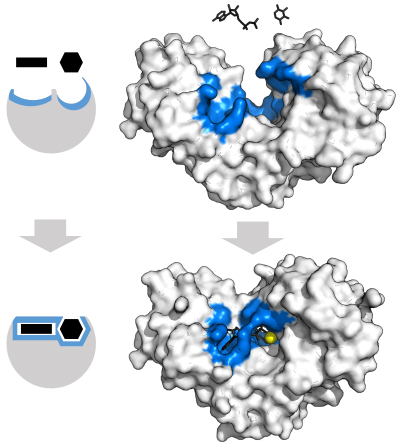
효소는 화학 반응을 촉매하기 전에 기질을 결합해야 한다.효소는 보통 어떤 기질에 결합하고 화학반응이 촉매작용을 하는지에 대해 매우 특이하다.특이성은 상보적인 형상, 전하 및 친수성/소수성 특성을 가진 포켓을 기질에 결합함으로써 달성된다.따라서 효소는 화학선택적, 위치선택적 및 입체특이적으로 [31]매우 유사한 기질분자를 구별할 수 있다.
가장 높은 특이성과 정확성을 보이는 효소 중 일부는 게놈의 복제와 발현에 관여한다.이러한 효소들 중 일부는 "교정" 메커니즘을 가지고 있다.여기서 DNA 중합효소 등의 효소는 제1공정에서 반응을 촉매한 후 [32]제2공정에서 제품이 올바른지 확인한다.이 2단계 공정은 고충실성 포유동물 중합효소 [1]: 5.3.1 반응에서 1억 번의 반응에서 평균 오차율이 1 미만인 결과를 초래한다.RNA 중합효소,[33] 아미노아실 tRNA 합성효소[34] 및 [35]리보솜에서도 유사한 교정 메커니즘이 발견된다.
반대로, 일부 효소는 효소 문란성을 나타내며, 광범위한 특이성을 가지며 생리적으로 관련된 다양한 기질에 작용한다.많은 효소들은 우연히(즉, 중성적으로) 발생한 작은 부작용을 가지고 있으며, 이것은 [36][37]새로운 기능의 진화적 선택의 출발점이 될 수 있다.
"잠금 및 열쇠" 모델
효소의 관찰된 특이성을 설명하기 위해, Emil Fischer는 1894년에 효소와 기질 둘 다 서로 [38]정확히 맞는 특정한 상호보완적 기하학적 형태를 가지고 있다고 제안했다.이것은 종종 "잠금 및 열쇠"[1]: 8.3.2 모델이라고 불립니다.이 초기 모델은 효소 특이성을 설명하지만 효소가 달성하는 [39]전이 상태의 안정화를 설명하지는 못합니다.
유도적합모델
1958년 다니엘 코슬랜드는 잠금 및 키 모델에 대한 수정을 제안했다. 효소는 다소 유연한 구조이기 때문에 활성 부위는 기질이 [40]효소와 상호작용할 때 기질과의 상호작용에 의해 지속적으로 재형성된다.그 결과 기질은 단순히 단단한 활성 부위에 결합하는 것이 아니라 활성 부위를 구성하는 아미노산 측쇄를 효소가 촉매 기능을 수행할 수 있는 정확한 위치로 성형한다.글리코시다아제와 같은 경우에 기질 분자는 활성 [41]부위에 들어갈 때 모양도 약간 변한다.활성 부위는 기판이 완전히 결합될 때까지 계속 변화하며, 이 시점에서 최종 모양과 전하 분포가 결정됩니다.[42]유도 적합은 구조 교정 [43]메커니즘을 통해 경쟁과 소음이 존재하는 경우 분자 인식의 충실도를 높일 수 있다.
촉매 작용
효소는 여러 가지 방법으로 반응을 가속할 수 있으며, 이 모든 것이 활성화 에너지(δG‡, 깁스 [44]자유 에너지)를 낮춥니다.
- 전환 상태를 안정화함으로써:
- 전이 상태의 전하 분포를 보완하는 전하 분포를 가진 환경을 조성하여 에너지를[45] 절감합니다.
- 대체 반응 경로를 제공함으로써:
- 기판과 일시적으로 반응하여 낮은 에너지 전이[46] 상태를 제공하기 위해 공유 중간체를 형성
- 기판 접지 상태를 불안정하게 함으로써:
효소는 이 메커니즘들 중 몇 가지를 동시에 사용할 수 있다.예를 들어 트립신 등의 단백질 분해효소는 촉매삼합체를 사용하여 공유 촉매작용을 수행하고, 옥시아니온 구멍을 사용하여 전이상태에서 전하 축적을 안정화하며, 배향수기질을 [50]사용하여 완전 가수분해한다.
다이내믹스
효소는 단단하고 정적인 구조가 아니다; 대신에 그들은 개별 아미노산 잔류물, 단백질 루프 또는 2차 구조의 단위를 형성하는 잔류물 그룹, 또는 전체 단백질 도메인과 같은 효소 구조의 일부의 움직임을 가지고 있다.이러한 움직임은 평형상태에서 서로 상호 변환하는 약간 다른 구조의 입체구조적 앙상블을 일으킨다.이 앙상블 내의 다른 상태는 효소 기능의 다른 측면과 연관될 수 있다.예를 들어 효소 디히드로폴산 환원효소의 다른 배치는 촉매 [51]공명 이론과 일치하는 촉매 회로의 기질 결합, 촉매 작용, 보조 인자 방출 및 생성물 방출 단계와 관련된다.
기판 프레젠테이션
기질 표시는 효소가 기질에서 격리되는 과정이다.효소는 핵 또는 세포질의 기질에서 떨어져 혈장막으로 격리될 수 있다.또는 막 내에서 효소는 그 기질에서 무질서한 영역에서 떨어진 지질 뗏목 안에 격리될 수 있다.효소가 방출되면 그것은 기질과 섞인다.또는 효소를 그 기질 근방에 격리하여 활성화시킬 수 있다.예를 들어 효소는 용해될 수 있으며 활성화 시 플라즈마막의 지질에 결합하고 플라즈마막의 분자에 작용한다.
알로스테릭 변조
알로스테릭 부위는 활성 부위와 구별되는 효소의 포켓으로 세포 환경의 분자에 결합합니다.그런 다음 이러한 분자는 활성 부위로 전달되는 효소의 구조 또는 역학에 변화를 일으켜 [52]효소의 반응 속도에 영향을 미친다.이런 식으로, 알로스테릭 상호작용은 효소를 억제하거나 활성화 시킬 수 있다.효소의 대사 경로에서 상류 또는 하류 대사물과의 알로스테릭 상호작용은 피드백 조절을 유발하며,[53] 나머지 경로를 통과하는 플럭스에 따라 효소의 활성을 변화시킨다.
보조 요인
일부 효소는 완전한 활성을 나타내기 위해 추가 성분이 필요하지 않습니다.다른 것들은 보조 인자라고 불리는 비단백질 분자가 [54]활성에 결합되어야 합니다.보조 인자는 무기(예: 금속 이온 및 철-황 클러스터) 또는 유기 화합물(예: 플라빈 및 헴)일 수 있습니다.이러한 보조 인자는 여러 가지 용도로 사용됩니다. 예를 들어, 금속 이온은 활성 [55]부위 내에서 친핵성 종을 안정시키는 데 도움이 될 수 있습니다.유기 보조 인자는 반응 중에 효소의 활성 부위에서 방출되는 조효소 또는 효소에 단단히 결합되어 있는 보철물 그룹일 수 있습니다.유기 보철기는 공유 결합될 수 있다(예를 들어 피루브산 카르복실화효소 [56]등의 효소의 비오틴).
보조 인자를 포함하는 효소의 예로는 활성 [57]부위의 일부로 결합된 아연 보조 인자를 사용하는 탄산 무수분해효소가 있습니다.이러한 단단하게 결합된 이온 또는 분자는 일반적으로 활성 부위에서 발견되며 촉매 [1]: 8.1.1 작용에 관여합니다.예를 들어 플라빈과 헴 보조 인자는 산화환원 반응에 [1]: 17 관여하는 경우가 많다.
보조 인자를 요구하지만 하나의 결합을 가지지 않는 효소는 아포엔자임 또는 아포단백질이라고 불린다.활성에 필요한 보조 인자와 함께 효소를 홀로엔자임(또는 할로엔자임)이라고 한다.홀로엔자임이라는 용어는 DNA 중합효소처럼 여러 단백질 서브유닛을 포함하는 효소에도 적용될 수 있다. 여기서 홀로엔자임은 활성에 [1]: 8.1.1 필요한 모든 서브유닛을 포함하는 완전한 복합체이다.
코엔자임
코엔자임은 효소에 느슨하거나 단단하게 결합될 수 있는 작은 유기 분자입니다.코엔자임은 한 효소에서 다른 [58]효소로 화학기를 운반한다.예로는 NADH, NADPH 및 아데노신 삼인산(ATP)이 있다.플라빈 모노뉴클레오티드(FMN), 플라빈 아데닌 디뉴클레오티드(FAD), 티아민 피로인산염(TPP) 및 테트라히드로폴레이트(THF)와 같은 일부 조효소는 비타민에서 유래한다.이러한 코엔자임은 체내에서 합성될 수 없으며, 밀접하게 관련된 화합물(비타민)은 식단에서 획득되어야 한다.운반되는 화학 그룹은 다음과 같습니다.
- NAD 또는+ NADP에 의해 운반되는 수소 이온(H−)
- 아데노신 삼인산에 의해 운반되는 인산기
- 조효소 A에 의해 운반되는 아세틸기
- 엽산에 의해 운반되는 포르밀, 메테닐 또는 메틸기
- S-아데노실메티오닌에[58] 의해 운반되는 메틸기
조효소는 효소 작용의 결과로 화학적으로 변화하기 때문에, 조효소를 많은 다른 효소에 공통적인 특별한 종류의 기질 또는 두 번째 기질로 간주하는 것이 유용하다.예를 들어, 약 1000개의 효소가 코엔자임 [59]NADH를 사용하는 것으로 알려져 있다.
코엔자임은 보통 지속적으로 재생되고 그 농도는 세포 내에서 일정한 수준으로 유지됩니다.예를 들어 NADPH는 메티오닌아데노실전달효소(methionine adenosyl transferase)에 의해 펜토오스인산 경로 및 S-아데노실메티오닌을 통해 재생된다.이러한 지속적인 재생은 소량의 코엔자임이 매우 집중적으로 사용될 수 있다는 것을 의미합니다.예를 들어, 인간의 몸은 매일 [60]자신의 몸무게를 ATP로 바꾼다.
열역학
모든 촉매와 마찬가지로 효소는 반응의 화학적 평형 위치를 바꾸지 않는다.효소가 있을 때, 반응은 효소가 없을 때와 같은 방향으로 더 [1]: 8.2.3 빠르게 진행됩니다.예를 들어, 무수탄산가수분해효소는 반응물의 [61]농도에 따라 어느 방향으로든 반응을 촉매한다.
-
(1)
-
(2)
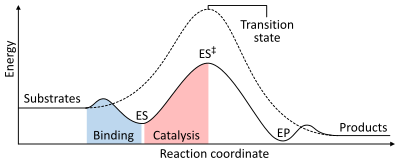
반응 속도는 전이 상태를 형성하기 위해 필요한 활성화 에너지에 따라 달라지며, 이 에너지는 생성물로 분해됩니다.효소는 전이 상태의 에너지를 낮춤으로써 반응 속도를 증가시킨다.우선 결합은 저에너지 효소-기질 복합체(ES)를 형성한다.둘째, 효소는 비촉매 반응(ES‡)에 비해 도달하는 데 더 적은 에너지를 필요로 하는 전이 상태를 안정화시킨다.마지막으로 효소 생성물 복합체(EP)가 분해되어 [1]: 8.3 생성물을 방출한다.
효소는 두 개 이상의 반응을 결합할 수 있으며, 따라서 열역학적으로 유리한 반응이 열역학적으로 불리한 반응을 "구동"하기 위해 사용될 수 있으므로 생성물의 결합된 에너지는 기질보다 낮다.예를 들어, ATP의 가수분해는 종종 다른 화학 [62]반응을 일으키는데 사용된다.
동력학
효소 동태는 효소가 기질을 결합하고 어떻게 [63]기질을 생산물로 변화시키는지에 대한 연구이다.운동 분석에 사용되는 속도 데이터는 일반적으로 효소 분석에서 얻습니다.1913년에 레오노르 미카엘리스와 모드 레오노라 멘텐은 미카엘리스-멘텐 [64]동역학의 양적 이론을 제안했다.Michaelis와 Menten의 주요 공헌은 효소 반응을 두 단계로 생각하는 것이었다.우선 기질은 효소에 가역적으로 결합해 효소-기질 복합체를 형성한다.이것은 때때로 그들의 명예로 미카엘리스-멘텐 콤플렉스라고 불린다.효소는 반응의 화학적 단계를 촉매하고 생성물을 방출합니다.이 연구는 G. E. Briggs와 J. B. S. Haldane에 의해 추가로 개발되었으며,[65] 그는 오늘날에도 여전히 널리 사용되는 운동 방정식을 도출했다.
효소율은 용액 상태와 기질 농도에 따라 달라집니다.효소반응의 최대속도를 구하기 위해 일정한 생성속도가 보일 때까지 기질농도를 증가시킨다.이는 오른쪽에 있는 포화 곡선에 표시됩니다.포화는 기질 농도가 증가함에 따라 유리효소가 기질 결합 ES 복합체로 점점 더 많이 전환되기 때문에 발생한다.효소의 최대반응속도(Vmax)에서는 모든 효소활성부위가 기질에 결합되어 ES복합체의 양이 [1]: 8.4 효소의 총량과 동일하다.
V는max 몇 가지 중요한 운동 파라미터 중 하나일 뿐이다.주어진 반응 속도를 달성하기 위해 필요한 기질의 양 또한 중요하다.이것은 효소가 최대 반응 속도의 절반에 도달하는 데 필요한 기질 농도인 Michaelis-Menten 상수(Km)에 의해 주어지며, 일반적으로 각 효소는 주어진 기질에 대해 특징적인M K를 가진다.또 다른 유용한 상수는 회전수라고도 불리는 k로cat,[1]: 8.4 이것은 초당 하나의 활성 부위에서 처리되는 기질 분자의 수입니다.
효소의 효율은 k/K로catm 나타낼 수 있다.이것은 특이성 상수라고도 불리며 첫 번째 불가역적 단계를 포함한 반응의 모든 단계에 대한 속도 상수를 포함합니다.특이성 상수는 친화력과 촉매 능력을 모두 반영하기 때문에 서로 다른 효소 또는 서로 다른 기질을 가진 동일한 효소를 비교하는데 유용하다.특이성 상수의 이론상 최대값은 확산 한계라고 불리며 약 108~109(Ms−1)입니다−1.이 때 효소와 그 기질이 충돌할 때마다 촉매작용이 일어나며, 생성물 형성 속도는 반응 속도에 의해 제한되지 않고 확산 속도에 의해 제한된다.이 성질을 가진 효소는 촉매적으로 완벽하거나 동태적으로 완벽하다고 불린다.이들 효소의 예로는 트리오스인산 이성질화효소, 탄산무수분해효소, 아세틸콜린에스테라아제, 카탈라아제, 푸마라아제, β-락타마아제,[1]: 8.4.2 슈퍼옥사이드 디스무타아제 등이 있다.그러한 효소의 교체는 [1]: 9.2 초당 수백만 개의 반응에 이를 수 있다.그러나 대부분의 효소는 완벽과는 거리가 멀다: c / m / { k t{\ {의 평균값은 약 - \ 10 {\rm {\rm} {1 - {\rmm}}}}}}}}}}}}}} {s[66]
마이클리스-멘텐 역학은 자유 확산과 열역학적으로 구동되는 무작위 충돌의 가정으로부터 도출된 질량 작용의 법칙에 의존한다.많은 생화학적 또는 세포적 과정은 고분자 밀집과 [67]제한된 분자 운동 때문에 이러한 조건에서 크게 벗어납니다.보다 최근에는 모델의 복잡한 확장이 이러한 [68]효과를 수정하려고 시도합니다.
억제
효소 반응 속도는 다양한 유형의 효소 억제제에 [69]: 73–74 의해 감소될 수 있다.
억제 유형
경쟁적인
경쟁억제제와 기질은 [70]효소에 동시에 결합할 수 없다.종종 경쟁 억제제는 효소의 실제 기질과 매우 유사합니다.예를 들어 디히드로폴산염의 테트라히드로폴산으로의 환원을 촉매하는 디히드로폴산 [71]환원효소의 경쟁억제제이다.디히드로폴산염과 이 약물의 구조 간 유사성은 첨부 그림에 나와 있다.이러한 종류의 억제는 높은 기질 농도로 극복할 수 있다.경우에 따라서는 억제제는 통상적인 기질의 결합부위 이외의 부위에 결합하여 알로스테릭 효과를 발휘하여 통상적인 결합부위의 형상을 변화시킬 [72]수 있다.
비경쟁적
비경쟁적 억제제는 기질이 결합하는 곳 이외의 부위에 결합한다.기질은 여전히 일반적인 친화력으로 결합하므로 K는m 동일하게 유지됩니다.그러나 억제제는 효소의 촉매효율을 감소시켜max V를 감소시킨다.경쟁저해와는 대조적으로 비경쟁저해는 높은 [69]: 76–78 기질농도로 극복할 수 없다.
경쟁력 없음
비경쟁적 억제제는 효소-기질 복합체에만 유리 효소에 결합할 수 없기 때문에 이들 억제제는 높은 기질 농도에서 가장 효과적이다.억제제의 존재 하에서 효소-기질 복합체는 [69]: 78 불활성이다.이런 종류의 억제는 드물다.[73]
혼재
혼합억제제가 알로스테릭 부위에 결합하고 기판과 억제제의 결합이 서로 영향을 준다.효소의 기능은 감소하지만 억제제에 결합했을 때 제거되지는 않는다.이 억제제 유형은 미카엘리스-멘텐 [69]: 76–78 방정식을 따르지 않는다.
되돌릴 수 없다
불가역적 억제제는 보통 [74]단백질에 공유 결합을 형성함으로써 효소를 영구적으로 비활성화한다.페니실린과[75][76] 아스피린은 이런 작용을 하는 일반적인 약이다.
억제제의 기능
많은 유기체에서 억제제는 피드백 메커니즘의 일부로 작용할 수 있다.만약 효소가 유기체에서 한 가지 물질을 너무 많이 생산한다면, 그 물질은 그것을 생산하는 경로의 시작 부분에서 효소의 억제제 역할을 할 수 있고, 충분한 양이 있을 때 물질의 생산을 늦추거나 멈추게 할 수 있다.이것은 부정적인 피드백의 한 형태입니다.구연산 회로와 같은 주요 대사 경로는 이 [1]: 17.2.2 메커니즘을 이용한다.
억제제는 효소의 기능을 조절하기 때문에 종종 약물로 사용된다.그러한 많은 약물은 위의 메토트렉세이트와 유사한 효소의 토종 기질을 닮은 가역적 경쟁 억제제이다; 다른 잘 알려진 예로는 높은 콜레스테롤을 [77]치료하는데 사용되는 스타틴과 [78]HIV와 같은 레트로바이러스 감염을 치료하는데 사용되는 단백질 분해효소 억제제가 있다.약물로 사용되는 돌이킬 수 없는 억제제의 일반적인 예는 염증 메신저 프로스타글란딘을 생성하는 COX-1 및 COX-2 효소를 억제하는 아스피린이다.[76]다른 효소 억제제는 독이다.예를 들어 시안화독은 효소 시토크롬c산화효소 활성부위에서 구리, 철과 결합하여 세포호흡을 [79]차단하는 비가역성 효소억제제이다.
효소 활성에 영향을 미치는 요인
효소는 단백질로 구성되기 때문에 pH, 온도, 기질 농도 등과 같은 많은 물리적 화학적 요인의 변화에 민감합니다.
아래 표는 다양한 [80]효소에 대한 pH 최적화를 보여줍니다.
| 효소 | 최적 pH | pH 설명 |
|---|---|---|
| 펩신 | 1.5–1.6 | 고산성 |
| 인베르타아제 | 4.5 | 산성 |
| 리파아제(위) | 4.0–5.0 | 산성 |
| 리파아제(카스터유) | 4.7 | 산성 |
| 리파아제(팬서) | 8.0 | 알칼리성 |
| 아밀라아제(몰트) | 4.6–5.2 | 산성 |
| 아밀라아제(판서) | 6.7–7.0 | 산중립 |
| 셀로비아제 | 5.0 | 산성 |
| 말타아제 | 6.1–6.8 | 산성 |
| 수크라아제 | 6.2 | 산성 |
| 카탈라아제 | 7.0 | 중립 |
| 우레아제 | 7.0 | 중립 |
| 콜린에스테라아제 | 7.0 | 중립 |
| 리보핵산가수분해효소 | 7.0–7.5 | 중립 |
| 후마라아제 | 7.8 | 알칼리성 |
| 트립신 | 7.8–8.7 | 알칼리성 |
| 아데노신3인산 | 9.0 | 알칼리성 |
| 아르기나아제 | 10.0 | 고알칼리성 |
생물학적 기능
효소는 생물 안에서 다양한 기능을 한다.그것들은 종종 키나아제 및 포스파타아제를 통해 신호 전달과 세포 조절에 필수적이다.[81]그들은 또한 근육 수축을 생성하기 위해 미오신이 ATP를 가수 분해하는 운동을 일으키고 세포 [82]골격의 일부로 세포 주위에 화물을 운반합니다.세포막의 다른 ATP는 활성 수송에 관여하는 이온 펌프이다.효소는 또한 [83]반딧불에서 빛을 내는 루시페라아제와 같은 더 이국적인 기능에도 관여합니다.바이러스는 또한 HIV 인테그라아제나 역전사효소 같은 세포를 감염시키기 위한 효소와 인플루엔자 바이러스 뉴라미니다아제 [84]같은 세포로부터의 바이러스 방출을 위한 효소를 포함할 수 있다.
효소의 중요한 기능은 동물의 소화기 계통에 있다.아밀라아제나 단백질분해효소 같은 효소는 큰 분자(녹말이나 단백질)를 작은 분자로 분해해 장에 흡수된다.예를 들어, 녹말 분자는 장에서 흡수되기에는 너무 크지만, 효소는 녹말 사슬을 말토오스나 포도당과 같은 더 작은 분자로 가수 분해하고, 이는 흡수될 수 있습니다.서로 다른 효소는 서로 다른 음식 물질을 소화한다.초식성 식단을 가진 반추동물에서 내장 속의 미생물은 식물섬유의 [85]셀룰로오스 세포벽을 파괴하기 위해 또 다른 효소인 셀룰라아제를 생산한다.
대사
여러 효소가 특정한 순서로 함께 작용하여 대사 [1]: 30.1 경로를 만들 수 있습니다.대사 경로에서 한 효소는 다른 효소의 생성물을 기질로 한다.촉매 반응 후 생성물은 다른 효소로 전달된다.때때로 하나 이상의 효소가 동시에 동일한 반응을 촉매할 수 있다; 이것은 더 복잡한 조절을 가능하게 할 수 있다: 예를 들어, 한 효소에 의해 제공되는 낮은 일정한 활성과 두 번째 [86]효소에 의해 유도되는 높은 활성이다.
효소는 이러한 경로에서 어떤 단계가 일어나는지 결정한다.효소가 없다면, 신진대사는 같은 단계를 거치지 않을 것이고 세포의 요구를 충족시키기 위해 조절될 수 없다.대부분의 중심 대사 경로는 몇 가지 주요 단계에서 조절되며, 전형적으로 ATP의 가수 분해와 관련된 활동을 하는 효소를 통해 조절됩니다.이 반응이 너무 많은 에너지를 방출하기 때문에, 열역학적으로 불리한 다른 반응들은 ATP 가수분해와 결합되어 전체 일련의 연결된 대사 [1]: 30.1 반응을 이끌 수 있습니다.
액티비티 관리
세포에서 [1]: 30.1.1 효소 활성을 조절하는 다섯 가지 주요 방법이 있다.
규정
효소는 다른 분자에 의해 활성화되거나 억제될 수 있다.예를 들어 대사경로의 최종생성물은 종종 경로의 첫 번째 효소 중 하나에 대한 억제제(보통 첫 번째 비가역적 단계, 커밋 단계라고 함)이며, 따라서 경로에 의해 만들어지는 최종생성물의 양을 조절한다.이러한 조절 메커니즘을 네거티브 피드백 메커니즘이라고 하는데, 이는 최종 생산물의 양이 자체 [87]: 141–48 농도에 의해 조절되기 때문입니다.네거티브 피드백 메커니즘은 세포의 요구에 따라 중간 대사물의 합성 속도를 효과적으로 조절할 수 있다.이를 통해 재료 및 에너지 절약의 효과적인 할당에 도움이 되며, 최종 제품의 과잉 생산을 방지할 수 있습니다.다른 항상성 장치와 마찬가지로, 효소 작용의 제어는 살아있는 [87]: 141 유기체의 안정적인 내부 환경을 유지하는 데 도움이 됩니다.
번역 후 수정
번역 후 수정의 예로는 인산화, 미리스토일화 및 글리코실화를 [87]: 149–69 들 수 있다.예를 들어, 인슐린에 대한 반응으로, 글리코겐 합성효소를 포함한 여러 효소의 인산화 작용은 글리코겐의 합성 또는 분해를 조절하고 세포가 [88]혈당의 변화에 반응하도록 한다.번역 후 변형의 또 다른 예는 폴리펩타이드 사슬의 절단이다.소화단백질가수분해효소인 키모트립신은 췌장에서 키모트립시노겐으로 비활성 형태로 생성되어 활성화되는 위장으로 운반된다.이것은 효소가 장으로 들어가기 전에 췌장이나 다른 조직들을 소화시키는 것을 막는다.효소에 대한 이러한 유형의 비활성 전구체는 자이모겐[87]: 149–53 또는 프로엔자임으로 알려져 있다.
양
효소 생산(효소 유전자의 전사 및 번역)은 세포 환경의 변화에 따라 세포에 의해 증가 또는 감소될 수 있다.이러한 형태의 유전자 조절은 효소 유도라고 불린다.예를 들어, 박테리아는 페니실린 [89]분자 내에서 중요한 베타-락탐 고리를 가수 분해하는 베타-락타마아제라고 불리는 효소가 유도되기 때문에 페니실린과 같은 항생제에 내성이 생길 수 있다.또 다른 예는 약물의 대사에 중요한 시토크롬 P450 산화효소라고 불리는 간에 있는 효소로부터 온다.이러한 효소의 유도 또는 억제는 약물 [90]상호작용을 일으킬 수 있다.효소 분해 [1]: 30.1.1 속도를 변화시킴으로써 효소 수준을 조절할 수도 있다.효소 유도의 반대는 효소 억제이다.
세포하 분포
효소는 다른 세포 구획에서 발생하는 다른 대사 경로로 구분될 수 있습니다.예를 들어 지방산은 세포졸, 소포체 및 골지 중 하나의 효소에 의해 합성되며, β-산화를 [91]통해 미토콘드리아의 에너지원으로서 다른 효소에 의해 사용된다.또한 효소의 다른 구획으로의 이동은 양성자화 정도(예를 들어 중성 세포질 및 산성 리소좀) 또는 산화 상태(예를 들어 과배질 산화 또는 세포질 감소)를 변화시켜 효소 활성에 [92]영향을 줄 수 있다.막 결합 유기물로 분할되는 것과는 대조적으로, 효소 아세포 국소화는 효소의 고분자 세포질 [93][94]필라멘트로의 중합에 의해 변경될 수 있다.
조직의 전문화
다세포 진핵생물에서, 다른 기관과 조직의 세포들은 다른 유전자 발현 패턴을 가지고 있고, 따라서 대사 반응에 사용할 수 있는 다른 효소 세트를 가지고 있습니다.이것은 유기체의 전체적인 신진대사를 조절하는 메커니즘을 제공한다.예를 들어 해당과정의 첫 번째 효소인 헥소키나제는 간 및 췌장에서 발현되는 글루코키나아제라는 특수한 형태를 가지며, 포도당에 대한 친화력은 낮지만 포도당 [95]농도에 더 민감하다.이 효소는 혈당을 감지하고 인슐린 [96]생성을 조절하는 데 관여합니다.
질병에 대한 관여

효소 활성의 엄격한 제어는 항상성에 필수적이기 때문에, 단일 중요 효소의 오작동(변성, 과잉 생산, 과소 생산 또는 결실)은 유전적인 질병으로 이어질 수 있습니다.인체에 존재하는 수천 가지 효소 중 한 가지만 제대로 작동하지 않으면 치명적일 수 있다.효소 결핍으로 인한 치명적인 유전 질환의 예로는 환자가 헥소사미니다아제를 [97][98]결핍하는 Tay-Sachs 질환이 있다.
효소 결핍의 한 예는 가장 흔한 종류의 페닐케톤뇨증이다.페닐알라닌 분해의 첫 번째 단계를 촉매하는 효소 페닐알라닌 하이드록실화효소의 많은 다른 단일 아미노산 돌연변이는 페닐알라닌과 관련 산물의 축적을 초래한다.일부 돌연변이는 활성 부위에 있어 결합과 촉매 작용을 직접적으로 방해하지만, 많은 돌연변이는 활성 부위에서 멀리 떨어져 단백질 구조를 불안정하게 하거나 올바른 올리고머화에 [99][100]영향을 줌으로써 활성을 감소시킨다.이 병을 [101]치료하지 않으면 지적 장애로 이어질 수 있다.또 다른 예는 콜린에스테르 약물을 분해하는 신체의 능력이 [102]저하되는 의사콜린에스테라아제 결핍이다.효소의 경구 투여는 췌장 기능[103] 부전 및 유당 [104]불내증과 같은 일부 기능적 효소 결핍을 치료하는데 사용될 수 있습니다.
효소 오작동이 질병을 일으킬 수 있는 또 다른 방법은 DNA 복구 효소를 코드하는 유전자의 생식계 돌연변이에서 비롯된다.이 효소들의 결함은 세포들이 게놈의 돌연변이를 복구하는 능력이 떨어지기 때문에 암을 유발한다.이것은 돌연변이의 느린 축적을 유발하고 암의 발생을 초래한다.이러한 유전성 암 증후군의 예로는 자외선에 [105][106]대한 최소한의 노출에도 반응하여 피부암에 걸리는 건피증이 있다.
진화
다른 단백질과 마찬가지로, 효소는 돌연변이와 배열 분리를 통해 시간이 지남에 따라 변합니다.신진대사에서 그들의 중심적인 역할을 감안할 때, 효소 진화는 적응에 중요한 역할을 한다.따라서 핵심 질문은 효소가 효소 활동을 함께 변화시킬 수 있는지 여부와 방법이다.진화는 또한 복제 없이 일어날 수 있지만, 많은 새로운 효소 활동이 유전자 복제와 복제 복제 복제의 돌연변이를 통해 진화했다는 것은 일반적으로 받아들여진다.활성을 변화시킨 효소의 한 예는 메티오닐 아미노펩티다아제(MAP)와 크레아틴아미노히드로라아제(Creatinohydrolase)의 조상이며, 메티오닌은 명백히 상동성이지만 매우 다른 반응을 촉매한다(MAP는 크레아티나아제 가수분해효소(Creatinaase)가 크레아틴을 사르코신 및 요소에 분해한다).또한 MAP은 금속 이온에 의존하지만 크레아티나아제는 의존하지 않기 때문에 시간이 [107]지남에 따라 이 특성도 손실되었다.효소 활성의 작은 변화는 효소들 사이에서 매우 흔하다.특히 기질결합특이성(위 참조)은 기질결합주머니 내의 단일 아미노산 변화에 따라 쉽고 빠르게 변화할 수 있다.이것은 키나아제 [108]같은 주요 효소 클래스에서 자주 나타난다.
인공(체외) 진화는 이제 산업적 응용을 위한 효소 활성 또는 특이성을 수정하는 데 일반적으로 사용됩니다(아래 참조).
산업용 응용 프로그램
효소는 매우 특정한 촉매가 필요할 때 화학 산업 및 기타 산업 분야에서 사용됩니다.일반적으로 효소는 촉매 작용을 위해 진화한 반응의 수와 유기 용매 및 고온에서의 안정성의 결여에 의해 제한됩니다.결과적으로, 단백질 공학은 활발한 연구 영역이며 합리적인 설계 또는 시험관 내 [109][110]진화를 통해 새로운 특성을 가진 새로운 효소를 창조하려는 시도를 포함한다.이러한 노력은 성공하기 시작했고,[111] 몇몇 효소는 자연에서 일어나지 않는 반응을 촉매하기 위해 "처음부터" 설계되었다.
| 어플 | 사용된 효소 | 사용하다 |
|---|---|---|
| 바이오 연료 산업 | 셀룰라아제 | 셀룰로오스를 [112]발효시킬 수 있는 설탕으로 분해하여 셀룰로오스 에탄올을 생성한다. |
| 리그나아제 | 바이오 연료 [112]생산을 위한 바이오매스 전처리. | |
| 생물 세제 | 단백질분해효소, 아밀라아제, 리파아제 | 세탁물과 [113]식기류에서 단백질, 녹말, 지방이나 기름때를 제거한다. |
| 만나나아제 | 일반적인 식품첨가물인 [113]과르껌에서 음식 얼룩을 제거한다. | |
| 양조업 | 아밀라아제, 글루카나아제, 단백질분해효소 | 맥아에서 [114]: 150–9 다당류와 단백질을 분리하세요. |
| 베타글루카나아제 | 와트 및 맥주 여과 [114]: 545 특성을 개선합니다. | |
| 아밀로그루코시다아제 및 풀라나아제 | 저열량 맥주를 만들어 발효성을 [114]: 575 조절한다. | |
| 아세트락틴탈탄산화효소(ALDC) | 디아세틸 생성을 [115]줄여 발효 효율을 높인다. | |
| 요리의 용도 | 빠빠인 | 고기를 [116]요리하기 위해 연하게 하다. |
| 유업 | 렌닌 | 치즈를 [117]제조할 때 단백질을 가수분해한다. |
| 리파아제 | 카망베르 치즈와 로케포트와 [118]같은 블루 치즈를 생산하세요. | |
| 식품 가공 | 아밀라아제 | 고과당 옥수수 [119]시럽을 만드는 것과 같이 녹말에서 설탕을 생산하세요. |
| 단백질분해효소 | 비스킷을 [120]만들 때처럼 밀가루의 단백질 수치를 낮추세요. | |
| 트립신 | 저알레르기성 [120]이유식을 제조하다. | |
| 셀룰라아제, 펙티나아제 | 과일 [121]주스를 맑게 하다. | |
| 분자생물학 | 핵산가수분해효소, DNA리가수분해효소 및 중합효소 | 제한 소화 및 중합효소 연쇄 반응을 사용하여 재조합 [1]: 6.2 DNA를 생성한다. |
| 제지업 | 자일라나아제, 헤미셀룰라아제 및 리그닌페옥시다아제 | 크래프트 [122]펄프에서 리그닌을 제거하세요. |
| 퍼스널 케어 | 단백질분해효소 | 감염을 방지하기 위해 콘택트렌즈의 [123]단백질을 제거하세요. |
| 전분 산업 | 아밀라아제 | 녹말을 포도당과 다양한 [124]시럽으로 바꿔라. |
「 」를 참조해 주세요.
효소 데이터베이스
레퍼런스
- ^ a b c d e f g h i j k l m n o p q r s t u Stryer L, Berg JM, Tymoczko JL (2002). Biochemistry (5th ed.). San Francisco: W.H. Freeman. ISBN 0-7167-4955-6.

- ^ Murphy JM, Farhan H, Eyers PA (2017). "Bio-Zombie: the rise of pseudoenzymes in biology". Biochem Soc Trans. 45 (2): 537–544. doi:10.1042/bst20160400. PMID 28408493.
- ^ Murphy JM, et al. (2014). "A robust methodology to subclassify pseudokinases based on their nucleotide-binding properties". Biochemical Journal. 457 (2): 323–334. doi:10.1042/BJ20131174. PMC 5679212. PMID 24107129.
- ^ Schomburg I, Chang A, Placzek S, Söhngen C, Rother M, Lang M, Munaretto C, Ulas S, Stelzer M, Grote A, Scheer M, Schomburg D (January 2013). "BRENDA in 2013: integrated reactions, kinetic data, enzyme function data, improved disease classification: new options and contents in BRENDA". Nucleic Acids Research. 41 (Database issue): D764–72. doi:10.1093/nar/gks1049. PMC 3531171. PMID 23203881.
- ^ Radzicka A, Wolfenden R (January 1995). "A proficient enzyme". Science. 267 (5194): 90–931. Bibcode:1995Sci...267...90R. doi:10.1126/science.7809611. PMID 7809611. S2CID 8145198.
- ^ Callahan BP, Miller BG (December 2007). "OMP decarboxylase—An enigma persists". Bioorganic Chemistry. 35 (6): 465–9. doi:10.1016/j.bioorg.2007.07.004. PMID 17889251.
- ^ de Réaumur RA (1752). "Observations sur la digestion des oiseaux". Histoire de l'Académie Royale des Sciences. 1752: 266, 461.
- ^ Williams HS (1904). A History of Science: in Five Volumes. Volume IV: Modern Development of the Chemical and Biological Sciences. Harper and Brothers.
- ^ Payen A, Persoz JF (1833). "Mémoire sur la diastase, les principaux produits de ses réactions et leurs applications aux arts industriels" [Memoir on diastase, the principal products of its reactions, and their applications to the industrial arts]. Annales de chimie et de physique. 2nd (in French). 53: 73–92.
- ^ Manchester KL (December 1995). "Louis Pasteur (1822–1895)–chance and the prepared mind". Trends in Biotechnology. 13 (12): 511–5. doi:10.1016/S0167-7799(00)89014-9. PMID 8595136.
- ^ Kühne 그 단어 Kühne W(1877년)의"효소"것입니다."Über 다쓰 Verhaltenverschiedener organisirter 운트 sog.Ungeformter Fermente"[다양한 소위 충분히 발달되지 않은 조직 섬유의 행동에].Verhandlungen 데 Naturhistorisch-medicinischen Vereins zu 하이델베르크.새로운 시리즈(독일어로).1(3):190–193.페이지 190에 관련 글:"음 Missverständnissen vorzubeugen und lästige Umschreibungen zuvermeiden schlägt Vortragender vor, 죽ungeformten 냄새 nicht organisirten Fermente,deren Wirkung ohne Anwesenheit 폰 Organismen undausserhalbderselben erfolgen kann,als 효소 zu bezeichnen."(번역:.오해를 없애고 번거로운 주변문구를 피하기 위해 [저자, 대학강사]는 유기체의 존재와 그 외부에서 작용이 일어날 수 있는 미형성 또는 비조직적 발효를 "효모"로 지정할 것을 제안한다.)
- ^ Holmes FL (2003). "Enzymes". In Heilbron JL (ed.). The Oxford Companion to the History of Modern Science. Oxford: Oxford University Press. p. 270. ISBN 9780199743766.
- ^ "Eduard Buchner". Nobel Laureate Biography. Nobelprize.org. Retrieved 23 February 2015.
- ^ "Eduard Buchner – Nobel Lecture: Cell-Free Fermentation". Nobelprize.org. 1907. Retrieved 23 February 2015.
- ^ 효소의 효소는 기질에 그 접미사"-ase"를 첨가하여 그 명명하기, 프랑스 과학자에 – 첫번째 효소 – 그의 책 Duclaux E(1899년)에서 이 연습을 도입함으로써 고립되는 디아스타아제의 발견자들을 기리기 위해 의도한 Duclaux(1840–1904), Émile 거슬러 올라간다.Traité 드 microbiologie:Diastases, 기타 venins[미생물학 Treatise:diastases, 독소, 그리고 독은](프랑스어로)toxines.프랑스 파리:마손과 무관참고 1장, 특히 페이지 9.
- ^ Willstätter R (1927). "Faraday lecture. Problems and methods in enzyme research". Journal of the Chemical Society (Resumed): 1359–1381. doi:10.1039/JR9270001359. 에 인용된.
- ^ "Nobel Prizes and Laureates: The Nobel Prize in Chemistry 1946". Nobelprize.org. Retrieved 23 February 2015.
- ^ Blake CC, Koenig DF, Mair GA, North AC, Phillips DC, Sarma VR (May 1965). "Structure of hen egg-white lysozyme. A three-dimensional Fourier synthesis at 2 Ångström resolution". Nature. 206 (4986): 757–61. Bibcode:1965Natur.206..757B. doi:10.1038/206757a0. PMID 5891407. S2CID 4161467.
- ^ Johnson LN, Petsko GA (1999). "David Phillips and the origin of structural enzymology". Trends Biochem. Sci. 24 (7): 287–9. doi:10.1016/S0968-0004(99)01423-1. PMID 10390620.
- ^ Moss GP. "Recommendations of the Nomenclature Committee of the International Union of Biochemistry and Molecular Biology on the Nomenclature and Classification of Enzymes by the Reactions they Catalyse". International Union of Biochemistry and Molecular Biology. Retrieved 28 August 2021.
- ^ Nomenclature Committee. "EC 2.7.1.1". International Union of Biochemistry and Molecular Biology (NC-IUBMB). School of Biological and Chemical Sciences, Queen Mary, University of London. Archived from the original on 1 December 2014. Retrieved 6 March 2015.
- ^ Mulder NJ (28 September 2007). "Protein Family Databases". eLS. Chichester, UK: John Wiley & Sons, Ltd. pp. a0003058.pub2. doi:10.1002/9780470015902.a0003058.pub2. ISBN 978-0-470-01617-6.
- ^ Anfinsen CB (July 1973). "Principles that govern the folding of protein chains". Science. 181 (4096): 223–30. Bibcode:1973Sci...181..223A. doi:10.1126/science.181.4096.223. PMID 4124164.
- ^ Dunaway-Mariano D (November 2008). "Enzyme function discovery". Structure. 16 (11): 1599–600. doi:10.1016/j.str.2008.10.001. PMID 19000810.
- ^ Petsko GA, Ringe D (2003). "Chapter 1: From sequence to structure". Protein structure and function. London: New Science. p. 27. ISBN 978-1405119221.
- ^ Chen LH, Kenyon GL, Curtin F, Harayama S, Bembenek ME, Hajipour G, Whitman CP (September 1992). "4-Oxalocrotonate tautomerase, an enzyme composed of 62 amino acid residues per monomer". The Journal of Biological Chemistry. 267 (25): 17716–21. doi:10.1016/S0021-9258(19)37101-7. PMID 1339435.
- ^ Smith S (December 1994). "The animal fatty acid synthase: one gene, one polypeptide, seven enzymes". FASEB Journal. 8 (15): 1248–59. doi:10.1096/fasebj.8.15.8001737. PMID 8001737. S2CID 22853095.
- ^ "The Catalytic Site Atlas". The European Bioinformatics Institute. Retrieved 4 April 2007.
- ^ a b Suzuki H (2015). "Chapter 7: Active Site Structure". How Enzymes Work: From Structure to Function. Boca Raton, FL: CRC Press. pp. 117–140. ISBN 978-981-4463-92-8.
- ^ Krauss G (2003). "The Regulations of Enzyme Activity". Biochemistry of Signal Transduction and Regulation (3rd ed.). Weinheim: Wiley-VCH. pp. 89–114. ISBN 9783527605767.
- ^ Jaeger KE, Eggert T (August 2004). "Enantioselective biocatalysis optimized by directed evolution". Current Opinion in Biotechnology. 15 (4): 305–13. doi:10.1016/j.copbio.2004.06.007. PMID 15358000.
- ^ Shevelev IV, Hübscher U (May 2002). "The 3' 5' exonucleases". Nature Reviews Molecular Cell Biology. 3 (5): 364–76. doi:10.1038/nrm804. PMID 11988770. S2CID 31605786.
- ^ Zenkin N, Yuzenkova Y, Severinov K (July 2006). "Transcript-assisted transcriptional proofreading". Science. 313 (5786): 518–20. Bibcode:2006Sci...313..518Z. doi:10.1126/science.1127422. PMID 16873663. S2CID 40772789.
- ^ Ibba M, Soll D (2000). "Aminoacyl-tRNA synthesis". Annual Review of Biochemistry. 69: 617–50. doi:10.1146/annurev.biochem.69.1.617. PMID 10966471.
- ^ Rodnina MV, Wintermeyer W (2001). "Fidelity of aminoacyl-tRNA selection on the ribosome: kinetic and structural mechanisms". Annual Review of Biochemistry. 70: 415–35. doi:10.1146/annurev.biochem.70.1.415. PMID 11395413.
- ^ Khersonsky O, Tawfik DS (2010). "Enzyme promiscuity: a mechanistic and evolutionary perspective". Annual Review of Biochemistry. 79: 471–505. doi:10.1146/annurev-biochem-030409-143718. PMID 20235827.
- ^ O'Brien PJ, Herschlag D (April 1999). "Catalytic promiscuity and the evolution of new enzymatic activities". Chemistry & Biology. 6 (4): R91–R105. doi:10.1016/S1074-5521(99)80033-7. PMID 10099128.
- ^ 피셔 E(1894년)."Einfluss der구성 Wirkung하는 효소 죽auf"[구성 enzymes의 작용에 미치는 영향].Berichte 역사 Deutschen Chemischen 게젤샤프트 베를린(독일어로)zu.27(3):2985–93. doi:10.1002/cber.18940270364.페이지 2992:"음, sagen,dass Enzym und Glucosid wie 슐로스 und Schlüssel zu einander passen müssen,)eine chemische Wirkung auf einanderausüben zu können ich 것이다 빌트zu gebrauchen ein."(이미지를 이용하려면부터, 나는을 하기 위해서 chemi을 발휘할 수 있는 효소와 당원질[즉, 미분 포도당]마치 자물쇠와 열쇠처럼, 딱 들어맞아야 한다 말할 것이다.서로 영향을 미치기 때문입니다.)
- ^ Cooper GM (2000). "Chapter 2.2: The Central Role of Enzymes as Biological Catalysts". The Cell: a Molecular Approach (2nd ed.). Washington (DC ): ASM Press. ISBN 0-87893-106-6.
- ^ Koshland DE (February 1958). "Application of a Theory of Enzyme Specificity to Protein Synthesis". Proceedings of the National Academy of Sciences of the United States of America. 44 (2): 98–104. Bibcode:1958PNAS...44...98K. doi:10.1073/pnas.44.2.98. PMC 335371. PMID 16590179.
- ^ Vasella A, Davies GJ, Böhm M (October 2002). "Glycosidase mechanisms". Current Opinion in Chemical Biology. 6 (5): 619–29. doi:10.1016/S1367-5931(02)00380-0. PMID 12413546.
- ^ Boyer R (2002). "Chapter 6: Enzymes I, Reactions, Kinetics, and Inhibition". Concepts in Biochemistry (2nd ed.). New York, Chichester, Weinheim, Brisbane, Singapore, Toronto.: John Wiley & Sons, Inc. pp. 137–8. ISBN 0-470-00379-0. OCLC 51720783.
- ^ Savir Y, Tlusty T (2007). Scalas E (ed.). "Conformational proofreading: the impact of conformational changes on the specificity of molecular recognition". PLOS ONE. 2 (5): e468. Bibcode:2007PLoSO...2..468S. doi:10.1371/journal.pone.0000468. PMC 1868595. PMID 17520027.
- ^ Fersht A (1985). Enzyme Structure and Mechanism. San Francisco: W.H. Freeman. pp. 50–2. ISBN 978-0-7167-1615-0.
- ^ Warshel A, Sharma PK, Kato M, Xiang Y, Liu H, Olsson MH (August 2006). "Electrostatic basis for enzyme catalysis". Chemical Reviews. 106 (8): 3210–35. doi:10.1021/cr0503106. PMID 16895325.
- ^ Cox MM, Nelson DL (2013). "Chapter 6.2: How enzymes work". Lehninger Principles of Biochemistry (6th ed.). New York, N.Y.: W.H. Freeman. p. 195. ISBN 978-1464109621.
- ^ Benkovic SJ, Hammes-Schiffer S (August 2003). "A perspective on enzyme catalysis". Science. 301 (5637): 1196–202. Bibcode:2003Sci...301.1196B. doi:10.1126/science.1085515. PMID 12947189. S2CID 7899320.
- ^ Jencks WP (1987). Catalysis in Chemistry and Enzymology. Mineola, N.Y: Dover. ISBN 978-0-486-65460-7.
- ^ Villa J, Strajbl M, Glennon TM, Sham YY, Chu ZT, Warshel A (October 2000). "How important are entropic contributions to enzyme catalysis?". Proceedings of the National Academy of Sciences of the United States of America. 97 (22): 11899–904. Bibcode:2000PNAS...9711899V. doi:10.1073/pnas.97.22.11899. PMC 17266. PMID 11050223.
- ^ Polgár L (October 2005). "The catalytic triad of serine peptidases". Cellular and Molecular Life Sciences. 62 (19–20): 2161–2172. doi:10.1007/s00018-005-5160-x. PMID 16003488. S2CID 3343824.
- ^ Ramanathan A, Savol A, Burger V, Chennubhotla CS, Agarwal PK (2014). "Protein conformational populations and functionally relevant substates". Acc. Chem. Res. 47 (1): 149–56. doi:10.1021/ar400084s. OSTI 1565147. PMID 23988159.
- ^ Tsai CJ, Del Sol A, Nussinov R (2009). "Protein allostery, signal transmission and dynamics: a classification scheme of allosteric mechanisms" (PDF). Mol Biosyst. 5 (3): 207–16. doi:10.1039/b819720b. PMC 2898650. PMID 19225609.
- ^ Changeux JP, Edelstein SJ (June 2005). "Allosteric mechanisms of signal transduction". Science. 308 (5727): 1424–8. Bibcode:2005Sci...308.1424C. doi:10.1126/science.1108595. PMID 15933191. S2CID 10621930.
- ^ de Bolster MW (1997). "Glossary of Terms Used in Bioinorganic Chemistry: Cofactor". International Union of Pure and Applied Chemistry. Archived from the original on 21 January 2017. Retrieved 30 October 2007.
- ^ Voet D, Voet J, Pratt C (2016). Fundamentals of Biochemistry. Hoboken, New Jersey: John Wiley & Sons, Inc. p. 336. ISBN 978-1-118-91840-1.
- ^ Chapman-Smith A, Cronan JE (September 1999). "The enzymatic biotinylation of proteins: a post-translational modification of exceptional specificity". Trends in Biochemical Sciences. 24 (9): 359–363. doi:10.1016/s0968-0004(99)01438-3. PMID 10470036.
- ^ Fisher Z, Hernandez Prada JA, Tu C, Duda D, Yoshioka C, An H, Govindasamy L, Silverman DN, McKenna R (February 2005). "Structural and kinetic characterization of active-site histidine as a proton shuttle in catalysis by human carbonic anhydrase II". Biochemistry. 44 (4): 1097–115. doi:10.1021/bi0480279. PMID 15667203.
- ^ a b Wagner AL (1975). Vitamins and Coenzymes. Krieger Pub Co. ISBN 0-88275-258-8.
- ^ "BRENDA The Comprehensive Enzyme Information System". Technische Universität Braunschweig. Retrieved 23 February 2015.
- ^ Törnroth-Horsefield S, Neutze R (December 2008). "Opening and closing the metabolite gate". Proceedings of the National Academy of Sciences of the United States of America. 105 (50): 19565–6. Bibcode:2008PNAS..10519565T. doi:10.1073/pnas.0810654106. PMC 2604989. PMID 19073922.
- ^ McArdle WD, Katch F, Katch VL (2006). "Chapter 9: The Pulmonary System and Exercise". Essentials of Exercise Physiology (3rd ed.). Baltimore, Maryland: Lippincott Williams & Wilkins. pp. 312–3. ISBN 978-0781749916.
- ^ Ferguson SJ, Nicholls D, Ferguson S (2002). Bioenergetics 3 (3rd ed.). San Diego: Academic. ISBN 0-12-518121-3.
- ^ Bisswanger H (2017). Enzyme kinetics : principles and methods (Third, enlarged and improved ed.). Weinheim, Germany. ISBN 9783527806461. OCLC 992976641.
- ^ 마이클 리스 L, Menten M(1913년)."다이 Kinetik Invertinwirkung der"[그 특성 Invertase 연대'의].Biochem.Z(독일어로).49:333–369., 마이클 리스 L, Menten 맥스, 존슨 KA, 구디 RS(2011년)."원래 미카엘리스:1913년 Michaelis–Menten의 종이 번역 일정".생화학. 50(39):8264–9. doi:10.1021/bi201284u.PMC3381512.PMID 21888353.
- ^ Briggs GE, Haldane JB (1925). "A Note on the Kinetics of Enzyme Action". The Biochemical Journal. 19 (2): 338–9. doi:10.1042/bj0190338. PMC 1259181. PMID 16743508.
- ^ Bar-Even A, Noor E, Savir Y, Liebermeister W, Davidi D, Tawfik DS, Milo R (2011). "The moderately efficient enzyme: evolutionary and physicochemical trends shaping enzyme parameters". Biochemistry. 50 (21): 4402–10. doi:10.1021/bi2002289. PMID 21506553.
- ^ Ellis RJ (October 2001). "Macromolecular crowding: obvious but underappreciated". Trends in Biochemical Sciences. 26 (10): 597–604. doi:10.1016/S0968-0004(01)01938-7. PMID 11590012.
- ^ Kopelman R (September 1988). "Fractal reaction kinetics". Science. 241 (4873): 1620–26. Bibcode:1988Sci...241.1620K. doi:10.1126/science.241.4873.1620. PMID 17820893. S2CID 23465446.
- ^ a b c d Cornish-Bowden A (2004). Fundamentals of Enzyme Kinetics (3 ed.). London: Portland Press. ISBN 1-85578-158-1.
- ^ Price NC (1979). "What is meant by 'competitive inhibition'?". Trends in Biochemical Sciences. 4 (11): N272–N273. doi:10.1016/0968-0004(79)90205-6.
- ^ Goodsell DS (1 August 1999). "The Molecular Perspective: Methotrexate". The Oncologist. 4 (4): 340–341. doi:10.1634/theoncologist.4-4-340. ISSN 1083-7159. PMID 10476546.
- ^ Wu P, Clausen MH, Nielsen TE (December 2015). "Allosteric small-molecule kinase inhibitors" (PDF). Pharmacology & Therapeutics. 156: 59–68. doi:10.1016/j.pharmthera.2015.10.002. PMID 26478442.
- ^ Cornish-Bowden A (July 1986). "Why is uncompetitive inhibition so rare? A possible explanation, with implications for the design of drugs and pesticides". FEBS Letters. 203 (1): 3–6. doi:10.1016/0014-5793(86)81424-7. PMID 3720956. S2CID 45356060.
- ^ Strelow JM (1 January 2017). "A Perspective on the Kinetics of Covalent and Irreversible Inhibition". SLAS Discovery. 22 (1): 3–20. doi:10.1177/1087057116671509. ISSN 2472-5552. PMID 27703080.
- ^ Fisher JF, Meroueh SO, Mobashery S (February 2005). "Bacterial resistance to beta-lactam antibiotics: compelling opportunism, compelling opportunity". Chemical Reviews. 105 (2): 395–424. doi:10.1021/cr030102i. PMID 15700950.
- ^ a b Johnson DS, Weerapana E, Cravatt BF (June 2010). "Strategies for discovering and derisking covalent, irreversible enzyme inhibitors". Future Medicinal Chemistry. 2 (6): 949–64. doi:10.4155/fmc.10.21. PMC 2904065. PMID 20640225.
- ^ Endo A (1 November 1992). "The discovery and development of HMG-CoA reductase inhibitors". J. Lipid Res. 33 (11): 1569–82. doi:10.1016/S0022-2275(20)41379-3. PMID 1464741.
- ^ Wlodawer A, Vondrasek J (1998). "Inhibitors of HIV-1 protease: a major success of structure-assisted drug design". Annual Review of Biophysics and Biomolecular Structure. 27: 249–84. doi:10.1146/annurev.biophys.27.1.249. PMID 9646869. S2CID 10205781.
- ^ Yoshikawa S, Caughey WS (May 1990). "Infrared evidence of cyanide binding to iron and copper sites in bovine heart cytochrome c oxidase. Implications regarding oxygen reduction". The Journal of Biological Chemistry. 265 (14): 7945–58. doi:10.1016/S0021-9258(19)39023-4. PMID 2159465.
- ^ Jain JL (May 1999). Fundamentals of biochemistry. New Delhi: S. Chand and Co. ISBN 8121903432. OCLC 818809626.
- ^ Hunter T (January 1995). "Protein kinases and phosphatases: the yin and yang of protein phosphorylation and signaling". Cell. 80 (2): 225–36. doi:10.1016/0092-8674(95)90405-0. PMID 7834742. S2CID 13999125.
- ^ Berg JS, Powell BC, Cheney RE (April 2001). "A millennial myosin census". Molecular Biology of the Cell. 12 (4): 780–94. doi:10.1091/mbc.12.4.780. PMC 32266. PMID 11294886.
- ^ Meighen EA (March 1991). "Molecular biology of bacterial bioluminescence". Microbiological Reviews. 55 (1): 123–42. doi:10.1128/MMBR.55.1.123-142.1991. PMC 372803. PMID 2030669.
- ^ De Clercq E (2002). "Highlights in the development of new antiviral agents". Mini Rev Med Chem. 2 (2): 163–75. doi:10.2174/1389557024605474. PMID 12370077.
- ^ Mackie RI, White BA (October 1990). "Recent advances in rumen microbial ecology and metabolism: potential impact on nutrient output". Journal of Dairy Science. 73 (10): 2971–95. doi:10.3168/jds.S0022-0302(90)78986-2. PMID 2178174.
- ^ Rouzer CA, Marnett LJ (2009). "Cyclooxygenases: structural and functional insights". J. Lipid Res. 50 Suppl: S29–34. doi:10.1194/jlr.R800042-JLR200. PMC 2674713. PMID 18952571.
- ^ a b c d Suzuki H (2015). "Chapter 8: Control of Enzyme Activity". How Enzymes Work: From Structure to Function. Boca Raton, FL: CRC Press. pp. 141–69. ISBN 978-981-4463-92-8.
- ^ Doble BW, Woodgett JR (April 2003). "GSK-3: tricks of the trade for a multi-tasking kinase". Journal of Cell Science. 116 (Pt 7): 1175–86. doi:10.1242/jcs.00384. PMC 3006448. PMID 12615961.
- ^ Bennett PM, Chopra I (1993). "Molecular basis of beta-lactamase induction in bacteria". Antimicrob. Agents Chemother. 37 (2): 153–8. doi:10.1128/aac.37.2.153. PMC 187630. PMID 8452343.
- ^ Skett P, Gibson GG (2001). "Chapter 3: Induction and Inhibition of Drug Metabolism". Introduction to Drug Metabolism (3 ed.). Cheltenham, UK: Nelson Thornes Publishers. pp. 87–118. ISBN 978-0748760114.
- ^ Faergeman NJ, Knudsen J (April 1997). "Role of long-chain fatty acyl-CoA esters in the regulation of metabolism and in cell signalling". The Biochemical Journal. 323 (Pt 1): 1–12. doi:10.1042/bj3230001. PMC 1218279. PMID 9173866.
- ^ Suzuki H (2015). "Chapter 4: Effect of pH, Temperature, and High Pressure on Enzymatic Activity". How Enzymes Work: From Structure to Function. Boca Raton, FL: CRC Press. pp. 53–74. ISBN 978-981-4463-92-8.
- ^ Noree C, Sato BK, Broyer RM, Wilhelm JE (August 2010). "Identification of novel filament-forming proteins in Saccharomyces cerevisiae and Drosophila melanogaster". The Journal of Cell Biology. 190 (4): 541–51. doi:10.1083/jcb.201003001. PMC 2928026. PMID 20713603.
- ^ Aughey GN, Liu JL (2015). "Metabolic regulation via enzyme filamentation". Critical Reviews in Biochemistry and Molecular Biology. 51 (4): 282–93. doi:10.3109/10409238.2016.1172555. PMC 4915340. PMID 27098510.
- ^ Kamata K, Mitsuya M, Nishimura T, Eiki J, Nagata Y (March 2004). "Structural basis for allosteric regulation of the monomeric allosteric enzyme human glucokinase". Structure. 12 (3): 429–38. doi:10.1016/j.str.2004.02.005. PMID 15016359.
- ^ Froguel P, Zouali H, Vionnet N, Velho G, Vaxillaire M, Sun F, Lesage S, Stoffel M, Takeda J, Passa P (March 1993). "Familial hyperglycemia due to mutations in glucokinase. Definition of a subtype of diabetes mellitus". The New England Journal of Medicine. 328 (10): 697–702. doi:10.1056/NEJM199303113281005. PMID 8433729.
- ^ Okada S, O'Brien JS (August 1969). "Tay–Sachs disease: generalized absence of a beta-D-N-acetylhexosaminidase component". Science. 165 (3894): 698–700. Bibcode:1969Sci...165..698O. doi:10.1126/science.165.3894.698. PMID 5793973. S2CID 8473726.
- ^ "Learning About Tay–Sachs Disease". U.S. National Human Genome Research Institute. Retrieved 1 March 2015.
- ^ Erlandsen H, Stevens RC (October 1999). "The structural basis of phenylketonuria". Molecular Genetics and Metabolism. 68 (2): 103–25. doi:10.1006/mgme.1999.2922. PMID 10527663.
- ^ Flatmark T, Stevens RC (August 1999). "Structural Insight into the Aromatic Amino Acid Hydroxylases and Their Disease-Related Mutant Forms". Chemical Reviews. 99 (8): 2137–2160. doi:10.1021/cr980450y. PMID 11849022.
- ^ "Phenylketonuria". Genes and Disease [Internet]. Bethesda (MD): National Center for Biotechnology Information (US). 1998–2015.
- ^ "Pseudocholinesterase deficiency". U.S. National Library of Medicine. Retrieved 5 September 2013.
- ^ Fieker A, Philpott J, Armand M (2011). "Enzyme replacement therapy for pancreatic insufficiency: present and future". Clinical and Experimental Gastroenterology. 4: 55–73. doi:10.2147/CEG.S17634. PMC 3132852. PMID 21753892.
- ^ Misselwitz B, Pohl D, Frühauf H, Fried M, Vavricka SR, Fox M (June 2013). "Lactose malabsorption and intolerance: pathogenesis, diagnosis and treatment". United European Gastroenterology Journal. 1 (3): 151–9. doi:10.1177/2050640613484463. PMC 4040760. PMID 24917953.
- ^ Cleaver JE (May 1968). "Defective repair replication of DNA in xeroderma pigmentosum". Nature. 218 (5142): 652–6. Bibcode:1968Natur.218..652C. doi:10.1038/218652a0. PMID 5655953. S2CID 4171859.
- ^ James WD, Elston D, Berger TG (2011). Andrews' Diseases of the Skin: Clinical Dermatology (11th ed.). London: Saunders/ Elsevier. p. 567. ISBN 978-1437703146.
- ^ Murzin, A. G. (1993). "Can homologous proteins evolve different enzymatic activities?". Trends in Biochemical Sciences. 18 (11): 403–405. doi:10.1016/0968-0004(93)90132-7. ISSN 0968-0004. PMID 8291080.
- ^ Ochoa D, Bradley D, Beltrao P (February 2018). "Evolution, dynamics and dysregulation of kinase signalling". Current Opinion in Structural Biology. 48: 133–140. doi:10.1016/j.sbi.2017.12.008. PMID 29316484.
- ^ Renugopalakrishnan V, Garduño-Juárez R, Narasimhan G, Verma CS, Wei X, Li P (November 2005). "Rational design of thermally stable proteins: relevance to bionanotechnology". Journal of Nanoscience and Nanotechnology. 5 (11): 1759–1767. doi:10.1166/jnn.2005.441. PMID 16433409.
- ^ Hult K, Berglund P (August 2003). "Engineered enzymes for improved organic synthesis". Current Opinion in Biotechnology. 14 (4): 395–400. doi:10.1016/S0958-1669(03)00095-8. PMID 12943848.
- ^ Jiang L, Althoff EA, Clemente FR, Doyle L, Röthlisberger D, Zanghellini A, Gallaher JL, Betker JL, Tanaka F, Barbas CF, Hilvert D, Houk KN, Stoddard BL, Baker D (March 2008). "De novo computational design of retro-aldol enzymes". Science. 319 (5868): 1387–91. Bibcode:2008Sci...319.1387J. doi:10.1126/science.1152692. PMC 3431203. PMID 18323453.
- ^ a b Sun Y, Cheng J (May 2002). "Hydrolysis of lignocellulosic materials for ethanol production: a review". Bioresource Technology. 83 (1): 1–11. doi:10.1016/S0960-8524(01)00212-7. PMID 12058826.
- ^ a b Kirk O, Borchert TV, Fuglsang CC (August 2002). "Industrial enzyme applications". Current Opinion in Biotechnology. 13 (4): 345–351. doi:10.1016/S0958-1669(02)00328-2. PMID 12323357.
- ^ a b c Briggs DE (1998). Malts and Malting (1st ed.). London: Blackie Academic. ISBN 978-0412298004.
- ^ Dulieu C, Moll M, Boudrant J, Poncelet D (2000). "Improved performances and control of beer fermentation using encapsulated alpha-acetolactate decarboxylase and modeling". Biotechnology Progress. 16 (6): 958–65. doi:10.1021/bp000128k. PMID 11101321. S2CID 25674881.
- ^ Tarté R (2008). Ingredients in Meat Products Properties, Functionality and Applications. New York: Springer. p. 177. ISBN 978-0-387-71327-4.
- ^ "Chymosin – GMO Database". GMO Compass. European Union. 10 July 2010. Archived from the original on 26 March 2015. Retrieved 1 March 2015.
- ^ Molimard P, Spinnler HE (February 1996). "Review: Compounds Involved in the Flavor of Surface Mold-Ripened Cheeses: Origins and Properties". Journal of Dairy Science. 79 (2): 169–184. doi:10.3168/jds.S0022-0302(96)76348-8.
- ^ Guzmán-Maldonado H, Paredes-López O (September 1995). "Amylolytic enzymes and products derived from starch: a review". Critical Reviews in Food Science and Nutrition. 35 (5): 373–403. doi:10.1080/10408399509527706. PMID 8573280.
- ^ a b "Protease – GMO Database". GMO Compass. European Union. 10 July 2010. Archived from the original on 24 February 2015. Retrieved 28 February 2015.
- ^ Alkorta I, Garbisu C, Llama MJ, Serra JL (January 1998). "Industrial applications of pectic enzymes: a review". Process Biochemistry. 33 (1): 21–28. doi:10.1016/S0032-9592(97)00046-0.
- ^ Bajpai P (March 1999). "Application of enzymes in the pulp and paper industry". Biotechnology Progress. 15 (2): 147–157. doi:10.1021/bp990013k. PMID 10194388. S2CID 26080240.
- ^ Begley CG, Paragina S, Sporn A (March 1990). "An analysis of contact lens enzyme cleaners". Journal of the American Optometric Association. 61 (3): 190–4. PMID 2186082.
- ^ Farris PL (2009). "Economic Growth and Organization of the U.S. Starch Industry". In BeMiller JN, Whistler RL (eds.). Starch Chemistry and Technology (3rd ed.). London: Academic. ISBN 9780080926551.
추가 정보
일반
어원과 역사
| 효소구조 및 메커니즘
동태와 억제
|




![{\displaystyle {\ce {CO2{}+H2O->[{\text{Carbonic anhydrase}}]H2CO3}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/cb4c8837b26e96fe552c17d863f93e0618cd998b)
![{\displaystyle {\ce {CO2{}+H2O<-[{\text{Carbonic anhydrase}}]H2CO3}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/618e95485aa1c3c44a29c557ac448ae5b544ff07)



 평균값은 약
평균값은 약