정합성
Conformational proofreading정합성 교정 또는 정합성 선택은 분자 인식기와 그 대상 사이에 구조적 불일치 또는 에너지 장벽이 도입되어 인식의 특수성과 품질을 향상시키는 분자 인식 시스템의 일반적인 메커니즘이다.[1][2][3][4][5][6]정합성 교정은 에너지의 소비를 요구하지 않으므로 어떤 분자 인식 시스템에서도 사용될 수 있다.정합성 교정은 인식자가 많은 유사한 경쟁자들 사이에서 적절한 목표를 선택해야 하는 시나리오에서 특히 유용하다.null
올바른 바인딩과 잘못된 바인딩의 밸런싱
분자 인식은 시끄럽고 혼잡한 생물학적 환경에서 이루어지며, 인식자는 종종 유사한 다양한 경쟁자들 사이에서 목표물을 선정하는 과제에 대처해야 한다.예를 들어, 리보솜은 구조적으로 유사한 많은 tRNA 중 mRNA 코돈과 일치하는 정확한 tRNA를 선택해야 한다.인식기와 정확한 표적이 자물쇠와 키처럼 완벽하게 일치한다면, 바인딩 시 변형이 필요하지 않기 때문에 바인딩 확률이 높을 것이다.동시에, 인식자는 유사한 구조를 가진 선수와 높은 확률로 결합할 수도 있다.인식자(잠금)와 키 사이에 구조적 불일치를 도입하면 정확한 표적에 대한 결합 확률은 감소하지만, 유사한 잘못된 표적에 대한 결합 확률은 더욱 감소하여 특수성이 개선된다.그러나 너무 많은 변형을 도입하면 정확한 표적에 대한 결합 확률을 현저히 감소시킨다.따라서 정확한 바인딩 확률을 최대화하는 것과 잘못된 바인딩 확률을 최소화하는 것 사이의 최적의 균형은 인식자가 목표치를 약간 벗어나 있을 때 달성된다.이는 유도 적합[7] 메커니즘과 같은 분자 인식 프로세스 중 순응적 변화가 인식의 특수성을 높이는 데 유리함을 시사한다.null
호몰로지 검색을 위해 호몰로 재조합에 의해 사용
일치 교정 메커니즘은 유사한 DNA 시퀀스를 구별하기 위해 동음이의 재조합 시스템에 활용된다.[3][4]동질 재조합은 동질 DNA 분자 간의 유전 물질의 교환을 용이하게 한다.이 중요한 과정은 매우 다양한 이질적 시퀀스 내에서 특정한 동질적 DNA 시퀀스를 검출할 것을 요구한다.이 검출은 대장균의 RecA 또는 다른 유기체의 슈퍼 패밀리의 구성원에 의해 매개된다.RecA는 먼저 한 가닥의 DNA를 따라 중합하고, 그 다음 이 단백질-DNA 필라멘트는 이중 가닥 DNA를 따라 동질성을 찾는다.RecA-DNA 필라멘트에서 베이스 간 거리는 이중 스트랜드의 베어 3.4 4(평균[8] 50 %)에 대해 크게 증가한다.이것은 이중 가닥의 DNA가 동질성을 검사하기 위해 같은 크기만큼 늘어나야 하기 때문에 탐색에 상당한 에너지 장벽이 된다.DNA 인식 프로세스를 신호 검출 문제로 공식화함으로써, 실험적으로 관찰된 RecA 유도 DNA 변형과 결합 정력제를 미세 조정하여 최적의 시퀀스 검출이 가능하도록 하는 것으로 나타났다.변형량은 동질 DNA 시퀀스에 대한 결합은 약간만 감소하는 반면 잘못된 시퀀스에 대한 결합은 크게 감소하는 것이다.이것은 정확히 일치 교정 메커니즘이다.null
동질 재조합에 의한 일치교정을 위한 실험적 증거
C의 그룹.데커(Delft University)는 자기와 광학 핀셋을 결합해 호몰로지 검색에 관련된 상호작용을 직접 조사했다.[9]그들은 동종학 검색과 인식은 나선형의 개방을 필요로 하며 따라서 DNA를 풀어 가속할 수 있다는 것을 발견했다.이것은 정확히 일치 교정 모델에 의해 예측된 에너지 장벽이다.이 데이터는 DNA 결합 사이트 사이의 거리에 따라 검색 프로세스의 충실도가 좌우되는 동종학 인식을 위한 물리적 그림을 나타낸다.저자들은 측정치에 대한 그들의 해석이 "순응적인 교정 체계와 유사하다"고 결론짓는다.여기서 recA 필라멘트가 아닌 dsDNA는 활성 검색 엔터티로 인식한다.dsDNA의 목표 경계 상태와 바인딩되지 않은 상태 사이에 큰 일치 불일치가 존재한다.대상 경계 상태는 위에서 논의한 바와 같이 정력적으로 불리한 중간 상태를 통해 접근한다.순응적 불일치는 인식 반응의 선택성을 향상시킨다."즉, 그들은 정력적인 장벽을 확인했고, 실제로 이중 가닥의 DNA가 이 장벽을 통과해야 하기 때문에 능동적인 참여자라는 것을 보여주었다.null
tRNA 디코딩을 위해 리보솜에 사용
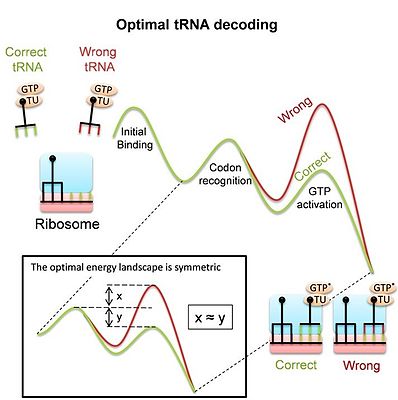
리보솜은 번역 과정에서 단백질을 합성하기 위해서는 코돈과 tRNA를 짝을 지어 mRNA를 해독해야 하는 복잡한 분자 기계다.디코딩은 체력의 주요 결정 요인이며, 많은 유사한 경쟁자들 사이에서 정확하고 빠른 올바른 tRNA를 선택해야 한다.대부분의 바인딩 이벤트는 비매칭("비인지") tRNAs에 의해 발생하며, 바인딩 사이트를 비우기 위해 리보솜은 가능한 한 빨리 이들을 거부해야 한다는 것을 명심해야 한다.동시에 리보솜은 일치하는 tRNA를 충분히 오랫동안 묶어두어야 단백질 합성 과정이 뒤따를 수 있다.tRNA 디코딩의 중요성에도 불구하고, 현대의 리보솜, 특히 디코딩 중 그 큰 순응적 변화가 디코더로서의 직무에 적응한 결과인지 아니면 다른 제약조건의 결과인지 최근까지 불분명했다.최근의 연구는[5] 경쟁하는 tRNA 기판들 간에 최적의 구별을 제공하는 에너지 환경을 도출했고, 따라서 최적의 tRNA 디코딩을 도출했다.최적의 경관은 대칭(이미지 참조)이다.이 연구는 원핵 리보솜의 측정된 지형이 실제로 대칭적이라는 것을 보여준다.이 모델은 디코딩 중 리보솜과 tRNA의 순응적 변화가 그러한 최적의 tRNA 디코더를 얻기 위한 수단임을 시사한다.동음 재조합과 tRNA 디코딩 모두 정합성 교정을 활용한다는 사실은 이것이 분자 인식 시스템에 의해 광범위하게 활용될 수 있는 일반적인 메커니즘임을 시사한다.null
다른 생물학적 시스템에서는
인체 자외선 손상 수리
최근의 연구는 일치 교정법이 인간의 DNA 수리 메커니즘에 의해 사용된다는 것을 보여준다.[10]이번 연구는 뉴클레오티드 절연수리(NER) 초기 단계에서 DNA 수리 단백질이 UV로 인한 손상을 인간 게놈에서 어떻게 스캔하느냐는 문제에 초점을 맞췄다.상세한 단일 분자 측정은 인간의 UV에 손상된 DNA 결합 단백질(UV-DDB)이 3D 검색을 수행하는 방법을 밝혀냈다.저자들은 "UV-DDB는 손상 부위에서 장수명 비운동성 UV-DDB 조광기(DDB1-DDB2)2를 형성하기 전에 별도의 단계로 DNA의 부지를 조사한다"고 밝혔다.손상되지 않은 DNA와 손상된 DNA의 과도 결합 분자에 대한 분리의 비율 분석은 세 개의 크기 순서에 걸쳐 여러 번의 체류 시간을 보여준다.이러한 중간 상태는 안정적인 손상 감지를 위한 궤적에서 분리된 UV-DDB 컨포머를 나타내는 것으로 생각된다.저자들은 상세한 운동 측정에서 UV-DDB가 복수의 매개체를 통해 정합성 교정 메커니즘을 사용하여 병변을 인식한다고 결론짓는다.null
기타 인식 체계
운동 교정과의 관계
운동 교정[11][12] 스키마에서는 정확하거나 부정확한 콤플렉스를 형성하는 동안 시간 지연(동일하게, 되돌릴 수 없는 중간 단계)이 도입된다.이 시간 지연은 두 단지의 생산률을 낮추지만 평형 한계를 넘어 충실도를 높인다.그 계획의 불가역성은 에너지원을 필요로 한다.운동 교정의 시간 지연은 순응 교정의 공간적 차이와 유사하다.그러나 순응 교정은 에너지를 소비하지 않는 평형 체계가 될 수 있다.null
참조
- ^ Savir Y & Tlusty T (2007). Scalas, Enrico (ed.). "Conformational Proofreading: The Impact of Conformational Changes on the Specificity of Molecular Recognition". PLOS ONE. 2 (5): e468. Bibcode:2007PLoSO...2..468S. doi:10.1371/journal.pone.0000468. PMC 1868595. PMID 17520027.
- ^ Savir Y, Tlusty T (2008). "Optimal Design of a Molecular Recognizer : Molecular Recognition as a Bayesian Signal Detection Problem". IEEE J Sel Topics Signal Process. 2 (3): 390–399. arXiv:1007.4527. Bibcode:2008ISTSP...2..390S. doi:10.1109/JSTSP.2008.923859. S2CID 7210763.
- ^ a b c Savir Y, Tlusty T (2010). "RecA-mediated homology search as a nearly optimal signal detection system". Molecular Cell. 40 (3): 388–96. arXiv:1011.4382. doi:10.1016/j.molcel.2010.10.020. PMID 21070965. S2CID 1682936.
- ^ a b c Rambo RP, Williams GJ, Tainer JA (2010). "Achieving Fidelity in Homologous Recombination Despite Extreme Complexity: Informed Decisions by Molecular Profiling". Molecular Cell. 40 (3): 347–48. doi:10.1016/j.molcel.2010.10.032. PMC 3003302. PMID 21070960.
- ^ a b c Savir, Yonatan; Tlusty, Tsvi (Apr 11, 2013). "The ribosome as an optimal decoder: a lesson in molecular recognition". Cell. 153 (2): 471–9. Bibcode:2013APS..MARY46006T. doi:10.1016/j.cell.2013.03.032. PMID 23582332.
- ^ Alon U (2008). "Journal Club". Nature. 453 (7196): 701. Bibcode:2008Natur.453..701A. doi:10.1038/453701e. S2CID 29639642.
- ^ Koshland, D. E. (1958). "Application of a Theory of Enzyme Specificity to Protein Synthesis". Proc Natl Acad Sci U S A. 44 (2): 98–104. Bibcode:1958PNAS...44...98K. doi:10.1073/pnas.44.2.98. PMC 335371. PMID 16590179.
- ^ Chen Z, Yang H, Pavletich NP (2008). "Mechanism of homologous recombination from the RecA-ssDNA/dsDNA structures". Nature. 453 (7194): 489–4. Bibcode:2008Natur.453..489C. doi:10.1038/nature06971. PMID 18497818. S2CID 4416531.
- ^ De Vlaminck I, van Loenhout MT, Zweifel L, den Blanken J, Hooning K, Hage S, Kerssemakers J, Dekker C (2012). "Mechanism of Homology Recognition in DNA Recombination from Dual-Molecule Experiments". Molecular Cell. 46 (5): 616–624. doi:10.1016/j.molcel.2012.03.029. PMID 22560720.
- ^ Ghodke H, Wang H, Hsieh CL, Woldemeskel S, Watkins SC, Rapić-Otrin V, Van Houten B (May 6, 2014). "Single-molecule analysis reveals human UV-damaged DNA-binding protein (UV-DDB) dimerizes on DNA via multiple kinetic intermediates". Proc Natl Acad Sci U S A. 111 (18): 1862–71. Bibcode:2014PNAS..111E1862G. doi:10.1073/pnas.1323856111. PMC 4020048. PMID 24760829.
- ^ Hopfield JJ (1974). "Kinetic Proofreading: A New Mechanism for Reducing Errors in Biosynthetic Processes Requiring High Specificity". Proc Natl Acad Sci U S A. 71 (10): 4135–4139. Bibcode:1974PNAS...71.4135H. doi:10.1073/pnas.71.10.4135. PMC 434344. PMID 4530290.
- ^ Ninio J (1975). "Kinetic amplification of enzyme discrimination Biochimie". Biochimie. 57 (5): 587–595. doi:10.1016/S0300-9084(75)80139-8. PMID 1182215.



