역전사효소
Reverse transcriptase| 역전사효소 (RNA의존성 DNA중합효소) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 | |||||||||
| 식별자 | |||||||||
| 기호. | RVT_1 | ||||||||
| 팜 | PF00078 | ||||||||
| 빠맘 클랜 | CL0027 | ||||||||
| 인터프로 | IPR000477 | ||||||||
| 프로 사이트 | PS50878 | ||||||||
| SCOP2 | 1hmv/SCOPe/SUPFAM | ||||||||
| CDD | cd00304 | ||||||||
| |||||||||
| RNA방향DNA중합효소 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| EC 번호 | 2.7.7.49 | ||||||||
| CAS 번호 | 9068-38-6 | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 엔트리 | ||||||||
| ExPASy | NiceZyme 뷰 | ||||||||
| 케그 | KEGG 엔트리 | ||||||||
| 메타사이크 | 대사 경로 | ||||||||
| 프라이머리 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBum | ||||||||
| 진 온톨로지 | AmiGO / QuickGO | ||||||||
| |||||||||
역전사효소(RT)는 RNA 템플릿에서 상보적 DNA(cDNA)를 생성하는 데 사용되는 효소이며 역전사라고 불리는 과정이다.역전사효소는 HIV와 B형 간염과 같은 바이러스에 의해 게놈을 복제하기 위해, 숙주 게놈 내에서 증식하기 위해 역전사 이동 유전 요소에 의해, 그리고 진핵 세포에 의해 선형 염색체 끝에 있는 텔로미어를 확장하기 위해 사용된다.널리 알려진 믿음과는 달리, RNA에서 DNA로의 정보 전달이 명시적으로 [2][3][4]가능하기 때문에, 이 과정은 고전적인 중심 교의에 의해 기술된 유전 정보의 흐름을 위반하지 않는다.
레트로바이러스 RT는 RNA 의존성 DNA 중합효소 활성, 리보핵산가수분해효소 H(RNase H) 및 DNA 의존성 DNA 중합효소 활성의 세 가지 순차적 생화학 활성을 가지고 있다.집합적으로, 이러한 활동은 효소가 단일 가닥 RNA를 이중 가닥 cDNA로 변환할 수 있게 한다.레트로바이러스와 레트로트랜스포존에서, 이 cDNA는 숙주 게놈에 통합될 수 있으며, 여기서 새로운 RNA 복사가 숙주 세포 전사를 통해 만들어질 수 있습니다.동일한 반응 시퀀스는 분자 복제, RNA 배열 분석, 중합효소 연쇄 반응 또는 게놈 분석에 사용하기 위해 RNA를 DNA로 변환하는 데 널리 사용됩니다.
역사
역전사효소는 위스콘신-매디슨[5] 대학의 하워드 테민에 의해 발견되었고, 1970년 MIT에서 데이비드 볼티모어에 의해 두 개의 RNA 종양 바이러스, 즉 쥐 백혈병 바이러스와 다시 루스 육종 [6]바이러스로부터 독립적으로 분리되었다.1975년 노벨 생리의학상을 레나토 둘베코와 공동 수상했다.
잘 연구된 역전사효소에는 다음이 포함됩니다.
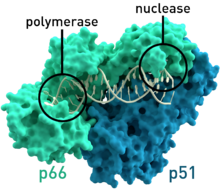
- 인간면역결핍바이러스형1(PDB: 1HMV)의 HIV-1 역전사효소에는 각각 66kDas [7]및 51kDas의 분자량을 갖는 2개의 서브유닛이 있다.
- Moloney murine 백혈병 바이러스의 M-MLV 역전사효소는 단일 75 kDa [8]단량체이다.
- 조류 골수아세포증 바이러스의 AMV 역전사효소도 63kDa 서브유닛과 95kDa [8]서브유닛의 2개의 서브유닛을 가진다.
- 진핵생물 염색체의 [9]텔로미어를 유지하는 텔로머라아제 역전사효소.
바이러스의 기능
효소는 복제 과정에서 역전사를 사용하는 바이러스에 의해 암호화되어 사용됩니다.레트로바이러스와 같은 역번역 RNA 바이러스는 그들의 RNA 게놈을 DNA로 역번역하는데 효소는 숙주 게놈에 통합되어 그것과 함께 복제된다.헤파드나바이러스와 같은 DNA 바이러스를 역변환하는 것은 RNA가 DNA 가닥을 조립하고 만드는 데 템플릿 역할을 할 수 있게 됩니다.HIV는 이 효소의 사용으로 인간을 감염시킨다.역전사효소 없이는 바이러스 게놈이 숙주 세포에 통합될 수 없어 [citation needed]복제에 실패할 수 있다.
역전사 또는 역전사 프로세스
역전사효소는 RNA 템플릿에서 이중 가닥 DNA를 생성합니다.
DNA의존성 DNA중합효소 활성이 결여된 역전사효소를 가진 바이러스 종에서는 호스트 부호화 DNA중합효소 δ에 의해 이중사슬 DNA를 생성할 수 있으며, 바이러스 DNA-RNA를 프라이머로 오인하여 프라이머 제거와 같은 메커니즘으로 이중사슬 DNA를 합성할 수 있다.Riginal RNA 템플릿.[citation needed]
역전사 또는 역전사로도 불리는 역전사의 과정은 매우 오류가 발생하기 쉬우며, 이 단계에서 돌연변이가 발생할 수 있습니다.이러한 돌연변이는 약물 내성을 유발할 수 있습니다.
레트로바이러스 역전사
VI급 ssRNA-RT 바이러스라고도 불리는 레트로바이러스는 DNA 중간체를 가진 RNA 역전사 바이러스이다.그들의 게놈은 5' 캡과 3' 폴리아데닐화 꼬리를 가진 양성의 단일 가닥 RNA 두 분자로 구성되어 있다.레트로바이러스의 예로는 인간면역결핍바이러스(HIV)와 인간T림프트로픽바이러스(HTLV)가 있다.이중 가닥 DNA의 생성은 세포에서[10] 다음과 같은 일련의 단계로 발생합니다.
- 리실 tRNA는 프라이머 역할을 하며 프라이머 결합 부위 또는 PBS라고 불리는 바이러스 RNA 게놈의 상보적인 부분으로 교배된다.
- 다음으로 역전사효소는 프라이머의 3' 말단에 DNA 뉴클레오티드를 첨가하여 바이러스 RNA의 U5(비부호화 영역)와 R 영역(RNA 분자의 양 끝에서 발견되는 직접적인 반복)을 보완하는 DNA를 합성한다.
- RNAse H라고 불리는 역전사효소 상의 도메인은 RNA의 5' 말단에서 U5 및 R 영역을 분해합니다.
- 그런 다음 tRNA 프라이머는 바이러스 게놈의 3' 말단으로 "점프"되고, 새로 합성된 DNA 가닥은 RNA의 상보적인 R 영역으로 교배된다.
- (2)에 첨가된 상보적 DNA(cDNA)는 더욱 확장된다.
- 대부분의 바이러스 RNA는 RNAse H에 의해 분해되고 PP 배열만 남습니다.
- 바이러스 RNA의 나머지 PP 조각을 프라이머로 사용하여 두 번째 DNA 가닥의 합성이 시작됩니다.
- tRNA 프라이머가 이탈하여 "점프"가 발생합니다.두 번째 가닥의 PBS는 첫 번째 가닥의 상보적인 PBS와 교배한다.
- 두 가닥은 원래의 바이러스 RNA 게놈의 완전한 이중 가닥 DNA 복사를 형성하기 위해 확장되며, 이것은 효소 인테그레이스에 의해 숙주의 게놈에 통합될 수 있습니다.
이중사슬 DNA의 생성은 또한 가닥 전이를 수반하며, 여기서 초기 RNA의존성 DNA 합성에서 게놈의 다른 끝의 수용체 템플릿 영역으로 짧은 DNA 생성물의 전위가 존재하며, 이는 나중에 DNA의존성 DNA [11]활성을 위해 역전사효소에 의해 도달되고 처리된다.
레트로바이러스 RNA는 5' 말단에서 3' 말단으로 배열된다.프라이머가 바이러스 RNA로 아닐되는 부위를 프라이머 결합 부위(PBS)라고 합니다.PBS 부위에 대한 RNA 5'end는 U5, PBS에 대한 RNA 3'end는 리더라고 불립니다.tRNA 프라이머는 14~22개의 뉴클레오티드 사이에서 분리되며 PBS에서 바이러스 RNA와 염기쌍의 이중화를 형성한다.PBS가 바이러스 RNA의 5' 말단 근처에 위치한다는 사실은 특이하다. 왜냐하면 역전사효소가 프라이머의 3' 끝에서 3' 방향으로 DNA를 합성하기 때문이다(새로 합성된 DNA 가닥에 대하여).따라서 프라이머와 역전사효소는 바이러스 RNA의 3' 말단으로 재배치되어야 합니다.이 위치를 바꾸기 위해서는 DNA 중합효소, 리보핵산가수분해효소 H(RNase H) 및 폴리뉴클레오티드 풀림 등을 포함한 다단계 및 다양한 효소가 필요하다.[12][13]
HIV 역전사효소는 또한 cDNA 합성 동안 바이러스 RNA를 분해하는 리보핵산가수분해효소 활성과 cDNA 가닥을 안티센스 DNA로 복사하여 이중 가닥 바이러스 DNA 중간체(vDNA)[14]를 형성하는 DNA 의존성 DNA 중합효소 활성을 가지고 있다.
세포 생활에서
역트랜스포존으로 알려진 진핵생물 게놈의 자기 복제 스트레칭은 RNA 중간체를 통해 게놈의 한 위치에서 다른 위치로 이동하기 위해 역전사효소를 이용합니다.그것들은 식물과 동물의 게놈에서 풍부하게 발견된다.텔로머라아제(Telomerase)는 인간을 포함한 많은 진핵생물에서 발견되는 또 다른 역전사효소이며, 이 RNA는 DNA [15]복제를 위한 템플릿으로 사용됩니다.
원핵생물의 역전사 효소에 대한 최초 보고는 1971년 프랑스(Beljanski et al., 1971a, 1972년)와 몇 년 후 소련(Romashchenko[16] 1977)에서 나왔다.이것들은 이후 역전사효소를 코드하는 별개의 배열인 박테리아 레트론의 일부로 광범위하게 설명되었고 msDNA 합성에 사용된다.DNA 합성을 시작하기 위해서는 프라이머가 필요하다.박테리아에서는 프라이머가 복제 [17]중에 합성된다.
오리건주의 Valerian Dolja는 바이러스의 다양성 때문에 역전사효소가 중심적인 [18]역할을 하면서, 바이러스가 세포 생명의 발달에 진화적인 역할을 했다고 주장한다.
구조.
역전사효소는 다른 바이러스 핵산 [19][20]중합효소에서 발견되는 것과 유사한 "오른쪽" 구조를 사용합니다.레트로바이러스 역전사효소에는 전사기능 외에 복제에 필수적인 RNase H족에 속하는 도메인이 있다.RNA 템플릿을 분해함으로써 다른 DNA 가닥을 [21]합성할 수 있습니다.소화에서 나온 일부 조각들은 또한 DNA 중합효소(동일한 효소 또는 숙주 단백질)의 프라이머 역할을 하며, 다른 (플러스) [19]가닥을 만드는 역할을 한다.
레플리케이션의 충실도
레트로 바이러스의 라이프 사이클에는, 3개의 다른 레플리케이션 시스템이 있습니다.첫 번째 과정은 바이러스 RNA에서 바이러스 DNA의 역전사효소 합성으로, 그 후 새롭게 만들어진 상보적인 DNA 가닥을 형성한다.두 번째 복제 과정은 숙주 세포 DNA 중합효소가 통합된 바이러스 DNA를 복제할 때 발생합니다. 마지막으로, RNA 중합효소 II는 프로바이러스 DNA를 RNA에 전사하고, RNA는 바이러스 DNA로 채워집니다.이러한 복제 단계 중 [22]하나 또는 모든 단계에서 변환이 발생할 수 있습니다.
역전사효소는 대부분의 다른 DNA 중합효소와는 달리 교정능력이 없기 때문에 RNA를 DNA로 전사할 때 오류율이 높다.이 높은 오류율을 통해 교정된 복제 형태에 비해 돌연변이가 빠른 속도로 축적될 수 있습니다.Promega에 의해 상업적으로 이용 가능한 역전사 효소는 매뉴얼에 따르면 AMV의 경우 17,000개 중 [23]1개, M-MLV의 경우 30,000개 중 1개 범위의 오류율을 가지고 있다.
단일핵산 다형성을 생성하는 것 외에, 역전사효소는 전사 융합, 엑손 셔플링 및 인공 안티센스 [24][25]전사 생성과 같은 과정에도 관여하는 것으로 나타났다.생체 내에서 완전히 입증될 수 있는 역전사효소의 템플릿 전환 활성은 [26]모델 유기체의 게놈에서 수천 개의 비주석 전사를 발견한 원인 중 하나였을 수 있다.
템플릿 스위칭
두 개의 RNA 게놈이 각 레트로바이러스 입자에 포장되어 있지만, 감염 후 각 바이러스는 오직 하나의 프로바이러스를 [27]생성한다.감염 후 역전사는 두 게놈 복사본 간의 템플릿 전환(복사 선택 재조합)[27]을 수반한다.RNA 전사 효소가 템플릿을 전환하는 이유를 제시하는 두 가지 모델이 있습니다.첫 번째 강제 복제 선택 모델은 역전사 효소가 흠집을 만났을 때 RNA 템플릿을 변화시켜 재조합이 바이러스 게놈 무결성을 유지하는 데 필수적이라는 것을 암시한다.두 번째 동적 선택 모델은 RNAse 기능과 중합효소 기능이 동기화 속도 면에서 일치하지 않을 때 역전사효소가 템플릿을 변경한다는 것을 시사하며, 이는 재조합이 무작위로 일어나며 게놈 손상에 반응하지 않음을 의미한다.Rawson 등의 연구는 두 가지 [27]재조합 모델을 모두 지원했다.각 복제 [28]주기마다 게놈당 5~14개의 재조합 이벤트가 발생합니다.템플릿 전환(재결합)은 게놈 무결성을 유지하고 손상된 [29][27]게놈을 복구하기 위한 복구 메커니즘으로서 필요한 것으로 보인다.
적용들

항바이러스제
HIV는 유전자 물질을 복제하고 새로운 바이러스(레트로바이러스 증식 서클의 일부)를 생성하기 위해 역전사효소를 사용하기 때문에 특정 약물이 그 과정을 방해하고 그 성장을 억제하도록 설계되어 있다.집합적으로 이러한 약물은 역전사효소 억제제로 알려져 있으며, 뉴클레오사이드 및 뉴클레오티드 유사체 디도부딘(상표명 레트로비르), 라미부딘(에피비르) 및 테노포비르(비라무네)[citation needed]와 같은 비뉴클레오시드 억제제를 포함한다.
분자생물학
역전사효소는 역전사 중합효소 연쇄반응이라고 불리는 기술로 RNA에 중합효소 연쇄반응 기술을 적용하는 연구에서 흔히 사용된다.기존의 PCR 기술은 DNA 가닥에만 적용될 수 있지만 역전사효소의 도움으로 RNA가 DNA로 전사될 수 있어 RNA 분자의 PCR 분석이 가능하다.역전사효소는 mRNA에서 cDNA 라이브러리를 생성하는 데도 사용됩니다.역전사효소의 상업적 이용가능성은 다른 효소와 함께 과학자들이 RNA를 복제, 배열, 특성화할 수 있게 해주었기 때문에 분자생물학 분야의 지식을 크게 향상시켰다.
「 」를 참조해 주세요.
레퍼런스
- ^ PDB: 3KLF;Tu X, Das K, Han Q, Bauman JD, Clark AD, Hou X, Frenkel YV, Gaffney BL, Jones RA, Boyer PL, Hughes SH, Sarafianos SG, Arnold E (October 2010). "Structural basis of HIV-1 resistance to AZT by excision". Nature Structural & Molecular Biology. 17 (10): 1202–9. doi:10.1038/nsmb.1908. PMC 2987654. PMID 20852643.
- ^ Crick F (August 1970). "Central dogma of molecular biology". Nature. 227 (5258): 561–3. Bibcode:1970Natur.227..561C. doi:10.1038/227561a0. PMID 4913914. S2CID 4164029.
- ^ Sarkar S (1996). The Philosophy and History of Molecular Biology: New Perspectives. Dordrecht: Kluwer Academic Publishers. pp. 187–232.
- ^ Danchin É, Pocheville A, Rey O, Pujol B, Blanchet S (2019). "Epigenetically facilitated mutational assimilation: epigenetics as a hub within the inclusive evolutionary synthesis". Biological Reviews. 94 (1): 259–282. doi:10.1111/brv.12453. PMC 6378602. S2CID 67861162.
- ^ Temin HM, Mizutani S (June 1970). "RNA-dependent DNA polymerase in virions of Rous sarcoma virus". Nature. 226 (5252): 1211–3. doi:10.1038/2261211a0. PMID 4316301. S2CID 4187764.
- ^ Baltimore D (June 1970). "RNA-dependent DNA polymerase in virions of RNA tumour viruses". Nature. 226 (5252): 1209–11. doi:10.1038/2261209a0. PMID 4316300. S2CID 4222378.
- ^ Ferris AL, Hizi A, Showalter SD, Pichuantes S, Babe L, Craik CS, Hughes SH (April 1990). "Immunologic and proteolytic analysis of HIV-1 reverse transcriptase structure" (PDF). Virology. 175 (2): 456–64. doi:10.1016/0042-6822(90)90430-y. PMID 1691562.
- ^ a b Konishi A, Yasukawa K, Inouye K (July 2012). "Improving the thermal stability of avian myeloblastosis virus reverse transcriptase α-subunit by site-directed mutagenesis" (PDF). Biotechnology Letters. 34 (7): 1209–15. doi:10.1007/s10529-012-0904-9. hdl:2433/157247. PMID 22426840. S2CID 207096569.
- ^ Autexier C, Lue NF (June 2006). "The structure and function of telomerase reverse transcriptase". Annual Review of Biochemistry. 75 (1): 493–517. doi:10.1146/annurev.biochem.75.103004.142412. PMID 16756500.
- ^ Bio-Medicine.org - 2009년 2월 17일 Retrovirus 취득
- ^ Telesnitsky A, Goff SP (1993). "Strong-stop strand transfer during reverse transcription". In Skalka MA, Goff SP (eds.). Reverse transcriptase (1st ed.). New York: Cold Spring Harbor. p. 49. ISBN 978-0-87969-382-4.
- ^ Bernstein A, Weiss R, Tooze J (1985). "RNA tumor viruses". Molecular Biology of Tumor Viruses (2nd ed.). Cold Spring Harbor, N.Y.: Cold Spring Harbor Laboratory.
- ^ Moelling K, Broecker F (April 2015). "The reverse transcriptase-RNase H: from viruses to antiviral defense". Annals of the New York Academy of Sciences. 1341 (1): 126–35. Bibcode:2015NYASA1341..126M. doi:10.1111/nyas.12668. PMID 25703292. S2CID 42378727.
- ^ Kaiser GE (January 2008). "The Life Cycle of HIV". Doc Kaiser's Microbiology Home Page. Community College of Baltimore Count. Archived from the original on 2010-07-26.
- ^ Krieger M, Scott MP, Matsudaira PT, Lodish HF, Darnell JE, Zipursky L, Kaiser C, Berk A (2004). Molecular cell biology. New York: W.H. Freeman and CO. ISBN 978-0-7167-4366-8.
- ^ Romashchenko AG, et al. (1977). "Otdelenie ot preparatov DNK-polimeraz I RNK-zavisimoy DNK-polimeraz; oshistka i svoystva fermenta". Proceedings of the USSR Academy of Sciences. 233: 734–737.
- ^ Hurwitz J, Leis JP (January 1972). "RNA-dependent DNA polymerase activity of RNA tumor viruses. I. Directing influence of DNA in the reaction". Journal of Virology. 9 (1): 116–29. doi:10.1128/JVI.9.1.116-129.1972. PMC 356270. PMID 4333538.
- ^ Arnold C (17 July 2014). "Could Giant Viruses Be the Origin of Life on Earth?". National Geographic. Retrieved 29 May 2016.
- ^ a b Sarafianos SG, Marchand B, Das K, Himmel DM, Parniak MA, Hughes SH, Arnold E (January 2009). "Structure and function of HIV-1 reverse transcriptase: molecular mechanisms of polymerization and inhibition". Journal of Molecular Biology. 385 (3): 693–713. doi:10.1016/j.jmb.2008.10.071. PMC 2881421. PMID 19022262.
- ^ Hansen JL, Long AM, Schultz SC (August 1997). "Structure of the RNA-dependent RNA polymerase of poliovirus". Structure. 5 (8): 1109–22. doi:10.1016/S0969-2126(97)00261-X. PMID 9309225.
- ^ Schultz SJ, Champoux JJ (June 2008). "RNase H activity: structure, specificity, and function in reverse transcription". Virus Research. 134 (1–2): 86–103. doi:10.1016/j.virusres.2007.12.007. PMC 2464458. PMID 18261820.
- ^ Bbenek K, Kunkel AT (1993). "The fidelity of retroviral reverse transcriptases". In Skalka MA, Goff PS (eds.). Reverse transcriptase. New York: Cold Spring Harbor Laboratory Press. p. 85. ISBN 978-0-87969-382-4.
- ^ "Promega kit instruction manual" (PDF). 1999. Archived from the original (PDF) on 2006-11-21.
- ^ Houseley J, Tollervey D (August 2010). "Apparent non-canonical trans-splicing is generated by reverse transcriptase in vitro". PLOS ONE. 5 (8): e12271. Bibcode:2010PLoSO...512271H. doi:10.1371/journal.pone.0012271. PMC 2923612. PMID 20805885.
- ^ Zeng XC, Wang SX (June 2002). "Evidence that BmTXK beta-BmKCT cDNA from Chinese scorpion Buthus martensii Karsch is an artifact generated in the reverse transcription process". FEBS Letters. 520 (1–3): 183–4, author reply 185. doi:10.1016/S0014-5793(02)02812-0. PMID 12044895. S2CID 24619868.
- ^ van Bakel H, Nislow C, Blencowe BJ, Hughes TR (2011). "Response to "The Reality of Pervasive Transcription"". PLOS Biology. 9 (7): e1001102. doi:10.1371/journal.pbio.1001102. PMC 3134445.
- ^ a b c d Rawson JM, Nikolaitchik OA, Keele BF, Pathak VK, Hu WS (November 2018). "Recombination is required for efficient HIV-1 replication and the maintenance of viral genome integrity". Nucleic Acids Research. 46 (20): 10535–10545. doi:10.1093/nar/gky910. PMC 6237782. PMID 30307534.
- ^ Cromer D, Grimm AJ, Schlub TE, Mak J, Davenport MP (January 2016). "Estimating the in-vivo HIV template switching and recombination rate". AIDS (London, England). 30 (2): 185–92. doi:10.1097/QAD.0000000000000936. PMID 26691546. S2CID 20086739.
- ^ Hu WS, Temin HM (November 1990). "Retroviral recombination and reverse transcription". Science. New York, N.Y. 250 (4985): 1227–33. Bibcode:1990Sci...250.1227H. doi:10.1126/science.1700865. PMID 1700865.
외부 링크
- 미국 국립의학도서관의 RNA+전사효소 제목(MeSH)
- 역전사효소 작용과 3가지 역전사효소 억제제의 애니메이션
- RCSB PDB 이달의 분자(2002년 9월)
- HIV 레플리케이션 3D 메디컬 애니메이션.(2008년 11월).Boehringer Ingelheim의 비디오.
- Goodsell DS. "Molecule of the Month: Reverse Transcriptase (Sep 2002)". Research Collaboratory for Structural Bioinformatics (RCSB) Protein Data Bank (PDB). Retrieved 2013-01-13.
- PDB for UniProt: P03366(인간면역결핍바이러스 Reverse transcriptase)에서 PDB에서 사용할 수 있는 모든 구조 정보의 개요는 PDBe-KB입니다.
- TWiV 904: 50년간 역전사 효소인 Vincent Racaniello가 콜드 스프링 하버 연구소로 이동해 데이비드 볼티모어, 존 코핀(과학자), 해롤드 바르무스와 1970년 레트로바이러스 역전사 효소의 발견과 그것이 생명 과학 연구에 미치는 영향에 대해 이야기를 나누고 있습니다.




