페닐알라닌 수산화효소
Phenylalanine hydroxylase| PAH | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| 식별자 | |||||||||||||||||||||||||
| 별칭 | PAH, PH, PKU, PKU1, 페닐알라닌 수산화효소 | ||||||||||||||||||||||||
| 외부 ID | OMIM: 612349 MGI: 97473 HomoloGene: 234 GeneCard: PAH | ||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| 직교체 | |||||||||||||||||||||||||
| 종 | 인간 | 마우스 | |||||||||||||||||||||||
| 엔트레스 | |||||||||||||||||||||||||
| 앙상블 | |||||||||||||||||||||||||
| 유니프로트 | |||||||||||||||||||||||||
| RefSeq(mRNA) | |||||||||||||||||||||||||
| RefSeq(단백질) | |||||||||||||||||||||||||
| 위치(UCSC) | Chr 12: 102.84 – 102.96Mb | Cr 10: 87.52 – 87.58Mb | |||||||||||||||||||||||
| PubMed 검색 | [3] | [4] | |||||||||||||||||||||||
| 위키다타 | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
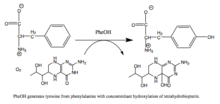
페닐알라닌 히드록실라아제(PAH) (EC 1.14.16.1)는 페닐알라닌의 방향족 사이드 체인의 히드록실화를 촉진시켜 티로신을 생성하는 효소다. PAH는 바이오테린 의존형 방향족 아미노산 하이드록실라아제의 3개 성분 중 하나로, 테트라하이드로비옵테린(BH4, pteridine coactor)을 사용하는 모노옥시게나제와 카탈루석을 위한 비히메 철이다. 반응하는 동안 분자 산소는 BH와4 페닐랄라닌 기질에 1개의 산소 원자를 순차적으로 결합하여 이질적으로 분해된다.[5] [6] 인간에게 있어서, 그것의 인코딩 유전자 PAH의 돌연변이는 대사 장애 페닐케톤뇨증을 초래할 수 있다.
효소 메커니즘
반응은 다음과 같은 단계를 거쳐 진행된다고 생각된다.
- Fe(II)-O-O-BH4 교량의 형성
- 페릴 옥소 수산화 중간 Fe(IV)를 산출하기 위한 O-O 본드의 이질성 분할=O
- Fe(IV)=O to hydroxylate penylalanine 기질에 대한 Tyrosine.[7]
철-페록시프테린 교량의 형성 및 갈라짐. 증거가 Fe(IV)=O를 히드록시 중간으로서 강력하게 지지하고 있지만,[8] 이질적 분할에 앞서 Fe(II)-O-O-BH4 교량의 형성에 기초하는 기계론적 세부사항은 여전히 논란으로 남아 있다. 철과 프테린 공동 인자 사이의 근접성이 다른 모델과 카탈루션 중에 철로 조정된 것으로 가정되는 물 분자의 수에 기초하여 두 가지 경로가 제안되었다. 한 모델에 따르면, 초기에는 철 다이옥시겐 복합체가 형성되어 FeO와2+2 FeO의3+2− 공명 하이브리드로서 안정화된다. 그러면 활성화된2 O는 BH를4 공격하여 전자-피질 프테린 링과 전자 부성 다이옥시겐 종 사이의 전하 분리를 특징으로 하는 전이 상태를 형성한다.[9] 이후 Fe(II)-O-O-BH4 브리지가 형성된다. 반면, BH4가 철의 첫 번째 조정 쉘에 위치하고 철이 어떤 물 분자와도 조정되지 않는다는 가정 하에 이 교량의 형성을 모델링하였다. 이 모델은 임계 매개체로 프테린 과산화물 과산화물과 관련된 다른 메커니즘을 예측한다.[10] 일단 형성되면 Fe(II)-O-O-BH4 브리지는 Fe(IV)=O와 4a-hydroxytetrahydrobiopterin에 대한 O-O 결합의 이질 분리를 통해 깨진다. 따라서 분자 산소는 프테린 링과 페닐알라닌을 히드산화하는데 사용되는 두 산소 원자의 근원이다.
페릴옥소 중간재를 이용한 페닐알라닌의 히드록실화. 이 메커니즘은 Fe(IV)=O(페록시프테린과는 반대로) 히드록시린 중간을 수반하기 때문에 페닐알라닌의 BH4 공actor와 히드록시화(hydroxylation)의 산화가 분해되어 BH의4 비생산적인 소비와 HO22 형성을 초래할 수 있다.[7] 그러나 생산성이 있을 때는 페닐알라닌에 Fe(IV)=O 중량을 첨가하여 철을 페릴에서 철 상태로 감소시키는 전기영역 방향족 대체 반응을 한다.[7] 처음에는 아렌 산화물 또는 급진적 중간체가 제안되었지만, 관련 트립토판과 티로신 수산화물 분석은 대신 Fe(IV)=O를 수산화군이 아닌 물 리간드로 조정해야 하는 양이온 중간을 통해 반응이 진행된다는 것을 시사했다.[7][11] 이 양이온 중간은 이후 1,2-하이드라이드 NIH 시프트를 거치며, 티로신 제품을 형성하기 위해 tautomerizes하는 디에논 중간이 생성된다.[12] PAH의 카르비놀아민 제품을 퀴노노이드 디하이드로바이오프테린(qBH2)에 수화시켜 프테린 공actor가 재생되며, 그 후 BH로4 환원된다.[13]
효소조절
PAH는 알로스테릭 규제의 모르핀 모델을 사용할 것을 제안한다.[14][15]
포유류 PAH는 두 개의 뚜렷한 구조의 테트라머로 구성된 평형 속에 존재하며, 평형의 일부로 하나 이상의 조광체 형태를 가지고 있다. 이 행동은 분열성 발기부전적 메커니즘과 일치한다.[15]
많은 연구에서 포유류 PAH가 pH와 리간드 결합과 같은 다양한 요인이 효소 활성과 단백질 안정성에 영향을 미치는 것으로 보고되는 PBGS(porphobilinogen synthase)에 필적하는 행동을 보인다는 연구 결과가 나왔다.[15]
구조
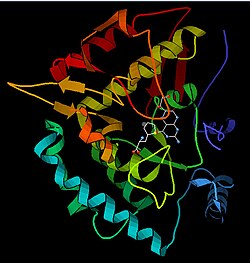
PAH 모노머(51.9 kDa)는 Phe-binding ACT 하위 도메인을 포함하는 규제 N-터미널 영역(1-117)과 촉매 도메인(리소스 118–427), 동일한 모노머의 과점화를 담당하는 C-터미널 영역(리소스 428–453)의 세 개의 구별 영역으로 구성된다. 광범위한 결정학적 분석이 수행되었으며, 특히 활성 부지를 검사하기 위해 피터린 및 철로 조정된 촉매 영역에 대해 수행되었다. N-단자 규제 영역의 구조도 결정되었으며, 호몰로ous tyrosine hydroxylase C-단자 4중자 영역의 해결된 구조와 함께 4중자 PAH의 구조 모델이 제안되었다.[13] X선 결정학을 이용하여 전신 쥐 PAH의 구조를 실험적으로 결정하여 효소의 자동 억제, 즉 휴식 상태 형태를 보여주었다.[16] 휴식상태 형태(RS-PAH)는 건축학적으로 활성화된 형태(A-PAH)와 구별된다.[17] 현재 A-PAH의 전신구조가 부족하지만, A-PAH의 특징인 Phe 안정화된 ACT-ACT 인터페이스가 결정되어 SACS 분석에 기초한 A-PAH의 구조모델이 제안되었다.[18][19]
촉매영역
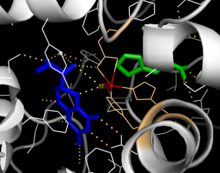
촉매 영역의 해결된 결정 구조는 글루탐산 잔류물 3개, 히스티딘 2개, 티로신 1개도 존재하며 철 제본으로 되어 있지만 활성 부위는 주로 소수성 잔류물에 의해 정렬된 개방되고 넓은 포켓으로 구성되어 있음을 나타낸다.[13] 철 원자의 조정 상태와 활성 사이트 내의 BH4에 대한 근접성에 대해 모순된 증거가 존재한다. 결정학적 분석에 따르면 Fe(II)는 물, His285, His290, Glu330(2-his-1-carboxylate face triad arrangement)에 의해 팔면 기하학으로 조정된다.[20] 결정 구조에 Phe 아날로그를 포함시키면 철이 6개에서 5개 조정 상태로 바뀌며, 단일 물 분자를 포함하고 Glu330에 대한 biddate 조율을 하며 산소가 결합할 수 있는 부지를 개방한다. 비록 프테린 공효소가 두 번째 조정 영역에 남아있지만, BH4는 동시에 철 원자 쪽으로 이동한다.[21] 한편, NMR과 분자 모델링 분석에 기초한 경쟁 모델은 BH4가 철로 직접 조정되는 동안 촉매 사이클 동안 모든 조정된 물 분자가 활성 부위에서 강제 배출된다는 것을 시사한다.[22] 위에서 논의한 바와 같이, 이러한 불일치를 해결하는 것은 PAH 촉매의 정확한 메커니즘을 결정하는 데 중요할 것이다.
N-단말 규제 영역
N-터미널 영역(리소스 1-117)의 규제 특성은 구조적 유연성에 의해 부여된다.[23] 수소/중수소 교환 분석은 Phe의 알로스테릭 결합이 전세계적으로 PAH의 순응을 변화시켜 규제 영역과 촉매 영역 사이의 인터페이스가 용제에 점점 노출됨에 따라 활성 부위가 덜 가려지게 된다는 것을 나타낸다.[23][24][25] 이 관찰은 전체 길이 PAH에 대해 초기에 낮은 타이로신 형성률을 보이는 운동 연구와 일치한다. 그러나 N-단자영역이 부족한 잘린 PAH에 대해서는 이 지연 시간이 관찰되지 않으며, 전신 효소가 Phe와 사전 결합되어 있는 경우에는 관찰되지 않는다. N단자 도메인을 삭제하면 Phe에 대한 친화력이 거의 2배 증가하면서 지연 시간도 제거된다; 4차 하이드로바이옵테린 공동요인에 대한 V나max K에서는m 아무런 차이가 관찰되지 않는다.[26] 세르16은 추가적인 규제를 제공한다; 이 잔류물의 인산화 작용은 효소 순응을 변화시키지는 않지만 알로스테릭 활성화에 필요한 페의 농도를 감소시킨다.[25] 이 N-단자 규제 영역은 박테리아 PAH에서 관찰되지 않지만 세린 생합성 경로의 효소인 인산염 탈수소효소의 규제 영역에 대한 상당한 구조적 호몰로학을 보여준다.[25]
테트라머라이징 도메인
원핵생물 PAH는 단핵생물인 반면 진핵생물 PAH는 동핵생성과 동핵생성형 사이의 평형상태에서 존재한다.[7][13] 조광 인터페이스는 동일한 모노머를 연결하는 대칭 관련 루프로 구성되는 반면, 겹치는 C-단자 사선화 영역은 촉매 및 사선화 영역의 다른 상대적 방향(Flatmark, Erlanden)으로 특징지어지는 순응적으로 구별되는 조광기의 연결을 매개한다. 그에 따른 테트라머 대칭의 왜곡은 디머라이제이션 인터페이스의 차등 표면적에 뚜렷이 나타나며, 테트라메릭 대칭 타이로신 수산화효소와 PAH를 구별한다.[13] 도메인 교환 메커니즘은 C-단자 알파-헬릭스들이 유연한 C-단자 5-레시듀 힌지 영역을 중심으로 상호 순응을 변경하여 코일 구조를 형성하고, 사선 형태를 향해 평형을 이동하는 디머로부터 테트라머 형성을 중재하기 위해 제안되었다.[7][13][27] PAH의 호모디메트릭과 호모테트라메릭 형태는 모두 촉매적으로 활성화되지만, 두 형태는 서로 다른 운동학 및 규제를 나타낸다. 디머는 촉매 효율 감소 외에도 L-Peh(고농도에서 효소를 활성화하는)에 대해 양의 협력성을 보이지 않으며, 이는 L-Phep가 조광기-다이머 상호작용에 영향을 줌으로써 PAH를 임의적으로 조절한다는 것을 시사한다.[27]
생물 함수
PAH는 페닐알라닌 신진대사의 중요한 효소로, 이산화탄소와 물에 대한 완전한 카타볼리즘의 속도제한 단계를 촉진한다.[13][28] 페닐칼라닌 관련 경로를 통한 플럭스의 조절은 페닐케톤뇨리아(아래 참조)에서 관찰된 아미노산의 높은 혈장 수준의 독성으로 입증되었듯이 포유류 대사에서 매우 중요하다. 페닐알라닌의 주요 공급원은 섭취된 단백질이지만, 이 풀의 비교적 적은 양이 단백질 합성에 사용된다.[28] 대신에, 섭취된 페닐알라닌의 대부분은 티로신을 형성하기 위해 PAH를 통해 카타볼화된다; 히드록실 그룹의 추가는 벤젠 링이 후속 카타볼릭 단계에서 깨질 수 있도록 한다. 대사물이 소변으로 배설되는 페닐피루브산염으로의 트랜스아미션은 페닐알라닌 교체의 또 다른 경로를 나타내지만 PAH를 통한 카타볼리즘이 지배적이다.[28]
인간에게 있어서 이 효소는 간과 신장에서 모두 발현되는데, 이러한 조직에서 이 효소가 차등 조절될 수 있다는 어떤 징후가 있다.[29] PAH는 방향족 아미노산 히드록실라아제 중에서도 특이하며, 반대로 티로신과 트립토판 히드록실라아제는 주로 중추신경계에서 발현되며 신경전달물질/호르몬 생합성에서 촉매율 제한 단계가 나타난다.[13]
질병 관련성
PAH의 돌연변이에 의한 PAH 활성의 결핍은 고페닐알라닌혈증(HPA)을 유발하며, 혈중 페닐알라닌 수치가 정상농도의 20배 이상 증가하면 대사질환 페닐케톤뇨리아(PKU)가 발생한다.[28] PKU는 유전자형과 표현형상 모두 이질적이다. 300개 이상의 구별되는 병원성 변종이 확인되었으며, 대부분은 촉매영역에 매핑되는 오식 변이에 해당한다.[13][20] 확인된 PAH 돌연변이의 코호트가 재조합 시스템으로 표현되었을 때, 효소는 운동 동작의 변화 및/또는 안정성의 저하를 보였으며, 이러한 돌연변이를 효소의 촉매 및 사선화 영역 모두에 대한 구조적 매핑과 일치했다.[13] BH4는4 약리학적 치료로 투여되었으며 유전자형이 일부 잔류 PAH 활동을 유발하지만 BH44 합성이나 재생에 결함이 없는 PKU 환자의 한 부분에 대해 페닐알라닌의 혈중 수치를 감소시키는 것으로 나타났다. 후속 연구는 특정 PAH 돌연변이의 경우, 초과 BH4가4 돌연변이 효소를 안정화시키는 약리학적 보호자 역할을 하며, 테트라머 조립이 중단되고 단백질 분해 및 집산에 대한 민감도가 증가한다는 것을 시사한다.[30] PAH locus에서 확인된 돌연변이는 Penylalanine Hydroxylase Locus Knowledgbase(PAHdb, https://web.archive.org/web/20130718162051/http://www.pahdb.mcgill.ca/))에 기록되어 있다.
페닐케톤뇨증은 돌이킬 수 없는 손상을 일으킬 수 있으므로 페닐알라닌 히드록실라아제의 결함은 개발 초기에 판별하는 것이 필수적이다. 원래 이것은 Guthrie Test라고 알려진 박테리아 억제 검사를 사용하여 수행되었다. 현재, PKU는 많은 나라에서 신생아 검사의 일부분이며, 페닐알라닌 수치가 높아진 것은 출생 직후 탠덤 질량 분광법으로 측정함으로써 확인된다. 개인을 낮은 페닐알라닌, 높은 티로신 식단에 올려놓으면 장기간의 발육 손상을 예방하는 데 도움이 될 수 있다.
관련효소
페닐알라닌 수산화효소는 두 가지 다른 효소와 밀접한 관련이 있다.
- 뇌와 위장관의 세로토닌 수치를 조절하는 트립토판 히드록실라아제(EC 번호 1.14.16.4)
- 뇌와 부신 메둘라의 도파민, 에피네프린, 노르에피네프린 수치를 조절하는 티로신 히드록실라아제(EC 번호 1.14.16.2)이다.
이 세 효소는 동음이의어, 즉 같은 고대 수산화효소로부터 진화한 것으로 생각된다.
참조
- ^ a b c GRCh38: 앙상블 릴리스 89: ENSG000001759 - 앙상블, 2017년 5월
- ^ a b c GRCm38: 앙상블 릴리스 89: ENSMUSG000020051 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ Fitzpatrick PF (1999). "Tetrahydropterin-dependent amino acid hydroxylases". Annual Review of Biochemistry. 68: 355–81. doi:10.1146/annurev.biochem.68.1.355. PMID 10872454.
- ^ Kaufman S (February 1958). "A new cofactor required for the enzymatic conversion of phenylalanine to tyrosine". The Journal of Biological Chemistry. 230 (2): 931–9. doi:10.1016/S0021-9258(18)70516-4. PMID 13525410.
- ^ a b c d e f Fitzpatrick PF (December 2003). "Mechanism of aromatic amino acid hydroxylation". Biochemistry. 42 (48): 14083–91. doi:10.1021/bi035656u. PMC 1635487. PMID 14640675.
- ^ Panay AJ, Lee M, Krebs C, Bollinger JM, Fitzpatrick PF (March 2011). "Evidence for a high-spin Fe(IV) species in the catalytic cycle of a bacterial phenylalanine hydroxylase". Biochemistry. 50 (11): 1928–33. doi:10.1021/bi1019868. PMC 3059337. PMID 21261288.
- ^ Bassan A, Blomberg MR, Siegbahn PE (January 2003). "Mechanism of dioxygen cleavage in tetrahydrobiopterin-dependent amino acid hydroxylases". Chemistry. 9 (1): 106–15. doi:10.1002/chem.200390006. PMID 12506369.
- ^ Olsson E, Martinez A, Teigen K, Jensen VR (March 2011). "Formation of the iron-oxo hydroxylating species in the catalytic cycle of aromatic amino acid hydroxylases". Chemistry. 17 (13): 3746–58. doi:10.1002/chem.201002910. PMID 21351297.
- ^ Bassan A, Blomberg MR, Siegbahn PE (September 2003). "Mechanism of aromatic hydroxylation by an activated FeIV=O core in tetrahydrobiopterin-dependent hydroxylases". Chemistry. 9 (17): 4055–67. doi:10.1002/chem.200304768. PMID 12953191.
- ^ Pavon JA, Fitzpatrick PF (September 2006). "Insights into the catalytic mechanisms of phenylalanine and tryptophan hydroxylase from kinetic isotope effects on aromatic hydroxylation". Biochemistry. 45 (36): 11030–7. doi:10.1021/bi0607554. PMC 1945167. PMID 16953590.
- ^ a b c d e f g h i j Flatmark T, Stevens RC (August 1999). "Structural Insight into the Aromatic Amino Acid Hydroxylases and Their Disease-Related Mutant Forms". Chemical Reviews. 99 (8): 2137–2160. doi:10.1021/cr980450y. PMID 11849022.
- ^ Selwood T, Jaffe EK (March 2012). "Dynamic dissociating homo-oligomers and the control of protein function". Archives of Biochemistry and Biophysics. 519 (2): 131–43. doi:10.1016/j.abb.2011.11.020. PMC 3298769. PMID 22182754.
- ^ a b c Jaffe EK, Stith L, Lawrence SH, Andrake M, Dunbrack RL (February 2013). "A new model for allosteric regulation of phenylalanine hydroxylase: implications for disease and therapeutics". Archives of Biochemistry and Biophysics. 530 (2): 73–82. doi:10.1016/j.abb.2012.12.017. PMC 3580015. PMID 23296088.
- ^ Arturo EC, Gupta K, Héroux A, Stith L, Cross PJ, Parker EJ, Loll PJ, Jaffe EK (March 2016). "First structure of full-length mammalian phenylalanine hydroxylase reveals the architecture of an autoinhibited tetramer". Proceedings of the National Academy of Sciences of the United States of America. 113 (9): 2394–9. doi:10.1073/pnas.1516967113. PMC 4780608. PMID 26884182.
- ^ Jaffe EK (August 2017). "New protein structures provide an updated understanding of phenylketonuria". Molecular Genetics and Metabolism. 121 (4): 289–296. doi:10.1016/j.ymgme.2017.06.005. PMC 5549558. PMID 28645531.
- ^ Patel D, Kopec J, Fitzpatrick F, McCorvie TJ, Yue WW (April 2016). "Structural basis for ligand-dependent dimerization of phenylalanine hydroxylase regulatory domain". Scientific Reports. 6 (1): 23748. doi:10.1038/srep23748. PMC 4822156. PMID 27049649.
- ^ Meisburger SP, Taylor AB, Khan CA, Zhang S, Fitzpatrick PF, Ando N (May 2016). "Domain Movements upon Activation of Phenylalanine Hydroxylase Characterized by Crystallography and Chromatography-Coupled Small-Angle X-ray Scattering". Journal of the American Chemical Society. 138 (20): 6506–16. doi:10.1021/jacs.6b01563. PMC 4896396. PMID 27145334.
- ^ a b Erlandsen H, Fusetti F, Martinez A, Hough E, Flatmark T, Stevens RC (December 1997). "Crystal structure of the catalytic domain of human phenylalanine hydroxylase reveals the structural basis for phenylketonuria". Nature Structural Biology. 4 (12): 995–1000. doi:10.1038/nsb1297-995. PMID 9406548. S2CID 6293946.
- ^ Andersen OA, Flatmark T, Hough E (July 2002). "Crystal structure of the ternary complex of the catalytic domain of human phenylalanine hydroxylase with tetrahydrobiopterin and 3-(2-thienyl)-L-alanine, and its implications for the mechanism of catalysis and substrate activation". Journal of Molecular Biology. 320 (5): 1095–108. doi:10.1016/S0022-2836(02)00560-0. PMID 12126628.
- ^ Teigen K, Frøystein NA, Martínez A (December 1999). "The structural basis of the recognition of phenylalanine and pterin cofactors by phenylalanine hydroxylase: implications for the catalytic mechanism". Journal of Molecular Biology. 294 (3): 807–23. doi:10.1006/jmbi.1999.3288. PMID 10610798.
- ^ a b Li J, Dangott LJ, Fitzpatrick PF (April 2010). "Regulation of phenylalanine hydroxylase: conformational changes upon phenylalanine binding detected by hydrogen/deuterium exchange and mass spectrometry". Biochemistry. 49 (15): 3327–35. doi:10.1021/bi1001294. PMC 2855537. PMID 20307070.
- ^ Li J, Ilangovan U, Daubner SC, Hinck AP, Fitzpatrick PF (January 2011). "Direct evidence for a phenylalanine site in the regulatory domain of phenylalanine hydroxylase". Archives of Biochemistry and Biophysics. 505 (2): 250–5. doi:10.1016/j.abb.2010.10.009. PMC 3019263. PMID 20951114.
- ^ a b c Kobe B, Jennings IG, House CM, Michell BJ, Goodwill KE, Santarsiero BD, Stevens RC, Cotton RG, Kemp BE (May 1999). "Structural basis of autoregulation of phenylalanine hydroxylase". Nature Structural Biology. 6 (5): 442–8. doi:10.1038/8247. PMID 10331871. S2CID 11709986.
- ^ Daubner SC, Hillas PJ, Fitzpatrick PF (December 1997). "Expression and characterization of the catalytic domain of human phenylalanine hydroxylase". Archives of Biochemistry and Biophysics. 348 (2): 295–302. doi:10.1006/abbi.1997.0435. PMID 9434741.
- ^ a b Bjørgo E, de Carvalho RM, Flatmark T (February 2001). "A comparison of kinetic and regulatory properties of the tetrameric and dimeric forms of wild-type and Thr427-->Pro mutant human phenylalanine hydroxylase: contribution of the flexible hinge region Asp425-Gln429 to the tetramerization and cooperative substrate binding". European Journal of Biochemistry. 268 (4): 997–1005. doi:10.1046/j.1432-1327.2001.01958.x. PMID 11179966.
- ^ a b c d Kaufman S (March 1999). "A model of human phenylalanine metabolism in normal subjects and in phenylketonuric patients". Proceedings of the National Academy of Sciences of the United States of America. 96 (6): 3160–4. doi:10.1073/pnas.96.6.3160. PMC 15912. PMID 10077654.
- ^ Lichter-Konecki U, Hipke CM, Konecki DS (August 1999). "Human phenylalanine hydroxylase gene expression in kidney and other nonhepatic tissues". Molecular Genetics and Metabolism. 67 (4): 308–16. doi:10.1006/mgme.1999.2880. PMID 10444341.
- ^ Muntau AC, Gersting SW (December 2010). "Phenylketonuria as a model for protein misfolding diseases and for the development of next generation orphan drugs for patients with inborn errors of metabolism". Journal of Inherited Metabolic Disease. 33 (6): 649–58. doi:10.1007/s10545-010-9185-4. PMID 20824346. S2CID 20843095.
추가 읽기
- Eisensmith RC, Woo SL (1993). "Molecular basis of phenylketonuria and related hyperphenylalaninemias: mutations and polymorphisms in the human phenylalanine hydroxylase gene". Human Mutation. 1 (1): 13–23. doi:10.1002/humu.1380010104. PMID 1301187. S2CID 19476605.
- Konecki DS, Lichter-Konecki U (August 1991). "The phenylketonuria locus: current knowledge about alleles and mutations of the phenylalanine hydroxylase gene in various populations". Human Genetics. 87 (4): 377–88. doi:10.1007/BF00197152. PMID 1679029. S2CID 25627287.
- Cotton RG (1991). "Heterogeneity of phenylketonuria at the clinical, protein and DNA levels". Journal of Inherited Metabolic Disease. 13 (5): 739–50. doi:10.1007/BF01799577. PMID 2246858. S2CID 21931016.
- Erlandsen H, Fusetti F, Martinez A, Hough E, Flatmark T, Stevens RC (December 1997). "Crystal structure of the catalytic domain of human phenylalanine hydroxylase reveals the structural basis for phenylketonuria". Nature Structural Biology. 4 (12): 995–1000. doi:10.1038/nsb1297-995. PMID 9406548. S2CID 6293946.
- Waters PJ, Parniak MA, Nowacki P, Scriver CR (1998). "In vitro expression analysis of mutations in phenylalanine hydroxylase: linking genotype to phenotype and structure to function". Human Mutation. 11 (1): 4–17. doi:10.1002/(SICI)1098-1004(1998)11:1<4::AID-HUMU2>3.0.CO;2-L. PMID 9450897.
- Waters PJ (April 2003). "How PAH gene mutations cause hyper-phenylalaninemia and why mechanism matters: insights from in vitro expression". Human Mutation. 21 (4): 357–69. doi:10.1002/humu.10197. PMID 12655545. S2CID 23769500.
외부 링크
- 페닐알라닌 수산화효소 결핍에 대한 GeneReviews/NCBI/NIH/UW 입력
- 인간 페닐알라닌 수산화효소 유전자 변형의 로커스별 데이터베이스
- 이달의 분자: 페닐알라닌 수산화효소
- PDBe-KB에서 UniProt: P00439(Human Penylalanine hydroxylase)에 대해 PDB에서 사용할 수 있는 모든 구조 정보의 개요.