펩티도글리칸
Peptidoglycan펩티도글리칸 또는 무레인은 당분과 아미노산으로 구성된 중합체로 대부분의 박테리아의 플라즈마 막 밖에 메쉬처럼 생긴 펩티도글리칸 층을 형성하여 세포벽을 형성한다. 설탕 성분은 β-(1,4) 연계 N-아세틸글루코사민(NAG)과 N-아세틸무라믹산(NAM)의 교번 잔류물로 구성된다. N-아세틸무라믹산에는 3-5개의 아미노산으로 구성된 펩타이드 체인이 부착되어 있다. 펩타이드 체인은 3D 망사 같은 층을 형성하는 또 다른 스트랜드의 펩타이드 체인과 교차 연결될 수 있다.[1] 펩티도글리컨은 세포질의 삼투압에 대항할 뿐만 아니라 구조적인 강도를 주는 박테리아 세포벽의 구조적 역할을 한다. 펩티도글리칸은 박테리아 세포 재생 중에 이항 핵분열에도 관여한다.
펩티도글리칸 층은 그램 양성 박테리아(20~80나노미터)가 그램 음성 박테리아(7~8나노미터)보다 실질적으로 두껍다.[2] pH 성장 조건에 따라 펩티도글리컨은 세포벽의 건조중량 그램 양성균의 약 40~90%를 형성하지만 그램 음성 균주의 약 10%만 형성한다. 따라서 높은 수준의 펩티도글리칸의 존재는 그램 양성 박테리아 특성화의 주요 결정 요인이다.[3] 그램 양성 변종에서는 애착 역할과 세로타입 목적으로 중요하다.[4] 그람 양성 박테리아와 그람 음성 박테리아 모두 약 2nm의 입자가 펩티도글리칸을 통과할 수 있다.[5]
구조
박테리아 세포벽의 펩티도글리칸 층은 N-아세틸글루코사민(GlcNAc 또는 NAGA)과 N-아세틸무라민산(MurNAc 또는 NAMA)이라는 두 개의 교차 아미노당의 선형 사슬에서 형성된 결정 격자 구조물이다. 교류당은 β-(1,4)-글리코시드 결합에 의해 연결된다. Each MurNAc is attached to a short (4- to 5-residue) amino acid chain, containing L-alanine, D-glutamic acid, meso-diaminopimelic acid, and D-alanine in the case of Escherichia coli (a Gram-negative bacterium) or L-alanine, D-glutamine, L-lysine, and D-alanine with a 5-glycine interbridge between tetrapeptides in the case of Staphylococcus aureus (그람 양성 박테리아). 펩티도글리칸은 자연에서 D-아미노산의 가장 중요한 공급원 중 하나이다.
서로 다른 선형 아미노당 체인의 아미노산 간 교차연계는 효소 DD-트랜스펙티다아제의 도움으로 발생하며 강인하고 단단한 3차원 구조를 낳는다. 특정한 아미노산 염기서열과 분자구조는 박테리아 종에 따라 다르다.[6]
펩티도글리컨의 구조. NAG = N-아세틸글루코사민(GlcNAc 또는 NAGA라고도 함), NAM = N-아세틸무라민산(MurNAc 또는 NAMA라고도 함)
생합성
펩티도글리칸 모노머는 시토솔에서 합성되어 멤브레인 캐리어 박토프레놀에 부착된다. 박토프레놀은 기존 펩티도글리칸에 삽입된 세포막을 가로질러 펩티도글리칸 모노머를 운반한다.[7]
펩티도글리칸 합성의 첫 단계에서 아미노산인 글루타민은 아미노 그룹을 과당 6인산염에 기증한다. 이것은 과당 6-인산염을 글루코사민-6-인산염으로 바꾼다. 2단계에서 아세틸 그룹은 아세틸 CoA에서 글루코사민-6-인산염의 아미노 그룹으로 전달되어 N-아세틸-글루코사민-6-인산염을 생성한다.[8] 합성 과정의 3단계에서 N-아세틸-글루코사민-6-인산염은 이소머화되며, N-아세틸-글루코사민-6-인산염은 N-아세틸-글루코사민-1-인산염으로 바뀐다.[8]
4단계에서는 현재 모노인산염인 N-아세틸-글루코사민-1-인산염이 UTP를 공격한다.우리딘 3인산염은 피리미딘 뉴클레오티드로서 에너지원으로 작용하는 능력을 가지고 있다. 이 특별한 반응에서, 모노인산염은 UTP를 공격한 후, 무기체 피로인산염이 방출되어 모노인산염으로 대체되어 UDP-N-아세틸글루코사민(2,4)을 생성한다.(UDP를 에너지원으로 사용할 때 무기인산염을 방출한다.) 이 초기 단계는 펩티도글리칸에서 NAG의 전구체를 만드는 데 사용된다.
5단계에서 UDP-N-아세틸글루코사민(UDP-GlcNAc) 중 일부는 글루코사민에 락틸 그룹을 추가하여 UDP-MurNAc(UDP-N-아세틸무라민산)로 변환된다. 또한 이 반응에서 C3 히드록실 그룹은 인산염의 알파 탄소로부터 인산염을 제거할 것이다. 이것은 6단계에서 NADPH에 의해 "락틸 모이티"로 감소될 에놀 파생상품이라고 불리는 것을 만든다.[8]
7단계에서 UDP-MurNAc는 대개 디펩타이드 D-알라닐-D-알라닌을 포함한 5개의 아미노산을 추가하여 UDP-MurNAc 펜타펩타이드로 변환된다.[8] 이러한 각각의 반응은 에너지 원천 ATP를 필요로 한다.[8] 이것은 모두 1단계라고 불린다.
2단계는 세포질 막에서 일어난다. 박토프레놀이라는 지질 운반체가 세포막을 통해 펩티도글리칸 전구체를 운반하는 막 안에 있다. 박토프레놀은 UDP-MurNAc 펜타를 공격해 PP-MurNac 펜타를 만들게 되는데, 이 펜타는 현재 지질인 PP-MurNac 펜타를 만들 것이다. UDP-GlcNAc는 이후 MurNAc로 이송되어 이당류인 Lipid-PP-MurNAc 펜타-GlcNAc를 생성하며 펩티도글리칸의 전구체도 만든다.[8] 이 분자가 어떻게 막을 통해 운반되는지는 아직도 이해되지 않는다. 그러나 일단 그곳에 있게 되면 자라나는 글리칸 체인에 첨가된다.[8] 다음 반응은 트랑글리코실화라고 알려져 있다. 그 반응에서 GlcNAc의 히드록실 그룹은 글리칸의 MurNAc에 부착될 것이며, 이것은 글리칸 체인의 지질-PP를 대체하게 될 것이다. 이것에 책임이 있는 효소는 트랜스글리코실라아제다.[8]
억제
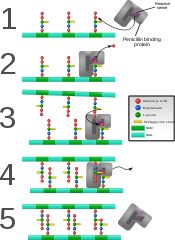
페니실린과 같은 일부 항균제는 페니실린 결합 단백질이나 DD-트랜스펩타이드제라고 알려진 박테리아 효소에 결합하여 펩티도글리칸의 생성을 방해한다.[4] 페니실린 결합 단백질은 펩티도글리칸의 올리고프티드 크로스링크 사이의 결합을 형성한다. 박테리아 세포가 이항 핵분열을 통해 번식하려면 100만 펩티도글리칸 서브유닛(NAM-NAG+올리고펩타이드) 이상이 기존 서브유닛에 부착되어야 한다.[9] 항생제와의 상호작용을 감소시키는 트랜스펩타이드에 대한 유전자 코딩의 돌연변이는 항생제 내성의 중요한 원천이다.[10] 무레이마이신은 인광-N-아세틸무라마모일-펜타페이드 반투명효소(MraY)의 천연기질 UDP-MurNAc-펜타펩타이드(UM5A)의 경쟁력 억제제 역할을 하는 뉴클레오시드 항생제의 하위급이다.[11]
눈물 속에서 발견되며 신체의 선천적인 면역체계의 일부를 구성하는 리소자임은 펩티도글리칸(위 참조)에서 β-(1,4)-글리코시드 결합을 깨뜨려 항균 효과를 발휘한다.
유사성 펩티도글리칸과 유사성
일부 고고학자는 유사 층의 유사 분포를 가지고 있는데, 여기서 설탕 잔류물은 β-(1,3)로 연결된 N-아세틸글루코사민과 N-아세틸탈로사미노루론산이다. 이것은 그러한 고고학의 세포벽을 리소자임에 무감각하게 만든다.[12]
참조
- ^ "Animation of Synthesis of Peptidoglycan Layer".
- ^ Purcell A (18 March 2016). "Bacteria". Basic Biology.
- ^ C. 마이클 호건 2010년 박테리아. 지구 백과사전. 에즈. 시드니 드래건과 C.J.클리블랜드, 워싱턴 DC, 국립과학환경위원회
- ^ a b Salton MR, Kim KS (1996). "Structure". In Baron S, et al. (eds.). Structure. In: Baron's Medical Microbiology (4th ed.). Univ of Texas Medical Branch. ISBN 978-0-9631172-1-2.
- ^ Demchick PH, Koch AL (1 February 1996). "The permeability of the wall fabric of Escherichia coli and Bacillus subtilis". Journal of Bacteriology. 178 (3): 768–73. doi:10.1128/jb.178.3.768-773.1996. PMC 177723. PMID 8550511.
- ^ Ryan KJ, Ray CG, eds. (2004). Sherris Medical Microbiology (4th ed.). McGraw Hill. ISBN 978-0-8385-8529-0.
- ^ "II. THE PROKARYOTIC CELL: BACTERIA". Archived from the original on 26 July 2010. Retrieved 1 May 2011.
- ^ a b c d e f g h White, D. (2007). The physiology and biochemistry of prokaryotes (3rd ed.). NY: Oxford University Press Inc.
- ^ Bauman R (2007). 2nd (ed.). Microbiology with Diseases by Taxonomy. Benjamin Cummings. ISBN 978-0-8053-7679-1.
- ^ Spratt BG (April 1994). "Resistance to antibiotics mediated by target alterations". Science. 264 (5157): 388–93. Bibcode:1994Sci...264..388S. doi:10.1126/science.8153626. PMID 8153626. S2CID 30578841.
- ^ Saeid Malek Zadeh (September 2, 2020). "Theoretical Study of Intermolecular Interactions between Critical Residues of Membrane Protein MraYAA and Promising Antibiotic Muraymycin D2". ACS Omega. 5 (36): 22739–22749. doi:10.1021/acsomega.0c01551. PMC 7495448. PMID 32954121. S2CID 221782183.
- ^ 마디건, M. T, J. M. 마틴코, P. V. 던랩, D. P. 클라크. 미생물의 브록 생물학. 12부. 샌프란시스코, CA: 피어슨/벤자민 커밍스, 2009.