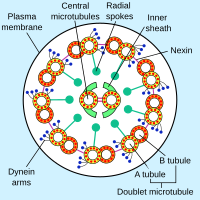
다이닌
Dynein다이닌은 세포 내 미세관을 따라 움직이는 세포골격 운동 단백질의 한 종류이다.그들은 ATP에 저장된 화학 에너지를 기계적인 작업으로 변환합니다.다이닌은 다양한 세포 화물을 운반하고, 유사분열에서 중요한 힘과 변위를 제공하며, 진핵 섬모 및 편모 박동을 촉진합니다.이러한 모든 기능은 역행 수송으로 알려진 마이크로튜브의 마이너스 끝을 향해 이동하는 다이네인의 능력에 의존합니다. 그래서 그것들은 "마이너스 엔드 지향 모터"라고 불립니다.반대로, 대부분의 키네신 운동 단백질은 전대 이동이라고 불리는 미세관의 플러스 엔드로 이동한다.
분류
| 다인 중쇄, N 말단 영역 1 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호. | DHC_N1 | ||||||||
| 팜 | PF08385 | ||||||||
| 인터프로 | IPR013594 | ||||||||
| |||||||||
| 다인 중쇄, N 말단 영역 2 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호. | DHC_N2 | ||||||||
| 팜 | PF08393 | ||||||||
| 인터프로 | IPR013602 | ||||||||
| |||||||||
| 다이네인 중쇄 및 다이네인 모터의 영역 D6 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호. | 다인_헤비 | ||||||||
| 팜 | PF03028 | ||||||||
| 인터프로 | IPR004273 | ||||||||
| |||||||||
| 다인광중간쇄(DLIC) | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||||
| 기호. | DLIC | ||||||||||
| 팜 | PF05783 | ||||||||||
| 빠맘 클랜 | CL0023 | ||||||||||
| |||||||||||
| 다이네인 경쇄 타입 1 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 결합된 펩타이드를 가진 인간 핀/lc8 이합체 구조 | |||||||||
| 식별자 | |||||||||
| 기호. | 다인_라이트 | ||||||||
| 팜 | PF01221 | ||||||||
| 인터프로 | IPR001372 | ||||||||
| 프로 사이트 | PDOC00953 | ||||||||
| SCOP2 | 1 bkq / SCOPe / SUPFAM | ||||||||
| |||||||||
| 바리케이드 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
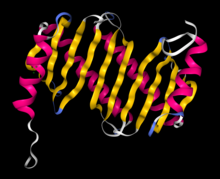 로드블록/LC7 단백질 구조 - RCSB PDB 1y4o | |||||||||
| 식별자 | |||||||||
| 기호. | Robl1, Robl2 | ||||||||
| 팜 | PF03259 | ||||||||
| 인터프로 | IPR016561 | ||||||||
| SCOP2 | 1y4o/SCOPe/SUPFAM | ||||||||
| |||||||||
다이네인은 세포질 다이네인과 축삭 다이네인의 두 그룹으로 나눌 수 있는데, 이들은 섬모 또는 편모 다이네인으로도 불린다.
- 축삭의
- 세포질의
기능.
액소네말 다이네인은 섬모와 편모의 축삭에서 미세관의 미끄러짐을 유발하며 이러한 구조를 가진 세포에서만 발견됩니다.
모든 동물 세포와 아마도 식물 세포에서도 발견되는 세포질 다이네인은 세포 세포 전달 및 중심체 조립과 같은 세포 생존에 필요한 기능을 수행합니다.[1]세포질 다이네인은 마이크로튜브를 따라 순차적으로 움직인다. 즉, 그 줄기 중 하나 또는 다른 줄기들은 항상 마이크로튜브에 부착되어 다이네인이 분리되지 않고 마이크로튜브를 따라 상당한 거리를 걸을 수 있다.
세포질 다이네인은 [1]골지 복합체와 세포 내 다른 기관들을 배치하는데 도움을 준다.또한 소포체, 엔도솜 및 리소솜에 의해 만들어진 소포와 같은 세포 기능에 필요한 화물을 운반하는 데 도움을 준다(Karp, 2005).다이네인은 염색체의 이동과 세포 [2][3]분열을 위한 유사분열 방추체 배치에 관여한다.다이나인은 역행 [1]축삭수송이라고 불리는 과정에서 뉴런의 축삭을 따라 세포체를 향해 소기관, 소포, 그리고 아마도 미세관 조각들을 운반한다.또한 다이네인 모터는 수상돌기에서 [4]역행하는 분해성 엔도좀의 수송에도 관여한다.
유사분열 스핀들 위치 결정
세포질 다이네인은 세포피질에 정착하여 중심체에서 나오는 아스트랄 미세관을 끌어당김으로써 방추체를 세포피질 부위에 위치시킨다.MIT 의대생인 키요미츠 토모미는 유사분열 중기 동안 다이네인이 세포 중앙의 염색체를 정렬하는 운동단백질 역할을 하는 것을 발견했다.다인인은 미세관과 염색체를 세포의 한쪽 끝으로 끌어당긴다.미세관의 끝이 세포막에 가까워지면, 그들은 다이네인을 세포의 다른 쪽에 보내는 화학 신호를 방출한다.이것은 반복하여 염색체가 세포 중앙에 오게 하는데,[5][6][7][8] 이것은 유사분열에서 필요하다.싹트기 효모는 이 과정을 연구하기 위한 강력한 모델 유기체였고 다이닌이 아스트랄 미세관의 끝과 끝부분을 목표로 하고 오프로딩 [9][10]메커니즘을 통해 세포 피질에 전달된다는 것을 보여주었다.
바이러스 리플리케이션
다이나인과 키네신은 둘 다 바이러스 복제 과정을 중재하기 위해 바이러스에 의해 이용될 수 있다.많은 바이러스는 침입 후 세포막을 [11]숙주하는 세포 내 복제 부위로 핵산/단백질 코어를 운반하기 위해 미세관 수송 시스템을 사용합니다.바이러스의 운동 특이적 결합 부위에 대해서는 많이 알려져 있지 않지만, 일부 바이러스는 제거되었을 때 다이낙틴 결합, 축삭수송(배양 중), 생체 [12]내 신경 침해를 감소시키는 프롤린이 풍부한 배열(바이러스 간 분화)을 포함하고 있는 것으로 알려져 있다.이는 프롤린이 풍부한 배열이 다이네인을 공선택하는 주요 결합 부위일 수 있음을 시사한다.
구조.

다이네인 모터의 각 분자는 다수의 작은 폴리펩타이드 서브유닛으로 이루어진 복합단백질 집합체이다.세포질 및 액소네말 다이네인은 동일한 구성 요소 중 일부를 포함하지만, 그들은 또한 몇 가지 고유한 서브 유닛을 포함합니다.
세포질 다이네인
약 1.5메가달톤(MDA)의 분자량을 가진 세포질 다이네인은 약 12개의 폴리펩타이드 서브유닛을 포함하는 이합체이다. 두 개의 동일한 "중쇄", 520 kDa 질량이 ATPase 활성을 포함하고 따라서 마이크로튜브를 따라 이동하는 역할을 한다; 두 개의 중간 사슬인 74 kDa.다이네인을 화물에 고정하기 위해 두 개의 53-59 kDa 경량 중간 체인 및 여러 개의 경량 체인.
각 다이닌 중쇄의 힘 생성 ATP 효소 활성은 다른 AAA 단백질과 관련된 큰 도넛 모양의 "머리"에 위치하고, 머리에서 나온 두 개의 돌기는 다른 세포질 구조와 연결된다.하나의 돌기인 코일 줄기는 분리 및 재접착의 반복 사이클을 통해 미소관의 표면에 결합하고 "걸어" 갑니다.또 다른 돌출물인 확장된 꼬리는 다이닌을 화물에 부착하는 경량 중간, 중간 및 경량 체인 서브유닛에 결합됩니다.완전한 세포질 다이네인 모터에서 쌍으로 구성된 중쇄의 교대로의 활동은 단일 다이네인 분자가 완전히 분리되지 않고 마이크로튜브를 따라 상당한 거리를 "걸어서" 화물을 운반할 수 있게 합니다.
다이네인의 아포상태에서 모터는 뉴클레오티드가 없고 AAA 도메인 링은 개방배치로 [14]존재하며 MTBD는 고친화성 [15]상태로 존재한다.AAA 도메인에 대한 많은 정보는 [16]아직 알려지지 않았지만, AAA1은 [17]다이닌에서 ATP 가수분해의 1차 부위로 잘 확립되어 있다.ATP는 AAA1에 바인드하면 AAA 도메인링의 '닫힘' 설정으로의 컨포메이션 변경, 버트레스 [14]이동 및 링커 [18][19]컨포메이션 변경을 시작합니다.링커는 구부러져 [14][19]AAA1에 바인드된 채로 AAA5에서AAA2로 이행합니다.줄기에서 부착된 하나의 알파나선은 버트리스에 의해 당겨지고, 나선은 코일코일 파트너에 [15][20]대해 반 헵타드 반복으로 미끄러져 [14]줄기가 꼬인다.그 결과 다이네인의 MTBD는 저친화성 상태가 되어 모터가 새로운 결합 [21][22]부위로 이동할 수 있게 된다.ATP의 가수 분해에 이어 줄기가 회전하여 [18]MT를 따라 다이닌을 더 이동합니다. 인산염의 방출에 따라 MTBD는 고친화성 상태로 돌아가 MT를 재결합하여 파워 [23]스트로크를 트리거합니다.링커는 직선 구성으로 돌아와 AAA2에서[24][25] AAA5로 돌아와 레버 [26]액션을 생성하여 파워[18] 스트로크에 의해 달성되는 다이네인의 최대 치환을 생성합니다.사이클은 ADP의 릴리스로 종료되어 AAA 도메인링이 "오픈"[22] 설정으로 돌아갑니다.
효모 다이네인은 분리 없이 미세관을 따라 걸을 수 있지만, 메타조안에서는 세포질 다이네인은 유사분열에서 필수적인 또 다른 멀티 서브 유닛 단백질인 다이낙틴과 화물 [27]어댑터의 결합에 의해 활성화되어야 한다.다이네인, 다이낙틴 및 카고 어댑터를 포함하는 이 3가지 복합체는 매우 가공적이며 카고의 세포 내 목적지에 도달하기 위해 분리 없이 장거리 도보 이동이 가능합니다.지금까지 확인된 화물 어댑터에는 BicD2, Hook3, FIP3 [27]및 Spindsly가 포함됩니다.Ras 슈퍼 패밀리의 일원인 광중간 체인은 다이네인 [28]모터에 여러 개의 화물 어댑터를 부착하는 역할을 합니다.다른 꼬리 서브유닛도 다이네인-다이낙틴-BicD2의 [29]저분해능 구조에서 증명된 바와 같이 이러한 상호작용을 촉진하는 데 도움을 줄 수 있다.
다이네인에 대한 세포 내 운동 조절의 주요 형태 중 하나는 다이낙틴이다.그것은 거의 모든 세포질 다이네인 [30]기능에 필요할 수 있다.현재 가장 잘 연구된 다이닌 파트너입니다.다이낙틴은 세포질 다이네인과 결합함으로써 세포 내 이동을 돕는 단백질이다.다이낙틴은 다른 단백질이 결합하는 발판 역할을 할 수 있다.또한 다이네인을 [31][32]위치시켜야 하는 위치에 위치시키는 모집 인자로도 기능합니다.또한 그것이 [33]키네신-2를 조절할 수 있다는 몇 가지 증거도 있다.다이낙틴 복합체는 20개 이상의 서브유닛으로 [29]구성되며, 그 중 p150(글루드)이 가장 [34]크다.다이낙틴 자체가 모터의 속도에 영향을 미친다는 확실한 증거는 없다.그러나 모터의 [35]처리능력에 영향을 미칩니다.결합 조절은 알로스테릭할 가능성이 높다. 실험 결과 다이네인 모터의 처리능력에 제공되는 강화는 마이크로튜브에 [36]대한 p150 서브유닛 결합 도메인에 의존하지 않는 것으로 나타났다.
액소네말다이닌
액소네말 다이닌은 하나, 두 개 또는 세 개의 동일하지 않은 중쇄를 포함하는 여러 가지 형태로 나타납니다(실륨의 유기체와 위치에 따라 다릅니다.각 중쇄는 다른 AAA 단백질과 유사한 것으로 여겨지는 도넛 모양의 구조를 가진 구상 운동 도메인, 미소관에 결합하는 코일형 코일 "스토크" 및 동일한 축삭의 인접한 미소관에 결합하는 확장된 꼬리(또는 "줄기")를 가진다.따라서 각 다이네인 분자는 섬모축소체의 인접한 2개의 미소관 사이에 가교를 형성한다.움직임을 일으키는 "파워 스트로크" 동안 AAA ATPase 모터 도메인은 미세관 결합 줄기가 화물 결합 꼬리에 대해 회전하게 하는 구조 변화를 겪으며, 그 결과 한 미세관이 다른 미세관에 대해 미끄러진다(Karp, 2005).이 슬라이딩은 섬모가 세포나 다른 입자를 두드리고 밀어내는 데 필요한 굽힘 운동을 생성합니다.반대 방향으로의 움직임을 담당하는 다이네인 분자 그룹은 섬모 또는 편모가 앞뒤로 움직일 수 있도록 아마도 조정된 방식으로 활성화되고 비활성화될 것입니다.레이디얼 스포크는 이 움직임을 동기화하는(또는 그 중 하나) 구조로 제안되었습니다.
편모 박동 주파수 및 섬모 파형에 있어 액소네말 다인 활성 조절이 매우 중요합니다.액소네말 다인 조절의 모드에는 인산화, 산화환원 및 칼슘이 포함됩니다.축삭에 가해지는 기계적 힘 또한 축삭 다인 기능에 영향을 미친다.액소네말 다이네인의 내측 팔과 외측 팔의 중쇄는 인산화/탈인산화되어 미세관의 미끄러짐 속도를 조절한다.다른 축삭 다이네인 팔과 관련된 티오레독신은 산화/환원되어 축삭 내에서 다이네인이 결합하는 위치를 조절한다.외부 축삭 다이나인 암의 중심점과 성분은 칼슘 농도의 변동을 감지합니다.칼슘 변동은 섬모 파형과 편모 박동 주파수를 변경하는 데 중요한 역할을 한다(King, 2012).[37]
역사
섬모와 편모의 움직임을 담당하는 단백질은 1963년에 처음 발견되었고 다이네인으로 명명되었다(Karp, 2005년.20년 후 편모 다이네인의 발견 이후 존재한다고 의심되었던 세포질 다이네인이 분리 및 확인되었다(Karp, 2005).
감수 분열 중 염색체 분리
세포의 반대 극에 대한 상동 염색체의 분리는 감수 분열의 첫 번째 분열 동안 일어난다.적절한 분리는 정상적인 염색체 보체를 가진 반수체 감수생성 제품을 생산하기 위해 필수적이다.키아스마타(크로스오버 재조합 이벤트)의 형성은 일반적으로 적절한 분리를 촉진하는 것으로 보인다.단, 키아스마타가 없을 때 다이네인이 [38]분리를 촉진하는 핵분열 효모인 시조당류 폼베.다이네인의 운동 서브 유닛인 Dhc1은 키아스마타의 [38]유무 모두에서 염색체 분리에 필요하다.다이네인 경쇄 Dlc1 단백질도 분리, 특히 키아스마타가 없을 때 필요합니다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b c Karp G, Beginnen K, Vogel S, Kuhlmann-Krieg S (2005). Molekulare Zellbiologie (in French). Springer. ISBN 978-3-540-23857-7.
- ^ Samora CP, Mogessie B, Conway L, Ross JL, Straube A, McAinsh AD (August 2011). "MAP4 and CLASP1 operate as a safety mechanism to maintain a stable spindle position in mitosis". Nature Cell Biology. 13 (9): 1040–50. doi:10.1038/ncb2297. PMID 21822276. S2CID 8869880.
- ^ Kiyomitsu T, Cheeseman IM (February 2012). "Chromosome- and spindle-pole-derived signals generate an intrinsic code for spindle position and orientation". Nature Cell Biology. 14 (3): 311–7. doi:10.1038/ncb2440. PMC 3290711. PMID 22327364.
- ^ Yap CC, Digilio L, McMahon LP, Wang T, Winckler B (April 2022). "Dynein is required for Rab7-dependent endosome maturation, retrograde dendritic transport, and degradation". The Journal of Neuroscience. doi:10.1523/JNEUROSCI.2530-21.2022. PMID 35474277.
- ^ https://www.researchgate.net/publication/325479623_Dynein-Dynactin-NuMA_clusters_generate_cortical_spindle-pulling_forces_as_a_multi-arm_ensemble doi: 10.7554/eLife.36559
- ^ Eshel D, Urrestarazu LA, Vissers S, Jauniaux JC, van Vliet-Reedijk JC, Planta RJ, Gibbons IR (December 1993). "Cytoplasmic dynein is required for normal nuclear segregation in yeast". Proceedings of the National Academy of Sciences of the United States of America. 90 (23): 11172–6. Bibcode:1993PNAS...9011172E. doi:10.1073/pnas.90.23.11172. PMC 47944. PMID 8248224.
- ^ Li YY, Yeh E, Hays T, Bloom K (November 1993). "Disruption of mitotic spindle orientation in a yeast dynein mutant". Proceedings of the National Academy of Sciences of the United States of America. 90 (21): 10096–100. Bibcode:1993PNAS...9010096L. doi:10.1073/pnas.90.21.10096. PMC 47720. PMID 8234262.
- ^ Carminati JL, Stearns T (August 1997). "Microtubules orient the mitotic spindle in yeast through dynein-dependent interactions with the cell cortex". The Journal of Cell Biology. 138 (3): 629–41. doi:10.1083/jcb.138.3.629. PMC 2141630. PMID 9245791.
- ^ Lee WL, Oberle JR, Cooper JA (February 2003). "The role of the lissencephaly protein Pac1 during nuclear migration in budding yeast". The Journal of Cell Biology. 160 (3): 355–64. doi:10.1083/jcb.200209022. PMC 2172672. PMID 12566428.
- ^ Lee WL, Kaiser MA, Cooper JA (January 2005). "The offloading model for dynein function: differential function of motor subunits". The Journal of Cell Biology. 168 (2): 201–7. doi:10.1083/jcb.200407036. PMC 2171595. PMID 15642746.
- ^ Valle-Tenney R, Opazo T, Cancino J, Goff SP, Arriagada G (August 2016). "Dynein Regulators Are Important for Ecotropic Murine Leukemia Virus Infection". Journal of Virology. 90 (15): 6896–6905. doi:10.1128/JVI.00863-16. PMC 4944281. PMID 27194765.
- ^ Zaichick SV, Bohannon KP, Hughes A, Sollars PJ, Pickard GE, Smith GA (February 2013). "The herpesvirus VP1/2 protein is an effector of dynein-mediated capsid transport and neuroinvasion". Cell Host & Microbe. 13 (2): 193–203. doi:10.1016/j.chom.2013.01.009. PMC 3808164. PMID 23414759.
- ^ PDB: 4RH7; Carter AP (February 2013). "Crystal clear insights into how the dynein motor moves". Journal of Cell Science. 126 (Pt 3): 705–13. doi:10.1242/jcs.120725. PMID 23525020.
- ^ a b c d Schmidt H, Zalyte R, Urnavicius L, Carter AP (February 2015). "Structure of human cytoplasmic dynein-2 primed for its power stroke". Nature. 518 (7539): 435–438. Bibcode:2015Natur.518..435S. doi:10.1038/nature14023. PMC 4336856. PMID 25470043.
- ^ a b Carter AP, Vale RD (February 2010). "Communication between the AAA+ ring and microtubule-binding domain of dynein". Biochemistry and Cell Biology. 88 (1): 15–21. doi:10.1139/o09-127. PMC 2894566. PMID 20130675.
- ^ Kardon JR, Vale RD (December 2009). "Regulators of the cytoplasmic dynein motor". Nature Reviews. Molecular Cell Biology. 10 (12): 854–65. doi:10.1038/nrm2804. PMC 3394690. PMID 19935668.
- ^ PDB: 1HN5; Mocz G, Gibbons IR (February 2001). "Model for the motor component of dynein heavy chain based on homology to the AAA family of oligomeric ATPases". Structure. London, England. 9 (2): 93–103. doi:10.1016/S0969-2126(00)00557-8. PMID 11250194.
- ^ a b c Roberts AJ, Numata N, Walker ML, Kato YS, Malkova B, Kon T, Ohkura R, Arisaka F, Knight PJ, Sutoh K, Burgess SA (February 2009). "AAA+ Ring and linker swing mechanism in the dynein motor". Cell. 136 (3): 485–95. doi:10.1016/j.cell.2008.11.049. PMC 2706395. PMID 19203583.
- ^ a b Roberts AJ, Malkova B, Walker ML, Sakakibara H, Numata N, Kon T, Ohkura R, Edwards TA, Knight PJ, Sutoh K, Oiwa K, Burgess SA (October 2012). "ATP-driven remodeling of the linker domain in the dynein motor". Structure. 20 (10): 1670–80. doi:10.1016/j.str.2012.07.003. PMC 3469822. PMID 22863569.
- ^ Kon T, Imamula K, Roberts AJ, Ohkura R, Knight PJ, Gibbons IR, Burgess SA, Sutoh K (March 2009). "Helix sliding in the stalk coiled coil of dynein couples ATPase and microtubule binding". Nature Structural & Molecular Biology. 16 (3): 325–33. doi:10.1038/nsmb.1555. PMC 2757048. PMID 19198589.
- ^ Carter AP (February 2013). "Crystal clear insights into how the dynein motor moves". Journal of Cell Science. 126 (Pt 3): 705–13. doi:10.1242/jcs.120725. PMID 23525020.
- ^ a b Bhabha G, Cheng HC, Zhang N, Moeller A, Liao M, Speir JA, Cheng Y, Vale RD (November 2014). "Allosteric communication in the dynein motor domain". Cell. 159 (4): 857–68. doi:10.1016/j.cell.2014.10.018. PMC 4269335. PMID 25417161.
- ^ Bhabha G, Johnson GT, Schroeder CM, Vale RD (January 2016). "How Dynein Moves Along Microtubules". Trends in Biochemical Sciences. 41 (1): 94–105. doi:10.1016/j.tibs.2015.11.004. PMC 4706479. PMID 26678005.
- ^ Gennerich A, Carter AP, Reck-Peterson SL, Vale RD (November 2007). "Force-induced bidirectional stepping of cytoplasmic dynein". Cell. 131 (5): 952–65. doi:10.1016/j.cell.2007.10.016. PMC 2851641. PMID 18045537.
- ^ Burgess SA, Knight PJ (April 2004). "Is the dynein motor a winch?". Current Opinion in Structural Biology. 14 (2): 138–46. doi:10.1016/j.sbi.2004.03.013. PMID 15093827.
- ^ Reck-Peterson SL, Yildiz A, Carter AP, Gennerich A, Zhang N, Vale RD (July 2006). "Single-molecule analysis of dynein processivity and stepping behavior". Cell. 126 (2): 335–48. doi:10.1016/j.cell.2006.05.046. PMC 2851639. PMID 16873064.
- ^ a b McKenney RJ, Huynh W, Tanenbaum ME, Bhabha G, Vale RD (July 2014). "Activation of cytoplasmic dynein motility by dynactin-cargo adapter complexes". Science. 345 (6194): 337–41. Bibcode:2014Sci...345..337M. doi:10.1126/science.1254198. PMC 4224444. PMID 25035494.
- ^ Schroeder CM, Ostrem JM, Hertz NT, Vale RD (October 2014). "A Ras-like domain in the light intermediate chain bridges the dynein motor to a cargo-binding region". eLife. 3: e03351. doi:10.7554/eLife.03351. PMC 4359372. PMID 25272277.
- ^ a b Urnavicius L, Zhang K, Diamant AG, Motz C, Schlager MA, Yu M, Patel NA, Robinson CV, Carter AP (March 2015). "The structure of the dynactin complex and its interaction with dynein". Science. 347 (6229): 1441–1446. Bibcode:2015Sci...347.1441U. doi:10.1126/science.aaa4080. PMC 4413427. PMID 25814576.
- ^ Karki S, Holzbaur EL (February 1999). "Cytoplasmic dynein and dynactin in cell division and intracellular transport". Current Opinion in Cell Biology. 11 (1): 45–53. doi:10.1016/S0955-0674(99)80006-4. PMID 10047518.
- ^ Moughamian AJ, Osborn GE, Lazarus JE, Maday S, Holzbaur EL (August 2013). "Ordered recruitment of dynactin to the microtubule plus-end is required for efficient initiation of retrograde axonal transport". The Journal of Neuroscience. 33 (32): 13190–203. doi:10.1523/JNEUROSCI.0935-13.2013. PMC 3735891. PMID 23926272.
- ^ Moughamian AJ, Holzbaur EL (April 2012). "Dynactin is required for transport initiation from the distal axon". Neuron. 74 (2): 331–43. doi:10.1016/j.neuron.2012.02.025. PMC 3347924. PMID 22542186.
- ^ Berezuk MA, Schroer TA (February 2007). "Dynactin enhances the processivity of kinesin-2". Traffic. 8 (2): 124–9. doi:10.1111/j.1600-0854.2006.00517.x. PMID 17181772. S2CID 46446471.
- ^ Schroer TA (8 October 2004). "Dynactin". Annual Review of Cell and Developmental Biology. 20: 759–79. doi:10.1146/annurev.cellbio.20.012103.094623. PMID 15473859.
- ^ King SJ, Schroer TA (January 2000). "Dynactin increases the processivity of the cytoplasmic dynein motor". Nature Cell Biology. 2 (1): 20–4. doi:10.1038/71338. PMID 10620802. S2CID 20349195.
- ^ Kardon JR, Reck-Peterson SL, Vale RD (April 2009). "Regulation of the processivity and intracellular localization of Saccharomyces cerevisiae dynein by dynactin". Proceedings of the National Academy of Sciences of the United States of America. 106 (14): 5669–74. Bibcode:2009PNAS..106.5669K. doi:10.1073/pnas.0900976106. PMC 2657088. PMID 19293377.
- ^ King SM (August 2012). "Integrated control of axonemal dynein AAA(+) motors". Journal of Structural Biology. 179 (2): 222–8. doi:10.1016/j.jsb.2012.02.013. PMC 3378790. PMID 22406539.
- ^ a b Davis L, Smith GR (June 2005). "Dynein promotes achiasmate segregation in Schizosaccharomyces pombe". Genetics. 170 (2): 581–90. doi:10.1534/genetics.104.040253. PMC 1450395. PMID 15802518.
추가 정보
- Karp G (2005). Cell and Molecular Biology: Concepts and Experiments (4th ed.). Hoboken, NJ: John Wiley and Sons. pp. 346–358. ISBN 978-0-471-19279-4.
- Schroer TA (2004). "Dynactin". Annual Review of Cell and Developmental Biology. 20: 759–79. doi:10.1146/annurev.cellbio.20.012103.094623. PMID 15473859.
외부 링크
- 진핵생물 선형 모티브 자원 모티브 클래스 LIG_Dynein_DLC8_1
- 론 베일의 세미나: "분자 운동 단백질"
- 미국 국립 의학 도서관 의학 주제 제목(MeSH)의 Dynein
- EC 3.6.4.2