다중복사 단일사슬 DNA
Multicopy single-stranded DNA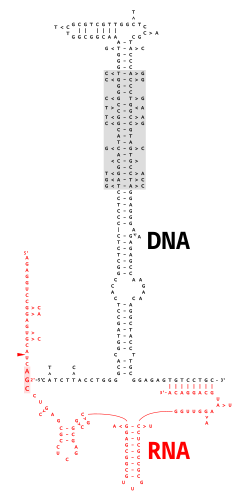
multicopy 홑가닥 DNA(msDNA)는 RNA 분자의 내부 구아노신에 2'-5' 포스포디에스터 결합을 통해 공유 결합되는 단가닥 DNA 분자로 구성된 염색체 외 위성 DNA의 일종이다.생성된 DNA/RNA 키메라에는 RNA 스플라이싱 중간체에서 발견되는 가지와 유사한 가지에 의해 결합된 두 개의 줄기 루프가 있습니다."retron"이라고 불리는 msDNA의 부호화 영역은 msDNA [2]합성에 필수적인 역전사효소의 한 유형을 부호화한다.
검출
myxobacteria에서 [3][4]msDNA가 발견되기 전에, 흙에 사는 박테리아 집단인 myxobacteria에서 역전사효소(RT)로 알려진 효소는 진핵 생물과 바이러스에만 존재한다고 생각되었다.그 발견은 그 지역에 대한 연구 증가로 이어졌다.그 결과 msDNA는 다양한 종류의 대장균과 병원성 [5]박테리아를 포함한 세균에 널리 분포하는 것으로 밝혀졌다.추가 연구는 HIV 인코딩 역전사 효소와 msDNA 코딩 영역에서 발견된 개방형 판독 프레임(ORF) 사이의 유사성을 발견했다.테스트 결과 레트론을 함유한 [6]균주의 조용액에서 역전사효소 활성의 존재가 확인되었다.RNase H 도메인은 레트론 ORF에서 잠정적으로 식별되었지만, 나중에 msDNA 합성에 필요한 RNase H 활성은 실제로 [7]숙주에 의해 공급된다는 것이 밝혀졌다.
리트론
msDNA의 발견은 역전사효소를 코드하는 유전자가 원핵생물, 진핵생물, 바이러스 그리고 심지어 고세균에서 발견되었기 때문에 역전사효소가 어디에서 유래했는지에 대한 광범위한 의문을 가져왔다.대장균에서 msDNA 생성을 코드하는 DNA 단편이 [8]발견된 후, 박테리오파지가 대장균에 [9]RT 유전자를 도입하는 데 책임이 있었을 것으로 추측되었다.이러한 발견은 역전사효소가 박테리아로부터의 바이러스 진화에 역할을 했다는 것을 암시하며, 한 가설은 역전사효소의 도움으로 바이러스가 단백질 외피를 획득하는 분리된 msDNA 유전자로 발생했을 수 있다고 말한다.거의 모든 RT 유전자가 레트로바이러스 복제 및/또는 트랜스포저블 요소의 이동에 기능하기 때문에, 리트론이 이동 가능한 유전 요소일 수 있다고 상상하는 것은 타당하지만, msDNA가 아직 박테리아 종들 사이에 산발적으로 분산되어 있다는 관찰된 사실 외에는 그러한 가설을 뒷받침하는 증거는 거의 없었다.수평 전달과 [5][10][11]수직 전달을 모두 암시하는 방식으로.retron 시퀀스 자체가 모바일 요소를 나타내는지는 알려지지 않았기 때문에 retron은 다른 가능한 활동에 대한 추측을 의도적으로 피하면서 msDNA를 생성하는 능력에 의해 기능적으로 정의된다.
기능.
msDNA의 기능은 세포 내에 많은 복사본이 존재하지만 알려지지 않았다.msDNA를 발현하지 않는 녹아웃 돌연변이는 생존 가능하기 때문에 msDNA의 생산은 실험실 조건에서 생명에 필수적이지 않다.msDNA의 과발현은 돌연변이 유발성이며,[10] 이는 전형적인 구조의 불일치 염기쌍에 의해 복구 단백질이 적정화된 결과이다.msDNA는 병원성 또는 스트레스 [12]상태에 적응하는 데 어느 정도 역할을 할 수 있다고 제안되었습니다.Myxoccus xanthus, Stigmatella aurantiaca [1]및 다른 많은[5][12] 박테리아로부터의 msDNA의 배열 비교는 모든 인지 [13]분자에서 발견되는 보존되고 초가변적인 염기서열을 연상시키는 보존되고 초가변적인 도메인을 보여준다.예를 들어, M. xanthus와 S. aurantiaca의 주요 msDNA는 42%[1]의 염기쌍 상동성을 공유하는 19개의 염기쌍 도메인 내에서 제외하고 94%의 염기쌍 상동성을 공유한다.이러한 영역의 존재는 myxobacteria가 무리 지어 결실체 형성을 포함한 복잡한 협력적 사회적 행동을 보이는 반면, 대장균과 다른 병원성 박테리아는 강화된 항생제 및 세제 내성을 보이는 바이오필름을 형성하기 때문에 중요하다.에너지의 상당한 개인 투자가 필요한 사회적 집합의 지속가능성은 일반적으로 그룹이 자기와 [14]비자아를 구별할 수 있도록 하는 모든 인식 메커니즘의 진화에 달려 있다.
생합성

msDNA의 생합성은 DNA/RNA 생화학에서 다른 곳에서 찾을 수 없는 독특한 경로를 따르는 것으로 알려져 있다.RNA 스플라이싱 중간체에서 발견된 분기 접합부와 2'-5' 분기 접합부의 유사성으로 인해 처음에는 스플라이시오솜 또는 리보자임 매개 결찰에 의한 분기 형성이 예상되었을 수 있다.그러나 놀랍게도 정제된 레트론 역전사효소를 사용한 무세포 시스템에서의 실험은 프라이머 [15]RNA의 특정 내부 G 잔기의 2'-OH 그룹에서 cDNA 합성이 직접 프라이밍된다는 것을 보여준다.RT는 전구체 RNA에서 특정 줄기-루프 구조를 인식하여 자체 레트론에 [16]매우 특이적인 RT에 의한 msDNA 합성을 생성한다.msDNA 합성의 프라이밍은 DNA 합성에 대한 우리의 이해에 매력적인 도전을 제공한다.DNA 중합효소(RT 포함)는 고도로 보존된 구조적 특징을 공유하며, 이는 활성 촉매 부위가 DNA를 템플릿으로 사용하는 DNA 중합효소 간에 거의 차이가 없다는 것을 의미한다.진핵 역 역전사 효소의 촉매 지역 3도메인은 오른손 그립의 3'-OH는 입문서는 여러분과 극은 산성 잔류물들 한 무리의 손바닥 betw에 위치한 polymerase,[17]의 활성화 부위에 묻혀함께 2중 가닥 primer-template를 여는"손가락","야자","엄지"이라고 부른 것으로 구성되어 있다.een집게손가락과 가운데손가락이 뭘까요?진핵생물 RT에서, RNase H 도메인은 엄지손가락 밑의 손목에 있지만, 레트론 RT는 RNase H 활성이 부족하다.중합효소 활성 부위에서 RNase H 활성 부위로 확장되는 핵산 결합 균열은 진핵생물 RT에서 길이가 약 60Ω으로, 거의 두 번의 나선 [18]회전에 해당한다.진핵생물 RT가 기존의 프라이머를 확장하면 성장 중인 DNA/RNA는 구분을 따라 이중나선나선을 이루며 이중나선이 RNase H 도메인을 통과함에 따라 템플릿 RNA가 소화되어 cDNA의 초기 가닥을 방출한다.그러나 msDNA 프라이머 확장의 경우 프라이밍 G의 3'-OH에 긴 RNA 가닥이 부착되어 있다.로 DNA합성이 진행되는 비록 그것은 2'-OH reaction,[16]는 DNA가닥의 추가 연장 문제가 제공하는 점화를 이용할 수 있도록 할 수 있는 RT-primer 템플릿 만들기 또는 콤플렉스에 대한 모델:가능하다,의 부피 RNA가닥은 3'-OH을 어떻게든 입체 장애로 인해 막혀 없이 바인딩 패인 흔적이 있는 나선형으로 급격히 떨어지기가 필요하다..이 문제를 해결하려면 msDNA 역전사효소에는 다른 [10]RT가 공유하지 않는 특수 기능이 필요합니다.
레퍼런스
- ^ a b c Dhundale A, Lampson B, Furuichi T, Inouye M, Inouye S (December 1987). "Structure of msDNA from Myxococcus xanthus: evidence for a long, self-annealing RNA precursor for the covalently linked, branched RNA". Cell. 51 (6): 1105–12. doi:10.1016/0092-8674(87)90596-4. PMID 2446773. S2CID 21762469.
- ^ Inouye S, Herzer PJ, Inouye M (February 1990). "Two independent retrons with highly diverse reverse transcriptases in Myxococcus xanthus". Proceedings of the National Academy of Sciences of the United States of America. 87 (3): 942–5. Bibcode:1990PNAS...87..942I. doi:10.1073/pnas.87.3.942. PMC 53385. PMID 1689062.
- ^ Yee T, Furuichi T, Inouye S, Inouye M (August 1984). "Multicopy single-stranded DNA isolated from a gram-negative bacterium, Myxococcus xanthus". Cell. 38 (1): 203–9. doi:10.1016/0092-8674(84)90541-5. PMID 6088065. S2CID 41165293.
- ^ Furuichi T, Inouye S, Inouye M (January 1987). "Biosynthesis and structure of stable branched RNA covalently linked to the 5' end of multicopy single-stranded DNA of Stigmatella aurantiaca". Cell. 48 (1): 55–62. doi:10.1016/0092-8674(87)90355-2. PMID 2431795. S2CID 32376617.
- ^ a b c Das R, Shimamoto T, Hosen SM, Arifuzzaman M (2011). "Comparative Study of different msDNA (multicopy single-stranded DNA) structures and phylogenetic comparison of reverse transcriptases (RTs): evidence for vertical inheritance" (PDF). Bioinformation. 7 (4): 176–9. doi:10.6026/97320630007176. PMC 3218519. PMID 22102774.
- ^ Lampson BC, Sun J, Hsu MY, Vallejo-Ramirez J, Inouye S, Inouye M (February 1989). "Reverse transcriptase in a clinical strain of Escherichia coli: production of branched RNA-linked msDNA" (PDF). Science. 243 (4894 Pt 1): 1033–8. Bibcode:1989Sci...243.1033L. doi:10.1126/science.2466332. PMID 2466332. Archived from the original (PDF) on 2014-12-22. Retrieved 2012-02-08.
- ^ Lima TM, Lim D (May 1995). "Isolation and characterization of host mutants defective in msDNA synthesis: role of ribonuclease H in msDNA synthesis". Plasmid. 33 (3): 235–8. doi:10.1006/plas.1995.1026. PMID 7568472.
- ^ Hsu MY, Inouye M, Inouye S (December 1990). "Retron for the 67-base multicopy single-stranded DNA from Escherichia coli: a potential transposable element encoding both reverse transcriptase and Dam methylase functions". Proceedings of the National Academy of Sciences of the United States of America. 87 (23): 9454–8. Bibcode:1990PNAS...87.9454H. doi:10.1073/pnas.87.23.9454. PMC 55184. PMID 1701261.
- ^ Inouye S.; Inouye M. (1993). "Bacterial Reverse Transcriptase". In Goff, Stephen and Anna M. Skalka (ed.). Reverse transcriptase. Cold Spring Harbor monograph series. Vol. 23. Plainview, N.Y: Cold Spring Harbor Laboratory Press. ISBN 978-0-87969-382-4.
- ^ a b c d Lampson BC, Inouye M, Inouye S (2005). "Retrons, msDNA, and the bacterial genome" (PDF). Cytogenetic and Genome Research. 110 (1–4): 491–9. doi:10.1159/000084982. PMID 16093702. S2CID 24854188. Archived from the original (PDF) on 2016-03-05. Retrieved 2012-02-08.
- ^ Simon DM, Zimmerly S (December 2008). "A diversity of uncharacterized reverse transcriptases in bacteria". Nucleic Acids Research. 36 (22): 7219–29. doi:10.1093/nar/gkn867. PMC 2602772. PMID 19004871.
- ^ a b Das R, Shimamoto T, Arifuzzaman M (2011). "A Novel msDNA (Multicopy Single-Stranded DNA) Strain Present in Yersinia frederiksenii ATCC 33641 Contig01029 Enteropathogenic Bacteria with the Genomic Analysis of It's Retron". Journal of Pathogens. 2011 (693769): 693769. doi:10.4061/2011/693769. PMC 3335539. PMID 22567337.
- ^ Sherman LA, Chattopadhyay S (1993). "The molecular basis of allorecognition". Annual Review of Immunology. 11: 385–402. doi:10.1146/annurev.iy.11.040193.002125. PMID 8476567.
- ^ Buss, Leo (2006). The Evolution of Individuality. Princeton University Press. ISBN 978-0-691-08469-5.
- ^ Shimamoto T, Kawanishi H, Tsuchiya T, Inouye S, Inouye M (June 1998). "In vitro synthesis of multicopy single-stranded DNA, using separate primer and template RNAs, by Escherichia coli reverse transcriptase". Journal of Bacteriology. 180 (11): 2999–3002. doi:10.1128/JB.180.11.2999-3002.1998. PMC 107272. PMID 9603895.
- ^ a b Inouye S, Hsu MY, Xu A, Inouye M (October 1999). "Highly specific recognition of primer RNA structures for 2'-OH priming reaction by bacterial reverse transcriptases". The Journal of Biological Chemistry. 274 (44): 31236–44. doi:10.1074/jbc.274.44.31236. PMID 10531319.
- ^ Jacobo-Molina A, Ding J, Nanni RG, Clark AD, Lu X, Tantillo C, Williams RL, Kamer G, Ferris AL, Clark P (July 1993). "Crystal structure of human immunodeficiency virus type 1 reverse transcriptase complexed with double-stranded DNA at 3.0 A resolution shows bent DNA". Proceedings of the National Academy of Sciences of the United States of America. 90 (13): 6320–4. Bibcode:1993PNAS...90.6320J. doi:10.1073/pnas.90.13.6320. PMC 46920. PMID 7687065.
- ^ Sarafianos SG, Das K, Tantillo C, Clark AD, Ding J, Whitcomb JM, Boyer PL, Hughes SH, Arnold E (March 2001). "Crystal structure of HIV-1 reverse transcriptase in complex with a polypurine tract RNA:DNA". The EMBO Journal. 20 (6): 1449–61. doi:10.1093/emboj/20.6.1449. PMC 145536. PMID 11250910.
추가 정보
- Lampson B, Inouye M, Inouye S (2001). The msDNAs of bacteria. Progress in Nucleic Acid Research and Molecular Biology. Vol. 67. pp. 65–91. doi:10.1016/S0079-6603(01)67025-9. ISBN 9780125400671. PMID 11525386.
- Zimmerly, Steven (2005). "Mobile introns and retroelements in bacteria". In Mullany, Peter (ed.). The Dynamic Bacterial Genome. Advances in Molecular and Cellular Microbiology. Vol. 8. Cambridge University Press. pp. 121–148. doi:10.1017/CBO9780511541544.004. ISBN 978-0-511-54154-4.

