가이드 RNA
Guide RNA가이드 RNA(gRNA)는 RNA 또는 DNA 표적화 효소의 가이드 역할을 하는 RNA 조각으로 복합체를 형성합니다.이러한 효소는 종종 표적 RNA나 DNA를 삭제, 삽입 또는 변경합니다.이러한 기능은 자연스럽게 발생하며 중요한 기능을 제공하지만 CRISPR-Cas9 및 CRISPR-Cas12와 같은 대상 편집용으로 사용할 수도 있습니다.
역사
RNA 편집 가이드 RNA는 1990년 B에 의해 발견되었다.라이슈마니아 타렌톨레라고 불리는 미토콘드리아에 있는 블룸, N. 바칼라라, L. 심슨[1].가이드 RNA는 맥시서클 DNA로 인코딩되며 mRNA의 편집 영역 내와 일치하는 염기서열을 포함합니다.베이스의 절단, 삽입 및 삭제를 가능하게 합니다.
프로티스트의 가이드 RNA
트리파노소마티드 원생체 및 다른 키네토플라스티드는 "RNA 편집"으로 알려진 새로운 전사 후 미토콘드리아 RNA 수정 과정을 가지고 있다.그들은 미토콘드리아에 고도로 조직된 DNA 세그먼트의 큰 부분을 가지고 있다.이 미토콘드리아 DNA는 원형이며 맥시사이클과 미니사이클로 나뉜다.세포는 부호화 영역과 비부호화 영역을 모두 가진 약 20~50개의 맥시클을 포함한다.코딩 영역은 보존성이 높고(16-17kb), 코딩되지 않은 영역은 종류에 따라 다릅니다.미니 사이클은 작지만 맥시사이클보다 더 많다.미니 사이클은 키네토플라스티드 DNA 질량의 95%를 차지한다.맥시시클은 "크립토겐"과 일부 gRNA를 부호화할 수 있다. 미니 사이클은 대부분의 gRNA를 부호화할 수 있다.250개 이상의 미니 동그라미로 최대 1000개의 gRNA를 부호화할 수 있습니다.심지어 만약 그들이 다른 시퀀스입니다. 반면에 다른gRNA 시퀀스pre-edited mRNA를 보완하지 않는다. 어떤 gRNA 유전자. Maxicircles과 분자 minicircles DNA의 편모의 단일 미토콘 도리아의 내부 객실의 기지에 위치해 있는 거대한 네트워크에 연쇄형은 일란성 삽입 및 삭제 사이트들을 보여 준다.[1]
맥시서클 전사물의 대부분은 배열 내의 여러 프레임 이동으로 인해 단백질로 변환될 수 없다.이러한 프레임 이동은 다른 세포로부터 미토콘드리아 단백질과 상동하는 미토콘드리아 단백질로 번역되는 열린 판독 프레임을 만드는 정확한 부위에 우리딘 잔기를 삽입 및 삭제함으로써 전사 후 보정된다.삽입 및 결실은 보완 배열의 형태로 편집 정보를 인코딩하는 짧은 안내 RNA(gRNA)에 의해 매개됩니다(GU 및 GC 염기쌍을 허용합니다).
gRNA-mRNA 복합체
가이드 RNA는 주로 DNA 맥시클의 유전자 간 영역에서 전사되며, 이들은 성숙한 mRNA에 대한 상보적이다.gRNA는 처음에 사전 편집된 mRNA와 상호작용한 후 보완 mRNA와 함께 5' 영역 염기쌍과 상호작용하는 것이 중요하다.gRNA의 3' 말단에는 mRNA의 A 및 G 리치 영역과 상호작용하여 안정적인 복합체를 형성하는 올리고 U 꼬리(길이 5-25 뉴클레오티드)가 포함되어 있다.이 초기 하이브리드는 [2]편집할 특정 mRNA 부위를 인식하는 데 도움이 됩니다.
기능.
미토콘드리아에서 두 개의 게놈이 존재하는데, 그 중 하나는 다른 게놈의 오류를 수정하는 배열 정보를 포함하고 있다.편집은 일반적으로 mRNA에서 3~5'로 진행됩니다.초기 편집 이벤트는 gRNA가 편집 부위의 바로 하류에서 상보적인 mRNA 염기서열을 가진 RNA 이중체를 형성할 때 발생합니다.그런 다음 gRNA-mRNA 앵커에 인접한 첫 번째 불일치 염기의 분열을 지시하는 다수의 리보핵단백질 복합체를 모집한다.우리딜전달효소는 3' 말단에 'U'를 삽입하고 RNA 연결효소는 2개의 절단단을 결합하는 역할을 한다.다음으로 인접한 업스트림 편집 사이트도 같은 방법으로 변경됩니다.보통 단일 gRNA는 여러 편집 사이트의 정보를 부호화하며(편집 "블록") 편집은 완전한 gRNA/mRNA 이중화를 생성합니다.이러한 변형 과정을 원래의 효소 캐스케이드 [2]모델이라고 한다.
"pan-edited"[3] mRNA의 경우 듀플렉스 언와인드 및 다른 gRNA가 편집된mRNA 시퀀스와 듀플렉스를 형성하여 다른 편집 라운드를 시작합니다.중복되는 gRNA는 편집 "도메인"을 형성합니다.일부 유전자에는 여러 편집 영역이 있습니다.특정 유전자의 편집 범위는 트리파노소마이드 종에 따라 다르다.변화는 3' 측에서 편집의 손실로 구성되며, 아마도 특정 gRNA를 인코딩하는 미니 원 시퀀스 클래스의 손실로 인해 발생할 수 있다.진화 과정에서 편집의 부분적, 그리고 경우에 따라서는 완전한 손실을 설명하기 위해 역방향 배치 모델이 제안되었다.대부분의 경우 편집 손실은 치명적이지만 오래된 실험실 균주에서는 손실이 발견되었습니다.이 고대 원생들의 오랜 진화 역사에 대한 편집의 유지는 선택적인 우위의 존재를 시사하며, 그 정확한 성격은 아직 불확실하다.
왜 트리파노소마티드가 mRNA를 생성하기 위해 이렇게 정교한 메커니즘을 사용하는지는 명확하지 않다.그것은 트라이파노소마티드의 조상인 보도니드에 존재하며, 키네토플라스티드와 같은 공통 조상에서 갈라진 유글로이드에 존재하지 않기 때문에 킨토플라스티드의 조상인 초기 미토콘드리아에서 유래했을 수 있다.
원생동물 Leishmania tarentolae에서는 이 과정을 사용하여 18개의 미토콘드리아 유전자 중 12개가 편집된다.그러한 유전자 중 하나가 Cyb이다.mRNA는 실제로 2회 연속으로 편집됩니다.첫 번째 편집의 경우 mRNA의 관련 시퀀스는 다음과 같습니다.
mRNA 5' AAAAAGGCUUAACUUUCUGU 3'
3' 끝은 염기쌍으로 gRNA(이 경우 gCyb-I gRNA)를 고정하는 데 사용됩니다(일부 G/U 쌍이 사용됩니다).5' 끝은 정확히 일치하지 않으며 세 가지 특정 핵산 분해효소 중 하나가 불일치 부위에서 mRNA를 분해한다.
gRNA 3' AAUAUAUAUAUAUAUAUA 5' mRNA 5'A 아가A A GCUAUAUACU 3'GUGU 3'
이제 mRNA는 각 편집 사이트에 U를 연속적으로 추가하여 다음 시퀀스를 제공함으로써 "복구"됩니다.
gRNA 3' AUAAUAUAUAUAUAUAUAUAUAUAUAUAUAUAUAUAUAUUAUUAUAUUUUUAUAUUUAUAUUAUUUAUUUAUUUUUUUAUAUUUUUAUAUAUAUAUAUAUAUAUAUUAUUUAUUUAUAUAUAUAUAUAUAUAUAUAUAUAUAU
이 특별한 유전자는 두 개의 겹치는 gRNA 편집 부위가 있다.이 섹션의 5' 끝은 다른 gRNA(gCyb-I gRNA)의 3' 앵커이다.
원핵생물의 유도 RNA
원핵생물에서의 CRISPR
박테리아와 고세균을 포함하는 대부분의 원핵생물들은 적응 면역 체계로 CAS 효소와 함께 CRISPR(클러스터링된 규칙적인 간격의 짧은 회문 반복)을 사용한다.원핵생물들이 파지에 의해 감염되어 공격을 막아낼 때, 특정 Cas 효소는 파지의 DNA를 절단하고 CRISPR 배열의 반복 사이에 부분을 통합할 것입니다.저장된 세그먼트는 미래의 바이러스 공격에서 인식될 수 있으며 Cas 효소는 그들의 RNA 복사를 연관된 CRISPR 세그먼트와 함께 gRNA로 사용하여 외래 서열을 식별하고 그것들을 무해하게 만들 것입니다.
구조.
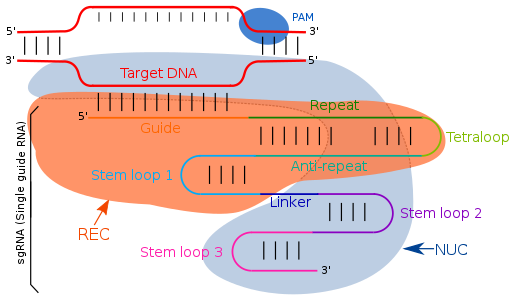
Guide RNA는 단순한 Watson-Crick 염기쌍을 통해 상보적 염기서열을 대상으로 합니다.Type II CRISPR/cas 시스템에서는 단일 가이드 RNA가 표적 특정 영역을 지시합니다.단일 가이드 RNA는 인공적으로 프로그램된 2개의 RNA 분자의 조합으로, 1개의 성분(tracrRNA)은 Cas9 엔도핵산가수분해효소 활성을 담당하며, 다른(crRNA)은 표적 특이 DNA 영역에 결합한다.따라서 트랜스활성화 RNA(tracrRNA)와 crRNA는 두 가지 핵심 성분이며 sgRNA를 형성하는 테트라루프에 의해 결합된다.TracrRNA는 그 자체로 줄기세포 구조를 가진 염기쌍으로 엔도핵산가수분해효소 효소에 결합한다.CRISPR 궤적을 전사하면 반복 배열로 인해 스페이서 측면 영역을 갖는 CRISPR RNA(crRNA)는 18~20개의 염기쌍으로 구성된다.crRNA는 Cas9에 의해 절단된 특정 상보적 표적 영역을 식별하며, 이들 모두 이펙터 복합체로 알려져 있다.가이드 RNA의 crRNA 시퀀스의 수정으로 결합 위치를 변경하여 사용자 정의 프로그램으로 정의할 수 있습니다.
적용들
gRNA 설계
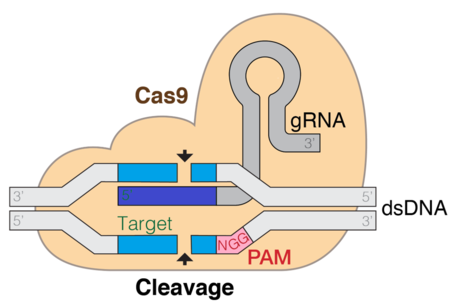
CRISPR-Cas9의 표적 특이성은 gRNA의 5' 말단에서 20-nt 시퀀스에 의해 결정된다.원하는 표적 배열은 원형사양인접 모티브(PAM)보다 선행되어야 합니다. 원형사양인접 모티브(PAM)는 일반적으로 2-6개의 염기쌍의 짧은 DNA 배열로 CRISPR-Cas9와 같은 CRISPR 시스템에 의해 분할을 목표로 하는 DNA 영역을 따릅니다.PAM은 Cas nuclease가 절단되는 데 필요하며 일반적으로 절단 부위 하류에서 3~4개의 뉴클레오티드가 발견된다.타깃에 대한 gRNA의 베이스 페어링 후 Cas9은 PAM의 약 3-nt 상류에서 이중 스트랜드 브레이크를 매개한다.
가이드 시퀀스의 GC 함량은 40~80%여야 합니다.GC 함량이 높으면 RNA-DNA 듀플렉스가 안정되고 오프타깃 하이브리드가 불안정해집니다.가이드 시퀀스의 길이는 17-24bp 사이여야 하며, 시퀀스가 짧을수록 오프타깃 효과가 최소화된다.17bp 미만의 가이드 시퀀스는 여러 개의 궤적을 대상으로 할 가능성이 있습니다.<https://www.youtube.com/watch?v=dXPDefej0Ps>
CRISPR Cas9
CRISPR(Clustered repectly interspaceed short palindromic repeates)/Cas9은 유전자 편집 및 유전자 치료에 사용되는 기술이다.Cas는 유도 RNA에 의해 지시된 특정 위치에서 DNA를 절단하는 핵산가수분해효소이다.이것은 이중 가닥 복구 경로에 따라 유전자 녹아웃 또는 녹아웃을 도입할 수 있는 표적 특이 기술이다.증거는 시험관내와 시험관내 모두 Cas9 및 표적 DNA 배열 결합을 위해 tracrRNA를 필요로 한다는 것을 보여준다.CRISPR CAS9 시스템은 세 가지 주요 단계로 구성됩니다.첫 번째 단계는 게놈 배열에서 외부 DNA 스페이서를 추가하여 CRISPR 궤적 영역의 염기를 확장하는 것입니다.cas1과 cas2와 같은 몇 가지 다른 단백질은 새로운 스페이서를 찾는데 도움을 준다.다음 단계는 CRISPR의 전사를 수반한다: CRISPR 반복 스페이서 어레이의 전사에 의해 프리 CRNA(precursor CRISPR RNA)가 발현된다.pre-crRNA에서 추가 변형이 이루어지면 짧은 crRNA를 형성하는 단일 스페이서 측면 영역으로 변환되며, RNA 성숙 과정은 I형과 II형이 유사하지만 III형이 다르며, 이 단계에서 트레이서로서의 aRNA를 첨가한다.세 번째 단계는 cas9 단백질의 결합과 DNA 세그먼트를 절단하도록 지시하는 것이다.Cas9 단백질은 이펙터 복합체를 형성하는 crRNA와 tracrRNA의 결합 형태에 결합합니다.이것은 핵산가수분해효소 [4]활성을 위해 그것을 지시하는 cas9 단백질의 가이드 RNA 역할을 한다.
RNA 돌연변이 유발
하나의 중요한 유전자 조절 방법은 RNA 돌연변이 발생으로, gRNA의 도움을 받아 RNA 편집을 통해 도입될 수 있다. Guide RNA는 특정 표적 부위에서 아데노신을 이노신으로 대체하고 유전자 [5]코드를 수정한다.아데노신탈아미나아제는 코돈과 다른 단백질 기능을 변화시킴으로써 전사 후 수정을 가져오는 RNA에 작용한다.가이드 RNA는 작은 핵소형 RNA이며, 리보 단백질과 함께 rRNA의 리보메틸화와 프리리보솜 RNA의 프듀우리딘 도입과 같은 세포 내 RNA 변화를 수행합니다. 가이드 RNA는 반감각 RNA 배열에 결합하고 RNA 변형을 조절합니다.일반적으로 작은 간섭 RNA(siRNA)와 마이크로 RNA(miRNA)가 표적 RNA 배열로 사용되며 크기가 작기 때문에 개조가 비교적 쉽다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b Blum, B.; Bakalara, N.; Simpson, L. (1990-01-26). "A model for RNA editing in kinetoplastid mitochondria: "guide" RNA molecules transcribed from maxicircle DNA provide the edited information". Cell. 60 (2): 189–198. doi:10.1016/0092-8674(90)90735-w. ISSN 0092-8674. PMID 1688737. S2CID 19656609.
- ^ a b Connell, Gregory J.; Byrne, Elaine M.; Simpson, Larry (1997-02-14). "Guide RNA-independent and Guide RNA-dependent Uridine Insertion into Cytochrome b mRNA in a Mitochondrial Lysate from Leishmania tarentolae ROLE OF RNA SECONDARY STRUCTURE". Journal of Biological Chemistry. 272 (7): 4212–4218. doi:10.1074/jbc.272.7.4212. ISSN 0021-9258. PMID 9020135.
- ^ Maslov, Dmitri A. (October 2010). "Complete set of mitochondrial pan-edited mRNAs in Leishmania mexicana amazonensis LV78". Molecular and Biochemical Parasitology. 173 (2): 107–114. doi:10.1016/j.molbiopara.2010.05.013. ISSN 0166-6851. PMC 2913609. PMID 20546801.
- ^ Karvelis, Tautvydas; Gasiunas, Giedrius; Miksys, Algirdas; Barrangou, Rodolphe; Horvath, Philippe; Siksnys, Virginijus (2013-05-01). "crRNA and tracrRNA guide Cas9-mediated DNA interference in Streptococcus thermophilus". RNA Biology. 10 (5): 841–851. doi:10.4161/rna.24203. ISSN 1547-6286. PMC 3737341. PMID 23535272.
- ^ Fukuda, Masatora; Umeno, Hiromitsu; Nose, Kanako; Nishitarumizu, Azusa; Noguchi, Ryoma; Nakagawa, Hiroyuki (2017-02-02). "Construction of a guide-RNA for site-directed RNA mutagenesis utilising intracellular A-to-I RNA editing". Scientific Reports. 7: 41478. Bibcode:2017NatSR...741478F. doi:10.1038/srep41478. ISSN 2045-2322. PMC 5288656. PMID 28148949.
추가 정보
- 시험관 내 RNA 방향 우리딘 삽입 RNA 편집 안내://www.jbc.org/content/272/7/4212.full
- Blum, Beat; Simpson, Larry (1990). "Guide RNAs in kinetoplastid mitochondria have a nonencoded 3′ oligo(U) tail involved in recognition of the preedited region". Cell. 62 (2): 391–397. doi:10.1016/0092-8674(90)90375-O. PMID 1695552. S2CID 2181338.
- Kurata, Morito; Wolf, Natalie K.; Lahr, Walker S.; Weg, Madison T.; Kluesner, Mitchell G.; Lee, Samantha; Hui, Kai; Shiraiwa, Masano; Webber, Beau R.; Moriarity, Branden S. (2018). "Highly multiplexed genome engineering using CRISPR/Cas9 gRNA arrays". PLOS ONE. 13 (9): e0198714. Bibcode:2018PLoSO..1398714K. doi:10.1371/journal.pone.0198714. PMC 6141065. PMID 30222773.
- Khan, Fehad J.; Yuen, Garmen; Luo, Ji (2019). "Multiplexed CRISPR/Cas9 gene knockout with simple crRNA:tracrRNA co-transfection". Cell & Bioscience. 9: 41. doi:10.1186/s13578-019-0304-0. PMC 6528186. PMID 31139343.
- Nishimasu, Hiroshi; Nureki, Osamu (2017). "Structures and mechanisms of CRISPR RNA-guided effector nucleases". Current Opinion in Structural Biology. 43: 68–78. doi:10.1016/j.sbi.2016.11.013. PMID 27912110.
- Chuai, Guohui; Ma, Hanhui; Yan, Jifang; Chen, Ming; Hong, Nanfang; Xue, Dongyu; Zhou, Chi; Zhu, Chenyu; Chen, Ke; Duan, Bin; Gu, Feng; Qu, Sheng; Huang, Deshuang; Wei, Jia; Liu, Qi (2018). "DeepCRISPR: Optimized CRISPR guide RNA design by deep learning". Genome Biology. 19 (1): 80. doi:10.1186/s13059-018-1459-4. PMC 6020378. PMID 29945655.