토포이소머라아제
Topoisomerase| 식별자 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| EC 번호 | 5.6.2.1 | ||||||||
| CAS 번호 | 80449-01-0 | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 엔트리 | ||||||||
| ExPASy | NiceZyme 뷰 | ||||||||
| 케그 | KEGG 엔트리 | ||||||||
| 메타사이크 | 대사 경로 | ||||||||
| 프라이머리 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBum | ||||||||
| |||||||||
| DNA토포이소머라아제, ATP의존성(타입II) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| EC 번호 | 5.6.2.2 | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 엔트리 | ||||||||
| ExPASy | NiceZyme 뷰 | ||||||||
| 케그 | KEGG 엔트리 | ||||||||
| 메타사이크 | 대사 경로 | ||||||||
| 프라이머리 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBum | ||||||||
| |||||||||
DNA 토포이소머라아제(또는 토포이소머라아제)는 DNA의 위상 상태 변화를 촉매하고, 이완되고 초코일 형태의 상호 변환, 연결(catenated)된 종과 연결되지 않은 [1][2]종, 그리고 매듭진 DNA와 매듭되지 않은 DNA이다.예를 들어, DNA 복제와 전사 중에 DNA 이중의 오버와잉을 초래할 수 있는 이중 나선 구조의 얽힌 특성 때문에 DNA의 위상 문제가 발생합니다.만약 변하지 않는다면, 이 비틀림은 결국 이러한 과정에 관여하는 DNA 또는 RNA 중합효소가 DNA 나선을 따라 지속하는 것을 멈출 것이다.두 번째 위상적 문제는 복제 중에 DNA가 연결되거나 엉킴으로써 발생합니다.해결되지 않은 채로 두면, 복제된 DNA 사이의 연결은 세포 분열을 방해할 것이다.DNA 토포이소머라아제는 이러한 유형의 위상 문제를 예방하고 교정합니다.그들은 DNA에 결합하고 DNA 가닥 중 하나(타입 I 토포이소머라아제) 또는 둘 다(타입 II 토포이소머라아제)의 당-인산 골격을 절단함으로써 이것을 한다.이 일시적인 파괴는 DNA를 풀거나 풀 수 있게 하고, 이러한 과정이 끝날 때, DNA 골격은 다시 봉합된다.DNA의 전체적인 화학적 구성 및 결합성은 변하지 않기 때문에 DNA 기질과 생성물은 화학적 이성질체로 위상만 다르다.
검출
최초의 DNA 토포이소머라아제는 1971년 James Wang에 의해 박테리아에서 발견되었고 처음에는 γ([3]오메가) 단백질로 명명되었다; 그것은 현재 대장균(E. coli) 토포이소머라아제 I(topo I)로 불리며 타입 IA의 효소군을 대표한다.그 후, James Champoux와 Renato Dulbecco에 [4]의해 진핵 세포(랫간)에서 유사한 활성이 발견되었다.; 책임이 있는 효소인 진핵 토포 I은 독특한 메커니즘을 가지고 있고 IB형 패밀리를 대표적이다.최초로 발견된 타입 II 토포이소머라아제는 1976년 [5]마틴 겔러트와 동료들에 의해 박테리아로부터의 DNA 자이라아제였고 니콜라스 코자렐리와 [6]동료들에 의해 특징지어졌습니다.DNA 자이라아제는 DNA에 음의 슈퍼코일의 도입을 촉매하고 이것을 하는 유일한 II형 효소이며, 다른 모든 것들은 DNA 이완을 촉매합니다.타입 II 효소는 ATP에 의존하며 두 DNA 가닥을 한 가닥이 아닌 순간적으로 절단한다는 점에서 타입 I과 기계적으로 구별된다.Type II 토포이소머라아제는 세균 바이러스와 진핵생물에서 [7][8][9]후속적으로 확인되었다.Topo EC 코드는 다음과 같습니다. ATP 비의존성(타입 I), EC 5.6.2.1, ATP 의존성(타입 II): EC 5.6.2.타입 I 토포이소머라아제 중 예외인 역자이라아제는 헬리케이스 도메인(EC 3.6.4.12)을 포함하고 ATP 의존적인 방식으로 양의 초코일을 도입한다.따라서 EC 5.6.2.2(표1)로 분류되는 유일한 I형 토포이소머라아제이다.
DNA토폴로지
DNA의 이중 나선 구조는 두 개의 폴리뉴클레오티드 가닥이 서로 얽혀 있는 것을 포함하며, 이것은 잠재적으로 위상적인 문제를 일으킨다.DNA 토폴로지는 이중 나선의 꼬임을 바꾸고 슈퍼코일,[10] 매듭, 카테난과 같은 DNA의 3차 구조를 발생시키는 두 DNA 가닥의 교차를 말한다.DNA의 이중 나선 구조와 관련된 잠재적 위상 문제는 1953년 제임스 왓슨, 프란시스 크릭, 로잘린드[11][12][13] 프랭클린에 의해 처음 설명되고 맥스 델브룩과 존 [14][15]케언스의 연구에 의해 더욱 발전된 직후에 인식되었다.폐쇄원형 이중사슬 DNA는 연결번호(Lk), 트위스트(Tw), 라이트(Writhe)의 3가지 매개변수로 설명할 수 있다(그림 1).여기서 Lk는 두 가닥이 연결된 횟수를 말하며, Tw는 나선축에 대해 측정된 DNA의 나선 회전수를 의미하며, Wr은 공간 내 DNA 나선의 경로의 코일링을 정량화하여 종종 '슈퍼 코일링'과 동일시한다.
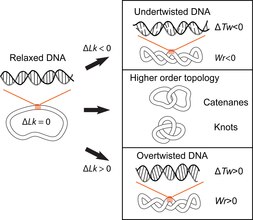
3개의 파라미터는 다음과 같이 관련되어 있습니다.Lk = Tw +Wr.이는 1959년[16] 켈루거 리아누에 의해 최초로 취득된 수학적 동일성으로, 켈루거 리아누 또는 켈루거 리아누로 불린다.화이트-풀러 [17][18]정리Lk는 Helix의 한쪽 또는 양쪽 Strand를 절단하지 않고는 변경할 수 없으며 Tw와 Wr은 상호 변환 가능하며 용액 조건에 따라 달라진다.슈퍼 코일링은 0이 아닌 연결 차이를 가진 DNA의 속어이며, 보다 정확하게는 특정 연결 차이(θ = δLk0/Lk, 여기서0 Lk는 완화 DNA 원의 평균 연결 수)로 불린다.DNA는 Lk의 Lk가 완화상태o(Lk-Lk = δLk, δLk > 0)보다0 높으면 양초코일이라고 하며, 이는 완화분자에 비해 Tw 및/또는 Wr이 증가함을 의미한다.반대로 분자의 Lk가 Lk0(δLk<0)보다 낮으면 DNA가 음의 초코일화된다.
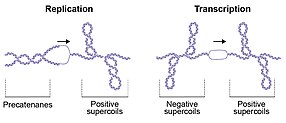
DNA에서 위상 교란의 결과는 이중 가닥이 분리되는 DNA 복제로 예시된다. 이러한 분리는 복제 포크 앞에 양성 슈퍼코일(DNA 오버와인드 또는 오버 트위스트)이 형성되고 뒤에 딸 가닥(프레테탄)이[10][19] 얽힌다(그림 2).양성 슈퍼코일이 완화되지 않으면 복제 포크의 진행이 방해되고, 딸 가닥의 연결을 해제하지 못하면 세포 분열에 [20]필요한 게놈 분리가 방지된다.RNA 중합효소에 의한 전사는 또한 전사 복합체 앞과 뒤에 양의 슈퍼코일을 생성한다(그림 2).이 효과는 1987년 [21]Leroy Liu와 James Wang이 기술한 바와 같이 트윈 슈퍼코일 도메인 모델로 알려져 있습니다.이러한 위상학적 동요는 DNA 대사가 진행되기 위해 해결되어야 하며, 세포는 세포 분열과 생기를 가능하게 하기 위해 게놈을 효율적으로 복제, 전사, 분할할 수 있게 한다.DNA의 매듭은 박테리오파지와 재조합 [10]반응의 산물로 발견될 수 있다.일반적으로, DNA의 매듭은 유해하며 (토포이소머라아제에 의해) 제거되어야 한다.DNA 카테난은 원형 분자의 복제에 의해 형성되며 세포 분열 동안 딸 분자의 적절한 분리를 가능하게 하기 위해 토포이소머라아제 또는 재조합 효소에 의해 분해될 필요가 있다.분해능을 필요로 하는 DNA 토폴로지의 해로운 측면 외에도, 유익한 측면도 있다.예를 들어 플라스미드 복제에는 원점의 음의 슈퍼코일이 필요하며, 이는 국소 용융을 촉진하고 복제를 시작하는 데 필요한 단일 가닥 DNA를 노출시킵니다.마찬가지로, 주요 박테리아 기원 오리C로부터의 복제의 시작도 음의 [22][23]슈퍼코일을 필요로 한다.또한 부분적으로 음의 슈퍼코일링에 의해 대장균 게놈의 콤팩트화를 실현한다.
종류들
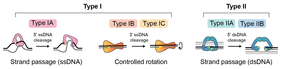
DNA 토포이소머라아제는 DNA의 위상 문제를 해결하기 위해 진화한 효소이다(표2).[10]그들은 DNA의 한 가닥 또는 두 가닥의 일시적인 파괴를 통해 이것을 한다.이에 따라 토포는 일시적인 단일 가닥 절단을 수반하는 반응을 촉매하는 유형 I과 일시적인 이중 가닥 절단을 수반하는 반응을 촉매하는 유형 II의 두 가지 유형으로 분류되었다(그림 3; 표 2).하위 유형은 이러한 분류 내에 존재합니다.
타입 I
이 효소들은 DNA의 일시적인 단일 가닥 절단을 통해 DNA 토폴로지의 변화를 촉매한다.반응은 단일 가닥 및 이중 가닥 DNA 기질 모두에서 발생할 수 있으며 '스위벨' 또는 '스트랜드 패스' 메커니즘을 통해 진행될 수 있다(그림 3).반응의 범위는 DNA 슈퍼코일 완화, 단일가닥 원의 매듭 해제 및 탈카테네이션(decatenation)을 포함한다.단, 적어도 하나의 파트너가 단일가닥 영역을 가지고 있는 경우.고생물 효소인 역자이라아제에서는 DNA의 양성 초코일이 가능하다.[24]
타입 IA
타입 IA는 단량체이며 DNA의 단가닥 세그먼트에 결합한다.이들은 효소의 티로신과 DNA의 5γ-인산 사이의 티로실-인산 결합 형성을 통해 일시적인 단일 가닥 분해를 유도한다.파손이 일어나는 DNA의 부분은 '게이트' 또는 'G-세그먼트'라고 불리며, 그 분할은 DNA의 또 다른 세그먼트인 '수송' 또는 'T-세그먼트'의 통과를 '스트랜드-패스'[25] 과정으로 허용한다.그 후 G세그먼트의 경계가 이어집니다.가닥 통과가 일어나려면 Topo IA는 DNA 게이트를 열고 T-segment 전달을 허용하는 구조 변화를 겪어야 합니다.DNA 완화 반응 중에 이 과정은 DNA의 연결 수를 +/-1로 변경합니다(그림 4).IA형 토포이소머라아제에는 원핵생물 토포I 및 III, 진핵생물 토포III 및 IIIβ 및 고생물 효소 역자이라아제가 포함된다.호열성 고세균에서 발생하는 역자이라아제는 헬리카아제와 결합된 IA 토포를 포함하고 있으며 DNA에 [24]양성 슈퍼코일을 도입할 수 있는 유일한 효소이다.역자이라아제를 코드하는 유전자는 또한 일부 호열성 박테리아 그룹에서도 발견되는데,[26] 이 그룹에서는 고세균의 수평 유전자 전달에 의해 전달되었을 가능성이 있다.
타입 IB
IB형 토포이소머라아제(Type IB topoisomerase)는 효소의 티로신과 DNA의 3γ-인산 사이의 티로실-인산 결합 형성을 통해 DNA의 일시적인 단가닥 절단을 포함한 반응을 촉매한다.이러한 효소는 가닥 통과 메커니즘을 사용하는 대신 온전한 [27]가닥 주위에 절단된 가닥의 '스위벨' 또는 '제어된 회전'을 통해 작동합니다.이 회전 제어 메커니즘은 먼저 Baccinia topo[27][28] I에 대해 설명되었으며, 흠집이 다시 결합되기 전에 효소 공동 내에서 '마찰'에 의해 제어되는 속도로 온전한 가닥 주위의 자유단 DNA 회전을 허용한다(그림 3).이로 인해 분할 이벤트 및 종교 이벤트별로 링크 번호가 가변적으로 변경됩니다.이 메커니즘은 IA형 효소의 메커니즘과 구별되며, 두 그룹의 효소는 구조적으로 진화적으로 관련이 없다.IB형 토포이소머라아제의 예로는 바이러스의 토포 I 외에 진핵생물 핵과 미토콘드리아 토포 I가 포함되지만, 생명체의 세 영역에서 모두 확인되었다.
타입 IC
IC형 토포이소머라아제는 IB형 효소와 유사한 메커니즘을 공유하지만 구조적으로 구별된다.유일한 대표자는 고온성 메타노피루스 칸들레리에서 [29]발견되는 토포 V이다.
타입 II
타입 II 토포이소머라아제는 DNA의 일시적인 이중가닥 절단을 통해 DNA 토폴로지의 변화를 촉매한다.반응은 이중 가닥 DNA 기질에서 발생하며 가닥 통과 메커니즘을 통해 진행됩니다(그림 5).반응의 범위는 DNA 완화, DNA 초코일링, 매듭 풀기, 탈연 등을 포함한다.모든 타입 II 토포이소머라아제는 DNA 이완을 촉매할 수 있는 반면, 원형 박테리아 토포이소머라아제인 자이라아제는 또한 음의 슈퍼코일을 도입할 수 있다.일반적으로 단량체인 I형 토포이소머라아제와는 대조적으로 II형 토포이소머라아제는 동질체 또는 헤테로테트라머이다.이들은 진화적, 구조적 및 기계적 고려사항에 따라 두 가지 하위 유형으로 분류된다.타입 II 토포스에 대한 일반적인 가닥 통과 메커니즘은 DNA 게이트에서 게이트 세그먼트(G-segment)라고 불리는 하나의 DNA 이중 결합에서 시작합니다.트랜스포트 세그먼트(T-segment)라고 불리는 또 다른 이중화물은 ATP 작동 클램프로 포착되어 양쪽 가닥의 5' 포스포티로신 결합을 수반하는 G-segment의 일시적인 단절을 거쳐 C-게이트를 통해 방출되고 G-segment가 재결합된다(그림 5).효소 교체는 ATP의 결합과 가수분해를 필요로 한다.

타입 IIA
타입 IIA 토포이소머라아제는 효소 내 티로신(각 서브유닛에 하나씩)과 반대되는 DNA 가닥에서 4개의 염기로 엇갈린 5'-인산염 사이의 티로실-인산 결합 형성을 통해 DNA의 일시적인 이중 가닥 분해를 촉매한다.가닥 통과 반응은 분자 내 또는 분자 간(그림 5)일 수 있으므로 각각 초코일과 매듭 또는 연결 해제의 변화를 허용한다.이 과정은 DNA의 연결 번호를 +/-2만큼 변경합니다.IIA 토포이소머라아제에는 세균 자이라아제 및 토포 IV 외에 진핵생물 토포 IIα 및 토포 IIβ가 포함된다.DNA 자이라아제는 다른 II형 효소와 동일한 이중 가닥 통과 메커니즘을 준수하지만, 음의 슈퍼코일을 DNA에 도입하는 능력과 관련된 독특한 특징을 가지고 있습니다.G 세그먼트는 효소를 감싸고 있는 훨씬 긴 DNA 조각(>100 bp)의 일부이며, 이 중 한 팔은 이중 가닥 절단을 통과하는 T 세그먼트를 형성합니다(그림 5).자이라아제의 경우 ATP 가수분해에서 발생하는 자유에너지의 상당량이 DNA의 비틀림 응력으로 변환된다. 즉, 슈퍼코일은 에너지 요구 [30]과정이다.또한 ATP가 없는 경우 자이라제는 느린 DNA 완화 반응으로 음의 슈퍼코일을 제거할 수 있다.
타입 IIB
IIB형 또한 효소의 티로신과 DNA의 반대 가닥의 5'-인산 사이의 티로실-인산 결합 형성을 통해 일시적인 이중가닥 절단을 촉매하지만, IIB 효소의 경우 이중가닥 절단은 2-염기 비틀림을 가진다.IIB형 효소는 중요한 구조적 차이를 나타내지만 진화적으로는 IIIA형 효소와 관련이 있다.이러한 차이는 단백질 '게이트'(C 게이트) 중 하나가 없는 것을 포함한다(그림 5).원래 고고학에서 발견된 그것들은 진핵생물, 특히 식물에서도 발견되어 왔다. 예를 들어 토포 VI와 토포 VII가 있다.Topo VI는 이 아형의 가장 잘 연구된 효소이며 선호 탈카테나아제인 [31]것으로 생각된다.
약물 표적으로
비전문가에게 토포이소머라아제의 가장 중요한 측면은 항균 및 항암 화학요법의 약물 표적으로서의 역할이다. 몇몇 토포이소머라아제 표적 항균 및 항암제는 2019년 세계보건기구 필수 의약품 모델 목록에 포함되어 있다.이러한 두드러진 이유는 그들의 반응이 DNA의 일시적인 파괴를 통해 진행되기 때문인데, 이는 약물 결합에 의해 안정화되면 게놈 DNA의 독성 단일 또는 이중 가닥 파괴 생성으로 인해 세포 사멸을 초래할 수 있다.토포 표적 약물의 대부분은 이러한 방식으로 작용한다. 즉, 효소-DNA 공유 분열 [32][33][34]중간체를 안정화시킨다.
항균 화합물
박테리아 토포 I과 같은 타입 I 토포는 생존 가능한 항생제 [35]표적이지만, 현재 임상 사용에서는 이러한 효소를 표적으로 하는 화합물이 없습니다.그러나 II형 효소인 DNA 자이라아제와 DNA 토포이소머라아제 IV는 널리 사용되는 플루오로퀴놀론 항생제의 표적으로 큰 성공을 거두고 있다(그림 6).
플루오로퀴놀론(FQ)
퀴놀론 항균 화합물은 1960년대에 처음 개발되어 1980년대부터 [36]임상적으로 사용되고 있다.시프로플록사신, 레보플록사신 및 묵시플록사신(그림 6)과 같은 FQ 유도체는 매우 성공적이었다.이 화합물들은 DNA-단백질 공유분할 중간체를 안정시키기 위해 절단 부위에서 표적(자이라아제 또는 토포 IV) 및 DNA와 상호작용함으로써 작용한다.구체적으로는 DNA에 인터칼레이션하여 토포이소머라아제 반응의 DNA 종교화 단계를 방지한다(그림 5).이는 여러 토포이소머라아제 표적 항암제에도 사용되는 매우 효과적인 억제 메커니즘이다.그들의 눈부신 성공에도 불구하고, FQ에 대한 저항은 심각한 [36]문제이다.키나졸리데오네와 이미다졸피라지논과 [37]같은 다양한 화합물들이 비슷한 방식으로 작용하며, 이들 중 일부는 미래에 FQ를 대체할 것으로 기대된다.
아미노쿠마린
노보비오신, 클로바이오신, 쿠메르마이신 A와1 같은 아미노코마린(그림 6)은 스트렙토마이세스의 자연산물로 자이라아제 및 토포 [37]IV의 ATP분해효소 반응을 억제한다.목표물에 대해 매우 강력할 수 있지만 투과성 및 독성 문제로 인해 FQ의 임상적 성공을 누리지 못했다.
단백질 억제제
FQ와 유사한 방식으로 분열 복합체를 안정화시키는 세균독소 CcdB, MccB17 [38][39][40]및 ParE를 포함한 자이라아제 단백질 억제제가 다수 있다.비록 이 단백질들이 항균 물질로 생존할 수는 없지만, 그들의 작용 방식은 새로운 항균 화합물의 개발을 자극할 수 있다.자이라아제의 다른 단백질 억제제는 분열 복합체를 안정화시키는 것이 아니라 토포이소머라아제에 의한 DNA 결합을 방지한다.여기에는 QnrB1 및 MfpA와 [42][43]같은 YacG[41] 및 펜타펩타이드 반복 단백질이 포함됩니다. 이러한 단백질 억제제는 또한 플루오로퀴놀론에 대한 내성을 제공합니다.
항암제
항암화학요법에서는 인간 토포I와 토포II(α 및 β 아이소폼 모두)를 대상으로 할 수 있다(그림 7).[32][33][44][45][46][47]이들 화합물의 대부분은 FQ와 유사한 방식으로 작용한다. 즉, DNA-단백질 공유분열 복합체를 안정화시킴으로써, 그들은 촉매 억제제와 [33][34][48]구별되는 토포이소머라아제 독으로 알려져 있다.몇몇 인간 토포이소머라아제 억제제는 세계보건기구의 필수 의약품 목록에 포함되어 있다.
캄토테신(CPT)
원래 Camptotheca acuminata 나무에서 유래한 Camptothecin(그림 7)은 인간의 토포 I을 표적으로 하며, Topotecan과 Irinotecan과 같은 유도체가 암 화학 [33]치료에 널리 사용된다.캄토테신 및 그 유도체는 토포 I의 분열 복합체를 안정화시킴으로써 DNA에서 단백질 매개 닉의 교화를 방지한다.이러한 계면 억제제는 절단된 DNA와의 상호작용과 효소에 대한 수소 결합에 의해 안정화된다.CPT 유도체가 단일 가닥 절단 복합체를 안정화시키지만, 복제 또는 전사 기계와의 후속 충돌은 독성 이중 가닥 DNA 파괴를 발생시키는 것으로 생각된다.이들 화합물은 대장, 난소, 폐, 유방, 자궁경부 등 암을 치료하기 위한 1차 또는 2차 치료제로 사용된다.그러나 CPT 유도체는 독성과 관련된 한계와 화학적 불안정성으로 인한 치료적 반감기가 제한된다.새로운 토포I 억제제인 인데노이소퀴놀린과 플루오로인데노이소퀴놀린은 CPT 유도체의 한계를 극복하고 현재 임상시험 [49]중이다.
Etoposide(VP-16)
에토포시드(그림7)와 그 근친관계인 테니포시드(VM-26)는 공유가열착체를 안정시켜 절단된 [46]DNA의 종교화를 방지함으로써 토포II를 표적으로 하는 야생만드라케의 뿌리줄기에서 얻은 에피포도필로톡신 유도체이다.이것들은 일반적으로 고환 종양, 소세포 폐암, 백혈병을 포함한 암을 치료하기 위해 다른 화학요법 약물과 함께 사용된다.에토포시드 치료는 주로 토포IIβ와 [46]관련된 특정 게놈 이동에서 발생하는 2차 백혈병을 야기할 수 있다.
독소루비신
독소루비신(그림7) 및 관련 유도체 다우노루비신, 에피루비신 및 이다루비신은 인간 토포II를 표적으로 하는 세균 Streptomyces에서[48] 얻은 안트라사이클린으로 다른 토포이소머라아제 독과 마찬가지로 절단 복합체를 안정화시킨다.미톡산트론은 화학 및 기능적으로 안트라센디온과 [47]유사한 합성 안트라센디온이다.안트라사이클린은 암 치료에 사용된 최초의 토포이소머라아제 억제제이며 유방암, 림프종, 백혈병, 암종, 육종 및 기타 [47]종양을 포함한 광범위한 암에 대해 가장 널리 사용되고 효과적인 치료제 중 하나이다.이러한 화합물은 DNA 간극제이며, 따라서 Topo II를 [33]독살하는 것 외에도 광범위한 세포 DNA 과정에 영향을 미칠 수 있다.추가적인 세포독성은 반응성 산소종을 생성하는 무연쇄와 관련된 산화환원 반응에서 비롯된다.Topo IIβ의 중독과 함께 활성산소의 생성은 안트라사이클린의 [33]용량 제한 심독성을 초래한다.
머바로네
메르바론은 티오바르비투르산 유도체이며, 관련된 여러 비스디옥소피페라진 유도체 중 하나인 덱스라독산(ICRF-187)은 토포II의 촉매 억제제로서 토포II의 촉매 사이클 완료를 막지만 DNA 분할 복합체를 안정화시키지 못한다.이러한 촉매 억제제는 세포독성을 나타내며 임상시험에서 테스트되었지만, 현재 암 [47]치료에는 임상적으로 사용되지 않습니다.단, Topo II에 의한 ATP 가수분해를 차단하는 덱스라독산은 안트라사이클린과 [50][51]관련된 심장독성을 방지하기 위해 사용된다.
| 토포이소머라아제 | 서브패밀리 타입 | 기능. | 멀티메트릭 | 금속 의존성 | ATP 의존성 | 단가닥인가 이중가닥인가? | 균열 극성 | 링크 번호 변경(L) |
|---|---|---|---|---|---|---|---|---|
| 토포이소머라아제 I (대장균) | 타입 IA | 슈퍼코일(+)을 제거합니다.게놈의 과도한 슈퍼코일을 방지하고 전사를 지원합니다. | 단량체 | 있음2+(Mg) | 아니요. | SS | ||
| 토포이소머라아제II (대장균) | (+) 슈퍼코일을 제거하며, 토포이소머라아제 IV와 겹치는 기능을 합니다. | |||||||
| 토포이소머라아제IIα (H. 사피엔스) | 슈퍼코일(+)을 제거한다.세포 DNA 복제에서 프리카테논의 언링크에 도움이 된다.단일 가닥 원의 매듭, 언노트, 인터링크는 물론 갭 또는 니킹된 이중 DNA 원의 매듭, 언노트, 디커넥션을 촉매할 수 있다. | |||||||
| 토포이소머라아제IIβ (H. 사피엔스) | 추정 RNA 토포이소머라아제인 것으로 나타났다.RNA 처리에 관여하다 | |||||||
| 역회전 (아카이아) | (+) 슈퍼코일을 제거하지만 (+) 슈퍼코일을 제거하여 포지티브 슈퍼코일을 도입합니다. | 단량체 및 헤테로다이머 | 네. | |||||
| 토포이소머라아제 I (H. 사피엔스) | 타입 IB | (+) 및 (-) 슈퍼코일 제거, 복제 및 전사 시 포크 이동 지원 | 단량체 | 아니요. | 아니요. | SS | 3' | ±n |
| 토포이소머라아제 I (바시니아 바이러스) | ||||||||
| 토포이소머라아제 V (아카이아) | 타입 IC | 슈퍼코일(+)과 슈퍼코일(-)을 완화합니다.DNA 복구에 관여하고 있습니다. | 단량체 | 아니요. | 아니요. | SS | 3' | ±n |
| 토포이소머라아제II(DNA 자이라아제) (대장균) | 타입 IIA | 슈퍼코일(-)을 생성합니다(이렇게 하는 것으로 알려진 유일한 토포이소머라아제) | 헤테로테트라머 | 있음2+(Mg) | 네. | DS | 5' | ±2 |
| 토포이소머라아제 IV (대장균) | 복제된 DNA를 제거하여 (-)보다 (+) 슈퍼코일을 빠르게 완화 | 헤테로테트라머 | ||||||
| 토포이소머라아제IIα (H. 사피엔스) | 필수, 복제 시 서로 얽힌 도터 듀플렉스의 연결을 해제하고 전사 중 DNA 이완에 기여 | 호모디머 | ||||||
| 토포이소머라아제IIβ (H. 사피엔스) | 뉴런의 재조합을 억제하거나 전사를 지원하는 역할 | 호모디머 | ||||||
| 토포이소머라아제 VI (아카이아) | 타입 IIB | (+) 슈퍼코일 및 (-) 슈퍼코일 완화, 레플리케이션 중간 분리 담당 | 헤테로테트라머 | 있음2+(Mg) | 네. | DS | 5' | ±2 |
전사조절에서의 토포이소머라아제 역할
적어도 하나의 토포이소머라아제인 DNA토포이소머라아제II 베타(토포Iβ)는 유전자 전사에서 조절 역할을 한다.Topo IIβ 의존성 이중 가닥 DNA 파괴와 DNA 손상 복구 기계의 구성요소는 신호 반응 유전자 [52][53][54][55]조절뿐만 아니라 즉각적인 초기 유전자의 신속한 발현에 중요하다.Topo IIβ는 다른 관련 [54]효소와 함께 고도로 전사되거나 긴 [56][57][58]유전자에서 일시 정지된 RNA 중합효소의 방출에 중요한 것으로 보인다.
전사 개시 시 Topo IIβ
신호조절유전자의 프로모터 영역에서 단기(10분~2시간)로 제한된 자극유도 DNA 이중사슬절단(DSB)은 토포IIβ에 의해 유도된다.이러한 DSB는 다수의 시스템에서 그러한 신호 반응성 유전자의 발현을 빠르게 상향 조절할 수 있게 한다(아래 표 참조).이러한 신호 조절 유전자는 에스트로겐, 혈청, 인슐린, 글루코콜티코이드(덱사메타손 등) 및 뉴런 활성화에 반응하여 활성화된 유전자를 포함한다.유도된 DNA 이중 가닥 파단이 복구되면, 신호 반응 유전자의 전사는 낮은 기저 [52]수준으로 돌아간다.

Topo IIβ 및 PARP-1은 신호응답성 유전자의 프로모터의 전사개시부위 부근에 적당한 수준으로 존재하는 것을 발견했다.신호 발생 후 Topo IIβ는 이중 스트랜드 절단을 일으켰고 PARP-1은 [55]전사를 촉진할 수 있는 히스톤 H1을 HMGB1/HMGA2로 대체하는 데 관여했다.Topo IIβ 및 PARP-1은 Topo IIβ 및 PARP-1로 조립된 DNA-PKCS, Ku70/Ku80 및 DNA 리가아제 IV를 포함한 비상동 단부 결합 DNA 복구 경로의 성분 및 이중 가닥 절단 부위에서 증가했다.이 조합은 모두 유전자의 프로모터 영역에서 단일 뉴클레오솜에 인접한 링커 DNA에 있었다(그림 참조).뉴클레오솜은 [55]유전자의 전사 시작 부위에 가까웠다.비상동 말단 결합 DNA 복구 경로의 구성 요소는 DNA 이중 가닥 파괴의 [52]폐쇄에 필수적이었다.
| 배위자 또는 활성화제 | DSB에 대해 평가된 유전자 | 신호유기활성화후DSB | 문자 변환에 DSB가 필요했습니다. | DSB 위치 | DSB에 존재하는 단백질 | 신호 유도 활성화 후 DSB 지속 시간 | 참조 |
|---|---|---|---|---|---|---|---|
| 에스트라디올 | ps2 | 네. | 네. | 발기인 | topo IIβ, PARP-1, DNA-PKCS, Ku70, Ku80 | 10분 | [59] |
| 인슐린 | FASN | 네. | 네. | 발기인 | topo IIβ, PARP-1, DNA-PKcs, Ku70, Ku80, 단백질인산가수분해효소1(PP1), P/CAF | 3시간 | [60] |
| 열충격 또는 혈청 | HSPA1B, JUN, FOS, EGR1, MYC | 네. | 네. | 프로모터 & POLII 일시정지 사이트 | DNA-PKCS, Ku70, δH2AX, TRIM28 | 30초~5분 | [61] |
| 덱사메타손 또는 에스트라디올 | PS2, MMTV, PLZF, HSD11B2 | 네. | 네. | 발기인 | topo IIβ, PARP-1, DNA-PKcs, Ku70, Ku80, BRG1 | 15분 | [62] |
| 배양된 1차 피질 뉴런의 KCl 또는 NMDA 활성화 | FOS, EGR1, NPAS4, NR4A1 | 네. | 네. | 발기인 | topo IIβ, PARP-1, DNA-PKcs, Ku70, Ku80, CTCF | 최대 2시간 | [52] |
| 공포 조절(쥐 해마 및 내측 전치피질 뉴런에서 평가) | 새로운 DSB와 상향 조절 발현을 가진 200개 이상의 유전자 | 네. | DSB는 문자 변환과 관련되어 있습니다. | 발기인 | 테스트되지 않았다 | 10분 후 30분에 두 번째 피크 | [63] |
유전자 발현 Topo IIβ 조절
RNA 중합효소 II는 유전자의 [64][65]전사 시작 부위의 하류에 약 30-60 뉴클레오티드인 일시정지 부위가 있는 경우가 많다.이러한 부위에서 RNA 중합효소 II의 일시정지 및 일시정지 방출의 제어는 유전자 전사에 조절 역할을 하는 것으로 생각됩니다.싱 외 [58]연구진이 지적한 바와 같이, "헬라 세포에서 고도로 발현된 유전자의 약 80%가 일시 정지된다."토포 IIβ 유도 DNA 이중사슬 파손은 RNA 중합효소 II의 일시정지 부위에서 발생하며, 정지 상태의 효율적인 방출과 유전자 [56][57][58]전사로의 진행에 필요한 것으로 보인다.그것이 발생하는 유전자의 경우 TOP2B에 의해 유도되는 DNA 이중사슬 파괴는 유전자 발현 조절 과정의 일부로 생각된다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b McKie SJ, Neuman KC, Maxwell A (April 2021). "DNA topoisomerases: Advances in understanding of cellular roles and multi-protein complexes via structure-function analysis". BioEssays. 43 (4): e2000286. doi:10.1002/bies.202000286. PMID 33480441. S2CID 231679533.
- ^ Sutormin DA, Galivondzhyan AK, Polkhovskiy AV, Kamalyan SO, Severinov KV, Dubiley SA (2021-03-15). "Diversity and Functions of Type II Topoisomerases". Acta Naturae. 13 (1): 59–75. doi:10.32607/actanaturae.11058. PMC 8084294. PMID 33959387.
- ^ Wang JC (February 1971). "Interaction between DNA and an Escherichia coli protein omega". Journal of Molecular Biology. 55 (3): 523–533. doi:10.1016/0022-2836(71)90334-2. PMID 4927945.
- ^ Champoux JJ, Dulbecco R (January 1972). "An activity from mammalian cells that untwists superhelical DNA--a possible swivel for DNA replication (polyoma-ethidium bromide-mouse-embryo cells-dye binding assay)". Proceedings of the National Academy of Sciences of the United States of America. 69 (1): 143–146. doi:10.1073/pnas.69.1.143. PMC 427563. PMID 4333036.
- ^ Gellert M, Mizuuchi K, O'Dea MH, Nash HA (November 1976). "DNA gyrase: an enzyme that introduces superhelical turns into DNA". Proceedings of the National Academy of Sciences of the United States of America. 73 (11): 3872–3876. Bibcode:1976PNAS...73.3872G. doi:10.1073/pnas.73.11.3872. PMC 431247. PMID 186775.
- ^ Sugino A, Peebles CL, Kreuzer KN, Cozzarelli NR (November 1977). "Mechanism of action of nalidixic acid: purification of Escherichia coli nalA gene product and its relationship to DNA gyrase and a novel nicking-closing enzyme". Proceedings of the National Academy of Sciences of the United States of America. 74 (11): 4767–4771. Bibcode:1977PNAS...74.4767S. doi:10.1073/pnas.74.11.4767. PMC 432036. PMID 200930.
- ^ Baldi MI, Benedetti P, Mattoccia E, Tocchini-Valentini GP (June 1980). "In vitro catenation and decatenation of DNA and a novel eucaryotic ATP-dependent topoisomerase". Cell. 20 (2): 461–467. doi:10.1016/0092-8674(80)90632-7. PMID 6248247. S2CID 42645648.
- ^ Liu LF, Liu CC, Alberts BM (October 1979). "T4 DNA topoisomerase: a new ATP-dependent enzyme essential for initiation of T4 bacteriophage DNA replication". Nature. 281 (5731): 456–461. Bibcode:1979Natur.281..456L. doi:10.1038/281456a0. PMID 226889. S2CID 4343962.
- ^ Stetler GL, King GJ, Huang WM (August 1979). "T4 DNA-delay proteins, required for specific DNA replication, form a complex that has ATP-dependent DNA topoisomerase activity". Proceedings of the National Academy of Sciences of the United States of America. 76 (8): 3737–3741. Bibcode:1979PNAS...76.3737S. doi:10.1073/pnas.76.8.3737. PMC 383908. PMID 226976.
- ^ a b c d Bates AD (2005). DNA topology. Anthony Maxwell (2nd ed.). Oxford: Oxford University Press. ISBN 978-0-19-154658-7. OCLC 64239232.
- ^ Watson JD, Crick FH (May 1953). "Genetical implications of the structure of deoxyribonucleic acid". Nature. 171 (4361): 964–967. Bibcode:1953Natur.171..964W. doi:10.1038/171964b0. PMID 13063483. S2CID 4256010.
- ^ Watson JD, Crick FH (April 1953). "Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid". Nature. 171 (4356): 737–738. Bibcode:1953Natur.171..737W. doi:10.1038/171737a0. PMID 13054692. S2CID 4253007.
- ^ Franklin RE, Gosling RG (July 1953). "Evidence for 2-chain helix in crystalline structure of sodium deoxyribonucleate". Nature. 172 (4369): 156–157. Bibcode:1953Natur.172..156F. doi:10.1038/172156a0. PMID 13072614. S2CID 4169572.
- ^ Cairns J (March 1963). "The bacterial chromosome and its manner of replication as seen by autoradiography". Journal of Molecular Biology. Elsevier. 6 (3): 208–213. doi:10.1016/s0022-2836(63)80070-4. PMID 14017761.
- ^ Delbrück M (September 1954). "On the Replication of Desoxyribonucleic Acid (Dna)". Proceedings of the National Academy of Sciences of the United States of America. 40 (9): 783–788. Bibcode:1954PNAS...40..783D. doi:10.1073/pnas.40.9.783. PMC 534166. PMID 16589559.
- ^ Călugăreanu G (1959). "L'intégrale de Gauss et l'analyse des nœuds tridimensionnels". Revue de Mathématiques Pure et Appliquées. 4: 5–20.
- ^ Fuller FB (April 1971). "The writhing number of a space curve". Proceedings of the National Academy of Sciences of the United States of America. 68 (4): 815–819. Bibcode:1971PNAS...68..815B. doi:10.1073/pnas.68.4.815. PMC 389050. PMID 5279522.
- ^ White JH (1969). "Self-Linking and the Gauss Integral in Higher Dimensions". American Journal of Mathematics. 91 (3): 693–728. doi:10.2307/2373348. ISSN 0002-9327. JSTOR 2373348.
- ^ Postow L, Crisona NJ, Peter BJ, Hardy CD, Cozzarelli NR (July 2001). "Topological challenges to DNA replication: conformations at the fork". Proceedings of the National Academy of Sciences of the United States of America. 98 (15): 8219–8226. Bibcode:2001PNAS...98.8219P. doi:10.1073/pnas.111006998. PMC 37424. PMID 11459956.
- ^ Sundin O, Varshavsky A (September 1981). "Arrest of segregation leads to accumulation of highly intertwined catenated dimers: dissection of the final stages of SV40 DNA replication". Cell. 25 (3): 659–669. doi:10.1016/0092-8674(81)90173-2. PMID 6269752. S2CID 24408315.
- ^ Liu LF, Wang JC (October 1987). "Supercoiling of the DNA template during transcription". Proceedings of the National Academy of Sciences of the United States of America. 84 (20): 7024–7027. Bibcode:1987PNAS...84.7024L. doi:10.1073/pnas.84.20.7024. PMC 299221. PMID 2823250.
- ^ Kraemer JA, Sanderlin AG, Laub MT (July 2019). "The Stringent Response Inhibits DNA Replication Initiation in E. coli by Modulating Supercoiling of oriC". mBio. 10 (4). doi:10.1128/mbio.01330-19. PMC 6606810. PMID 31266875.
- ^ von Freiesleben U, Rasmussen KV (September 1992). "The level of supercoiling affects the regulation of DNA replication in Escherichia coli". Research in Microbiology. 143 (7): 655–663. doi:10.1016/0923-2508(92)90060-2. PMID 1488550.
- ^ a b Kikuchi A, Asai K (1984). "Reverse gyrase--a topoisomerase which introduces positive superhelical turns into DNA". Nature. 309 (5970): 677–681. Bibcode:1984Natur.309..677K. doi:10.1038/309677a0. PMID 6328327. S2CID 4242694.
- ^ Liu LF, Liu CC, Alberts BM (March 1980). "Type II DNA topoisomerases: enzymes that can unknot a topologically knotted DNA molecule via a reversible double-strand break". Cell. 19 (3): 697–707. doi:10.1016/s0092-8674(80)80046-8. PMID 6244895. S2CID 8921868.
- ^ Brochier-Armanet C, Forterre P (May 2007). "Widespread distribution of archaeal reverse gyrase in thermophilic bacteria suggests a complex history of vertical inheritance and lateral gene transfers". Archaea. 2 (2): 83–93. doi:10.1155/2006/582916. PMC 2686386. PMID 17350929.
- ^ a b Stewart L, Redinbo MR, Qiu X, Hol WG, Champoux JJ (March 1998). "A model for the mechanism of human topoisomerase I". Science. 279 (5356): 1534–1541. Bibcode:1998Sci...279.1534S. doi:10.1126/science.279.5356.1534. PMID 9488652.
- ^ Stivers JT, Harris TK, Mildvan AS (April 1997). "Vaccinia DNA topoisomerase I: evidence supporting a free rotation mechanism for DNA supercoil relaxation". Biochemistry. 36 (17): 5212–5222. doi:10.1021/bi962880t. PMID 9136883.
- ^ Baker NM, Rajan R, Mondragón A (February 2009). "Structural studies of type I topoisomerases". Nucleic Acids Research. 37 (3): 693–701. doi:10.1093/nar/gkn1009. PMC 2647283. PMID 19106140.
- ^ Bates AD, Berger JM, Maxwell A (August 2011). "The ancestral role of ATP hydrolysis in type II topoisomerases: prevention of DNA double-strand breaks". Nucleic Acids Research. 39 (15): 6327–6339. doi:10.1093/nar/gkr258. PMC 3159449. PMID 21525132.
- ^ McKie SJ, Desai PR, Seol Y, Allen AM, Maxwell A, Neuman KC (January 2022). "Topoisomerase VI is a chirally-selective, preferential DNA decatenase". eLife. 11. doi:10.7554/eLife.67021. PMC 8837201. PMID 35076393.
- ^ a b Pommier Y (January 2013). "Drugging topoisomerases: lessons and challenges". ACS Chemical Biology. 8 (1): 82–95. doi:10.1021/cb300648v. PMC 3549721. PMID 23259582.
- ^ a b c d e f Pommier Y, Leo E, Zhang H, Marchand C (May 2010). "DNA topoisomerases and their poisoning by anticancer and antibacterial drugs". Chemistry & Biology. 17 (5): 421–433. doi:10.1016/j.chembiol.2010.04.012. PMC 7316379. PMID 20534341.
- ^ a b Pommier Y, Marchand C (December 2011). "Interfacial inhibitors: targeting macromolecular complexes". Nature Reviews. Drug Discovery. 11 (1): 25–36. doi:10.1038/nrd3404. PMC 7380715. PMID 22173432.
- ^ Tse-Dinh YC (2015). "Targeting bacterial topoisomerase I to meet the challenge of finding new antibiotics". Future Medicinal Chemistry. 7 (4): 459–471. doi:10.4155/fmc.14.157. PMC 4415981. PMID 25875873.
- ^ a b Bush NG, Diez-Santos I, Abbott LR, Maxwell A (December 2020). "Quinolones: Mechanism, Lethality and Their Contributions to Antibiotic Resistance". Molecules. 25 (23): 5662. doi:10.3390/molecules25235662. PMC 7730664. PMID 33271787.
- ^ a b Maxwell A, Bush NG, Germe T, McKie SJ (2018). "Non-quinolone topoisomerase inhibitors". In Fong IW, Drlica K (eds.). Antimicrobial resistance and implications for the twenty-first century. New York: Springer. pp. 593–618. ISBN 978-0-387-72417-1. OCLC 227210110.
- ^ Collin F, Maxwell A (August 2019). "The Microbial Toxin Microcin B17: Prospects for the Development of New Antibacterial Agents". Journal of Molecular Biology. 431 (18): 3400–3426. doi:10.1016/j.jmb.2019.05.050. PMC 6722960. PMID 31181289.
- ^ Jiang Y, Pogliano J, Helinski DR, Konieczny I (May 2002). "ParE toxin encoded by the broad-host-range plasmid RK2 is an inhibitor of Escherichia coli gyrase". Molecular Microbiology. 44 (4): 971–979. doi:10.1046/j.1365-2958.2002.02921.x. PMID 12010492. S2CID 40019620.
- ^ Smith AB, Maxwell A (2006). "A strand-passage conformation of DNA gyrase is required to allow the bacterial toxin, CcdB, to access its binding site". Nucleic Acids Research. 34 (17): 4667–4676. doi:10.1093/nar/gkl636. PMC 1635281. PMID 16963775.
- ^ Vos SM, Lyubimov AY, Hershey DM, Schoeffler AJ, Sengupta S, Nagaraja V, Berger JM (July 2014). "Direct control of type IIA topoisomerase activity by a chromosomally encoded regulatory protein". Genes & Development. 28 (13): 1485–1497. doi:10.1101/gad.241984.114. PMC 4083091. PMID 24990966.
- ^ Feng L, Mundy JE, Stevenson CE, Mitchenall LA, Lawson DM, Mi K, Maxwell A (March 2021). "The pentapeptide-repeat protein, MfpA, interacts with mycobacterial DNA gyrase as a DNA T-segment mimic". Proceedings of the National Academy of Sciences of the United States of America. 118 (11). doi:10.1073/pnas.2016705118. PMC 7980463. PMID 33836580.
- ^ Mazurek Ł, Ghilarov D, Michalczyk E, Pakosz Z, Metelev M, Czyszczoń W, et al. (February 2021). "Pentapeptide repeat protein QnrB1 requires ATP hydrolysis to rejuvenate poisoned gyrase complexes". Nucleic Acids Research. 49 (3): 1581–1596. doi:10.1093/nar/gkaa1266. PMC 7897471. PMID 33434265.
- ^ Nelson EM, Tewey KM, Liu LF (March 1984). "Mechanism of antitumor drug action: poisoning of mammalian DNA topoisomerase II on DNA by 4'-(9-acridinylamino)-methanesulfon-m-anisidide". Proceedings of the National Academy of Sciences of the United States of America. 81 (5): 1361–1365. Bibcode:1984PNAS...81.1361N. doi:10.1073/pnas.81.5.1361. PMC 344833. PMID 6324188.
- ^ Pommier Y, Tanizawa A, Kohn KW (1994). "Mechanisms of topoisomerase I inhibition by anticancer drugs". DNA Topoisomerases: Topoisomerase-Targeting Drugs. Advances in Pharmacology. Vol. 29B. Elsevier. pp. 73–92. doi:10.1016/s1054-3589(08)61132-1. ISBN 978-0-12-032930-4. PMID 8996602.
- ^ a b c Vann KR, Oviatt AA, Osheroff N (June 2021). "Topoisomerase II Poisons: Converting Essential Enzymes into Molecular Scissors". Biochemistry. 60 (21): 1630–1641. doi:10.1021/acs.biochem.1c00240. PMC 8209676. PMID 34008964.
- ^ a b c d Murphy MB, Mercer SL, Deweese JE (January 2017). "Inhibitors and Poisons of Mammalian Type II Topoisomerases". Advances in Molecular Toxicology. Vol. 11. Elsevier. pp. 203–240. doi:10.1016/b978-0-12-812522-9.00005-1. ISBN 9780128125229.
- ^ a b Hande KR (October 1998). "Clinical applications of anticancer drugs targeted to topoisomerase II". Biochimica et Biophysica Acta (BBA) - Gene Structure and Expression. 1400 (1–3): 173–184. doi:10.1016/s0167-4781(98)00134-1. PMID 9748560.
- ^ Pommier Y, Cushman M (May 2009). "The indenoisoquinoline noncamptothecin topoisomerase I inhibitors: update and perspectives". Molecular Cancer Therapeutics. 8 (5): 1008–1014. doi:10.1158/1535-7163.mct-08-0706. PMC 2888777. PMID 19383846.
- ^ Speyer JL, Green MD, Kramer E, Rey M, Sanger J, Ward C, et al. (September 1988). "Protective effect of the bispiperazinedione ICRF-187 against doxorubicin-induced cardiac toxicity in women with advanced breast cancer". The New England Journal of Medicine. 319 (12): 745–752. doi:10.1056/nejm198809223191203. PMID 3137469.
- ^ Cvetković RS, Scott LJ (2005). "Dexrazoxane: a review of its use for cardioprotection during anthracycline chemotherapy". Drugs. 65 (7): 1005–1024. doi:10.2165/00003495-200565070-00008. PMID 15892593.
- ^ a b c d Madabhushi R, Kim TK (March 2018). "Emerging themes in neuronal activity-dependent gene expression". Mol Cell Neurosci. 87: 27–34. doi:10.1016/j.mcn.2017.11.009. PMC 5894330. PMID 29254824.
- ^ Bunch H, Jeong J, Kang K, Jo DS, Cong AT, Kim D, Kim D, Cho DH, Lee YM, Chen BP, Schellenberg MJ, Calderwood SK (October 2021). "BRCA1-BARD1 regulates transcription through modulating topoisomerase IIβ". Open Biol. 11 (10): 210221. doi:10.1098/rsob.210221. PMC 8492178. PMID 34610268.
- ^ a b Austin CA, Cowell IG, Khazeem MM, Lok D, Ng HT (December 2021). "TOP2B's contributions to transcription". Biochem Soc Trans. 49 (6): 2483–2493. doi:10.1042/BST20200454. PMID 34747992.
- ^ a b c Ju BG, Lunyak VV, Perissi V, Garcia-Bassets I, Rose DW, Glass CK, Rosenfeld MG (June 2006). "A topoisomerase II beta-mediated dsDNA break required for regulated transcription". Science. 312 (5781): 1798–802. doi:10.1126/science.1127196. PMID 16794079.
- ^ a b Dellino GI, Palluzzi F, Chiariello AM, Piccioni R, Bianco S, Furia L, De Conti G, Bouwman BA, Melloni G, Guido D, Giacò L, Luzi L, Cittaro D, Faretta M, Nicodemi M, Crosetto N, Pelicci PG (June 2019). "Release of paused RNA polymerase II at specific loci favors DNA double-strand-break formation and promotes cancer translocations". Nat Genet. 51 (6): 1011–1023. doi:10.1038/s41588-019-0421-z. PMID 31110352.
- ^ a b Gittens WH, Johnson DJ, Allison RM, Cooper TJ, Thomas H, Neale MJ (October 2019). "A nucleotide resolution map of Top2-linked DNA breaks in the yeast and human genome". Nat Commun. 10 (1): 4846. doi:10.1038/s41467-019-12802-5. PMC 6813358. PMID 31649282.
- ^ a b c Singh S, Szlachta K, Manukyan A, Raimer HM, Dinda M, Bekiranov S, Wang YH (March 2020). "Pausing sites of RNA polymerase II on actively transcribed genes are enriched in DNA double-stranded breaks". J Biol Chem. 295 (12): 3990–4000. doi:10.1074/jbc.RA119.011665. PMC 7086017. PMID 32029477.
- ^ Ju BG, Lunyak VV, Perissi V, Garcia-Bassets I, Rose DW, Glass CK, Rosenfeld MG (June 2006). "A topoisomerase IIbeta-mediated dsDNA break required for regulated transcription". Science. 312 (5781): 1798–802. doi:10.1126/science.1127196. PMID 16794079.
- ^ Wong RH, Chang I, Hudak CS, Hyun S, Kwan HY, Sul HS (March 2009). "A role of DNA-PK for the metabolic gene regulation in response to insulin". Cell. 136 (6): 1056–72. doi:10.1016/j.cell.2008.12.040. PMC 2768498. PMID 19303849.
- ^ Bunch H, Lawney BP, Lin YF, Asaithamby A, Murshid A, Wang YE, Chen BP, Calderwood SK (December 2015). "Transcriptional elongation requires DNA break-induced signalling". Nat Commun. 6: 10191. doi:10.1038/ncomms10191. PMC 4703865. PMID 26671524.
- ^ Trotter KW, King HA, Archer TK (August 2015). "Glucocorticoid Receptor Transcriptional Activation via the BRG1-Dependent Recruitment of TOP2β and Ku70/86". Mol Cell Biol. 35 (16): 2799–817. doi:10.1128/MCB.00230-15. PMC 4508321. PMID 26055322.
- ^ Stott RT, Kritsky O, Tsai LH (2021). "Profiling DNA break sites and transcriptional changes in response to contextual fear learning". PLoS One. 16 (7): e0249691. doi:10.1371/journal.pone.0249691. PMC 8248687. PMID 34197463.
- ^ Dollinger R, Gilmour DS (July 2021). "Regulation of Promoter Proximal Pausing of RNA Polymerase II in Metazoans". J Mol Biol. 433 (14): 166897. doi:10.1016/j.jmb.2021.166897. PMID 33640324.
- ^ Price DH (May 2018). "Transient pausing by RNA polymerase II". Proc Natl Acad Sci U S A. 115 (19): 4810–4812. doi:10.1073/pnas.1805129115. PMC 5949015. PMID 29691322.
추가 정보
- Wang JC (2009). Untangling the Double Helix: DNA entanglement and the action of the DNA topoisomerases. Cold Spring Harbor: Cold Spring Harbor Laboratory Press. p. 245. ISBN 978-0-87969-879-9.
외부 링크
- 미국 국립의학도서관의 DNA+토포이소머라아제(MeSH)





