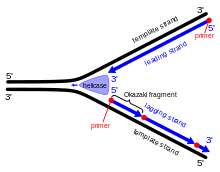
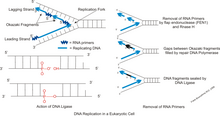
오카자키 조각
Okazaki fragments오카자키 조각은 DNA 뉴클레오티드의 짧은 배열(진핵생물에서는 약 150에서 200개의 염기쌍)로, 불연속적으로 합성되고 나중에 DNA 연결 효소에 의해 결합되어 DNA 복제 [1]중에 지연된 가닥을 생성한다.그것들은 1960년대에 일본의 분자생물학자 레이지와 쓰네코 오카자키에 의해 동료들의 도움으로 발견되었다.
DNA 복제 중에 이중 나선은 풀리고 상보적인 가닥은 효소 DNA 헬리카제에 의해 분리되어 DNA 복제 포크라고 알려진 것을 생성한다.이 포크에 이어, DNA 프리마아제와 DNA 중합효소가 새로운 상보적 가닥을 만들기 위해 작용하기 시작합니다.이들 효소는 5' ~ 3' 방향에서만 작동할 수 있기 때문에, 두 개의 풀린 템플릿 가닥은 다른 방식으로 [2]복제된다.하나의 가닥인 선두 가닥은 템플릿 가닥의 방향성이 3'에서 5'이므로 연속 복제 과정을 거치고 선두 가닥을 조립하는 중합효소가 중단 없이 복제 포크를 따를 수 있다.그러나 지연된 가닥은 템플릿 가닥의 방향성이 5' ~ 3'이므로 연속적으로 생성할 수 없으며, 이는 중합효소가 복제 포크에서 역방향으로 작용해야 함을 의미합니다.이로 인해 지연된 가닥을 생성하는 과정에서 주기적으로 중단이 발생합니다.프라이머라아제와 중합효소는 포크의 반대 방향으로 움직이기 때문에 효소는 DNA 헬리케이스가 가닥을 끊는 동안 멈추었다가 다시 시작해야 합니다.일단 조각이 만들어지면, DNA 연결효소는 그것들을 하나의 연속된 [3]가닥으로 연결한다.새로운 가닥 중 하나는 연속적으로 형성되고 다른 하나는 [4]연속적으로 형성되지 않기 때문에 전체 복제 프로세스는 "반연속적"으로 간주됩니다.
[2] 1960년대에 레이지 씨와 오카자키 쓰네코 씨는 대장균의 DNA 복제를 포함한 실험을 실시했습니다.이 시기 이전에는 복제는 두 가닥의 연속적인 과정이라고 일반적으로 생각되었지만 대장균과 관련된 발견은 새로운 복제 모델로 이어졌다.과학자들은 DNA의 맥박 라벨을 붙이고 비연속 복제를 가리키는 변화를 관찰함으로써 불연속적인 복제 과정이 있다는 것을 발견했다.
실험
사카베 기와코, 오카자키 레이지, 오카자키 쓰네코의 연구는 DNA 복제가 불연속 과정이라는 가설을 뒷받침하는 실험 증거를 제공했다.기존에는 복제는 3'에서 5', 5'에서 3'의 방향 모두 연속적인 것으로 일반적으로 받아들여졌습니다.3'와 5'는 핵산 중의 디옥시리보스 고리 위에 특이적으로 번호가 매겨진 탄소로, 가닥의 방향성 또는 방향을 말한다.1967년 오카자키 쓰네코와 오가와 도루는 복제효소인 DNA 중합효소를 이용해 3~5' 방향으로 연속 복제를 보인 메커니즘은 발견되지 않았다고 주장했다.연구팀은 불연속 복제를 사용하면 복제 지점에서 합성된 짧은 DNA 가닥이 오래된 [5]가닥에 5'3' 방향으로 부착될 수 있다는 가설을 세웠다.
연구팀은 DNA가 실험적으로 사용하는 복제 방법을 구별하기 위해 대장균 염색체의 새로 복제된 영역을 펄스 라벨링하고 변성시킨 후 DNA를 추출했다.많은 수의 방사성 쇼트 유닛은 복제 방법이 중단적일 가능성이 높다는 것을 의미했습니다.이 가설은 [6]짧은 DNA 가닥을 연결하는 효소인 폴리뉴클레오티드 연결효소의 발견으로 더욱 뒷받침되었다.
1968년 레이지와 오카자키 쓰네코는 초기 DNA 가닥의 추가 증거를 수집했다.그들은 만약 폴리뉴클레오티드 연결효소에 의해 함께 연결된 짧은 DNA 사슬을 포함하는 불연속적인 복제가 DNA 합성에 사용되는 메커니즘이라면, "새로운 합성된 짧은 DNA 사슬은 연결효소의 기능이 일시적으로 손상된 조건에서 세포에 축적될 것"이라고 가설을 세웠다. 대장균은 박테리오파지 T4 t에 감염되었다.모자는 온도에 민감한 폴리뉴클레오티드 연결효소를 생성한다.T4 파지에 감염된 세포들은 높은 온도에 노출되었을 때 가설에서 예측한 바와 같이 짧은, 새롭게 합성된 DNA 사슬을 많이 축적했다.이 실험은 폴리뉴클레오티드 연결효소에 의한 불연속 복제와 결합에 대한 Okazakis의 가설을 더욱 뒷받침했다.그것은 또한 [7]추출 과정에서 짧은 체인이 생산되었다는 개념을 반증했다.
오카자키 부부의 실험은 DNA의 복제 과정과 나중에 오카자키 조각으로 알려지게 된 짧고 새롭게 합성된 DNA 사슬의 존재에 대한 광범위한 정보를 제공했다.
패스
오카자키 단편을 처리하기 위해 짧은 플랩 경로와 긴 플랩 경로의 두 가지 경로가 제안되었다.
짧은 플랩 경로
진핵생물에서의 짧은 플랩 경로에서 지연된 DNA 가닥은 짧은 간격으로 프라이밍된다.짧은 경로에서만 핵산가수분해효소 FEN1이 관여한다.Pol δ는 하류 Okazaki 단편을 자주 만나 RNA/DNA 이니시에이터 프라이머를 5' 플랩으로 치환한다.FEN1 5'-3' 엔도핵산가수분해효소는 5' 플랩이 변위된 것을 인식하여 결찰용 기질을 형성한다.이 방법에서는 Pol a-합성 프라이머를 제거합니다.연구에[which?] 따르면 FEN1에서는 핵산가수분해효소가 5' 플랩에서 기저부로 이동하여 분할 전 형태인 '추적; 모델'을 제안한다.Pol does은 치환된 플랩을 절단하기 위해 핵산가수분해효소 활성을 처리하지 않는다.FEN1은 형성 직후 짧은 플랩을 절단합니다.상보적 프라이머 또는 비오틴 결합 스트렙타비딘 부분 중 하나로 DNA 플랩의 5' 끝을 차단할 때 분할이 억제된다.DNA 연결효소는 FEN1에 의해 만들어진 틈을 봉합하고 그것은 기능적인 연속적인 이중 가닥의 DNA를 생성한다. PCNA는 FEN1과 DNA 연결효소에 대한 단백질의 효소적 기능을 시뮬레이션한다.이 상호작용은 지연된 DNA 가닥의 적절한 결속을 만드는 데 중요하다.순차적 가닥 치환 및 Pol δ 및 FEN1에 의한 분할은 결찰 전에 전체 이니시에이터 RNA를 제거하는 데 도움이 됩니다.이니시에이터 프라이머를 탈거하려면 많은 변위가 필요하며 분할 반응이 필요합니다.플랩이 생성되고 처리되며 짧은 플랩 경로에 의해 숙성됩니다.
긴 플랩 경로
경우에 따라서는, FEN1은 단시간 밖에 지속되지 않고, 레플리케이션 콤플렉스에서 해방됩니다.이로 인해 분할이 지연되어 Pol δ에 의해 치환된 플랩이 길어진다.RPA가 충분한 길이에 도달하면 안정적으로 결합할 수 있습니다.RPA 결합 플랩이 FEN1로 재인자화되면 는 처리를 위해 또 다른 핵산 분해 효소를 필요로 하며, 이는 대체 핵산 분해 효소인 DNA2로 확인되었다.DNA2는 DEN1 과잉 발현에 결함이 있습니다.DNA2는 긴 플랩을 처리하기 위해 FEN1과 함께 작동하는 것을 보여주었다.DNA2는 긴 플랩에서 RPA를 분리할 수 있으며, 이것은 FEN1과 같은 메커니즘을 사용하여 이루어집니다.플랩을 묶고 플랩의 5' 끝을 꿰어 줍니다.핵산가수분해효소는 플랩을 절단하여 RPA에 결합하기엔 너무 짧으며 플랩이 너무 짧으면 FEN1 및 결찰에 사용할 수 있습니다.이를 롱 플랩 방식이라고 합니다.DNA2는 핵산가수분해효소 활성의 백업으로 FEN1로 작용할 수 있지만 효율적인 과정은 아니다.
대체 경로
최근까지 오카자키 파편을 처리하는 경로는 2개뿐이었다.그러나, 현재의 조사에서는, 오카자키 조각화와 DNA 복제를 위한 새로운 경로가 존재하는 것이 판명되었다.이 대체 경로에는 Pol δ 및 FEN1과 동일한 플랩 제거 과정을 수행하는 Pif1과 [8]Pol δ 효소가 포함된다.
단편 형성에 관여하는 효소
프라이마아제
프리마아제는 지연된 가닥에 RNA 프라이머를 첨가하여 오카자키 단편을 5'에서 3'까지 합성할 수 있다.그러나 프리마아제는 DNA 중합효소가 선두 가닥의 DNA를 합성하는 속도보다 훨씬 낮은 속도로 RNA 프라이머를 생성한다.또한 RNA 프라이머에 이어 오카자키 단편을 만들기 위해 지연된 가닥의 DNA 중합효소를 지속적으로 재활용해야 한다.이는 선행 가닥보다 지연 가닥 합성의 속도를 훨씬 느리게 만듭니다.이를 해결하기 위해 프리마아제는 일시적인 정지 신호로 작용하여 DNA 복제 중에 복제 포크의 진행을 잠시 중지합니다.이 분자 과정은 선두 가닥이 뒤처진 [9]가닥을 추월하는 것을 막습니다.
DNA중합효소γ
이 단계에서 5'~3' 방향으로 DNA를 합성하는 효소에 의해 새로운 DNA가 만들어진다.DNA 중합효소는 연속 가닥으로 만들어지는 선도 가닥과 DNA 합성으로 작은 조각으로 만들어지는 지연 가닥 모두에 필수적입니다.이 과정은 새로 합성된 조각의 확장과 RNA 및 DNA 세그먼트의 배출을 위해 발생합니다.합성은 DNA 중합효소α-프리마아제 및 DNA 중합효소γ의 두 가지 다른 중합효소와의 3상으로 이루어진다.이 과정은 클램프 로더 복제 효과에 의해 RNA 및 DNA 프라이머에서 중합효소α-프리마아제가 변위하는 것으로 시작되며, 이 효과는 슬라이딩 클램프를 DNA로 이끈다.이후 DNA 중합효소 δ가 홀로엔자임 형태로 들어가 합성이 시작된다.이전 오카자키 조각의 5'가 도착할 때까지 합성 과정이 계속됩니다.도착 후, 오카자키 단편 처리가 진행되어, 새롭게 합성된 단편과 후행 가닥이 결합됩니다.DNA 중합효소 δ의 마지막 기능은 FEN1/RAD27 5' 플랩핵산가수분해효소 활성의 보충제 역할을 하는 것이다.rad27-p 대립 유전자는 대부분의 조합에서 치명적이지만 rad27-p 중합효소 및 exo1과 함께 사용할 수 있었다.rad27-p 중합효소 및 exo1은 CAN 1 복제 돌연변이의 강한 상승효과를 나타낸다.이 돌연변이가 가능한 유일한 이유는 이중 가닥 절단 복구 유전자 RAD50, RAD51, RAD52 때문이다.RAD27/FEN1은 래깅된 스트랜드에서 스트랜드 분출량을 최소화하여 인접한 Okazaki 조각 사이에 흠집을 만듭니다.
DNA리가아제I
지연된 가닥 합성 중에 DNA 연결효소 I은 DNA 중합효소 δ에 의한 RNA 프라이머의 DNA 뉴클레오티드로 치환된 후 오카자키 단편을 연결한다.결속되지 않은 오카자키 조각은 [10]DNA를 절단하는 이중가닥을 일으킬 수 있다.소수의 이중 스트랜드 절단만이 허용되며, 수복할 수 있는 것은 극소수뿐이므로 충분한 결찰 실패는 셀에 치명적일 수 있습니다.
추가 연구는 오카자키 단편을 결합하는 DNA 연결효소 I의 기능에 대한 증식 세포핵항원(PCNA)의 보충적 역할을 포함한다.DNA 리가아제 I의 PCNA 결합 부위가 비활성화되면 오카자키 단편을 연결하는 DNA 리가아제 I의 능력이 심각하게 저하된다.따라서 PCNA-DNA 중합효소 δ복합체가 오카자키 단편을 합성한 후 DNA 중합효소 δ가 방출된다.그 후, DNA 리가아제 I은 PCNA에 결합하고, PCNA는 뒤떨어진 가닥의 틈새에 고정되며, 포스포디에스테르 [11][12][13]결합의 형성을 촉매한다.
플랩 핵산가수분해효소 1
플랩핵산가수분해효소 1(FEN1)은 오카자키 단편 처리를 담당한다.그것은 DNA 중합효소와 함께 작용하여 오카자키 단편의 RNA 프라이머를 제거하고 DNA 중합효소가 지연된 가닥 합성 중에 가닥을 치환할 때 5' 리보뉴클레오티드 및 5' 플랩을 제거할 수 있다.이러한 플랩의 제거에는 닉 변환이라고 불리는 프로세스가 포함되어 결찰에 대한 닉이 생성됩니다.따라서 긴 연속 DNA 가닥을 형성할 때 오카자키 단편의 성숙에 FEN1의 기능이 필요하다.마찬가지로 DNA 염기 복구 중에 손상된 뉴클레오티드는 플랩으로 치환되고 FEN1에 [14][15]의해 제거된다.
DNA2핵산가수분해효소
DNA2 엔도핵산가수분해효소는 특정 구조를 가지고 있지 않으며 그 특성은 잘 특성화되지 않았지만 자유단(sssDNA)을 가진 단일 가닥 DNA로 언급될 수 있다.DNA2 엔도핵산가수분해효소는 오카자키 공정에서 FEN1을 떠나는 긴 DNA 플랩을 분해하는 데 필수적이다.Dna2 엔도핵산가수분해효소는 Okazaki fragments에서 이니시에이터 RNA 세그먼트를 제거하는 역할을 합니다.또한 Dna2 엔도핵산가수분해효소는 다양한 DNA 대사 중에 생성되는 중간체에서 중추적인 역할을 하며 텔로미어 [16][17][18][19][20]유지에 기능한다.
DNA2 엔도핵산가수분해효소는 말단 RNA 세그먼트가 5'에서 3' 방향으로 전이되기 때문에 5' 끝에서 결합하면 활성화된다.단일 가닥이 있는 DNA결합단백질 RPA의 존재하에서는 DNA5'플랩이 너무 길어지고 닉이 FEN1의 기질로서 더 이상 적합하지 않게 된다.이것에 의해, FEN1이 5 인치 플랩을 떼어낼 수 없게 됩니다.따라서 Dna2의 역할은 이들 조각의 3⁄ 끝을 감소시켜 FEN1이 플랩을 절단하고 오카자키 조각의 성숙을 보다 효율적으로 하는 것이다.오카자키 공정에서는 Dna2 헬리케이스와 엔도핵산가수분해효소는 분리할 수 없다.Dna2 엔도핵산가수분해효소는 활성의 5' 꼬리 포크 구조에 의존하지 않는다.비생산적인 바인딩은 FEN1 분할 및 추적에 대한 블록을 생성하는 것으로 알려져 있습니다.ATP는 활성을 감소시키지만 3' 말단 라벨의 분비를 촉진하는 것으로 알려져 있다.연구에 따르면 Dna2 엔도핵산가수분해효소 및 FEN1의 새로운 모델이 오카자키 [19][17][16][21]단편 성숙에 부분적으로 관여하는 것으로 나타났다.
생물학적 기능
오카자키 단편으로 알려진 새로 합성된 DNA는 새로운 DNA 가닥을 형성하는 DNA 연결효소에 의해 결합된다.DNA를 합성할 때 생성되는 두 가닥이 있다.선행 가닥은 연속적으로 합성되어 이 과정에서 연장되어 지연 가닥(오카자키 조각)에 사용되는 템플릿이 노출된다.DNA 복제 과정에서 DNA와 RNA 프라이머를 DNA의 뒤떨어진 가닥에서 제거해 오카자키 단편을 결합시킨다.이 과정은 매우 일반적이기 때문에, 오카자키 숙성은 DNA 복제가 1회 완료되는 동안 약 100만회 정도 발생합니다.오카자키 성숙을 위해서는 RNA 프라이머가 결합할 단편에 세그먼트를 생성해야 합니다.이것은 지연된 가닥의 DNA 합성을 위한 구성 블록으로 사용됩니다.템플릿 가닥에서 중합효소는 복제 포크와 반대 방향으로 합성됩니다.템플릿이 중단되면 Okazaki fragment가 생성됩니다.오카자키 단편의 성숙 결함으로 DNA의 가닥이 끊어지고 다른 형태의 염색체 이상이 발생할 수 있다.염색체의 이러한 돌연변이는 외모, 세트의 수, 또는 개별 염색체의 수에 영향을 미칠 수 있습니다.염색체는 각각의 특정 종에 대해 고정되어 있기 때문에, 그것은 또한 DNA를 변화시키고 그 종의 유전자에 결함을 일으킬 수 있다.
원핵생물과 진핵생물의 차이
이 섹션에는 차이점인 고고학에 대한 정보가 누락되어 있습니다. PMID12612604, PMID258614667; 경로와 효소의 세균 차이(이전 부분은 진핵 효소에 대해서만 설명).(2022년 7월) |
오카자키 조각은 원핵생물과 [22]진핵생물에 모두 존재한다.진핵 생물의 DNA 분자는 더 크고 보통 복제의 여러 기원을 가지고 있다는 점에서 원핵 생물의 원형 분자와 다르다.이것은 각 진핵생물 염색체가 복제의 여러 기원을 가진 DNA의 많은 복제 단위로 구성된다는 것을 의미한다.그에 비해, 원핵 DNA는 복제의 기원이 하나밖에 없다.진핵생물에서, DNA를 따라 수없이 많은 이러한 복제 포크는 복제하는 동안 DNA에서 "거품"을 형성합니다.레플리케이션 포크는 Autonomous Replicating Sequence(ARS; 자율 복제 시퀀스)라고 불리는 특정 포인트로 형성됩니다.진핵생물들은 클램프 로더 복합체와 증식세포 [23]핵항원이라고 불리는 6단위 클램프를 가지고 있다.복제 포크의 효율적인 이동은 또한 ATP 의존성 클램프 로더 복합체에 의해 지연 DNA 가닥에 새로 준비된 부위에 슬라이딩 클램프의 신속한 배치에 매우 의존한다.즉, 오카자키 조각의 분할 생성이 선두 가닥의 DNA의 연속 합성에 보조를 맞출 수 있습니다.이러한 클램프 로더 복합체는 모든 진핵생물의 특징이며 원핵생물과 진핵생물의 [24]오카자키 조각 합성에 있어 약간의 차이를 분리한다.원핵생물과 진핵생물의 오카자키 조각의 길이도 다르다.원핵생물에는 진핵생물보다 상당히 긴 오카자키 조각이 있다.진핵생물들은 일반적으로 100에서 200개의 뉴클레오티드 길이의 오카자키 조각들을 가지고 있는 반면, 원핵생물 대장균의 조각들은 2,000개의 뉴클레오티드 길이일 수 있다.이 불일치의 원인은 불명확합니다.
각각의 진핵생물 염색체는 복제의 여러 기원을 가진 DNA의 많은 복제 단위로 구성되어 있다.이에 비해 원핵형 대장균 염색체는 복제의 기원이 하나밖에 없다.원핵생물에서의 복제는 세포질 안에서 일어나며, 이것은 모두 약 100에서 200개 이상의 뉴클레오티드로 이루어진 복제를 시작합니다.진핵생물 DNA 분자는 약 50,000개 이상의 상당히 많은 수의 복제자를 가지고 있다; 하지만, 복제는 모든 복제자에서 동시에 일어나지 않는다.진핵생물에서, DNA 복제는 핵에서 일어난다.단 하나의 복제 DNA 분자에 있는 복막 복제 형태에서 DNA 복제의 시작은 멀티 서브유닛 단백질에 의해 멀어진다.이 복제는 느리고, 때때로 초당 100개의 뉴클레오티드가 첨가된다.
우리는 원핵세포가 구조가 더 단순하고, 핵도 없고, 세포소기관도 없고, 단일 염색체의 형태로 DNA도 거의 없다는 것을 알 수 있다.진핵세포는 여러 개의 세포와 더 많은 DNA가 선형 염색체로 배열된 핵을 가지고 있다.우리는 또한 크기가 이러한 원핵세포와 진핵세포 사이의 또 다른 차이점임을 알 수 있다.평균적인 진핵 세포는 원핵 세포보다 약 25배 더 많은 DNA를 가지고 있다.복제는 진핵 세포보다 원핵 세포에서 훨씬 더 빨리 일어난다; 박테리아는 때때로 40분밖에 걸리지 않는 반면, 동물 세포는 400시간까지 걸릴 수 있다.진핵생물들은 또한 그들의 마지막 염색체 끝에 있는 텔로미어를 복제하기 위한 뚜렷한 작업을 가지고 있다.원핵생물들은 원형의 염색체를 가지고 있어 끝이 없는 합성을 일으킨다.원핵생물은 지속적으로 일어나는 짧은 복제 과정을 가지고 있다; 반면에 진핵생물은 세포 주기의 S 단계 동안에만 DNA 복제를 수행한다.
유사점은 DNA 복제를 위한 단계입니다.원핵생물과 진핵생물 모두에서 복제는 DNA 헬리케이스라고 불리는 효소에 의해 DNA를 풀어서 이루어진다.새로운 가닥들은 DNA 중합효소라고 불리는 효소에 의해 만들어진다.이 두 가지 모두 반보수적 복제라고 불리는 유사한 패턴을 따르는데, 각각의 DNA 가닥이 다른 방향으로 생산되고, 이것은 앞서고 뒤처지는 가닥을 만듭니다.이러한 낙후된 가닥은 곧 결합되는 오카자키 조각의 생성에 의해 합성된다.이 두 유기체 모두 작은 RNA 가닥을 포함하는 새로운 DNA 가닥을 시작합니다.
테크놀로지에서의 사용
오카자키 조각과 관련된 의학적 개념
세포는 유전자 배열에 돌연변이가 생기지 않도록 여러 단계를 거치지만, 오카자키 단편이 성숙하는 동안 특정 결손이나 다른 유전적 변화가 눈에 띄지 않을 수 있다.Okazaki fragments는 지연된 가닥에 대한 뉴클레오티드의 집합이기 때문에, 원래 가닥의 결실, 삽입 또는 복제를 포함한 어떠한 변화도 검출 및 고정되지 않으면 돌연변이를 일으킬 수 있다.돌연변이의 다른 원인으로는 DNA 복제를 돕는 단백질 문제가 있다.예를 들어 프리마아제와 관련된 돌연변이는 RNA 프라이머 제거에 영향을 미치며 DNA 가닥을 더 취약하고 파손되기 쉽게 만들 수 있다.또 다른 돌연변이는 오카자키 단편 배열의 편집과 유전자 물질로의 단백질의 통합을 저해하는 중합효소α에 관한 것이다.두 가지 변화 모두 염색체 이상, 의도하지 않은 유전자 재배열, 그리고 나중에 [25]다양한 암으로 이어질 수 있다.
살아있는 유기체에 대한 단백질 돌연변이의 영향을 테스트하기 위해, 연구원들은 DNA 복제와 관련된 또 다른 단백질 돌연변이인 플랩 핵산가수분해효소 1 또는 FEN1에 대해 호모 접합이 되도록 유전자 변형 실험 생쥐를 만들었다.결과는 특정 유전자 변화에 따라 달라졌다.호모 접합 녹아웃 돌연변이 생쥐는 "세포 증식의 실패"와 "조기 배아 사망률"(27)을 경험했다.돌연변이 F343A와 F344A(FFAA라고도 함)를 가진 쥐는 범세포감소증과 폐성형부전증을 포함한 출생 합병증으로 인해 출생 직후 사망했다.이는 FFAA 돌연변이가 FEN1이 PCNA(증식세포 핵항원)와 상호작용하는 것을 방해하여 결과적으로 오카자키 단편의 성숙 동안 그 목적을 완수할 수 없기 때문이다.이 단백질과의 상호작용은 FEN1의 생물학적 기능에서 핵심 분자 기능으로 간주된다.FFAA 돌연변이는 RNA 프라이머 제거 및 장기 염기쌍 복구의 결함을 유발하며, 이 중 DNA에 많은 손상을 일으킨다.주의 깊게 관찰한 결과, FFAA FEN1 돌연변이에 대해 호모 접합된 세포는 성숙에 부분적인 결함만을 보이는 것으로 보이며, 이는 돌연변이에 대해 헤테로 접합된 생쥐가 게놈에 여러 작은 흠집이 있음에도 불구하고 성인까지 생존할 수 있다는 것을 의미한다.그러나 필연적으로 이러한 상처는 미래의 DNA 복제를 방해합니다. 왜냐하면 이러한 상처는 복제 포크를 붕괴시키고 실제 DNA 염기서열에서 이중 가닥 절단을 일으키기 때문입니다.시간이 지나면서, 이러한 상처들은 또한 심각한 돌연변이와 암을 야기할 수 있는 완전한 염색체 손상을 야기한다.다른 돌연변이는 변경된 버전의 중합효소 α와 함께 구현되어 유사한 [25]결과를 초래했다.
레퍼런스
- ^ Balakrishnan L, Bambara RA (February 2013). "Okazaki fragment metabolism". Cold Spring Harbor Perspectives in Biology. 5 (2): a010173. doi:10.1101/cshperspect.a010173. PMC 3552508. PMID 23378587.
- ^ a b Okazaki T (2017-05-11). "Days weaving the lagging strand synthesis of DNA - A personal recollection of the discovery of Okazaki fragments and studies on discontinuous replication mechanism". Proceedings of the Japan Academy. Series B, Physical and Biological Sciences. 93 (5): 322–338. Bibcode:2017PJAB...93..322O. doi:10.2183/pjab.93.020. PMC 5489436. PMID 28496054.
- ^ Cooper, Geoffrey M (2000). "DNA Replication".
{{cite journal}}:Cite 저널 요구 사항journal=(도움말) - ^ MacNeill SA (October 2001). "DNA replication: partners in the Okazaki two-step". Current Biology. 11 (20): R842–4. doi:10.1016/s0960-9822(01)00500-0. PMID 11676941. S2CID 15853820.
- ^ Ogawa T, Okazaki T (1980). "Discontinuous DNA transcription". Annual Review of Biochemistry. 49: 421–57. doi:10.1146/annurev.bi.49.070180.002225. PMID 6250445.
- ^ Okazaki R, Okazaki T, Sakabe K, Sugimoto K, Sugino A (February 1968). "Mechanism of DNA chain growth. I. Possible discontinuity and unusual secondary structure of newly synthesized chains". Proceedings of the National Academy of Sciences of the United States of America. 59 (2): 598–605. Bibcode:1968PNAS...59..598O. doi:10.1073/pnas.59.2.598. PMC 224714. PMID 4967086.
- ^ Sugimoto K, Okazaki T, Okazaki R (August 1968). "Mechanism of DNA chain growth, II. Accumulation of newly synthesized short chains in E. coli infected with ligase-defective T4 phages". Proceedings of the National Academy of Sciences of the United States of America. 60 (4): 1356–62. Bibcode:1968PNAS...60.1356S. doi:10.1073/pnas.60.4.1356. PMC 224926. PMID 4299945.
- ^ Pike JE, Henry RA, Burgers PM, Campbell JL, Bambara RA (December 2010). "An alternative pathway for Okazaki fragment processing: resolution of fold-back flaps by Pif1 helicase". The Journal of Biological Chemistry. 285 (53): 41712–23. doi:10.1074/jbc.M110.146894. PMC 3009898. PMID 20959454.
- ^ Lee JB, Hite RK, Hamdan SM, Xie XS, Richardson CC, van Oijen AM (February 2006). "DNA primase acts as a molecular brake in DNA replication" (PDF). Nature. 439 (7076): 621–4. Bibcode:2006Natur.439..621L. doi:10.1038/nature04317. PMID 16452983. S2CID 3099842.
- ^ Soza, Samuela; Leva, Valentina; Vago, Riccardo; Ferrari, Giovanni; Mazzini, Giuliano; Biamonti, Giuseppe; Montecucco, Alessandra (April 2009). "DNA Ligase I Deficiency Leads to Replication-Dependent DNA Damage and Impacts Cell Morphology without Blocking Cell Cycle Progression". Molecular and Cellular Biology. 29 (8): 2032–2041. doi:10.1128/MCB.01730-08. PMC 2663296. PMID 19223467.
- ^ Jin YH, Ayyagari R, Resnick MA, Gordenin DA, Burgers PM (January 2003). "Okazaki fragment maturation in yeast. II. Cooperation between the polymerase and 3'-5'-exonuclease activities of Pol delta in the creation of a ligatable nick". The Journal of Biological Chemistry. 278 (3): 1626–33. doi:10.1074/jbc.M209803200. PMID 12424237.
- ^ Levin DS, Bai W, Yao N, O'Donnell M, Tomkinson AE (November 1997). "An interaction between DNA ligase I and proliferating cell nuclear antigen: implications for Okazaki fragment synthesis and joining". Proceedings of the National Academy of Sciences of the United States of America. 94 (24): 12863–8. Bibcode:1997PNAS...9412863L. doi:10.1073/pnas.94.24.12863. PMC 24229. PMID 9371766.
- ^ Levin DS, McKenna AE, Motycka TA, Matsumoto Y, Tomkinson AE (2000). "Interaction between PCNA and DNA ligase I is critical for joining of Okazaki fragments and long-patch base-excision repair". Current Biology. 10 (15): 919–22. doi:10.1016/S0960-9822(00)00619-9. PMID 10959839. S2CID 14089939.
- ^ Jin YH, Obert R, Burgers PM, Kunkel TA, Resnick MA, Gordenin DA (April 2001). "The 3'-->5' exonuclease of DNA polymerase delta can substitute for the 5' flap endonuclease Rad27/Fen1 in processing Okazaki fragments and preventing genome instability". Proceedings of the National Academy of Sciences of the United States of America. 98 (9): 5122–7. doi:10.1073/pnas.091095198. PMC 33174. PMID 11309502.
- ^ Liu Y, Kao HI, Bambara RA (2004). "Flap endonuclease 1: a central component of DNA metabolism". Annual Review of Biochemistry. 73: 589–615. doi:10.1146/annurev.biochem.73.012803.092453. PMID 15189154.
- ^ a b Bae, Sung-Ho; Kim, Dong Wook; Kim, Jiyoung; Kim, Jeong-Hoon; Kim, Do-Hyung; Kim, Hee-Dai; Kang, Ho-Young; Seo, Yeon-Soo (2002-07-19). "Coupling of DNA Helicase and Endonuclease Activities of Yeast Dna2 Facilitates Okazaki Fragment Processing". Journal of Biological Chemistry. 277 (29): 26632–26641. doi:10.1074/jbc.M111026200. PMID 12004053.
- ^ a b Bae, Sung-Ho; Seo, Yeon-Soo (2000-12-01). "Characterization of the Enzymatic Properties of the Yeast Dna2 Helicase/Endonuclease Suggests a New Model for Okazaki Fragment Processing". Journal of Biological Chemistry. 275 (48): 38022–38031. doi:10.1074/jbc.M006513200. PMID 10984490.
- ^ Kang, Young-Hoon; Lee, Chul-Hwan; Seo, Yeon-Soo (2010-02-04). "Dna2 on the road to Okazaki fragment processing and genome stability in eukaryotes". Critical Reviews in Biochemistry and Molecular Biology. 45 (2): 71–96. doi:10.3109/10409230903578593. PMID 20131965. S2CID 23897130.
- ^ a b Stewart, Jason A.; Campbell, Judith L.; Bambara, Robert A. (2009-03-27). "Significance of the Dissociation of Dna2 by Flap Endonuclease 1 to Okazaki Fragment Processing in Saccharomyces cerevisiae". Journal of Biological Chemistry. 284 (13): 8283–8291. doi:10.1074/jbc.M809189200. PMC 2659186. PMID 19179330.
- ^ Duxin, Julien P.; Moore, Hayley R.; Sidorova, Julia; Karanja, Kenneth; Honaker, Yuchi; Dao, Benjamin; Piwnica-Worms, Helen; Campbell, Judith L.; Monnat, Raymond J. (2012-06-22). "Okazaki Fragment Processing-independent Role for Human Dna2 Enzyme during DNA Replication". Journal of Biological Chemistry. 287 (26): 21980–21991. doi:10.1074/jbc.M112.359018. PMC 3381158. PMID 22570476.
- ^ Ayyagari R, Gomes XV, Gordenin DA, Burgers PM (January 2003). "Okazaki fragment maturation in yeast. I. Distribution of functions between FEN1 AND DNA2". The Journal of Biological Chemistry. 278 (3): 1618–25. doi:10.1074/jbc.M209801200. PMID 12424238.
- ^ "MCB 150 Frequently Asked Questions - Are Okazaki fragments unique to eukaryotes? Or is it universal, so it's present in bacterial DNA replication as well?".
- ^ "유핵 DNA 복제"분자-플랜트-바이오테크놀로지. multilab.biz, n.d.Web. 2011년 3월 29일
- ^ Matsunaga F, Norais C, Forterre P, Myllykallio H (February 2003). "Identification of short 'eukaryotic' Okazaki fragments synthesized from a prokaryotic replication origin". EMBO Reports. 4 (2): 154–8. doi:10.1038/sj.embor.embor732. PMC 1315830. PMID 12612604.
- ^ a b Zheng L, Shen B (February 2011). "Okazaki fragment maturation: nucleases take centre stage". Journal of Molecular Cell Biology. 3 (1): 23–30. doi:10.1093/jmcb/mjq048. PMC 3030970. PMID 21278448.
추가 정보
- Inman, Ross B.; Schnös, Maria (March 1971). "Structure of branch points in replicating DNA: Presence of single-stranded connections in λ DNA branch points". Journal of Molecular Biology. 56 (2): 319–325. doi:10.1016/0022-2836(71)90467-0. PMID 4927949.
- Thommes, Pia; Hubscher, Ulrich (December 1990). "Eukaryotic DNA replication. Enzymes and proteins acting at the fork". European Journal of Biochemistry. 194 (3): 699–712. doi:10.1111/j.1432-1033.1990.tb19460.x. PMID 2269294.
- "염색체 이상"Biology Reference, www.biologyreference.com/Ce-Co/Chromosome-Aberrations.html.
쿠퍼, 제프리 M. "DNA 복제"현재 신경학 및 신경과학 보고서, 미국 국립 의학 도서관, 1970년 1월 1일, www.ncbi.nlm.nih.gov/books/NBK9940/.