람다파지
Lambda phage| 에셰리치아바이러스 람다 | |
|---|---|
 | |
| Escherichia 바이러스 람다종의 바이러스 입자의 전자 현미경 사진 | |
| 바이러스 분류 | |
| (순위 미지정): | 바이러스 |
| 영역: | 듀플로드나바이러스 |
| 왕국: | 흥공비래 |
| 문: | 우로비리코타 |
| 클래스: | 카우도비리케테스 |
| 주문: | 카우도비랄레스 |
| 패밀리: | 십호바이러스과 |
| 속: | 람다바이러스 |
| 종류: | 에셰리치아바이러스 람다 |

엔테로박테리아파지γ(lambda phage, coliphage,, 공식명칭 에셰리치아바이러스 람다)는 세균성 대장균(E. coli)에 감염되는 세균 바이러스이다.그것은 [2]1950년 에스더 레더버그에 의해 발견되었다.야생형 바이러스는 용원균을 통해 숙주의 게놈에 상주하거나 용원균을 통해 세포를 죽이고 분해하여 자손을 낳는 용원균에 들어갈 수 있는 온대성 생명주기를 가지고 있다.특정 부위에서 돌연변이된 람다 균주는 세포를 용해시킬 수 없습니다; 대신, 그들은 성장하여 이미 [3]용해된 세포를 과잉 감염시킨 후에 용해 사이클로 들어갑니다.
파지 [4]입자는 머리(캡시드라고도 함), 꼬리 및 꼬리 섬유로 구성됩니다(아래 바이러스의 이미지 참조).머리에는 파지의 이중 가닥 선형 DNA 게놈이 들어있다.감염되는 동안, 파지 입자는 숙주 대장균을 인식하고 결합하며, 파지 머리의 DNA가 꼬리를 통해 박테리아 세포의 세포질로 배출되도록 합니다.보통, 람다 DNA가 복제되고 세포 내에서 새로운 파지 입자가 생성되는 "용해 주기"가 뒤따른다.그 후 세포 용해가 일어나 조립된 바이러스온을 포함한 세포 성분을 환경으로 방출합니다.그러나 특정 조건에서 파지 DNA는 용원 경로에서 숙주 세포 염색체에 통합될 수 있다.이 상태에서 δDNA는 프로파지라고 불리며 숙주에 명백한 해를 끼치지 않고 숙주의 게놈 내에 상주한다.프로파지가 존재할 때 숙주는 용종이라고 불립니다.이 프로파지는 용존이 스트레스 상태에 들어가면 용해 사이클로 들어갈 수 있다.
해부학

바이러스 입자는 꼬리 섬유를 가질 수 있는 머리와 꼬리로 구성되어 있다.전체 입자는 총 1000개 이상의 단백질 분자와 파지헤드에 위치한 하나의 DNA 분자를 가진 12-14개의 다른 단백질로 구성됩니다.하지만, L과 M 단백질이 비리온의 [5]일부인지는 아직 완전히 명확하지 않다.모든 특징적인 람도이드 파지는 파지 HK022를 제외하고 N단백질 매개 전사 방지 메커니즘을 가지고 있다.

게놈은 이중 가닥, 선형 DNA의 48,490쌍의 염기쌍을 포함하고 있으며, 양 [7]끝에는 12개의 염기 단일 가닥 세그먼트가 있다.이 두 개의 단일 가닥 세그먼트는 cos 사이트라고 불리는 것의 "스틱 엔드"입니다.세포질 부위는 숙주 세포질의 DNA를 순환시킵니다.따라서 원형 형태의 파지 게놈은 길이가 48,502쌍이다.[7]람다 게놈은 대장균 염색체에 삽입되어 프로파지라고 불립니다.상세한 것에 대하여는, 이하의 항을 참조해 주세요.
라이프 사이클
감염
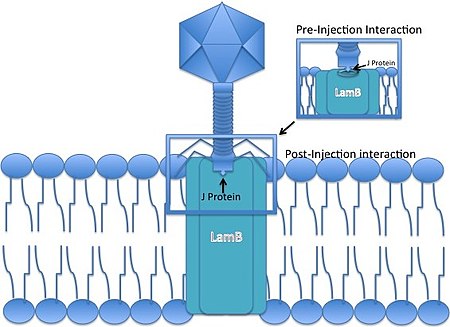
람다 파지는 비수축 꼬리 파지로, 감염 사건 동안 박테리아 세포막을 통해 DNA를 '강제'할 수 없다는 것을 의미합니다.대신 숙주 세포에 침입하기 위해 기존의 경로를 사용해야 하며, 숙주 세포에 DNA가 들어갈 수 있도록 꼬리의 끝이 특정 기공과 상호작용하도록 진화했습니다.
- 박테리오파지 람다는 꼬리 끝의 J단백질을 통해 대장균 세포에 결합합니다.J단백질은 말토스 오퍼론의 일부인 대장균의 [8]말토스 외막 포린(램B 유전자의 산물)과 상호작용한다.
- 선형 파지 게놈은 외막을 통해 주입된다.
- DNA는 내막의[9][10] 만노스 투과효소 복합체를 통과한다(manX에 의해 암호화됨).YZ 유전자)와 즉시 Cos 부위인 12-base G-C가 풍부한 응집성 "접착성 끝"을 사용하여 순환한다.단일 가닥 바이러스 DNA 말단은 숙주 DNA 연결효소에 의해 결합된다.일반적으로 [11]12bp 람다 응집 말단이 생물학적 DNA의 첫 직접 뉴클레오티드 배열의 대상이었다는 사실은 인정되지 않는다.
- 숙주 DNA 자이라아제는 원형 염색체에 음의 슈퍼코일을 넣어 A-T가 풍부한 영역을 풀어 전사를 촉진합니다.
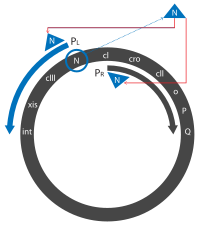
- 전사는 '즉시 조기' 전사를 생산하는 구성L P, PR, PR' 프로모터로부터 시작됩니다.처음에 이들은 N과 크로 유전자를 발현시켜 N과 크로와 짧은 비활성 단백질을 생성한다.
- 크로가 OR3에 결합하여 P 프로모터에RM 대한 접근을 방지하고 cI 유전자의 발현을 방지합니다.N은 두 개의 Nut(N 이용) 부위에 결합하고, 하나는 P 판독L 프레임의 N 유전자에 결합하고, 다른 하나는 P 판독R 프레임의 크로 유전자에 결합한다.
- N 단백질은 항암제이며, 초기에 전사된 mRNA의 특정 부위에서 전사 RNA 중합효소를 결합함으로써 기능합니다.RNA 중합효소가 이러한 영역을 전사할 때, N을 모집하고 여러 숙주 Nus 단백질과 복합체를 형성합니다.이 복합체는 대부분의 종단 시퀀스를 건너뜁니다.확장된 전사물('말기' 전사물)은 cII 및 cII 유전자와 함께 N 및 크로 유전자와 ciI 유전자와 나중에 논의된 xis, int, O, P 및 Q 유전자를 포함합니다.
- cII 단백질은 경쟁 억제제 역할을 함으로써 FtSH(막 결합 필수 대장균 단백질 효소)에 의한 단백질 분해로부터 cII 단백질을 보호하는 역할을 한다.이러한 억제는 용원성을 선호하는 정균 상태를 유도할 수 있다.cII는 또한 cII [12]단백질을 직접 안정화시킨다.
초기 감염 시 cII의 안정성은 파지의 라이프스타일을 결정하며, 안정적인 cII는 용원성 경로로 이어지는 반면, cII가 분해되면 파지는 용해 경로로 들어갑니다.낮은 온도, 세포의 기아, 높은 다중 감염(MOI)은 용원성을 선호하는 것으로 알려져 있습니다(나중에 논의 참조).
항종양법
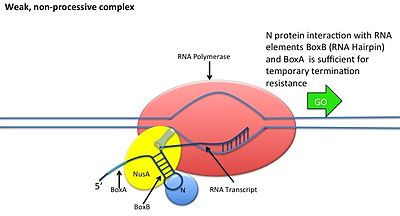
이것은 N단백질이 DNA와 상호작용하지 않고 일어난다; 대신 단백질은 새로 전사된 mRNA에 결합한다.견과류 사이트에는 3개의 보존된 "박스"가 있으며, 그 중 박스 B만이 필수적입니다.
- 박스B RNA 시퀀스는 pL 및 pR 전사체의 5' 말단 근처에 있습니다.전사될 때 각 배열은 N 단백질이 결합할 수 있는 헤어핀 루프 구조를 형성합니다.
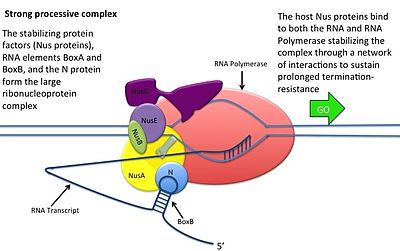
- N단백질은 각 전사체의 박스B에 결합하고 RNA 루핑을 통해 전사 RNA 중합효소와 접촉한다.N-RNAP 복합체는 여러 숙주 Nus(N 이용 물질) 단백질의 후속 결합에 의해 안정화된다(전사 종료/항피인자 및 이상하게도 리보솜 서브유닛 포함).
- 전체 복합체(mRNA의 결합된 Nut 부위 포함)는 전사를 계속하며 종단 시퀀스를 건너뛸 수 있습니다.
용혈 수명 주기
이는 대부분의 감염 후 파지가 따르는 라이프 사이클로, cII 단백질은 분해로 인해 충분히 높은 농도에 도달하지 못하기 때문에 촉진제가 활성화되지 않습니다.
- 람다 게놈(OP)의 복제를 위한 xis, int, Q, 유전자를 포함한 '말기 초기' 기록들이 계속 작성되고 있다.크로(Cro)는 억제제 부위를 지배하며("억제자" 섹션 참조), P 프로모터RM(용원 사이클의 촉진제)로부터의 합성을 억제한다.
- O 및 P 단백질은 파지 염색체의 복제를 시작합니다("용해 복제" 참조).
- 또 다른 항거미네이터인 Q는 Qut 사이트에 바인드됩니다.
- P 프로모터로부터의R' 전사는 이제 용해와 머리와 꼬리 단백질을 위한 mRNA를 생성하기 위해 확장될 수 있다.
- 구조 단백질과 파지 게놈은 새로운 파지 입자로 자가 조립됩니다.
- S, R, Rz, Rz1 유전자의 산물은 세포 용해를 일으킨다.S는 작은 막 단백질인 홀린으로 단백질의 배열에 의해 결정될 때 갑자기 막에 구멍을 뚫는다.R은 S구멍을 통해 빠져나가 세포벽을 쪼개는 효소인 엔돌리신이다.Rz와 Rz1은 세포벽을 분해한 후 외막을 어떻게든 파괴하는 복합체를 형성하는 막 단백질이다.야생형 람다의 경우 감염 개시 후 약 50분에 용해가 발생하며 100마리 안팎의 비리온을 방출한다.
오른쪽 문자 변환
오른쪽 전사는 O, P, Q 유전자를 나타냅니다.O와 P는 복제를 시작하고 Q는 P에서 머리R’, 꼬리, 용융 유전자를 발현시키는 또 다른 항암제이다.
리틱 리플리케이션
- 처음 몇 개의 복제 사이클 동안 람다 게놈은 γ 복제(원 대 원)를 거친다.
- 이것은 O 유전자에 위치한 오리 부위에서 시작된다.O단백질은 오리 부위를 결합하고, P단백질은 O단백질뿐만 아니라 숙주 복제기계의 DnaB 서브유닛도 결합한다.이것은 숙주 DNA 중합효소를 효과적으로 명령한다.
- 곧 파지 M13에서 사용하는 것과 같은 롤링 서클 복제로 전환된다.DNA가 손상되고 3' 끝부분이 프라이머 역할을 합니다.이것은 파지 게놈의 단일 복사본을 방출하는 것이 아니라 많은 게놈 복사본을 가진 하나의 긴 분자, 즉 콘사이트머를 방출하는 것에 주목하십시오.
- 이 콘크리트들은 포장될 때 cos 사이트에서 절단됩니다.원형 파지 DNA에서는 패키징이 발생할 수 없으며, 규조체 DNA에서만 발생합니다.
항종양
Q는 효과 면에서 N과 유사하다: Qut 부위에서 Q는 RNA 중합효소에 결합하고 결과 복합체는 종료자를 무시할 수 있다. 그러나 메커니즘은 매우 다르다. Q 단백질은 먼저 mRNA [14]배열이 아닌 DNA 배열과 연관된다.
- Qut 부위는 P 프로모터와R’ 매우 가까우며, RNA 중합효소 홀로엔자임에서 γ인자가 방출되지 않을 정도로 충분히 가깝다.Qut 사이트의 일부가 -10 Pribnow 박스와 비슷하여 홀로엔자임이 일시 중지됩니다.
- 다음으로 Q단백질은 γ인자의 일부를 결합 및 치환하여 전사가 다시 시작됩니다.
- 머리와 꼬리 유전자가 전사되고 대응하는 단백질이 스스로 조립됩니다.
왼쪽 문자 변환
왼쪽 전사는 gam, red, xis 및 int 유전자를 나타냅니다.Gam과 적색 단백질은 재조합에 관여한다.또한 Gam은 호스트 RecBCD 핵산가수분해효소가 롤링 서클 복제의 3' 엔드를 저하시키는 것을 억제한다는 점에서 중요합니다.Int와 xis는 용원화에 필수적인 통합 및 제거 단백질이다.
삽입 및 절제의 xis 및 int 조절
- xis와 int는 동일한 mRNA 조각에서 발견되므로, 대략 동일한 농도의 xis와 int 단백질이 생산된다.이것은 (처음에는) 숙주 게놈에서 삽입된 게놈을 제거하는 결과입니다.
- P 프로모터의L mRNA는 mRNA의 sib 부분에서 스템루프와 함께 안정적인 2차 구조를 형성한다.이것은 RNAase에 대한 mRNA의 3' (sib) 말단을 목표로 한다.III 분해는 xis mRNA보다 int mRNA의 유효 농도가 낮으므로(int cistron이 sib 시퀀스에 보다 sib 시퀀스에 가까울 때), 따라서 int보다 xis 농도가 더 높게 관찰된다.
- int보다 높은 농도의 xis는 파지 게놈의 삽입이나 절제를 초래하지 않으며, 진화적으로 선호되는 작용으로, 사전 삽입된 모든 파지 게놈을 삽입(따라서 경쟁을 감소)하게 하고 파지 게놈이 숙주의 게놈에 삽입되는 것을 막는다.
용원성(또는 용원성) 라이프 사이클
용원성 라이프 사이클은 cI 단백질이 소수의 감염 후 프로모터를 활성화하기에 충분한 농도에 도달하면 시작됩니다.
- 람다 게놈의 복제를 위한 xis, int, Q, 유전자를 포함한 '말기 초기' 기록들이 계속 작성되고 있다.
- 안정화 cII는 P, PI, Pantiq 프로모터의RE 전사를 촉진하는 역할을 합니다.
- Pantiq 프로모터는 P 프로모터R 전사체의 Q 유전자 메시지에 대한 안티센스 mRNA를 생성함으로써 Q 생성을 차단한다.PRE 프로모터는 P 프로모터R 전사체의 크로 부분에 안티센스 mRNA를 생성하여 크로 생산을 감소시키고 cI 유전자의 전사체를 가지고 있다.이는 cI 억제기 생산을 켜면서 표현됩니다.P 프로모터는 IntI 유전자를 발현시켜 Int 단백질을 고농도로 만든다.이 int 단백질은 파지 DNA를 숙주 염색체에 통합합니다("프로피지 통합" 참조).
- Q가 없으면 P 프로모터의 판독R' 프레임이 확장되지 않으므로 용해 단백질이나 구조 단백질이 생성되지 않습니다.(xis보다 훨씬 높은) int의 수치가 높아지면 람다 게놈이 호스트 게놈에 삽입됩니다(그림 참조).cI의 생성은 P 프로모터의R OR1 및 OR2 부위에 cI가 결합되어 크로 및 기타 초기 유전자 발현을 꺼지게 합니다.cI는 또한 P 프로모터에L 결합하여 거기서도 전사를 꺼줍니다.
- 크로가 부족하면 OR3 부위가 결합되지 않으므로 P 프로모터의RM 전사가 발생하여 cI 수준을 유지할 수 있습니다.
- PL 및 PR 프로모터의 전사가 부족하면 cII 및 cII가 더 이상 생성되지 않습니다.
- cII 및 cII 농도가 감소하면 Pantiq, PRE, P로부터의I 전사가 불필요해지기 때문에 촉진되지 않는다.
- PRM 및 PR' 프로모터만 활성 상태로 유지되며, 전자는 cI 단백질을 생성하며 후자는 짧은 비활성 전사체입니다.게놈은 숙주 게놈에 삽입된 상태로 휴면 상태로 남아 있다.
프로파지는 호스트의 모든 후속 세포 분할과 복제됩니다.이 휴면 상태에서 발현되는 파지 유전자는 용해 사이클로의 진입을 막기 위해 다른 파지 유전자(구조 및 용해 유전자 등)의 발현을 억제하는 단백질의 코드입니다.이 억제 단백질들은 숙주 세포가 스트레스를 받을 때 분해되어 억제된 파지 유전자의 발현을 초래한다.스트레스는 기아, 독극물(항생제 등) 또는 숙주를 손상시키거나 파괴할 수 있는 다른 요인에서 발생할 수 있습니다.스트레스에 응답하여 활성화된 프로파지는 새로 발현된 유전자 생성물 중 하나에 의해 숙주세포의 DNA에서 제거되어 그 용해 경로로 들어간다.
Prophage 통합
파지γ의 통합은 att라고λ 불리는 세균과 파지 게놈의 특별한 부착 부위에서 일어난다.gal과 bio operon 사이에서는 attB라고 불리며 B-O-B' 부분으로 구성되며, 원형 파지 게놈에서 상보적인 배열은 attP라고 불리며 P-O-P' 부분으로 구성된다.통합 자체는 홀리데이 접합을 통한 순차적 교환(유전자 재조합 참조)이며 파지 단백질 Int와 세균 단백질 IHF(통합 숙주 인자)를 모두 필요로 한다.Int와 IHF는 모두 atP에 결합하고 파지와 숙주 DNA의 부위 특이적 재조합을 위해 설계된 DNA-단백질 복합체인 인타솜을 형성한다.원래의 B-O-B' 배열은 B-O-P'-phage DNA-P-O-B'로 통합됨으로써 변화한다.파지 DNA는 이제 숙주의 [15]게놈의 일부가 되었다.
용원 유지
- 용원성은 cI에 의해서만 유지된다.cI는 P와L P로부터의R 전사를 억제하는 동시에 P로부터의RM 자신의 발현을 업 레귤레이션 및 제어한다.그러므로 그것은 용원성 파지에 의해 발현되는 유일한 단백질이다.
- 이것은 P 및R P 연산자에 의해L 조정됩니다.두 연산자 모두 cI에 대해 OL1, OL2, OL3, P에LR 대해 OR1, OR2, OR3의 3개의 바인딩 사이트가 있습니다.
- cI는 OR1에 가장 잘 결합합니다. 여기서 결합하면 P로부터의R 전사가 억제됩니다.cI가 쉽게 이량화되기 때문에 cI와 OR1의 결합은 OR2와의 cI의 결합 친화력을 크게 증가시키며, 이는 OR1의 결합 직후에 발생한다.이것은 OR2의 cI의 N 말단 도메인이 P에 대한RM RNA 중합효소의 결합을 단단하게 하고 따라서 cI가 자체 전사를 자극하기 때문에 P와RM 다른 방향으로 전사를 활성화한다.훨씬 더 높은 농도로 존재할 때, 그것은 또한 OR3에 결합하여 P로부터의RM 전사를 억제하고, 따라서 음의 피드백 루프에서 자체 레벨을 조절한다.
- P 연산자에L 대한 cI 결합은 cI 전사에 직접적인 영향을 미치지 않는다는 점을 제외하고는 매우 유사합니다.그러나 OR3와 OL3에 결합된 cI 이합체는 그 자신의 발현을 추가적으로 억제하기 위해 이들 사이의 DNA를 구부려 사량화한다.
- cI의 존재는 다른L 람다 파지의 P 및 PR 촉진제를 억제하기 때문에 중복 감염에 대한 면역성을 유발한다.
인덕션
용종의 전형적인 유도는 감염된 세포에 자외선을 조사하는 것과 관련이 있다.용원체가 DNA 손상을 입거나 숙주의 SOS 응답을 자극하는 모든 상황은 유도로 이어진다.
- 휴면 파지 게놈을 가진 숙주 세포는 높은 스트레스 환경으로 인해 DNA 손상을 경험하고 SOS 응답을 받기 시작한다.
- RecA(세포단백질)는 DNA 손상을 검출하고 활성화된다.현재는 RecA*로 매우 구체적인 공동프로세서입니다.
- 일반적으로 RecA*는 LexA(전사억제제)를 결합하여 LexA 자동단백질 활성을 활성화하여 LexA억제제를 파괴하고 DNA 복구단백질을 생산한다.용원세포에서는 이 반응이 포착되어 RecA*가 cI 자동이동을 자극합니다.이는 cI가 오토리베이지 사이트에서 LexA의 구조를 모방하기 때문입니다.
- 분해된 cI는 더 이상 이합화되지 않고 DNA 결합에 대한 친화력을 잃습니다.
- PR 및 PL 프로모터는 더 이상 억제되지 않고 켜지며, 세포는 용해성 발현 시퀀스로 돌아갑니다(SOS 응답을 받는 세포에서는 cII가 안정적이지 않습니다).하지만 한가지 눈에 띄는 차이가 있다.
유도 시 파지 게놈 절제 제어
- 파지 게놈은 여전히 숙주 게놈에 삽입되어 있고 DNA 복제가 일어나려면 절제가 필요하다.단, 일반적인L P 프로모터 문자 이외의 sib 섹션은 더 이상 이 판독 프레임에 포함되지 않는다(그림 참조).
- P 프로모터L mRNA의 어떤 sib 도메인도 3' 말단에서 헤어핀 루프를 초래하지 않으며, 전사물은 더 이상 RNAase를 대상으로 하지 않는다.III 열화
- 새로운 온전한 전사물은 xis와 int의 복사본이 하나이므로, 거의 동일한 농도의 xis와 int 단백질이 생산된다.
- 동일한 농도의 xis 및 int는 복제 및 나중에 파지 생성을 위해 삽입된 게놈을 숙주 게놈에서 제거하는 결과를 초래합니다.
다중성 재활성화 및 예방적 재활성화
다중성 재활성화(MR)는 각각 비활성화된 게놈 손상을 포함하는 여러 바이러스 게놈이 감염된 세포 내에서 상호작용하여 생존 가능한 바이러스 게놈을 형성하는 과정이다.MR은 원래 파지 T4와 함께 발견되었지만 이후 파지 β(다른 수많은 세균 및 포유류 바이러스에서도[16] 발견됨)에서 발견되었다.자외선에 의해 불활성화된 파지의 MR은 숙주 또는 감염 [17]파지의 재조합 기능에 따라 달라진다.양쪽 재조합 시스템이 없으면 MR이 손실됩니다.
대장균 숙주가 상동성 프로파지의 용원성인 경우, 프로파지 [18]재활성화라고 하는 현상인 자외선 조사 파지의 생존이 증가한다.MR과 유사한 재조합보수공정에 의해 파지 appears의 프로파지 재활성화가 발생하는 것으로 보인다.
리프레서
파지 람다에서 발견된 억제제는 매우 단순한 시스템에 의해 유전자 발현에 대한 제어 수준의 주목할 만한 예이다.그것은 Barbara J에 의해 발견되었듯이 상호 배타적인 발현 아래 있는 두 개의 유전자를 가진 '이진 스위치'를 형성한다. 마이어.[19]
람다 억제 유전자 시스템은 (염색체의 왼쪽에서 오른쪽으로) 다음과 같이 구성된다.
- cI유전자
- OR3
- OR2
- OR1
- 크로유전자
람다 억제제는 cI [20]단백질로도 알려진 자가 조립 이합체입니다.그것은 DNA를 나선-돌림-나선 결합 모티브로 결합시킨다.그것은 cI 단백질과 Cro 단백질의 전사를 조절한다.
람다 파지의 라이프 사이클은 cI와 Cro 단백질에 의해 제어된다.람다 파지는 cI 단백질이 우세하면 용원 상태에 머물지만 크로 단백질이 우세하면 용해 사이클로 전환된다.
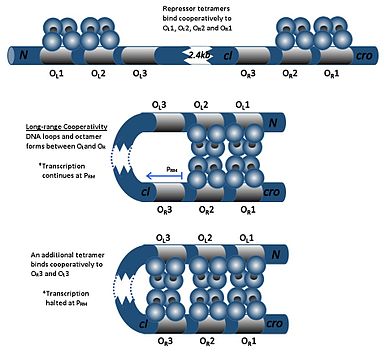
cI 디머는 O1R > O2R > O3의R 순서로R O1, O2R, O3의R 3개의 오퍼레이터 중 하나에 바인드 할 수 있습니다.cI 이합체가 O1에R 결합하면 두 번째 cI 이합체가 O2에R 결합하는 것을 강화하는데, 이것은 협동성이라고 불리는 효과이다.따라서R O1과 O2는R 거의 항상 cI에 의해 동시에 점유됩니다.단, cI 농도가 높을R 때만 점유되는 cI와 O3의 친화력은 증가하지 않습니다.
고농도의 cI에서는, 다이머는 오퍼레이터L O1 및L O2(R 오퍼레이터로부터 다운스트림 2 kb 이상)에도 바인드 됩니다.cI 이합체가 O1, O2L, O1R, O2에R 결합하면L DNA 내에서 루프가 유도되어 이들 이합체가 결합되어 옥타머가 형성됩니다.이것은 장거리 협력이라고 불리는 현상입니다.옥타머가 형성되면 cI 이합체가 O3 및R O3에 공동으로L 결합되어 cI의 전사를 억제할 수 있다.이 자동음성 조절은 억제기 분자의 안정적인 최소 농도를 보장하며, SOS 신호가 발생할 경우 보다 효율적인 프로파지 [21]유도를 가능하게 합니다.
- cI 단백질이 없는 경우 크로 유전자는 전사될 수 있다.
- cI단백질의 존재하에서는 cI유전자만이 전사될 수 있다.
- 고농도의 cI에서는 양쪽 유전자의 전사가 억제된다.
단백질 기능 개요
| 단백질 | 라이프 사이클에서의 기능 | 프로모터 지역 | 묘사 |
|---|---|---|---|
| cII | 조절 단백질 CII.용장성, cII 안정성 | PL. | (클리어 3) HflB(FtSH) 결합단백질은 단백질 분해효소 분해로부터 cII를 보호한다. |
| cII | Lysogeny, 문자 변환 액티베이터 | PR. | (클리어 2) P, PRE 및I P 프로모터로부터의AQ 문자 변환을 활성화하고 cI 및 int를 문자 변환합니다.세포성 HflB(FtSH) 단백질 분해효소(특히 건강한 세포 및 SOS 응답을 받는 세포)에 대한 민감성으로 인해 안정성이 낮다.cII가 높으면 파지가 통합과 용원성으로 밀려나는 반면, cII가 낮으면 용해가 발생합니다. |
| cI | 리프레서, 리소게니 유지 | PRM, PRE | (Clear 1) 전사억제제로 O1R, O2R, O3와 결합한다R(친화성R O1R > O2R = O3, 즉 O1과 우선적으로 결합한다R).저농도에서는 P 프로모터가R 차단됩니다(크로 생성 방지).고농도에서는 O3 결합을 통해R 자체 생산을 하향 조절합니다.억제제는 또한 P 프로모터로부터의L 전사를 억제한다.SOS 응답을 받는 셀에서 RecA*에 의해 분할되기 쉽습니다. |
| 크로 | 리프레서 조작자의 용해, 제어 | PR. | 전사 억제제는 O3, O2R 및 O1과R 결합한다R(친화성R O3 > O2R = O1R, 즉 O3와 우선적으로 결합한다R).저농도에서는 pRM 프로모터가 차단됩니다(cI 생성을 방해).고농도에서는 O2 및 O1R 결합을 통해R 자체 생산을 하향 조절합니다.공동 바인딩 없음(cI 바인딩의 경우 아래 c.f) |
| O | 용해, DNA 복제 | PR. | 레플리케이션 단백질 O.오리 부위에서 결합하여 파지 람다 DNA 복제를 시작합니다. |
| P | 용해, DNA 복제 | PR. | O 및 DnaB 서브유닛에 결합하여 Page Lambda DNA 복제를 시작합니다.이러한 결합은 숙주 DNA 중합효소에 대한 제어를 제공합니다. |
| 게임 | 용해, DNA 복제 | PL. | 호스트 RecBCD 핵산가수분해효소가 3' 엔드를 저하시키는 것을 금지합니다.롤링 서클 복제를 계속할 수 있습니다. |
| S | 용해 | PR'. | Holin은 용해 중에 막에 구멍을 뚫는 막 단백질이다. |
| R | 용해 | PR'. | 홀린에 의해 생성된 구멍을 통해 세포 밖으로 나와 세포벽을 갈라내는 효소인 리소자임인 엔돌리신. |
| Rz 및 Rz1 | 용해 | PR'. | 세포벽의 엔돌리신에 의한 분해에 따라 외세포막을 파괴하는 막단백질복합체를 형성한다.스패닌, Rz1(외막 서브유닛) 및 Rz(내막 서브유닛). |
| F | 용해 | PR'. | 파지캡시드 머리 단백질. |
| D | 용해 | PR'. | 머리 장식 단백질. |
| E | 용해 | PR'. | 주요 두부 단백질. |
| C | 용해 | PR'. | 미량 캡시드 단백질. |
| B | 용해 | PR'. | 간문단백질B. |
| A | 용해 | PR'. | 큰 터미네이스 단백질. |
| J | 용해 | PR'. | 숙주특이성 단백질 J. |
| M V U G L T Z | 용해 | PR'. | 마이너 테일 단백질 M. |
| K | 용해 | PR'. | 엔도펩티드가수분해효소일 수도 있어 |
| H | 용해 | PR'. | 꼬리줄은 단백질 H를 측정한다. |
| I | 용해 | PR'. | 꼬리 조립 단백질 I. |
| FI | 용해 | PR'. | DNA패킹단백질 FI. |
| FII | 용해 | PR'. | 꼬리 부착 단백질. |
| tfa | 용해 | PR'. | 테일 파이버 어셈블리 단백질. |
| 인트 | 게놈 통합, 절제 | PI, PL | Integrase는 숙주의 게놈에 파지 게놈을 삽입하는 것을 관리합니다.낮은 농도 조건에서는 효과가 없습니다.xis의 농도가 낮고 농도가 높으면 파지 게놈이 삽입됩니다.xis와 int의 농도가 높으면(그리고 거의 같음) 숙주의 게놈에서 파이지 게놈이 제거됩니다. |
| xis | 게놈 절제 | PI, PL | 절제효소 및 int 단백질 조절기로, 숙주의 게놈에 대한 파지 게놈의 절제 및 삽입을 관리합니다. |
| N | 후기 초기 유전자의 전사를 위한 항암제 | PL. | 항테르미네이터, RNA 결합 단백질 및 RNA 중합효소 보조 인자는 (Nut 부위에서) RNA를 결합하고 견과류 부위를 전사한 초기 RNApol로 이동합니다.이 RNApol 변형은 종단 부위의 인식을 방해하므로, 정상적인 RNA 중합효소 종단 신호는 무시되고 RNA 합성은 말단 파지 유전자(cI, cII, xis, int, O, P, Q)로 계속된다. |
| Q | 후기 용혈 유전자의 전사를 위한 항암제 | PR. | 항테르미네이터, DNA 결합 단백질 및 RNApol 보조 인자는 (Qut 부위에서) DNA를 결합하고 시작 RNApol로 이동합니다.이 RNApol 수정은 종료 시퀀스의 인식을 변경하므로 정상적인 것은 무시됩니다. 약 20,000bp 떨어진 특수 Q 종료 시퀀스가 효과적입니다.Q-확장된 전사물은 파지구조단백질(A-F, Z-J)과 용해유전자(S, R, Rz 및 Rz1)를 포함한다.용원 중 P 안티센스 mRNA에 의해antiq 하향 조절됩니다. |
| RecA | SOS 응답 | 숙주단백질 | DNA 복구 단백질, SOS 응답 중 공단백질, LexA 및 cI 자동 클리어 및 용해를 촉진합니다. |
용혈성 vs 용혈성
여기서 중요한 차이는 두 가지 결정 사이의 차이입니다; 용원균과 감염에 대한 용융, 그리고 지속적인 용원균 또는 용융.후자는 유도 섹션에 자세히 나와 있는 것처럼 셀의 SOS 응답에서 RecA가 활성화됨으로써만 결정됩니다.전자는 또한 이것의 영향을 받습니다; 어떤 cI 단백질도 축적되지 않기 때문에 SOS 응답을 받는 세포는 항상 용해됩니다.단, 감염에 대한 초기 용해/용원 결정은 cII 및 cII 단백질에 의존하기도 한다.
충분한 영양소를 가진 세포에서는 단백질 분해효소 활성도가 높아 CII가 [22]분해된다.이것은 용해 생활로 이어진다.영양소가 제한된 세포에서는 단백질 분해효소 활성이 낮아 cII가 안정적이다.이는 용원성 라이프스타일로 이어진다.cII는 직접적으로 그리고 관련된 단백질 분해효소에 대한 경쟁적 억제제로서 작용함으로써 cII를 안정시키는 것으로 보인다.이것은 "문제"에 있는 세포, 즉 영양소가 부족하고 더 휴면 상태에 있는 세포가 용존화될 가능성이 더 높다는 것을 의미한다.이것은 파지가 더 나은 시기에 떨어질 때까지 박테리아에 잠복해 있을 수 있기 때문에 선택될 것이다. 그래서 파지는 더 많은 자원을 이용할 수 있고 더 많은 전염성 세포와 더 가까운 곳에 있을 가능성이 높기 때문에 더 많은 복사본을 만들 수 있다.
람다의 용융-용해 결정을 위한 완전한 생물물리학 모델은 아직 개발되지 않았다.컴퓨터 모델링과 시뮬레이션은 감염 중의 무작위 과정이 개별 [23]세포 내에서 용해 또는 용원성 선택을 촉진한다는 것을 시사한다.그러나 최근의 실험들은 감염 전에 존재하는 세포들 사이의 물리적 차이가 세포가 용해될지 용존이 [24]될지를 미리 결정한다는 것을 암시한다.
유전적인 도구로서
람다 파지는 모델 유기체로 많이 사용되어 왔고 미생물 유전학, 그리고 나중에는 분자 유전학에서 유용한 도구의 풍부한 원천이 되었습니다.재조합 DNA 복제를 위한 벡터로서의 그것의 적용, 게이트웨이 방법에 의한 복제 DNA의 혼합을 위한 부위 특이적 재조합 효소(int)의 사용, 그리고 재조합이라 불리는 DNA 공학 방법에서 단백질 Red 알파(exo라고도 함), 베타 및 감마(gamma)를 포함한 Red 오퍼론의 적용을 포함한다.람다파지의 48KB DNA 조각은 생산적인 감염에 필수적이지 않고 외래 DNA로 대체될 수 있다. 람다파지는 플라스미드보다 박테리아에 더 쉽게 들어갈 수 있기 때문에 숙주의 DNA를 파괴하거나 일부가 될 수 있는 유용한 벡터이다. 람다파지는 조작되어 항암 백신, 나노 입자로 사용될 수 있다.인간 아스파틸(아스파라기닐)β-히드록실화효소(ASPH, HAH).[25]람다 파지는 또한 특수 변환 연구에서도 중요한 역할을 해왔다.
「 」를 참조해 주세요.
레퍼런스
- ^ Padilla-Sanchez, Victor (2021-07-16), Bacteriophage Lambda Structural Model at Atomic Resolution, doi:10.5281/zenodo.5134493, retrieved 2021-07-24
- ^ Esther Lederberg, "대장균 균주 K-12의 용원성, 미생물 유전학 회보, v.1, 페이지 5-8(1950년 1월);에 이어
- ^ Griffiths A, Miller J, Suzuki D, Lewontin R, Gelbart W (2000). An Introduction to Genetic Analysis (7th ed.). New York: W. H. Freeman. ISBN 978-0-7167-3520-5. Retrieved 19 May 2017.
- ^ Wang, Chang; Zeng, Jianwei; Wang, Jiawei (2022-04-07). "Structural basis of bacteriophage lambda capsid maturation". Structure (London, England: 1993). 30 (4): 637–645.e3. doi:10.1016/j.str.2021.12.009. ISSN 1878-4186. PMID 35026161.
- ^ a b c Rajagopala SV, Casjens S, Uetz P (September 2011). "The protein interaction map of bacteriophage lambda". BMC Microbiology. 11: 213. doi:10.1186/1471-2180-11-213. PMC 3224144. PMID 21943085.
- ^ Casjens, S. R., & Hendrix, R. W. (2015년)박테리오파지 람다: 초기 개척자이며 여전히 관련이 있다.바이러스학, 479-480, 310–330.doi:10.1016/j.virol.2015.02.010
- ^ a b 캠벨, A.M. 박테리오파지.In: Neidhardt, FC 등(1996) 대장균 및 살모넬라 티푸스:세포 및 분자생물학 (ASM Press, 워싱턴 DC)
- ^ Werts C, Michel V, Hofnung M, Charbit A (February 1994). "Adsorption of bacteriophage lambda on the LamB protein of Escherichia coli K-12: point mutations in gene J of lambda responsible for extended host range". Journal of Bacteriology. 176 (4): 941–7. doi:10.1128/jb.176.4.941-947.1994. PMC 205142. PMID 8106335.
- ^ Erni B, Zanolari B, Kocher HP (April 1987). "The mannose permease of Escherichia coli consists of three different proteins. Amino acid sequence and function in sugar transport, sugar phosphorylation, and penetration of phage lambda DNA". The Journal of Biological Chemistry. 262 (11): 5238–47. doi:10.1016/S0021-9258(18)61180-9. PMID 2951378.
- ^ Liu, Xueli; Zeng, Jianwei; Huang, Kai; Wang, Jiawei (2019-06-17). "Structure of the mannose transporter of the bacterial phosphotransferase system". Cell Research. 29 (8): 680–682. doi:10.1038/s41422-019-0194-z. ISSN 1748-7838. PMC 6796895. PMID 31209249.
- ^ Casjens, S. R., & Hendrix, R. W. (2015년)박테리오파지 람다: 초기 개척자이며 여전히 관련이 있다.바이러스학, 479-480, 310–330.doi:10.1016/j.virol.2015.02.010
- ^ Kobiler O, Rokney A, Oppenheim AB (April 2007). "Phage lambda CIII: a protease inhibitor regulating the lysis-lysogeny decision". PLOS ONE. 2 (4): e363. Bibcode:2007PLoSO...2..363K. doi:10.1371/journal.pone.0000363. PMC 1838920. PMID 17426811.
- ^ a b Santangelo TJ, Artsimovitch I (May 2011). "Termination and antitermination: RNA polymerase runs a stop sign". Nature Reviews. Microbiology. 9 (5): 319–29. doi:10.1038/nrmicro2560. PMC 3125153. PMID 21478900.
- ^ Deighan P, Hochschild A (February 2007). "The bacteriophage lambdaQ anti-terminator protein regulates late gene expression as a stable component of the transcription elongation complex". Molecular Microbiology. 63 (3): 911–20. doi:10.1111/j.1365-2958.2006.05563.x. PMID 17302807.
- ^ Groth AC, Calos MP (January 2004). "Phage integrases: biology and applications". Journal of Molecular Biology. 335 (3): 667–78. doi:10.1016/j.jmb.2003.09.082. PMID 14687564.
- ^ Michod RE, Bernstein H, Nedelcu AM (May 2008). "Adaptive value of sex in microbial pathogens". Infection, Genetics and Evolution. 8 (3): 267–85. doi:10.1016/j.meegid.2008.01.002. PMID 18295550.
- ^ Huskey RJ (April 1969). "Multiplicity reactivation as a test for recombination function". Science. 164 (3877): 319–20. Bibcode:1969Sci...164..319H. doi:10.1126/science.164.3877.319. PMID 4887562. S2CID 27435591.
- ^ Blanco M, Devoret R (March 1973). "Repair mechanisms involved in prophage reactivation and UV reactivation of UV-irradiated phage lambda". Mutation Research. 17 (3): 293–305. doi:10.1016/0027-5107(73)90001-8. PMID 4688367.
- ^ "바바라 J. Meyer", HHMI Interactive.
- ^ Burz DS, Beckett D, Benson N, Ackers GK (July 1994). "Self-assembly of bacteriophage lambda cI repressor: effects of single-site mutations on the monomer-dimer equilibrium". Biochemistry. 33 (28): 8399–405. doi:10.1021/bi00194a003. PMID 8031775.
- ^ Ptashne, Mark (2004)유전자 전환, 페이지 112콜드 스프링 하버 연구소, 뉴욕ISBN 978-0879697167.
- ^ Ptashne M(1986)'유전자 전환'유전자 제어와 파지 람다.셀프레스 ISBN 0-86542-315-6
- ^ Arkin A, Ross J, McAdams HH (August 1998). "Stochastic kinetic analysis of developmental pathway bifurcation in phage lambda-infected Escherichia coli cells". Genetics. 149 (4): 1633–48. doi:10.1093/genetics/149.4.1633. PMC 1460268. PMID 9691025.
- ^ St-Pierre F, Endy D (December 2008). "Determination of cell fate selection during phage lambda infection". Proceedings of the National Academy of Sciences of the United States of America. 105 (52): 20705–10. Bibcode:2008PNAS..10520705S. doi:10.1073/pnas.0808831105. PMC 2605630. PMID 19098103.
- ^ Fuller, Steven; Stewart, Solomon; Lebowitz, Michael; Malhotra, Kanam; Semenuk, Mark; Biswas, Biswajit; Ghanbari, Hossein (2013). "Immunogenicity of a lambda phage-based anti-cancer vaccine targeting HAAH". Journal for ImmunoTherapy of Cancer. 1 (Suppl 1): 210. doi:10.1186/2051-1426-1-S1-P210. PMC 3991175.
추가 정보
- James Watson, Tania Baker, Stephen Bell, Alexander Gann, Michael Levine, Richard Losick Molecular Biology of the Gene (국제판) - 제6판
- Mark Ptashne과 Nancy Hopkins, "람다 파지 억제기에 의해 제어되는 연산자", PNAS, v.60, n.4, 페이지 1282–1287(1968).
- 바바라 J.Meyer, Dennis G. Kleid 및 Mark Ptashne, "Lambda Repressor Turns Off Its Own Gene", PNAS, v.72, n.12, 페이지 4785–4789(1975년 12월).
- Brüssow H, Hendrix RW (January 2002). "Phage genomics: small is beautiful". Cell. 108 (1): 13–6. doi:10.1016/S0092-8674(01)00637-7. PMID 11792317.
- Dodd IB, Shearwin KE, Egan JB (April 2005). "Revisited gene regulation in bacteriophage lambda". Current Opinion in Genetics & Development. 15 (2): 145–52. doi:10.1016/j.gde.2005.02.001. PMID 15797197.
- Friedman DI, Court DL (April 2001). "Bacteriophage lambda: alive and well and still doing its thing". Current Opinion in Microbiology. 4 (2): 201–7. doi:10.1016/S1369-5274(00)00189-2. PMID 11282477.
- Gottesman, M. and Weisberg, R.A. 2004 "Little lambda - who make you?" , Micro and Mol Biol Revs, 68, 796-813 (온라인의 미생물학 및 분자생물학 리뷰, 미국 미생물학회에서 이용 가능)
- Hendrix RW, Smith MC, Burns RN, Ford ME, Hatfull GF (March 1999). "Evolutionary relationships among diverse bacteriophages and prophages: all the world's a phage". Proceedings of the National Academy of Sciences of the United States of America. 96 (5): 2192–7. Bibcode:1999PNAS...96.2192H. doi:10.1073/pnas.96.5.2192. PMC 26759. PMID 10051617.
- Kitano H (March 2002). "Systems biology: a brief overview" (PDF). Science. 295 (5560): 1662–4. Bibcode:2002Sci...295.1662K. doi:10.1126/science.1069492. PMID 11872829. S2CID 2703843.
- Ptashne, M. "유전자 스위치: Page Lambda Revisited", 2003년 제3판
- Ptashne M (June 2005). "Regulation of transcription: from lambda to eukaryotes". Trends in Biochemical Sciences. 30 (6): 275–9. doi:10.1016/j.tibs.2005.04.003. PMID 15950866.
- Snyder, L. and Champness, W. "박테리오파지 람다의 분자 유전학", 2007년 제3판 (박테리오파지 람다에 대한 유익하고 잘 설명된 개요 포함)
- Splasho, 람다의 온라인 개요(라이프사이클의 모든 단계에서 활성화된 유전자를 보여준다)
외부 링크
- Life Cycle, Lambda Lifecycle의 기본 애니메이션(일부 단백질 및 전사 세부 정보를 사용하여 감염 및 용혈/용혈 경로를 설명함)
- 파지 람다에 의한 용융과 용원성을 모두 보여주는 MIT의 시간 경과 현미경 영상
- 람다 파지 수명 주기(람다 박테리오파지 수명 주기의 기본 시각적 시연)
- GenBank 람다파지 게놈
- UniProt의 람다 파지 참조 프로테옴
- NCBI의 람다 파지 단백질 구조(박테리오파지 람다에 대한 단백질 구조의 3D 표시)