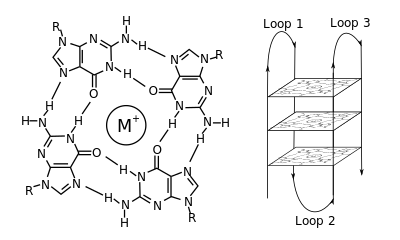
G-쿼드플렉스
G-quadruplex분자생물학에서 구아닌이 [2]풍부한 배열에 의해 핵산에서 G-쿼드플렉스 2차 구조(G4)를 형성한다.그것들은 나선형으로 생겼고 하나,[3] 두[4] 개 또는 네 개의 [5]가닥에서 형성될 수 있는 구아닌 테트라드를 포함합니다.단분자 형태는 종종 말단소체 영역으로 더 잘 알려진 염색체 끝 근처와 여러 유전자의 전사 조절 영역, 미생물과[6][7] 인간의 [9]종양유전자를 포함한 척추동물 모두에서 자연적으로 발생합니다.4개의 구아닌 염기는 Hoogsteen 수소 결합을 통해 결합하여 구아닌 테트라드(G-테트라드 또는 G-쿼텟)라고 불리는 정사각형 평면 구조를 형성할 수 있으며, 2개 이상의 구아닌 테트라드(G-tracts, 구아닌의 연속 런)가 서로 겹쳐 G-쿼드루플렉스를 형성할 수 있다.
G-쿼드플렉스를 형성하기 위한 배치 및 결합은 랜덤이 아니며 매우 특이한 기능적 목적을 제공합니다.4중 구조는 각 테트라드 [3]쌍 사이의 중앙 채널에 있는 양이온, 특히 칼륨의 존재에 의해 더욱 안정화됩니다.DNA, RNA, LNA 및 PNA로 형성될 수 있으며 분자 내, 2분자 또는 4분자일 [10]수 있습니다.테트라드를 형성하는 가닥 또는 가닥의 일부의 방향에 따라 구조는 평행 또는 반평행으로 설명될 수 있다.G-쿼드플렉스 구조는 DNA 또는 RNA 배열 [11][12]모티브에서 계산적으로 예측될 수 있지만, 그들의 실제 구조는 게놈당 10만 개 이상에 달할 수 있는 모티브 내에서 그리고 모티브 간에 상당히 달라질 수 있다.기본적인 유전자 과정에서의 그들의 활동은 텔로미어, 유전자 조절,[13][14] 기능 유전체 연구에서의 활발한 연구 영역이다.
역사
구아닌 관련성이 높은 구조의 식별은 1960년대 초에 구아닌과 [15]관련된 겔 유사 물질의 식별을 통해 명확해졌다.보다 구체적으로,[16] 이 연구는 1980년대에 DNA의 진핵생물 텔로미어 영역에서 나중에 확인되었던 구아닌의 높은 연관성을 가진 네 가닥 DNA 구조를 상세하게 설명했습니다.G-쿼드플렉스 구조를 발견하는 것의 중요성은 "G-쿼드플렉스가 그렇게 쉽게 체외에서 형성된다면, 자연은 그것들을 생체 내에서 사용하는 방법을 발견했을 것이다"라는 문구를 통해 설명되었다. - 노벨 화학상 수상자 애런 클룩(1982)2007년 4월 [7]켄터키주 루이빌에서 열린 제1회 국제 G-쿼드루플렉스 회의에서 인간, 침팬지, 마우스, 랫드의 유전자 프로모터 내 G-쿼드루플렉스(pG4) 형성 배열을 대규모 게놈 전체 분석에서 확산시킨 후 G-쿼드루플렉스의 생체 내 기능에 대한 관심이 급증했다.2006년, 몇몇 박테리아 게놈의 유전자 촉진제 내에서 G-쿼드루플렉스의 유병률은 G-쿼드루플렉스 매개 유전자 [6]조절을 예측한 것으로 보고되었다.생체 내 G-쿼드루플렉스가 풍부할 때, 이러한 구조는 종양유전자의 프로모터 영역 및 DNA 가닥의 텔로미어 영역과의 상호작용을 통해 생물학적으로 관련된 역할을 한다.현재 연구는 특정 종양유전자에 대한 이러한 G-Quadruplex 구조의 생물학적 기능을 확인하고 G-Quadruplex와의 상호작용에 기초한 암의 효과적인 치료법을 발견하는 것으로 구성되어 있다.
토폴로지
테트라드 형성에 관여하는 핵산 배열의 길이는 사중체가 접히는 방식을 결정한다.3개 이상의 구아닌 염기로 이루어진 단일 연속 실행만으로 구성된 짧은 시퀀스는 4개의 개별 가닥을 4중으로 형성해야 한다.이러한 4중합체는 4분자로 설명되며, 이는 4개의 개별 가닥의 요구사항을 반영합니다.G4 DNA라는 용어는 원래 감수분열에서 [5]역할을 할 수 있는 이러한 4분자 구조를 위해 남겨졌다.그러나 현재 분자생물학에서 사용되고 있는 바와 같이, G4라는 용어는 어떤 분자성의 G-쿼드플렉스를 의미할 수 있다.구아닌 영역이 하나 이상의 염기로 분리된 3개 이상의 구아닌 염기의 연속된 2개의 런을 포함하는 긴 배열은 4중체를 형성하기에 충분한 구아닌 염기를 제공하기 위해 2개의 그러한 배열만 필요로 한다.G가 풍부한 두 개의 분리된 가닥으로 형성된 이러한 구조를 쌍분자 사중합체라고 합니다.마지막으로, 구아닌 염기의 4개의 서로 다른 런을 포함하는 배열은 스스로 안정적인 4중 구조를 형성할 수 있으며, 전체적으로는 단일 가닥에서 형성된 4중합체를 분자 내 [17]4중합체라고 한다.
구아닌 염기의 개별 실행이 2분자 또는 분자 내 4중합체에 배치되는 방법에 따라 4중합체는 다양한 루프 구성을 가진 [18]여러 위상 중 하나를 채택할 수 있다.DNA의 모든 가닥이 같은 방향으로 진행된다면, 4중합체는 평행이라고 불립니다.분자 내 4중합체의 경우, 이는 존재하는 모든 루프 영역이 4중합체의 측면에 위치한 프로펠러 타입이어야 함을 의미합니다.구아닌 염기의 1개 이상의 런이 구아닌 염기의 다른 런과 반대인 5'-3' 방향을 가지면 4중합체는 역평행 위상을 채택한 것으로 간주된다.분자 내 역평행 4중체에서 구아닌 염기의 루프 결합은 대각선 방향의 구아닌 염기의 2개의 대각선 방향 반대 방향 또는 측면(엣지 방향) 유형의 루프 결합으로 구아닌 염기쌍의 2개의 인접 런을 결합한다.
이중 가닥 DNA로 형성된 사중극에서, 가능한 가닥간 위상 또한 [20]논의되었다.인터스트랜드 사중극에는 dsDNA의 양쪽 가닥에서 발생하는 구아닌이 포함되어 있습니다.
게놈의 구조와 기능적 역할
인간 게놈의 염기서열 분석 후, 4중체를 형성할 수 있는 가능성이 있는 구아닌이 풍부한 염기서열이 많이 발견되었다.[21]세포 유형과 세포 주기에 따라, 히스톤 단백질에 단단히 감긴 DNA로 구성된 염색질의 DNA 결합 단백질, 그리고 다른 환경 조건과 스트레스와 같은 매개 인자는 4중합체의 동적 형성에 영향을 미친다.예를 들어 분자 크로밍의 열역학에 대한 정량적 평가는 역평행 g-quadruplex가 분자 [22]크로밍에 의해 안정화됨을 나타낸다.이 효과는 DNA의 수화 변화와 Hoogsteen 염기쌍 [23]결합에 대한 영향에 의해 매개되는 것으로 보인다.이러한 사중합체는 염색체 말단에서 쉽게 발생하는 것으로 보였다.또한 서로 배타적인 헤어핀 또는 G-쿼드플렉스 구조를 형성할 가능성이 있는 RNA 배열의 전사 중 g-쿼드플렉스 형성 경향은 헤어핀 형성 [24]배열의 위치에 크게 의존한다.
복구 효소는 선형 염색체의 끝을 손상된 DNA로 자연스럽게 인식하고 세포에 해로운 영향을 미치도록 처리하기 때문에, 선형 염색체의 끝부분에서 명확한 신호 전달과 엄격한 조절이 필요하다.텔로미어는 이 시그널링을 제공하기 위해 기능합니다.텔로미어는 구아닌이 풍부하고 g-쿼드루플렉스를 형성하는 경향이 있으며 염색체의 말단에 위치하고 이러한 취약한 말단을 불안정으로부터 보호함으로써 게놈 무결성을 유지하는 데 도움을 준다.
이들 텔로머 영역은 이중가닥 CCCTAA의 긴 영역으로 특징지어집니다.TTAGGG 반복.반복은 10~50개의 단일 가닥 TTAGG 반복으로 3' 돌출되는 것으로 끝납니다.헤테로다이머 복합 리보핵단백질 효소 텔로머라아제는 DNA 가닥의 3' 말단에서 TTAGGG 반복을 추가합니다.이들 3' 말단 돌출부에서 G가 풍부한 돌출부는 돌출부가 4회 TTAGGG 반복보다 길면 G-쿼드루플렉스와 같은 2차 구조를 형성할 수 있다.이러한 구조의 존재는 텔로머라아제 [25]복합체에 의한 텔로미어 신장을 방지한다.
텔로미어 4중접속
다양한 유기체의 텔로미어 반복은 체외에서 이러한 4중 구조를 형성하고,[26][27] 그 후 체내에서도 형성되는 것으로 나타났다.인간 텔로미어 반복(모든 척추동물에 동일)은 배열(GGTTAG)의 많은 반복으로 구성되며, 이 구조에 의해 형성된 4중극체는 크기가 5~8nm인 구슬 같은 구조로 형성될 수 있으며 NMR, TEM 및 X선 결정 구조 [28]결정에 의해 잘 연구되었다.텔로미어에서 이러한 4중합체의 형성은 텔로미어의 길이를 유지하는 역할을 하고 모든 암의 약 85%에 관여하는 효소 텔로머라아제의 활성을 감소시키는 것으로 나타났다.이것은 텔로메스타틴을 포함한 약물 발견의 활성 대상이다.
비텔로미어 4중플렉스
포플렉스는 텔로미어 이외의 장소에 존재합니다.인간, 침팬지, 쥐, 쥐의 게놈을 분석한 결과 비말단성 영역에서 엄청난 수의 잠재적 G-쿼드루플렉스(pG4) 생성 서열이 나타났다.유전자 촉진제에서 많은 수의 비-텔로머 G-쿼드루플렉스가 발견되었고,[6][7] 종 전체에 걸쳐 보존되었다.마찬가지로, 대장균과 수백 개의 다른 미생물 게놈에서 대량의 G-4중합체가 발견되었다.또한 척추동물과 마찬가지로 G-쿼드루플렉스는 유전자 촉진제 [6]내에서 농축되었다.또한 RNA 중합효소 [29]II의 큰 서브유닛을 코드하는 유전자에서 식물과 조류에서 10억 년 이상 보존된 G-4중합체 궤적이 발견되었다.이러한 연구들은 G-쿼드플렉스 매개 유전자 조절을 예측했지만, 모든 pG4가 생체 내에서 형성될 가능성은 낮다.프로토-oncogenic c-myc는 유전자 [30][31]활동에 중요한 핵산가수분해효소 과민성 영역에서 4중합체를 형성한다.프로모터 영역에서 G-쿼드루플렉스를 형성하는 것으로 나타나는 다른 유전자는 닭β-글로빈 유전자, 인간 유비퀴틴 리가아제 RFP2 및 프로토온코제네스 c-kit, bcl-2, VEGF, H-ras [32][33][34]및 N-ras를 포함한다.
4중 접기 규칙에 기초한 게놈 전체 조사가 수행되었으며, 인간 게놈에서 376,000개의 추정 4중 접기 배열(PQS)을 확인했지만, 이들 모두가 생체 [35]내를 형성하지는 않을 것이다.유사한 연구는 원핵생물, 즉 [36]대장균에서 추정 G-쿼드루플렉스를 확인했다.포플렉스가 어떻게 유전자 활동에 영향을 미칠 수 있는지에 대한 몇 가지 가능한 모델들이 있는데, 상향 조절이나 하향 조절이 그것이다.프로모터 내 또는 그 부근에서 G-쿼드플렉스 형성이 유전자의 전사를 차단하여 비활성화하는 모델 중 하나가 아래에 제시되어 있다.또 다른 모델에서는 비부호화 DNA 가닥에 형성된 4중합체는 부호화 DNA 가닥의 개방적인 배열을 유지하고 각 유전자의 발현을 향상시키는 데 도움이 된다.
기능.
포플렉스 형성은 면역글로불린 중쇄 [5]전환에 역할을 한다고 제안되어 왔다.세포들이 형성되는 사중복선을 해결하기 위한 메커니즘을 진화함에 따라(즉, 풀림)4중합체 형성은 세포에 잠재적으로 손상을 줄 수 있다. 헬리케이스 WRN과 블룸 증후군 단백질은 DNA G-4중합체 [37]분해에 높은 친화력을 가진다.또한 DEAH/RHA 헬리케이스 DHX36은 주요 G-쿼드루플렉스 분해 [38][39]효소로 확인되었다.2009년 전이억제단백질 NM23H2(NME2)는 c-myc 유전자의 프로모터에서 G-quadruplex와 직접 상호작용하고 c-myc를 [40][41]전사적으로 조절하는 것으로 밝혀졌다.보다 최근에 NM23H2는 인간 텔로머라아제(hTERT) 유전자의 프로모터에서 G-쿼드루플렉스와 상호작용하고 hTERT 발현을 조절하는 것으로 보고되었다. 2019년 텔로미어 결합인자-2(TRF2 또는 TERF2)는 인간 TRADPlex에서 수천 개의 비텔로머 G-쿼드루플렉스에 결합하는 것으로 나타났다.hTERT와 [42]같은 유전자의 후생유전학적 조절을 포함하여 4중체를 양성 및 음성 전사 조절에 포함시키는 많은 연구가 있다.또한 면역로글로빈 중유전자와 병원성 [44]나이세리아의 필린 항원 변이계의 프로그래밍된 재조합을 가능하게 하는 G-쿼드루플렉스의 기능도 보고되었다.번역 제어에 있어서의 4중 구조의 역할은 충분히 조사되지 않았습니다.인간[45] 세포에서 G-쿼드루플렉스 구조의 직접 가시화와 G-쿼드루플렉스에[46] 결합된 RNA 헬리케이스의 공동 결정 구조는 세포 생물학과의 관련성에 대한 중요한 확인을 제공했다.텔로미어 복제 및 기능에서 포플렉스의 잠재적인 긍정적 역할과 부정적 역할은 여전히 논란의 여지가 있습니다.T-루프와 G-쿼드루플렉스는 텔로미어 말단을 보호하고 텔로미어 [47]길이를 조절하는 2개의 3차 DNA 구조로 설명된다.
G-쿼드플렉스 구조 형성을 통한 게놈 조절
많은 게놈 조절 과정이 G-쿼드플렉스 구조의 형성과 연관되어 있는데, 이는 AP [48]사이트로 알려진 아푸린성/아피리미딘성 부위의 DNA 수복에 있어 G-쿼드플렉스 구조가 큰 역할을 하기 때문이다.AP 부위를 지도화하는 새로운 기술은 AP-seq로 알려져 있으며, AP-seq는 비오틴 라벨이 부착된 알데히드 반응 프로브(ARP)를 이용하여 AP 부위 손상이 [49]현저한 게놈의 특정 영역에 태그를 붙인다.ChIP-시퀀싱으로 알려진 또 다른 게놈 전체 매핑 배열 방법은 AP 부위의 손상과 AP 엔도핵산가수분해효소 1(APE1)을 모두 매핑하기 위해 이용되었다.이러한 게놈 전체 매핑 배열 방법인 ChIP 염기서열 분석과 ARP는 AP 부위 손상 발생이 무작위적이지 않음을 나타냅니다.AP 부위 손상은 또한 특정 활성 프로모터와 인핸서 마커를 포함하는 게놈의 특정 영역에서도 더 흔했으며, 일부는 폐선암과 [50]대장암을 일으키는 영역과 관련이 있었다.AP 부위 손상은 게놈의 PQS 부위에서 우세한 것으로 확인되었으며, G-4중플렉스 구조의 형성은 DNA 복구 과정인 [50]BER에 의해 조절되고 촉진된다.미토콘드리아 내 성분들이 감소하기 시작하면서 세포의 염기절제 복구 과정이 노화와 함께 감소하는 것으로 입증되었고, 이는 알츠하이머병(AD)[51]과 같은 많은 질병들의 형성을 초래할 수 있다.이러한 G-쿼드플렉스 구조는 초헬리시티를 통해 DNA의 프로모터 영역에서 형성된다고 하며, 이는 DNA의 이중 나선 구조의 해체를 선호하고, 차례로 가닥을 루프로 하여 구아닌이 풍부한 영역에서 G-쿼드플렉스 구조는 구아닌이 풍부한 [52]영역에서 형성된다.BER 경로는 산화적 DNA 염기 손상을 나타내며, 여기서 8-옥소구아닌-DNA 글리코실라아제 1(OGG1), APE1 및 G-쿼드루플렉스와 같은 구조가 복구에 큰 역할을 한다.이들 효소는 구아닌 [53]염기로의 산화 스트레스 하에서 형성되는 7,8-디히드로-8-옥소구아닌(8-oxoG)과 같은 특정 DNA 병변을 복구하기 위해 BER에 참여합니다.
G4 형성에 대한 내인성 산화 DNA 염기 손상의 역할
G-쿼드루플렉스의 구아닌(G) 염기는 레독스 전위가 가장 낮아 게놈 내 내인성 산화 DNA 염기 손상인 8-옥소구아닌(8-oxoG) 형성에 더 민감하다.구아닌은 다른 뉴클레오티드 [54]염기보다 전자 환원 전위가 낮기 때문에 8-옥소-2'-디옥시구아노신(8-oxo-dG)은 DNA 산화의 주요 산물로 알려져 있다.그 농도는 세포 [55]내의 산화적 응력을 측정하는 데 사용된다.DNA가 산화 손상을 입었을 때, 이온화 방사 후 구아닌의 가능한 구조적 변화는 에놀 형태인 8-OH-Gua를 발생시킨다.이 산화물은 원래 손상 구아닌인 8-oxo-Gua에서 호변이성 시프트를 통해 형성되며 구조의 변화를 일으키는 DNA 손상을 나타낸다.이 형태를 통해 베이스 절제 수리(BER) 효소 OGG1이 APE1의 도움을 받아 산화적 손상을 결합 및 제거하여 AP 부위를 [53][51]만들 수 있습니다.또한 AP 부위는 DNA 손상으로 인해 퓨린염기 또는 피리미딘염기가 없는 DNA 내 위치이며 세포 내 가장 일반적인 내인성 DNA 손상 유형입니다.AP 사이트는 자발적으로 생성되거나 8-OH-Gua와 [49]같이 변경된 염기의 분할 후에 생성될 수 있습니다.AP 사이트를 생성하면 듀플렉스 DNA를 용융하여 PQS를 마스크 해제하고 G-쿼드플렉스 폴드를 채택할 수 있습니다.게놈 전체 ChIP 염기서열 분석, 세포 기반 분석 및 시험관 내 생화학 분석을 사용하여 산화 DNA 염기 유래 AP 부위와 G-쿼드루플렉스의 [50]형성 사이에 연결이 이루어졌다.
질병에 대한 DNA 산화의 기여
또한 8-oxo-dG의 농도는 세포 내 산화스트레스의 바이오마커로 알려져 있으며 과도한 산화스트레스는 발암 및 기타 [56]질병과 관련되어 있다.8-oxo-dG가 생성되면 OGG1을 비활성화할 수 있어 구아닌의 [50]산화에 의한 DNA 손상 복구를 방지할 수 있다.가능한 불활성화는 복구되지 않은 DNA 손상을 근육과 같은 비복제 세포에 모이게 하고 [55]노화를 일으킬 수도 있습니다.또한 8-oxo-dG와 같은 산화적 DNA 손상은 유전자 발현 변조 또는 [55]돌연변이의 유도를 통해 발암에 기여한다.BER에 의해 8-oxo-dG가 수복되는 조건으로 수복단백질의 일부가 남아 후생유전학적 변화 또는 유전자 [57][58]발현 변조를 초래할 수 있다.사람의 티미딘인산화효소 유전자에 8-oxo-dG를 삽입한 결과, BER에 의해 8-oxo-dG를 검사하지 않고 방치할 경우 돌연변이가 빈번해지고 결국 [50][51]발암으로 이어질 수 있다고 판단되었다.
유전자 조절에서의 APE1의 역할
AP 엔도핵산가수분해효소 1(APE1)은 G-4중합체 구조의 촉진 및 형성을 담당하는 효소이다.APE1은 주로 BER 경로를 통해 AP 사이트에 발생한 손상을 복구하는 업무를 담당합니다.APE1은 AP 사이트 손상이 [58]DNA에 대한 가장 반복적인 내인성 손상의 유형으로 알려져 있기 때문에 매우 중요한 것으로 여겨진다.구아닌과 같은 특정 퓨린 염기의 산화는 [55]염기서열에서 뉴클레오티드가 일치하지 않음으로써 DNA 기능을 손상시키는 산화 뉴클레오티드를 형성합니다.이는 8-옥소구아닌과 같이 산화 구조를 형성하는 PQS 배열에서 더 흔하다.세포가 산화적 스트레스와 손상을 인지하면, 그 부위에 OGG1을 모집하고, 그 주된 기능은 BER [50]경로를 시작하는 것이다.OGG1은 산화 염기를 분해하여 주로 음의 슈퍼헬리시티 [52]프로세스를 통해 AP 사이트를 생성함으로써 이를 실현합니다.다음으로 이 AP 사이트는 APE1 바인딩을 활성화하도록 셀에 신호를 보냅니다.APE1 바인딩은 오픈 듀플렉스 [56]영역에 바인드됩니다.그런 다음 APE1의 결합은 해당 영역에서 G-준중구조의 형성을 안정화함으로써 중요한 역할을 한다.이는 [59]스탠드의 접힘에 의한 G-쿼드플렉스 구조의 형성을 촉진합니다.이 루프 프로세스는 Hoogsteen 베이스 페어링에 의해 함께 고정되는 4개의 베이스를 근접하게 가져옵니다.이 단계 후에 APE1은 염색질 상의 여러 리신 잔기에 의해 아세틸화되어 아세틸화 APE1(AcAPE1)[59]을 형성한다.AcAPE1은 유전자 [60]발현을 조절할 수 있도록 전사 인자(TF)를 손상 부위에 로드하는 기능을 하는 전사 공활성제 또는 코어프레서 역할을 하기 때문에 BER 경로에 매우 중요하다.AcAPE1은 또한 APE1이 시퀀스로부터의 해리를 지연시킴으로써 더 오랜 시간 동안 결합할 수 있도록 하여 복구 프로세스를 더 효율적으로 [61]할 수 있도록 하기 때문에 매우 중요합니다.AcAPE1의 탈아세틸화는 이러한 TF의 로딩의 원동력이며, 여기서 APE1은 G-쿼드플렉스 [62]구조에서 분리된다.한 연구가 세포에서 APE1과 AcAPE1의 존재를 하향 조정했을 때, G-쿼드플렉스 구조의 형성은 억제되었고, 이는 이러한 구조의 형성에 있어 APE1의 중요성을 증명한다.그러나, 모든 G-쿼드플렉스 구조가 형성을 위해 APE1을 필요로 하는 것은 아니며, 사실 그들 중 일부는 부재 [50]중에 더 큰 G-쿼드플렉스 구조를 형성했다.따라서 APE1은 게놈 조절에서 g-quadruplex 구조의 형성을 안정화하고 AP 사이트에 전사 인자를 로드하는 두 가지 중요한 역할을 한다는 결론을 내릴 수 있다.
암
텔로미어
진핵세포, 특히 텔로미어, 5' 미번역 가닥 및 전위 핫스팟에서 G-쿼드플렉스 형성 배열이 우세하다.G-쿼드루플렉스는 정상적인 세포 기능을 억제할 수 있으며, 건강한 세포에서는 헬리케이스에 의해 쉽고 쉽게 풀린다.그러나 돌연변이 헬리케이스가 있는 암세포에서는 이러한 복합체가 풀리지 않고 세포의 잠재적 손상으로 이어진다.이것은 손상되고 암세포의 복제를 일으킨다.치료적 진보를 위해, 암세포의 G-4중합체를 안정화시키는 것은 세포의 [63]성장과 복제를 억제할 수 있다.
주최자 지역
DNA의 텔로머 영역에서 G-4중합체의 연관성과 함께, G-4중합체 구조는 다양한 인간 프로토톤 유전자 프로모터 영역에서 확인되었다.이러한 종양유전자의 프로모터 영역에 가장 많이 존재하는 구조는 평행사슬 G-4중 DNA [64]구조인 경향이 있다.이러한 종양유전자의 일부는 c-KIT, PDGF-A, c-Myc 및 VEGF를 포함하며, 이는 암의 성장과 발달에서 이 2차 구조의 중요성을 보여준다.G-쿼드플렉스 구조의 형성은 종양유전자의 다른 프로모터 영역에 따라 어느 정도 다르지만, 이러한 구조의 일관된 안정화는 암 발생에서 [65]발견되었다.현재의 치료 연구는 조절되지 않은 세포 성장과 분열을 막기 위해 G-4중구조의 안정화를 목표로 하는 데 적극적으로 초점을 맞추고 있다.
하나의 특정 유전자 영역인 c-myc 경로는 단백질 생성물인 c-myc의 조절에 필수적인 역할을 한다.본 제품에서 c-Myc 단백질은 아포토시스, 세포 성장 또는 발달 과정 및 인간 텔로머라아제 역전사효소에 [66]대한 전사 제어로 기능한다.2009년 암세포에서 C-Myc 프로모터 G-쿼드루플렉스와 NM23H2의 상호작용이 C-Myc를 조절하는 것으로 나타났다.
인간 텔로머라아제 역전사효소(hTERT)를 통한 c-myc의 조절은 후생유전변형이 NM23H2-G-쿼드루플렉스 [42]연관성에 의존한 전사인자 NM23H2와의 상호작용에 의해 프로모터를 통해 직접 조절된다.최근 hTERT 후생유전자 조절은 hTERT 프로모터 G-quadruplex와 텔로머 인자 TRF2의 [67]상호작용을 통해 매개되는 것으로 보고되었다.
또 다른 유전자 경로는 혈관신생의 과정이나 새로운 혈관의 형성에 관여하는 VEGF 유전자인 혈관내피성장인자를 다룬다.VEGF 유전자의 프로모터 영역의 폴리퓨린로 연구를 통해 분자 내 G-4중합체 구조의 형성이 확인되었다.생체 내 G-quadruplex 기능의 역할에 대한 최근 연구를 통해 G-quadruplex 구조의 안정화는 VEGF 유전자 전사를 조절하는 것으로 나타났다.분자 내 G-4중합체 구조는 대부분 이 특정 [68]경로의 프로모터 영역에서 풍부한 구아닌 배열을 통해 형성된다.사이클린 의존성 세포주기 체크포인트 키나제 억제제-1 CDKN1A(일명 p21) 유전자는 프로모터 G-쿼드루플렉스를 가지고 있다.이 G-quadruplex와 TRF2(TERF2)의 상호작용으로 p21의 후생적 조절이 이루어졌으며, G-quadruplex 결합 리간드 360A를 [69]사용하여 테스트되었다.
저산소 유도인자 1,, HIF-1,는 저산소증 존재 하에서 저산소 반응 요소(HRE)와의 결합을 통해 암 시그널링에 관여하여 혈관신생 과정을 시작한다.이 특정 유전자 경로에 대한 최근의 연구를 통해, 폴리푸린과 폴리피리미딘 영역은 이 특정 유전자의 전사와 분자 내 G-4중합체 구조의 형성을 가능하게 한다.그러나 G-쿼드루플렉스의 형성이 이 유전자의 발현을 긍정적 [70]또는 부정적으로 조절하는지를 결정하기 위해서는 더 많은 연구가 필요하다.
c-kit 종양 유전자는 RTK를 코드하는 경로를 다루며, RTK는 특정 유형의 암에서 발현 수준이 높아진 것으로 나타났다.이 프로모터 영역의 풍부한 구아닌 배열은 다양한 4중체를 형성할 수 있는 능력을 보여주었다.이 경로에 대한 현재 연구는 c-kit 경로에서 이러한 특정 4중합성 형성의 생물학적 기능을 발견하는 데 초점을 맞추고 있으며, 이 4중합성 배열은 다양한 [34]종에서 발견되었다.
RET 종양 유전자는 특정 유형의 암에 풍부했던 키나아제 전사에서 기능을 한다.이 경로에 대한 프로모터 영역의 구아닌 풍부 배열은 이 수용체 티로신 키나제의 베이스라인 전사의 필요성을 제기한다.특정 유형의 암에서는 RET 단백질이 발현 수준을 증가시켰다.이 경로에 대한 연구는 촉진제 영역에서 G-쿼드루플렉스의 형성과 치료 [71]치료를 위한 적용 가능한 대상을 제시했다.
PDGF-A를 포함하는 또 다른 종양유전자 경로인 혈소판 유래 성장인자는 상처 치유 과정을 포함하며 세포의 승모판 발생 성장인자로 기능한다.높은 수준의 PDGF 발현은 세포 성장과 암과 관련이 있다.PDGF-A의 프로모터 영역에 구아닌이 풍부한 배열이 존재함에 따라 분자 내 평행 G-쿼드플렉스 구조를 형성할 수 있는 능력을 보였으며, PDGF-A의 전사 조절에서 역할을 할 것으로 제안되었다.그러나 TMPyP4와 이 프로모터 [72]배열의 상호작용으로 인해 이 영역 내에서 G-쿼드플렉스 구조의 존재도 확인되었다.
치료학
텔로미어는 일반적으로 G-쿼드플렉스로 구성되며 치료 연구와 발견의 중요한 표적으로 남아 있습니다.이들 복합체는 포르피린 고리에 대한 친화력이 높아 효과적인 항암제가 된다.그러나 TMPyP4는 암세포 텔로미어와 정상 이중가닥 DNA(dsDNA)에 대한 비선택성 때문에 사용이 제한되었다.TMPyP4와 유사한 문제를 해결하기 위해 TMPyP4보다 [73]효과적으로 암 성장을 억제하는 G 4중 DNA만을 대상으로 하는 5Me로 합성되었다.
리간드 설계 및 개발은 G-쿼드루플렉스의 풍부함과 이들의 다중 구조 차이 때문에 치료 시약에 대한 중요한 연구 분야로 남아있다.Quindoline 유도체를 포함하는 배위자 중 하나인 SYUIQ-05는 프로모터 영역의 G-쿼드루플렉스의 안정화 기능을 이용하여 c-Myc 단백질 생성물 및 인간 텔로머라아제 역전사효소(hTERT)의 생산을 모두 억제한다.이 영역을 대상으로 하는 이러한 주요 경로는 텔로머라아제 연장의 부족을 초래하여 세포 발달을 저해한다.보다 효율적인 항종양 [66]활성으로 원치 않는 반응을 최소화하기 위해 단일 유전자 표적을 발견하기 위한 추가 연구가 여전히 필요하다.
포플렉스를 결합하는 리간드
G-쿼드플렉스 형성을 유도하거나 안정화시키는 한 가지 방법은 G-쿼드플렉스 구조에 결합할 수 있는 분자를 도입하는 것이다.작은 분자와 단백질일 수 있는 많은 리간드는 G-4중합체에 결합할 수 있다.이러한 배위자는 자연적으로 발생하거나 합성될 수 있습니다.이것은 유전학, 생화학, 약리학에서 점점 더 큰 연구 분야가 되었다.
양이온성 포르피린은 분자 텔로메스타틴뿐만 아니라 G-쿼드루플렉스와도 상호 작용적으로 결합하는 것으로 나타났다.
G-쿼드루플렉스는 전형적으로 전위 핫스팟에서 발견되기 때문에 G-쿼드루플렉스에 대한 리간드의 결합은 항암 추적에 필수적이다.BCL-2 프로모터에서 4중으로 선택적으로 결합하는 배위자 MM41은 중앙 코어 및 입체적으로 분기하는 4개의 사이드 체인으로 형성된다.리간드의 모양은 사중합체와 핵산의 고리가 겹쳐져 있는 사중합체와 밀접하게 일치하기 때문에 매우 중요합니다.결합 시, MM41의 중심 발색단은 3' 말단 G-쿼텟 위에 위치하고 배위자의 측쇄는 4중합체의 루프에 관련된다.4중주단과 색소체는 γ-γ 결합으로 결합되어 있고, 사이드 체인과 루프는 결합되어 있지 않지만 근접해 있습니다.이 결합을 강하게 하는 것은 리간드 측쇄와 [74]더 잘 결합하기 위한 루프 위치의 유동성입니다.
양이온성 포르피린인 TMPyP4는 c-Myc를 억제하는 데 도움이 되는 더 잘 알려진 G4 결합 리간드이다.TMPyP4가 G4에 바인드하는 방법은 MM41과 비슷하며 링은 외부 G쿼트에 스태킹되고 사이드 [75]체인은 G4의 루프에 관련지어집니다.
G-쿼드루플렉스에 결합하는 리간드를 설계할 때, 리간드는 병렬 접힌 G-쿼드루플렉스에 대한 친화력이 더 높다.배위자가 작을수록 전자 밀도가 더 높기 때문에 측쇄가 작은 배위자는 사중합체에 더 잘 결합하는 것으로 밝혀졌다.또, 측쇄가 작은 리간드의 수소 결합이 짧고, 따라서 강하다.이동성 측쇄를 가진 리간드는 G4 루프와 리간드 측쇄의 배치가 [76]정렬될 수 있기 때문에 중앙 발색단 주위로 회전할 수 있는 리간드는 G-쿼드루플렉스와 더욱 강하게 관련된다.
4중 예측 기술
포플렉스를 형성할 수 있는 능력을 가진 시퀀스를 식별하고 예측하는 것은 그 역할을 더욱 이해하는 데 중요한 도구입니다.일반적으로 간단한 패턴 매치는 가능한 스트랜드 내 4중합성 배열 d(GNGNG3+1-73+1-73+1-73+)를 탐색하기 위해 사용된다.여기서 N은 임의의 뉴클레오티드 염기(구아닌 [77]포함)이다.이 규칙은 온라인 알고리즘에서 널리 사용되고 있습니다.이 규칙은 G-4중합체 형성 부위를 효과적으로 식별하지만 트리플렉스 형성[78] 및 C-스트랜드 i-모티프 [79]형성이 가능한 불완전한 호모푸린 미러 반복의 서브셋도 식별한다.또한, 이러한 배열은 4중[4] 및 3중[80] DNA 구조를 형성하는 암묵적 중간체인 슬립 및 폴드백 구조를 형성하는 능력도 가지고 있다.한 연구에서,[81] 이러한 모티브의 염기쌍당 관찰된 수(즉, 빈도)가 완전한 게놈 서열을 사용할 수 있는 음메타조아에서 빠르게 증가한 것으로 밝혀졌다.이는 비 B구조 형성을 억제할 수 있는 시스템의 진화에 의해 시퀀스가 포지티브 선택 하에 있을 수 있음을 시사한다.
G-쿼드플렉스 연구 방법
G-쿼드플렉스의 계산 예측을 지원하기 위해 많은 실험 방법이 개발되었다.이 방법들은 크게 생물물리법과 생화학법의 [82]두 종류로 정의될 수 있다.
생화학적 방법
생화학적 기법은 긴 시퀀스 맥락에서 G-쿼드플렉스 형성을 조사하기 위해 사용되었다.본 발명의 DNA 중합효소 정지 어세이에서는 DNA 템플릿 중 G-쿼드루플렉스의 형성은 바리케이드로서 작용하여 중합효소 실속을 일으킬 수 있으며, 이로 인해 프라이머 [83]확장이 정지된다.디메틸 황산염(DMS)에 이어 피페리딘 분해 분석을 실시하는 것은 G-쿼드루플렉스의 형성이 DMS에 의한 N7 구아닌 메틸화를 금지하고, 피페리딘 분해 [84]후 DNA G-쿼드루플렉스 영역에서 관찰되는 보호 패턴을 가져온다는 사실에 기초한다.
생물물리학적 방법
G-쿼드플렉스 구조의 토폴로지는 특정 [85]파장에서 양의 또는 음의 원형 이색성(CD) 신호를 모니터링하여 확인할 수 있습니다.병렬 G-쿼드루플렉스는 각각 240nm와 262nm에서 음의 CD신호와 양의 CD신호를 갖는 반면 역평행 G-쿼드루플렉스는 각각 262nm와 295nm에서 이들 신호를 배치한다.G-quadruplex 형성을 검증하려면 비 G-quadruplex 안정화(Li+) 및 G-quadruplex 안정화 조건(K+ 또는 G-quadruplex 리간드 등)에서 CD 실험을 수행하고 원 UV 영역(180-230 nm)을 향해 스캔해야 합니다.마찬가지로 295nm의 [86]UV신호를 관찰함으로써 G-쿼드플렉스 구조의 내열성을 확인할 수 있다.G-쿼드플렉스 용융 시 295 nm에서의 UV 흡광도가 감소하여 G-쿼드플렉스 구조의 특징인 저색소 변화를 일으킨다.G-쿼드루플렉스의 검출을 위한 또 다른 접근방식은 나노포어 기반의 방법을 포함한다.첫째, 생물학적 나노포어는 G-쿼드루플렉스와 단백질 나노캐비티의 [87]크기 배제 및 특이적 상호작용에 기초하여 G-쿼드루플렉스를 검출할 수 있는 것으로 나타났다.이 새로운 접근법은 G-쿼드플렉스의 라벨 없는 검출, dsDNA에서의 매핑 및 G-쿼드플렉스 [88]형성을 감시하기 위한 고체 나노포어와 DNA 나노 기술을 결합한다.
신경 장애에서의 역할
G-쿼드루플렉스는 두 가지 주요 메커니즘을 통해 신경학적 장애와 관련이 있다.첫 번째는 C9orf72 유전자와 근위축성 측삭경화증(ALS) 또는 전두엽성 치매(FTD)의 경우와 같이 직접적으로 질병을 일으키는 G-4중구 구조의 형성을 유도하는 유전자 내 G-반복의 확장을 통해서이다.두 번째 메커니즘은 연약한 X 정신지체 유전자 1(FMR1) 유전자와 연약한 X [89]증후군에서 볼 수 있듯이 G-쿼드플렉스 결합 단백질의 발현에 영향을 미치는 돌연변이를 통해 이루어진다.
C9orf72 유전자는 신경세포질과 시냅스 전 말단에서 [90]뇌 전체에 발견되는 단백질 C9orf72를 코드화한다.C9orf72 유전자의 돌연변이는 FTD와 [91]ALS의 발달과 관련이 있다.이 두 질환은 C9orf72 유전자의 첫 번째 인트론 내에서 GGGCC(GC)42 반복과 인과관계가 있다.보통 일반인은 약 242~8GC의 반복 횟수를 가지지만 FTD 또는 ALS를 가진 사람은 500~수천GC의42 [92][93]반복 횟수를 가집니다.이러한 반복실험의 전사된 RNA는 안정적인 G-쿼드루플렉스를 형성하는 것으로 나타났으며, DNA의 GC 반복실험은42 혼합된 병렬-반파라렐 G-쿼드루플렉스 구조도 [94][95]형성할 수 있다는 증거가 있다.GC 반복을 포함하는 이러한42 RNA 전사물은 뉴클레오린을 포함한 다양한 단백질을 결합하고 분리하는 것으로 나타났다.뉴클레오린은 핵 내의 리보솜의 합성 및 성숙에 관여하며, 변이된 RNA 전사체에 의한 뉴클레오린의 분리는 핵소체 기능과 리보솜 RNA [96]합성을 저해한다.
취약 X 정신지체단백질(FMRP)은 FMR1 유전자에 의해 코드화된 널리 발현되는 단백질로 뉴런의 G-쿼드플렉스 2차 구조에 결합하고 시냅스 [97]가소성에 관여한다.FMRP는 번역의 네거티브 레귤레이터로서 작용하며, 그 결합은 mRNA 전사체의 G-쿼드플렉스 구조를 안정화시켜 뉴런의 수상돌기에서 mRNA의 리보솜 신장을 억제하고 전사체의 [98][99]발현 타이밍을 제어한다.이 유전자의 돌연변이는 연약한 X 증후군, 자폐증, 그리고 다른 신경학적 [100]장애의 발생을 야기할 수 있다.구체적으로, 연약 X 증후군은 FMR1 유전자의 엑손 13 내에서 CGG 반복 횟수가 50회에서 200회 이상으로 증가하여 발생합니다.이러한 반복 팽창은 유전자의 전사를 막는 FMR1의 DNA 메틸화와 다른 후생적 헤테로크로마틴 변형을 촉진하여 병리적으로 FMRP의 [101][102]낮은 수치를 초래한다.
치료적 접근법
반센스 매개 개입과 소분자 배위자는 G-4중합체 확장 반복과 관련된 신경학적 질병을 목표로 하는 데 사용되는 일반적인 전략이다.따라서 이러한 기술은 특히 기능성 메커니즘이 있는 신경계 질환, 즉 변경된 유전자 생성물이 새로운 기능 또는 유전자의 새로운 발현을 가지고 있을 때 C9orf72(염색체9 공개판독범위72)[103]에서 검출되었다.
안티센스 치료는 합성된 핵산 가닥이 특정 유전자에 의해 생성된 mRNA와 직접적이고 구체적으로 결합하기 위해 사용되는 과정이며, 이것은 그것을 불활성화시킬 것이다.안티센스 올리고뉴클레오티드(ASO)는 G-쿼드루플렉스 GGGCC 확장 반복 영역의 C9orf72 RNA를 목표로 하는 데 일반적으로 사용되며, 이는 C9orf72 [104][105][106]세포 모델에서 독성을 낮췄다.ASO는 이전에 기능성 메커니즘을 가진 다른 신경 질환에서 정상적인 표현형을 복원하는 데 사용되었지만, 유일한 차이점은 G-4중플렉스 확장 반복 [107][108][109][110]영역이 없을 때 사용되었다는 것이다.
일반적으로 사용되는 또 다른 기술은 소분자 배위자의 이용이다.이것들은 신경학적 장애를 일으키는 G-4중플렉스 영역을 목표로 하는 데 사용될 수 있다.방향족 고리를 통해 상호작용할 수 있는 약 1,000개의 다양한 G-쿼드플렉스 배위자가 존재한다. 이는 소분자 배위자가 G-쿼드플렉스 영역 내의 평면 말단 테트라드에 쌓일 수 있게 한다.치료기술로서 소분자 리간드를 사용하는 단점은 G-쿼드루플렉스의 1차 배열, 배향, 열역학적 안정성 및 핵산 스트랜드 화학량계의 가변성으로 인해 특이성을 관리하기 어렵다는 것이다.현재 [when?]단일 G-4분자 [111][112]배열에 대해 완벽하게 특이할 수 있는 단일 소분자 배위자는 없습니다.단, TMPyP4로 알려진 양이온성 포르피린은 C9orf72 GGGCC 반복영역에 결합할 수 있으며, 이로 인해 G-쿼드루플렉스 반복영역이 펼쳐지고 단백질과의 상호작용이 상실되어 기능이 [113]상실된다.주로 납으로 구성된 소분자 리간드는 GGGCC 반복 영역도 대상으로 할 수 있으며, 궁극적으로 근위축성 측삭경화증(ALS) 환자로부터 파생된 뉴런 세포에서 반복 관련 비 ATG 번역과 RNA 포시를 모두 감소시켰다.이는 소분자 배위자가 GGGCC 영역을 대상으로 하는 효과적이고 효율적인 과정이며, 소분자 배위자 결합의 특이성이 과학계에 실현 가능한 목표라는 증거를 제공한다.
금속 착체는 G4 DNA 결합체로서 특히 적합하며 따라서 잠재적 약물로 적합하게 만드는 많은 특징을 가지고 있다.금속은 대부분의 G4 바인더에서 구조적인 역할을 하지만, 정전기적 상호작용이나 [114]핵염기와의 직접적인 조정에 의해 G4와 직접 상호작용하는 예도 있다.
레퍼런스
- ^ Capra, John A.; Paeschke, Katrin; Singh, Mona; Zakian, Virginia A.; Stormo, Gary D. (22 July 2010). "G-Quadruplex DNA Sequences Are Evolutionarily Conserved and Associated with Distinct Genomic Features in Saccharomyces cerevisiae". PLOS Computational Biology. 6 (7): e1000861. Bibcode:2010PLSCB...6E0861C. doi:10.1371/journal.pcbi.1000861. PMC 2908698. PMID 20676380.
- ^ Routh ED, Creacy SD, Beerbower PE, Akman SA, Vaughn JP, Smaldino PJ (March 2017). "A G-quadruplex DNA-affinity Approach for Purification of Enzymaticacvly Active G4 Resolvase1". Journal of Visualized Experiments. 121 (121). doi:10.3791/55496. PMC 5409278. PMID 28362374.
- ^ a b Largy E, Mergny J, Gabelica V (2016). "Chapter 7. Role of Alkali Metal Ions in G-Quadruplex Nucleic Acid Structure and Stability". In Astrid S, Helmut S, Roland KO S (eds.). The Alkali Metal Ions: Their Role in Life (PDF). Metal Ions in Life Sciences. Vol. 16. Springer. pp. 203–258. doi:10.1007/978-3-319-21756-7_7. PMID 26860303.
- ^ a b Sundquist WI, Klug A (December 1989). "Telomeric DNA dimerizes by formation of guanine tetrads between hairpin loops". Nature. 342 (6251): 825–9. Bibcode:1989Natur.342..825S. doi:10.1038/342825a0. PMID 2601741. S2CID 4357161.
- ^ a b c Sen D, Gilbert W (July 1988). "Formation of parallel four-stranded complexes by guanine-rich motifs in DNA and its implications for meiosis". Nature. 334 (6180): 364–6. Bibcode:1988Natur.334..364S. doi:10.1038/334364a0. PMID 3393228. S2CID 4351855.
- ^ a b c d Rawal P, Kummarasetti VB, Ravindran R, Kumar N, Halder K, Sharma R, Mukerji M, Das SK, Chowdhury S (2006). "Genome-wide Prediction of G4 DNA as Regulatory Motifs: Role in Escherichia Coli Global Regulation". Genome Research. 16 (5): 644‐655. doi:10.1101/gr.4508806. PMC 1457047. PMID 16651665.
- ^ a b c d Borman S (May 28, 2007). "Ascent of quadruplexes nucleic acid structures become promising drug targets". Chemical and Engineering News. 85 (22): 12–17. doi:10.1021/cen-v085n009.p012a.
- ^ Verma A, Halder K, Halder R, Yadav VK, Rawal P, Thakur RK, Mohd F, Sharma A, Chowdhury S (2008). "Genome-wide Computational and Expression Analyses Reveal G-quadruplex DNA Motifs as Conserved Cis-Regulatory Elements in Human and Related Species". Journal of Medicinal Chemistry. 51 (18): 5641‐5649. doi:10.1021/jm800448a. PMID 18767830.
- ^ Han H, Hurley LH (April 2000). "G-quadruplex DNA: a potential target for anti-cancer drug design". Trends in Pharmacological Sciences. 21 (4): 136–42. doi:10.1016/s0165-6147(00)01457-7. PMID 10740289.
- ^ Bochman ML, Paeschke K, Zakian VA (November 2012). "DNA secondary structures: stability and function of G-quadruplex structures". Nature Reviews. Genetics. 13 (11): 770–80. doi:10.1038/nrg3296. PMC 3725559. PMID 23032257.
- ^ Yadav VK, Abraham JK, Mani P, Kulshrestha R, Chowdhury S (2008). "QuadBase: Genome-Wide Database of G4 DNA--occurrence and Conservation in Human, Chimpanzee, Mouse and Rat Promoters and 146 Microbes". Nucleic Acids Research. 36 (Database): D381‐D385. doi:10.1093/nar/gkm781. PMC 2238983. PMID 17962308.
- ^ Dhapola P, Chowdhury S (July 2016). "QuadBase2: Web Server for Multiplexed Guanine Quadruplex Mining and Visualization". Nucleic Acids Research. 44 (W1): W277‐W283. doi:10.1093/nar/gkw425. PMC 4987949. PMID 27185890.
- ^ Rhodes D, Lipps HJ (October 2015). "G-quadruplexes and their regulatory roles in biology". Nucleic Acids Research. 43 (18): 8627–37. doi:10.1093/nar/gkv862. PMC 4605312. PMID 26350216.
- ^ Borman S (November 2009). "Promoter quadruplexes folded DNA structures in gene-activation sites may be useful cancer drug targets". Chemical and Engineering News. 87 (44): 28–30. doi:10.1021/cen-v087n044.p028.
- ^ Gellert M, Lipsett MN, Davies DR (December 1962). "Helix formation by guanylic acid". Proceedings of the National Academy of Sciences of the United States of America. 48 (12): 2013–8. Bibcode:1962PNAS...48.2013G. doi:10.1073/pnas.48.12.2013. PMC 221115. PMID 13947099.
- ^ Henderson E, Hardin CC, Walk SK, Tinoco I, Blackburn EH (December 1987). "Telomeric DNA oligonucleotides form novel intramolecular structures containing guanine-guanine base pairs". Cell. 51 (6): 899–908. doi:10.1016/0092-8674(87)90577-0. PMID 3690664. S2CID 37343642.
- ^ Simonsson T (April 2001). "G-quadruplex DNA structures--variations on a theme". Biological Chemistry. 382 (4): 621–8. doi:10.1515/BC.2001.073. PMID 11405224. S2CID 43536134.
- ^ Burge S, Parkinson GN, Hazel P, Todd AK, Neidle S (2006). "Quadruplex DNA: sequence, topology and structure". Nucleic Acids Research. 34 (19): 5402–15. doi:10.1093/nar/gkl655. PMC 1636468. PMID 17012276.
- ^ Cao K, Ryvkin P, Johnson FB (May 2012). "Computational detection and analysis of sequences with duplex-derived interstrand G-quadruplex forming potential". Methods. 57 (1): 3–10. doi:10.1016/j.ymeth.2012.05.002. PMC 3701776. PMID 22652626.
- ^ Kudlicki AS (2016). "G-Quadruplexes Involving Both Strands of Genomic DNA Are Highly Abundant and Colocalize with Functional Sites in the Human Genome". PLOS ONE. 11 (1): e0146174. Bibcode:2016PLoSO..1146174K. doi:10.1371/journal.pone.0146174. PMC 4699641. PMID 26727593.
- ^ Murat P, Balasubramanian S (April 2014). "Existence and consequences of G-quadruplex structures in DNA". Current Opinion in Genetics & Development. 25 (25): 22–9. doi:10.1016/j.gde.2013.10.012. PMID 24584093.
- ^ Miyoshi D, Karimata H, Sugimoto N (June 2006). "Hydration regulates thermodynamics of G-quadruplex formation under molecular crowding conditions". Journal of the American Chemical Society. 128 (24): 7957–63. doi:10.1021/ja061267m. PMID 16771510.
- ^ Zheng KW, Chen Z, Hao YH, Tan Z (January 2010). "Molecular crowding creates an essential environment for the formation of stable G-quadruplexes in long double-stranded DNA". Nucleic Acids Research. 38 (1): 327–38. doi:10.1093/nar/gkp898. PMC 2800236. PMID 19858105.
- ^ Endoh T, Rode AB, Takahashi S, Kataoka Y, Kuwahara M, Sugimoto N (February 2016). "Real-Time Monitoring of G-Quadruplex Formation during Transcription". Analytical Chemistry. 88 (4): 1984–9. doi:10.1021/acs.analchem.5b04396. PMID 26810457.
- ^ Wang Q, Liu JQ, Chen Z, Zheng KW, Chen CY, Hao YH, Tan Z (August 2011). "G-quadruplex formation at the 3' end of telomere DNA inhibits its extension by telomerase, polymerase and unwinding by helicase". Nucleic Acids Research. 39 (14): 6229–37. doi:10.1093/nar/gkr164. PMC 3152327. PMID 21441540.
- ^ Schaffitzel C, Berger I, Postberg J, Hanes J, Lipps HJ, Plückthun A (July 2001). "In vitro generated antibodies specific for telomeric guanine-quadruplex DNA react with Stylonychia lemnae macronuclei". Proceedings of the National Academy of Sciences of the United States of America. 98 (15): 8572–7. Bibcode:2001PNAS...98.8572S. doi:10.1073/pnas.141229498. PMC 37477. PMID 11438689.
- ^ Paeschke K, Simonsson T, Postberg J, Rhodes D, Lipps HJ (October 2005). "Telomere end-binding proteins control the formation of G-quadruplex DNA structures in vivo". Nature Structural & Molecular Biology. 12 (10): 847–54. doi:10.1038/nsmb982. PMID 16142245. S2CID 6079323.
- ^ Kar, Anirban; Jones, Nathan; Arat, N. Özlem; Fishel, Richard; Griffith, Jack D. (2018-06-15). "Long repeating (TTAGGG) n single-stranded DNA self-condenses into compact beaded filaments stabilized by G-quadruplex formation". Journal of Biological Chemistry. 293 (24): 9473–9485. doi:10.1074/jbc.RA118.002158. ISSN 0021-9258. PMC 6005428. PMID 29674319.
- ^ Volná, Adriana; Bartas, Martin; Karlický, Václav; Nezval, Jakub; Kundrátová, Kristýna; Pečinka, Petr; Špunda, Vladimír; Červeň, Jiří (2021). "G-Quadruplex in Gene Encoding Large Subunit of Plant RNA Polymerase II: A Billion-Year-Old Story". International Journal of Molecular Sciences. 22 (14): 7381. doi:10.3390/ijms22147381. PMC 8306923. PMID 34299001.
- ^ Simonsson T, Pecinka P, Kubista M (March 1998). "DNA tetraplex formation in the control region of c-myc". Nucleic Acids Research. 26 (5): 1167–72. doi:10.1093/nar/26.5.1167. PMC 147388. PMID 9469822.
- ^ Siddiqui-Jain A, Grand CL, Bearss DJ, Hurley LH (September 2002). "Direct evidence for a G-quadruplex in a promoter region and its targeting with a small molecule to repress c-MYC transcription". Proceedings of the National Academy of Sciences of the United States of America. 99 (18): 11593–8. Bibcode:2002PNAS...9911593S. doi:10.1073/pnas.182256799. PMC 129314. PMID 12195017.
- ^ Huppert JL, Balasubramanian S (14 December 2006). "G-quadruplexes in promoters throughout the human genome". Nucleic Acids Research. 35 (2): 406–13. doi:10.1093/nar/gkl1057. PMC 1802602. PMID 17169996.
- ^ Dai J, Dexheimer TS, Chen D, Carver M, Ambrus A, Jones RA, Yang D (February 2006). "An intramolecular G-quadruplex structure with mixed parallel/antiparallel G-strands formed in the human BCL-2 promoter region in solution". Journal of the American Chemical Society. 128 (4): 1096–8. doi:10.1021/ja055636a. PMC 2556172. PMID 16433524.
- ^ a b Fernando H, Reszka AP, Huppert J, Ladame S, Rankin S, Venkitaraman AR, Neidle S, Balasubramanian S (June 2006). "A conserved quadruplex motif located in a transcription activation site of the human c-kit oncogene". Biochemistry. 45 (25): 7854–60. doi:10.1021/bi0601510. PMC 2195898. PMID 16784237.
- ^ Huppert JL, Balasubramanian S (2005). "Prevalence of quadruplexes in the human genome". Nucleic Acids Research. 33 (9): 2908–16. doi:10.1093/nar/gki609. PMC 1140081. PMID 15914667.
- ^ Rawal P, Kummarasetti VB, Ravindran J, Kumar N, Halder K, Sharma R, Mukerji M, Das SK, Chowdhury S (May 2006). "Genome-wide prediction of G4 DNA as regulatory motifs: role in Escherichia coli global regulation". Genome Research. 16 (5): 644–55. doi:10.1101/gr.4508806. PMC 1457047. PMID 16651665.
- ^ Kamath-Loeb A, Loeb LA, Fry M (2012). Cotterill S (ed.). "The Werner syndrome protein is distinguished from the Bloom syndrome protein by its capacity to tightly bind diverse DNA structures". PLOS ONE. 7 (1): e30189. Bibcode:2012PLoSO...730189K. doi:10.1371/journal.pone.0030189. PMC 3260238. PMID 22272300.
- ^ Vaughn JP, Creacy SD, Routh ED, Joyner-Butt C, Jenkins GS, Pauli S, Nagamine Y, Akman SA (November 2005). "The DEXH protein product of the DHX36 gene is the major source of tetramolecular quadruplex G4-DNA resolving activity in HeLa cell lysates". The Journal of Biological Chemistry. 280 (46): 38117–20. doi:10.1074/jbc.C500348200. PMID 16150737.
- ^ Chen MC, Ferré-D'Amaré AR (15 August 2017). "Structural Basis of DEAH/RHA Helicase Activity". Crystals. 7 (8): 253. doi:10.3390/cryst7080253.
- ^ a b Thakur RK, Kumar P, Halder K, Verma A, Kar A, Parent JL, Basundra R, Kumar A, Chowdhury S (January 2009). "Metastases Suppressor NM23-H2 Interaction With G-quadruplex DNA Within c-MYC Promoter Nuclease Hypersensitive Element Induces c-MYC Expression". Nucleic Acids Research. 37 (1): 172‐183. doi:10.1093/nar/gkn919. PMC 2615625. PMID 19033359.
- ^ Borman S (November 2009). "Promoter Quadruplexes Folded DNA structures in gene-activation sites may be useful cancer drug targets". Chemical and Engineering News. 87 (44): 28–30. doi:10.1021/cen-v087n044.p028.
- ^ a b c Saha D, Singh A, Hussain T, Srivastava V, Sengupta S, Kar A, Dhapola P, Ummanni R, Chowdhury S (July 2017). "Epigenetic Suppression of Human Telomerase ( hTERT) Is Mediated by the Metastasis Suppressor NME2 in a G-quadruplex-dependent Fashion". The Journal of Biological Chemistry. 292 (37): 15205‐15215. doi:10.1074/jbc.M117.792077. PMC 5602382. PMID 28717007.
- ^ Mukherjee AK, Sharma S, Bagri S, Kutum R, Kumar P, Hussain A, Singh P, Saha D, Kar A, Dash D, Chowdhury S (November 2019). "Telomere Repeat-Binding Factor 2 Binds Extensively to Extra-Telomeric G-quadruplexes and Regulates the Epigenetic Status of Several Gene Promoters". The Journal of Biological Chemistry. 294 (47): 17709–17722. doi:10.1074/jbc.RA119.008687. PMC 6879327. PMID 31575660.
- ^ Maizels N, Gray LT (April 2013). Rosenberg SM (ed.). "The G4 genome". PLOS Genetics. 9 (4): e1003468. doi:10.1371/journal.pgen.1003468. PMC 3630100. PMID 23637633.
- ^ Biffi G, Tannahill D, McCafferty J, Balasubramanian S (March 2013). "Quantitative visualization of DNA G-quadruplex structures in human cells". Nature Chemistry. 5 (3): 182–6. Bibcode:2013NatCh...5..182B. doi:10.1038/nchem.1548. PMC 3622242. PMID 23422559.
- ^ Chen MC, Tippana R, Demeshkina NA, Murat P, Balasubramanian S, Myong S, Ferré-D'Amaré AR (June 2018). "Structural basis of G-quadruplex unfolding by the DEAH/RHA helicase DHX36". Nature. 558 (7710): 465–469. Bibcode:2018Natur.558..465C. doi:10.1038/s41586-018-0209-9. PMC 6261253. PMID 29899445.
- ^ Rice C, Skordalakes E (2016). "Structure and function of the telomeric CST complex". Computational and Structural Biotechnology Journal. 14: 161–7. doi:10.1016/j.csbj.2016.04.002. PMC 4872678. PMID 27239262.
- ^ Hänsel-Hertsch, Robert; Beraldi, Dario; Lensing, Stefanie V.; Marsico, Giovanni; Zyner, Katherine; Parry, Aled; Di Antonio, Marco; Pike, Jeremy; Kimura, Hiroshi; Narita, Masashi; Tannahill, David (October 2016). "G-quadruplex structures mark human regulatory chromatin". Nature Genetics. 48 (10): 1267–1272. doi:10.1038/ng.3662. ISSN 1546-1718. PMID 27618450. S2CID 20967177.
- ^ a b Poetsch, Anna R. (2020). "AP-Seq: A Method to Measure Apurinic Sites and Small Base Adducts Genome-Wide". The Nucleus. Methods in Molecular Biology (Clifton, N.J.). Vol. 2175. pp. 95–108. doi:10.1007/978-1-0716-0763-3_8. ISBN 978-1-0716-0762-6. ISSN 1940-6029. PMID 32681486. S2CID 220631202.
- ^ a b c d e f g Roychoudhury, Shrabasti; Pramanik, Suravi; Harris, Hannah L.; Tarpley, Mason; Sarkar, Aniruddha; Spagnol, Gaelle; Sorgen, Paul L.; Chowdhury, Dipanjan; Band, Vimla; Klinkebiel, David; Bhakat, Kishor K. (May 26, 2020). "Endogenous oxidized DNA bases and APE1 regulate the formation of G-quadruplex structures in the genome". Proceedings of the National Academy of Sciences of the United States of America. 117 (21): 11409–11420. doi:10.1073/pnas.1912355117. ISSN 1091-6490. PMC 7260947. PMID 32404420.
- ^ a b c d Canugovi, Chandrika; Shamanna, Raghavendra A.; Croteau, Deborah L.; Bohr, Vilhelm A. (2014-06-01). "Base excision DNA repair levels in mitochondrial lysates of Alzheimer's disease". Neurobiology of Aging. 35 (6): 1293–1300. doi:10.1016/j.neurobiolaging.2014.01.004. ISSN 0197-4580. PMC 5576885. PMID 24485507.
- ^ a b Sun, Daekyu; Hurley, Laurence H. (2009-05-14). "The importance of negative superhelicity in inducing the formation of G-quadruplex and i-motif structures in the c-Myc promoter: implications for drug targeting and control of gene expression". Journal of Medicinal Chemistry. 52 (9): 2863–2874. doi:10.1021/jm900055s. ISSN 0022-2623. PMC 2757002. PMID 19385599.
- ^ a b Hill, J. W. (2001-01-15). "Stimulation of human 8-oxoguanine-DNA glycosylase by AP-endonuclease: potential coordination of the initial steps in base excision repair". Nucleic Acids Research. 29 (2): 430–438. doi:10.1093/nar/29.2.430. ISSN 1362-4962. PMC 29662. PMID 11139613.
- ^ Burrows, Cynthia J.; Muller, James G. (1998). "Oxidative Nucleobase Modifications Leading to Strand Scission". Chemical Reviews. 98 (3): 1109–1152. doi:10.1021/cr960421s. ISSN 0009-2665. PMID 11848927.
- ^ a b c d Poetsch, Anna R. (2020-01-07). "The genomics of oxidative DNA damage, repair, and resulting mutagenesis". Computational and Structural Biotechnology Journal. 18: 207–219. doi:10.1016/j.csbj.2019.12.013. ISSN 2001-0370. PMC 6974700. PMID 31993111.
- ^ a b Fleming, Aaron M.; Burrows, Cynthia J. (2017-10-11). "8-Oxo-7,8-dihydro-2′-deoxyguanosine and abasic site tandem lesions are oxidation prone yielding hydantoin products that strongly destabilize duplex DNA". Organic & Biomolecular Chemistry. 15 (39): 8341–8353. doi:10.1039/C7OB02096A. ISSN 1477-0539. PMC 5636683. PMID 28936535.
- ^ "Epigenetics", Wikipedia, 2020-12-16, retrieved 2020-12-16
- ^ a b Kitsera, Nataliya; Rodriguez-Alvarez, Marta; Emmert, Steffen; Carell, Thomas; Khobta, Andriy (2019-09-19). "Nucleotide excision repair of abasic DNA lesions". Nucleic Acids Research. 47 (16): 8537–8547. doi:10.1093/nar/gkz558. ISSN 0305-1048. PMC 6895268. PMID 31226203.
- ^ a b Roychoudhury, Shrabasti; Nath, Somsubhra; Song, Heyu; Hegde, Muralidhar L.; Bellot, Larry J.; Mantha, Anil K.; Sengupta, Shiladitya; Ray, Sutapa; Natarajan, Amarnath; Bhakat, Kishor K. (2016-12-19). "Human Apurinic/Apyrimidinic Endonuclease (APE1) Is Acetylated at DNA Damage Sites in Chromatin, and Acetylation Modulates Its DNA Repair Activity". Molecular and Cellular Biology. 37 (6). doi:10.1128/mcb.00401-16. ISSN 0270-7306. PMC 5335514. PMID 27994014.
- ^ Chattopadhyay, Ranajoy; Das, Soumita; Maiti, Amit K.; Boldogh, Istvan; Xie, Jingwu; Hazra, Tapas K.; Kohno, Kimitoshi; Mitra, Sankar; Bhakat, Kishor K. (2008-09-22). "Regulatory Role of Human AP-Endonuclease (APE1/Ref-1) in YB-1-Mediated Activation of the Multidrug Resistance Gene MDR1". Molecular and Cellular Biology. 28 (23): 7066–7080. doi:10.1128/mcb.00244-08. ISSN 0270-7306. PMC 2593380. PMID 18809583.
- ^ Bhakat, K. K. (2003-12-01). "Role of acetylated human AP-endonuclease (APE1/Ref-1) in regulation of the parathyroid hormone gene". The EMBO Journal. 22 (23): 6299–6309. doi:10.1093/emboj/cdg595. ISSN 1460-2075. PMC 291836. PMID 14633989.
- ^ Yamamori, Tohru; DeRicco, Jeremy; Naqvi, Asma; Hoffman, Timothy A.; Mattagajasingh, Ilwola; Kasuno, Kenji; Jung, Saet-Byel; Kim, Cuk-Seong; Irani, Kaikobad (2009-11-24). "SIRT1 deacetylates APE1 and regulates cellular base excision repair". Nucleic Acids Research. 38 (3): 832–845. doi:10.1093/nar/gkp1039. ISSN 0305-1048. PMC 2817463. PMID 19934257.
- ^ Neidle S (July 2016). "Quadruplex Nucleic Acids as Novel Therapeutic Targets" (PDF). Journal of Medicinal Chemistry. 59 (13): 5987–6011. doi:10.1021/acs.jmedchem.5b01835. PMID 26840940.
- ^ Chen Y, Yang D (September 2012). Sequence, stability, and structure of G-quadruplexes and their interactions with drugs. Current Protocols in Nucleic Acid Chemistry. Vol. Chapter 17. pp. 17.5.1–17.5.17. doi:10.1002/0471142700.nc1705s50. ISBN 978-0471142706. PMC 3463244. PMID 22956454.
- ^ Brooks TA, Kendrick S, Hurley L (September 2010). "Making sense of G-quadruplex and i-motif functions in oncogene promoters". The FEBS Journal. 277 (17): 3459–69. doi:10.1111/j.1742-4658.2010.07759.x. PMC 2971675. PMID 20670278.
- ^ a b Ou TM, Lin J, Lu YJ, Hou JQ, Tan JH, Chen SH, Li Z, Li YP, Li D, Gu LQ, Huang ZS (August 2011). "Inhibition of cell proliferation by quindoline derivative (SYUIQ-05) through its preferential interaction with c-myc promoter G-quadruplex". Journal of Medicinal Chemistry. 54 (16): 5671–9. doi:10.1021/jm200062u. PMID 21774525.
- ^ Sharma S, Mukherjee AK, Roy SS, Bagri S, Lier S, Verma M, Sengupta A, Kumar M, Nesse G, Pandey DP, Chowdhury S (January 2020). "Human Telomerase Expression is under Direct Transcriptional Control of the Telomere-binding-factor TRF2" (PDF). bioRxiv. doi:10.1101/2020.01.15.907626. S2CID 214472968.
- ^ Sun D, Guo K, Rusche JJ, Hurley LH (2005-10-12). "Facilitation of a structural transition in the polypurine/polypyrimidine tract within the proximal promoter region of the human VEGF gene by the presence of potassium and G-quadruplex-interactive agents". Nucleic Acids Research. 33 (18): 6070–80. doi:10.1093/nar/gki917. PMC 1266068. PMID 16239639.
- ^ Hussain T, Saha D, Purohit G, Mukherjee AK, Sharma S, Sengupta S, Dhapola P, Maji B, Vedagopuram S, Horikoshi NT, Horikoshi N, Pandita RK, Bhattacharya S, Bajaj A, Riou JF, Pandita TK, Chowdhury S (September 2017). "Transcription Regulation of CDKN1A (p21/CIP1/WAF1) by TRF2 Is Epigenetically Controlled Through the REST Repressor Complex". Scientific Reports. 7 (1): 11541. Bibcode:2017NatSR...711541H. doi:10.1038/s41598-017-11177-1. PMC 5599563. PMID 28912501.
- ^ De Armond R, Wood S, Sun D, Hurley LH, Ebbinghaus SW (December 2005). "Evidence for the presence of a guanine quadruplex forming region within a polypurine tract of the hypoxia inducible factor 1alpha promoter". Biochemistry. 44 (49): 16341–50. doi:10.1021/bi051618u. PMID 16331995.
- ^ Guo K, Pourpak A, Beetz-Rogers K, Gokhale V, Sun D, Hurley LH (August 2007). "Formation of pseudosymmetrical G-quadruplex and i-motif structures in the proximal promoter region of the RET oncogene". Journal of the American Chemical Society. 129 (33): 10220–8. doi:10.1021/ja072185g. PMC 2566970. PMID 17672459.
- ^ Qin Y, Rezler EM, Gokhale V, Sun D, Hurley LH (2007-11-26). "Characterization of the G-quadruplexes in the duplex nuclease hypersensitive element of the PDGF-A promoter and modulation of PDGF-A promoter activity by TMPyP4". Nucleic Acids Research. 35 (22): 7698–713. doi:10.1093/nar/gkm538. PMC 2190695. PMID 17984069.
- ^ Chilakamarthi U, Koteshwar D, Jinka S, Vamsi Krishna N, Sridharan K, Nagesh N, Giribabu L (November 2018). "Novel Amphiphilic G-Quadruplex Binding Synthetic Derivative of TMPyP4 and Its Effect on Cancer Cell Proliferation and Apoptosis Induction". Biochemistry. 57 (46): 6514–6527. doi:10.1021/acs.biochem.8b00843. PMID 30369235. S2CID 53093959.
- ^ Ohnmacht SA, Marchetti C, Gunaratnam M, Besser RJ, Haider SM, Di Vita G, Lowe HL, Mellinas-Gomez M, Diocou S, Robson M, Šponer J, Islam B, Pedley RB, Hartley JA, Neidle S (June 2015). "A G-quadruplex-binding compound showing anti-tumour activity in an in vivo model for pancreatic cancer". Scientific Reports. 5: 11385. Bibcode:2015NatSR...511385O. doi:10.1038/srep11385. PMC 4468576. PMID 26077929.
- ^ Siddiqui-Jain A, Grand CL, Bearss DJ, Hurley LH (September 2002). "Direct evidence for a G-quadruplex in a promoter region and its targeting with a small molecule to repress c-MYC transcription". Proceedings of the National Academy of Sciences of the United States of America. 99 (18): 11593–8. Bibcode:2002PNAS...9911593S. doi:10.1073/pnas.182256799. PMC 129314. PMID 12195017.
- ^ Collie GW, Promontorio R, Hampel SM, Micco M, Neidle S, Parkinson GN (February 2012). "Structural basis for telomeric G-quadruplex targeting by naphthalene diimide ligands". Journal of the American Chemical Society. 134 (5): 2723–31. doi:10.1021/ja2102423. PMID 22280460.
- ^ Todd AK, Johnston M, Neidle S (2005). "Highly prevalent putative quadruplex sequence motifs in human DNA". Nucleic Acids Research. 33 (9): 2901–7. doi:10.1093/nar/gki553. PMC 1140077. PMID 15914666.
- ^ Frank-Kamenetskii MD, Mirkin SM (1995). "Triplex DNA structures". Annual Review of Biochemistry. 64 (9): 65–95. doi:10.1146/annurev.bi.64.070195.000433. PMID 7574496.
- ^ Guo K, Gokhale V, Hurley LH, Sun D (August 2008). "Intramolecularly folded G-quadruplex and i-motif structures in the proximal promoter of the vascular endothelial growth factor gene". Nucleic Acids Research. 36 (14): 4598–608. doi:10.1093/nar/gkn380. PMC 2504309. PMID 18614607.
- ^ Mirkin SM, Lyamichev VI, Drushlyak KN, Dobrynin VN, Filippov SA, Frank-Kamenetskii MD (1987). "DNA H form requires a homopurine-homopyrimidine mirror repeat". Nature. 330 (6147): 495–7. Bibcode:1987Natur.330..495M. doi:10.1038/330495a0. PMID 2825028. S2CID 4360764.
- ^ Smith SS (2010). "Evolutionary expansion of structurally complex DNA sequences". Cancer Genomics & Proteomics. 7 (4): 207–15. PMID 20656986.
- ^ Kwok, Chun Kit; Merrick, Catherine J. (2017-10-01). "G-Quadruplexes: Prediction, Characterization, and Biological Application". Trends in Biotechnology. 35 (10): 997–1013. doi:10.1016/j.tibtech.2017.06.012. ISSN 0167-7799. PMID 28755976.
- ^ Han, H.; Hurley, L. H.; Salazar, M. (1999-01-01). "A DNA polymerase stop assay for G-quadruplex-interactive compounds". Nucleic Acids Research. 27 (2): 537–542. doi:10.1093/nar/27.2.537. ISSN 0305-1048. PMC 148212. PMID 9862977.
- ^ Sun, Daekyu; Hurley, Laurence H. (2009-10-23), Biochemical Techniques for the Characterization of G-Quadruplex Structures: EMSA, DMS Footprinting, and DNA Polymerase Stop Assay, Methods in Molecular Biology, vol. 608, Humana Press, pp. 65–79, doi:10.1007/978-1-59745-363-9_5, ISBN 9781588299505, PMC 2797547, PMID 20012416
- ^ Paramasivan, Sattanathan; Rujan, Iulian; Bolton, Philip H. (2007-12-01). "Circular dichroism of quadruplex DNAs: Applications to structure, cation effects and ligand binding". Methods. Quadruplex DNA. 43 (4): 324–331. doi:10.1016/j.ymeth.2007.02.009. ISSN 1046-2023. PMID 17967702.
- ^ Mergny, Jean-Louis; Phan, Anh-Tuan; Lacroix, Laurent (1998-09-11). "Following G-quartet formation by UV-spectroscopy". FEBS Letters. 435 (1): 74–78. doi:10.1016/s0014-5793(98)01043-6. ISSN 0014-5793. PMID 9755862. S2CID 1306129.
- ^ An, Na; Fleming, Aaron M.; Middleton, Eric G.; Burrows, Cynthia J. (2014-09-15). "Single-molecule investigation of G-quadruplex folds of the human telomere sequence in a protein nanocavity". Proceedings of the National Academy of Sciences. 111 (40): 14325–14331. Bibcode:2014PNAS..11114325A. doi:10.1073/pnas.1415944111. ISSN 0027-8424. PMC 4209999. PMID 25225404.
- ^ Bošković, Filip; Zhu, Jinbo; Chen, Kaikai; Keyser, Ulrich F. (2019-10-02). "Monitoring G-Quadruplex Formation with DNA Carriers and Solid-State Nanopores". Nano Letters. 19 (11): 7996–8001. Bibcode:2019NanoL..19.7996B. doi:10.1021/acs.nanolett.9b03184. ISSN 1530-6984. PMID 31577148. S2CID 203638480.
- ^ Simone R, Fratta P, Neidle S, Parkinson GN, Isaacs AM (June 2015). "G-quadruplexes: Emerging roles in neurodegenerative diseases and the non-coding transcriptome". FEBS Letters. 589 (14): 1653–68. doi:10.1016/j.febslet.2015.05.003. PMID 25979174.
- ^ C9orf72 염색체 9개 개방 판독범위 72 [호모 사피엔스] - 유전자 - NCBI
- ^ Ratnavalli E, Brayne C, Dawson K, Hodges JR (June 2002). "The prevalence of frontotemporal dementia". Neurology. 58 (11): 1615–21. doi:10.1212/WNL.58.11.1615. PMID 12058088. S2CID 45904851.
- ^ Rutherford NJ, Heckman MG, Dejesus-Hernandez M, Baker MC, Soto-Ortolaza AI, Rayaprolu S, Stewart H, Finger E, Volkening K, Seeley WW, Hatanpaa KJ, Lomen-Hoerth C, Kertesz A, Bigio EH, Lippa C, Knopman DS, Kretzschmar HA, Neumann M, Caselli RJ, White CL, Mackenzie IR, Petersen RC, Strong MJ, Miller BL, Boeve BF, Uitti RJ, Boylan KB, Wszolek ZK, Graff-Radford NR, Dickson DW, Ross OA, Rademakers R (December 2012). "Length of normal alleles of C9ORF72 GGGGCC repeat do not influence disease phenotype". Neurobiology of Aging. 33 (12): 2950.e5–7. doi:10.1016/j.neurobiolaging.2012.07.005. PMC 3617405. PMID 22840558.
- ^ Beck J, Poulter M, Hensman D, Rohrer JD, Mahoney CJ, Adamson G, Campbell T, Uphill J, Borg A, Fratta P, Orrell RW, Malaspina A, Rowe J, Brown J, Hodges J, Sidle K, Polke JM, Houlden H, Schott JM, Fox NC, Rossor MN, Tabrizi SJ, Isaacs AM, Hardy J, Warren JD, Collinge J, Mead S (March 2013). "Large C9orf72 hexanucleotide repeat expansions are seen in multiple neurodegenerative syndromes and are more frequent than expected in the UK population". American Journal of Human Genetics. 92 (3): 345–53. doi:10.1016/j.ajhg.2013.01.011. PMC 3591848. PMID 23434116.
- ^ Fratta P, Mizielinska S, Nicoll AJ, Zloh M, Fisher EM, Parkinson G, Isaacs AM (December 2012). "C9orf72 hexanucleotide repeat associated with amyotrophic lateral sclerosis and frontotemporal dementia forms RNA G-quadruplexes". Scientific Reports. 2: 1016. Bibcode:2012NatSR...2E1016F. doi:10.1038/srep01016. PMC 3527825. PMID 23264878.
- ^ Reddy K, Zamiri B, Stanley SY, Macgregor RB, Pearson CE (April 2013). "The disease-associated r(GGGGCC)n repeat from the C9orf72 gene forms tract length-dependent uni- and multimolecular RNA G-quadruplex structures". The Journal of Biological Chemistry. 288 (14): 9860–6. doi:10.1074/jbc.C113.452532. PMC 3617286. PMID 23423380.
- ^ Haeusler AR, Donnelly CJ, Periz G, Simko EA, Shaw PG, Kim MS, Maragakis NJ, Troncoso JC, Pandey A, Sattler R, Rothstein JD, Wang J (March 2014). "C9orf72 nucleotide repeat structures initiate molecular cascades of disease". Nature. 507 (7491): 195–200. Bibcode:2014Natur.507..195H. doi:10.1038/nature13124. PMC 4046618. PMID 24598541.
- ^ Darnell, J. C., Jensen, K. B., Jin, P., Brown, V., Warren, S. T., Darnell. R. B. (November 2001). "Fragile X Mental Retardation Protein Targets G Quartet mRNAs Important for Neuronal Function". Cell. 107 (4): 489–499. doi:10.1016/S0092-8674(01)00566-9. PMID 11719189. S2CID 8203054.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Ceman S, O'Donnell WT, Reed M, Patton S, Pohl J, Warren ST (December 2003). "Phosphorylation influences the translation state of FMRP-associated polyribosomes". Human Molecular Genetics. 12 (24): 3295–305. doi:10.1093/hmg/ddg350. PMID 14570712.
- ^ Fähling M, Mrowka R, Steege A, Kirschner KM, Benko E, Förstera B, Persson PB, Thiele BJ, Meier JC, Scholz H (February 2009). "Translational regulation of the human achaete-scute homologue-1 by fragile X mental retardation protein". The Journal of Biological Chemistry. 284 (7): 4255–66. doi:10.1074/jbc.M807354200. PMID 19097999.
- ^ "취약 X 정신지체" 인간 유전자 요약
- ^ Pieretti, M., Zhang, F., Fu, Y., Warren, S. T., Oostra, B. A., Caskey, C. T., Nelson, D. L. (August 1991). "Absence of expression of the FMR-1 gene in fragile X syndrome". Cell. 66 (4): 816–822. doi:10.1016/0092-8674(91)90125-I. PMID 1878973. S2CID 31455523.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Sutcliffe JS, Nelson DL, Zhang F, Pieretti M, Caskey CT, Saxe D, Warren ST (September 1992). "DNA methylation represses FMR-1 transcription in fragile X syndrome". Human Molecular Genetics. 1 (6): 397–400. doi:10.1093/hmg/1.6.397. PMID 1301913.
- ^ Mizielinska S, Isaacs AM (October 2014). "C9orf72 amyotrophic lateral sclerosis and frontotemporal dementia: gain or loss of function?". Current Opinion in Neurology. 27 (5): 515–23. doi:10.1097/WCO.0000000000000130. PMC 4165481. PMID 25188012.
- ^ Donnelly CJ, Zhang PW, Pham JT, Haeusler AR, Heusler AR, Mistry NA, Vidensky S, Daley EL, Poth EM, Hoover B, Fines DM, Maragakis N, Tienari PJ, Petrucelli L, Traynor BJ, Wang J, Rigo F, Bennett CF, Blackshaw S, Sattler R, Rothstein JD (October 2013). "RNA toxicity from the ALS/FTD C9ORF72 expansion is mitigated by antisense intervention". Neuron. 80 (2): 415–28. doi:10.1016/j.neuron.2013.10.015. PMC 4098943. PMID 24139042.
- ^ Lagier-Tourenne C, Baughn M, Rigo F, Sun S, Liu P, Li HR, Jiang J, Watt AT, Chun S, Katz M, Qiu J, Sun Y, Ling SC, Zhu Q, Polymenidou M, Drenner K, Artates JW, McAlonis-Downes M, Markmiller S, Hutt KR, Pizzo DP, Cady J, Harms MB, Baloh RH, Vandenberg SR, Yeo GW, Fu XD, Bennett CF, Cleveland DW, Ravits J (November 2013). "Targeted degradation of sense and antisense C9orf72 RNA foci as therapy for ALS and frontotemporal degeneration". Proceedings of the National Academy of Sciences of the United States of America. 110 (47): E4530–9. Bibcode:2013PNAS..110E4530L. doi:10.1073/pnas.1318835110. PMC 3839752. PMID 24170860.
- ^ Sareen D, O'Rourke JG, Meera P, Muhammad AK, Grant S, Simpkinson M, Bell S, Carmona S, Ornelas L, Sahabian A, Gendron T, Petrucelli L, Baughn M, Ravits J, Harms MB, Rigo F, Bennett CF, Otis TS, Svendsen CN, Baloh RH (October 2013). "Targeting RNA foci in iPSC-derived motor neurons from ALS patients with a C9ORF72 repeat expansion". Science Translational Medicine. 5 (208): 208ra149. doi:10.1126/scitranslmed.3007529. PMC 4090945. PMID 24154603.
- ^ Wheeler TM, Leger AJ, Pandey SK, MacLeod AR, Nakamori M, Cheng SH, Wentworth BM, Bennett CF, Thornton CA (August 2012). "Targeting nuclear RNA for in vivo correction of myotonic dystrophy". Nature. 488 (7409): 111–5. Bibcode:2012Natur.488..111W. doi:10.1038/nature11362. PMC 4221572. PMID 22859208.
- ^ Lee JE, Bennett CF, Cooper TA (March 2012). "RNase H-mediated degradation of toxic RNA in myotonic dystrophy type 1". Proceedings of the National Academy of Sciences of the United States of America. 109 (11): 4221–6. Bibcode:2012PNAS..109.4221L. doi:10.1073/pnas.1117019109. PMC 3306674. PMID 22371589.
- ^ Carroll JB, Warby SC, Southwell AL, Doty CN, Greenlee S, Skotte N, Hung G, Bennett CF, Freier SM, Hayden MR (December 2011). "Potent and selective antisense oligonucleotides targeting single-nucleotide polymorphisms in the Huntington disease gene / allele-specific silencing of mutant huntingtin". Molecular Therapy. 19 (12): 2178–85. doi:10.1038/mt.2011.201. PMC 3242664. PMID 21971427.
- ^ Gagnon KT, Pendergraff HM, Deleavey GF, Swayze EE, Potier P, Randolph J, Roesch EB, Chattopadhyaya J, Damha MJ, Bennett CF, Montaillier C, Lemaitre M, Corey DR (November 2010). "Allele-selective inhibition of mutant huntingtin expression with antisense oligonucleotides targeting the expanded CAG repeat". Biochemistry. 49 (47): 10166–78. doi:10.1021/bi101208k. PMC 2991413. PMID 21028906.
- ^ 캠벨, N.H., 파텔, M. 등(2009).G- 4중 루프의 리간드 인식에서 선택적.생화학. 48. 1675-1680.
- ^ S.A. 옴마흐트 및 S. Neidle.(2014).소분자 사중자 표적 약물 발견바이오오그메드, 케미컬24번지 2602-2612번지
- ^ Zamiri, B., Reddy, K. 등 (2014년)TMPyP4 포르피린은 C9orf72 유전자의 질병과 관련된 r(GGGCC)n 반복의 RNA G-쿼드플렉스 구조를 왜곡하고 RNA 결합 단백질의 상호작용을 차단한다.J. Biol. 제289호 4653-4659호
- ^ Vilar R (2018). "Chapter 12. Nucleic Acid Quadruplexes and Metallo-Drugs". In Sigel A, Sigel H, Freisinger E, Sigel RK (eds.). Metallo-Drugs: Development and Action of Anticancer Agents. Metal Ions in Life Sciences. Vol. 18. pp. 325–349. doi:10.1515/9783110470734-018. ISBN 9783110470734. PMID 29394031.
추가 정보
- Ren J, Wang J, Han L, Wang E, Wang J (October 2011). "Kinetically grafting G-quadruplexes onto DNA nanostructures for structure and function encoding via a DNA machine". Chemical Communications. 47 (38): 10563–5. doi:10.1039/c1cc13973h. PMID 21858307.
- Johnson JE, Smith JS, Kozak ML, Johnson FB (August 2008). "In vivo veritas: using yeast to probe the biological functions of G-quadruplexes". Biochimie. 90 (8): 1250–63. doi:10.1016/j.biochi.2008.02.013. PMC 2585026. PMID 18331848.
- Huppert JL, Balasubramanian S (2005). "Prevalence of quadruplexes in the human genome". Nucleic Acids Research. 33 (9): 2908–16. doi:10.1093/nar/gki609. PMC 1140081. PMID 15914667.
- Todd AK, Johnston M, Neidle S (2005). "Highly prevalent putative quadruplex sequence motifs in human DNA". Nucleic Acids Research. 33 (9): 2901–7. doi:10.1093/nar/gki553. PMC 1140077. PMID 15914666.
- Burge S, Parkinson GN, Hazel P, Todd AK, Neidle S (2006). "Quadruplex DNA: sequence, topology and structure". Nucleic Acids Research. 34 (19): 5402–15. doi:10.1093/nar/gkl655. PMC 1636468. PMID 17012276.
- Siddiqui-Jain A, Grand CL, Bearss DJ, Hurley LH (September 2002). "Direct evidence for a G-quadruplex in a promoter region and its targeting with a small molecule to repress c-MYC transcription". Proceedings of the National Academy of Sciences of the United States of America. 99 (18): 11593–8. Bibcode:2002PNAS...9911593S. doi:10.1073/pnas.182256799. PMC 129314. PMID 12195017.
- Rawal P, Kummarasetti VB, Ravindran J, Kumar N, Halder K, Sharma R, Mukerji M, Das SK, Chowdhury S (May 2006). "Genome-wide prediction of G4 DNA as regulatory motifs: role in Escherichia coli global regulation". Genome Research. 16 (5): 644–55. doi:10.1101/gr.4508806. PMC 1457047. PMID 16651665.
- Hou X, Guo W, Xia F, Nie FQ, Dong H, Tian Y, Wen L, Wang L, Cao L, Yang Y, Xue J, Song Y, Wang Y, Liu D, Jiang L (June 2009). "A biomimetic potassium responsive nanochannel: G-quadruplex DNA conformational switching in a synthetic nanopore". Journal of the American Chemical Society. 131 (22): 7800–5. doi:10.1021/ja901574c. PMID 19435350.
- Neidle & Balasubramanian, ed. (2006). Quadruplex Nucleic Acids. ISBN 978-0-85404-374-3. Archived from the original on 2007-09-30.
- Rowland GB, Barnett K, Dupont JI, Akurathi G, Le VH, Lewis EA (December 2013). "The effect of pyridyl substituents on the thermodynamics of porphyrin binding to G-quadruplex DNA". Bioorganic & Medicinal Chemistry. 21 (23): 7515–22. doi:10.1016/j.bmc.2013.09.036. PMID 24148836.
외부 링크
- 나노포어 및 Aptamer 바이오센서 그룹{NAB 그룹}
4중 웹 사이트
- G-Quadruplex World – G-Quadruplex 분야에서 일하는 사람들에게 출판물 및 기타 관심 정보를 소개하는 웹 사이트
- Greglist – 잠재적인 G4D 조절 유전자를 나열하는 데이터베이스
- Fourplex 정보 데이터베이스:IGIB의 쿼드베이스
- GRSDB - RNA 처리 사이트 근처에 있는 G-쿼드루플렉스의 데이터베이스.
- GRS_UTRDB Wayback Machine에서 아카이브된 2011-07-20 - UTRs의 G-Quadruplex 데이터베이스.
- G-쿼드플렉스 리소스 사이트
- 비 B DB의 비 B 모티브 검색 도구 - 사용자의 DNA 배열에서 모티브를 형성하는 G-4중플렉스 및 기타 비 B DNA를 예측하는 웹 서버.
G-쿼드플렉스 모티브를 예측하는 도구
- QGRS Mapper: Bagga 그룹의 뉴클레오티드 배열과 NCBI 유전자의 G-쿼드루플렉스를 예측하기 위한 웹 기반 애플리케이션.
- 쿼드파인더:Maiti의 그룹, IGIB, Delhi, India의 DNA/RNA 배열에서 G 4중 모티브 예측 및 분석 도구[영구 데드링크]
- [1] Mergny 그룹의 G4Hunter이지만 사용자는 R에서 코드를 실행해야 합니다.
- [2] pqsfinder: R에서 잠재적인 4중첩 생성 시퀀스를 위한 완전하고 불완전한 검색 도구입니다.
- [3] pqsfinder: 최신 R/Bioconductor 패키지를 사용한 온라인 검색 도구