박테리오파지
Bacteriophage
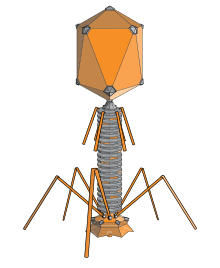

박테리오파지(/bækˈtʊriɪfeʒdʒ/)는 박테리아와 고균 내에서 감염되고 복제되는 두플로드나비리아 바이러스입니다.이 용어는 박테리아와 그리스어 파게인(phagein)에서 유래되었는데, 이는 "식욕을 한다"는 뜻입니다.박테리오파지는 DNA 또는 RNA 게놈을 캡슐화하는 단백질로 구성되어 있으며 단순하거나 정교한 구조를 가지고 있을 수 있습니다.그들의 게놈은 적게는 4개의 유전자(예: MS2)와 많게는 수백 개의 유전자를 암호화할 수 있습니다.파지는 세포질에 게놈을 주입한 후에 박테리아 내에서 복제됩니다.
박테리오파지는 생물권에서 [2]가장 흔하고 다양한 개체 중 하나입니다.박테리오파지는 어디에나 있는 바이러스로, 박테리아가 존재하는 곳에서 발견됩니다.지구상에 박테리아를 포함한 지구상의 모든 생물체를 [3]합친 것보다 많은 10개 이상의31 박테리오파지가 있는 것으로 추정됩니다.바이러스는 세계 해양의 물기둥에서 가장 풍부한 생물체이며,[5] [4]원핵생물 다음으로 두 번째로 큰 바이오매스 성분으로, 표면의 미생물 매트에서 밀리리터당 최대 9x10개의8 비리온이 발견되었으며,[6] 파지에 의해 해양 세균의 70%까지 감염될 수 있습니다.
파지는 [7][8]20세기 후반부터 프랑스뿐만 아니라 구소련과 중유럽에서 항생제의 대안으로 사용되어 왔습니다.그들은 많은 박테리아의 다제내성 균주에 대한 가능한 치료법으로 간주됩니다(파지 [9][10][11][12]치료 참조).
파지는 파지 암호화된 단백질의 세균 발현을 통해 간접적으로 그리고 선천적 면역과 세균 [13]제거에 직접적으로 영향을 줌으로써 면역 체계와 상호작용하는 것으로 알려져 있습니다.페이지-호스트 상호작용은 점점 더 중요한 [14]연구 분야가 되고 있습니다.
분류
박테리오파지는 생물권에서 풍부하게 발생하며, 유전체와 생활방식이 다릅니다.파지는 형태학과 핵산에 따라 국제바이러스분류위원회(ICTV)에 의해 분류됩니다.


| 주문 | 가족 | 형태학 | 핵산 | 예 |
|---|---|---|---|---|
| 벨프리비랄레스 | 투리비루스과 | 포락선, 등각선 | 선형 dsDNA | |
| 카우도비랄레스속 | 아커만비루스과 | 껍질이 없는 수축성이 있는 꼬리 | 선형 dsDNA | |
| 아우토키비루스과 | 비포장, 비수축성 꼬리 (짧은) | 선형 dsDNA | ||
| 체이스비루스과 | 선형 dsDNA | |||
| 데메렉비루스과 | 선형 dsDNA | |||
| 드렉슬레르비루스과 | 선형 dsDNA | |||
| 구엔리비르과 | 선형 dsDNA | |||
| 히렐레비루스과 | 껍질이 없는 수축성이 있는 꼬리 | 선형 dsDNA | ||
| 묘비루스과 | 껍질이 없는 수축성이 있는 꼬리 | 선형 dsDNA | T4, 뮤, P1, P2 | |
| 시포비루스과 | 비포장, 비수축성 꼬리 (긴 | 선형 dsDNA | ◦,T5,HK97,N15 | |
| 포도비루스과 | 비포장, 비수축성 꼬리 (짧은) | 선형 dsDNA | T7, T3, Φ29, P22 | |
| 산트리비루스과 | 선형 dsDNA | |||
| 살라스마비리과 | 선형 dsDNA | |||
| 스키토비루스과 | 선형 dsDNA | |||
| 조벨비루스과 | 선형 dsDNA | |||
| 할로파니비랄레스 | 스파에롤리포비루스과 | 포락선, 등각선 | 선형 dsDNA | |
| 시물로바이러스과 | 포락선, 등각선 | 선형 dsDNA | ||
| 맛슈시타비루스과 | 포락선, 등각선 | 선형 dsDNA | ||
| 할로루비랄레스 | 플레올리포비루스과 | 포락선, 다형 | 원형 ssDNA, 원형 dsDNA 또는 선형 dsDNA | |
| 칼라마비랄레스 | 텍티비루스과 | 봉투 없음, 등각 | 선형 dsDNA | |
| 리가멘비랄레스 | 리포트릭스비루스과 | 봉 모양의 봉 모양의 봉 | 선형 dsDNA | 아시디아누스 필라멘트 바이러스 1 |
| 루디비르과 | 봉투 미장착 봉형 | 선형 dsDNA | Sulfolobus Islandicus 막대모양 바이러스 1 | |
| 민디비랄레스 | 시스토비루스과 | 포박, 구형 | 선형 dsRNA | Ⅱ6 |
| 노르치비랄레스 | 앳킨스비루스과 | 봉투 없음, 등각 | 선형 ssRNA | |
| 두인비루스과 | 봉투 없음, 등각 | 선형 ssRNA | ||
| 피어스비루스과 | 봉투 없음, 등각 | 선형 ssRNA | MS2, Qβ | |
| 솔스피비루스과 | 봉투 없음, 등각 | 선형 ssRNA | ||
| 쁘띠비랄레스 | 마이크로비루스과 | 봉투 없음, 등각 | 원형 ssDNA | ΔX174 |
| 프리마비랄레스속 | 트리스트로마비루스과 | 봉 모양의 봉 모양의 봉 | 선형 dsDNA | |
| 티믈로비랄레스 | 블루메비루스과 | 봉투 없음, 등각 | 선형 ssRNA | |
| 스타이츠비루스과 | 봉투 없음, 등각 | 선형 ssRNA | ||
| 투불라비랄레스 | 이노비리과 | 봉투 미포함, 필라멘트 | 원형 ssDNA | M13 |
| 폴리노비루스과 | 봉투 미포함, 필라멘트 | 원형 ssDNA | ||
| 플렉트로비루스과 | 봉투 미포함, 필라멘트 | 원형 ssDNA | ||
| 비나비랄레스 | 코르티코비루스과 | 봉투 없음, 등각 | 원형 dsDNA | PM2 |
| 두르나비랄레스 | 피코비르나바이러스과 (제안) | 봉투 없음, 등각 | 선형 dsRNA | |
| 미할당 | 암풀라비루스과 | 병 모양의 봉투 | 선형 dsDNA | |
| 오토리키비루스과 | 봉투 없음, 등각 | 선형 dsDNA | ||
| 비카우다비루스과 | 봉투없음 레몬모양 | 원형 dsDNA | ||
| 클라바비루스과 | 봉투 미장착 봉형 | 원형 dsDNA | ||
| 핀레이크비루스과 | 봉투 없음, 등각 | 원형 ssDNA | 플립.[15] | |
| 푸셀로바이러스과 | 봉투없음 레몬모양 | 원형 dsDNA | ||
| 글로불로바이러스과 | 포락선, 등각선 | 선형 dsDNA | ||
| 구타비루스과 | 외피, 난형 | 원형 dsDNA | ||
| 할스피비루스과 | 봉투없음 레몬모양 | 선형 dsDNA | ||
| 플라스마비리과 | 포락선, 다형 | 원형 dsDNA | ||
| 포르토글로보비루스과 | 포락선, 등각선 | 원형 dsDNA | ||
| 타스피비루스과 | 봉투없음 레몬모양 | 선형 dsDNA | ||
| 스피라비루스과 | 봉투 미포함 막대형 | 원형 ssDNA |
피코비르나바이러스과의 구성원들은 박테리아를 감염시키지만 [16]포유류는 감염시키지 않는다는 의견이 있습니다.
또한 레비비리케스속("Leviviricetes")에는 분류되지 않은 속들이 많이 있습니다.선형 ssRNA 유전체를[17] 포함하는 침파바이러스, 호글리바이러스, 마흐라바이러스, 메이즈바이러스, 니체바이러스, 스컬루비루스, 스크럽노바이러스, 테티바이러스 및 위누나바이러스는 선형 dsDNA 유전체를 포함하는 카우도비루스목의 할당되지 않은 릴리바이러스 속.
역사

1896년, 어니스트 핸버리 행킨은 인도의 갠지스강과 야무나강의 물속에 있는 어떤 것이 콜레라에 대한 두드러진 항균작용을 가지고 있고 그것은 아주 미세한 자기 [18]필터를 통과할 수 있다고 보고했습니다.1915년, 영국 세균학자인 런던 브라운 연구소의 책임자인 프레드릭 트워트는 박테리아를 감염시키고 죽이는 작은 박테리아를 발견했습니다.그는 에이전트가 다음 중 하나일 것이라고 믿었습니다.
Twort의 연구는 자금 부족과 항생제의 발견뿐만 아니라 제1차 세계대전의 시작으로 중단되었습니다.
독자적으로 프랑스계 캐나다 미생물학자 펠릭스 드'1917년 9월 3일 파리의 파스퇴르 연구소에서 근무하던 헤렐은 "이질균의 보이지 않는 길항성 미생물"을 발견했다고 발표했습니다.데렐에게 그의 발견의 본질에 대해서는 의문의 여지가 없었습니다: "순식간에 나는 이해했습니다: 나의 명확한 점들을 야기한 것은 사실 보이지 않는 미생물이었습니다...박테리아에 [20]기생하는 바이러스"라고 말했습니다. 데렐은 이 바이러스를 박테리오파지, 박테리아를 잡아먹는 동물이라고 불렀습니다. (그리스어 파지인에서 유래한 말로 "식욕하다"라는 뜻입니다.그는 또한 이질을 앓고 있던 [21]한 남자가 박테리오파지에 의해 건강하게 회복되었다는 극적인 이야기를 기록했습니다.D'였습니다.박테리오파지에 대한 많은 연구를 수행하고 파지 [22]요법의 개념을 소개한 Herelle.1919년 프랑스 파리에서 데렐은 박테리오파지의 첫 번째 임상 적용을 실시하였고,[23] 1922년 미국에서 처음으로 보고된 사용 사례.
파지 연구에 대하여 노벨상을 수여함.
1969년, 막스 델브뤼크, 알프레드 허쉬, 살바도르 루리아는 바이러스의 복제와 그들의 유전자 [24]구조를 발견한 공로로 노벨 생리학·의학상을 수상했습니다.특히 1952년 허쉬-체이스 실험의 기여자로서 허쉬의 연구는 단백질이 아닌 DNA가 생명체의 유전적 물질이라는 설득력 있는 증거를 제공했습니다.델브뤼크와 루리아는 루리아-델브뤼크 실험을 수행했는데, 이 실험은 통계적으로 박테리아의 돌연변이가 무작위로 발생하여 라마르크 원리가 아닌 다윈 [citation needed]원리를 따른다는 것을 증명했습니다.
사용하다
파지테라피

파지는 항균제로 밝혀졌으며, 구 조지아 소비에트 공화국(조르지 엘리아바가 박테리오파지의 공동 발견자인 펠릭스 디'의 도움을 받아 그곳을 개척함)에서 사용되었습니다.1920년대와 1930년대에 세균 감염 치료를 위해 헤렐(Hérelle).그들은 붉은 [citation needed]군대의 병사들을 치료하는 것을 포함하여 광범위하게 사용되었습니다.그러나 서양에서 일반적으로 사용하기 위해 버려졌던 이유는 다음과 같습니다.
- 항생제가 발견되고 널리 판매되었습니다.그들은 만들고, 보관하고, 처방하기가 더 쉬웠습니다.
- 파지에 대한 의학적 실험이 수행되었지만, 파지에 대한 기본적인 이해 부족은 이러한 [25]실험의 타당성에 의문을 제기했습니다.
- 소련에서 연구에 대한 발표는 주로 러시아어나 조지아어로 이루어졌고 여러 해 동안 국제적으로 지켜지지 않았습니다.
러시아,[26] 그루지야, 중부와 동유럽의 다른 지역에서는 냉전이 끝난 이후에도 파지의 사용이 계속되고 있습니다.첫 번째 조절된 무작위 이중맹검 임상시험은 2009년 6월 Journal of Wound Care에서 보고되었는데, 이 시험은 박테리오파지 칵테일의 안전성과 인간 [27]환자의 감염된 다리 정맥 궤양을 치료하기 위한 효능을 평가했습니다.FDA는 이 연구를 임상 1상 시험으로 승인했습니다.그 연구의 결과는 박테리오파지의 치료적 적용의 안전성을 증명했지만, 효과를 보여주지는 못했습니다.저자들은 표준 상처 치료의 일부인 특정 화학물질(예: 락토페린 또는 은)의 사용이 박테리오파지 [27]생존을 방해했을 수 있다고 설명했습니다.그 직후 서유럽에서 [28]또 다른 대조군 임상시험(Pseudomonas aeruginosa에 의한 귀 감염의 치료)이 2009년 8월 임상이비인후과 저널에 보고되었습니다.이 연구는 박테리오파지 제제가 인간의 만성 귀 감염 치료에 안전하고 효과적이었다는 결론을 내렸습니다.또한, 박테리오파지의 감염된 화상 및 상처, [28]낭포성 섬유증 관련 폐 감염 등과 같은 다양한 질병에 대한 효능을 평가하는 동물 및 기타 실험적 임상 시험들이 다수 진행되어 왔습니다.반면, Inoviridae의 파지는 폐렴과 낭포성 섬유증에 관련된 생물막을 복잡하게 만들고 질병을 근절하기 위한 약물로부터 박테리아를 보호하여 지속적인 [29]감염을 촉진하는 것으로 나타났습니다.
한편, 박테리오파지 연구자들은 항생제 내성을 극복하기 위해 조작된 바이러스를 개발하고, 바이오필름 매트릭스를 분해하는 효소, 파지 구조 단백질, 세균 [5][6][7]세포벽의 용해를 담당하는 효소를 코딩하는 파지 유전자를 조작해 왔습니다.크기가 작고 꼬리가 짧은 T4 파지가 인체 [30]내 대장균 검출에 도움이 될 수 있다는 결과가 나왔습니다.
파지 칵테일의 치료 효능은 다제내성(MDR) 바우만니의 비강 감염을 동반한 마우스 모델에서 평가되었습니다.파지 칵테일로 치료한 쥐들은 감염 [31]후 7일 동안 치료받지 않은 쥐들에 비해 2.3배나 높은 생존율을 보였습니다.2017년 MDRA 바우만니에 의해 췌장이 손상된 환자는 여러 항생제를 투여받았지만, 이에도 불구하고 환자의 건강은 4개월 동안 계속 악화되었습니다.효과적인 항생제 없이 환자는 MDR A. 바우만니에 대해 효과적인 것으로 입증된 9가지 다른 파지가 포함된 파지 칵테일을 사용하여 파지 치료를 받았습니다.일단 이 치료를 받으면 환자의 하향 임상 궤도가 역전되어 [32]건강 상태로 돌아갔습니다.
D'Herele는 "세균이 번성하는 곳, 하수구, 파이프에서 나오는 쓰레기를 잡는 강, 그리고 회복 중인 [33]환자의 대변에서 발견된다는 것을 빠르게 알게 되었습니다."여기에는 인도의 갠지스 [34]강을 포함해 전통적으로 치유의 힘을 가진 것으로 여겨졌던 강들이 포함됩니다.
다른.
식품 산업 – 파지는 식품을 안전하게 하고 부패균을 [35]미리 예방하는 데 점점 더 많이 사용되고 있습니다.2006년부터 미국 식품의약국(FDA)과 미국 농무부(USDA)는 몇몇 박테리오파지 제품을 승인했습니다.LMP-102 (Intralytix)는 바로 먹을 수 있는 가금류 및 육류 제품을 처리하기 위해 승인되었습니다.같은 해 FDA는 리스테리아 모노사이토제네스 박테리아를 죽이기 위해 치즈에 박테리오파지를 사용하여 LISTEX를 승인했습니다. 일반적으로 GRAS(안전)[36] 상태로 인정되는 것입니다.2007년 7월, 같은 박테리오파지가 모든 [37]식품에 사용되도록 승인되었습니다.2011년 USDA는 LISTEX가 깨끗한 라벨 처리 보조제이며 [38]USDA에 포함되어 있음을 확인했습니다.식품 안전 분야의 연구는 다양한 [citation needed]식품에 있는 다른 식품 매개 병원체를 통제하기 위해 용균 파지가 실행 가능한 선택인지에 대한 연구를 계속하고 있습니다.
진단 – 2011년 FDA는 시험관 내 [39]진단을 위한 최초의 박테리오파지 기반 제품을 승인했습니다.KeyPath MRSA/MSSA 혈액 배양 검사는 양성 혈액 배양물에서 황색포도상구균을 검출하고 메티실린 내성 또는 민감도를 결정하기 위해 박테리오파지 혼합물을 사용합니다.표준 미생물 식별 및 민감도 검사 방법의 경우 2~3일에 비해 약 5시간 만에 결과가 반환됩니다.이것은 [40]FDA에 의해 승인된 최초의 가속 항생제 감수성 시험이었습니다.
생물무기와 독소 대응 – 서방의 정부 기관들은 몇 년 동안 탄저균과 보툴리누스 [41]중독과 같은 생물무기와 독소 대응을 위한 파지를 이용하기 위해 조지아와 구 소련에 도움을 요청해 왔습니다.미국의 연구 그룹들 사이에서 발전이 계속되고 있습니다. 다른 용도로는 식물과 채소 생산물을 부패와 세균성 질병의 확산으로부터 보호하기 위한 원예에서의 스프레이 적용이 있습니다.박테리오파지에 대한 다른 적용은 예를 들어 병원에서 환경 표면에 대한 생물학적 살상 및 임상 환경에서 사용하기 전 카테터 및 의료 장치에 대한 예방적 치료입니다.수술을 위한 유니폼, 커튼, 또는 심지어 봉합과 같은 건조한 표면에 적용되는 파지에 대한 기술은 현재 존재합니다.임상 이비인후과에서[28] 보고된 임상시험들은 중이염을 가진 애완견의 수의학적 치료에 성공을 보여줍니다.
SEPTIC 박테리아 감지 및 식별 방법은 파지 감염 시 이온 방출 및 그 역학을 이용하고 [42]높은 특이도와 검출 속도를 제공합니다.
파지 디스플레이는 표면 단백질과 연결된 다양한 펩티드를 가진 파지 라이브러리를 포함하는 파지의 다른 용도입니다.각 파지 유전체는 표면에 표시된 단백질의 변이체(따라서 이름)를 암호화하여 펩타이드 변이체와 그 암호화 유전자 사이의 연결을 제공합니다.라이브러리의 변형 파지는 고정화된 분자(예를 들어, 보툴리즘 독소)에 대한 결합 친화성을 통해 선택되어 중화시킬 수 있습니다.경계가 있고 선택된 파지는 민감한 박테리아 균주를 재감염함으로써 증식될 수 있으므로 추가 연구를 위해 그 [43]안에 암호화된 펩타이드를 회수할 수 있습니다.
항균성 약물 발견 – 파지 단백질은 종종 항균성을 가지며 펩티도미메틱, [44]즉 펩타이드를 모방하는 약물의 리드 역할을 할 수 있습니다.파지-리간드 기술은 파지 단백질을 박테리아 및 박테리아 성분(예: 내독소)의 결합 및 박테리아 용해와 [45]같은 다양한 응용에 사용합니다.
기초 연구 – 박테리오파지는 진화와 [46]생태의 원리를 연구하기 위한 중요한 모델 생물입니다.
해악츠
낙농업
환경에 존재하는 박테리오파지는 치즈가 발효되지 못하게 할 수 있습니다.이를 방지하기 위해 혼합형 선발 문화와 문화 순환 체제를 [47]사용할 수 있습니다.배양 미생물의 유전공학은 특히 락토코커스 락티스와 스트렙토코커스 써모필러스를 포함하며, 파지 내성을 개선하기 위한 유전학적 분석과 수정을 위해 연구되어 왔습니다.이것은 특히 플라스미드 및 재조합 염색체 [48][35]변형에 초점을 맞추고 있습니다.
일부 연구는 박테리오파지가 식품 매개 병원체에 대한 항균제로서의 가능성과 유제품 산업에서의 생물막 형성에 초점을 맞추고 있습니다.항생제 내성의 확산이 유제품 업계의 주요 관심사이기 때문에, 파지는 유망한 [49]대안의 역할을 할 수 있습니다.
복제

박테리오파지의 수명 주기는 분해 주기 또는 용해 주기인 경향이 있습니다.또한 일부 페이지는 유사 용종성 [13]행동을 보여줍니다.
T4 파지와 같은 용해성 파지를 사용하면 세균 세포는 깨져서 열린 상태가 되고 비리온을 즉시 복제한 후 파괴됩니다.세포가 파괴되자마자, 파지 자손은 [13]감염시킬 새로운 숙주를 찾을 수 있습니다.리틱 파지는 파지 치료에 더 적합합니다.일부 용해된 파지는 용해 억제(lysis inhibitation)로 알려진 현상을 겪는데, 세포 외 파지 농도가 높으면 완성된 파지 자손이 즉시 세포 밖으로 빠져나가지 않습니다.이 메커니즘은 저온 파지가 휴면하는 것과 동일하지 않고 보통 일시적입니다.[citation needed]
반대로, 라이소겐 사이클은 숙주 세포의 즉각적인 용해를 초래하지 않습니다.라이소제닉을 겪을 수 있는 파지는 온대 파지로 알려져 있습니다.그들의 바이러스 게놈은 숙주 DNA와 통합되어 상대적으로 해롭지 않게 복제되거나 심지어 플라스미드로 확립될 수도 있습니다.이 바이러스는 숙주 상태가 악화될 때까지 휴면 상태를 유지하며, 아마도 영양소가 고갈되어 있을 것이고, 그 후에 내인성 파지(프로파지로 알려져 있음)가 활성화됩니다.이 시점에서 이들은 생식 주기를 시작하여 숙주 세포의 용해를 초래합니다.라이소제닉 사이클이 숙주 세포가 계속해서 생존하고 번식할 수 있게 해주기 때문에, 바이러스는 세포의 모든 자손에게 복제됩니다.용질 주기와 용질 주기를 따르는 것으로 알려진 박테리오파지의 예로는 대장균의 [50]파지 람다가 있습니다.
때때로 프로파지는 세포유전체 변환이라 불리는 현상에서 박테리아 게놈에 새로운 기능을 추가함으로써 휴면 상태에 있는 동안 숙주 박테리아에 이점을 제공할 수도 있습니다.박테리오파지에 의한 코리네박테리움 디프테리아나 비브리오 콜레라균의 무해한 균주가 각각 디프테리아나 콜레라를 일으키는 맹독성 균주로 전환되는 것이 그 예입니다.[51][52]이러한 독소-인코딩 프로파지를 표적화함으로써 특정 세균 감염을 방지하기 위한 전략이 [53]제안되었습니다.
부착 및 관통
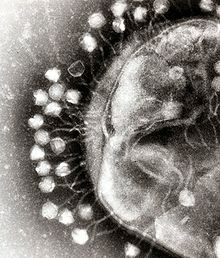
박테리아 세포는 면역 숙주 방어와 [54]항생제 둘 다로부터 박테리아 세포를 보호하는 중요한 독성 요인인 다당류 세포벽에 의해 보호됩니다.숙주 세포에 들어가기 위해, 박테리오파지는 지방 다당류, 테이초산, 단백질, 또는 심지어 편모를 포함한 박테리아의 표면의 특정한 수용체에 결합합니다. 이 특이성은 박테리오파지가 그들이 결합할 수 있는 수용체를 가지고 있는 특정한 박테리아만 감염시킬 수 있다는 것을 의미하고, 이것은 다시 그 박테리아의 숙주 범위를 결정합니다. 다당류 분해 효소는 촘촘하게 프로그램된 파지 감염 [citation needed]과정의 초기 단계에서 숙주의 캡슐 외층을 효소적으로 분해하는 비리온 관련 단백질입니다.숙주 성장 조건은 또한 파지의 부착 [55]및 침입 능력에 영향을 미칩니다.파지 비리온은 독립적으로 이동하지 않기 때문에 혈액, 림프관 순환, 관개, 토양수 [citation needed]등 용액에 있을 때 정확한 수용체와의 무작위 접촉에 의존해야 합니다.
마이오바이러스 박테리오파지는 피하주사기와 같은 움직임을 이용하여 그들의 유전자 물질을 세포에 주입합니다.적절한 수용체와 접촉한 후, 꼬리 섬유는 베이스 플레이트를 세포 표면에 더 가깝게 만들기 위해 굴곡합니다.이것은 가역적 결합이라고 알려져 있습니다.일단 완전히 붙으면, 비가역적 결합이 시작되고 아마도 [6]꼬리에 존재하는 ATP의 도움으로 꼬리가 수축하여 박테리아 [56]막을 통해 유전 물질을 주입합니다.주입은 옆으로 가서 세포에 더 가까이 수축하고 뒤로 밀어 올림으로써 샤프트에서 일종의 굽힘 운동을 통해 이루어집니다.포도바이러스는 마이오바이러스처럼 긴 꼬리의 피복이 없기 때문에, 대신에 그들은 유전 물질을 삽입하기 전에 세포막의 한 부분을 분해하기 위해 작고 이빨처럼 생긴 꼬리 섬유를 효소적으로 사용합니다.
단백질과 핵산의 합성
몇 분 안에 박테리아 리보솜은 바이러스 mRNA를 단백질로 변환하기 시작합니다.RNA 기반 파지의 경우, RNA 복제효소는 공정 초기에 합성됩니다.단백질은 박테리아 RNA 중합효소를 변형시켜서 바이러스 mRNA를 우선적으로 전사합니다.숙주의 정상적인 단백질과 핵산 합성은 방해받고, 대신 바이러스 제품을 생산하도록 강요됩니다.이 제품들은 계속해서 세포 내에서 새로운 비리온의 일부가 되고, 새로운 비리온의 집합에 기여하는 조력자 단백질, 또는 세포 용해에 관여하는 단백질이 됩니다.1972년, 월터 피어스(벨기에 겐트 대학교)는 최초로 유전자의 완전한 뉴클레오타이드 서열을 확립했고 1976년 박테리오파지 [57]MS2의 바이러스 게놈을 확립했습니다. 일부 dsDNA 박테리오파지는 파지 감염 동안 단백질 번역을 조절하는 것으로 생각되는 [58]리보솜 단백질을 암호화합니다.
비리온 어셈블리
T4 파지의 경우, 새로운 바이러스 입자의 구성은 파지 형태 생성 [59]동안 촉매 작용하는 헬퍼 단백질의 도움을 수반합니다.베이스 플레이트가 먼저 조립되고 그 위에 꼬리가 세워집니다.별도로 구성된 헤드 캡시드는 자발적으로 테일과 조립됩니다.파지 T4 비리온이 조립되는 동안 파지 유전자에 의해 암호화된 형태유전학적 단백질은 특징적인 서열로 서로 상호작용합니다.바이러스 감염 동안 생성된 이러한 단백질 각각의 양에서 적절한 균형을 유지하는 것은 정상적인 파지 T4 형태 생성에 [60]매우 중요한 것으로 보입니다.DNA는 머리 [61]속에 효율적으로 채워집니다.이 모든 과정은 약 15분 정도 걸립니다.
비리온 방출
파지는 세포 용해를 통해, 압출을 통해, 또는 경우에 따라서는 싹이 트기도 합니다.꼬리를 가진 파지에 의해 용해는 세포벽 펩티도글리칸을 공격하고 분해하는 엔돌리신이라고 불리는 효소에 의해 이루어집니다.완전히 다른 파지 종류인 필라멘트 파지는 숙주 세포가 계속해서 새로운 바이러스 입자를 분비하도록 만듭니다.방출된 바이러스는 무료로 설명되며, 결함이 없는 한 새로운 박테리아를 감염시킬 수 있습니다.싹은 특정 마이코플라즈마 파지와 관련이 있습니다.비리온 방출과는 대조적으로, 리소제닉 사이클을 보여주는 페이지는 숙주를 죽이지 않고,[62] 프로파지로서 장기 거주자가 됩니다.
의사소통
2017년의 연구는 박테리오파지 Ⅱ3T가 숙주 박테리아를 죽이는 대신 다른 박테리오파지들이 휴면 상태에 있도록 신호를 보내는 짧은 바이러스 단백질을 만든다고 밝혔습니다.중재자는 [63][64]이 단백질을 발견한 연구자들에 의해 붙여진 이름입니다.
유전체 구조
환경에 있는 수백만 개의 다른 파지들을 고려할 때 파지 유전체들은 다양한 형태와 크기로 나옵니다.MS2와 같은 RNA 파지는 몇 킬로 염기로 가장 작은 유전체를 가지고 있습니다.하지만, T4와 같은 몇몇 DNA 파지들은 수백 개의 유전자를 가진 큰 유전체를 가지고 있을지도 모릅니다;[65] 캡시드의 크기와 모양은 유전체의 크기에 따라 다릅니다.가장 큰 박테리오파지 유전체는 735kb [66]크기에 이릅니다.

박테리오파지 유전체는 매우 모자이크일 수 있습니다. 즉, 많은 파지 종의 게놈은 수많은 개별 모듈로 구성되어 있는 것으로 보입니다.이러한 모듈은 다른 파지 종에서 다른 배열로 발견될 수 있습니다.마이코박테리오파지, 마이코박테리오파지, 마이코박테리오파지, 마이코박테리오파지는 이러한 모자이크의 훌륭한 예를 제공합니다.이러한 마이코박테리오파지에서, 유전적 분류는 부위-특이적 재조합 및 비합법적 재조합의 반복된 예(세균 숙주 유전자 [68]서열의 파지 유전체 획득의 결과)의 결과일 수 있습니다.박테리아 바이러스의 게놈을 형성하는 진화적 메커니즘은 다른 가족들 사이에서 다양하며 핵산의 종류, 바이러스 구조의 특징, 바이러스 수명 [69]주기의 모드에 따라 다릅니다.
일부 해양 로제오박터 파지는 게놈 DNA에 디옥시티미딘(dT) 대신 디옥시우리딘(dU)을 함유하고 있습니다.이 특이한 성분이 제한 엔도뉴클레오타이드와 CRISPR/Cas 시스템과 같은 박테리아 방어 메커니즘을 회피하는 메커니즘이라는 증거가 있습니다. 이 메커니즘은 침입하는 파지 내에서 서열을 인식하고 절단하도록 진화하여 비활성화시킵니다.다른 파지들은 특이한 뉴클레오티드를 사용하는 것으로 오랫동안 알려져 왔습니다.1963년, Takahashi와 Marmur는 [70]유전체에서 dT를 치환하는 dU를 갖는 Bacillus 파지를 확인하였고, 1977년 Kirnos et al.은 아데닌([71]A) 대신 2-아미노아데닌(Z)을 포함하는 시아노파지를 확인하였습니다.
시스템생물학
시스템 생물학 분야는 보통 계산 도구와 [72]모델링을 사용하여 유기체 내의 복잡한 상호 작용 네트워크를 조사합니다.예를 들어, 박테리아 숙주 세포로 들어가는 파지 유전체는 수많은 숙주 유전자의 발현이나 숙주의 대사에 영향을 미칠 수 있는 수백 개의 파지 단백질을 발현할 수 있습니다.이 모든 복잡한 상호작용은 컴퓨터 [72]모델에서 설명되고 시뮬레이션될 수 있습니다.
예를 들어, 온대 파지 PaP3에 의한 녹농균 감염은 숙주 유전자의 38%(2160/5633)의 발현을 변화시켰습니다.이러한 효과들 중 많은 것들이 아마도 간접적일 것이고,[73] 따라서 박테리아와 파지 사이의 직접적인 상호작용을 확인하는 것이 과제가 됩니다.
파지와 숙주 사이의 단백질-단백질 상호작용을 지도화하기 위한 여러 시도가 있었습니다.예를 들어, 박테리오파지 람다는 숙주인 대장균과 수십 개의 상호작용에 의해 상호작용하는 것으로 밝혀졌습니다.다시 말하지만, 이러한 많은 상호작용의 중요성은 여전히 명확하지 않지만, 이 연구들은 여러 주요 상호작용과 많은 간접적 상호작용이 그 역할이 [74]특성화되지 않은 채로 남아 있다는 것을 암시합니다.
호스트 저항
박테리오파지는 박테리아에 대한 주요한 위협이고 원핵생물은 감염을 차단하거나 숙주 세포 내에서 박테리오파지의 복제를 차단하기 위해 수많은 메커니즘을 진화시켜 왔습니다.CRISPR 시스템은 레트론과 [75]이들에 의해 암호화된 항독소 시스템과 같은 메커니즘 중 하나입니다.Thoeris 방어 시스템은 NAD+[76] 분해를 통한 박테리아 안티파지 내성에 대한 독특한 전략을 전개하는 것으로 알려져 있습니다.
박테리오파지-숙주 공생
온대 파지는 염색체 외 에피솜으로 숙주에 유전적 물질을 통합하는 박테리오파지(bacteriophage) 또는 지질학적 [77][78][79]주기 동안의 프로파지(prophage)입니다.일부 온대지방은 항생제 내성 유전자(ARG)[78][80]의 전달 또는 도입을 통한 항생제 내성 부여, 식세포증([83][84][85]phagocytosis)[81][82]으로부터 숙주를 보호, 초감염 배제를 통한 [77][86]2차 감염으로부터 숙주를 보호, 숙주 병원성 강화,또는 박테리아의 대사나 [87][88][89][90]성장을 강화할 수 있습니다.박테리오파지-숙주 공생은 파지 [91]게놈을 수동적으로 복제하면서 선택적인 이점을 제공함으로써 박테리아에 도움을 줄 수 있습니다.
환경에서
메타게노믹스는 [92]이전에는 불가능했던 박테리오파지를 수중에서 검출할 수 있게 해주었습니다.
또한 박테리오파지는 특히 지표수와 지하수 상호작용이 일어나는 하천 시스템에서 수문 추적 및 모델링에 사용되어 왔습니다.파지는 지하수를 통과할 때 흡수가 현저히 적고 매우 낮은 [93]농도에서 쉽게 검출되기 때문에 보다 일반적인 염료 마커보다 사용이 선호됩니다.오염되지 않은 물은 [94]ml 당 약 2×108 박테리오파지를 함유할 수 있습니다.
박테리오파지는 주로 [95]형질전환을 통해 자연 환경에서 수평적인 유전자 전달에 광범위하게 기여하는 것으로 생각됩니다.메타게노믹스 기반의 연구는 또한 다양한 환경에서 온 바이롬이 다제내성을 [96]유발할 수 있는 유전자를 포함하여 항생제 내성 유전자를 가지고 있다는 것을 밝혀냈습니다.
인휴먼
비록 파지가 사람을 감염시키지는 않지만, 우리의 광범위한 마이크로바이옴을 고려할 때, 인체에는 셀 수 없이 많은 파지 입자들이 있습니다.우리의 파지 인구는 "건강한 내장 파지홈"(HGP)과 "질병에 걸린 인간 파지홈"(DHP)[97]을 포함하여 인간 파지홈이라고 불려왔습니다.건강한 인간의 능동적 파지좀(즉, 복제되지 않는 통합된 프로파지와 대조적으로 능동적으로 복제하는 것)은 수십에서 수천 개의 [98]다양한 바이러스로 구성되는 것으로 추정됩니다.박테리오파지와 박테리아가 인간 내장 마이크로바이옴에서 적대적으로 그리고 [99]유익하게 상호작용한다는 증거가 있습니다.
예비 연구에 따르면 일반 박테리오파지는 건강한 개인의 평균 62%에서 발견되는 반면 궤양성 대장염(UC)과 크론병(CD)[97] 환자에서는 유병률이 평균 42%와 54% 감소했습니다. [99]노인에게서도 파지의 풍부함이 감소할 수 있습니다.
전세계적으로 발견되는 인간의 장에서 가장 흔한 파지는 crAssage입니다.CrAsphage는 출생 직후에 산모에서 아이로 전송되며, 국소적으로 전송될 수도 있음을 시사하는 몇 가지 증거가 있습니다.각 사용자는 자신만의 고유한 crAsphage 클러스터를 개발합니다.CrAs와 유사한 파지는 [99]인간 이외에도 영장류에도 존재할 수 있습니다.
일반적으로 연구되는 박테리오파지
셀 수 없이 많은 파지 중에서 미생물 유전 초기에 발견된 역사적으로 중요한 파지를 포함하여 세부적으로 연구된 것은 극히 일부에 불과합니다.이것들, 특히 T-phage는 유전자 구조와 기능의 중요한 원리를 발견하는데 도움을 주었습니다.
참고 항목
- 박테리보어
- CrAsphage
- 크리스퍼
- DNA 바이러스
- 대식세포
- 파지생태학
- 파아지 모노그래프 (파아지 및 파아지 관련 모노그래프의 포괄적 목록, 1921-현재)
- 파지미드
- 폴리파지
- RNA 바이러스
- 전환
- 비리오메
- 바이러스, 다른 바이러스를 감염시키는 바이러스
참고문헌
- ^ Padilla-Sanchez V (2021). "Structural Model of Bacteriophage T4". WikiJournal of Science. 4 (1): 5. doi:10.15347/WJS/2021.005. S2CID 238939621.
- ^ a b McGrath S, van Sinderen D, eds. (2007). Bacteriophage: Genetics and Molecular Biology (1st ed.). Caister Academic Press. ISBN 978-1-904455-14-1.
- ^ LaFee S, Buschman H (25 April 2017). "Novel Phage Therapy Saves Patient with Multidrug-Resistant Bacterial Infection". UC Health – UC San Diego. Retrieved 13 May 2018.
- ^ Suttle CA (September 2005). "Viruses in the sea". Nature. 437 (7057): 356–361. Bibcode:2005Natur.437..356S. doi:10.1038/nature04160. PMID 16163346. S2CID 4370363.
- ^ a b Wommack KE, Colwell RR (March 2000). "Virioplankton: viruses in aquatic ecosystems". Microbiology and Molecular Biology Reviews. 64 (1): 69–114. doi:10.1128/MMBR.64.1.69-114.2000. PMC 98987. PMID 10704475.
- ^ a b c m Prescott L (1993). Microbiology. Wm. C. Brown Publishers. ISBN 0-697-01372-3.
- ^ a b – Bunting J (1997). "The Virus that Cures". BBC Horizon. BBC Worldwide Ltd. OCLC 224991186. 러시아와 서양의 파지의학사에 관한 다큐멘터리
- ^ Borrell B, Fishchetti V (August 2012). "Science talk: Phage factor". Scientific American. pp. 80–83. JSTOR 26016042.
- ^ Kortright KE, Chan BK, Koff JL, Turner PE (February 2019). "Phage Therapy: A Renewed Approach to Combat Antibiotic-Resistant Bacteria". Cell Host & Microbe. 25 (2): 219–232. doi:10.1016/j.chom.2019.01.014. PMID 30763536. S2CID 73439131.
- ^ Gordillo Altamirano FL, Barr JJ (April 2019). "Phage Therapy in the Postantibiotic Era". Clinical Microbiology Reviews. 32 (2). doi:10.1128/CMR.00066-18. PMC 6431132. PMID 30651225.
- ^ González-Mora A, Hernández-Pérez J, Iqbal HM, Rito-Palomares M, Benavides J (September 2020). "Bacteriophage-Based Vaccines: A Potent Approach for Antigen Delivery". Vaccines. 8 (3): 504. doi:10.3390/vaccines8030504. PMC 7565293. PMID 32899720.
- ^ Keen EC (2012). "Phage therapy: concept to cure". Frontiers in Microbiology. 3: 238. doi:10.3389/fmicb.2012.00238. PMC 3400130. PMID 22833738.
- ^ a b c Popescu M, Van Belleghem JD, Khosravi A, Bollyky PL (September 2021). "Bacteriophages and the Immune System". Annual Review of Virology. 8 (1): 415–435. doi:10.1146/annurev-virology-091919-074551. PMID 34014761.
- ^ Stone E, Campbell K, Grant I, McAuliffe O (June 2019). "Understanding and Exploiting Phage-Host Interactions". Viruses. 11 (6): 567. doi:10.3390/v11060567. PMC 6630733. PMID 31216787.
- ^ Laanto E, Mäntynen S, De Colibus L, Marjakangas J, Gillum A, Stuart DI, et al. (August 2017). "Virus found in a boreal lake links ssDNA and dsDNA viruses". Proceedings of the National Academy of Sciences of the United States of America. 114 (31): 8378–8383. Bibcode:2017PNAS..114.8378L. doi:10.1073/pnas.1703834114. PMC 5547622. PMID 28716906.
- ^ Krishnamurthy SR, Wang D (March 2018). "Extensive conservation of prokaryotic ribosomal binding sites in known and novel picobirnaviruses". Virology. 516: 108–114. doi:10.1016/j.virol.2018.01.006. PMID 29346073.
- ^ Callanan J, Stockdale SR, Adriaenssens EM, Kuhn JH (January 2021). "Rename one class (Leviviricetes - formerly Allassoviricetes), rename one order (Norzivirales - formerly Levivirales), create one new order (Timlovirales), and expand the class to a total of six families, 420 genera and 883 species". ResearchGate. doi:10.13140/RG.2.2.25363.40481.
- ^ Hankin EH (1896). "L'action bactericide des eaux de la Jumna et du Gange sur le vibrion du cholera". Annales de l'Institut Pasteur (in French). 10: 511–23.
- ^ Twort FW (1915). "An Investigation on the Nature of Ultra-Microscopic Viruses". The Lancet. 186 (4814): 1241–43. doi:10.1016/S0140-6736(01)20383-3.
- ^ d'Hérelles F (1917). "Sur un microbe invisible antagoniste des bacilles dysentériques" (PDF). Comptes Rendus de l'Académie des Sciences de Paris. 165: 373–5. Archived (PDF) from the original on 11 May 2011. Retrieved 5 September 2010.
- ^ d'Hérelles F (1949). "The bacteriophage" (PDF). Science News. 14: 44–59. Retrieved 5 September 2010.
- ^ Keen EC (December 2012). "Felix d'Herelle and our microbial future". Future Microbiology. 7 (12): 1337–1339. doi:10.2217/fmb.12.115. PMID 23231482.
- ^ Aswani VH, Shukla SK (June 2021). "An Early History of Phage Therapy in the United States: Is it Time to Reconsider?". Clinical Medicine & Research. 19 (2): 82–89. doi:10.3121/cmr.2021.1605. PMC 8231696. PMID 34172535.
- ^ "The Nobel Prize in Physiology or Medicine 1969". Nobel Foundation. Retrieved 28 July 2007.
- ^ Kutter E, De Vos D, Gvasalia G, Alavidze Z, Gogokhia L, Kuhl S, Abedon ST (January 2010). "Phage therapy in clinical practice: treatment of human infections". Current Pharmaceutical Biotechnology. 11 (1): 69–86. doi:10.2174/138920110790725401. PMID 20214609. S2CID 31626252.
- ^ Golovin S (2017). "Бактериофаги: убийцы в роли спасителей" [Bacteriophages: killers as saviors]. Наука и жизнь [Nauka I Zhizn (Science and life)] (in Russian) (6): 26–33.
- ^ a b Rhoads DD, Wolcott RD, Kuskowski MA, Wolcott BM, Ward LS, Sulakvelidze A (June 2009). "Bacteriophage therapy of venous leg ulcers in humans: results of a phase I safety trial". Journal of Wound Care. 18 (6): 237–8, 240–3. doi:10.12968/jowc.2009.18.6.42801. PMID 19661847.
- ^ a b c Wright A, Hawkins CH, Anggård EE, Harper DR (August 2009). "A controlled clinical trial of a therapeutic bacteriophage preparation in chronic otitis due to antibiotic-resistant Pseudomonas aeruginosa; a preliminary report of efficacy". Clinical Otolaryngology. 34 (4): 349–357. doi:10.1111/j.1749-4486.2009.01973.x. PMID 19673983.
- ^ Sweere JM, Van Belleghem JD, Ishak H, Bach MS, Popescu M, Sunkari V, et al. (March 2019). "Bacteriophage trigger antiviral immunity and prevent clearance of bacterial infection". Science. 363 (6434): eaat9691. doi:10.1126/science.aat9691. PMC 6656896. PMID 30923196.
- ^ Tawil N, Sacher E, Mandeville R, Meunier M (May 2012). "Surface plasmon resonance detection of E. coli and methicillin-resistant S. aureus using bacteriophages" (PDF). Biosensors & Bioelectronics. 37 (1): 24–29. doi:10.1016/j.bios.2012.04.048. PMID 22609555. Archived (PDF) from the original on 2 February 2023.
- ^ Cha K, Oh HK, Jang JY, Jo Y, Kim WK, Ha GU, et al. (10 April 2018). "Characterization of Two Novel Bacteriophages Infecting Multidrug-Resistant (MDR) Acinetobacter baumannii and Evaluation of Their Therapeutic Efficacy in Vivo". Frontiers in Microbiology. 9: 696. doi:10.3389/fmicb.2018.00696. PMC 5932359. PMID 29755420.
- ^ Schooley RT, Biswas B, Gill JJ, Hernandez-Morales A, Lancaster J, Lessor L, et al. (October 2017). "Development and Use of Personalized Bacteriophage-Based Therapeutic Cocktails To Treat a Patient with a Disseminated Resistant Acinetobacter baumannii Infection". Antimicrobial Agents and Chemotherapy. 61 (10). doi:10.1128/AAC.00954-17. PMC 5610518. PMID 28807909.
- ^ Kuchment A (2012), The Forgotten Cure: The past and future of phage therapy, Springer, p. 11, ISBN 978-1-4614-0250-3
- ^ Deresinski S (April 2009). "Bacteriophage therapy: exploiting smaller fleas". Clinical Infectious Diseases. 48 (8): 1096–1101. doi:10.1086/597405. PMID 19275495.
- ^ a b O'Sullivan L, Bolton D, McAuliffe O, Coffey A (March 2019). "Bacteriophages in Food Applications: From Foe to Friend". Annual Review of Food Science and Technology. Annual Reviews. 10 (1): 151–172. doi:10.1146/annurev-food-032818-121747. PMID 30633564. S2CID 58620015.
- ^ 미국 FDA/CFSAN: 기관 응답서, GRAS 고시 No. 000198
- ^ (미국 FDA/CFSAN: 기관 응답서, GRAS 고시 번호 000218)
- ^ "FSIS Directive 7120: Safe and Suitable Ingredients Used in the Production of Meat, Poultry, and Egg Products" (PDF). Food Safety and Inspection Service. Washington, DC: United States Department of Agriculture. Archived from the original (PDF) on 18 October 2011.
- ^ "FDA 510(k) Premarket Notification". U.S. Food and Drug Administration.
- ^ "FDA clears first test to quickly diagnose and distinguish MRSA and MSSA". U.S. Food and Drug Administration. 6 May 2011. Archived from the original on 5 January 2012.
- ^ Vaisman D (25 May 2007). "Studying anthrax in a Soviet-era lab – with Western funding". The New York Times.
- ^ Dobozi-King M, Seo S, Kim JU, Young R, Cheng M, Kish LB (2005). "Rapid detection and identification of bacteria: SEnsing of Phage-Triggered Ion Cascade (SEPTIC)" (PDF). Journal of Biological Physics and Chemistry. 5: 3–7. doi:10.4024/1050501.jbpc.05.01. Archived from the original (PDF) on 26 September 2018. Retrieved 19 December 2016.
- ^ Smith GP, Petrenko VA (April 1997). "Phage Display". Chemical Reviews. 97 (2): 391–410. doi:10.1021/cr960065d. PMID 11848876.
- ^ Liu J, Dehbi M, Moeck G, Arhin F, Bauda P, Bergeron D, et al. (February 2004). "Antimicrobial drug discovery through bacteriophage genomics". Nature Biotechnology. 22 (2): 185–191. doi:10.1038/nbt932. PMID 14716317. S2CID 9905115.
- ^ "Technological background Phage-ligand technology". bioMérieux.
- ^ Keen EC (January 2014). "Tradeoffs in bacteriophage life histories". Bacteriophage. 4 (1): e28365. doi:10.4161/bact.28365. PMC 3942329. PMID 24616839.
- ^ Atamer Z, Samtlebe M, Neve H, J Heller K, Hinrichs J (16 July 2013). "Review: elimination of bacteriophages in whey and whey products". Frontiers in Microbiology. 4: 191. doi:10.3389/fmicb.2013.00191. PMC 3712493. PMID 23882262.
- ^ Coffey A, Ross RP (August 2002). "Bacteriophage-resistance systems in dairy starter strains: molecular analysis to application". Antonie van Leeuwenhoek. Springer. 82 (1–4): 303–321. doi:10.1023/a:1020639717181. PMID 12369198. S2CID 7217985.
- ^ Fernández L, Escobedo S, Gutiérrez D, Portilla S, Martínez B, García P, Rodríguez A (November 2017). "Bacteriophages in the Dairy Environment: From Enemies to Allies". Antibiotics. [MDPI]. 6 (4): 27. doi:10.3390/antibiotics6040027. PMC 5745470. PMID 29117107.
- ^ Mason KA, Losos JB, Singer SR, Raven PH, Johnson GB (2011). Biology. New York: McGraw-Hill. p. 533. ISBN 978-0-07-893649-4.
- ^ Mokrousov I (January 2009). "Corynebacterium diphtheriae: genome diversity, population structure and genotyping perspectives". Infection, Genetics and Evolution. 9 (1): 1–15. doi:10.1016/j.meegid.2008.09.011. PMID 19007916.
- ^ Charles RC, Ryan ET (October 2011). "Cholera in the 21st century". Current Opinion in Infectious Diseases. 24 (5): 472–477. doi:10.1097/QCO.0b013e32834a88af. PMID 21799407. S2CID 6907842.
- ^ Keen EC (December 2012). "Paradigms of pathogenesis: targeting the mobile genetic elements of disease". Frontiers in Cellular and Infection Microbiology. 2: 161. doi:10.3389/fcimb.2012.00161. PMC 3522046. PMID 23248780.
- ^ Drulis-Kawa Z, Majkowska-Skrobek G, Maciejewska B (2015). "Bacteriophages and phage-derived proteins--application approaches". Current Medicinal Chemistry. 22 (14): 1757–1773. doi:10.2174/0929867322666150209152851. PMC 4468916. PMID 25666799.
- ^ Gabashvili IS, Khan SA, Hayes SJ, Serwer P (October 1997). "Polymorphism of bacteriophage T7". Journal of Molecular Biology. 273 (3): 658–667. doi:10.1006/jmbi.1997.1353. PMID 9356254.
- ^ Maghsoodi A, Chatterjee A, Andricioaei I, Perkins NC (December 2019). "How the phage T4 injection machinery works including energetics, forces, and dynamic pathway". Proceedings of the National Academy of Sciences of the United States of America. 116 (50): 25097–25105. Bibcode:2019PNAS..11625097M. doi:10.1073/pnas.1909298116. PMC 6911207. PMID 31767752.
- ^ Fiers W, Contreras R, Duerinck F, Haegeman G, Iserentant D, Merregaert J, et al. (April 1976). "Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene". Nature. 260 (5551): 500–507. Bibcode:1976Natur.260..500F. doi:10.1038/260500a0. PMID 1264203. S2CID 4289674.
- ^ Mizuno CM, Guyomar C, Roux S, Lavigne R, Rodriguez-Valera F, Sullivan MB, et al. (February 2019). "Numerous cultivated and uncultivated viruses encode ribosomal proteins". Nature Communications. 10 (1): 752. Bibcode:2019NatCo..10..752M. doi:10.1038/s41467-019-08672-6. PMC 6375957. PMID 30765709.
- ^ Snustad DP (August 1968). "Dominance interactions in Escherichia coli cells mixedly infected with bacteriophage T4D wild-type and amber mutants and their possible implications as to type of gene-product function: catalytic vs. stoichiometric". Virology. 35 (4): 550–563. doi:10.1016/0042-6822(68)90285-7. PMID 4878023.
- ^ Floor E (February 1970). "Interaction of morphogenetic genes of bacteriophage T4". Journal of Molecular Biology. 47 (3): 293–306. doi:10.1016/0022-2836(70)90303-7. PMID 4907266.
- ^ Petrov AS, Harvey SC (July 2008). "Packaging double-helical DNA into viral capsids: structures, forces, and energetics". Biophysical Journal. 95 (2): 497–502. Bibcode:2008BpJ....95..497P. doi:10.1529/biophysj.108.131797. PMC 2440449. PMID 18487310.
- ^ Henrot C, Petit MA (November 2022). "Signals triggering prophage induction in the gut microbiota". Molecular Microbiology. 118 (5): 494–502. doi:10.1111/mmi.14983. PMC 9827884. PMID 36164818. S2CID 252542284.
- ^ Callaway E (2017). "Do you speak virus? Phages caught sending chemical messages". Nature. doi:10.1038/nature.2017.21313.
- ^ Erez Z, Steinberger-Levy I, Shamir M, Doron S, Stokar-Avihail A, Peleg Y, et al. (January 2017). "Communication between viruses guides lysis-lysogeny decisions". Nature. 541 (7638): 488–493. Bibcode:2017Natur.541..488E. doi:10.1038/nature21049. PMC 5378303. PMID 28099413.
- ^ Black LW, Thomas JA (2012). "Condensed Genome Structure". Viral Molecular Machines. Advances in Experimental Medicine and Biology. Vol. 726. pp. 469–87. doi:10.1007/978-1-4614-0980-9_21. ISBN 978-1-4614-0979-3. PMC 3559133. PMID 22297527.
- ^ Al-Shayeb B, Sachdeva R, Chen LX, Ward F, Munk P, Devoto A, et al. (February 2020). "Clades of huge phages from across Earth's ecosystems". Nature. 578 (7795): 425–431. Bibcode:2020Natur.578..425A. doi:10.1038/s41586-020-2007-4. PMC 7162821. PMID 32051592.
- ^ Häuser R, Blasche S, Dokland T, Haggård-Ljungquist E, von Brunn A, Salas M, et al. (2012). "Bacteriophage protein-protein interactions". Advances in Virus Research. 83: 219–298. doi:10.1016/B978-0-12-394438-2.00006-2. ISBN 9780123944382. PMC 3461333. PMID 22748812.
- ^ Morris P, Marinelli LJ, Jacobs-Sera D, Hendrix RW, Hatfull GF (March 2008). "Genomic characterization of mycobacteriophage Giles: evidence for phage acquisition of host DNA by illegitimate recombination". Journal of Bacteriology. 190 (6): 2172–2182. doi:10.1128/JB.01657-07. PMC 2258872. PMID 18178732.
- ^ Krupovic M, Prangishvili D, Hendrix RW, Bamford DH (December 2011). "Genomics of bacterial and archaeal viruses: dynamics within the prokaryotic virosphere". Microbiology and Molecular Biology Reviews. 75 (4): 610–635. doi:10.1128/MMBR.00011-11. PMC 3232739. PMID 22126996.
- ^ Takahashi I, Marmur J (February 1963). "Replacement of thymidylic acid by deoxyuridylic acid in the deoxyribonucleic acid of a transducing phage for Bacillus subtilis". Nature. 197 (4869): 794–795. Bibcode:1963Natur.197..794T. doi:10.1038/197794a0. PMID 13980287. S2CID 4166988.
- ^ Kirnos MD, Khudyakov IY, Alexandrushkina NI, Vanyushin BF (November 1977). "2-aminoadenine is an adenine substituting for a base in S-2L cyanophage DNA". Nature. 270 (5635): 369–370. Bibcode:1977Natur.270..369K. doi:10.1038/270369a0. PMID 413053. S2CID 4177449.
- ^ a b Klipp E (2009). Systems biology: a textbook. Weinheim: Wiley-VCH. ISBN 978-3-527-31874-2. OCLC 288986435.
- ^ Zhao X, Chen C, Shen W, Huang G, Le S, Lu S, et al. (January 2016). "Global Transcriptomic Analysis of Interactions between Pseudomonas aeruginosa and Bacteriophage PaP3". Scientific Reports. 6: 19237. Bibcode:2016NatSR...619237Z. doi:10.1038/srep19237. PMC 4707531. PMID 26750429.
- ^ Blasche S, Wuchty S, Rajagopala SV, Uetz P (December 2013). "The protein interaction network of bacteriophage lambda with its host, Escherichia coli". Journal of Virology. 87 (23): 12745–12755. doi:10.1128/JVI.02495-13. PMC 3838138. PMID 24049175.
- ^ Bobonis J, Mitosch K, Mateus A, Karcher N, Kritikos G, Selkrig J, et al. (September 2022). "Bacterial retrons encode phage-defending tripartite toxin-antitoxin systems". Nature. 609 (7925): 144–150. Bibcode:2022Natur.609..144B. doi:10.1038/s41586-022-05091-4. PMID 35850148. S2CID 250643138.
- ^ Ka D, Oh H, Park E, Kim JH, Bae E (June 2020). "Structural and functional evidence of bacterial antiphage protection by Thoeris defense system via NAD+ degradation". Nature Communications. 11 (1): 2816. Bibcode:2020NatCo..11.2816K. doi:10.1038/s41467-020-16703-w. PMC 7272460. PMID 32499527.
- ^ a b Cieślik M, Bagińska N, Jończyk-Matysiak E, Węgrzyn A, Węgrzyn G, Górski A (May 2021). "Temperate Bacteriophages-The Powerful Indirect Modulators of Eukaryotic Cells and Immune Functions". Viruses. 13 (6): 1013. doi:10.3390/v13061013. PMC 8228536. PMID 34071422.
- ^ a b Wendling CC, Refardt D, Hall AR (February 2021). "Fitness benefits to bacteria of carrying prophages and prophage-encoded antibiotic-resistance genes peak in different environments". Evolution; International Journal of Organic Evolution. 75 (2): 515–528. doi:10.1111/evo.14153. PMC 7986917. PMID 33347602.
- ^ Kirsch JM, Brzozowski RS, Faith D, Round JL, Secor PR, Duerkop BA (September 2021). "Bacteriophage-Bacteria Interactions in the Gut: From Invertebrates to Mammals". Annual Review of Virology. 8 (1): 95–113. doi:10.1146/annurev-virology-091919-101238. PMC 8484061. PMID 34255542.
- ^ Brenciani A, Bacciaglia A, Vignaroli C, Pugnaloni A, Varaldo PE, Giovanetti E (January 2010). "Phim46.1, the main Streptococcus pyogenes element carrying mef(A) and tet(O) genes". Antimicrobial Agents and Chemotherapy. 54 (1): 221–229. doi:10.1128/AAC.00499-09. PMC 2798480. PMID 19858262.
- ^ Jahn MT, Arkhipova K, Markert SM, Stigloher C, Lachnit T, Pita L, et al. (October 2019). "A Phage Protein Aids Bacterial Symbionts in Eukaryote Immune Evasion". Cell Host & Microbe. 26 (4): 542–550.e5. doi:10.1016/j.chom.2019.08.019. PMID 31561965. S2CID 203580138.
- ^ Leigh BA (October 2019). "Cooperation among Conflict: Prophages Protect Bacteria from Phagocytosis". Cell Host & Microbe. 26 (4): 450–452. doi:10.1016/j.chom.2019.09.003. PMID 31600498. S2CID 204243652.
- ^ Ali Y, Koberg S, Heßner S, Sun X, Rabe B, Back A, et al. (2014). "Temperate Streptococcus thermophilus phages expressing superinfection exclusion proteins of the Ltp type". Frontiers in Microbiology. 5: 98. doi:10.3389/fmicb.2014.00098. PMC 3952083. PMID 24659988.
- ^ McGrath S, Fitzgerald GF, van Sinderen D (January 2002). "Identification and characterization of phage-resistance genes in temperate lactococcal bacteriophages". Molecular Microbiology. 43 (2): 509–520. doi:10.1046/j.1365-2958.2002.02763.x. PMID 11985726. S2CID 7084706.
- ^ Douwe M, McGrath J, Fitzgerald S, van Sinderen GF. Identification and Characterization of Lactococcal-Prophage-Carried Superinfection Exclusion Genes▿ †. American Society for Microbiology (ASM). OCLC 679550931.
- ^ Brüssow H, Canchaya C, Hardt WD (September 2004). "Phages and the evolution of bacterial pathogens: from genomic rearrangements to lysogenic conversion". Microbiology and Molecular Biology Reviews. 68 (3): 560–602, table of contents. doi:10.1128/MMBR.68.3.560-602.2004. PMC 515249. PMID 15353570.
- ^ Edlin G, Lin L, Kudrna R (June 1975). "Lambda lysogens of E. coli reproduce more rapidly than non-lysogens". Nature. 255 (5511): 735–737. Bibcode:1975Natur.255..735E. doi:10.1038/255735a0. PMID 1094307. S2CID 4156346.
- ^ Sekulovic O, Fortier LC (February 2015). Schaffner DW (ed.). "Global transcriptional response of Clostridium difficile carrying the CD38 prophage". Applied and Environmental Microbiology. 81 (4): 1364–1374. doi:10.1128/AEM.03656-14. PMC 4309704. PMID 25501487.
- ^ Rossmann FS, Racek T, Wobser D, Puchalka J, Rabener EM, Reiger M, et al. (February 2015). "Phage-mediated dispersal of biofilm and distribution of bacterial virulence genes is induced by quorum sensing". PLOS Pathogens. 11 (2): e1004653. doi:10.1371/journal.ppat.1004653. PMC 4338201. PMID 25706310.
- ^ Obeng N, Pratama AA, Elsas JD (June 2016). "The Significance of Mutualistic Phages for Bacterial Ecology and Evolution" (PDF). Trends in Microbiology. 24 (6): 440–449. doi:10.1016/j.tim.2015.12.009. PMID 26826796.
- ^ Li G, Cortez MH, Dushoff J, Weitz JS (July 2020). "When to be temperate: on the fitness benefits of lysis vs. lysogeny". Virus Evolution. 6 (2): veaa042. bioRxiv 10.1101/709758. doi:10.1093/ve/veaa042. PMC 9532926. PMID 36204422.
- ^ Breitbart M, Salamon P, Andresen B, Mahaffy JM, Segall AM, Mead D, et al. (October 2002). "Genomic analysis of uncultured marine viral communities". Proceedings of the National Academy of Sciences of the United States of America. 99 (22): 14250–14255. Bibcode:2002PNAS...9914250B. doi:10.1073/pnas.202488399. PMC 137870. PMID 12384570.
- ^ Martin C (1988). "The Application of Bacteriophage Tracer Techniques in South West Water". Water and Environment Journal. 2 (6): 638–642. doi:10.1111/j.1747-6593.1988.tb01352.x.
- ^ Bergh O, Børsheim KY, Bratbak G, Heldal M (August 1989). "High abundance of viruses found in aquatic environments". Nature. 340 (6233): 467–468. Bibcode:1989Natur.340..467B. doi:10.1038/340467a0. PMID 2755508. S2CID 4271861.
- ^ Keen EC, Bliskovsky VV, Malagon F, Baker JD, Prince JS, Klaus JS, Adhya SL (January 2017). "Novel "Superspreader" Bacteriophages Promote Horizontal Gene Transfer by Transformation". mBio. 8 (1): e02115–16. doi:10.1128/mBio.02115-16. PMC 5241400. PMID 28096488.
- ^ Lekunberri I, Subirats J, Borrego CM, Balcázar JL (January 2017). "Exploring the contribution of bacteriophages to antibiotic resistance". Environmental Pollution. 220 (Pt B): 981–984. doi:10.1016/j.envpol.2016.11.059. hdl:10256/14115. PMID 27890586.
- ^ a b Manrique P, Bolduc B, Walk ST, van der Oost J, de Vos WM, Young MJ (September 2016). "Healthy human gut phageome". Proceedings of the National Academy of Sciences of the United States of America. 113 (37): 10400–10405. Bibcode:2016PNAS..11310400M. doi:10.1073/pnas.1601060113. PMC 5027468. PMID 27573828.
- ^ Minot S, Sinha R, Chen J, Li H, Keilbaugh SA, Wu GD, et al. (October 2011). "The human gut virome: inter-individual variation and dynamic response to diet". Genome Research. 21 (10): 1616–1625. doi:10.1101/gr.122705.111. PMC 3202279. PMID 21880779.
- ^ a b c Kirsch JM, Brzozowski RS, Faith D, Round JL, Secor PR, Duerkop BA (September 2021). "Bacteriophage-Bacteria Interactions in the Gut: From Invertebrates to Mammals". Annual Review of Virology. 8 (1): 95–113. doi:10.1146/annurev-virology-091919-101238. PMC 8484061. PMID 34255542.
- ^ Strauss JH, Sinsheimer RL (July 1963). "Purification and properties of bacteriophage MS2 and of its ribonucleic acid". Journal of Molecular Biology. 7 (1): 43–54. doi:10.1016/S0022-2836(63)80017-0. PMID 13978804.
- ^ Miller ES, Kutter E, Mosig G, Arisaka F, Kunisawa T, Rüger W (March 2003). "Bacteriophage T4 genome". Microbiology and Molecular Biology Reviews. 67 (1): 86–156, table of contents. doi:10.1128/MMBR.67.1.86-156.2003. PMC 150520. PMID 12626685.
- ^ Ackermann HW, Krisch HM (6 April 2014). "A catalogue of T4-type bacteriophages". Archives of Virology. 142 (12): 2329–2345. doi:10.1007/s007050050246. PMID 9672598. S2CID 39369249.
서지학
- Hauser AR, Mecsas J, Moir DT (July 2016). "Beyond Antibiotics: New Therapeutic Approaches for Bacterial Infections". Clinical Infectious Diseases. 63 (1): 89–95. doi:10.1093/cid/ciw200. PMC 4901866. PMID 27025826.
- Strathdee S, Patterson T (2019). The Perfect Predator. Hachette Books. ISBN 978-0316418089.
- Häusler T (2006). Viruses vs. superbugs : a solution to the antibiotics crisis?. London: Macmillan. ISBN 978-1-4039-8764-8.
외부 링크
- Abedon ST. "The Bacteriophage Ecology Group". The Ohio State University. Archived from the original on 3 June 2013.
- Tourterel C, Blouin Y. "Bacteriophages illustrations and genomics". Orsay phage web site. Archived from the original on 29 October 2013. Retrieved 24 October 2013.
- "QuipStories: Bacteriophages get a foothold on their prey" (PDF). PDBe.
- Flatow I (April 2008). "Using 'Phage' Viruses to Help Fight Infection". Science Friday podcast. NPR. Archived from the original on 17 April 2008.
- "Animation of a scientifically correct T4 bacteriophage targeting E. coli bacteria". YouTube.
- "T4 Bacteriophage targeting E. coli bacteria". Animation by Hybrid Animation Medical. 21 December 2009.
- 박테리오파지: 그들은 뭐지. 피츠버그대 그레이엄 해트풀 교수의 유투브 발표



