DMSO 환원효소
DMSO reductase| 황산화디메틸 환원효소 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| EC 번호 | 1.8.5.3 | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 입력 | ||||||||
| 엑스퍼시 | 나이스자이메 뷰 | ||||||||
| 케그 | KEG 입력 | ||||||||
| 메타사이크 | 대사통로 | ||||||||
| 프리암 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBsum | ||||||||
| |||||||||
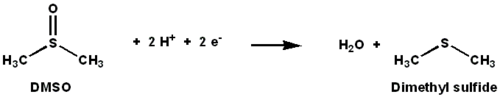
DMSO 환원효소는 몰리브데넘 함유 효소로 황산화디메틸(DMSO)을 디메틸화이드(DMS)로 환원시키는 효소다.이 효소는 일부 박테리아에서 혐기성 조건 하에서 단자 환원효소 역할을 하며, DMSO는 단자 전자 수용체 역할을 한다.반응 과정에서 DMSO의 산소 원자는 몰리브덴으로 전달된 후 물로 환원된다.
DMSOR(DMSO 환원효소)와 다른 DMSO 환원효소 제품군은 박테리아와 고고학 고유의 것이다.혐기성 산화 인산화 및 무기기증자 기반 석회성 호흡에서 이 계열의 효소.이 효소들은 옥소이온을 분해하도록 설계되었다.[1][2][3]DMSOR는 반응에서 두 개의 전자와 한 개의 산소 원자의 전달을 촉진한다.DMSOR의 활성 사이트는 달리 생물학에서는 드물게 몰리브덴을 포함하고 있다.[2]
3차 구조 및 활성 현장
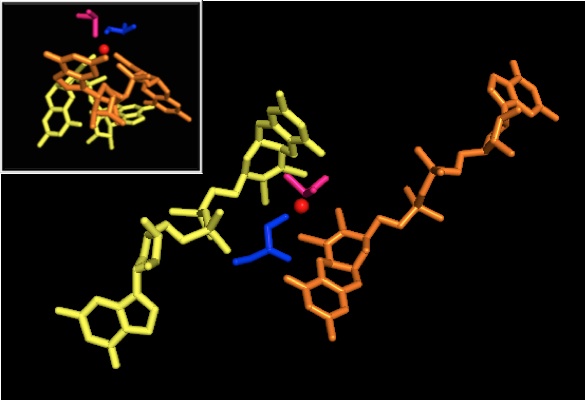
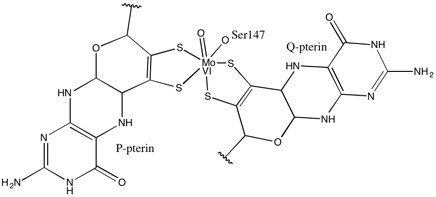
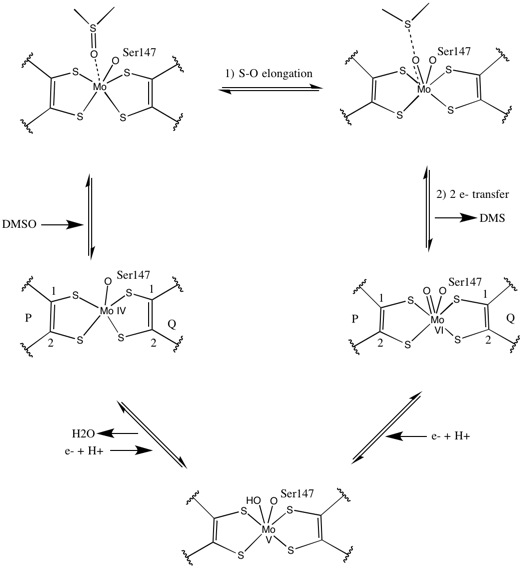
DMSOR의 다른 구성원의 경우, DMSOR의 3차 구조는 Mo-surrounding 도메인 I-IV로 구성되며, 도메인 IV는 활성 사이트의 피라노프틴디티올렌 모코팩터(P- 및 Q-pterin)와 크게 상호작용한다.[2][3]DMSO 환원효소 제품군의 구성원들은 그들의 활성 사이트에 대해 서로 다르다.[3]DMSOR의 경우, 두 개의 피라놉테린 공actor가 제공하는 두 개의 디티올렌에서 Mo 센터가 발견된다.몰리브도프테린이라고 불리는 이러한 유기농 공작용제는 GMP와 연결되어 디뉴클레오티드 형태를 만들어낸다.추가로 다섯 번째 캡 모양의 리간드는 세린-147 잔여물의 사이드 체인 O로 효소를 더 나아가 타입 III DMSO 환원효소로 분류한다.InType I과 II Serine은 각각 시스테인과 아스파테이트 잔류물로 대체된다.반응이 진행됨에 따라 IV, V, VI 사이에서 변동하는 Mo의 redox 상태에 따라 활성 사이트 Mo core도 각각 아쿠아, 수산화, 옥소 그룹의 산소 원자에 결합할 수 있다.연구에 따르면 Mo 코어를 조정하는 데 사용되는 아미노산 아미노산의 특정 정체는 촉매변환에 대한 효소의 메커니즘에서 핵심 결정인 산소군 레독스 중간점 전위와 양성 상태에 큰 영향을 미친다.[2]
메커니즘
초기 동위원소 DMSO18 연구는 R. sphaeride의 DMSOR를 위한 이중 산소환원효소 메커니즘을 확립했다.이 메커니즘에서 라벨이 표시된 O는18 기질에서 Mo로 전달되며, 그 다음 O를18 1,3,5-트리자-7-인산아다만탄탄(PTA)로 전송하여 PTAO를18 산출한다.[6]유사한 메커니즘에서, DMSO는 O를 Mo에게 전달하고, 그 결과 Mo(VI)O 중심이 감소하여 물을 산출한다.[7]
합성 Mo bis-dithiolene 복합체에 대한 연구는 산소 전달, 전자 전달이라고 제안한다.S K-edge XAS와 DFT를 이용하여, 이 모델 연구들은 결합된 S-O 분열과 전자 전자를 지적한다.속도는 기질 X-O 결합 강도를 감소시키고 기질 양성자 친화력을 증가시키는 것에 비례한다.[8]
X선 결정학은 효소의 전반적인 3차 구조가 반응 진행을 통해 일정하게 유지된다는 것을 입증했다.그러나 R. Spaeroides의 DMSOR에 대해 수행된 여러 가지 다른 실험은 4개의 잠재적 디티올렌 리간드의 조정 활동에 대해 서로 다른 결과를 보고했다.한 X선 결정학 연구는 산화된 형태의 Mo-S 리간드 4개 모두에 대해 등거리 조율을 결론을 내렸지만, 다른 연구는 비대칭 Mo-S 거리를 특징으로 했다.전자파파자기공명(EPR) 연구뿐만 아니라 두 연구 모두 Mo 활성 부위가 위치 및 잠재적 리간드 좌표의 정도 측면에서 매우 유연하다고 예측했다.[7][9]
크게 비대칭인 두 개의 피라노프테린 공효소를 제안하는 데이터는 반응 메커니즘을 제안하는 데 사용되었다.활성 부지의 완전히 산화된 Mo VI 형태에서, 황소군 및 세린 리간드는 Mo 중심에서 1.7 A 거리에서 조정되었다.P-pterin의 S1, S2, Q-pterin의 S1은 Mo로부터 2.4 A 떨어진 곳에 위치하였고, Q-pterin의 S2는 3.1 A 떨어진 곳에 위치하였다.이러한 프테린 비대칭은 옥소군 바로 맞은편에 위치한 S2-Mo 결합을 약화시키는 옥소군의 전이 효과의 결과일 수 있다.[7]
대조적으로, 활성 부위의 완전히 축소된 Mo IV 형태의 구조는 S1과 S2 P-pterin 및 S1 Q-pterin이 완전한 조정을 유지하였지만, Q-pterin의 S2가 금속 중심에서 멀어짐으로써 조정력 저하를 나타냈다.리간드-모 본드 길이의 이러한 변화는 DMSO 기질에서 Mo로 직접 산소 전달 메커니즘을 제안하는 것과 일치한다. 감소된 효소 형태의 디티올렌 조율이 약하면 S=O의 직접 결합을 촉진할 수 있다.Mo의 감소와 옥소군의 양성에서는 사이토크롬 전자원이 활성 부위 위의 우울증에 결합하여 Mo 중심을 직접 감소시킬 수 있거나, 또는 이 사이토크롬이 Q-pterin에 근접하여 잘 접합된 폴리펩타이드 루프에 결합할 수 있고, Q-pterin이 이러한 전자전달을 중재할 수 있다고 제안한다..[7]
셀룰러 위치 및 규정
R. sphaeroides에서 DMSOR는 pterin을 넘어서는 추가적인 공작용제가 필요 없는 단일 서브 유닛 수용성 단백질이다.대장균에서 DMSOR는 세포막 내에 내장되어 있으며, 3개의 고유한 서브유닛을 가지고 있는데, 그 중 하나는 특징적인 프테린 코팩터를 포함하고 있으며, 다른 하나는 4Fe:4S 군집을 포함하고 있으며, 최종 트랜섬브레인 서브유닛은 메나퀴놀을 결합하고 산화시킨다.메나퀴놀에서 4Fe:4S 클러스터로 e-를 전송하고 마지막으로 pterin-Mo 활성 사이트로 전송하면 ATP 생성에 사용되는 양성자 그라데이션이 생성된다.[7]
DMSOR는 주로 전사적 수준에서 규제된다.도르 유전자에 의해 인코딩되며, 신호 캐스케이드에 의해 활성화되었을 때 발현되는데, 도르스, 도르R, 도르C 단백질의 규제 하에 있다.해당 DorS, DorR, DorC 발기인들에 대한 lacZ 융합(리포터 유전자) 연구 결과 산소환경 감소로 DorR과 DorC의 발현이 증가했지만 DorS 발현에는 산소농도의 영향을 받지 않았다.DMSO의 농도가 증가함에 따라 DorC 표현도 증가했다.[10]
환경영향
DMSOR의 산물인 DMS는 유황 순환의 성분이다.DMS는 메탄설폰산염으로 산화되는데, 이 메탄설폰산염은 핵의 대체 공급원인 먼지가 없는 탁 트인 대양 위에 구름 응결을 핵으로 만든다.구름 형성은 지구의 알베도를 증가시키고 대기 온도를 조절하는 데 중요한 요소로서, 이 효소와 그것이 촉매하는 반응은 기후 통제 개척에 도움이 될 수 있다.[11]
참조
- ^ Kappler U, Schäfer H (2014). "Chapter 11. Transformations of Dimethylsulfide". In Kroneck PM, Torres ME (eds.). The Metal-Driven Biogeochemistry of Gaseous Compounds in the Environment. Metal Ions in Life Sciences. Vol. 14. Springer. pp. 279–313. doi:10.1007/978-94-017-9269-1_11. ISBN 978-94-017-9268-4. PMID 25416398.
- ^ a b c d McEwan AG, Kappler U (2004). "The DMSO Reductase Family of Microbial Molybdenum Enzymes" (PDF). Australian Biochemist. 35 (3): 17–20. Archived from the original (PDF) on 2014-03-07. Retrieved 2014-02-27.
- ^ a b c d e McEwan AG, Ridge JP, McDevitt CA, Hugenholtz P (2002). "The DMSO Reductase Family of Microbial Molybdenum Enzymes; Molecular Properties and Role in the Dissimilatory Reduction of Toxic Elements". Geomicrobiology Journal. 19 (1): 3–21. doi:10.1080/014904502317246138.
- ^ PDB: 1DMS; Schneider F, Löwe J, Huber R, Schindelin H, Kisker C, Knäblein J (October 1996). "Crystal structure of dimethyl sulfoxide reductase from Rhodobacter capsulatus at 1.88 A resolution". Journal of Molecular Biology. 263 (1): 53–69. doi:10.1006/jmbi.1996.0555. PMID 8890912.
- ^ PDB: 4DMR; McAlpine AS, McEwan AG, Bailey S (January 1998). "The high resolution crystal structure of DMSO reductase in complex with DMSO". Journal of Molecular Biology. 275 (4): 613–23. doi:10.1006/jmbi.1997.1513. PMID 9466935.
- ^ Schultz BE, Hille R, Holm RH (1995), "Direct oxygen atom transfer in the mechanism of action of Rhodobacter sphaeroides dimethyl sulfoxide reductase", Journal of the American Chemical Society, 117 (2): 827–828, doi:10.1021/ja00107a031, ISSN 0002-7863
- ^ a b c d e Kisker C, Schindelin H, Rees DC (1997). "Molybdenum-cofactor-containing enzymes: structure and mechanism" (PDF). Annual Review of Biochemistry. 66: 233–67. doi:10.1146/annurev.biochem.66.1.233. PMID 9242907.
- ^ Tenderholt AL, Wang JJ, Szilagyi RK, Holm RH, Hodgson KO, Hedman B, Solomon EI (June 2010). "Sulfur K-edge X-ray absorption spectroscopy and density functional calculations on Mo(IV) and Mo(VI)=O bis-dithiolenes: insights into the mechanism of oxo transfer in DMSO reductase and related functional analogues". Journal of the American Chemical Society. 132 (24): 8359–71. doi:10.1021/ja910369c. PMC 2907113. PMID 20499905.
- ^ McAlpine AS, McEwan AG, Shaw AL, Bailey S (1997). "Molybdenum active centre of DMSO reductase from Rhodobacter capsulatus: crystal structure of the oxidised enzyme at 1.82-A resolution and the dithionite-reduced enzyme at 2.8-A resolution". JBIC. 2 (6): 690–701. doi:10.1007/s007750050185.
- ^ Gunsalus RP (November 1992). "Control of electron flow in Escherichia coli: coordinated transcription of respiratory pathway genes". Journal of Bacteriology. 174 (22): 7069–74. PMC 207394. PMID 1331024.
- ^ Sarkar B (21 March 2002). Heavy Metals In The Environment. CRC Press. p. 456. ISBN 978-0-8247-4475-5.