합성생물학
Synthetic biology| 에 관한 일련의 기사의 일부 |
| 합성생물학 |
|---|
| 합성 생물 회로 |
| 게놈 편집 |
| 인공 세포 |
| 이종생물학 |
| 기타 토픽 |
합성생물학(SynBio)은 새로운 생물학적 부품, 장치 및 시스템을 만들거나 자연에서 이미 발견된 시스템을 재설계하는 다원적 연구 분야입니다.
생명공학, 유전공학, 분자생물학, 분자공학, 시스템생물학, 막과학, 생물물리학, 화학 및 생물공학, 제어공학, 진화생물학과 같은 다양한 분야의 방법론을 포괄하는 과학의 한 분야이다.y를 클릭합니다.
보다 강력한 유전공학 능력과 DNA 합성 및 염기서열 분석 비용 감소로 인해, 합성 생물학 분야는 빠르게 성장하고 있다.2016년에는 40개국에서 350개 이상의 기업이 합성 생물 애플리케이션에 적극적으로 종사하고 있습니다.이들 기업 모두 세계 시장에서 [1]약 39억달러의 순자산을 보유하고 있습니다.
정의.
합성 생물학은 현재 일반적으로 받아들여지는 정의가 없다.다음은 몇 가지 예입니다.
- "새로운(따라서 합성)[2] 생명체를 창조하기 위해 물리공학과 유전공학의 혼합물을 사용하는 것"
- "화학적으로 합성된 DNA 설계에서 생물학, 공학 및 관련 분야의 지식과 방법을 결합하여 새롭고 강화된 특징과 특징을 [3]가진 유기체를 만드는 것을 목표로 하는 새로운 연구 분야"
- "생물 모듈, 생물 시스템 및 생물 기계의 설계 및 구축 또는 유용한 [4]목적을 위해 기존 생물 시스템의 재설계"
- "자연에는 존재하지 않는 새로운 기능을 가진 예측 가능하고 견고한 시스템을 생산하기 위해 시스템 설계의 엔지니어링 패러다임을 생물학적 시스템에 적용" (유럽위원회, 2005) 이것은 리보솜과[5] 같은 생체 분자 시스템에 기초한 분자 조립자의 가능성을 포함할 수 있다.
합성생물학은 전통적으로 두 가지 다른 접근법, 즉 위에서 아래로와 아래로 나뉘어져 왔다.
- 하향식 접근법은 살아있는 세포에 새로운 기능을 부여하기 위해 대사 및 유전 공학 기술을 사용하는 것을 포함한다.
- 상향식 접근법은 종종 인공 세포를 만들 목적으로 '살아있지 않은' 생체 분자 구성 [6]요소들을 모아서 체외에서 새로운 생물학적 시스템을 만드는 것을 포함한다.
따라서 생물학적 시스템은 모듈별로 조립됩니다.세포 없는 단백질 발현 시스템은 멤브레인 기반 분자 기계와 마찬가지로 종종 [7][8][9]사용된다.이러한 접근방식의 차이를 메우기 위한 노력은 혼성 생물/합성 [10]세포를 형성하고 생물 세포군과 합성 세포군 [11]간의 엔지니어링 통신을 통해 증가하고 있다.
역사
1910: Stéphane Leduc의 출판물 Théori physicalo-chimique de la vie et généations [12]aspratanées에서 "합성생물학"이라는 용어의 최초 식별 가능한 사용.그는 1912년 [13]또 다른 출판물인 La Biologicalie Synthétique에서도 이 용어를 언급했다.
1961년: Jacob과 Monod는 대장균의 라크 오퍼론에 대한 연구에서 분자 네트워크에 의한 세포 조절을 가정하고 분자 [14]성분으로부터 새로운 시스템을 조립할 수 있는 능력을 구상했다.
1973년: 플라스미드에서 DNA의 첫 분자 복제와 증폭은 합성생물학의 [15]시작을 구성하는 코헨, 보이어 등에 의해 P.N.A.S.에 발표되었다.
1978년: Arber, Nathans 및 Smith는 제한 효소의 발견으로 노벨 생리의학상을 수상하여 Szybalski가 Gene 저널에 사설 코멘트를 제공하도록 이끌었습니다.
제한핵산가수분해효소에 대한 연구는 우리가 쉽게 재조합 DNA 분자를 구성하고 개별 유전자를 분석할 수 있게 해줄 뿐만 아니라 기존의 유전자가 기술되고 분석될 뿐만 아니라 새로운 유전자 배열이 구성되고 [16]평가될 수 있는 합성생물학의 새로운 시대로 우리를 이끌었다.
1988: 내열성 DNA 중합효소를 이용한 중합효소 연쇄반응(PCR)에 의한 최초의 DNA 증폭은 Mullis 등에 의해 사이언스에 발표되었다.[17]이것은 각 PCR 사이클 후에 새로운 DNA 중합효소를 첨가하는 것을 피했고, 따라서 DNA 돌연변이 유발과 조립을 크게 단순화했다.
2000: Nature의 두 논문은 대장균 [18][19]세포 내의 유전자를 결합함으로써 합성 생물학적 회로, 유전자 전환 스위치와 생체 시계를 보고했습니다.
2003: 가장 널리 사용되는 표준화된 DNA 부품인 바이오브릭 플라스미드는 Tom [20]Knight에 의해 발명되었습니다.이 부품들은 다음 해에 MIT에서 열리는 국제 유전자 공학 기계 경연대회(iGEM)의 중심이 될 것입니다.
2003년: 연구자들은 [21]대장균의 아르테미시닌 전구 경로를 설계한다.
2004년: 최초의 합성생물학 국제회의인 Synthetic Biology 1.0(SB1.0)이 미국 매사추세츠 공과대학에서 개최됩니다.
2005년:[22] 연구자들은 대장균의 광감지 회로를 개발했습니다.또 다른 그룹은 다세포 패턴 [23]형성이 가능한 회로를 설계한다.
2006년: 연구자들은 종양 [24]세포의 세균 침투를 촉진하는 합성 회로를 개발했습니다.
2010년: 연구자들은 M. mycoides JCVI-syn1.[25][26]0이라는 최초의 합성 박테리아 게놈을 사이언스에 발표했다.게놈은 효모 재조합을 이용하여 화학적으로 합성된 DNA로 만들어진다.
2011년: 기능성 합성 염색체 팔은 [27]효모로 제조되었다.
2012년: Charpentier와 Doudna 연구소는 DNA [28]분열을 목표로 하는 CRISPR-Cas9 박테리아 면역 프로그래밍을 Science에 발표했다.이 기술은 진핵생물 유전자 편집을 대폭 간소화하고 확대했다.
2019년: ETH 취리히의 과학자들은 Caulobacter ethensis-2.0이라는 이름의 최초의 박테리아 게놈이 완전히 컴퓨터에 의해 만들어졌다고 보고하고 있다. 그러나 C. ethensis-2.0과 관련된 실행 가능한 형태는 [29][30]아직 존재하지 않는다.
2019년: 연구자들은 20개의 아미노산을 [31][32]암호화하기 위해 박테리아 게놈에 있는 64개의 코돈의 자연수를 59개의 코돈으로 줄임으로써 박테리아 대장균의 변종인 새로운 합성(아마도 인공적인) 형태의 생존 생물의 생산을 보고한다.
2020년: 과학자들은 개구리 세포에서 파생되어 [33]AI에 의해 설계된 최초의 프로그래밍 가능한 이종 로봇 합성 유기체를 만들었다.
2021년: 과학자들은 제노봇이 환경 내에서 느슨한 세포를 모아 새로운 [34]제노봇을 형성함으로써 자가 복제를 할 수 있다고 보고했습니다.
전망
기술자들이 기술(다른 말로 하면, 주어진 시스템 생물 공학 또는 그것의 생물학적 공학을 포함하)[35]합성 생물학에서와고 건설하는 정보를 처리하는데 공학적 살아 있는 생물학적 시스템을 설계할 수 있는 궁극적인 골로 넓은 재정의와 생명 공학의 확대,,를 조작하는 화학 물질을 포함한다 생물학 보고 있다., fa재료와 구조를 소금에 절이고, 에너지를 생산하고, 음식을 제공하고, 인간의 건강을 유지하고 강화하며, 생물학적 시스템(생물의학 공학 참조)과 환경에 [36]대한 기초 지식을 발전시킨다.
합성생물학 연구는 당면한 문제에 대한 접근법에 따라 광범위한 분류로 세분될 수 있다: 생물학적 부분의 표준화, 생체 분자 공학, 게놈 공학, 대사 [citation needed]공학.
생체 분자 공학은 살아있는 세포에 새로운 기술적 기능을 제시하기 위해 도입될 수 있는 기능 단위의 툴킷을 만드는 것을 목표로 하는 접근법을 포함한다.유전자 공학은 합성 염색체 또는 마이코플라스마 연구소와 같은 최소 유기체를 구성하는 방법을 포함한다.
생체분자 디자인은 생체분자 구성요소의 새로운 디자인과 가법적 결합에 대한 일반적인 아이디어를 말한다.이러한 접근법 각각은 유사한 작업을 공유한다. 즉, 이전 [37][38]수준에서 보다 단순한 부분을 발명하여 보다 복잡한 수준에서 보다 합성적인 실체를 개발하는 것이다.부자연스러운 시스템에서 이러한 외생 경로를 최적화하려면 원하는 [39]제품의 최고 농도를 위해 선택하기 위해 개별 생체 분자 구성 요소의 반복적인 미세 조정이 필요합니다.
반면, "재작성자"는 생물학적 시스템의 환원 불가능성을 테스트하는 데 관심이 있는 합성 생물학자입니다.자연 생물학적 시스템의 복잡성 때문에, 이해, 제어, [40]조작이 용이한 가공된 대용품을 제공하기 위해, 관심 있는 자연 시스템을 처음부터 재구축하는 것이 더 간단할 것이다.리라이터는 리팩터링에서 영감을 얻습니다.리팩터링은 때때로 컴퓨터 소프트웨어를 개선하기 위해 사용됩니다.
테크놀로지의 유효화
몇 가지 새로운 가능 기술은 합성 생물학의 성공에 매우 중요했습니다.개념에는 생물학적 부품의 표준화와 합성 [41]시스템에서 이러한 부품을 사용할 수 있도록 하기 위한 계층적 추상화가 포함된다.기본적인 기술로는 DNA를 읽고 쓰는 기술(시퀀싱과 조작)이 있습니다.정확한 모델링과 컴퓨터 지원 설계(CAD)를 위해서는 여러 조건에서 측정이 필요합니다.
DNA 및 유전자 합성
올리고뉴클레오티드("올리고스") 합성 비용의 급격한 감소와 PCR의 등장으로 인해 올리고스의 DNA 구성 크기가 게놈 [42]수준으로 증가했다.2000년에, 연구원들은 화학적으로 합성된 [43]60에서 80미터로 9.6 kbp (킬로 bp) C형 간염 바이러스 게놈을 합성했다고 보고했다.2002년 스토니브룩 대학 연구진은 7741bp 폴리오바이러스 게놈을 2년에 [44]걸쳐 합성하는 데 성공했다.2003년에 박테리오파지 Phi X 174의 5386 bp 게놈이 약 2주 [45]만에 조립되었다.2006년 J. Craig Venter Institute의 같은 팀이 새로운 최소 박테리아인 마이코플라스마 연구소의 합성 게놈을 만들고 특허를 냈습니다. 그리고 살아있는 [46][47][48]세포에서 기능하도록 하는 작업을 하고 있었습니다.
2007년에는 여러 회사가 최대 2000개의 염기쌍(bp) 길이의 유전자 염기서열 합성을 bp당 약 1달러의 가격과 2주 [49]미만의 반환 시간에 제공하고 있는 것으로 보고되었다.PCR 및 DNA 불일치 오류 수정과 결합된 포토 리소그래피 또는 잉크젯 제조 DNA 칩에서 수확된 올리고뉴클레오티드는 유전자 발현을 개선하거나 새로운 아미노산을 통합하기 위해 유전자 시스템에서 코돈의 저렴한 대규모 변화를 가능하게 한다(조지 M 참조). Church's and Anthony Forster의 합성 세포 프로젝트)[50][51]이것은 스크래치에서 합성하는 접근방식을 선호한다.
또한, CRISPR/Cas 시스템은 유전자 편집의 유망한 기술로 떠올랐다.그것은 "거의 30년 동안 합성 생물학 분야에서 가장 중요한 혁신"[52]으로 묘사되었다.다른 방법들이 유전자 서열을 편집하는 데 몇 달에서 몇 년이 걸리는 반면, CRISPR은 그 시간을 몇 [52]주까지 단축시킨다.그러나 이 제품의 사용 편의성과 접근성 때문에 특히 바이오 [53][54][55]해킹에서의 사용을 둘러싸고 윤리적인 문제가 제기되었습니다.
시퀀싱
DNA 배열은 DNA 분자 내의 뉴클레오티드 염기 순서를 결정한다.합성 생물학자들은 DNA 염기서열 분석을 여러 가지 방법으로 사용한다.첫째, 대규모 게놈 배열 분석 노력은 자연발생 유기체에 대한 정보를 계속 제공한다.이 정보는 합성 생물학자들이 부품과 장치를 구성할 수 있는 풍부한 기판을 제공합니다.둘째, 시퀀싱을 통해 제조된 시스템이 의도한 대로인지 확인할 수 있습니다.셋째, 빠르고 저렴하며 신뢰할 수 있는 시퀀싱은 합성 시스템과 [56]유기체의 신속한 검출과 식별을 촉진할 수 있습니다.
모듈러성
가장 많이[57]: 22–23 사용되는 표준화된 DNA 부품은 2003년 [20]Tom Knight에 의해 발명된 BioBrick 플라스미드이다.바이오브릭은 매사추세츠주 캠브리지에 있는 표준 생체 부품 등록소에 보관되어 있습니다.BioBrick 표준은 전 세계 수만 명의 학생들이 국제 유전공학 기계([57]: 22–23 iGEM) 대회에서 사용하고 있습니다.
중복 유전자라고 불리는 두 개의 유전자 요소 사이의 배열 중복은 그들의 개별적인 [58]조작을 막을 수 있다.게놈 모듈성을 높이기 위해 게놈 리팩터링 또는 개선 "외부 시스템 [59]기능을 유지하면서 미래 사용을 위한 기존 시스템의 내부 구조"를 합성 생물학 분야에 [58]걸쳐 채택하고 있다.질소 고정 클러스터[60] 및 타입 III 분비[61] 시스템과 박테리오파지[59] [62]T7, δX174를 포함한 리팩터링의 몇 가지 주목할 만한 예.
DNA가 정보 저장에 가장 중요한 반면, 세포 활동의 상당 부분은 단백질에 의해 수행된다.도구는 세포의 특정 부위에 단백질을 보내고 서로 다른 단백질을 연결할 수 있다.단백질 파트너 간의 상호작용 강도는 몇 초간의 수명(동적 시그널링 이벤트에 바람직함)에서 돌이킬 수 없는 상호작용(디바이스 안정성에 바람직함 또는 가혹한 조건에 대한 탄력성)까지 조정할 수 있어야 한다.코일 코일,[63] SH3 도메인-펩타이드[64] 결합 또는 SpyTag/SpyCatcher와[65] 같은 상호 작용이 이러한 제어를 제공합니다.또한 빛(광산소 전압 감지 도메인 사용) 또는 화학적으로 유도된 이합체화에 [66]의해 세포 투과성 소분자와 같은 세포 내 단백질-단백질 상호작용을 조절하는 것이 필요하다.
살아있는 세포에서 분자 모티브는 업스트림 및 다운스트림 구성요소와 함께 더 큰 네트워크에 내장되어 있습니다.이러한 컴포넌트에 의해 모델링 모듈의 시그널링 기능이 변경될 수 있습니다.초감응 모듈의 경우 모듈의 감도 기여도는 모듈이 [67][68]단독으로 유지하는 감도와 다를 수 있습니다.
모델링.
모델은 제조 전에 시스템 동작을 더 잘 예측함으로써 엔지니어링된 생물학적 시스템의 설계에 정보를 제공합니다.합성 생물학은 생물학적 분자가 기질을 결합하고 반응을 촉매하는 방법, DNA가 세포를 특정하는 데 필요한 정보를 어떻게 암호화하고 다성분 통합 시스템이 어떻게 동작하는지에 대한 더 나은 모델로부터 이익을 얻는다.유전자 조절 네트워크의 멀티스케일 모델은 합성 생물학 응용에 초점을 맞춘다.시뮬레이션은 유전자 조절 [69]네트워크의 전사, 번역, 조절 및 유도에서 모든 생체 분자 상호작용을 모델링할 수 있습니다.[70] [71]
미세유체학
미세유체학, 특히 액체 미세유체학은 새로운 구성 요소를 구성하고 분석 [72][73]및 특성화하는 데 사용되는 새로운 도구입니다.그것은 선별 [74]검사에 널리 사용된다.
합성 전사인자
연구는 DNA 전사 메커니즘의 구성 요소를 고려해왔다.합성 생물 회로를 만드는 과학자들의 한 가지 바람은 단세포 생물과 다세포 생물에서 합성 DNA의 전사를 통제할 수 있는 것이다.한 연구에서는 여러 전사인자 [75]복합체 간의 협업 능력 및 전사 출력 영역에서 합성 전사인자(sTF)의 조정 가능성을 테스트했습니다.연구진은 특정 운영자 DNA 배열 부위에 대한 친화력을 감소시키고, 따라서 관련 부위 고유 활동을 감소시키기 위해 sTF의 DNA 특정 성분인 아연 핑거라고 불리는 기능 영역을 돌연변이 시킬 수 있었다.그들은 또한 아연 손가락을 진핵생물 번역 [75]메커니즘인 복합 형성 sTF의 구성 요소로 사용했다.
적용들
바이오센서
바이오센서는 중금속이나 독소의 존재와 같은 주변 현상을 보고할 수 있는 공학적 유기체, 보통 박테리아를 말한다.그러한 시스템 중 하나는 Aliivrio fischeri의 [76]Lux 오퍼론이며, 이것은 박테리아 생물 발광의 원천인 효소를 코드하고 특정 환경 [77]자극에 반응하여 발광 유전자를 발현하기 위해 응답 프로모터 뒤에 배치될 수 있다.그러한 센서 중 하나는 특정 석유 오염 물질을 감지하기 위해 감광성 컴퓨터 칩에 생물 발광 박테리아 코팅으로 구성되어 있습니다.박테리아가 오염물질을 감지하면 빛을 [78]발한다.유사한 메커니즘의 또 다른 예로는 TNT 및 그 주요 분해물 DNT를 검출하여 결과적으로 녹색 형광단백질(GFP)[79]을 제조할 수 있는 엔지니어링된 E.coli 리포터 균주에 의한 지뢰 검출이 있다.
변형된 유기체는 환경 신호를 감지하고 검출할 수 있는 출력 신호를 보낼 수 있으며 진단 목적에 도움이 됩니다.마이크로베 코호트가 [80]사용되었습니다.
바이오센서는 SARS-CoV-2 등의 병원성 시그니처를 검출하기 위해서도 사용할 수 있어 [81][82]착용할 수 있습니다.
음식과 음료
세포 농업은 전통적인 농업에서 유래했을 단백질, 지방, 조직을 생산하는 새로운 방법을 만들고 설계하기 위해 생명공학, 조직공학, 분자생물학, 합성생물학을 조합하여 세포 배양으로부터 농산물을 생산하는 것에 초점을 맞추고 있다.대부분의 산업은 해로운 환경 영향(예: 육류 생산), 동물 복지, 식품 안전 및 인간의 건강과 관련된 상당한 세계적인 문제와 관련된 가축을 사육하고 도축하기 보다는 세포 배양으로 생산된 고기, 우유, 계란과 같은 동물 제품에 초점을 맞추고 있다.세포 농업은 생물 기반 경제의 분야이다.세포농업의 가장 잘 알려진 개념은 배양육이다.(전문...)
그러나 모든 합성영양제품이 동물성 식품인 것은 아니다.예를 들어 2021년 현재 [85][86][87]상품화에 가까운 합성커피 제품도 있다.식품과 음료의 생산에 사용될 수 있는 합성 생물학을 기반으로 한 유사한 연구 및 생산 분야는 다음과 같다.
자재
광합성 미생물 세포는 거미줄을 [92][93]합성 생산하기 위한 단계로 사용되어 왔다.
생물 컴퓨터
생물학적 컴퓨터는 합성 생물학에서 지배적인 패러다임인 컴퓨터와 같은 작업을 수행할 수 있는 공학적 생물학적 시스템을 말한다.연구자들은 다수의 [94]유기체에 다양한 논리 게이트를 구축하고 특성을 부여했으며 살아있는 세포에서 아날로그와 디지털 연산을 모두 시연했다.그들은 박테리아가 아날로그 및/또는 디지털 [95][96]연산을 모두 수행하도록 설계될 수 있다는 것을 입증했다.인간 세포 연구는 2007년 [97]포유류의 세포에서 작동하는 보편적인 논리 평가자를 입증했다.그 후,[98] 연구자들은 이 패러다임을 이용하여 2011년에 인간의 암세포를 발견하고 죽이기 위해 생물학적 디지털 연산을 사용하는 개념 증명 치료법을 시연했다.또 다른 연구진은 2016년 컴퓨터 공학의 원리가 박테리아 [99]세포의 디지털 회로 설계를 자동화하는데 사용될 수 있다는 것을 증명했다.2017년 연구진은 인간 [100]세포에서 디지털 연산을 조작하는 'BLADE(Boolean logic and assculation through DNA instraction)' 시스템을 시연했다.2019년, 연구자들은 생물 시스템에 퍼셉트론을 구현하여 이러한 [101]시스템에 기계 학습의 길을 열었다.
세포 변환
세포는 유전자 회로라고 불리는 상호작용하는 유전자와 단백질을 사용하여 환경 신호에 반응, 의사 결정, 의사소통과 같은 다양한 기능을 구현합니다.DNA, RNA, 합성생물학자가 설계한 유전자 회로는 전사, 전사 후 및 번역 수준을 포함한 여러 수준에서 유전자 발현을 제어할 수 있습니다.
전통적인 대사 공학은 외래 유전자의 조합의 도입과 직접적인 진화에 의한 최적화에 의해 강화되어 왔다.여기에는 항말라리아제인 아르테미시닌의 [102]상업적 생산을 위한 엔지니어링 대장균 및 효모가 포함된다.
살아있는 세포는 새로운 DNA로 변형될 수 있지만, 전체 유기체는 아직 처음부터 만들어지지 않았다.여러 가지 방법으로 합성 DNA 성분과 심지어 전체 합성 게놈을 만들 수 있지만, 일단 원하는 유전자 코드를 얻으면, 그것은 성장과 [103]번영을 통해 원하는 새로운 능력이나 표현형을 나타낼 것으로 기대되는 살아있는 세포에 통합된다.세포 변환은 생물학적 회로를 만드는 데 사용되며, 생물학적 [18][19]회로는 원하는 출력을 내도록 조작될 수 있습니다.
합성 생물학을 재료 과학에 통합함으로써, 세포를 미세한 분자 주조 공장으로서 유전적으로 암호화된 특성을 가진 물질을 생산하는 것이 가능해질 것이다.리엔지니어링은 프로그램 가능한 나노물질의 플랫폼으로 바이오필름 세포외물질의 아밀로이드 성분인 Curli 섬유를 생산했다.이러한 나노 파이버는 기질과의 접착, 나노 입자 템플릿 제작, 단백질 고정화 [104]등 특정 기능을 위해 유전적으로 구성되었습니다.
설계단백질
자연 단백질은 예를 들어, 유도 진화에 의해, 기존 단백질의 기능과 일치하거나 개선된 새로운 단백질 구조를 생산할 수 있다.한 그룹은 헤모글로빈과 유사한 성질의 산소를 결합할 수 있지만 [106]일산화탄소와 결합하지 않는 나선 다발을 생성했다.유사한 단백질 구조가 생성되어 다양한 산화환원효소 활성을 지원하였고, 다른 단백질 구조는 구조적이고 순차적으로 새로운 ATP [108]효소를 형성하였다.또 다른 그룹은 불활성 소분자 클로자핀 N-옥시드에 의해 활성화될 수 있는 G-단백질 결합 수용체 패밀리를 생성했지만, 천연 리간드인 아세틸콜린에는 둔감했다. 이러한 수용체를 DREADD라고 한다.[109]새로운 기능성 또는 단백질 특이성도 계산 접근법을 사용하여 설계할 수 있다.한 연구는 두 가지 다른 계산 방법, 즉 염기서열 데이터베이스를 채굴하는 생물 정보학 및 분자 모델링 방법과 효소 특이성을 재프로그래밍하는 계산 효소 설계 방법을 사용할 수 있었다.두 방법 모두 [110]설탕에서 더 긴 사슬 알코올을 생산하기 위해 100배 이상의 특이성을 가진 효소를 설계했습니다.
또 다른 일반적인 연구는 20개의 아미노산 천연 세트의 팽창이다.정지 코돈을 제외하고 61개의 코돈이 확인되었지만, 일반적으로 모든 유기체에서 코드화된 아미노산은 20개뿐입니다.특정 코돈은 O-메틸티로신과 같은 비표준 아미노산 또는 4-플루오로페닐알라닌과 같은 외인성 아미노산을 포함한 대체 아미노산을 코드하도록 설계되어 있다.일반적으로 이러한 프로젝트는 다른 유기체의 재코딩된 난센스 억제제 tRNA-아미노아실 tRNA 합성효소 쌍을 사용한다. 그러나 대부분의 경우 상당한 엔지니어링이 필요하다.[111]
다른 연구원들은 정상 20개의 아미노산 세트를 줄임으로써 단백질 구조와 기능을 조사했다.제한된 단백질 배열 라이브러리는 아미노산 그룹이 단일 아미노산으로 [112]대체될 수 있는 단백질을 생성함으로써 만들어집니다.예를 들어 단백질 내의 여러 무극성 아미노산은 모두 하나의 무극성 [113]아미노산으로 치환할 수 있다.한 프로젝트는 9개의 아미노산만 [114]사용해도 Chorismate 뮤타아제 엔지니어링 버전이 여전히 촉매 활성을 보인다는 것을 입증했다.
연구원과 기업은 높은 활성, 최적의 산출량 및 효과를 가진 산업용 효소를 합성하기 위해 합성 생물학을 실습합니다.이러한 합성 효소는 세제나 유당 없는 유제품과 같은 제품들을 더 비용 [115]효율적으로 만드는 것을 목표로 한다.합성 생물학에 의한 대사 공학의 향상은 제약 및 발효 화학 물질을 발견하기 위해 산업에서 사용되는 생명 공학 기술의 한 예이다.합성 생물학은 생화학적 생산에서 모듈러 경로 시스템을 조사하고 대사 생산량을 증가시킬 수 있다.인공 효소 활성과 대사 반응 속도 및 수율에 대한 후속 효과는 "세포 특성을 개선하기 위한 효율적인 새로운 전략"을 개발할 수 있다.산업적으로 중요한 생화학적 생산을 위해.[116]
설계된 핵산 시스템
과학자들은 디지털 정보를 하나의 합성 DNA에 부호화할 수 있다.2012년에는 조지 M. 처치는 합성 생물학에 관한 그의 책 중 하나를 DNA로 암호화했다.5.3Mb의 데이터는 합성 [117]DNA에 저장되는 이전의 가장 큰 양의 정보보다 1000배 이상 많았다.비슷한 프로젝트가 윌리엄 셰익스피어의 완전한 소네트를 [118]DNA로 암호화했다.보다 일반적으로 NUPACK,[119] ViennaRNA,[120] Ribosome Binding Site Calculator,[121] Cello [122]및 Non-Repeative Parts[123] Calculator와 같은 알고리즘은 새로운 유전자 시스템의 설계를 가능하게 합니다.
많은 기술은 인공적인 뉴클레오티드와 아미노산을 시험관내 및 생체내 핵산과 단백질에 통합하기 위해 개발되어 왔다.예를 들어, 2014년 5월, 연구원들은 박테리아 DNA에 두 개의 새로운 인공 뉴클레오티드를 성공적으로 도입했다고 발표했다.배양 배지에 개별 인공 뉴클레오티드를 포함시킴으로써, 그들은 박테리아를 24번 교환할 수 있었다; 그들은 mRNA나 인공 [124][125][126]뉴클레오티드를 사용할 수 있는 단백질을 생성하지 않았다.
우주 탐사
합성 생물학은 지구에서 [127][128][129]보내지는 제한된 화합물로부터 우주 비행사들을 위한 자원을 생산하는 데 도움을 줄 수 있기 때문에 NASA의 관심을 높였다.특히 화성에서는 합성생물학이 현지 자원에 기반한 생산 과정을 이끌 수 있어 지구에 [127]대한 의존도가 낮은 점령된 전초기지를 개발하는 데 강력한 도구가 될 수 있다.작물의 특정 [130]환경 요인에 대한 복원력을 높이기 위해 채용된 것과 유사한 기술을 사용하여 혹독한 화성 환경에 대처할 수 있는 식물 품종 개발에 착수했습니다.
합성생명체
합성생물학의 중요한 주제 중 하나는 생체분자 및/또는 그 화학적 유사체로부터 체외로 생성된 가상의 유기체와 관련된 합성생명체이다.합성 생명 실험은 생명의 기원을 조사하거나, 생명의 특성 중 일부를 연구하거나, 또는 더 야심차게 생명이 없는 구성 요소로부터 생명을 재현하려고 시도합니다.합성생물학은 의약품 제조부터 오염된 땅과 [132]물의 해독까지 중요한 기능을 수행할 수 있는 생물을 창조하려고 한다.의학에서는 디자이너의 생물학적 부품을 새로운 종류의 치료와 진단 [132]도구의 출발점으로 사용할 수 있는 가능성을 제시합니다.
살아있는 "인공 세포"는 에너지를 포착하고, 이온 구배를 유지하고, 고분자를 포함할 수 있을 뿐만 아니라 정보를 저장하고, [133]돌연변이를 할 수 있는 완전한 합성 세포로 정의되어 왔다.아무도 그런 [133]세포를 만들 수 없었다.
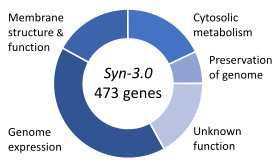
2010년 크레이그 벤터에 의해 완전히 합성된 박테리아 염색체가 만들어졌고 그의 팀은 유전적으로 비워진 박테리아 숙주 [25]세포에 그것을 도입했다.숙주 세포는 성장하고 [134][135]복제할 수 있었다.마이코플라스마 연구소는 완전히 조작된 게놈을 가진 유일한 살아있는 유기체이다.
'인공' 확장된 DNA 코드를 가진 최초의 살아있는 유기체는 2014년에 제시되었다; 연구팀은 게놈을 추출하고 확장된 유전 코드를 가진 염색체로 대체된 대장균을 사용했다.첨가된 뉴클레오시드는 d5SICS와 dNaM이다.[126]
2019년 5월, 연구자들은 20개의 [31][32]아미노산을 암호화하기 위해 박테리아 게놈에 있는 64개의 코돈의 자연수를 59개의 코돈으로 줄임으로써 박테리아 대장균의 변형인 새로운 합성(인공일 가능성이 있는) 형태의 생존 생물의 창조를 보고했다.
2017년에는 FabriCell,[137] MaxSynBio[138], BaSyC [139]등 여러 국가의 국가별 합성 세포 기구에 이어 합성 세포 구축을 위한 국제 Build-a-Cell 대규모 연구 협력이 시작되었습니다.[136]유럽의 합성 세포 노력은 2019년에 SynCell로 통합되었다.EU의 이니셔티브.[140]
약물 전달 플랫폼
공학적 박테리아 기반 플랫폼
박테리아는 오랫동안 암 치료에 사용되어 왔다.비피도박테륨과 클로스트리듐은 선택적으로 종양을 군집화하고 크기를 [141]줄인다.최근 합성 생물학자들은 특정 암 상태를 감지하고 반응하기 위해 박테리아를 재프로그래밍했다.대부분의 경우 박테리아는 치료용 분자를 종양에 직접 전달하여 목표 외의 영향을 최소화하는데 사용됩니다.종양세포를 목표로 하기 위해 종양을 특이적으로 인식할 수 있는 펩타이드를 박테리아 표면에 발현시켰다.사용되는 펩타이드는 인간 표피 성장인자 수용체[142] 2를 특이적으로 표적으로 하는 아피보디 분자와 합성 접착제를 [143]포함한다.또 다른 방법은 AND 논리 게이트를 박테리아에 [144]구축함으로써 박테리아가 종양 미세 환경, 예를 들어 저산소증을 감지하도록 하는 것입니다.그런 다음 박테리아는 용융이나[145] 세균 분비 [146]시스템을 통해서만 표적 치료 분자를 종양에 방출한다.용해는 면역체계를 자극하고 성장을 조절할 수 있다는 장점이 있다.여러 유형의 분비 시스템을 사용할 수 있으며 다른 전략도 사용할 수 있습니다.이 시스템은 외부 신호에 의해 유도된다.유도 물질에는 화학 물질, 전자파 또는 광파가 포함됩니다.
이러한 치료법에는 여러 종과 균주가 적용된다.가장 일반적으로 사용되는 박테리아는 살모넬라 티푸스, 대장균, 비피도박테리아, 스트렙토코커스, 유산균, 리스테리아, 서브틸리스균이다.이들 종들은 각각 그들만의 특성을 가지고 있으며, 조직 정착, 면역 체계와의 상호작용, 적용의 용이성 측면에서 암 치료에 독특하다.
셀 기반 플랫폼
면역체계는 암에 중요한 역할을 하고 암세포를 공격하는데 이용될 수 있다.세포 기반 치료법은 주로 T세포를 조작하여 면역요법에 초점을 맞춘다.
T세포 수용체는 암 에피토프를 검출하기 위해 조작되고 '훈련'되었다.키메라항원수용체(CARs)는 세포의 증식을 활성화 및 트리거할 수 있는 세포내 T세포 시그널링 도메인에 융합된 항체의 단편으로 구성된다.2세대 CAR 기반 치료법이 [citation needed]FDA에 의해 승인되었다.
유전자 스위치는 치료의 안전성을 높이기 위해 설계되었습니다.환자가 심각한 부작용을 [147]보일 경우 치료를 종료하기 위해 킬 스위치가 개발되었습니다.메커니즘은 시스템을 보다 세밀하게 제어하고 [148][149]정지 및 재활성화할 수 있습니다.T세포의 수는 치료 지속성과 중증도에 중요하기 때문에 치료제의 효과와 안전성에 영향을 [150]미치도록 T세포의 성장도 조절된다.
비록 몇몇 메커니즘이 안전과 제어를 개선할 수 있지만, 제한은 세포에 큰 DNA 회로를 유도하는 어려움과 세포에 외부 성분, 특히 단백질을 도입하는 것과 관련된 위험을 포함한다.
오르가노이드
합성 생물학은 의학 연구와 [151]이식에 응용되는 실험실에서 배양된 장기인 유기체에 사용되어 왔다.
바이오프린트된 장기
의료 분야에는 3D 바이오프린팅을 위한 몇 가지 응용 프로그램이 있습니다.기관연화증(TBM)으로 알려진 희귀 호흡기 질환이 있는 유아 환자에게 3D 프린팅으로 [152]만들어진 기관 부목을 투여했습니다. 3D 바이오프린팅을 사용하여 신체 여러 부위의 조직을 재구성할 수 있습니다.말기 방광 질환 환자들은 손상된 [153]장기를 재건하기 위해 조작된 방광 조직을 사용하여 치료될 수 있다.이 기술은 뼈, 피부, 연골, 근육 [154]조직에도 적용될 수 있다.3D 바이오프린팅 기술의 장기적인 목표 중 하나는 전체 장기를 재구성하는 것이지만, 완전히 기능하는 [155]장기를 프린팅하는 데는 거의 성공하지 못했습니다.이식 가능한 스텐트와 달리 장기는 복잡한 형태를 가지고 있고 바이오프린트가 상당히 어렵습니다.예를 들어 생체 인쇄된 심장은 구조 요구 사항뿐만 아니라 혈관 형성, 기계적 부하 및 전기 신호 전파 [156]요구 사항도 충족해야 합니다.이스라엘 연구원들은 2019년에 [157]인간의 세포로 토끼 크기의 심장을 만들었다.
고기와 같은 바이오프린트 식품에 대해서는 #식음료를 참조하십시오.
기타 이식 및 유도 재생
인간의 재생을[relevant?] 유도하는 합성 생물학 기반 방법과 이식 가능한 인공 장기의 창조에 대한 연구와 개발이 진행 중이다.
나노입자, 인공세포 및 마이크로액적
합성 생물학은 나노 입자를 만드는데 사용될 수 있는데, 나노 입자는 약물 전달뿐만 아니라 다른 [158]목적에도 사용될 수 있다.보완연구개발은 생물세포의 기능을 모방한 합성세포를 모색하고 만들어냈다.응용 분야에는 심장 [159][160][161]마비를 일으키는 아테롬성 경화성 플라크의 일부를 혈액 세포가 안쪽에서 바깥쪽으로 갉아먹게 하는 디자이너 나노 입자 같은 약이 포함됩니다.예를 들어, 조류 세포용 합성 마이크로액적 또는 상승성 조류 세균 다세포 구상 미생물 원자로는 수소 경제 생명공학으로 [162][163]수소를 생산하는 데 사용될 수 있다.
윤리
이 섹션은 업데이트해야 합니다.(2019년 1월) |
새로운 생명체의 창조와 기존 생명체의 조작은 합성생물학 분야에서 윤리적 문제를 제기했고 활발하게 [164][165]논의되고 있다.
일반적인 윤리적 질문은 다음과 같습니다.
- 자연을 훼손하는 것이 도덕적으로 옳은가?
- 새 생명을 창조할 때 신 노릇을 하는 건가요?
- 만약 합성 유기체가 실수로 탈출한다면 어떻게 될까요?
- 개인이 합성 생물학을 오용하여 유해한 실체(예: 생물학적 무기)를 만든다면?
- 누가 합성생물학의 산물을 통제하고 접근할 것인가?
- 이러한 혁신을 통해 누가 이익을 얻을 수 있을까요?투자자들?의료 환자요?산업 농가?
- 특허제도는 생물에 대한 특허를 허용합니까?인간의 [166]HIV 내성 유전자와 같은 유기체의 일부분은 어떨까요?
- 만약 새로운 창조물이 도덕적 또는 법적 지위를 가질 자격이 있다면?
합성생물학의 윤리적 측면은 3가지 주요 특징을 가지고 있다: 생물안전성, 생물안전성, 그리고 새로운 [167]생명체의 창조.언급된 다른 윤리적 문제에는 새로운 창조물의 규제, 새로운 창조물의 특허 관리, 이익 분배 및 연구 [168][164]무결성이 포함됩니다.
재조합 DNA 및 유전자 변형 생물(GMO) 기술에 대한 윤리적 문제가 표면화되었고 유전자 공학 및 병원체 연구에 대한 광범위한 규제가 많은 관할 지역에서 시행되었습니다.에이미 구트만 전 대통령 생명윤리위원회 위원장은 합성생물학 전반, 특히 유전공학을 지나치게 규제하려는 유혹을 피해야 한다고 주장했다.Gutmann에 따르면, "규제 절약은 새로운 기술에서 특히 중요합니다...불확실성과 미지의 공포를 바탕으로 혁신을 억누르려는 유혹이 특히 크다.법적 및 규제적 구속이라는 둔탁한 수단은 새로운 혜택의 분배를 저해할 뿐만 아니라 연구원들이 효과적인 보호장치를 [169]개발하는 것을 방해함으로써 보안과 안전에 역효과를 가져올 수 있다.
생명의 창조
한 가지 윤리적 질문은 때로는 "신 놀이"로 알려진 새로운 생명체를 만드는 것이 받아들여질 수 있는가 하는 것입니다.현재 자연계에 존재하지 않는 새로운 생명체의 생성은 소규모이며 잠재적 유익성과 위해성은 아직 알려지지 않았으며 대부분의 [164]연구에 대해 신중한 검토와 감독이 보장된다.많은 지지자들은 인공 생명체를 만드는 것의 큰 잠재적 가치를 농업, 의학, 학술적 지식 특히 표현합니다.새로운 실체의 창조는 자연현상을 연구함으로써 현재 알려진 것 이상으로 과학적 지식을 넓힐 수 있다.그러나 인공 생명체가 자연의 "순수"를 감소시키고(즉, 인간의 개입과 조작에 의해 자연이 부패될 수 있음), 생물 다양성과 자연에 초점을 맞춘 이상 대신 더 공학적 원리의 채택에 잠재적으로 영향을 미칠 수 있다는 우려가 있다.일부에서는 인공생물이 자연으로 방출되면 자연종을 물리치고 자원을 확보함으로써 생물다양성을 저해할 수 있다고 우려한다.또 다른 관심사는 새로 만들어진 실체가 고통, 지각, 자기 인식을 느낄 경우 윤리적인 처우를 하는 것입니다.이러한 권리가 어떻게 관리되거나 집행될지에 대한 합의가 이루어지지 않았지만, 그러한 생명체가 도덕적 또는 법적 권리를 부여받아야 하는지에 대한 논쟁이 계속되고 있다.
합성 생물학에 대한 윤리적 지원
는 합성 생물학의 특정 응용 프로그램을 지원하는 윤리와 도덕적 원리 기존의 농업(고기 생산 등), 동물 복지는 식량 안보와 인간 health,[170][171][172][173][174]뿐만 아니라의 잠재적으로 감소의 해로운 환경적 영향의 상당한 세계적인 문제 중 그들의 잠재력 mititgation을 포함한다.노동 요구와, v. 인간의질병 치료법, 인간의 고통 감소 및 장기 수명 연장.
바이오세이프티 및 바이오컨테인먼트
생물학적 안전 대책을 고려할 때 윤리적으로 가장 적합한 것은 무엇입니까?자연환경에서 합성생물이 우연히 유입되는 것을 어떻게 피할 수 있을까요?이러한 질문에는 많은 윤리적 고려와 비판적 고려가 이루어졌다.바이오 세이프티는 생물학적 봉쇄를 의미할 뿐만 아니라 잠재적으로 위험한 생물학적 물질로부터 대중을 보호하기 위해 취해진 진보를 의미한다.그러한 우려는 중요하고 답변이 없는 채로 남아 있지만, 합성 생물학의 모든 산물이 생물학적 안전이나 환경에 대한 부정적인 결과를 우려하는 것은 아니다.대부분의 합성 기술은 양성이고 "자연스럽지 않은" 특성 때문에 외부 세계에서 번영할 수 없다는 주장이 있다. 야생에서 적합성에 유리한 트랜스제닉 마이크로베이트의 예가 아직 없기 때문이다.
일반적으로 기존의 위험 통제, 위험 평가 방법론 및 기존 유전자 변형 생물(GMO)에 대해 개발된 규제는 합성 생물에 충분한 것으로 간주된다.실험실 맥락에서 "외부" 생물 격납 방법에는 개인 보호 장비뿐만 아니라 생체 안전 캐비닛과 글로브 박스를 통한 물리적 격납이 포함된다.농업적인 맥락에서 그들은 GMO의 생물학적 억제 방법과 유사한 격리 거리와 꽃가루 장벽을 포함합니다. 합성 유기체는 통제되지 않은 환경에서 성장을 제한하거나 수평 유전자 이동을 막는 "내적" 생물학적 억제 방법으로 설계될 수 있기 때문에 증가된 위험 제어를 제공할 수 있습니다.자연 생물내인성 바이오콘테인먼트의 예로는 보조생물, 생물학적 킬 스위치, 수정 또는 합성 유전자를 자손에게 복제하거나 전달할 수 없는 유기체, 대체 생화학(예를 들어 DNA [175][176]대신 인공 제노핵산(XNA) 사용)을 들 수 있다.보조 영양증에 관해서는 박테리아와 효모는 모든 생명에 중요한 아미노산인 히스티딘을 생산할 수 없도록 조작할 수 있다.따라서 그러한 유기체는 히스티딘이 풍부한 실험실 환경에서만 배양될 수 있으며, 바람직하지 않은 영역으로 확산될 수 있다는 두려움을 없애준다.
생물 보안
일부 윤리적 문제는 생합성 기술이 사회 및/또는 환경에 해를 끼치기 위해 의도적으로 사용될 수 있는 생물 보안과 관련이 있다.합성 생물학은 윤리적 문제와 생물 보안 문제를 제기하기 때문에, 인류는 잠재적인 유해한 창조물을 어떻게 다루어야 하는지, 그리고 사악한 생합성 기술을 막기 위해 어떤 종류의 윤리적 조치를 취할 수 있는지를 고려하고 계획해야 한다.때문에 앞서 재조합 DNA에 유전자 변형 유기체(GMO)논쟁과 유전 공학과 병원 균 연구의 광대한 규제가 이미 장소에는 많은 관할 구역에서 자란닸다 하지만 합성 생물학과 생명 공학 companies,[177][178]을 규제한 예외를 제외하고 이 사안을 새 것처럼 비치지 않는다.s.[179]
유럽 연합
유럽연합이 후원하는 프로젝트[180] SYNBIOSAFE는 합성 생물학을 관리하는 방법에 대한 보고서를 발표했다.2007년 논문은 안전, 보안, 윤리 및 과학-사회 인터페이스와 관련된 주요 이슈를 식별했으며, 이 프로젝트에서는 공교육과 과학자, 기업, 정부 및 윤리학자 [181][182]간의 지속적인 대화로 정의했다.SYNBIOSAFE가 파악한 주요 보안 문제에는 합성 DNA를 판매하는 기업과 아마추어 생물학자들의 바이오해킹 커뮤니티가 포함되어 있습니다.중요한 윤리적 문제들은 새로운 생명체의 창조와 관련이 있었다.
후속 보고서에서는 생물 보안, 특히 이중 사용 과제에 초점을 맞췄다.예를 들어, 합성 생물학은 의료 치료의 더 효율적인 생산으로 이어질 수 있지만, 유해 병원체(예: 천연두)[183]의 합성 또는 수정으로 이어질 수도 있다.오픈 소스 바이오 테크놀로지의 분산과 확산으로 인해 바이오 안전성 및 바이오 [184]보안에 대한 잠재적 우려를 추적, 규제 또는 완화하는 것이 어렵기 때문에 바이오 해킹 커뮤니티는 여전히 특별한 우려의 원천이다.
또 다른 유럽 이니셔티브인 COSY는 대중의 인식과 [185][186][187]의사소통에 초점을 맞추고 있다.COSY와 SYNBIOSAFE는 합성 생물학과 그 사회적 파장을 보다 널리 알리기 위해 2009년 10월에 [180]38분짜리 다큐멘터리 영화인 SYNBIOSAFE를 출판했습니다.
국제합성생물학회는 자율규제를 [188]제안했다.이것은 합성 생물 산업, 특히 DNA 합성 회사들이 실행해야 할 구체적인 조치들을 제안합니다.2007년, 주요 DNA 합성 회사의 과학자들이 이끄는 그룹은 "DNA 합성 [177]산업을 위한 효과적인 감독 체계를 개발하기 위한 실용적인 계획"을 발표했다.
미국
2009년 1월 알프레드 P. Sloan Foundation은 Woodrow Wilson Center, Hastings Center 및 J. Craig Venter Institute에 자금을 지원하여 합성 [189]생물학의 대중적 인식, 윤리 및 정책적 의미를 조사했습니다.
2009년 7월 9일부터 10일까지 미국 국립과학원 과학기술법위원회는 "합성생물학의 신흥 분야에서의 기회와 과제"[190]에 관한 심포지엄을 소집했다.
최초의 합성 게놈의 발표와 그에 따른 "생명"에 대한 언론의 보도가 있은 후, 버락 오바마 대통령은 합성 [191]생물학을 연구하기 위해 생명윤리 문제 연구를 위한 대통령 직속 위원회를 설립했다.위원회는 일련의 회의를 소집하여 2010년 12월에 "새로운 방향:'합성생물학과 신기술의 윤리'위원회는 "벤터의 업적은 비교적 큰 게놈을 정확하게 합성하고 다른 게놈을 대체할 수 있다는 것을 증명하는 데 있어 상당한 기술적 진보를 기록했지만,[192] 그것은 "생명체의 창조"에 해당하지 않는다"고 말했다.그것은 합성 생물학이 잠재적인 위험과 보상을 창출하는 새로운 분야라고 언급했다.위원회는 정책이나 감독 변경을 권고하지 않았으며 연구의 계속적인 자금 지원 및 모니터링, 새로운 윤리 문제 연구 및 [179]공교육을 위한 새로운 자금 지원을 요구했다.
합성 생물학은 생물학적 진보를 위한 주요 도구로서 "생물 무기를 개발하는 잠재력, 인간의 건강에 대한 예상하지 못한 부정적인 영향... 그리고 잠재적인 환경 영향"[193]을 초래한다.그러한 기술의 확산은 또한 생화학 무기의 생산을 보다 광범위한 국가 [194]및 비국가 행위자들에게 가능하게 할 수 있다.이러한 보안 문제는 정책 입법을 통해 바이오 테크놀로지의 산업 사용을 규제함으로써 피할 수 있습니다.유전자 조작에 관한 연방 지침은 "대통령의 생명윤리위원회가 제안하고 있다...화학적으로 합성된 게놈에서 자기조절세포가 만들어졌다는 발표에 대응하여, 과학을 규제하기 위한 18가지 권고안을 제시하였다.'[193]대중 교육'을 위한 것입니다.
반대
2012년 3월 13일, Friends of the Earth, International Center for Technology Assessment 및 ETC Group을 포함한 100개 이상의 환경 및 시민 사회 단체가 The Principle for the Inthetic Biology를 발표했다.이 성명서는 보다 강력한 규제와 엄격한 생물안전 대책이 확립될 때까지 합성생물의 방출과 상업적 사용을 세계적으로 유예할 것을 요구하고 있다.이 단체들은 특히 인간 게놈이나 인간 마이크로바이옴에 [195][196]대한 합성생물학 사용을 전면 금지할 것을 요구하고 있다.Richard Lewontin은 The Principle for the Supervisor of Synthetic Biology에서 논의된 감독에 대한 안전 원칙의 일부는 타당하지만, 성명서의 권고사항의 주요 문제는 "일반 대중은 그러한 권고사항의 의미 있는 실현을 강제할 수 있는 능력이 결여되어 있다"[197]는 것이라고 썼다.
건강과 안전
합성생물학의 위험에는 노동자와 일반인에 대한 생물안전 위험, 해를 입히기 위한 유기체의 고의적인 엔지니어링에서 비롯된 생물안전 위험 및 환경적 위험이 포함된다.바이오 안전성 위험은 기존의 바이오 테크놀로지 분야와 유사하며, 주로 병원균과 독성 화학물질에 노출되지만, 새로운 합성 유기체는 새로운 [198][175]위험을 가질 수 있다.생물 보안의 경우, 합성 또는 재설계된 유기체가 이론적으로 생물 테러리즘에 사용될 수 있다는 우려가 있다.잠재적인 위험에는 알려진 병원체를 처음부터 다시 만들고, 기존의 병원체를 더 위험하게 만들고, 유해한 생화학물질을 [199]생성하기 위해 미생물을 조작하는 것이 포함됩니다.마지막으로, 환경적 위험에는 합성 [200][201]유기체의 농업적 사용으로 인한 토지 이용의 잠재적 변화를 포함하여 생물 다양성과 생태계 서비스에 대한 악영향을 포함한다.합성 생물학은 의도적이거나 의도하지 않게 사람에게 해를 입히거나 환경을 해칠 수 있는 방식으로 사용될 수 있는 가능성을 가진 이중 사용 기술의 한 예이다.종종 "과학자, 주최 기관 및 자금 지원 기관"은 계획된 연구가 오용될 수 있는지 여부를 고려하고 때로는 [202]오용 가능성을 줄이기 위한 조치를 시행한다.
GMO에 대한 기존 위험 분석 시스템은 일반적으로 합성 유기체에 대해 충분한 것으로 간주되지만, 개별 유전자 [176][203]배열로부터 "상향적"으로 구축된 유기체는 어려움이 있을 수 있다.합성생물학은 일반적으로 GMO와 바이오테크놀로지에 대한 기존 규제와 하류 상업용 제품에 대한 규제를 적용받지만 [204][205]합성생물학에 특정한 규제는 없다.
「 」를 참조해 주세요.
레퍼런스
- ^ Bueso, F. Y.; Tangney, M. (2017). "Synthetic Biology in the Driving Seat of the Bioeconomy". Trends in Biotechnology. 35 (5): 373–378. doi:10.1016/j.tibtech.2017.02.002. PMID 28249675.
- ^ Hunter, D (2013). "How to object to radically new technologies on the basis of justice: the case of synthetic biology". Bioethics. 27 (8): 426–434. doi:10.1111/bioe.12049. PMID 24010854. S2CID 23895514.
- ^ Gutmann, A (2011). "The ethics of synthetic biology: guiding principles for emerging technologies". Hastings Center Report. 41 (4): 17–22. doi:10.1002/j.1552-146x.2011.tb00118.x. PMID 21845917. S2CID 20662786.
- ^ Nakano T, Eckford AW, Haraguchi T (12 September 2013). Molecular Communication. Cambridge University Press. ISBN 978-1-107-02308-6.
- ^ "Productive Nanosystems: A Technology Roadmap" (PDF). Foresight Institute.
- ^ Schwille P (September 2011). "Bottom-up synthetic biology: engineering in a tinkerer's world". Science. 333 (6047): 1252–4. Bibcode:2011Sci...333.1252S. doi:10.1126/science.1211701. PMID 21885774. S2CID 43354332.
- ^ Noireaux V, Libchaber A (December 2004). "A vesicle bioreactor as a step toward an artificial cell assembly". Proceedings of the National Academy of Sciences of the United States of America. 101 (51): 17669–74. Bibcode:2004PNAS..10117669N. doi:10.1073/pnas.0408236101. PMC 539773. PMID 15591347.
- ^ Hodgman CE, Jewett MC (May 2012). "Cell-free synthetic biology: thinking outside the cell". Metabolic Engineering. 14 (3): 261–9. doi:10.1016/j.ymben.2011.09.002. PMC 3322310. PMID 21946161.
- ^ Elani Y, Law RV, Ces O (June 2015). "Protein synthesis in artificial cells: using compartmentalisation for spatial organisation in vesicle bioreactors". Physical Chemistry Chemical Physics. 17 (24): 15534–7. Bibcode:2015PCCP...1715534E. doi:10.1039/C4CP05933F. PMID 25932977.
- ^ Elani Y, Trantidou T, Wylie D, Dekker L, Polizzi K, Law RV, Ces O (March 2018). "Constructing vesicle-based artificial cells with embedded living cells as organelle-like modules". Scientific Reports. 8 (1): 4564. Bibcode:2018NatSR...8.4564E. doi:10.1038/s41598-018-22263-3. PMC 5852042. PMID 29540757.
- ^ Lentini R, Martín NY, Forlin M, Belmonte L, Fontana J, Cornella M, Martini L, Tamburini S, Bentley WE, Jousson O, Mansy SS (February 2017). "Two-Way Chemical Communication between Artificial and Natural Cells". ACS Central Science. 3 (2): 117–123. doi:10.1021/acscentsci.6b00330. PMC 5324081. PMID 28280778.
- ^ Duc, Stéphane Le (October 26, 1910). Théorie physico-chimique de la vie et générations spontanées. A. Poinat. OL 23348076M – via The Open Library.
- ^ Leduc S (1912). Poinat A (ed.). La biologie synthétique, étude de biophysique.
- ^ Jacob, F.C. & Monod, J. 유전자 활성 조절에 대해콜드 스프링 하버.공감. 수량.Biol. 26, 193~211(1961)
- ^ Cohen SN, Chang AC, Boyer HW, Helling RB (1973). "Construction of biologically functional bacterial plasmids in vitro". Proc. Natl. Acad. Sci. USA. 70 (11): 3240–3244. Bibcode:1973PNAS...70.3240C. doi:10.1073/pnas.70.11.3240. PMC 427208. PMID 4594039.
- ^ Szybalski W, Skalka A (November 1978). "Nobel prizes and restriction enzymes". Gene. 4 (3): 181–2. doi:10.1016/0378-1119(78)90016-1. PMID 744485.
- ^ Saiki RK, Gelfand DH, Stoffel S, Scharf SJ, Higuchi R, Horn GT, Mullis KB, Erlich HA (1988). "Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase". Science. 239 (4839): 487–491. doi:10.1126/science.239.4839.487. PMID 2448875.
- ^ a b Elowitz MB, Leibler S (January 2000). "A synthetic oscillatory network of transcriptional regulators". Nature. 403 (6767): 335–8. Bibcode:2000Natur.403..335E. doi:10.1038/35002125. PMID 10659856. S2CID 41632754.
- ^ a b Gardner TS, Cantor CR, Collins JJ (January 2000). "Construction of a genetic toggle switch in Escherichia coli". Nature. 403 (6767): 339–42. Bibcode:2000Natur.403..339G. doi:10.1038/35002131. PMID 10659857. S2CID 345059.
- ^ a b Knight T (2003). "Tom Knight (2003). Idempotent Vector Design for Standard Assembly of Biobricks". hdl:1721.1/21168.
{{cite journal}}:Cite 저널 요구 사항journal=(도움말) - ^ 마틴, V. J., 피테라, D. J., 위더스, S. T., 뉴먼, J. D. & 키슬링, J. D.테르페노이드 생산을 위한 대장균의 메발론산 경로 엔지니어링.Nature Biotech. 21, 796–802(2003).
- ^ Levskaya, A.; et al. (2005). ""Synthetic biology " engineering Escherichia coli to see light". Nature. 438 (7067): 441–442. Bibcode:2005Natur.438..441L. doi:10.1038/nature04405. PMID 16306980. S2CID 4428475.
- ^ Basu, S., Gerchman, Y., Collins, C. H., Arnold, F. H. & Weiss, R. "프로그램된 패턴 형성을 위한 합성 다세포 시스템입니다.네이처 434
- ^ Anderson, J. C.; Clarke, E. J.; Arkin, A. P.; Voigt, C. A. (2006). "Environmentally controlled invasion of cancer cells by engineered bacteria". J. Mol. Biol. 355 (4): 619–627. doi:10.1016/j.jmb.2005.10.076. PMID 16330045.
- ^ a b Gibson DG, Glass JI, Lartigue C, Noskov VN, Chuang RY, Algire MA, Benders GA, Montague MG, Ma L, Moodie MM, Merryman C, Vashee S, Krishnakumar R, Assad-Garcia N, Andrews-Pfannkoch C, Denisova EA, Young L, Qi ZQ, Segall-Shapiro TH, Calvey CH, Parmar PP, Hutchison CA, Smith HO, Venter JC (July 2010). "Creation of a bacterial cell controlled by a chemically synthesized genome". Science. 329 (5987): 52–6. Bibcode:2010Sci...329...52G. doi:10.1126/science.1190719. PMID 20488990.
- ^ "American scientist who created artificial life denies 'playing God'". The Telegraph. May 2010. Archived from the original on 2022-01-12.
- ^ Dymond, J. S.; et al. (2011). "Synthetic chromosome arms function in yeast and generate phenotypic diversity by design". Nature. 477 (7365): 816–821. Bibcode:2011Natur.477..471D. doi:10.1038/nature10403. PMC 3774833. PMID 21918511.
- ^ Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E (2012). "A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity". Science. 337 (6096): 816–821. Bibcode:2012Sci...337..816J. doi:10.1126/science.1225829. PMC 6286148. PMID 22745249.
- ^ ETH Zurich (1 April 2019). "First bacterial genome created entirely with a computer". EurekAlert!. Retrieved 2 April 2019.
- ^ Venetz, Jonathan E.; et al. (1 April 2019). "Chemical synthesis rewriting of a bacterial genome to achieve design flexibility and biological functionality". Proceedings of the National Academy of Sciences of the United States of America. 116 (16): 8070–8079. doi:10.1073/pnas.1818259116. PMC 6475421. PMID 30936302.
- ^ a b Zimmer, Carl (15 May 2019). "Scientists Created Bacteria With a Synthetic Genome. Is This Artificial Life? - In a milestone for synthetic biology, colonies of E. coli thrive with DNA constructed from scratch by humans, not nature". The New York Times. Retrieved 16 May 2019.
- ^ a b Fredens, Julius; et al. (15 May 2019). "Total synthesis of Escherichia coli with a recoded genome". Nature. 569 (7757): 514–518. Bibcode:2019Natur.569..514F. doi:10.1038/s41586-019-1192-5. PMC 7039709. PMID 31092918.
- ^ Sokol, Joshua (2020-04-03). "Meet the Xenobots: Virtual Creatures Brought to Life". The New York Times.
- ^ Kriegman, Sam; Blakiston, Douglas; Levin, Michael; Bongard, Josh (7 December 2021). "Kinematic self-replication in reconfigurable organisms". Proceedings of the National Academy of Sciences of the United States of America. 118 (49). doi:10.1073/pnas.2112672118. PMC 8670470. PMID 34845026.
- ^ Zeng, Jie (Bangzhe). "On the concept of systems bio-engineering". Coomunication on Transgenic Animals, June 1994, CAS, PRC. 6.
- ^ Chopra, Paras; Akhil Kamma. "Engineering life through Synthetic Biology". In Silico Biology. 6.
- ^ Channon K, Bromley EH, Woolfson DN (August 2008). "Synthetic biology through biomolecular design and engineering". Current Opinion in Structural Biology. 18 (4): 491–8. doi:10.1016/j.sbi.2008.06.006. PMID 18644449.
- ^ Duran-Nebreda, Salva; Pla, Jordi; Vidiella, Blai; Piñero, Jordi; Conde-Pueyo, Nuria; Solé, Ricard (2021-01-15). "Synthetic Lateral Inhibition in Periodic Pattern Forming Microbial Colonies". ACS Synthetic Biology. 10 (2): 277–285. doi:10.1021/acssynbio.0c00318. ISSN 2161-5063. PMC 8486170. PMID 33449631. S2CID 231624252.
- ^ van Warmerdam, Travis (December 25, 2021). "Exploring biotechnological advancements in the exogenous biological production of industrial small molecules and pharmaceuticals". YourBioHelper.com.
{{cite web}}: CS1 maint :url-status (링크) - ^ Stone, M (2006). "Life Redesigned to Suit the Engineering Crowd" (PDF). Microbe. 1 (12): 566–570. S2CID 7171812. Archived from the original (PDF) on 2018-06-12.
- ^ Baker D, Church G, Collins J, Endy D, Jacobson J, Keasling J, Modrich P, Smolke C, Weiss R (June 2006). "Engineering life: building a fab for biology". Scientific American. 294 (6): 44–51. Bibcode:2006SciAm.294f..44B. doi:10.1038/scientificamerican0606-44. PMID 16711359.
- ^ Kosuri S, Church GM (May 2014). "Large-scale de novo DNA synthesis: technologies and applications". Nature Methods. 11 (5): 499–507. doi:10.1038/nmeth.2918. PMC 7098426. PMID 24781323.
- ^ Blight KJ, Kolykhalov AA, Rice CM (December 2000). "Efficient initiation of HCV RNA replication in cell culture". Science. 290 (5498): 1972–4. Bibcode:2000Sci...290.1972B. doi:10.1126/science.290.5498.1972. PMID 11110665.
- ^ Couzin J (July 2002). "Virology. Active poliovirus baked from scratch". Science. 297 (5579): 174–5. doi:10.1126/science.297.5579.174b. PMID 12114601. S2CID 83531627.
- ^ Smith HO, Hutchison CA, Pfannkoch C, Venter JC (December 2003). "Generating a synthetic genome by whole genome assembly: phiX174 bacteriophage from synthetic oligonucleotides". Proceedings of the National Academy of Sciences of the United States of America. 100 (26): 15440–5. Bibcode:2003PNAS..10015440S. doi:10.1073/pnas.2237126100. PMC 307586. PMID 14657399.
- ^ Wade, Nicholas (2007-06-29). "Scientists Transplant Genome of Bacteria". The New York Times. ISSN 0362-4331. Retrieved 2007-12-28.
- ^ Gibson DG, Benders GA, Andrews-Pfannkoch C, Denisova EA, Baden-Tillson H, Zaveri J, Stockwell TB, Brownley A, Thomas DW, Algire MA, Merryman C, Young L, Noskov VN, Glass JI, Venter JC, Hutchison CA, Smith HO (February 2008). "Complete chemical synthesis, assembly, and cloning of a Mycoplasma genitalium genome". Science. 319 (5867): 1215–20. Bibcode:2008Sci...319.1215G. doi:10.1126/science.1151721. PMID 18218864. S2CID 8190996.
- ^ Ball, Philip (2016). "Man Made: A History of Synthetic Life". Distillations. 2 (1): 15–23. Retrieved 22 March 2018.
- ^ Pollack, Andrew (2007-09-12). "How Do You Like Your Genes? Biofabs Take Orders". The New York Times. ISSN 0362-4331. Retrieved 2007-12-28.
- ^ "Synthetic Biology Projects". arep.med.harvard.edu. Retrieved 2018-02-17.
- ^ Forster AC, Church GM (2006-08-22). "Towards synthesis of a minimal cell". Molecular Systems Biology. 2 (1): 45. doi:10.1038/msb4100090. PMC 1681520. PMID 16924266.
- ^ a b Basulto, Dominic (November 4, 2015). "Everything you need to know about why CRISPR is such a hot technology". Washington Post. Retrieved 5 December 2015.
- ^ Kahn, Jennifer (November 9, 2015). "The Crispr Quandary". New York Times. Retrieved 5 December 2015.
- ^ Ledford, Heidi (June 3, 2015). "CRISPR, the disruptor". Nature. Nature News. 522 (7554): 20–4. Bibcode:2015Natur.522...20L. doi:10.1038/522020a. PMID 26040877.
- ^ Higginbotham, Stacey (4 December 2015). "Top VC Says Gene Editing Is Riskier Than Artificial Intelligence". Fortune. Retrieved 5 December 2015.
- ^ Rollie; et al. (2012). "Designing biological systems: Systems Engineering meets Synthetic Biology". Chemical Engineering Science. 69 (1): 1–29. doi:10.1016/j.ces.2011.10.068.
- ^ a b Freemont PS, Kitney RI (2012). Synthetic Biology – A Primer. World Scientific. doi:10.1142/p837. ISBN 978-1-84816-863-3.
- ^ a b Wright, Bradley W.; Molloy, Mark P.; Jaschke, Paul R. (2021-10-05). "Overlapping genes in natural and engineered genomes". Nature Reviews Genetics. 23 (3): 154–168. doi:10.1038/s41576-021-00417-w. ISSN 1471-0064. PMC 8490965. PMID 34611352.
- ^ a b Chan, Leon Y.; Kosuri, Sriram; Endy, Drew (2005). "Refactoring bacteriophage T7". Molecular Systems Biology. 1: 2005.0018. doi:10.1038/msb4100025. ISSN 1744-4292. PMC 1681472. PMID 16729053.
- ^ Temme, Karsten; Zhao, Dehua; Voigt, Christopher A. (2012-05-01). "Refactoring the nitrogen fixation gene cluster from Klebsiella oxytoca". Proceedings of the National Academy of Sciences of the United States of America. 109 (18): 7085–7090. Bibcode:2012PNAS..109.7085T. doi:10.1073/pnas.1120788109. ISSN 1091-6490. PMC 3345007. PMID 22509035.
- ^ Song, Miryoung; Sukovich, David J.; Ciccarelli, Luciano; Mayr, Julia; Fernandez-Rodriguez, Jesus; Mirsky, Ethan A.; Tucker, Alex C.; Gordon, D. Benjamin; Marlovits, Thomas C.; Voigt, Christopher A. (2017-05-09). "Control of type III protein secretion using a minimal genetic system". Nature Communications. 8 (1): 14737. Bibcode:2017NatCo...814737S. doi:10.1038/ncomms14737. ISSN 2041-1723. PMC 5436071. PMID 28485369.
- ^ Jaschke, Paul R.; Lieberman, Erica K.; Rodriguez, Jon; Sierra, Adrian; Endy, Drew (2012-10-15). "A fully decompressed synthetic bacteriophage øX174 genome assembled and archived in yeast". Virology. 434 (2): 278–284. doi:10.1016/j.virol.2012.09.020. PMID 23079106.
- ^ Woolfson DN, Bartlett GJ, Bruning M, Thomson AR (August 2012). "New currency for old rope: from coiled-coil assemblies to α-helical barrels". Current Opinion in Structural Biology. 22 (4): 432–41. doi:10.1016/j.sbi.2012.03.002. PMID 22445228.
- ^ Dueber JE, Wu GC, Malmirchegini GR, Moon TS, Petzold CJ, Ullal AV, Prather KL, Keasling JD (August 2009). "Synthetic protein scaffolds provide modular control over metabolic flux". Nature Biotechnology. 27 (8): 753–9. doi:10.1038/nbt.1557. PMID 19648908. S2CID 2756476.
- ^ Reddington SC, Howarth M (December 2015). "Secrets of a covalent interaction for biomaterials and biotechnology: SpyTag and SpyCatcher". Current Opinion in Chemical Biology. 29: 94–9. doi:10.1016/j.cbpa.2015.10.002. PMID 26517567.
- ^ Bayle JH, Grimley JS, Stankunas K, Gestwicki JE, Wandless TJ, Crabtree GR (January 2006). "Rapamycin analogs with differential binding specificity permit orthogonal control of protein activity". Chemistry & Biology. 13 (1): 99–107. doi:10.1016/j.chembiol.2005.10.017. PMID 16426976.
- ^ Altszyler E, Ventura A, Colman-Lerner A, Chernomoretz A (October 2014). "Impact of upstream and downstream constraints on a signaling module's ultrasensitivity". Physical Biology. 11 (6): 066003. Bibcode:2014PhBio..11f6003A. doi:10.1088/1478-3975/11/6/066003. PMC 4233326. PMID 25313165.
- ^ Altszyler E, Ventura AC, Colman-Lerner A, Chernomoretz A (2017). "Ultrasensitivity in signaling cascades revisited: Linking local and global ultrasensitivity estimations". PLOS ONE. 12 (6): e0180083. arXiv:1608.08007. Bibcode:2017PLoSO..1280083A. doi:10.1371/journal.pone.0180083. PMC 5491127. PMID 28662096.
- ^ Carbonell-Ballestero M, Duran-Nebreda S, Montañez R, Solé R, Macía J, Rodríguez-Caso C (December 2014). "A bottom-up characterization of transfer functions for synthetic biology designs: lessons from enzymology". Nucleic Acids Research. 42 (22): 14060–14069. doi:10.1093/nar/gku964. PMC 4267673. PMID 25404136.
- ^ Kaznessis YN (November 2007). "Models for synthetic biology". BMC Systems Biology. 1 (1): 47. doi:10.1186/1752-0509-1-47. PMC 2194732. PMID 17986347.
- ^ Tuza ZA, Singhal V, Kim J, Murray RM (December 2013). "An in silico modeling toolbox for rapid prototyping of circuits in a biomolecular "breadboard" system.". 52nd IEEE Conference on Decision and Control. doi:10.1109/CDC.2013.6760079.
- ^ Elani Y (June 2016). "Construction of membrane-bound artificial cells using microfluidics: a new frontier in bottom-up synthetic biology". Biochemical Society Transactions. 44 (3): 723–30. doi:10.1042/BST20160052. PMC 4900754. PMID 27284034.
- ^ Gach PC, Iwai K, Kim PW, Hillson NJ, Singh AK (October 2017). "Droplet microfluidics for synthetic biology". Lab on a Chip. 17 (20): 3388–3400. doi:10.1039/C7LC00576H. OSTI 1421856. PMID 28820204.
- ^ Vinuselvi P, Park S, Kim M, Park JM, Kim T, Lee SK (2011-06-03). "Microfluidic technologies for synthetic biology". International Journal of Molecular Sciences. 12 (6): 3576–93. doi:10.3390/ijms12063576. PMC 3131579. PMID 21747695.
- ^ a b Khalil AS, Lu TK, Bashor CJ, Ramirez CL, Pyenson NC, Joung JK, Collins JJ (August 2012). "A synthetic biology framework for programming eukaryotic transcription functions". Cell. 150 (3): 647–58. doi:10.1016/j.cell.2012.05.045. PMC 3653585. PMID 22863014.
- ^ de Almeida PE, van Rappard JR, Wu JC (September 2011). "In vivo bioluminescence for tracking cell fate and function". American Journal of Physiology. Heart and Circulatory Physiology. 301 (3): H663–71. doi:10.1152/ajpheart.00337.2011. PMC 3191083. PMID 21666118.
- ^ Close DM, Xu T, Sayler GS, Ripp S (2011). "In vivo bioluminescent imaging (BLI): noninvasive visualization and interrogation of biological processes in living animals". Sensors. 11 (1): 180–206. doi:10.3390/s110100180. PMC 3274065. PMID 22346573.
- ^ Gibbs WW (1997). "Critters on a Chip". Scientific American. Retrieved 2 Mar 2009.
- ^ Belkin, Shimshon; Yagur-Kroll, Sharon; Kabessa, Yossef; Korouma, Victor; Septon, Tali; Anati, Yonatan; Zohar-Perez, Cheinat; Rabinovitz, Zahi; Nussinovitch, Amos (April 2017). "Remote detection of buried landmines using a bacterial sensor". Nature Biotechnology. 35 (4): 308–310. doi:10.1038/nbt.3791. ISSN 1087-0156. PMID 28398330. S2CID 3645230.
- ^ Danino T, Prindle A, Kwong GA, Skalak M, Li H, Allen K, Hasty J, Bhatia SN (May 2015). "Programmable probiotics for detection of cancer in urine". Science Translational Medicine. 7 (289): 289ra84. doi:10.1126/scitranslmed.aaa3519. PMC 4511399. PMID 26019220.
- ^ "Face masks that can diagnose COVID-19". medicalxpress.com. Retrieved 11 July 2021.
- ^ Nguyen, Peter Q.; Soenksen, Luis R.; Donghia, Nina M.; Angenent-Mari, Nicolaas M.; de Puig, Helena; Huang, Ally; Lee, Rose; Slomovic, Shimyn; Galbersanini, Tommaso; Lansberry, Geoffrey; Sallum, Hani M.; Zhao, Evan M.; Niemi, James B.; Collins, James J. (28 June 2021). "Wearable materials with embedded synthetic biology sensors for biomolecule detection". Nature Biotechnology. 39 (11): 1366–1374. doi:10.1038/s41587-021-00950-3. ISSN 1546-1696. PMID 34183860. S2CID 235673261.
- ^ "Japanese scientists produce first 3D-bioprinted, marbled Wagyu beef". New Atlas. 25 August 2021. Retrieved 21 September 2021.
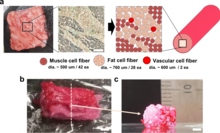
- ^ Kang, Dong-Hee; Louis, Fiona; Liu, Hao; Shimoda, Hiroshi; Nishiyama, Yasutaka; Nozawa, Hajime; Kakitani, Makoto; Takagi, Daisuke; Kasa, Daijiro; Nagamori, Eiji; Irie, Shinji; Kitano, Shiro; Matsusaki, Michiya (24 August 2021). "Engineered whole cut meat-like tissue by the assembly of cell fibers using tendon-gel integrated bioprinting". Nature Communications. 12 (1): 5059. Bibcode:2021NatCo..12.5059K. doi:10.1038/s41467-021-25236-9. ISSN 2041-1723. PMC 8385070. PMID 34429413.
- ^ Lavars, Nick (20 September 2021). "Lab-grown coffee cuts out the beans and deforestation". New Atlas. Retrieved 18 October 2021.
- ^ "Eco-friendly, lab-grown coffee is on the way, but it comes with a catch". The Guardian. 16 October 2021. Retrieved 26 October 2021.
- ^ "Sustainable coffee grown in Finland – VTT News". www.vttresearch.com. Retrieved 18 October 2021.
- ^ "Growing food with air and solar power: More efficient than planting crops". phys.org. Retrieved 11 July 2021.
- ^ Leger, Dorian; Matassa, Silvio; Noor, Elad; Shepon, Alon; Milo, Ron; Bar-Even, Arren (29 June 2021). "Photovoltaic-driven microbial protein production can use land and sunlight more efficiently than conventional crops". Proceedings of the National Academy of Sciences. 118 (26): e2015025118. doi:10.1073/pnas.2015025118. ISSN 0027-8424. PMC 8255800. PMID 34155098. S2CID 235595143.
- ^ "World-first artificial synthesis of starch from CO2 outperforms nature". New Atlas. 28 September 2021. Retrieved 18 October 2021.
- ^ Cai, Tao; Sun, Hongbing; Qiao, Jing; Zhu, Leilei; Zhang, Fan; Zhang, Jie; Tang, Zijing; Wei, Xinlei; Yang, Jiangang; Yuan, Qianqian; Wang, Wangyin; Yang, Xue; Chu, Huanyu; Wang, Qian; You, Chun; Ma, Hongwu; Sun, Yuanxia; Li, Yin; Li, Can; Jiang, Huifeng; Wang, Qinhong; Ma, Yanhe (24 September 2021). "Cell-free chemoenzymatic starch synthesis from carbon dioxide". Science. 373 (6562): 1523–1527. Bibcode:2021Sci...373.1523C. doi:10.1126/science.abh4049. PMID 34554807. S2CID 237615280.
- ^ "Spider silk made by photosynthetic bacteria". phys.org. Archived from the original on 7 August 2020. Retrieved 16 August 2020.
- ^ Foong, Choon Pin; Higuchi-Takeuchi, Mieko; Malay, Ali D.; Oktaviani, Nur Alia; Thagun, Chonprakun; Numata, Keiji (2020-07-08). "A marine photosynthetic microbial cell factory as a platform for spider silk production". Communications Biology. Springer Science and Business Media LLC. 3 (1): 357. doi:10.1038/s42003-020-1099-6. ISSN 2399-3642. PMC 7343832. PMID 32641733.
- ^ Singh V (December 2014). "Recent advances and opportunities in synthetic logic gates engineering in living cells". Systems and Synthetic Biology. 8 (4): 271–82. doi:10.1007/s11693-014-9154-6. PMC 4571725. PMID 26396651.
- ^ Purcell O, Lu TK (October 2014). "Synthetic analog and digital circuits for cellular computation and memory". Current Opinion in Biotechnology. Cell and Pathway Engineering. 29: 146–55. doi:10.1016/j.copbio.2014.04.009. PMC 4237220. PMID 24794536.
- ^ Daniel R, Rubens JR, Sarpeshkar R, Lu TK (May 2013). "Synthetic analog computation in living cells". Nature. 497 (7451): 619–23. Bibcode:2013Natur.497..619D. doi:10.1038/nature12148. PMID 23676681. S2CID 4358570.
- ^ Rinaudo K, Bleris L, Maddamsetti R, Subramanian S, Weiss R, Benenson Y (July 2007). "A universal RNAi-based logic evaluator that operates in mammalian cells". Nature Biotechnology. 25 (7): 795–801. doi:10.1038/nbt1307. PMID 17515909. S2CID 280451.
- ^ Xie Z, Wroblewska L, Prochazka L, Weiss R, Benenson Y (September 2011). "Multi-input RNAi-based logic circuit for identification of specific cancer cells". Science. 333 (6047): 1307–11. Bibcode:2011Sci...333.1307X. doi:10.1126/science.1205527. PMID 21885784. S2CID 13743291.
- ^ Nielsen AA, Der BS, Shin J, Vaidyanathan P, Paralanov V, Strychalski EA, Ross D, Densmore D, Voigt CA (April 2016). "Genetic circuit design automation". Science. 352 (6281): aac7341. doi:10.1126/science.aac7341. PMID 27034378.
- ^ Weinberg BH, Pham NT, Caraballo LD, Lozanoski T, Engel A, Bhatia S, Wong WW (May 2017). "Large-scale design of robust genetic circuits with multiple inputs and outputs for mammalian cells". Nature Biotechnology. 35 (5): 453–462. doi:10.1038/nbt.3805. PMC 5423837. PMID 28346402.
- ^ Pandi A, Koch M, Voyvodic PL, Soudier P, Bonnet J, Kushwaha M, Faulon JL (August 2019). "Metabolic perceptrons for neural computing in biological systems". Nature Communications. 10 (1): 3880. Bibcode:2019NatCo..10.3880P. doi:10.1038/s41467-019-11889-0. PMC 6713752. PMID 31462649.
- ^ Westfall PJ, Pitera DJ, Lenihan JR, Eng D, Woolard FX, Regentin R, Horning T, Tsuruta H, Melis DJ, Owens A, Fickes S, Diola D, Benjamin KR, Keasling JD, Leavell MD, McPhee DJ, Renninger NS, Newman JD, Paddon CJ (January 2012). "Production of amorphadiene in yeast, and its conversion to dihydroartemisinic acid, precursor to the antimalarial agent artemisinin". Proceedings of the National Academy of Sciences of the United States of America. 109 (3): E111–8. Bibcode:2012PNAS..109E.111W. doi:10.1073/pnas.1110740109. PMC 3271868. PMID 22247290.
- ^ Connor, Steve (28 March 2014). "Eureka! Scientists unveil giant leap towards synthetic life". The Independent. Archived from the original on 2022-05-26. Retrieved 2015-08-06.
- ^ Nguyen PQ, Botyanszki Z, Tay PK, Joshi NS (September 2014). "Programmable biofilm-based materials from engineered curli nanofibres". Nature Communications. 5: 4945. Bibcode:2014NatCo...5.4945N. doi:10.1038/ncomms5945. PMID 25229329.
- ^ Kuhlman B, Dantas G, Ireton GC, Varani G, Stoddard BL, Baker D (November 2003). "Design of a novel globular protein fold with atomic-level accuracy". Science. 302 (5649): 1364–8. Bibcode:2003Sci...302.1364K. doi:10.1126/science.1089427. PMID 14631033. S2CID 1939390.
- ^ Koder RL, Anderson JL, Solomon LA, Reddy KS, Moser CC, Dutton PL (March 2009). "Design and engineering of an O(2) transport protein". Nature. 458 (7236): 305–9. Bibcode:2009Natur.458..305K. doi:10.1038/nature07841. PMC 3539743. PMID 19295603.
- ^ Farid TA, Kodali G, Solomon LA, Lichtenstein BR, Sheehan MM, Fry BA, Bialas C, Ennist NM, Siedlecki JA, Zhao Z, Stetz MA, Valentine KG, Anderson JL, Wand AJ, Discher BM, Moser CC, Dutton PL (December 2013). "Elementary tetrahelical protein design for diverse oxidoreductase functions". Nature Chemical Biology. 9 (12): 826–833. doi:10.1038/nchembio.1362. PMC 4034760. PMID 24121554.
- ^ Wang, MS; Hecht, MH (2020). "A Completely De Novo ATPase from Combinatorial Protein Design". Journal of the American Chemical Society. 142 (36): 15230–15234. doi:10.1021/jacs.0c02954. ISSN 0002-7863. PMID 32833456. S2CID 221308093.
- ^ Armbruster BN, Li X, Pausch MH, Herlitze S, Roth BL (March 2007). "Evolving the lock to fit the key to create a family of G protein-coupled receptors potently activated by an inert ligand". Proceedings of the National Academy of Sciences of the United States of America. 104 (12): 5163–8. Bibcode:2007PNAS..104.5163A. doi:10.1073/pnas.0700293104. PMC 1829280. PMID 17360345.
- ^ Mak WS, Tran S, Marcheschi R, Bertolani S, Thompson J, Baker D, Liao JC, Siegel JB (November 2015). "Integrative genomic mining for enzyme function to enable engineering of a non-natural biosynthetic pathway". Nature Communications. 6: 10005. Bibcode:2015NatCo...610005M. doi:10.1038/ncomms10005. PMC 4673503. PMID 26598135.
- ^ Wang Q, Parrish AR, Wang L (March 2009). "Expanding the genetic code for biological studies". Chemistry & Biology. 16 (3): 323–36. doi:10.1016/j.chembiol.2009.03.001. PMC 2696486. PMID 19318213.
- ^ Davidson, AR; Lumb, KJ; Sauer, RT (1995). "Cooperatively folded proteins in random sequence libraries". Nature Structural Biology. 2 (10): 856–864. doi:10.1038/nsb1095-856. PMID 7552709. S2CID 31781262.
- ^ Kamtekar S, Schiffer JM, Xiong H, Babik JM, Hecht MH (December 1993). "Protein design by binary patterning of polar and nonpolar amino acids". Science. 262 (5140): 1680–5. Bibcode:1993Sci...262.1680K. doi:10.1126/science.8259512. PMID 8259512.
- ^ Walter KU, Vamvaca K, Hilvert D (November 2005). "An active enzyme constructed from a 9-amino acid alphabet". The Journal of Biological Chemistry. 280 (45): 37742–6. doi:10.1074/jbc.M507210200. PMID 16144843.
- ^ "Synthetic Biology Applications". www.thermofisher.com. Retrieved 2015-11-12.
- ^ Liu Y, Shin HD, Li J, Liu L (February 2015). "Toward metabolic engineering in the context of system biology and synthetic biology: advances and prospects". Applied Microbiology and Biotechnology. 99 (3): 1109–18. doi:10.1007/s00253-014-6298-y. PMID 25547833. S2CID 954858.
- ^ Church GM, Gao Y, Kosuri S (September 2012). "Next-generation digital information storage in DNA". Science. 337 (6102): 1628. Bibcode:2012Sci...337.1628C. doi:10.1126/science.1226355. PMID 22903519. S2CID 934617.
- ^ "Huge amounts of data can be stored in DNA". Sky News. 23 January 2013. Archived from the original on 2016-05-31. Retrieved 24 January 2013.
- ^ Zadeh, Joseph N.; Steenberg, Conrad D.; Bois, Justin S.; Wolfe, Brian R.; Pierce, Marshall B.; Khan, Asif R.; Dirks, Robert M.; Pierce, Niles A. (2011-01-15). "NUPACK: Analysis and design of nucleic acid systems". Journal of Computational Chemistry. 32 (1): 170–173. doi:10.1002/jcc.21596. PMID 20645303. S2CID 33709556.
- ^ Lorenz, Ronny; Bernhart, Stephan H.; Höner zu Siederdissen, Christian; Tafer, Hakim; Flamm, Christoph; Stadler, Peter F.; Hofacker, Ivo L. (2011-11-24). "ViennaRNA Package 2.0". Algorithms for Molecular Biology. 6 (1): 26. doi:10.1186/1748-7188-6-26. ISSN 1748-7188. PMC 3319429. PMID 22115189.
- ^ Salis, Howard M.; Mirsky, Ethan A.; Voigt, Christopher A. (October 2009). "Automated design of synthetic ribosome binding sites to control protein expression". Nature Biotechnology. 27 (10): 946–950. doi:10.1038/nbt.1568. ISSN 1546-1696. PMC 2782888. PMID 19801975.
- ^ Nielsen, A. A. K.; Der, B. S.; Shin, J.; Vaidyanathan, P.; Paralanov, V.; Strychalski, E. A.; Ross, D.; Densmore, D.; Voigt, C. A. (2016-04-01). "Genetic circuit design automation". Science. 352 (6281): aac7341. doi:10.1126/science.aac7341. ISSN 0036-8075. PMID 27034378.
- ^ Hossain, Ayaan; Lopez, Eriberto; Halper, Sean M.; Cetnar, Daniel P.; Reis, Alexander C.; Strickland, Devin; Klavins, Eric; Salis, Howard M. (2020-07-13). "Automated design of thousands of nonrepetitive parts for engineering stable genetic systems". Nature Biotechnology. 38 (12): 1466–1475. doi:10.1038/s41587-020-0584-2. ISSN 1546-1696. PMID 32661437. S2CID 220506228.
- ^ Pollack, Andrew (May 7, 2014). "Researchers Report Breakthrough in Creating Artificial Genetic Code". New York Times. Retrieved May 7, 2014.
- ^ Callaway, Ewen (May 7, 2014). "First life with 'alien' DNA". Nature. doi:10.1038/nature.2014.15179. S2CID 86967999. Retrieved May 7, 2014.
- ^ a b Malyshev DA, Dhami K, Lavergne T, Chen T, Dai N, Foster JM, Corrêa IR, Romesberg FE (May 2014). "A semi-synthetic organism with an expanded genetic alphabet". Nature. 509 (7500): 385–8. Bibcode:2014Natur.509..385M. doi:10.1038/nature13314. PMC 4058825. PMID 24805238.
- ^ a b Verseux, C.; Paulino-Lima, I.; Baque, M.; Billi, D.; Rothschild, L. (2016). "Synthetic Biology for Space Exploration: Promises and Societal Implications". Ambivalences of Creating Life. Ethics of Science and Technology Assessment. Vol. 45. Springer-Verlag. pp. 73–100. doi:10.1007/978-3-319-21088-9_4. ISBN 978-3-319-21087-2.
- ^ Menezes, A; Cumbers, J; Hogan, J; Arkin, A (2014). "Towards synthetic biological approaches to resource utilization on space missions". Journal of the Royal Society, Interface. 12 (102): 20140715. doi:10.1098/rsif.2014.0715. PMC 4277073. PMID 25376875.
- ^ Montague M, McArthur GH, Cockell CS, Held J, Marshall W, Sherman LA, Wang N, Nicholson WL, Tarjan DR, Cumbers J (December 2012). "The role of synthetic biology for in situ resource utilization (ISRU)". Astrobiology. 12 (12): 1135–42. Bibcode:2012AsBio..12.1135M. doi:10.1089/ast.2012.0829. PMID 23140229.
- ^ GSFC, Bill Steigerwald. "NASA - Designer Plants on Mars". www.nasa.gov. Retrieved 2020-05-29.
- ^ Hutchison CA, Chuang RY, Noskov VN, Assad-Garcia N, Deerinck TJ, Ellisman MH, Gill J, Kannan K, Karas BJ, Ma L, Pelletier JF, Qi ZQ, Richter RA, Strychalski EA, Sun L, Suzuki Y, Tsvetanova B, Wise KS, Smith HO, Glass JI, Merryman C, Gibson DG, Venter JC (March 2016). "Design and synthesis of a minimal bacterial genome". Science. 351 (6280): aad6253. Bibcode:2016Sci...351.....H. doi:10.1126/science.aad6253. PMID 27013737.
- ^ a b Connor, Steve (1 December 2014). "Major synthetic life breakthrough as scientists make the first artificial enzymes". The Independent. London. Archived from the original on 2022-05-26. Retrieved 2015-08-06.
- ^ a b Deamer D (July 2005). "A giant step towards artificial life?". Trends in Biotechnology. 23 (7): 336–8. doi:10.1016/j.tibtech.2005.05.008. PMID 15935500.
- ^ "Scientists Reach Milestone On Way To Artificial Life". NPR. 2010-05-20. Retrieved 2010-06-09.
- ^ Venter, JC. "From Designing Life to Prolonging Healthy Life". YouTube. University of California Television (UCTV). Archived from the original on 2021-12-21. Retrieved 1 February 2017.
- ^ "Build-a-Cell". Retrieved 4 Dec 2019.
- ^ "FabriCell". Retrieved 8 Dec 2019.
- ^ "MaxSynBio - Max Planck Research Network in Synthetic Biology". Retrieved 8 Dec 2019.
- ^ "BaSyC". Retrieved 8 Dec 2019.
- ^ "SynCell EU". Retrieved 8 Dec 2019.
- ^ Zu C, Wang J (August 2014). "Tumor-colonizing bacteria: a potential tumor targeting therapy". Critical Reviews in Microbiology. 40 (3): 225–35. doi:10.3109/1040841X.2013.776511. PMID 23964706. S2CID 26498221.
- ^ Gujrati V, Kim S, Kim SH, Min JJ, Choy HE, Kim SC, Jon S (February 2014). "Bioengineered bacterial outer membrane vesicles as cell-specific drug-delivery vehicles for cancer therapy". ACS Nano. 8 (2): 1525–37. doi:10.1021/nn405724x. PMID 24410085.
- ^ Piñero-Lambea C, Bodelón G, Fernández-Periáñez R, Cuesta AM, Álvarez-Vallina L, Fernández LÁ (April 2015). "Programming controlled adhesion of E. coli to target surfaces, cells, and tumors with synthetic adhesins". ACS Synthetic Biology. 4 (4): 463–73. doi:10.1021/sb500252a. PMC 4410913. PMID 25045780.
- ^ Deyneko, I.V.; Kasnitz, N.; Leschner, S.; Weiss, S. (2016). "Composing a tumor specific bacterial promoter". PLOS ONE. 11 (5): e0155338. Bibcode:2016PLoSO..1155338D. doi:10.1371/journal.pone.0155338. PMC 4865170. PMID 27171245.
- ^ Rice, KC; Bayles, KW (2008). "Molecular control of bacterial death and lysis". Microbiol Mol Biol Rev. 72 (1): 85–109. doi:10.1128/mmbr.00030-07. PMC 2268280. PMID 18322035.
- ^ Ganai, S.; Arenas, R. B.; Forbes, N. S. (2009). "Tumour-targeted delivery of TRAIL using Salmonella typhimurium enhances breast cancer survival in mice". Br. J. Cancer. 101 (10): 1683–1691. doi:10.1038/sj.bjc.6605403. PMC 2778534. PMID 19861961.
- ^ 존스, B.S., 램, L.S., 골드만, F. & Di Stasi, A.자살 유전자 이식을 통한 세포 치료제 안전성 향상.앞. 약골. 5, 254 (2014).
- ^ Wei, P; Wong, WW; Park, JS; Corcoran, EE; Peisajovich, SG; Onuffer, JJ; Weiss, A; LiWA (2012). "Bacterial virulence proteins as tools to rewire kinase pathways in yeast and immune cells". Nature. 488 (7411): 384–388. Bibcode:2012Natur.488..384W. doi:10.1038/nature11259. PMC 3422413. PMID 22820255.
- ^ Danino, T.; Mondragon-Palomino, O.; Tsimring, L.; Hasty, J. (2010). "A synchronized quorum of genetic clocks". Nature. 463 (7279): 326–330. Bibcode:2010Natur.463..326D. doi:10.1038/nature08753. PMC 2838179. PMID 20090747.
- ^ Chen, Y. Y.; Jensen, M. C.; Smolke, C. D. (2010). "Genetic control of mammalian T-cell proliferation with synthetic RNA regulatory systems". Proc. Natl. Acad. Sci. U.S.A. 107 (19): 8531–6. Bibcode:2010PNAS..107.8531C. doi:10.1073/pnas.1001721107. PMC 2889348. PMID 20421500.
- ^ Hofer, Moritz; Lutolf, Matthias P. (May 2021). "Engineering organoids". Nature Reviews Materials. 6 (5): 402–420. Bibcode:2021NatRM...6..402H. doi:10.1038/s41578-021-00279-y. ISSN 2058-8437. PMC 7893133. PMID 33623712.
- ^ Zopf DA, Hollister SJ, Nelson ME, Ohye RG, Green GE (May 2013). "Bioresorbable airway splint created with a three-dimensional printer". The New England Journal of Medicine. 368 (21): 2043–5. doi:10.1056/NEJMc1206319. PMID 23697530.
- ^ Atala A, Bauer SB, Soker S, Yoo JJ, Retik AB (April 2006). "Tissue-engineered autologous bladders for patients needing cystoplasty". Lancet. 367 (9518): 1241–6. doi:10.1016/S0140-6736(06)68438-9. PMID 16631879. S2CID 17892321.
- ^ Hong N, Yang GH, Lee J, Kim G (January 2018). "3D bioprinting and its in vivo applications". Journal of Biomedical Materials Research Part B: Applied Biomaterials. 106 (1): 444–459. doi:10.1002/jbm.b.33826. PMID 28106947.
- ^ Sommer AC, Blumenthal EZ (September 2019). "Implementations of 3D printing in ophthalmology". Graefe's Archive for Clinical and Experimental Ophthalmology. 257 (9): 1815–1822. doi:10.1007/s00417-019-04312-3. PMID 30993457. S2CID 116884575.
- ^ Cui H, Miao S, Esworthy T, Zhou X, Lee SJ, Liu C, et al. (July 2018). "3D bioprinting for cardiovascular regeneration and pharmacology". Advanced Drug Delivery Reviews. 132: 252–269. doi:10.1016/j.addr.2018.07.014. PMC 6226324. PMID 30053441.
- ^ Freeman D (April 19, 2019). "Israeli scientists create world's first 3D-printed heart using human cells". NBC News. Retrieved 2019-04-20.
- ^ Edmundson, Matthew C.; Capeness, Michael; Horsfall, Louise (25 December 2014). "Exploring the potential of metallic nanoparticles within synthetic biology". New Biotechnology. 31 (6): 572–578. doi:10.1016/j.nbt.2014.03.004. ISSN 1871-6784. PMID 24681407. S2CID 15790244.
- ^ "Nanoparticle chomps away plaques that cause heart attacks". Michigan State University. 27 January 2020. Archived from the original on 29 January 2020. Retrieved 31 January 2020.
- ^ "Nanoparticle helps eat away deadly arterial plaque". New Atlas. 28 January 2020. Archived from the original on 1 March 2020. Retrieved 13 April 2020.
- ^ Flores, Alyssa M.; Hosseini-Nassab, Niloufar; Jarr, Kai-Uwe; Ye, Jianqin; Zhu, Xingjun; Wirka, Robert; Koh, Ai Leen; Tsantilas, Pavlos; Wang, Ying; Nanda, Vivek; Kojima, Yoko; Zeng, Yitian; Lotfi, Mozhgan; Sinclair, Robert; Weissman, Irving L.; Ingelsson, Erik; Smith, Bryan Ronain; Leeper, Nicholas J. (February 2020). "Pro-efferocytic nanoparticles are specifically taken up by lesional macrophages and prevent atherosclerosis". Nature Nanotechnology. 15 (2): 154–161. Bibcode:2020NatNa..15..154F. doi:10.1038/s41565-019-0619-3. PMC 7254969. PMID 31988506.
- ^ "Research creates hydrogen-producing living droplets, paving way for alternative future energy source". phys.org. Archived from the original on 16 December 2020. Retrieved 9 December 2020.
- ^ Xu, Zhijun; Wang, Shengliang; Zhao, Chunyu; Li, Shangsong; Liu, Xiaoman; Wang, Lei; Li, Mei; Huang, Xin; Mann, Stephen (25 November 2020). "Photosynthetic hydrogen production by droplet-based microbial micro-reactors under aerobic conditions". Nature Communications. 11 (1): 5985. doi:10.1038/s41467-020-19823-5. ISSN 2041-1723. PMC 7689460. PMID 33239636.
- ^ a b c Newson, AJ (2015). "Synthetic Biology: Ethics, Exceptionalism and Expectations". Macquarie Law Journal. 15: 45.
- ^ U. N. Environment (2019-03-04). "Frontiers 2018/19: Emerging Issues of Environmental Concern". UNEP - UN Environment Programme. Retrieved 2021-01-03.
- ^ Staff, Agencies (November 2018). "World's first gene-edited babies created in China, claims scientist". The Guardian.
- ^ Hayry, Mattie (April 2017). "Synthetic Biology and Ethics: Past, Present, and Future". Cambridge Quarterly of Healthcare Ethics. 26 (2): 186–205. doi:10.1017/S0963180116000803. PMID 28361718. S2CID 29992939.
- ^ Jin, Shan; et al. (September 2019). "Synthetic biology applied in the agrifood sector: Public perceptions, attitudes and implications for future studies". Trends in Food Science and Technology. 91: 454–466. doi:10.1016/j.tifs.2019.07.025. S2CID 199632852.
- ^ Amy, Gutmann (2012). "The Ethics of Synthetic Biology". The Hastings Center Report. 41 (4): 17–22. doi:10.1002/j.1552-146X.2011.tb00118.x. PMID 21845917. S2CID 20662786.
- ^ Bryant, Christopher J (3 August 2020). "Culture, meat, and cultured meat". Journal of Animal Science. 98 (8): skaa172. doi:10.1093/jas/skaa172. ISSN 0021-8812. PMC 7398566. PMID 32745186.
- ^ Hong, Tae Kyung; Shin, Dong-Min; Choi, Joonhyuk; Do, Jeong Tae; Han, Sung Gu (May 2021). "Current Issues and Technical Advances in Cultured Meat Production: AReview". Food Science of Animal Resources. 41 (3): 355–372. doi:10.5851/kosfa.2021.e14. ISSN 2636-0772. PMC 8112310. PMID 34017947.
- ^ Treich, Nicolas (1 May 2021). "Cultured Meat: Promises and Challenges". Environmental and Resource Economics. 79 (1): 33–61. doi:10.1007/s10640-021-00551-3. ISSN 1573-1502. PMC 7977488. PMID 33758465.
- ^ Bryant, Christopher J (1 August 2020). "Culture, meat, and cultured meat". Journal of Animal Science. 98 (8): skaa172. doi:10.1093/jas/skaa172. PMC 7398566. PMID 32745186.
- ^ Treich, Nicolas (May 2021). "Cultured Meat: Promises and Challenges". Environmental and Resource Economics. 79 (1): 33–61. doi:10.1007/s10640-021-00551-3. PMC 7977488. PMID 33758465.
- ^ a b Howard, John; Murashov, Vladimir; Schulte, Paul (2016-10-18). "Synthetic biology and occupational risk". Journal of Occupational and Environmental Hygiene. 14 (3): 224–236. doi:10.1080/15459624.2016.1237031. ISSN 1545-9624. PMID 27754800. S2CID 205893358.
- ^ a b European Commission. Directorate General for Health Consumers (2016-02-12). Opinion on synthetic biology II: Risk assessment methodologies and safety aspects. EU Directorate-General for Health and Consumers. Publications Office. doi:10.2772/63529. ISBN 9789279439162.
- ^ a b Bügl H, Danner JP, Molinari RJ, Mulligan JT, Park HO, Reichert B, Roth DA, Wagner R, Budowle B, Scripp RM, Smith JA, Steele SJ, Church G, Endy D (June 2007). "DNA synthesis and biological security". Nature Biotechnology. 25 (6): 627–9. doi:10.1038/nbt0607-627. PMID 17557094. S2CID 7776829.
- ^ "Ethical Issues in Synthetic Biology: An Overview of the Debates" (PDF).
- ^ a b 대통령 직속 생명윤리문제연구위원회, 2010년 12월 합성생물학과 신기술의 윤리성 2012-04-14.
- ^ a b "Synbiosafe". Synbiosafe.
- ^ Schmidt M, Ganguli-Mitra A, Torgersen H, Kelle A, Deplazes A, Biller-Andorno N (December 2009). "A priority paper for the societal and ethical aspects of synthetic biology" (PDF). Systems and Synthetic Biology. 3 (1–4): 3–7. doi:10.1007/s11693-009-9034-7. PMC 2759426. PMID 19816794.
- ^ 슈미트 M. 켈레 AGanguli A, de Vfrient H. (Eds.) 2009."합성 생물학입니다. 「테크노사이언스와 그 사회적 결과」.스프링거 학술 출판사
- ^ Kelle A (December 2009). "Ensuring the security of synthetic biology-towards a 5P governance strategy". Systems and Synthetic Biology. 3 (1–4): 85–90. doi:10.1007/s11693-009-9041-8. PMC 2759433. PMID 19816803.
- ^ Schmidt M (June 2008). "Diffusion of synthetic biology: a challenge to biosafety" (PDF). Systems and Synthetic Biology. 2 (1–2): 1–6. doi:10.1007/s11693-008-9018-z. PMC 2671588. PMID 19003431.
- ^ COSY: 합성생물학 커뮤니케이션
- ^ Kronberger N, Holtz P, Kerbe W, Strasser E, Wagner W (December 2009). "Communicating Synthetic Biology: from the lab via the media to the broader public". Systems and Synthetic Biology. 3 (1–4): 19–26. doi:10.1007/s11693-009-9031-x. PMC 2759424. PMID 19816796.
- ^ Cserer A, Seiringer A (December 2009). "Pictures of Synthetic Biology : A reflective discussion of the representation of Synthetic Biology (SB) in the German-language media and by SB experts". Systems and Synthetic Biology. 3 (1–4): 27–35. doi:10.1007/s11693-009-9038-3. PMC 2759430. PMID 19816797.
- ^ 2011년 7월 19일 뮌헨 웨이백 머신에서 보관된 "합성 생물 보안을 위한 기술 솔루션" 보고서, 2008년
- ^ Parens E., Johnston J., Moses J. 합성생물학의 윤리적 문제.2009.
- ^ "Policy and Global Affairs at the National Academies". sites.nationalacademies.org.
- ^ 대통령 직속 생명윤리문제연구위원회, 2010년 12월 FAQ
- ^ 합성생물학 F.A.Q. 생명윤리문제연구위원회
- ^ a b Erickson B, Singh R, Winters P (September 2011). "Synthetic biology: regulating industry uses of new biotechnologies". Science. 333 (6047): 1254–6. Bibcode:2011Sci...333.1254E. doi:10.1126/science.1211066. PMID 21885775. S2CID 1568198.
- ^ Kelley M. Sayler (June 8, 2021). Defense Primer: Emerging Technologies (PDF) (Report). Congressional Research Service. Retrieved July 22, 2021.
- ^ 하버드 매거진의 캐서린 쉬에입니다.2014년 9월~10월 합성생물학 뉴메나게리
- ^ Scidev.net의 Yojana Sharma 2012년 3월 15일NGO는 합성 생물학의 국제적 규제를 요구한다
- ^ 새로운 합성 생물학: 누가 이득을 보는가? (2014-05-08), 리차드 C. Lewontin, 뉴욕리뷰 오브 북스
- ^ Howard, John; Murashov, Vladimir; Schulte, Paul (2017-01-24). "Synthetic Biology and Occupational Risk". Journal of Occupational and Environmental Hygiene. 14 (3): 224–236. doi:10.1080/15459624.2016.1237031. PMID 27754800. S2CID 205893358. Retrieved 2018-11-30.
- ^ National Academies Of Sciences, Engineering; Division on Earth Life Studies; Board On Life, Sciences; Board on Chemical Sciences Technology; Committee on Strategies for Identifying Addressing Potential Biodefense Vulnerabilities Posed by Synthetic Biology (2018-06-19). Biodefense in the Age of Synthetic Biology. National Academies of Sciences, Engineering, and Medicine. doi:10.17226/24890. ISBN 9780309465182. PMID 30629396. S2CID 90767286.
- ^ "Future Brief: Synthetic biology and biodiversity". European Commission. September 2016. pp. 14–15. Retrieved 2019-01-14.
- ^ Final opinion on synthetic biology III: Risks to the environment and biodiversity related to synthetic biology and research priorities in the field of synthetic biology. EU Directorate-General for Health and Food Safety. 2016-04-04. pp. 8, 27. ISBN 9789279549731. Retrieved 2019-01-14.
- ^ El Karoui, Meriem; Hoyos-Flight, Monica; Fletcher, Liz (2019). "Future Trends in Synthetic Biology—A Report". Frontiers in Bioengineering and Biotechnology. 7: 175. doi:10.3389/fbioe.2019.00175. ISSN 2296-4185. PMC 6692427. PMID 31448268.
- ^ Bailey, Claire; Metcalf, Heather; Crook, Brian (2012). "Synthetic biology: A review of the technology, and current and future needs from the regulatory framework in Great Britain" (PDF). UK Health and Safety Executive. Retrieved 2018-11-29.
- ^ Pei, Lei; Bar‐Yam, Shlomiya; Byers‐Corbin, Jennifer; Casagrande, Rocco; Eichler, Florentine; Lin, Allen; Österreicher, Martin; Regardh, Pernilla C.; Turlington, Ralph D. (2012), "Regulatory Frameworks for Synthetic Biology", Synthetic Biology, John Wiley & Sons, Ltd, pp. 157–226, doi:10.1002/9783527659296.ch5, ISBN 9783527659296
- ^ Trump, Benjamin D. (2017-11-01). "Synthetic biology regulation and governance: Lessons from TAPIC for the United States, European Union, and Singapore". Health Policy. 121 (11): 1139–1146. doi:10.1016/j.healthpol.2017.07.010. ISSN 0168-8510. PMID 28807332.
참고 문헌
- Church, George; Regis, Ed (2012). How Synthetic Biology will Reinvent Nature and Ourselves. New York, NY: Basic Books. ISBN 978-0465021758.
- 유럽위원회(2016년) 합성생물학과 생물다양성; 환경정책을 위한 과학(2016년); 미래개요 15.브리스톨, UWE, Science Communication Unit에 의해 European Communications DG Environment용으로 제작되었습니다.유럽위원회 - 환경정책을 위한 과학, PDF, 36페이지.
- Venter, Craig (2013). Life at the Speed of Light: The Double Helix and the Dawn of Digital Life. New York, NY: Penguin Books. ISBN 978-0670025404.
외부 링크
- 합성 생물학 서적 인기 과학 서적 및 교과서
- 합성생물학 개요합성생물학의 개념, 개발 및 응용에 대한 간결한 개요
- 합성 생물학에 관한 협업 개요 기사
- 논란이 되고 있는 DNA 스타트업은 고객이 생명체를 만들 수 있도록 하고 싶다(2015-01-03), 샌프란시스코 크로니클
- 살아있다, 하지만 생명이다: 합성생물학과 창조의 미래 (2016년 9월 28일), 세계과학축제