인간 마이크로바이옴
Human microbiome인간 마이크로바이옴은 피부, 유선, 정액, 자궁, 난포, 폐, 침, 구강 점막, 결막, 담도, 위장관을 포함한 [1]해당 해부학적 부위와 함께 인체 조직과 생체유체에 존재하는 모든 미생물들의 집합체이다.인간 마이크로바이오타의 종류에는 박테리아, 고세균, 곰팡이, 원생동물,비록 미세동물은 인간의 몸에서도 살 수 있지만, 그들은 전형적으로 이 정의에서 제외된다.유전체학에서 인간 마이크로바이옴이라는 용어는 때때로 상주 미생물의 [2]집단 게놈을 가리키는 데 사용되기도 하지만, 인간 메타제놈이라는 용어는 같은 [1]의미를 가지고 있다.
인간은 인간의 [3]세포와 거의 같은 크기의 비인간 세포를 가진 많은 미생물에 의해 식민지화되어 있다.인간을 식민지로 만드는 어떤 미생물들은 인간을 해치지 않고 공존한다는 것을 의미하며, 다른 미생물들은 인간의 [2]: 700 [4]숙주와 상호주의적 관계를 맺고 있다.반대로, 일부 비병원성 미생물은 트리메틸아민과 같은 대사물을 통해 숙주를 해칠 수 있는데, 트리메틸아민은 인체가 FMO3 [5][6]매개 산화를 통해 트리메틸아민 N-옥시드로 변환됩니다.특정 미생물은 숙주에게 유용한 것으로 알려진 작업을 수행하지만, 대부분의 미생물의 역할은 잘 알려져 있지 않다.존재할 것으로 예상되는 것, 그리고 정상적인 상황에서 질병을 일으키지 않는 것은 때때로 정상적인 식물군 또는 정상적인 [2]미생물군으로 간주된다.
Human Microbiom Project (HMP)는 특히 피부, 입, 코,[2] 소화관, 질에 일반적으로 서식하는 미생물군에 초점을 맞춘 인간 미생물군의 게놈 염기서열 분석 프로젝트를 수행했다.그것은 최초 [7]결과를 발표했던 2012년에 이정표에 도달했다.
용어.
−4500 — – — – −4000 — – — – −3500 — – — – −3000 — – — – −2500 — – — – −2000 — – — – −1500 — – — – −1000 — – — – −500 — – — – 0 — |
| |||||||||||||||||||||||||||||||||||||||||||||
플로라 또는 마이크로플로라로 널리 알려져 있지만, 이것은 기술적인 용어로는 잘못된 명칭이다. 왜냐하면 뿌리 플로라는 식물과 관련이 있고, 바이오타는 특정 생태계에 있는 유기체의 총집합을 의미하기 때문이다.최근에는 microbiota라는 용어가 더 적합하지만, 그 사용이 세균이나 다른 미생물에 대한 식물군의 확립된 사용과 인식을 약화시키지는 않았다.두 용어 모두 다른 [4]문헌에서 사용되고 있다.
상대수
2014년 현재, 인체에 있는 미생물 세포가 인간의 세포보다 약 10배 많다는 것이 대중 매체와 과학 문헌에 보도되었다. 이 수치는 인간 마이크로바이옴이 약 100조 개의 박테리아 세포를 포함하고 있고 성인 인간이 일반적으로 약 10조 개의 인간 세포를 가지고 있다는 추정에 기초하였다.2014년에 .[8], 미국 아카데미 미생물학, 그리고 의 또는 최근의 연구는 인간 세포의 수를 새로운 측정치를 약 37.2조 –에,microbial-to-human 세포의 비율을 의미하며, 도착했다고를 자주 묻는 질문이 미생물 세포의 수와 인간 세포의 수는 모두 추정을 강조했다를 출간했다.igi100조 박테리아 세포의 nal 추정치는 정확하며, 3:[8][9]1에 가깝다.2016년에 다른 그룹은 이 비율에 대한 새로운 추정치를 약 1:1(1.3:1), "표준 70kg[[10][3]150파운드] 남성의 모집단에 대한 불확실성과 53%의 변동"으로 발표했다.
보다 최근의 추정치는 모든 인간 세포에 대해 1.3:1의 박테리아 세포 비율이 있는 반면, 화지와 바이러스의 수는 최소한 한 단계 이상 박테리아 세포보다 많다.박테리아 유전자(내장에 있는 박테리아 1000종이 한 종당 2000개의 유전자로 가정)의 수는 약 2만 개의 인간 [11]유전자의 100배인 200만 개로 추정된다.
스터디
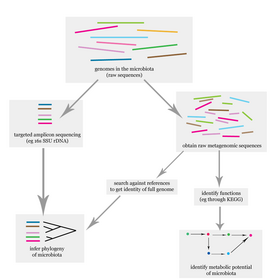
인간 마이크로바이옴을 설명하는 문제는 기본적으로 박테리아, 진핵생물,[12] 바이러스를 포함하는 미생물 집단의 구성원을 확인하는 것입니다.이는 주로 디옥시리보핵산(DNA) 기반 연구를 사용하여 이루어지지만 리보핵산(RNA), 단백질 및 대사물 기반 연구도 수행됩니다.[12][13]DNA 기반 마이크로바이옴 연구는 일반적으로 표적 엠프리콘 연구 또는 최근에는 산탄총 메타게노믹 연구로 분류될 수 있다.전자는 특정 알려진 마커 유전자에 초점을 맞추고 분류학적으로 주로 유용한 반면, 후자는 [12]공동체의 기능적 잠재력을 연구하는 데 사용될 수 있는 전체 메타제노믹 접근법이다.인간 마이크로바이옴 연구에는 있지만 다른 메타제노믹 연구에는 없는 과제 중 하나는 [14]숙주 DNA를 연구에 포함시키지 않는 것이다.
단순히 인간 마이크로바이옴의 구성을 설명하는 것 외에, 인간 마이크로바이옴과 관련된 주요 질문 중 하나는 "핵심"이 있는지 여부, 즉 대부분의 [15][16]인간들과 공유되는 공동체의 하위 집합이 있는지 여부이다.핵심이 있으면 특정 커뮤니티 구성을 질병 상태와 연관지을 수 있는데, 이는 HMP의 목표 중 하나이며, 인간 마이크로바이옴(내장 마이크로바이오타 등)은 단일 피험자 내에서나 개인 간에 매우 가변적인 것으로 알려져 있으며, 이러한 현상은 [4]생쥐에서도 관찰된다.
2012년 6월 13일, 미국 국립보건원(NIH)의 프란시스 [7]콜린스 소장에 의해 HMP의 주요 이정표가 발표되었다.이날 발표에는 네이처에 게재된[17][18] 일련의 공동 논문과 공공과학도서관(PLoS)의 여러 저널이 함께 게재됐다.HMP의 연구진은 건강한 인간의 정상적인 미생물 구성을 게놈 배열 기술을 이용해 지도화함으로써 기준 데이터베이스와 인간의 정상적인 미생물 변이의 경계를 만들었다.건강한 미국 자원봉사자 242명을 대상으로 입, 코, 피부, 장, 질 등 신체 부위 15개에서 18개까지 5000개 이상의 샘플을 채취했다.인간과 미생물을 포함한 모든 DNA를 DNA 염기서열 분석기로 분석했습니다.미생물 게놈 데이터는 세균 특이 리보솜 RNA, 16S rRNA를 식별하여 추출하였다.연구원들은 10,000종 이상의 미생물이 인간의 생태계를 점유하고 있다고 계산했고, 그들은 그 속들의 81-99%를 확인했습니다.
처리후분석
통계 분석은 얻어진 결과를 검증하는 데 필수적입니다(그룹 간 차이를 크기 조정하기 위해 ANOVA를 사용할 수 있습니다). 그래픽 도구와 쌍을 이루면 결과를 쉽게 시각화하고 [19]이해할 수 있습니다.
일단 메타제놈이 조립되면 마이크로바이옴의 기능적 잠재력을 추론할 수 있다.이러한 유형의 분석에 대한 계산상의 어려움은 단일 게놈보다 더 크다. 왜냐하면 보통 메타제놈 조립자는 품질이 떨어지고, 많은 회복된 유전자는 불완전하거나 조각화되기 때문이다.유전자 동정 단계 후,[20] 데이터는 오르톨로그 데이터베이스에 대한 표적 유전자의 다중 정렬에 의해 기능적 주석을 실시하기 위해 사용될 수 있다.
마커 유전자 분석
프라이머를 이용해 특정 유전자 영역을 공략하고 미생물 계통 발생을 판별하는 기술이다.유전적 영역은 상세한 식별을 제공할 수 있는 매우 가변적인 영역으로 특징지어진다. 즉, 보존된 영역으로 구분되며, PCR에서 사용되는 프라이머의 결합 부위로 기능한다.박테리아와 고세균을 특징짓는 데 사용되는 주요 유전자는 16S rRNA 유전자이며, 곰팡이 식별은 내부 전사 스페이서(ITS)에 기초한다.이 기술은 빠르고 그렇게 비싸지 않으며 미생물 샘플의 낮은 분해능 분류를 얻을 수 있습니다; 이것은 숙주 DNA에 의해 오염될 수 있는 샘플에 최적입니다. 프라이머 친화력은 모든 DNA 배열에 따라 다르며, 증폭 반응 중에 편향을 초래할 수 있습니다; 사실, 저농도 샘플은 오버암에 취약합니다.PCR 사이클이 증가할 경우 다른 오염 미생물이 과잉으로 나타날 수 있으므로 도화 오류.따라서 프라이머 선택을 최적화하면 시료에 존재하는 미생물과 그 상대적인 [21]함량에 대한 완전한 지식이 필요하지만 이러한 오류를 줄일 수 있습니다.
마커 유전자 분석은 프라이머 선택에 의해 영향을 받을 수 있습니다; 이러한 종류의 분석에서는 잘 검증된 프로토콜(지구 마이크로바이옴 프로젝트에서 사용되는 프로토콜 등)을 사용하는 것이 바람직합니다.마커 유전자 엠프리콘 분석에서 가장 먼저 해야 할 일은 배열 오류를 제거하는 것입니다. 많은 배열 플랫폼은 매우 신뢰할 수 있지만, 대부분의 명백한 배열 다양성은 배열 과정에서의 오류에 기인합니다.이 현상을 줄이기 위해 첫 번째 접근법은 시퀀스를 운영 분류 단위(OTU)로 클러스터링하는 것이다.이 프로세스는 유사한 시퀀스(통상 97%의 유사성 임계값)를 추가 분석 단계에서 사용할 수 있는 단일 피쳐로 통합한다.그러나 이 방법은 SNP를 단일 클러스터로 클러스터링하기 때문에 폐기한다.e OTU. 또 다른 접근방식은 올리고타이핑이다.올리고타이핑은 작은 뉴클레오티드 변화를 검출하기 위한 16s rRNA 시퀀싱의 위치 고유 정보를 포함하고 밀접하게 관련된 구별된 분류군을 구별하지 않는다.이 방법들은 [21]OTU가 아닌 샘플당 DNA 배열과 다른 배열의 카운트의 표를 출력으로 제공한다.
분석의 또 다른 중요한 단계는 데이터의 미생물 배열에 분류학적 이름을 할당하는 것이다.이는 약 80%의 속 수준에서 정확도에 도달할 수 있는 기계 학습 접근방식을 사용하여 수행될 수 있다.다른 일반적인 분석 패키지는 참조 데이터베이스와 정확히 일치하는 분류학적 분류를 지원하며, 더 큰 특이성을 제공하지만 민감도는 낮다.분류되지 않은 미생물은 소기관 [21]배열을 추가로 확인해야 합니다.
계통학적 분석
계통학적 추론을 이용하는 많은 방법들은 진핵생물의 경우 16SRNA 유전자를 사용하고 진핵생물의 경우 18SRNA 유전자를 사용한다.계통비교법(PCS)은 미생물의 여러 특성을 비교한 것이다.원칙은 그것들이 밀접하게 관련될수록 그들이 공유하는 특성의 수가 많아지는 것이다.일반적으로 PCS는 보다 유의한 결과를 얻기 위해 계통일반화최소제곱(PGLS) 또는 기타 통계분석과 결합됩니다.조상들의 상태 재구성은 특성이 알려지지 않은 분류군의 특성 값을 귀속시키기 위해 마이크로바이옴 연구에서 사용된다.이것은 일반적으로 사용 가능한 데이터베이스에 의존하는 PICRUSt에서 수행됩니다.계통발생학적 변수는 연구의 유형에 따라 연구자에 의해 선택된다. 즉, 중요한 생물학적 정보를 가진 일부 변수의 선택을 통해 [22]분석할 데이터의 차원을 줄일 수 있다.
계통 인식 거리는 보통 UniFrac 또는 유사한 도구(예: Soresen 지수 또는 Rao의 D)를 사용하여 서로 다른 군집 간의 차이를 정량화하기 위해 수행된다.이 모든 방법들은 오류를 일으키고 먼 종들의 상관관계를 이끌 수 있기 때문에 수평 유전자 전달에 의해 부정적인 영향을 받는다.HGT의 부정적인 영향을 줄이기 위한 다른 방법들이 있다: 추정적인 HGT [22]사건의 확률을 평가하기 위해 다중 유전자 또는 계산 도구를 사용하는 것이다.
종류들
박테리아

박테리아와 효모와 같은 미생물 집단은 신체의 다양한 부분에 있는 피부와 점막 표면에 서식합니다.그들의 역할은 정상적이고 건강한 인간의 생리의 일부를 형성하지만, 미생물 수가 그들의 전형적인 범위를 넘어 증가하거나(종종 면역 체계 손상으로 인해), 또는 미생물이 일반적으로 식민지화되거나 멸균되지 않은(예: 혈액, 하부 호흡기 또는 복부) 신체 부위에 서식하고 있는 경우(각각 균혈증/패혈증, 폐렴 및 복막염을 [medical citation needed]유발할 수 있습니다.)
Human Microbiom Project는 개개인이 수천 종류의 박테리아를 숙주한다는 것을 알아냈습니다. 각기 다른 신체 부위가 그들만의 독특한 공동체를 가지고 있습니다.피부와 질 부위는 입과 내장에 비해 다양성이 작아 가장 풍부한 것으로 나타났다.인체 내 특정 부위의 세균 구성은 유형뿐만 아니라 풍부하게 사람마다 다르다.입 안에서 발견되는 같은 종의 박테리아는 여러 아형이며, 입 안에서 뚜렷하게 다른 위치에 사는 것을 선호한다.이전에는 잘 이해된 것으로 생각되었던 인간의 장내 장형도 분류군의 [23][24]경계가 불분명한 광범위한 공동체에서 왔다.
인간의 내장에는 500~1,000종의 박테리아가 살고 있는 것으로 추정되지만, 박테리오타, 박테리오타 등이 우세한 것으로 추정되고 있지만, 또한 Pseudomonadota, Verucic brobiota, Actinobacteriota, Fusobacteriota, 그리고 Cyanobacteria도 [25]있다.
Actinomyces viscosus와 A. naeslundii와 같은 많은 종류의 박테리아들이 플라그라고 불리는 끈적끈적한 물질의 일부인 입 안에 산다.만약 칫솔질을 통해 이것이 제거되지 않는다면, 그것은 미적분학으로 굳어진다.이 박테리아는 치아 에나멜을 녹이는 산을 분비해 충치를 일으킨다.
질 미세 꽃은 대부분 다양한 유산균 종으로 이루어져 있다.이 종들 중 가장 흔한 것은 락토바실루스 애시드필루스라고 오랫동안 생각되어 왔지만, 나중에 L. iners가 가장 흔하고, L. crispatus가 그 다음이라는 것이 증명되었다.질에서 발견되는 다른 락토바실리는 L. jensenii, L. delbruekii 및 L. gasseri이다.질세포의 교란은 세균성 질염이나 칸디다증("효모 감염")과 같은 감염을 초래할 수 있습니다.
고세균
고세균은 인간의 장에 존재하지만, 이 장기에 있는 엄청난 종류의 박테리아와 대조적으로, 고세균의 수는 훨씬 [26]더 제한적이다.지배적인 그룹은 메타노겐, 특히 메타노브레비박터 스미스와 메타노세라 스타드마네이다.[27]그러나 메타노겐에 의한 군집화는 가변적이며, 인간의 약 50%만이 이러한 [28]유기체의 개체군을 쉽게 발견할 수 있다.
일부 메타노겐의 존재와 인간 치주 [31]질환 사이의 관계가 제안되었지만, 2007년 현재 고고학적 병원체의 명확한 예는 알려져 [29][30]있지 않다.
곰팡이
곰팡이, 특히 효모는 인간의 내장에 [32][33][34][35]존재한다.이들 중 가장 잘 연구된 것은 면역력이 저하된 [33][34][35]숙주에서도 병원성이 되는 능력으로 인해 칸디다 종이다.효모는 또한 말라세지아 종과 같은 [32]피부에도 존재하는데, 말라세지아는 [36][37]피지선에서 분비되는 기름을 소비합니다.
바이러스
바이러스, 특히 박테리아 바이러스는 다양한 신체 부위를 식민지로 만듭니다.이러한 식민지화된 부위는 피부,[38] 내장,[39] 폐,[40][41] 구강을 포함한다.바이러스 집단은 몇몇 질병과 연관되어 있으며 단순히 세균 [42][43][44]집단을 반영하는 것이 아니다.
해부학적 영역
피부.
건강한 사람 10명당 20개의 피부 부위를 조사한 결과, 19개의 박테리아 균에서 205개의 속들이 확인되었으며, 대부분의 염기서열은 4개의 균에 할당되었다.방선균류(51.8%), 바실로타(24.4%), 의사도모나도타(16.5%), 박테로이드타(6.3%)[45] 순으로 나타났다.건강한 인간의 피부에는 많은 진균속들이 존재하며, 신체 부위에 따라 다소 차이가 있지만, 병리학적 조건에서는 특정 속들이 영향을 받는 [32]지역에서 지배적인 경향이 있다.예를 들어 말라세지아는 아토피 피부염에, 아크레모늄은 비듬에 영향을 받는 [32]메스에 우세하다.
피부는 병원성 미생물의 침입을 막는 장벽 역할을 한다.인간의 피부는 피부 안이나 피부에 상주하는 미생물을 포함하고 있으며 거주성 또는 일시적인 것일 수 있습니다.상주 미생물 유형은 인체의 피부 유형에 따라 다릅니다.대부분의 미생물은 피부의 표면 세포에 살거나 분비선과 결합하는 것을 선호합니다.기름이나 땀샘과 같은 분비선은 미생물에게 물, 아미노산, 지방산을 공급한다.또한, 기름샘과 연관된 상주 박테리아는 종종 그램 양성이고 [2]병원성일 수 있습니다.
결막
결막에는 [32][46]보통 소량의 박테리아와 곰팡이가 존재한다.그램 양성 구균(예: 포도상구균 및 연쇄상구균)과 그램 음성 막대 및 구균(예: 해모필루스 및 네이세리아)이 있다.[46]곰팡이속에는 칸디다, 아스페르길루스, 페니실륨 [32]등이 있다.눈물샘은 지속적으로 분비되어 결막을 촉촉하게 유지하며, 간헐적으로 깜박이는 것은 결막을 윤활하고 이물질을 씻어냅니다.눈물에는 리소자임 등의 살균제가 함유되어 있어 미생물이 리소자임에서 살아남아 상피 표면에 정착하는 데 어려움이 있다.
위장관
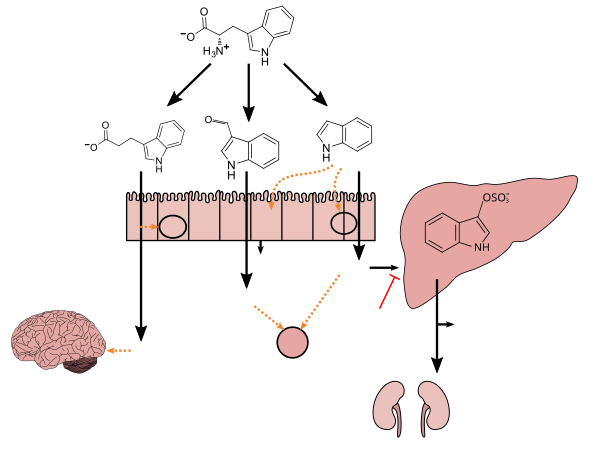
인간위장관 마이크로바이오타에 의한 트립토판 대사(Tryptophan metricota)) |
인간의 경우,[51] 위장 마이크로바이옴의 조성은 출생 중에 확립된다.제왕절개에 의한 출산이나 질 분만 또한 장의 미생물 구성에 영향을 미친다.질관을 통해 태어난 아기들은 [52]산모에게서 발견되는 것과 유사한 비병원적이고 유익한 내장 미생물군을 가지고 있다.그러나 제왕절개로 분만된 아기들의 내장 마이크로바이오타에는 대장균이나 포도상구균과 같은 병원성 세균이 더 많이 들어있어 비병원성 유익한 내장 마이크로바이오타 [53]발생에 더 오랜 시간이 걸린다.
일부 내장 미생물군과 인간 사이의 관계는 단지 보상적인 관계일 뿐만 아니라, 오히려 상호주의적인 [2]관계입니다.어떤 인간의 내장 미생물은 식이섬유를 아세트산이나 낙산과 같은 짧은 사슬 지방산으로 발효시킴으로써 [4][54]숙주에게 이로운 작용을 한다.장내 세균은 또한 비타민 B와 비타민 K를 합성하고 담즙산, 스테롤,[2][54] 그리고 이세균을 대사하는 역할도 한다.SCFA와 그들이 생산하는 다른 화합물의 체계적 중요성은 호르몬과 같고 장내 세균 자체는 내분비 [54]기관처럼 기능하는 것으로 보이며 장내 세균의 조절 장애는 염증 [4][55]및 자가 면역 조건의 숙주와 상관관계가 있다.
인간의 내장 미생물의 구성은 시간이 지남에 따라, 식단이 변화할 때, 그리고 전반적인 건강이 [4][55]변화함에 따라 변화한다.7월 2016년에서 15사람의 무선 제어되는 재판의 체계적인 리뷰는 생균제 박테리아의 비피 도박테륨과 젖산 속에서 특정 상업적으로 이용 가능한 변종(Blongum, Bbreve, Binfantis, L.helveticus, L.rhamnosus, L.plantarum, L.casei), 입으로 109–1010 식민지 formi에 대해 일일 복용량에서 찍은 사진을 발견했다.쇼핑 단위(CFU)는 1-2개월 동안 불안, 우울증, 자폐 스펙트럼 장애, 강박증 등 특정 중추신경계 장애에서 치료 효과(행동 결과 개선)를 가지고 기억력의 [56]특정 측면을 개선한다.하지만, 내장 마이크로바이오타의 구성 변화는 또한 건강에 해로운 영향과 관련이 있는 것으로 밝혀졌다.Musso et al.에 의해 발행된 기사에서, 비만인 무비대의 내장 미생물군이 건강한 [57]사람보다 더 많은 바실로타를 가지고 있고 더 적은 박테로이도타를 가지고 있다는 것이 발견되었다.미생물 비율의 변화는 음식에서 에너지를 더 효율적으로 추출하는 박테리아 증가에 기여할 수 있다고 믿어진다.연구원들은 비만한 쥐의 마이크로바이오타를 마른 쥐와 비교하기 위해 산탄총 시퀀싱을 사용했다.그들은 비만 생쥐의 게놈이 사람 [58]몸만으로 소화하기 어려운 다당류를 분해할 수 있는 효소를 코드하는 풍부한 유전자로 구성되어 있다는 것을 발견했다.
게다가 고든 등의 연구는 비만을 일으키는 것이 그 반대라기보다는 미생물이라는 것을 확인시켜 주었다.이것은 식이요법 유도 비만 쥐나 군살 조절 쥐의 내장 마이크로바이오타를 마이크로바이옴이 없는 군살 없는 쥐에게 이식함으로써 이루어졌다.그들은 DIO 생쥐 내장 마이크로바이오타를 이식받은 쥐들이 같은 [59]식단을 먹였을 때 살코기 생쥐 마이크로바이오타를 이식받은 쥐들보다 체지방이 훨씬 더 높다는 것을 발견했다.
2013년 Ridaura 등에 의해 완료된 별도의 연구는 무균 생쥐에 인간의 배설물을 처음으로 이식하는 것을 수행했다.채취된 인분은 체지방 비율이 확연히 다른 성인 여성 쌍둥이에게서 비롯됐다.연구자들은 기본적으로 비만 표현형과 군살 표현형을 쥐에게 전달할 수 있었고, 동시에 둘 다 저지방 쥐에게 먹이를 줄 수 있었다.비만 쌍둥이에게서 유래한 대변을 가진 쥐들은 전체 몸과 지방량을 증가시켰으며, 반면 마른 쌍둥이에게서 유래한 대변을 가진 쥐들은 비만과 같은 특징이나 [60]증상을 나타내지 않았다.
요도 및 방광
비뇨기 [61][62]계통은 요로 감염의 징후를 보일 때 소변에 있는 박테리아를 검출하기 위해 표준 임상 미생물 배양 방법을 오랫동안 사용해 온 것에 비추어 볼 때 예상치 못한 발견이다; 이러한 검사에서는 박테리아가 [63]존재하지 않는 것이 일반적이다.일반적인 배양법은 일반적으로 [63]존재하는 많은 종류의 박테리아와 다른 미생물을 발견하지 못하는 것으로 보인다.2017년 기준, 요로장애인과 건강한 [61][62][64]사람간 미생물의 차이 유무를 판단하기 위해 배열을 정하는 방법을 사용하였다.비뇨기계가 아닌 방광의 마이크로바이옴을 적절하게 평가하기 위해서는 소변 검체를 방광에서 직접 채취해야 하며,[65] 이는 종종 카테터로 이루어진다.
질
질 마이크로바이오타는 질에 군집하는 종과 속들을 말한다.이 유기체들은 감염으로부터 보호하고 질 [66]건강을 유지하는데 중요한 역할을 한다.폐경 전 여성에게서 발견되는 가장 풍부한 질 미생물은 과산화수소와 젖산을 [34][66][67]만들어 병원균을 억제하는 락토바실루스속이다.세균의 종류 구성과 비율은 생리 주기의 [68][69][needs update]단계에 따라 달라집니다.민족성은 또한 질 식물군에 영향을 미친다.과산화수소를 생성하는 락토바실리의 발생은 아프리카계 미국인 여성에게서 더 낮고 질 pH는 [70]더 높다.성관계와 항생제와 같은 다른 영향력 있는 요소들은 락토바실리의 [67]손실과 관련이 있다.게다가 콘돔과의 성관계는 락토바실리의 수치를 변화시키고 질내 대장균의 [67]수치를 증가시키는 것으로 나타났다.정상적이고 건강한 질 마이크로바이오타의 변화는 칸디다증이나 박테리아 [67]질염과 같은 [71]감염의 징후이다.칸디다 알비칸은 락토바실루스 종의 성장을 억제하는 반면, 과산화수소를 생성하는 락토바실루스 종은 칸디다 알비칸의 질과 내장의 [32][34][35]성장과 독성을 억제한다.
질에서 검출된 곰팡이속에는 [32]칸디다, 피치아, 유로튬, 얼터나리아, 로도토룰라, 클래도스포리움이 있다.
태반
최근까지 태반은 무균 기관으로 간주되었지만 상응하는 비병원성 박테리아 종과 속은 태반 [72][73][74]조직에 존재하는 것으로 확인되었다.하지만, 태반에서 마이크로바이옴의 존재는 여러 연구에서 비판받았듯이 논란이 되고 있다.소위 "평면 마이크로바이옴"이라고 불리는 것은 저바이오매스 샘플이 쉽게 [75][76][77]오염되기 때문에 리젠트의 오염에서 파생된 것일 수 있습니다.
자궁
최근까지 여성의 상부 생식관은 무균 환경으로 간주되었다.다양한 미생물이 건강하고 증세가 없는 생식기 여성의 자궁에 살고 있다.자궁의 마이크로바이옴은 질이나 [78]위장관의 마이크로바이옴과 크게 다르다.
구강
인간의 입안에 존재하는 환경은 그곳에서 발견되는 특징적인 미생물의 성장을 가능하게 한다.그것은 적당한 [2]온도뿐만 아니라 수분과 영양분을 공급합니다.입 안에 있는 미생물은 입에서 위로의 기계적 홍수에 저항하기 위해 치아와 잇몸에 달라붙어 산성에 민감한 미생물이 염산에 [2][34]의해 파괴된다.
구강 내 혐기성 세균에는 다음이 포함된다.악티노마이세스, 아라크니아, 박테로이데스, 비피도박테륨, 에우박테륨, 후소박테륨, 유산균, 렙토트리치아, 펩토크쿠스, 프로피오니박테륨, 셀레노모나스, 트레포네마 및 베일로넬라.[79][needs update]입에서 자주 발견되는 곰팡이속에는 칸디다, 클래도스포리움, 아스페르길루스, 후사리움, 글로무스, 얼터나리아, 페니실륨,[32] 크립토코쿠스 등이 있다.
박테리아는 바이오필름의 경질 및 연질 구강조직에 축적되어 환경요인과 [80]항균제로부터 보호받으면서 구강환경에서 부착하고 분투할 수 있습니다.침은 생물막의 형성을 위한 세균의 재결합을 가능하게 하고 생물막의 [81]축적을 분리함으로써 성장을 제어할 수 있는 중요한 생물막의 항상성 역할을 한다.그것은 또한 영양소와 온도 조절 수단을 제공한다.바이오필름의 위치는 노출된 영양소의 [82]종류를 결정합니다.
구강세균은 환경을 감지하고 숙주를 회피하거나 수정하는 메커니즘을 진화시켰다.그러나 매우 효율적인 선천적 숙주 방어 시스템은 지속적으로 세균 집단을 감시하고 국소 조직의 세균 침입을 방지한다.치석 박테리아와 선천적인 숙주 방어 [83]시스템 사이에 동적 평형이 존재한다.
숙주 구강과 구강 미생물 사이의 이러한 동력은 [84]체내로의 진입을 제공하기 때문에 건강과 질병에 중요한 역할을 합니다.건강한 균형은 구강 미생물이 병원균의 성장과 부착을 제한하고 숙주가 병원균이 [84][80]번성할 수 있는 환경을 제공하는 공생 관계를 나타냅니다.면역 상태의 변화, 상주 미생물의 이동 및 영양소의 가용성과 같은 생태학적 변화는 숙주가 구강 및 전신 질환에 [80]걸리기 쉬운 상호 관계에서 기생 관계로 변화한다.당뇨병과 심혈관 질환과 같은 전신 질환은 구강 [84]건강 악화와 관련이 있다.특히 관심을 끄는 것은 구강 미생물이 구강 내 두 가지 주요 질병인 충치와 치주 [83]질환에서 차지하는 역할이다.치주염의 병원균 집락은 과도한 면역 반응을 유발하여 치주 포켓, 즉 치아와 [80]잇몸 사이의 깊어진 공간을 야기한다.이것은 혐기성 [80]병원균을 위한 영양분을 가진 혈액이 풍부한 보호 저장고 역할을 한다.몸의 다양한 부위의 전신 질환은 구강 미생물이 치주 주머니와 [84]구막을 우회하여 혈액으로 유입되면서 발생할 수 있다.
지속적인 적절한 구강 [84]위생은 구강 및 전신 질환을 예방하는 주요 방법입니다.바이오필름의 밀도와 [82]질병을 일으키는 잠재적 병원성 세균의 과잉 증식을 감소시킨다.그러나 구강 마이크로바이옴, 유전학, 면역 반응의 변화가 만성 [82]감염을 일으키는 요인이 되기 때문에 적절한 구강 위생으로는 충분하지 않을 수 있다.항생제의 사용은 이미 퍼진 감염을 치료할 수 있지만 생체막 [82]내의 박테리아에 대해서는 효과가 없다.
비강
건강한 코 마이크로바이옴은 코리네박테륨과 포도상구균에 의해 지배된다.점막 마이크로바이옴은 바이러스 [85]감염을 조절하는 데 중요한 역할을 한다.
폐
구강과 같이, 상부와 하부 호흡 시스템은 미생물을 제거하는 기계적 억제를 가지고 있습니다.고블렛 세포는 지속적으로 움직이는 섬모상피세포를 [2]통해 미생물을 가둬 호흡기관 밖으로 내보내는 점액을 생성한다.또, 리소자임 [2]효소를 함유하는 코 점액에 의해 살균 효과가 발생한다.상·하부 호흡기는 자체 미생물군을 [86]가지고 있는 것으로 보인다.폐세균 마이크로바이오타는 프리보텔라, 스핑고모나스, 슈도모나스, 아시네토박터, 후소박터, 메가세라, 베이요넬라, 포도상구균, 스트렙토코커스 등 9개 주요 세균속에 속한다.호흡기에서 "정상적인 생물"로 여겨지는 박테리아 중 일부는 특히 면역력이 약한 개인에게 심각한 질병을 일으킬 수 있습니다; 이것들은 Streptococcus pyogenes, Hemophilus influence,[citation needed] Streptoccus pneumoniae, Neisseria meningitidis, 그리고 Staphyloccus aure를 포함합니다.폐마이코바이옴을 구성하는 곰팡이속에는 [32]칸디다, 말라세지아, 네오사토리아, 사카로미세스, 아스페르길루스 등이 있다.
낭포성 섬유증이 [32][87]있는 사람들에게서 호흡기의 세균과 진균 속들의 비정상적인 분포가 관찰된다.이들의 세균은 항생제에 내성이 있고 성장이 느린 박테리아를 포함하고 있으며,[87] 이러한 병원균의 빈도는 나이에 따라 변한다.
담도
전통적으로 담도는 일반적으로 멸균된 것으로 간주되어 왔으며 담즙에 미생물이 존재하는 것은 병리학적 과정의 지표이다.이 가정은 정상적인 담관으로부터 박테리아 균주를 할당하지 못함으로써 확인되었다.2013년부터는 정상적인 담도미생물군이 외인성 미생물에 [88]의한 담도 콜로니케이션으로부터 보호하는 별도의 기능층이라는 논문이 발표되기 시작했다.
질병과 죽음
인간의 몸은 필수 [89]영양소의 원천으로서 무수한 박테리아 유전자에 의존한다.메타제노믹스와 역학 연구 모두 제2형 당뇨병과 비만에서 염증성 장질환, 파킨슨병, 그리고 [90]우울증과 같은 정신 건강 상태에 이르기까지 광범위한 질병을 예방하는 데 인간 마이크로바이옴의 중요한 역할을 보여준다.내장 미생물군과 다른 박테리아 사이의 공생 관계는 개인의 면역 [91]반응에 영향을 미칠 수 있다.초기 단계이지만, 마이크로바이옴 기반 치료도 유망하며, 특히 내약품성 C. 디피실 감염[92] 치료와 당뇨병 [93]치료에서 가장 두드러진다.
클로스트리디오이데스 디피실 감염
C. 디피실이라는 박테리아가 압도적으로 존재하면, 보통 항생제 투여에 의해 유발된 것으로 여겨지는 미생물과의 이상 증상과 관련된 위장관의 감염으로 이어진다.항생제의 사용은 보통 병원성 박테리아가 우위를 [94]확립하는 것을 막는 위장관 내의 유익한 장내 균을 근절한다.C. 디피실 감염에 대한 전통적인 치료법에는 항생제의 추가 투여가 포함되지만, 평균 유효율은 20~30%[95]이다.건강한 장내 박테리아의 중요성을 인식하면서, 연구원들은 분변 마이크로바이오타 이식이라고 알려진 시술로 눈을 돌렸는데, 여기서 C. 디피실 감염과 같은 위장 질환을 앓고 있는 환자들은 정상적인 기능을 하는 장내 마이크로바이오타를 [96]회복하기 위해 건강한 사람으로부터 분변 함량을 받는다.분변 마이크로바이오타 이식은 항생제가 효과가 없거나 항생제에 이어 [97][98]질병이 재발하는 CDI에 걸린 사람들에게 약 85-90% 효과적이다.CDI를 앓고 있는 대부분의 사람들은 FMT 치료 [99][94][100]한 번으로 회복된다.
암
암은 일반적으로 숙주 유전과 환경적 요인에 의한 질병이지만, 미생물은 인간 [101]암의 약 20%에 관련된다.특히 대장암의 잠재적 인자의 경우, 박테리아 밀도가 소장에 비해 100만 배 높고, 소장에 비해 약 12배 더 많은 암이 발생하며, 대장암과 [102]직장암에서 미생물의 병원성 역할을 확립할 수 있다.미생물 밀도는 대장암 [102]평가에서 예후 도구로 사용될 수 있다.
마이크로바이오타는 (i) 종양세포 증식과 죽음의 균형을 바꾸는 것, (ii) 면역체계 기능을 조절하는 것, (ii) 숙주가 생산하는 요인, 식품 및 의약품의 [101]대사에 영향을 미치는 세 가지 방법으로 발암에 영향을 미칠 수 있다.피부, 구인두, 호흡기, 소화기 및 비뇨기 계통과 같은 경계 표면에서 발생하는 종양은 미생물군을 가지고 있다.종양 부위에 상당한 미생물이 존재한다고 해서 연관성이나 인과관계가 확립되는 것은 아니다.대신, 미생물들은 종양 산소 긴장이나 영양 프로파일이 도움이 된다는 것을 발견할 수 있다.특정 미생물의 개체수 감소 또는 유도된 산화 스트레스 [101][102]또한 위험을 증가시킬 수 있다.지구상에 존재하는 10여30 종의 미생물 중 10종은 국제암연구기구에 의해 인간의 발암물질로 [101]지정됐다.미생물은 숙주의 세포 증식을 직접적으로 촉진하거나 발암에 [101]기여하는 방법으로 급성 또는 만성 염증을 촉진하는 것을 포함하여 숙주의 면역체계를 상향 또는 하향 조정할 수 있다.
면역기능의 관계와 염증의 발생에 관해서는 점막 표면장벽은 환경상의 위험이 있으므로 항상성을 유지하기 위해 신속하게 복구해야 한다.숙주나 미생물의 탄력성이 저하되어 악성 종양에 대한 저항력이 저하되어 염증과 암을 유발할 수 있습니다.일단 장벽이 뚫리면, 미생물은 다양한 [101]경로를 통해 염증이나 면역 억제 프로그램을 유도할 수 있다.예를 들어 암 관련 미생물은 종양 마이크로바이러스 내에서 NF-γβ 시그널링을 활성화하는 것으로 보인다.뉴클레오티드 결합 올리고머화 도메인 유사 수용체(NLR) 패밀리 구성원 NOD-2, NLRP3, NLRP6 및 NLRP12와 같은 다른 패턴 인식 수용체는 대장암을 [101]중재하는 역할을 할 수 있다.마찬가지로 헬리코박터균은 위에서 [101][102]만성 염증 반응을 일으키기 때문에 위암의 위험을 높이는 것으로 보인다.
염증성 장질환
염증성 장질환은 궤양성 대장염과 크론병 두 가지 다른 질병으로 구성되어 있으며, 이 두 가지 질환 모두 장내 미생물에 장애를 일으킨다.이 소화불량증은 [103][104]장내 미생물 다양성 감소의 형태로 나타나며,[103] 개인의 선천적인 면역 반응을 변화시키는 숙주 유전자의 결함과 관련이 있다.
인간면역결핍바이러스
HIV 질환의 진행은 장내 미생물군의 구성과 기능에 영향을 미치며 HIV 음성, HIV 양성 및 포스트 ART HIV 양성 [citation needed]모집단 간에 현저한 차이가 있습니다.HIV는 단단한 접합부에 영향을 줌으로써 장상피 장벽 기능의 무결성을 감소시킨다.이 붕괴는 장 상피 전체에 걸쳐 전이를 허용하는데,[105] 이것은 HIV에 걸린 사람들에게서 나타나는 염증의 증가에 기여하는 것으로 생각됩니다.
질내 미생물은 HIV의 감염에 관여하며, 여성이 [106]질내 세균의 비정상적인 균형을 특징으로 하는 세균성 질병에 걸렸을 때 감염과 전염의 위험이 높아진다.질 내 소염성 사이토카인과 CCR5 + CD4+ 세포가 증가하면 감염성이 향상된다.그러나 질 유산균의 수치가 높아지면서 감염성의 감소가 나타나며, 이는 항염증 상태를 [105]촉진한다.
죽음.
죽음과 함께, 생체의 마이크로바이옴은 붕괴되고 네크로바이옴이라는 다른 미생물의 구성은 복잡한 물리적 분해 과정의 중요한 활성 성분으로 자리매김합니다.시간의 경과에 따른 예측 가능한 변화는 [107][108]사망 시간을 결정하는 데 도움이 될 것으로 생각됩니다.
환경 건강
2009년 연구는 인간의 개입으로 인한 생물(미생물군 포함)의 감소가 인간의 건강, 병원 안전 절차, 식품 설계 및 [109]질병 치료를 저해할 수 있는지에 대해 의문을 제기했다.
이행
예비 조사에 따르면, 한 국가에서 다른 나라로 이민을 갈 때, 예를 들어 태국 이민자들이[110] 미국에 정착했을 때 또는 라틴 아메리카인들이 미국으로 [111]이민 왔을 때, 마이크로바이오타의 즉각적인 변화가 일어날 수 있다.미생물 다양성의 손실은 비만인 개인과 이민자들의 자녀들에게서 [110][111]더 컸다.
「 」를 참조해 주세요.
참고 문헌
- 에드 용포함 수: 우리 안에 있는 미생물들과 웅장한 인생관.368쪽, 2016년 8월 9일 Ecco 발행, ISBN0062368591.
레퍼런스
- ^ a b Marchesi JR, Ravel J (2015). "The vocabulary of microbiome research: a proposal". Microbiome. 3: 31. doi:10.1186/s40168-015-0094-5. PMC 4520061. PMID 26229597.
Microbiome
This term refers to the entire habitat, including the microorganisms (bacteria, archaea, lower and higher eurkaryotes, and viruses), their genomes (i.e., genes), and the surrounding environmental conditions. This definition is based on that of "biome," the biotic and abiotic factors of given environments. Others in the field limit the definition of microbiome to the collection of genes and genomes of members of a microbiota. It is argued that this is the definition of metagenome, which combined with the environment constitutes the microbiome. - ^ a b c d e f g h i j k Sherwood L, Willey J, Woolverton C (2013). Prescott's Microbiology (9th ed.). New York: McGraw Hill. pp. 713–721. ISBN 9780073402406. OCLC 886600661.
- ^ a b Sender R, Fuchs S, Milo R (January 2016). "Are We Really Vastly Outnumbered? Revisiting the Ratio of Bacterial to Host Cells in Humans". Cell. 164 (3): 337–40. doi:10.1016/j.cell.2016.01.013. PMID 26824647.
- ^ a b c d e f Quigley EM (September 2013). "Gut bacteria in health and disease". Gastroenterology & Hepatology. 9 (9): 560–9. PMC 3983973. PMID 24729765.
- ^ Falony G, Vieira-Silva S, Raes J (2015). "Microbiology Meets Big Data: The Case of Gut Microbiota-Derived Trimethylamine". Annual Review of Microbiology. 69: 305–21. doi:10.1146/annurev-micro-091014-104422. PMID 26274026.
we review literature on trimethylamine (TMA), a microbiota-generated metabolite linked to atherosclerosis development.
- ^ Gaci N, Borrel G, Tottey W, O'Toole PW, Brugère JF (November 2014). "Archaea and the human gut: new beginning of an old story". World Journal of Gastroenterology. 20 (43): 16062–78. doi:10.3748/wjg.v20.i43.16062. PMC 4239492. PMID 25473158.
Trimethylamine is exclusively a microbiota-derived product of nutrients (lecithin, choline, TMAO, L-carnitine) from normal diet, from which seems originate two diseases, trimethylaminuria (or Fish-Odor Syndrome) and cardiovascular disease through the proatherogenic property of its oxidized liver-derived form.
- ^ a b "NIH Human Microbiome Project defines normal bacterial makeup of the body". NIH News. 13 June 2012.
- ^ a b 미국 미생물학회 FAQ: 2016년 12월 31일 웨이백 머신에 보관된 인간 미생물학 FAQ 2014년 1월
- ^ 2014년 2월, Microbe Magazine의 유다 L. 로스너.인간의 신체 세포보다 10배 많은 미생물 세포?
- ^ 네이처 뉴스의 앨리슨 애벗입니다.2016년 1월 8일 과학자들은 인간의 세포보다 우리 몸에 더 많은 박테리아가 있다는 신화를 깨뜨렸다.
- ^ Gilbert JA, Blaser MJ, Caporaso JG, Jansson JK, Lynch SV, Knight R (April 2018). "Current understanding of the human microbiome". Nature Medicine. 24 (4): 392–400. doi:10.1038/nm.4517. PMC 7043356. PMID 29634682.
- ^ a b c Peterson J, Garges S, Giovanni M, McInnes P, Wang L, Schloss JA, et al. (December 2009). "The NIH Human Microbiome Project". Genome Research. 19 (12): 2317–23. doi:10.1101/gr.096651.109. PMC 2792171. PMID 19819907.
- ^ Kuczynski J, Lauber CL, Walters WA, Parfrey LW, Clemente JC, Gevers D, Knight R (December 2011). "Experimental and analytical tools for studying the human microbiome". Nature Reviews. Genetics. 13 (1): 47–58. doi:10.1038/nrg3129. PMC 5119550. PMID 22179717.
- ^ Vestheim H, Jarman SN (July 2008). "Blocking primers to enhance PCR amplification of rare sequences in mixed samples - a case study on prey DNA in Antarctic krill stomachs". Frontiers in Zoology. 5: 12. doi:10.1186/1742-9994-5-12. PMC 2517594. PMID 18638418.
- ^ Tap J, Mondot S, Levenez F, Pelletier E, Caron C, Furet JP, et al. (October 2009). "Towards the human intestinal microbiota phylogenetic core". Environmental Microbiology. 11 (10): 2574–84. doi:10.1111/j.1462-2920.2009.01982.x. PMID 19601958.
- ^ Hamady M, Knight R (July 2009). "Microbial community profiling for human microbiome projects: Tools, techniques, and challenges". Genome Research. 19 (7): 1141–52. doi:10.1101/gr.085464.108. PMC 3776646. PMID 19383763.
- ^ Methé BA, Nelson KE, Pop M, Creasy HH, Giglio MG, Huttenhower C, et al. (Human Microbiome Project Consortium) (June 2012). "A framework for human microbiome research". Nature. 486 (7402): 215–21. Bibcode:2012Natur.486..215T. doi:10.1038/nature11209. PMC 3377744. PMID 22699610.
- ^ The Human Microbiome Project Consortium (June 2012). "Structure, function and diversity of the healthy human microbiome". Nature. 486 (7402): 207–14. Bibcode:2012Natur.486..207T. doi:10.1038/nature11234. PMC 3564958. PMID 22699609.
- ^ Quince C, Walker AW, Simpson JT, Loman NJ, Segata N (September 2017). "Shotgun metagenomics, from sampling to analysis" (PDF). Nature Biotechnology. 35 (9): 833–844. doi:10.1038/nbt.3935. hdl:2164/10167. PMID 28898207. S2CID 19041044.
- ^ Claesson MJ, Clooney AG, O'Toole PW (October 2017). "A clinician's guide to microbiome analysis". Nature Reviews. Gastroenterology & Hepatology. 14 (10): 585–595. doi:10.1038/nrgastro.2017.97. PMID 28790452. S2CID 24644894.
- ^ a b c Knight R, Vrbanac A, Taylor BC, Aksenov A, Callewaert C, Debelius J, et al. (July 2018). "Best practices for analysing microbiomes". Nature Reviews. Microbiology. 16 (7): 410–422. doi:10.1038/s41579-018-0029-9. PMID 29795328. S2CID 43936002.
- ^ a b Washburne AD, Morton JT, Sanders J, McDonald D, Zhu Q, Oliverio AM, Knight R (June 2018). "Methods for phylogenetic analysis of microbiome data". Nature Microbiology. 3 (6): 652–661. doi:10.1038/s41564-018-0156-0. PMID 29795540. S2CID 43962376.
- ^ PLoS Human Microbyome Project Collection 원고 요약 Wayback Machine에서 2014년 3월 4일 아카이브 2012년 6월 13일
- ^ "Consortium of Scientists Map the Human Body's Bacterial Ecosystem". ucsf.edu.
- ^ Sommer F, Bäckhed F (April 2013). "The gut microbiota--masters of host development and physiology". Nature Reviews. Microbiology. 11 (4): 227–38. doi:10.1038/nrmicro2974. PMID 23435359. S2CID 22798964.
- ^ Eckburg PB, Bik EM, Bernstein CN, Purdom E, Dethlefsen L, Sargent M, et al. (June 2005). "Diversity of the human intestinal microbial flora". Science. 308 (5728): 1635–8. Bibcode:2005Sci...308.1635E. doi:10.1126/science.1110591. PMC 1395357. PMID 15831718.
- ^ Duncan SH, Louis P, Flint HJ (April 2007). "Cultivable bacterial diversity from the human colon". Letters in Applied Microbiology. 44 (4): 343–50. doi:10.1111/j.1472-765X.2007.02129.x. PMID 17397470. S2CID 43706882.
- ^ Florin TH, Zhu G, Kirk KM, Martin NG (October 2000). "Shared and unique environmental factors determine the ecology of methanogens in humans and rats". The American Journal of Gastroenterology. 95 (10): 2872–9. CiteSeerX 10.1.1.606.4187. doi:10.1111/j.1572-0241.2000.02319.x. PMID 11051362. S2CID 1087298.
- ^ Eckburg PB, Lepp PW, Relman DA (February 2003). "Archaea and their potential role in human disease". Infection and Immunity. 71 (2): 591–6. doi:10.1128/IAI.71.2.591-596.2003. PMC 145348. PMID 12540534.
- ^ Cavicchioli R, Curmi PM, Saunders N, Thomas T (November 2003). "Pathogenic archaea: do they exist?". BioEssays. 25 (11): 1119–28. doi:10.1002/bies.10354. PMID 14579252.
- ^ Lepp PW, Brinig MM, Ouverney CC, Palm K, Armitage GC, Relman DA (April 2004). "Methanogenic Archaea and human periodontal disease". Proceedings of the National Academy of Sciences of the United States of America. 101 (16): 6176–81. Bibcode:2004PNAS..101.6176L. doi:10.1073/pnas.0308766101. PMC 395942. PMID 15067114.
- ^ a b c d e f g h i j k Cui L, Morris A, Ghedin E (July 2013). "The human mycobiome in health and disease". Genome Medicine. 5 (7): 63. doi:10.1186/gm467. PMC 3978422. PMID 23899327.
Figure 2: Distribution of fungal genera in different body sites
{{cite journal}}:외부 링크quote= - ^ a b Martins N, Ferreira IC, Barros L, Silva S, Henriques M (June 2014). "Candidiasis: predisposing factors, prevention, diagnosis and alternative treatment" (PDF). Mycopathologia. 177 (5–6): 223–40. doi:10.1007/s11046-014-9749-1. hdl:10198/10147. PMID 24789109. S2CID 795450.
Candida species and other microorganisms are involved in this complicated fungal infection, but Candida albicans continues to be the most prevalent. In the past two decades, it has been observed an abnormal overgrowth in the gastrointestinal, urinary and respiratory tracts, not only in immunocompromised patients, but also related to nosocomial infections and even in healthy individuals. There is a widely variety of causal factors that contribute to yeast infection which means that candidiasis is a good example of a multifactorial syndrome.
- ^ a b c d e Wang ZK, Yang YS, Stefka AT, Sun G, Peng LH (April 2014). "Review article: fungal microbiota and digestive diseases". Alimentary Pharmacology & Therapeutics. 39 (8): 751–66. doi:10.1111/apt.12665. PMID 24612332. S2CID 22101484.
In addition, GI fungal infection is reported even among those patients with normal immune status. Digestive system-related fungal infections may be induced by both commensal opportunistic fungi and exogenous pathogenic fungi. ... Candida sp. is also the most frequently identified species among patients with gastric IFI. ... It was once believed that gastric acid could kill microbes entering the stomach and that the unique ecological environment of the stomach was not suitable for microbial colonisation or infection. However, several studies using culture-independent methods confirmed that large numbers of acid-resistant bacteria belonging to eight phyla and up to 120 species exist in the stomach, such as Streptococcus sp., Neisseria sp. and Lactobacillus sp. etc.26, 27 Furthermore, Candida albicans can grow well in highly acidic environments,28 and some genotypes may increase the severity of gastric mucosal lesions.29
- ^ a b c Erdogan A, Rao SS (April 2015). "Small intestinal fungal overgrowth". Current Gastroenterology Reports. 17 (4): 16. doi:10.1007/s11894-015-0436-2. PMID 25786900. S2CID 3098136.
Small intestinal fungal overgrowth (SIFO) is characterized by the presence of excessive number of fungal organisms in the small intestine associated with gastrointestinal (GI) symptoms. Candidiasis is known to cause GI symptoms particularly in immunocompromised patients or those receiving steroids or antibiotics. However, only recently, there is emerging literature that an overgrowth of fungus in the small intestine of non-immunocompromised subjects may cause unexplained GI symptoms. Two recent studies showed that 26 % (24/94) and 25.3 % (38/150) of a series of patients with unexplained GI symptoms had SIFO. The most common symptoms observed in these patients were belching, bloating, indigestion, nausea, diarrhea, and gas. ... Fungal-bacterial interaction may act in different ways and may either be synergistic or antagonistic or symbiotic [29]. Some bacteria such as Lactobacillus species can interact and inhibit both the virulence and growth of Candida species in the gut by producing hydrogen peroxide [30]. Any damage to the mucosal barrier or disruption of GI microbiota with chemotherapy or antibiotic use, inflammatory processes, activation of immune molecules and disruption of epithelial repair may all cause fungal overgrowth [27].
- ^ Marcon MJ, Powell DA (April 1992). "Human infections due to Malassezia spp". Clinical Microbiology Reviews. 5 (2): 101–19. doi:10.1128/CMR.5.2.101. PMC 358230. PMID 1576583.
- ^ Roth RR, James WD (1988). "Microbial ecology of the skin". Annual Review of Microbiology. 42 (1): 441–64. doi:10.1146/annurev.mi.42.100188.002301. PMID 3144238.
- ^ Hannigan GD, Meisel JS, Tyldsley AS, Zheng Q, Hodkinson BP, SanMiguel AJ, et al. (October 2015). "The human skin double-stranded DNA virome: topographical and temporal diversity, genetic enrichment, and dynamic associations with the host microbiome". mBio. 6 (5): e01578-15. doi:10.1128/mBio.01578-15. PMC 4620475. PMID 26489866.
- ^ Minot S, Sinha R, Chen J, Li H, Keilbaugh SA, Wu GD, et al. (October 2011). "The human gut virome: inter-individual variation and dynamic response to diet". Genome Research. 21 (10): 1616–25. doi:10.1101/gr.122705.111. PMC 3202279. PMID 21880779.
- ^ Young JC, Chehoud C, Bittinger K, Bailey A, Diamond JM, Cantu E, et al. (January 2015). "Viral metagenomics reveal blooms of anelloviruses in the respiratory tract of lung transplant recipients". American Journal of Transplantation. 15 (1): 200–9. doi:10.1111/ajt.13031. PMC 4276431. PMID 25403800.
- ^ Abeles SR, Robles-Sikisaka R, Ly M, Lum AG, Salzman J, Boehm TK, Pride DT (September 2014). "Human oral viruses are personal, persistent and gender-consistent". The ISME Journal. 8 (9): 1753–67. doi:10.1038/ismej.2014.31. PMC 4139723. PMID 24646696.
- ^ Ly M, Abeles SR, Boehm TK, Robles-Sikisaka R, Naidu M, Santiago-Rodriguez T, Pride DT (May 2014). "Altered oral viral ecology in association with periodontal disease". mBio. 5 (3): e01133-14. doi:10.1128/mBio.01133-14. PMC 4030452. PMID 24846382.
- ^ Monaco CL, Gootenberg DB, Zhao G, Handley SA, Ghebremichael MS, Lim ES, et al. (March 2016). "Altered Virome and Bacterial Microbiome in Human Immunodeficiency Virus-Associated Acquired Immunodeficiency Syndrome". Cell Host & Microbe. 19 (3): 311–22. doi:10.1016/j.chom.2016.02.011. PMC 4821831. PMID 26962942.
- ^ Norman JM, Handley SA, Baldridge MT, Droit L, Liu CY, Keller BC, et al. (January 2015). "Disease-specific alterations in the enteric virome in inflammatory bowel disease". Cell. 160 (3): 447–60. doi:10.1016/j.cell.2015.01.002. PMC 4312520. PMID 25619688.
- ^ Grice EA, Kong HH, Conlan S, Deming CB, Davis J, Young AC, et al. (NISC Comparative Sequencing Program) (May 2009). "Topographical and temporal diversity of the human skin microbiome". Science. 324 (5931): 1190–2. Bibcode:2009Sci...324.1190G. doi:10.1126/science.1171700. PMC 2805064. PMID 19478181.
- ^ a b "The Normal Bacterial Flora of Humans". textbookofbacteriology.net.
- ^ a b c d e f g h i Zhang LS, Davies SS (April 2016). "Microbial metabolism of dietary components to bioactive metabolites: opportunities for new therapeutic interventions". Genome Med. 8 (1): 46. doi:10.1186/s13073-016-0296-x. PMC 4840492. PMID 27102537.
Lactobacillus spp. convert tryptophan to indole-3-aldehyde (I3A) through unidentified enzymes [125]. Clostridium sporogenes convert tryptophan to IPA [6], likely via a tryptophan deaminase. ... IPA also potently scavenges hydroxyl radicals
표 2: 미생물 대사물: 그 합성, 작용 메커니즘 및 건강과 질병에 미치는 영향
그림 1: 인돌과 그 대사물이 숙주의 생리학 및 질병에 작용하는 분자 메커니즘 - ^ Wikoff WR, Anfora AT, Liu J, Schultz PG, Lesley SA, Peters EC, Siuzdak G (March 2009). "Metabolomics analysis reveals large effects of gut microflora on mammalian blood metabolites". Proc. Natl. Acad. Sci. U.S.A. 106 (10): 3698–3703. Bibcode:2009PNAS..106.3698W. doi:10.1073/pnas.0812874106. PMC 2656143. PMID 19234110.
Production of IPA was shown to be completely dependent on the presence of gut microflora and could be established by colonization with the bacterium Clostridium sporogenes.
IPA 대사도 - ^ "3-Indolepropionic acid". Human Metabolome Database. University of Alberta. Retrieved 12 June 2018.
- ^ Chyan YJ, Poeggeler B, Omar RA, Chain DG, Frangione B, Ghiso J, Pappolla MA (July 1999). "Potent neuroprotective properties against the Alzheimer beta-amyloid by an endogenous melatonin-related indole structure, indole-3-propionic acid". J. Biol. Chem. 274 (31): 21937–21942. doi:10.1074/jbc.274.31.21937. PMID 10419516. S2CID 6630247.
[Indole-3-propionic acid (IPA)] has previously been identified in the plasma and cerebrospinal fluid of humans, but its functions are not known. ... In kinetic competition experiments using free radical-trapping agents, the capacity of IPA to scavenge hydroxyl radicals exceeded that of melatonin, an indoleamine considered to be the most potent naturally occurring scavenger of free radicals. In contrast with other antioxidants, IPA was not converted to reactive intermediates with pro-oxidant activity.
- ^ Yang I, Corwin EJ, Brennan PA, Jordan S, Murphy JR, Dunlop A (2016). "The Infant Microbiome: Implications for Infant Health and Neurocognitive Development". Nursing Research. 65 (1): 76–88. doi:10.1097/NNR.0000000000000133. PMC 4681407. PMID 26657483.
- ^ Mueller NT, Bakacs E, Combellick J, Grigoryan Z, Dominguez-Bello MG (February 2015). "The infant microbiome development: mom matters". Trends in Molecular Medicine. 21 (2): 109–17. doi:10.1016/j.molmed.2014.12.002. PMC 4464665. PMID 25578246.
- ^ Wall R, Ross RP, Ryan CA, Hussey S, Murphy B, Fitzgerald GF, Stanton C (4 March 2009). "Role of gut microbiota in early infant development". Clinical Medicine. Pediatrics. 3: 45–54. doi:10.4137/cmped.s2008. PMC 3676293. PMID 23818794.
- ^ a b c Clarke G, Stilling RM, Kennedy PJ, Stanton C, Cryan JF, Dinan TG (August 2014). "Minireview: Gut microbiota: the neglected endocrine organ". Molecular Endocrinology. 28 (8): 1221–38. doi:10.1210/me.2014-1108. PMC 5414803. PMID 24892638.
- ^ a b Shen S, Wong CH (April 2016). "Bugging inflammation: role of the gut microbiota". Clinical & Translational Immunology. 5 (4): e72. doi:10.1038/cti.2016.12. PMC 4855262. PMID 27195115.
- ^ Wang H, Lee IS, Braun C, Enck P (October 2016). "Effect of Probiotics on Central Nervous System Functions in Animals and Humans: A Systematic Review". Journal of Neurogastroenterology and Motility. 22 (4): 589–605. doi:10.5056/jnm16018. PMC 5056568. PMID 27413138.
These probiotics showed efficacy in improving psychiatric disorder-related behaviors including anxiety, depression, autism spectrum disorder (ASD), obsessive-compulsive disorder, and memory abilities, including spatial and non-spatial memory. Because many of the basic science studies showed some efficacy of probiotics on central nervous system function, this background may guide and promote further preclinical and clinical studies. ... According to the qualitative analyses of current studies, we can provisionally draw the conclusion that B. longum, B. breve, B. infantis, L. helveticus, L. rhamnosus, L. plantarum, and L. casei were most effective in improving CNS function, including psychiatric disease-associated functions (anxiety, depression, mood, stress response) and memory abilities
- ^ Musso G, Gambino R, Cassader M (October 2010). "Obesity, diabetes, and gut microbiota: the hygiene hypothesis expanded?". Diabetes Care. 33 (10): 2277–84. doi:10.2337/dc10-0556. PMC 2945175. PMID 20876708.
- ^ Harsch IA, Konturek PC (April 2018). "The Role of Gut Microbiota in Obesity and Type 2 and Type 1 Diabetes Mellitus: New Insights into "Old" Diseases". Medical Sciences. 6 (2): 32. doi:10.3390/medsci6020032. PMC 6024804. PMID 29673211.
- ^ Turnbaugh PJ, Bäckhed F, Fulton L, Gordon JI (April 2008). "Diet-induced obesity is linked to marked but reversible alterations in the mouse distal gut microbiome". Cell Host & Microbe. 3 (4): 213–23. doi:10.1016/j.chom.2008.02.015. PMC 3687783. PMID 18407065.
- ^ Ridaura VK, Faith JJ, Rey FE, Cheng J, Duncan AE, Kau AL, et al. (September 2013). "Gut microbiota from twins discordant for obesity modulate metabolism in mice". Science. 341 (6150): 1241214. doi:10.1126/science.1241214. PMC 3829625. PMID 24009397.
- ^ a b Drake MJ, Morris N, Apostolidis A, Rahnama'i MS, Marchesi JR (April 2017). "The urinary microbiome and its contribution to lower urinary tract symptoms; ICI-RS 2015". Neurourology and Urodynamics. 36 (4): 850–853. doi:10.1002/nau.23006. hdl:1983/3b024f95-9f86-406a-9be3-ce35984b8de1. PMID 28444712. S2CID 27636043.
- ^ a b Aragón IM, Herrera-Imbroda B, Queipo-Ortuño MI, Castillo E, Del Moral JS, Gómez-Millán J, et al. (January 2018). "The Urinary Tract Microbiome in Health and Disease". European Urology Focus. 4 (1): 128–138. doi:10.1016/j.euf.2016.11.001. PMID 28753805.
- ^ a b Schmiemann G, Kniehl E, Gebhardt K, Matejczyk MM, Hummers-Pradier E (May 2010). "The diagnosis of urinary tract infection: a systematic review". Deutsches Ärzteblatt International. 107 (21): 361–7. doi:10.3238/arztebl.2010.0361. PMC 2883276. PMID 20539810.
- ^ Al, Kait F.; Denstedt, John D.; Daisley, Brendan A.; Bjazevic, Jennifer; Welk, Blayne K.; Pautler, Stephen E.; Gloor, Gregory B.; Reid, Gregor; Razvi, Hassan; Burton, Jeremy P. (September 2020). "Ureteral Stent Microbiota Is Associated with Patient Comorbidities but Not Antibiotic Exposure". Cell Reports Medicine. 1 (6): 100094. doi:10.1016/j.xcrm.2020.100094. PMC 7659606. PMID 33205072.
- ^ Wolfe AJ, Brubaker L (February 2019). "Urobiome updates: advances in urinary microbiome research". Nature Reviews. Urology. 16 (2): 73–74. doi:10.1038/s41585-018-0127-5. PMC 6628711. PMID 30510275.
- ^ a b Petrova MI, Lievens E, Malik S, Imholz N, Lebeer S (2015). "Lactobacillus species as biomarkers and agents that can promote various aspects of vaginal health". Frontiers in Physiology. 6: 81. doi:10.3389/fphys.2015.00081. PMC 4373506. PMID 25859220.
- ^ a b c d Witkin SS, Linhares IM, Giraldo P (June 2007). "Bacterial flora of the female genital tract: function and immune regulation". Best Practice & Research. Clinical Obstetrics & Gynaecology. 21 (3): 347–54. doi:10.1016/j.bpobgyn.2006.12.004. PMID 17215167.
- ^ Todar K (2012). "The Normal Bacterial Flora of Humans". Todar's Online Textbook of Bacteriology. Madison, WI: Kenneth Todar. Retrieved 6 April 2012.
- ^ Onderdonk AB, Zamarchi GR, Walsh JA, Mellor RD, Muñoz A, Kass EH (February 1986). "Methods for quantitative and qualitative evaluation of vaginal microflora during menstruation". Applied and Environmental Microbiology. 51 (2): 333–9. Bibcode:1986ApEnM..51..333O. doi:10.1128/AEM.51.2.333-339.1986. PMC 238869. PMID 3954346.
- ^ Antonio MA, Hawes SE, Hillier SL (December 1999). "The identification of vaginal Lactobacillus species and the demographic and microbiologic characteristics of women colonized by these species". The Journal of Infectious Diseases. 180 (6): 1950–6. doi:10.1086/315109. PMID 10558952.
- ^ Usyk M, Zolnik CP, Castle PE, Porras C, Herrero R, Gradissimo A, et al. (March 2020). "Cervicovaginal microbiome and natural history of HPV in a longitudinal study". PLOS Pathogens. 16 (3): e1008376. doi:10.1371/journal.ppat.1008376. PMC 7098574. PMID 32214382.
- ^ Fox C, Eichelberger K (December 2015). "Maternal microbiome and pregnancy outcomes". Fertility and Sterility. 104 (6): 1358–63. doi:10.1016/j.fertnstert.2015.09.037. PMID 26493119.
- ^ Wassenaar TM, Panigrahi P (December 2014). "Is a foetus developing in a sterile environment?". Letters in Applied Microbiology. 59 (6): 572–9. doi:10.1111/lam.12334. PMID 25273890. S2CID 206169539.
- ^ Schwiertz A (2016). Microbiota of the human body : implications in health and disease. Switzerland: Springer. p. 1. ISBN 978-3-319-31248-4.
- ^ Tamburini S, Shen N, Wu HC, Clemente JC (July 2016). "The microbiome in early life: implications for health outcomes". Nature Medicine. 22 (7): 713–22. doi:10.1038/nm.4142. PMID 27387886. S2CID 2462790.
- ^ de Goffau MC, Lager S, Sovio U, Gaccioli F, Cook E, Peacock SJ, et al. (August 2019). "Human placenta has no microbiome but can contain potential pathogens". Nature. 572 (7769): 329–334. Bibcode:2019Natur.572..329D. doi:10.1038/s41586-019-1451-5. PMC 6697540. PMID 31367035.
- ^ Eisenhofer R, Minich JJ, Marotz C, Cooper A, Knight R, Weyrich LS (February 2019). "Contamination in Low Microbial Biomass Microbiome Studies: Issues and Recommendations". Trends in Microbiology. 27 (2): 105–117. doi:10.1016/j.tim.2018.11.003. PMID 30497919. S2CID 54166123.
- ^ Franasiak JM, Scott RT (December 2015). "Reproductive tract microbiome in assisted reproductive technologies". Fertility and Sterility. 104 (6): 1364–71. doi:10.1016/j.fertnstert.2015.10.012. PMID 26597628.
- ^ Sutter VL (1984). "Anaerobes as normal oral flora". Reviews of Infectious Diseases. 6 (Suppl 1): S62-566. doi:10.1093/clinids/6.Supplement_1.S62. PMID 6372039.
- ^ a b c d e Kumar PS (December 2013). "Oral microbiota and systemic disease". Anaerobe. 24: 90–3. doi:10.1016/j.anaerobe.2013.09.010. PMID 24128801.
- ^ Arweiler NB, Netuschil L (May 2016). "The Oral Microbiota". In Schwiertz A (ed.). Microbiota of the Human Body: Implications in Health and Disease. Advances in Experimental Medicine and Biology. Vol. 902. Springer, Cham. pp. 45–60. doi:10.1007/978-3-319-31248-4_4. ISBN 978-3-319-31248-4. PMID 27161350.
- ^ a b c d Avila M, Ojcius DM, Yilmaz O (August 2009). "The oral microbiota: living with a permanent guest". DNA and Cell Biology. 28 (8): 405–11. doi:10.1089/dna.2009.0874. PMC 2768665. PMID 19485767.
- ^ a b Rogers AH, ed. (2008). Molecular Oral Microbiology. Caister Academic Press. ISBN 978-1-904455-24-0.
- ^ a b c d e Zarco MF, Vess TJ, Ginsburg GS (March 2012). "The oral microbiome in health and disease and the potential impact on personalized dental medicine". Oral Diseases. 18 (2): 109–20. doi:10.1111/j.1601-0825.2011.01851.x. PMID 21902769. S2CID 24411104.
- ^ Rhoades NS, Pinski AN, Monsibais AN, Jankeel A, Doratt BM, Cinco IR, et al. (August 2021). "Acute SARS-CoV-2 infection is associated with an increased abundance of bacterial pathogens, including Pseudomonas aeruginosa in the nose". Cell Reports. 36 (9): 109637. doi:10.1016/j.celrep.2021.109637. ISSN 2211-1247. PMC 8361213. PMID 34433082.
- ^ Wing Ho Man; Wouter A A de Steenhuijsen Piters; Debby Bogaert (20 March 2017). "The microbiota of the respiratory tract: gatekeeper to respiratory health". Nature Reviews Microbiology. 15 (5): 259–270. doi:10.1038/NRMICRO.2017.14. hdl:20.500.11820/f1137874-9c51-401b-bca4-e2a5da3e219b. ISSN 1740-1534. PMC 7097736. PMID 28316330.Wikidata: Q34553608
- ^ a b Beringer PM, Appleman MD (November 2000). "Unusual respiratory bacterial flora in cystic fibrosis: microbiologic and clinical features" (PDF). Current Opinion in Pulmonary Medicine. 6 (6): 545–50. doi:10.1097/00063198-200011000-00015. PMID 11100967. S2CID 845977. Archived from the original (PDF) on 16 October 2013.
- ^ Verdier J, Luedde T, Sellge G (June 2015). "Biliary Mucosal Barrier and Microbiome". Viszeralmedizin. 31 (3): 156–61. doi:10.1159/000431071. PMC 4569210. PMID 26468308.
- ^ Yu B, Yu B, Yu L (June 2020). "Commentary: Reconciling Hygiene and Cleanliness: A New Perspective from Human Microbiome". Indian Journal of Microbiology. 60 (2): 259–261. doi:10.1007/s12088-020-00863-w. PMC 7105528. PMID 32255860.
- ^ 코프랜드 CS우리 안의 세계: 건강과 인간 마이크로바이옴.뉴올리언스의 헬스케어 저널, 2017년 9월~10월호.
- ^ Honda K, Littman DR (July 2016). "The microbiota in adaptive immune homeostasis and disease". Nature. 535 (7610): 75–84. Bibcode:2016Natur.535...75H. doi:10.1038/nature18848. PMID 27383982. S2CID 4461492.
- ^ Liubakka A, Vaughn BP (July 2016). "Clostridium difficile Infection and Fecal Microbiota Transplant". AACN Advanced Critical Care. 27 (3): 324–337. doi:10.4037/aacnacc2016703. PMC 5666691. PMID 27959316.
- ^ Burton JH, Johnson M, Johnson J, Hsia DS, Greenway FL, Heiman ML (July 2015). "Addition of a Gastrointestinal Microbiome Modulator to Metformin Improves Metformin Tolerance and Fasting Glucose Levels". Journal of Diabetes Science and Technology. 9 (4): 808–14. doi:10.1177/1932296815577425. PMC 4525649. PMID 25802471.
- ^ a b Bakken JS, Borody T, Brandt LJ, Brill JV, Demarco DC, Franzos MA, et al. (December 2011). "Treating Clostridium difficile infection with fecal microbiota transplantation". Clinical Gastroenterology and Hepatology. 9 (12): 1044–9. doi:10.1016/j.cgh.2011.08.014. PMC 3223289. PMID 21871249.
- ^ Gough E, Shaikh H, Manges AR (November 2011). "Systematic review of intestinal microbiota transplantation (fecal bacteriotherapy) for recurrent Clostridium difficile infection". Clinical Infectious Diseases. 53 (10): 994–1002. doi:10.1093/cid/cir632. PMID 22002980.
- ^ Brown WR (August 2014). "Fecal microbiota transplantation in treating Clostridium difficile infection". Journal of Digestive Diseases. 15 (8): 405–8. doi:10.1111/1751-2980.12160. PMID 24825534. S2CID 44651256.
- ^ Burke KE, Lamont JT (August 2013). "Fecal transplantation for recurrent Clostridium difficile infection in older adults: a review". Journal of the American Geriatrics Society. 61 (8): 1394–8. doi:10.1111/jgs.12378. PMID 23869970. S2CID 34998497.
- ^ Drekonja D, Reich J, Gezahegn S, Greer N, Shaukat A, MacDonald R, et al. (May 2015). "Fecal Microbiota Transplantation for Clostridium difficile Infection: A Systematic Review". Annals of Internal Medicine. 162 (9): 630–8. doi:10.7326/m14-2693. PMID 25938992. S2CID 1307726.
- ^ Brandt LJ, Borody TJ, Campbell J (September 2011). "Endoscopic fecal microbiota transplantation: "first-line" treatment for severe clostridium difficile infection?". Journal of Clinical Gastroenterology. 45 (8): 655–7. doi:10.1097/MCG.0b013e3182257d4f. PMID 21716124.
- ^ Kelly CR, de Leon L, Jasutkar N (February 2012). "Fecal microbiota transplantation for relapsing Clostridium difficile infection in 26 patients: methodology and results". Journal of Clinical Gastroenterology. 46 (2): 145–9. doi:10.1097/MCG.0b013e318234570b. PMID 22157239. S2CID 30849491.
- ^ a b c d e f g h Garrett WS (April 2015). "Cancer and the microbiota". Science. 348 (6230): 80–6. Bibcode:2015Sci...348...80G. doi:10.1126/science.aaa4972. PMC 5535753. PMID 25838377.
- ^ a b c d Gagnière J, Raisch J, Veziant J, Barnich N, Bonnet R, Buc E, et al. (January 2016). "Gut microbiota imbalance and colorectal cancer". World Journal of Gastroenterology. 22 (2): 501–18. doi:10.3748/wjg.v22.i2.501. PMC 4716055. PMID 26811603.
- ^ a b Sartor RB, Mazmanian SK (July 2012). "Intestinal Microbes in Inflammatory Bowel Diseases". The American Journal of Gastroenterology Supplements. 1 (1): 15–21. doi:10.1038/ajgsup.2012.4.
- ^ Hold GL, Smith M, Grange C, Watt ER, El-Omar EM, Mukhopadhya I (February 2014). "Role of the gut microbiota in inflammatory bowel disease pathogenesis: what have we learnt in the past 10 years?". World Journal of Gastroenterology. 20 (5): 1192–210. doi:10.3748/wjg.v20.i5.1192. PMC 3921503. PMID 24574795.
- ^ a b Zilberman-Schapira G, Zmora N, Itav S, Bashiardes S, Elinav H, Elinav E (June 2016). "The gut microbiome in human immunodeficiency virus infection". BMC Medicine. 14 (1): 83. doi:10.1186/s12916-016-0625-3. PMC 4891875. PMID 27256449.
- ^ Petrova MI, van den Broek M, Balzarini J, Vanderleyden J, Lebeer S (September 2013). "Vaginal microbiota and its role in HIV transmission and infection". FEMS Microbiology Reviews. 37 (5): 762–92. doi:10.1111/1574-6976.12029. PMID 23789590.
- ^ Intagliata C (22 December 2016). ""Necrobiome" Reveals a Corpse's Time of Death". Scientific American. Retrieved 26 March 2018.
- ^ Young E (10 December 2015). "Meet the Necrobiome: The Waves of Microbes That Will Eat Your Corpse". The Atlantic. Retrieved 26 March 2018.
- ^ Harmon K (16 December 2009). "Bugs Inside: What Happens When the Microbes That Keep Us Healthy Disappear?". Scientific American. Retrieved 27 December 2008.
- ^ a b Vangay P, Johnson AJ, Ward TL, Al-Ghalith GA, Shields-Cutler RR, Hillmann BM, et al. (November 2018). "US Immigration Westernizes the Human Gut Microbiome". Cell. 175 (4): 962–972.e10. doi:10.1016/j.cell.2018.10.029. PMC 6498444. PMID 30388453.
- ^ a b Kaplan RC, Wang Z, Usyk M, Sotres-Alvarez D, Daviglus ML, Schneiderman N, et al. (November 2019). "Gut microbiome composition in the Hispanic Community Health Study/Study of Latinos is shaped by geographic relocation, environmental factors, and obesity". Genome Biology. 20 (1): 219. doi:10.1186/s13059-019-1831-z. PMC 6824043. PMID 31672155.
외부 링크
- 2015~2016년 미국 자연사 박물관 '당신 안의 비밀 세계' 전시
- FAQ: Human Microbiom, 2014년 1월 미국 미생물학회