계산생물학
Computational biology이 문서에서는 문법, 스타일, 응집력, 톤 또는 철자를 복사 편집해야 할 수 있습니다.할수 . (2022년 6월 ( 템플릿메시지의에 대해 알아보기) |
계산생물학은 생물학적 시스템과 [1]관계를 이해하기 위해 데이터 분석, 수학적 모델링 및 계산 시뮬레이션을 사용하는 것을 말한다.컴퓨터 공학, 생물학, 빅데이터의 교차로인 이 분야는 또한 응용 수학, 화학,[2] 유전학의 기초를 가지고 있다.그것은 컴퓨터를 만들기 위해 생명공학을 사용하는 컴퓨터 공학의 하위 분야인 생물 컴퓨팅과는 다릅니다.
역사
생물 정보학, 즉 생물 시스템의 정보학 과정을 분석하는 것은 1970년대 초에 시작되었다.이 시기에 인공지능 연구는 새로운 알고리즘을 만들기 위해 인간 두뇌의 네트워크 모델을 사용하고 있었다.이러한 생물학적 데이터의 사용은 생물학 연구자들이 자신들의 분야에서 [3]큰 데이터 세트를 평가하고 비교하기 위해 컴퓨터를 사용하도록 강요했다.
1982년까지 연구원들은 펀치 카드를 통해 정보를 공유했다.데이터의 양은 1980년대 말까지 기하급수적으로 증가했고, 관련 [3]정보를 신속하게 해석하기 위한 새로운 계산 방법이 필요했다.
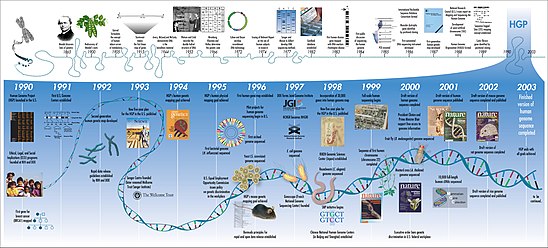
아마도 컴퓨터 생물학의 가장 잘 알려진 예인 인간 게놈 프로젝트는 [4]1990년에 공식적으로 시작되었다.2003년까지 이 프로젝트는 인간 게놈의 약 85%를 매핑하여 초기 [5]목표를 달성했습니다.그러나 작업은 계속 진행되었고 2021년에는 "완전 게놈" 수준에 도달했으며, 잠재적 [6][7]문제로 커버되는 베이스는 0.3%에 불과했다.사라진 Y염색체는 2022년 1월에 추가되었다.
1990년대 후반부터 컴퓨터 생물학은 생물학에서 중요한 부분이 되어 수많은 하위 [8]분야로 이어졌다.오늘날, 국제 컴퓨터 생물학 협회는 21개의 다른 '특별한 관심의 공동체'를 인정하고 있으며, 각각은 더 [9]큰 분야의 한 부분을 대표한다.컴퓨터 생물학은 인간 게놈의 염기서열 분석뿐만 아니라 인간 두뇌의 정확한 모델 작성, 게놈의 3D 구조 지도 작성, 생체 시스템 [3]모델 작성에도 도움을 주었습니다.
적용들
해부학
컴퓨터 해부학은 형태학의 가시적 또는 총 해부학적 (\ 척도의 해부학적 형태와 형태를 연구하는 학문이다.여기에는 생물학적 구조를 모델링하고 시뮬레이션하기 위한 계산, 수학 및 데이터 분석 방법의 개발이 포함됩니다.의료 영상촬영 장치보다는 영상촬영 중인 해부학적 구조에 초점을 맞춥니다.자기공명영상 등의 기술을 통한 밀도 높은 3D 측정이 가능해짐에 따라 컴퓨터 해부학은 3D 형태소 척도의 해부학적 좌표계를 추출하기 위한 의료 영상 및 생물 공학 분야의 하위 분야로 부상했다.
컴퓨터 해부학의 원래 공식은 변환을 [10]통해 작용한 예로부터 형태와 형태의 생성 모델이다.미분형성 그룹은 R의 해부학적 구성 중 것으로의 흐름의과 오일러의 속도를 통해 생성된 좌표 변환을 통해 다른 좌표계를 연구하는 데 사용된다그것은 형태 통계학 및 형태 측정학과 관련이 있으며, 미분 동형이 좌표계를 매핑하는 데 사용된다는 구별과 관련이 있으며, 그 연구는 미분 동형 측정학으로 알려져 있다.
데이터 및 모델링
수학 생물학은 생물 시스템의 구조, 발달, 행동을 지배하는 시스템을 조사하기 위해 살아있는 유기체의 수학적 모델을 사용하는 것입니다.이것은 실험 [11]생물학의 경험적 사고방식보다는 문제에 대한 보다 이론적인 접근을 수반한다.수학 생물학은 이산 수학, 위상(계산 모델링에도 유용), 베이지안 통계, 선형 대수 및 부울 [12]대수학을 활용합니다.
이러한 수학적 접근방식은 생물학적 데이터를 저장, 검색 및 분석하기 위한 데이터베이스와 다른 방법을 만들 수 있게 했다. 이 분야는 생물정보학으로 알려져 있다.보통, 이 과정은 유전학과 유전자 분석을 포함한다.
대규모 데이터 세트를 수집하고 분석함으로써 데이터 마이닝,[12] 컴퓨터 모델 구축 및 생체 시스템의 시각적 시뮬레이션을 참조하는 컴퓨터 생체 모델링과 같은 증가하는 연구 분야에 자리를 내주고 있습니다.이를 통해 연구자들은 그러한 시스템이 다른 환경에 어떻게 반응할지를 예측할 수 있으며, 시스템이 "외부 및 내부 [13]섭동에 대해 상태와 기능을 유지할 수 있는지" 판단하는 데 유용하다.현재의 기술은 작은 생물학적 시스템에 초점을 맞추고 있지만, 연구자들은 대규모 네트워크를 분석하고 모델링할 수 있는 접근방식을 연구하고 있습니다.대다수의 연구원들은 이것이 새로운 약을 만들고 유전자 [13]치료법을 만드는 현대 의학적인 접근을 개발하는데 필수적일 것이라고 믿고 있다.유용한 모델링 방법은 esyN [14]등의 도구를 통해 Petri net을 사용하는 것입니다.
유사한 노선을 따라, 최근 수십 년 동안 이론 생태학은 경험 생태학자들이 사용하는 통계 모델로부터 분리된 분석 모델을 주로 다루어 왔다.그러나, 계산 방법은 생태학적 분석에서 계산 통계에서 방법의 적용을 증가시키는 것 외에 생태학적 시스템의 시뮬레이션을 통해 생태학적 이론을 개발하는 데 도움을 주었다.
시스템 생물학
시스템 생물학은 발생 성질을 발견하는 것을 목표로 세포 수준에서 전체 모집단에 이르는 다양한 생물학적 시스템 간의 상호작용을 계산하는 것으로 구성됩니다.이 과정은 보통 네트워크 세포 신호와 대사 경로를 포함한다.시스템 생물학은 세포 수준에서 이러한 복잡한 상호작용을 연구하기 위해 종종 생물학적 모델링과 그래프 이론의 [12]계산 기술을 사용합니다.
진화생물학
컴퓨터 생물학은 다음과 같은 방법으로 진화생물학을 지원했습니다.
- DNA 데이터를 사용하여 계산 계통학으로 생명의 나무를 재구성하는 것
- 인구통계학적 또는 선택적 이력에 대한 추론을 위해 DNA 데이터에 모집단 유전학 모델(전진[15] 시간 또는 후진 시간)을 적합시킨다.
- 진화할 가능성을 예측하기 위해 제1원칙부터 진화 시스템의 집단 유전학 모델을 구축한다.
유전체학
컴퓨터 유전체학은 세포와 유기체의 게놈에 대한 연구이다.인간 게놈 프로젝트는 컴퓨터 유전체학의 한 예이다.이 프로젝트는 인간 게놈 전체를 일련의 데이터로 배열하는 것을 목표로 하고 있다.일단 완전히 시행되면, 의사들은 개별 환자의 [16]게놈을 분석할 수 있게 될 것이다.이것은 개인의 기존 유전자 패턴에 기초한 치료를 처방하는 개인 맞춤형 의학의 가능성을 열어준다.연구자들은 동물, 식물, 박테리아, 그리고 다른 모든 종류의 [17]생명체의 게놈 배열을 찾고 있다.
게놈을 비교하는 주요 방법 중 하나는 배열 호몰로지에 의한 것입니다.호몰로지는 공통의 조상으로부터 유래한 다른 유기체의 생물학적 구조와 뉴클레오티드 배열에 대한 연구이다.연구에 따르면 새로 배열된 원핵생물 게놈의 80~90%가 이런 방식으로 [17]확인될 수 있다.
배열 정렬은 생물학적 배열 또는 유전자 간의 유사성을 비교하고 검출하는 또 다른 과정이다.배열 정렬은 두 유전자의 가장 긴 공통적 후속 계산이나 특정 [18]질병의 변종 비교와 같은 많은 생물 정보학 애플리케이션에서 유용합니다.
컴퓨터 유전체학에서 손대지 않은 프로젝트는 인간 [17]게놈의 약 97%를 구성하는 유전자간 영역의 분석이다.연구자들은 계산 및 통계 방법의 개발과 ENCODE 및 Roadmap 에피게노믹스 프로젝트와 같은 대규모 컨소시엄 프로젝트를 통해 인간 게놈의 비코딩 영역의 기능을 이해하기 위해 노력하고 있습니다.
개별 유전자가 분자, 세포, 그리고 유기체 수준에서 유기체의 생물학에 어떻게 기여하는지를 이해하는 것은 유전자 존재학으로 알려져 있다.Gene Ontology Consortium의 사명은 분자 수준에서 보다 큰 경로, 세포 및 유기체 수준의 시스템에 이르기까지 최신의 포괄적이고 계산적인 생물학적 시스템 모델을 개발하는 것입니다.Gene Ontology 자원은 인간에서 [19]박테리아까지 다양한 유기체에서 생성된 유전자(또는 유전자에 의해 생성된 단백질과 비코드 RNA 분자)의 기능에 대한 현재의 과학적 지식의 컴퓨터 표현을 제공합니다.
3D 유전체학은 진핵 세포 내에서 유전자의 구성과 상호작용에 초점을 맞춘 컴퓨터 생물학의 하위 섹션이다.GAM은 DNA를 조사하기 위해 핵에서 스트립을 잘라내는 과정인 저온처리를 레이저 미세해부와 결합해 게놈 내 염색질과 DNA의 3D 거리를 측정한다.핵 프로파일은 단순히 핵에서 떼어낸 이 스트립 또는 슬라이스이다.각 핵 프로파일은 DNA의 기본 단위인 특정 뉴클레오티드의 배열인 게놈 창을 포함한다. GAM은 [20]세포 전체에 걸쳐 복잡하고 다원적인 인핸서 크로마틴 접촉의 게놈 네트워크를 포착한다.
신경과학
컴퓨터 신경과학은 신경계의 정보처리 특성 측면에서 뇌 기능을 연구하는 학문이다.신경과학의 일부로서, 신경계의 [21]특정 측면을 조사하기 위해 뇌를 모델링하는 것으로 보입니다.뇌의 모델은 다음과 같습니다.
- 현실적인 두뇌 모델:이 모델들은 세포 수준에서 가능한 한 많은 세부 사항을 포함하여 뇌의 모든 측면을 나타내는 것으로 보입니다.현실적 모델은 뇌에 대한 가장 많은 정보를 제공하지만 오류의 여지도 가장 크다.뇌 모델에 변수가 많을수록 오류가 더 많이 발생할 가능성이 있습니다.이 모델들은 과학자들이 알지 못하는 세포 구조의 일부를 설명하지 않는다.현실적인 뇌 모델은 가장 계산량이 많고 [22]구현 비용이 많이 듭니다.
- 뇌 모델의 심플화:이러한 모델은 신경계의 특정 물리적 특성을 평가하기 위해 모델의 범위를 제한하는 것으로 보인다.이를 통해 집중적인 계산 문제를 해결할 수 있으며 현실적인 뇌 모델에서 [22]발생할 수 있는 오류의 양을 줄일 수 있습니다.
이러한 계산의 속도를 높이기 위해 현재 사용되는 알고리즘과 데이터 구조를 개선하는 것은 컴퓨터 신경 과학자들의 작업이다.
컴퓨터 신경정신과학은 정신 질환에 관련된 뇌 메커니즘의 수학적 및 컴퓨터 보조 모델링을 사용하는 새로운 분야이다.몇 가지 이니셔티브는 컴퓨터 모델링이 정신 기능과 기능 [23][24][25]부전을 발생시킬 수 있는 신경 회로를 이해하는 데 중요한 기여를 한다는 것을 입증했다.
약리학
컴퓨터 약리학은 "특정 유전자형과 질병 사이의 연관성을 찾고 약물 [26]데이터를 선별하기 위한 게놈 데이터의 효과에 대한 연구"이다.제약업계는 약물 데이터를 분석하는 방법의 변화가 필요합니다.약사들은 마이크로소프트 엑셀을 사용하여 약물의 효과와 관련된 화학 및 게놈 데이터를 비교할 수 있었다.그러나 업계는 Excel 바리케이드라고 불리는 것에 도달했다.이는 스프레드시트에서 액세스할 수 있는 셀 수가 제한되어 있기 때문에 발생합니다.이러한 개발은 컴퓨터 약리학의 필요성으로 이어졌다.과학자들과 연구원들은 이러한 방대한 데이터 세트를 분석하기 위한 계산 방법을 개발한다.이를 통해 주목할 만한 데이터 포인트를 효율적으로 비교할 수 있으며 보다 정확한 약을 [27]개발할 수 있다.
분석가들은 주요 의약품들이 특허로 인해 실패할 경우, 그 컴퓨터 생물학은 현재 시판 중인 의약품들을 대체하기 위해 필요할 것이라고 예측하고 있다.컴퓨터 생물학 박사과정 학생들은 박사후 직책을 맡기보다는 산업분야에서 경력을 쌓을 것을 권장받고 있다.이는 주요 제약회사들이 [27]신약 생산에 필요한 대규모 데이터 세트에 대해 보다 자격 있는 분석가를 필요로 하는 직접적인 결과입니다.
마찬가지로, 컴퓨터 종양학은 알고리즘 접근법을 통해 암의 미래 돌연변이를 결정하는 것을 목표로 한다.이 분야의 연구는 로봇 공학 및 기타 감지 장치를 사용하여 수백만 개의 데이터가 가리키는 높은 처리량 측정의 사용으로 이어졌습니다.이 데이터는 DNA, RNA 및 기타 생물학적 구조에서 수집됩니다.주목되는 분야에는 종양의 특징 결정, 암을 유발하는 결정론적 분자 분석, 그리고 인간 게놈이 종양과 [28][29]암의 인과관계에 대한 이해 등이 포함된다.
기술
컴퓨터 생물학자들은 연구를 수행하기 위해 광범위한 소프트웨어와 알고리즘을 사용합니다.
비지도 학습
비지도 학습은 레이블이 없는 데이터에서 패턴을 찾는 알고리즘의 한 유형이다.한 가지 예는 n개의 데이터 점을 k개의 군집으로 분할하는 것을 목적으로 하는 k-평균 군집 분석입니다. 이 군집에서는 각 데이터 점이 가장 가까운 평균을 갖는 군집에 속합니다.또 다른 버전은 k-중간 알고리즘입니다. 이 알고리즘은 군집 중심 또는 군집 중심을 선택할 때 군집의 평균뿐만 아니라 집합의 데이터 점 중 하나를 선택합니다.
알고리즘은, 다음의 순서에 따릅니다.
- k개의 구별되는 데이터 점을 랜덤하게 선택합니다.이것이 초기 클러스터입니다.
- 각 점과 각 'k' 군집 사이의 거리를 측정합니다(이것은 각 점 k로부터의 점 거리입니다).
- 각 점을 가장 가까운 군집에 할당합니다.
- 각 군집의 중심(중간체)을 찾습니다.
- 클러스터가 더 이상 변경되지 않을 때까지 반복합니다.
- 각 클러스터 내의 변동을 합산하여 클러스터 품질을 평가합니다.
- 서로 다른 k 값을 사용하여 공정을 반복합니다.
- 그래프에서 분산이 가장 작은 "팔꿈치"를 찾아서 "k"에 대한 최적의 값을 선택합니다.
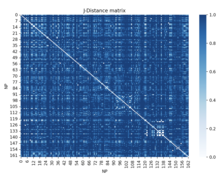
생물학의 한 예는 게놈의 3D 매핑에 사용됩니다.유전자 발현 옴니버스로부터 [30]생쥐의 염색체 13개 HIST1 영역 정보를 수집한다.이 정보에는 특정 게놈 영역에서 나타나는 핵 프로파일에 대한 데이터가 포함되어 있다.이 정보를 사용하여 Jaccard 거리를 사용하여 모든 위치 사이의 정규화된 거리를 찾을 수 있습니다.
그래프 분석
그래프 분석 또는 네트워크 분석은 서로 다른 개체 간의 연결을 나타내는 그래프에 대한 연구입니다.그래프는 단백질-단백질 상호작용 네트워크, 조절 네트워크, 대사 및 생화학 네트워크 등 생물학에서 모든 종류의 네트워크를 나타낼 수 있습니다.이러한 네트워크를 분석하는 방법은 여러 가지가 있습니다.그 중 하나는 중앙집중성을 그래프로 보는 것입니다.그래프에서 중심점을 찾으면 노드 순위가 그래프에서 노드 인기 또는 중심점에 할당됩니다.이것은 어떤 노드가 가장 중요한지 알아내는 데 도움이 될 수 있습니다.이것은 생물학에서 많은 면에서 매우 유용할 수 있다.예를 들어 주어진 기간 동안 유전자의 활성에 대한 데이터를 가지고 있다면, 네트워크 전체에서 어떤 유전자가 가장 활성화 되어 있는지 또는 네트워크 전체에서 어떤 유전자가 다른 유전자와 가장 많이 상호 작용하는지 확인하기 위해 정도 중심성을 사용할 수 있습니다.이것은 특정 유전자가 네트워크에서 어떤 역할을 하는지 이해하는 데 도움을 줄 수 있다.
그래프에서 중심점을 계산하는 방법은 여러 가지가 있으며, 이 모든 방법은 중심점에 대한 다양한 정보를 제공할 수 있습니다.생물학에서 중심점을 찾는 것은 유전자 조절, 단백질 상호작용 그리고 신진대사 네트워크와 [31]같은 많은 다양한 상황에서 적용될 수 있습니다.
지도 학습
지도 학습은 레이블이 지정된 데이터에서 학습하고 레이블이 없는 미래 데이터에 레이블을 할당하는 방법을 배우는 알고리즘의 한 유형입니다.생물학에서 감독 학습은 분류하는 방법을 알고 더 많은 데이터를 분류할 때 도움이 될 수 있습니다.
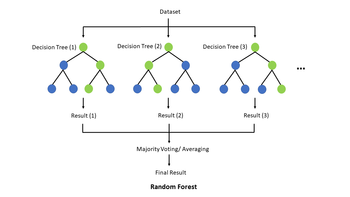
일반적인 지도 학습 알고리즘은 랜덤 포레스트입니다. 랜덤 포레스트는 다수의 의사결정 트리를 사용하여 모델을 훈련시켜 데이터 세트를 분류합니다.랜덤 포레스트의 기초를 형성하는 의사결정 트리는 데이터의 특정 알려진 특징을 사용하여 데이터 집합을 분류하거나 라벨을 붙이는 것을 목적으로 하는 구조이다.이것의 실용적인 생물학적 예는 개인의 유전자 데이터를 가져다가 그 개인이 특정 질병이나 암에 걸릴지 여부를 예측하는 것이다.각 내부 노드에서 알고리즘은 데이터셋을 정확히 하나의 특징(이전 예의 특정 유전자)으로 체크하고 그 결과에 따라 왼쪽 또는 오른쪽으로 분기합니다.그런 다음 각 리프 노드에서 결정 트리가 데이터 세트에 클래스 레이블을 할당합니다.따라서 실제로는 알고리즘이 입력 데이터 세트를 기반으로 특정 루트-리프 경로를 Decision Tree를 통해 이동하므로 데이터 세트가 분류됩니다.일반적으로 의사 결정 트리에는 예/아니오처럼 이산 값을 갖는 대상 변수가 있으며, 이 경우 이를 분류 트리라고 하지만 대상 변수가 연속형이면 회귀 트리라고 합니다.의사 결정 트리를 구성하려면 먼저 대상 변수의 가장 좋은 예측 변수를 식별하기 위한 교육 세트를 사용하여 의사 결정 트리를 훈련해야 합니다.
오픈 소스 소프트웨어
오픈 소스 소프트웨어는 컴퓨터 생물학용 플랫폼을 제공합니다.이 플랫폼에서는 모든 사람이 연구용으로 개발된 소프트웨어에 액세스하여 이점을 얻을 수 있습니다.PLOS는 오픈[citation needed] 소스 소프트웨어를 사용하는 4가지 주요 이유를 들고 있습니다.
- 재현성:이를 통해 연구자들은 생물학적 데이터 간의 관계를 계산하는 데 사용되는 정확한 방법을 사용할 수 있습니다.
- 개발 속도 향상: 개발자 및 연구자는 사소한 작업에 대해 기존 코드를 재창조할 필요가 없습니다.대신 기존 프로그램을 사용하여 대규모 프로젝트의 개발 및 구현 시간을 절약할 수 있습니다.
- 품질 향상:같은 주제를 연구하는 여러 연구자로부터 입력을 받는 것은 코드에 오류가 없다는 것을 보증하는 층을 제공한다.
- 장기적인 가용성:오픈 소스 프로그램은 어떤 기업이나 특허에도 얽매이지 않습니다.이를 통해 여러 웹 페이지에 게시할 수 있으며 나중에 사용할 [32]수 있습니다.
조사.
컴퓨터 생물학과 관련된 몇 개의 큰 컨퍼런스가 있다.몇 가지 주목할 만한 예로는 분자생물학을 위한 인텔리전트 시스템, 계산생물학 유럽회의, 계산분자생물학 연구 등이 있습니다.
컴퓨터 생물학에 관한 수많은 저널도 있다.일부 주목할 만한 예로는 Journal of Computational Biology와 PLOS Computational Biology가 있습니다.이는 컴퓨터 생물학 분야에서 주목할 만한 연구 프로젝트를 많이 보유한 동료 검토 오픈 액세스 저널입니다.소프트웨어 리뷰, 오픈 소스 소프트웨어 튜토리얼, 다가오는 컴퓨터 생물학 [citation needed]컨퍼런스에 대한 정보를 제공합니다.
관련 필드
계산생물학, 생물정보학 및 수리생물학은 모두 생명과학에 대한 학제간 접근법으로서 수학과 정보과학 같은 양적 분야로부터 도출된다.NIH는 계산/수학적 생물학을 생물학에서 이론 및 실험 질문을 다루기 위한 계산/수학적 접근법의 사용으로 설명하고, 반대로 생물정보학은 복잡한 생명과학 [1]데이터를 이해하기 위한 정보과학의 적용으로 설명한다.
구체적으로 NIH는 다음을 정의합니다.
컴퓨터 생물학:생물학적,[1] 행동적, 사회적 시스템 연구에 대한 데이터 분석 및 이론적 방법, 수학적 모델링 및 계산 시뮬레이션 기술의 개발과 적용.
생물정보학:생물학적,[1] 의료적, 행동적 또는 건강적 데이터의 사용을 확대하기 위한 계산 도구와 접근법의 연구, 개발 또는 적용(이러한 데이터의 취득, 저장, 정리, 보관, 분석 또는 시각화를 포함한다).
각 분야가 구별되는 반면,[1] 그들의 인터페이스에는 상당한 중복이 있을 수 있기 때문에, 많은 사람들에게 생물정보학과 계산생물학은 상호 교환적으로 사용되는 용어이다.
계산 생물학과 진화 계산이라는 용어는 비슷한 이름을 가지고 있지만 혼동해서는 안 된다.컴퓨터 생물학과는 달리 진화적 계산은 생물학적 데이터를 모델링하고 분석하는 것과 관련이 없습니다.대신 여러 종에 걸친 진화의 개념을 바탕으로 알고리즘을 만듭니다.때때로 유전자 알고리즘이라고 불리는 이 분야의 연구는 컴퓨터 생물학에 적용될 수 있다.진화적 계산은 본질적으로 계산생물학의 일부가 아니지만, 계산적 진화생물학은 그것의 [33]하위 분야이다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b c d e "NIH working definition of bioinformatics and computational biology" (PDF). Biomedical Information Science and Technology Initiative. 17 July 2000. Archived from the original (PDF) on 5 September 2012. Retrieved 18 August 2012.
- ^ "About the CCMB". Center for Computational Molecular Biology. Retrieved 18 August 2012.
- ^ a b c Hogeweg, Paulien (7 March 2011). "The Roots of Bioinformatics in Theoretical Biology". PLOS Computational Biology. 3. 7 (3): e1002021. Bibcode:2011PLSCB...7E2021H. doi:10.1371/journal.pcbi.1002021. PMC 3068925. PMID 21483479.
- ^ "The Human Genome Project". The Human Genome Project. 22 December 2020. Retrieved 13 April 2022.
- ^ "Human Genome Project FAQ". Genome.gov. Retrieved 2022-04-20.
- ^ "T2T-CHM13v1.1 - Genome - Assembly - NCBI". www.ncbi.nlm.nih.gov. Retrieved 2022-04-20.
- ^ "Genome List - Genome - NCBI". www.ncbi.nlm.nih.gov. Retrieved 2022-04-20.
- ^ Bourne, Philip (2012). "Rise and Demise of Bioinformatics? Promise and Progress". PLOS Computational Biology. 8 (4): e1002487. Bibcode:2012PLSCB...8E2487O. doi:10.1371/journal.pcbi.1002487. PMC 3343106. PMID 22570600.
- ^ "COSI Information". www.iscb.org. Retrieved 2022-04-21.
- ^ Grenander, Ulf; Miller, Michael I. (1998-12-01). "Computational Anatomy: An Emerging Discipline". Q. Appl. Math. 56 (4): 617–694. doi:10.1090/qam/1668732.
- ^ "Mathematical Biology Faculty of Science". www.ualberta.ca. Retrieved 2022-04-18.
- ^ a b c "The Sub-fields of Computational Biology". Ninh Laboratory of Computational Biology. 2013-02-18. Retrieved 2022-04-18.
- ^ a b Kitano, Hiroaki (14 November 2002). "Computational systems biology". Nature. 420 (6912): 206–10. Bibcode:2002Natur.420..206K. doi:10.1038/nature01254. PMID 12432404. S2CID 4401115. ProQuest 204483859.
- ^ Favrin, Bean (2 September 2014). "esyN: Network Building, Sharing and Publishing". PLOS ONE. 9 (9): e106035. Bibcode:2014PLoSO...9j6035B. doi:10.1371/journal.pone.0106035. PMC 4152123. PMID 25181461.
- ^ Antonio Carvajal-Rodríguez (2012). "Simulation of Genes and Genomes Forward in Time". Current Genomics. 11 (1): 58–61. doi:10.2174/138920210790218007. PMC 2851118. PMID 20808525.
- ^ "Genome Sequencing to the Rest of Us". Scientific American.
- ^ a b c Koonin, Eugene (6 March 2001). "Computational Genomics". Curr. Biol. 11 (5): 155–158. doi:10.1016/S0960-9822(01)00081-1. PMID 11267880. S2CID 17202180.
- ^ "Sequence Alignment - an overview ScienceDirect Topics". www.sciencedirect.com. Retrieved 2022-04-18.
- ^ "Gene Ontology Resource". Gene Ontology Resource. Retrieved 2022-04-18.
- ^ Beagrie, Robert A.; Scialdone, Antonio; Schueler, Markus; Kraemer, Dorothee C. A.; Chotalia, Mita; Xie, Sheila Q.; Barbieri, Mariano; de Santiago, Inês; Lavitas, Liron-Mark; Branco, Miguel R.; Fraser, James (March 2017). "Complex multi-enhancer contacts captured by genome architecture mapping". Nature. 543 (7646): 519–524. Bibcode:2017Natur.543..519B. doi:10.1038/nature21411. ISSN 1476-4687. PMC 5366070. PMID 28273065.
- ^ "Computational Neuroscience Neuroscience". www.bu.edu.
- ^ a b Sejnowski, Terrence; Christof Koch; Patricia S. Churchland (9 September 1988). "Computational Neuroscience". Science. 4871. 241 (4871): 1299–306. Bibcode:1988Sci...241.1299S. doi:10.1126/science.3045969. PMID 3045969.
- ^ Dauvermann, Maria R.; Whalley, Heather C.; Schmidt, Andrã©; Lee, Graham L.; Romaniuk, Liana; Roberts, Neil; Johnstone, Eve C.; Lawrie, Stephen M.; Moorhead, Thomas W. J. (2014). "Computational Neuropsychiatry – Schizophrenia as a Cognitive Brain Network Disorder". Frontiers in Psychiatry. 5: 30. doi:10.3389/fpsyt.2014.00030. PMC 3971172. PMID 24723894.
- ^ Tretter, F.; Albus, M. (December 2007). "'Computational Neuropsychiatry' of Working Memory Disorders in Schizophrenia: The Network Connectivity in Prefrontal Cortex - Data and Models". Pharmacopsychiatry. 40 (S 1): S2–S16. doi:10.1055/S-2007-993139. S2CID 18574327.
- ^ Marin-Sanguino, A.; Mendoza, E. (2008). "Hybrid Modeling in Computational Neuropsychiatry". Pharmacopsychiatry. 41: S85–S88. doi:10.1055/s-2008-1081464. PMID 18756425.
- ^ Price, Michael (2012-04-13). "Computational Biologists: The Next Pharma Scientists?".
- ^ a b Jessen, Walter (2012-04-15). "Pharma's shifting strategy means more jobs for computational biologists".
- ^ Barbolosi, Dominique; Ciccolini, Joseph; Lacarelle, Bruno; Barlesi, Fabrice; Andre, Nicolas (2016). "Computational oncology--mathematical modelling of drug regimens for precision medicine". Nature Reviews Clinical Oncology. 13 (4): 242–254. doi:10.1038/nrclinonc.2015.204. PMID 26598946. S2CID 22492353.
- ^ Yakhini, Zohar (2011). "Cancer Computational Biology". BMC Bioinformatics. 12: 120. doi:10.1186/1471-2105-12-120. PMC 3111371. PMID 21521513.
- ^ 링크
- ^ Koschützki, Dirk; Schreiber, Falk (2008-05-15). "Centrality Analysis Methods for Biological Networks and Their Application to Gene Regulatory Networks". Gene Regulation and Systems Biology. 2: 193–201. doi:10.4137/grsb.s702. ISSN 1177-6250. PMC 2733090. PMID 19787083.
- ^ Prlić, Andreas; Lapp, Hilmar (2012). "The PLOS Computational Biology Software Section". PLOS Computational Biology. 8 (11): e1002799. Bibcode:2012PLSCB...8E2799P. doi:10.1371/journal.pcbi.1002799. PMC 3510099.
- ^ Foster, James (June 2001). "Evolutionary Computation". Nature Reviews Genetics. 2 (6): 428–436. doi:10.1038/35076523. PMID 11389459. S2CID 205017006.

 해부학적 구성 중
해부학적 구성 중