Phi X 174
Phi X 174| 에스케리치아바이러스 φX174 | |
|---|---|
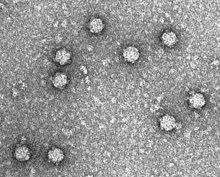 | |
| phX174 페이지의 전자 마이크로그래프 | |
| 바이러스 분류 | |
| (랭킹되지 않음):null | 바이러스 |
| 영역: | 모노드나비리아 |
| 킹덤: | 산게르비레아과 |
| 망울: | 픽스비리코타 |
| 클래스: | 말그란다비리케테스속 |
| 순서: | 쁘띠비르목 |
| 패밀리: | 미생물과 |
| 속: | 신스하메르바이러스 |
| 종: | 에스케리치아바이러스 φX174 |
phi X 174(또는 φX174) 박테리오파지는 대장균을 감염시키는 단일 가닥 DNA(ssDNA) 바이러스로서, 최초로 DNA 기반 게놈의 서열을 정리한 것이다.이 작품은 1977년 프레드 생거와 그의 팀에 의해 완성되었다.[1]1962년에 월터 피어스와 로버트 신스하이머는 이미 φX174 DNA의 물리적, 공칭적으로 닫힌 원형성을 입증했다.[2]노벨상 수상자인 아서 코른베르크(Arthur Kornberg)는 ⅡX174를 모델로 삼아 정제 효소에 의해 시험관에서 합성된 DNA가 자연 바이러스의 모든 특징을 만들어 낼 수 있다는 것을 먼저 증명해 합성생물학의 시대를 열었다.[3][4]1972–1974년, 제라드 허위츠, 수 위크너, 그리고 협력자들과의 리드 위크너는 단일 좌초된 형태의 바이러스를 이중 좌초된 복제 양식으로의 변환을 촉진하기 위해 효소를 생산하는 데 필요한 유전자를 확인했다.[5]2003년에 크레이그 벤터 그룹으로부터 genomeX174의 게놈은 합성 올리고뉴클레오티드로부터 체외에서 완전히 조립된 최초의 게놈이라는 보고를 받았다.[6]φX174 바이러스 입자도 체외에서 성공적으로 조립되었다.[7]2012년, 그것의 매우 겹치는 게놈은 어떻게 완전히 분해될 수 있고 여전히 기능할 수 있는지를 보여주었다.[8]null
게놈
이 박테리오파지는 5,386개의 뉴클레오티드의 [+] 감각의 원형 단일 가닥 DNA 게놈을 가지고 있다.[9]게놈 GC 함량은 44%, 뉴클레오티드의 95%는 코딩 유전자에 속한다.게놈의 밸런스 베이스 패턴 때문에 Illumina sequencer의 제어 DNA로 사용된다.null
유전자
φX174는 발견된 순서에 따라 알파벳의 연속 문자로 명명된 11개의 유전자를 암호화하고, 큰 A유전자 내에서 대체 시작 코돈인 A*를 제외한다.시작 코돈은 ATT로 변경될 수 있지만 다른 순서는 아니기 때문에 A*에 대한 약간의 의심은 있지만, 오직 유전자 A*와 K만이 필수적이지 않은 것으로 생각된다.[10]현재 ATT는 여전히 대장균 내에서[11] 단백질을 생산할 수 있을 가능성이 있으며 따라서 이 유전자는 사실 필수적일 수 있다는 것이 알려져 있다.null
φX174 게놈의 전반부는 11개 유전자 중 8개가 적어도 하나의 뉴클레오티드에 의해 겹치는 높은 수준의 유전자가[12] 특징이다.[1]모든 유전자 중첩이 제거된 리팩터링 페이지는 야생형으로부터의 적합성을 감소시켰지만, 이러한 중복은 필수적이지 않은 것으로 나타났다.[13]null
페이지 ⅡX174는 '합성에 의한 증명' 접근법을 통해 미발견 유전정보의 부재를 규명하기 위해 사용해 왔다.[14]null
성적 증명서
2020년에는 φX174의 녹취록이 생성되었다.[15]φX174 transcriptome의 눈에 띄는 특징은 최대 4개의 Ro 독립형(내성형) 터미네이터와 1개의 Ro 의존형 터미네이터를 직렬로 하는 최대 4개의 상대적으로 약한 프로모터 시리즈다.null
단백질
φX174는 11개의 단백질을 암호화한다.null
| 단백질 | 사본 | 함수[16] |
|---|---|---|
| A | — | Rolling circle 복제를 시작하기 위한 Nicks RF DNA; 단일 가닥 원형 DNA를 형성하기 위한 선형 페이징 DNA의 끝단 결합 |
| A* | — | 호스트 세포 DNA 복제 억제, 초감염 페이징 차단, 필수적이지 않음 |
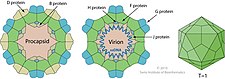
| B | 프로캡시드로60번길 | 프로캡시드 조립체 내부 비계단백질 |
| C | — | DNA포장 |
| D | 프로캡시드로240번길 | 프로캡시드 조립체에 관여하는 외부 비계단백질 |
| E | — | 숙주 세포 용해 |
| F | 처녀자리 60 | 주요캡시드단백질 |
| G | 처녀자리 60 | 주요 스파이크 단백질 |
| H | 처녀자리 12 | DNA 파일럿 단백질(또는 미세 스파이크 단백질) |
| J | 처녀자리 60 | 새로운 단일 가닥의 페이징 DNA에 결합; 페이징 DNA를 프로캡시드로 동반 |
| K | — | 버스트 크기 최적화(필수 없음) |
프로테오메
질량분광법을 이용한 모든 xX174 단백질의 식별이 최근 보고되었다.[13]null
감염 주기
감염은 G단백질이 박테리아 숙주세포 표면의 리포폴리사당체에 결합하면서 시작된다.H단백질(또는 DNA 파일럿 단백질)은 예측된 N단자 투과영역 나선을 통해 E.coli 박테리아의[17] 박테리아막을 통해 바이러스 게놈을 조종한다.[18]그러나 H 단백질이 다기능 단백질이라는 것은 명백해졌다.[19]이것은 coupleX174의 유일한 바이러스 캡시드 단백질로서 몇 가지 이유로 결정 구조가 결여되어 있다.방향족 함량이 낮고 글리신 함량이 높아 단백질 구조가 매우 유연하며, 또한 개별 수소 원자(글리신용 R 그룹)는 단백질 결정학에서 검출하기 어렵다.또한 H단백질은 예측된 N단자 투과선 나선이 박테리아 벽을 통해 쉽게 구멍을 뚫기 때문에 고농도에서 박테리아 숙주의 용해를 유도한다.생물정보학적으로, 이 단백질은 알려진 전사 인자에 대한 유의미한 동질감을 갖는 네 개의 예측 코일 영역을 포함한다.또한, 다른 바이러스성 단백질의 최적 합성을 위해 de novo H 단백질이 필요하다고 판단되었다.[20]바이러스 결합을 막는 H단백질 돌연변이는 내부 비계단백질인 B단백질이 과잉 공급될 때 극복할 수 있다.null
DNA는 5배 정점에 있는 친수 채널을 통해 배출된다.[21]이 지역에는 H단백질이 서식하고 있는 것으로 파악되지만 실험 증거는 정확한 위치를 확인하지 못했다.일단 숙주 박테리아 안으로 들어가면, [+] ssDNA 게놈의 복제는 음의 DNA 중간을 통해 진행된다.이것은 페이징 게놈 슈퍼코일과 그와 같은 슈퍼코일로 형성된 2차 구조가 프리모솜 단백질 복합체를 끌어당기면서 이루어진다.이것은 게놈 주위를 한 번 반투명하고, 양의 원래의 게놈에서 나온 [-]ssDNA를 합성한다.바이러스로 포장하는 [+]ssDNA 게놈은 이것으로부터 롤링 서클 메커니즘에 의해 생성된다.이중 좌초된 슈퍼코일 게놈을 바이러스인코딩 A단백질에 의해 양성 가닥에 흠집을 내고, 세균 DNA 중합효소(DNAP)를 갈라진 부위에 끌어들이는 메커니즘이다.DNAP는 양성 DNA를 만들기 위해 음의 가닥을 템플릿으로 사용한다.그것은 게놈을 중심으로 반투명화되면서 이미 합성된 DNA의 바깥쪽 가닥을 대체하고, SSBP 단백질에 의해 즉시 코팅된다.A 단백질은 원점 배열을 인식할 때마다 완전한 게놈을 분해한다.null
D단백질이 가장 풍부한 유전자 기록물인 만큼 바이러스성 프로캡시드에서 가장 많은 단백질이다.마찬가지로 F, J, G에 대한 유전자 성분은 H에 비해 풍부하다. 이러한 구조 단백질에 대한 스토이치측정법이 5:5:1이기 때문이다.프리모솜은 효소 헬리코아제를 템플릿에 부착/바느질하는 단백질 복합체다.프리모솜은 DNA 합성을 위한 RNA 프라이머를 가닥에 준다.null
유전학 및 다양성
φX174는 다른 마이크로바이러대, 특히 NC 페이징(예: NC1, NC7, NC11, NC16, NC37, NC5, NC41, NC56, NC51 등)과 밀접하게 연관되어 있으며, G4와 유사한 페이징과 더욱 먼 관계가 있으며 α3과 같은 페이지는 더욱 관련이 있다.로키타 외 연구진 2006은 그들의 관계에 대한 계통생성 트리를 제시하였다.[22]null
사용하다
실험진화
φX174는 많은 진화 실험에서 모델 유기체로 사용되어 왔다.[23]null
생명공학
ΦX174 is regularly used as a positive control in DNA sequencing due to its relatively small genome size in comparison to other organisms, its relatively balanced nucleotide content — about 23% G, 22% C, 24% A, and 31% T, i.e., 45% G+C and 55% A+T, see the accession NC_001422.1[9] for its 5,386 nucleotide long sequence.Illumina의 염기서열 분석 기구는 controlX174를 양성 대조군으로 사용하며,[24] Illumina 염기서열 분석 실행 한 번으로 4X174 게놈을 수 백만 번 이상 덮을 수 있어 역사상 가장 고도로 염기서열화된 게놈일 가능성이 높다.[citation needed]null
φX174는 혈액매개성 바이러스에 대한 개인 보호장비의 저항성을 시험하는 데도 사용된다.[25]null
φX174도 바이러스 캡시드G 단백질의 펩타이드 디스플레이(페이지 디스플레이)가 가능하도록 변형했다.[26]null
합성생물학
φX174 게놈은 효모에서 복제된 최초의 페이지로,[8] 게놈 수정을 위한 편리한 드라이독스를 제공한다.[27]φX174는 또한 완전히 분해된 최초의 게놈으로 모든 유전자 중첩을 제거했다.[12]이러한 변화의 영향은 호스트 부착력, 단백질 표현 오류 규제 및 열 민감도를 현저히 감소시키는 결과를 낳았다.[13]null
참고 항목
참조
- ^ a b Sanger F, Air GM, Barrell BG, Brown NL, Coulson AR, Fiddes CA, et al. (February 1977). "Nucleotide sequence of bacteriophage phi X174 DNA". Nature. 265 (5596): 687–95. Bibcode:1977Natur.265..687S. doi:10.1038/265687a0. PMID 870828. S2CID 4206886.
- ^ Fiers W, Sinsheimer RL (October 1962). "The structure of the DNA of bacteriophage phi-X174. III. Ultracentrifugal evidence for a ring structure". Journal of Molecular Biology. 5 (4): 424–34. doi:10.1016/S0022-2836(62)80031-X. PMID 13945085.
- ^ 국립 과학 의학 도서관의 프로필.아서 코른베르크 신문.1959-1970년 "시험관에서의 생명 만들기"연결하다[non-primary source needed]
- ^ Goulian M, Kornberg A, Sinsheimer RL (December 1967). "Enzymatic synthesis of DNA, XXIV. Synthesis of infectious phage phi-X174 DNA". Proceedings of the National Academy of Sciences of the United States of America. 58 (6): 2321–8. Bibcode:1967PNAS...58.2321G. doi:10.1073/pnas.58.6.2321. JSTOR 58720. PMC 223838. PMID 4873588.
- ^ Wickner S, Hurwitz J (October 1974). "Conversion of phiX174 viral DNA to double-stranded form by purified Escherichia coli proteins". Proceedings of the National Academy of Sciences of the United States of America. 71 (10): 4120–4. doi:10.1073/pnas.71.10.4120. PMC 434340. PMID 4610569.
- ^ Smith HO, Hutchison CA, Pfannkoch C, Venter JC (December 2003). "Generating a synthetic genome by whole genome assembly: phiX174 bacteriophage from synthetic oligonucleotides". Proceedings of the National Academy of Sciences of the United States of America. 100 (26): 15440–5. Bibcode:2003PNAS..10015440S. doi:10.1073/pnas.2237126100. JSTOR 3149024. PMC 307586. PMID 14657399.
- ^ Cherwa JE, Organtini LJ, Ashley RE, Hafenstein SL, Fane BA (September 2011). "In VITRO ASSEMBLY of the øX174 procapsid from external scaffolding protein oligomers and early pentameric assembly intermediates". Journal of Molecular Biology. 412 (3): 387–96. doi:10.1016/j.jmb.2011.07.070. PMID 21840317.
- ^ a b c Jaschke PR, Lieberman EK, Rodriguez J, Sierra A, Endy D (December 2012). "A fully decompressed synthetic bacteriophage øX174 genome assembled and archived in yeast". Virology. 434 (2): 278–84. doi:10.1016/j.virol.2012.09.020. PMID 23079106.
- ^ a b c 엔토박테리아 페이지는 phiX174 센츄 라토, 완전한 게놈."전체 게놈: 등록 NC_001422", 국립 생명공학 정보 센터.2016년 1월 30일에 회수됨.
- ^ Baas PD, Liewerink H, van Teeffelen HA, van Mansfeld AD, van Boom JH, Jansz HS (June 1987). "Alteration of the ATG start codon of the A protein of bacteriophage phi X174 into an ATT codon yields a viable phage indicating that A protein is not essential for phi X174 reproduction". FEBS Letters. 218 (1): 119–25. doi:10.1016/0014-5793(87)81030-x. PMID 2954853. S2CID 24174007.
- ^ Hecht A, Glasgow J, Jaschke PR, Bawazer LA, Munson MS, Cochran JR, et al. (April 2017). "Measurements of translation initiation from all 64 codons in E. coli". Nucleic Acids Research. 45 (7): 3615–3626. doi:10.1093/nar/gkx070. PMC 5397182. PMID 28334756.
- ^ a b Wright, Bradley W.; Molloy, Mark P.; Jaschke, Paul R. (5 October 2021). "Overlapping genes in natural and engineered genomes". Nature Reviews Genetics: 1–15. doi:10.1038/s41576-021-00417-w. ISSN 1471-0064. PMC 8490965. PMID 34611352.
- ^ a b c Wright BW, Ruan J, Molloy MP, Jaschke PR (November 2020). "Genome Modularization Reveals Overlapped Gene Topology Is Necessary for Efficient Viral Reproduction". ACS Synthetic Biology. 9 (11): 3079–3090. doi:10.1021/acssynbio.0c00323. PMID 33044064. S2CID 222300240.
- ^ Jaschke PR, Dotson GA, Hung KS, Liu D, Endy D (November 2019). "Definitive demonstration by synthesis of genome annotation completeness". Proceedings of the National Academy of Sciences of the United States of America. 116 (48): 24206–24213. doi:10.1073/pnas.1905990116. PMC 6883844. PMID 31719208.
- ^ Logel DY, Jaschke PR (August 2020). "A high-resolution map of bacteriophage ϕX174 transcription". Virology. 547: 47–56. doi:10.1016/j.virol.2020.05.008. PMID 32560904. S2CID 219459208.
- ^ Fane BA, Brentlinger KL, Burch AD, Chen M, Hafenstein S, Moore E, Novak CR, Uchiyama A (2006). "ɸX174 et al., the Microviridae". In Calender R (ed.). The Bacteriophages (2nd ed.). New York: Oxford Univ. Press. p. 130. ISBN 978-0195148503.
- ^ Jazwinski SM, Lindberg AA, Kornberg A (July 1975). "The lipopolysaccharide receptor for bacteriophage phiX174 and S13". Virology. 66 (1): 268–82. doi:10.1016/0042-6822(75)90197-x. PMID 1094681.
- ^ Tusnády GE, Simon I (September 2001). "The HMMTOP transmembrane topology prediction server". Bioinformatics. 17 (9): 849–50. doi:10.1093/bioinformatics/17.9.849. PMID 11590105.
- ^ Cherwa JE, Young LN, Fane BA (March 2011). "Uncoupling the functions of a multifunctional protein: the isolation of a DNA pilot protein mutant that affects particle morphogenesis". Virology. 411 (1): 9–14. doi:10.1016/j.virol.2010.12.026. PMID 21227478.
- ^ Ruboyianes MV, Chen M, Dubrava MS, Cherwa JE, Fane BA (October 2009). "The expression of N-terminal deletion DNA pilot proteins inhibits the early stages of phiX174 replication". Journal of Virology. 83 (19): 9952–6. doi:10.1128/JVI.01077-09. PMC 2748053. PMID 19640994.
- ^ McKenna R, Xia D, Willingmann P, Ilag LL, Krishnaswamy S, Rossmann MG, et al. (January 1992). "Atomic structure of single-stranded DNA bacteriophage phi X174 and its functional implications". Nature. 355 (6356): 137–43. Bibcode:1992Natur.355..137M. doi:10.1038/355137a0. PMC 4167681. PMID 1370343.
- ^ Rokyta DR, Burch CL, Caudle SB, Wichman HA (February 2006). "Horizontal gene transfer and the evolution of microvirid coliphage genomes". Journal of Bacteriology. 188 (3): 1134–42. doi:10.1128/JB.188.3.1134-1142.2006. PMC 1347346. PMID 16428417.
- ^ Wichman HA, Brown CJ (August 2010). "Experimental evolution of viruses: Microviridae as a model system". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 365 (1552): 2495–501. doi:10.1098/rstb.2010.0053. PMC 2935103. PMID 20643739.
- ^ "Using a PhiX Control for HiSeq® Sequencing Runs". Illumina. Archived from the original on 9 January 2019. Retrieved 8 January 2019.
- ^ "PPE-Info – Standard Details". wwwn.cdc.gov. Retrieved 8 February 2019.
- ^ Christakos KJ, Chapman JA, Fane BA, Campos SK (January 2016). "PhiXing-it, displaying foreign peptides on bacteriophage ΦX174". Virology. 488: 242–8. doi:10.1016/j.virol.2015.11.021. PMC 6191337. PMID 26655242.
- ^ Ando H, Lemire S, Pires DP, Lu TK (September 2015). "Engineering Modular Viral Scaffolds for Targeted Bacterial Population Editing". Cell Systems. 1 (3): 187–196. doi:10.1016/j.cels.2015.08.013. PMC 4785837. PMID 26973885.
외부 링크
- Goodsell D (February 2000). "Bacteriophage phiX174". Molecule of the Month. RCSB-PDB.