실시간 중합효소 연쇄반응
Real-time polymerase chain reaction실시간 중합효소 연쇄반응(실시간 PCR, qPCR)은 중합효소 연쇄반응(PCR)에 기초한 분자생물학의 실험실 기술이다.그것은 기존의 PCR처럼 끝이 아닌 PCR 동안(즉, 실시간) 표적 DNA 분자의 증폭을 모니터링한다.실시간 PCR은 정량적(정량적 실시간 PCR) 및 반정량적(즉, 일정량의 DNA 분자 이상/이하)으로 사용할 수 있다(반정량적 실시간 PCR).
실시간 PCR에서의 PCR 생성물 검출을 위한 두 가지 일반적인 방법은 (1) 임의의 이중사슬 DNA와 상호 작용하는 비특이적 형광염료와 (2) 형광 리포터로 라벨링된 올리고뉴클레오티드로 이루어진 배열 특이적 DNA 프로브이며, (2) 형광 리포터와 보체의 하이브리드화 후에만 검출을 가능하게 한다.ary 시퀀스
MIQE(Minimum Information for Publicative Real-Time PCR Experiments) 가이드라인은 qPCR을 정량적 실시간 PCR에 사용하고 RT-qPCR을 역문자 변환-QPCR에 [1]사용할 것을 제안한다.약어 "RT-PCR"은 일반적으로 실시간 PCR이 아닌 역전사 중합효소 연쇄 반응을 나타내지만, 모든 저자가 이 [2]관례를 따르는 것은 아니다.
배경
모든 유기체의 세포는 유전자 전사(단일 가닥 RNA)의 교체를 통해 유전자 발현을 조절한다.세포에서 발현된 유전자의 양은 샘플에 존재하는 유전자의 RNA 전사물의 복사본 수로 측정될 수 있습니다.소량의 RNA로부터 유전자 발현을 강하게 검출해 정량화하기 위해서는 유전자 전사 증폭이 필요하다.중합효소 연쇄반응(PCR)은 DNA를 증폭하는 일반적인 방법입니다. RNA 기반 PCR의 경우 RNA 샘플은 먼저 역전사 효소와 함께 상보적 DNA(cDNA)로 역전사됩니다.
소량의 DNA를 증폭하기 위해 DNA 템플릿, 적어도 1쌍의 특정 프라이머, 디옥시리보뉴클레오티드 트리포스페이트, 적절한 완충액 및 열안정성 DNA 중합효소를 사용하여 기존 PCR과 동일한 방법론을 사용한다.이 혼합물에 불소포자 마크가 있는 물질을 원하는 파장에서 들뜬 후 불소포자의 형광을 측정하기 위한 센서를 포함한 열사이클러에서 첨가함으로써 하나 이상의 특정 제품에 대해 발생률을 측정할 수 있다.이를 통해 각 PCR 사이클에서 증폭된 제품의 생성 속도를 측정할 수 있습니다.이렇게 생성된 데이터는 컴퓨터 소프트웨어로 분석하여 여러 샘플에서 상대적 유전자 발현(또는 mRNA 복사 번호)을 계산할 수 있습니다.정량적 PCR은 샘플 [3]내 특정 DNA 배열의 존재와 풍부함을 결정하기 위해 샘플 내 DNA의 검출 및 정량화에 적용할 수도 있다.이 측정은 각 증폭 사이클 후에 이루어지며, 이것이 이 방법을 실시간 PCR(즉, 즉시 또는 동시 PCR)이라고 부르는 이유입니다.
정량적 PCR과 DNA 마이크로어레이는 유전자 발현을 연구하는 현대적 방법론이다.mRNA 농도를 측정하기 위해 차등 표시, RNase 보호 분석 및 노던 블롯과 같은 이전 방법이 사용되었습니다.노던 블롯은 샘플에서 mRNA 전사체의 풍부함을 시각화함으로써 유전자의 발현 수준을 추정하기 위해 종종 사용된다.이 방법에서 정제 RNA는 아가로스겔 전기영동에 의해 분리되어 고체 매트릭스(나일론막 등)로 이행되어 관심 유전자에 상보적인 특정 DNA 또는 RNA 프로브에 의해 프로빙된다.이 기술은 유전자 발현을 평가하기 위해 여전히 사용되고 있지만, 상대적으로 많은 양의 RNA를 필요로 하며 mRNA [4]수준의 질적 또는 반정량적 정보만 제공한다.정량 방법의 변화로부터 발생하는 추정 오차는 DNA 무결성, 효소 효율성 및 다른 많은 요인들의 결과일 수 있다.이 때문에, 많은 표준화 시스템(종종 정규화 방법이라고 불린다)이 개발되고 있습니다.일부는 총 유전자 발현을 수량화하기 위해 개발되었지만, 가장 흔한 것은 거의 일정한 발현 수준으로 선택된 정규화 유전자라고 불리는 또 다른 유전자와 관련하여 연구되고 있는 특정 유전자를 수량화하는 것을 목표로 한다.이러한 유전자들은 기본적인 세포 생존과 관련된 기능이 보통 구성적인 유전자 [5][6]발현을 의미하기 때문에 종종 하우스키핑 유전자에서 선택된다.이를 통해 연구자는 관심 유전자의 발현 비율을 선택한 정규화자의 발현으로 나누어 보고할 수 있으며, 따라서 실제로 절대 발현 수준을 알지 않고도 전자를 비교할 수 있다.
가장 일반적으로 사용되는 정규화 유전자는 튜불린, 글리세린알데히드-3-인산탈수소효소, 알부민, 시클로필린, 리보솜 RNA의 [4]분자를 코드하는 유전자이다.
기본 원칙
실시간 PCR은 적어도 1개의 소정 파장의 광빔으로 각 시료를 조명하고 들뜬 형광체가 방출하는 형광을 검출하는 능력을 가진 서멀 사이클러로 이루어진다.또한 열 사이클러는 샘플을 빠르게 가열 및 냉각할 수 있으므로 핵산과 DNA 중합효소의 물리적 화학적 특성을 활용할 수 있습니다.
PCR 프로세스는 일반적으로 25~50회 반복되는 일련의 온도 변화로 구성됩니다.이러한 주기는 일반적으로 약 95°C에서 첫 번째 단계는 핵산의 이중 사슬 분리를 허용하고, 두 번째 단계는 약 50~60°C에서 프라이머와 DNA [7]템플릿의 결합을 허용하며, 세 번째 단계는 68~72°C에서 DNA 중합효소에 의해 수행되는 중합 과정을 촉진한다.배열 단계와 변성 단계 사이의 변화 중에 효소가 DNA 앰플리콘을 복제할 수 있기 때문에 단편들의 크기가 작기 때문에 보통 이 유형의 PCR에서는 마지막 단계가 생략된다.또한 4단계 PCR에서는 비특정 염료 사용 [8]시 프라이머 이합체의 존재로 인해 발생하는 신호를 줄이기 위해 예를 들어 80°C의 온도에서 각 사이클에서 몇 초 동안만 지속되는 단온 단계에서 형광을 측정한다.각 사이클에 사용되는 온도와 타이밍은 DNA 합성에 사용되는 효소, 반응에서 2가 이온 및 디옥시리보뉴클레오티드 트리포스페이트(dNTPs)의 농도 및 프라이머의 [9]결합 온도와 같은 다양한 매개변수에 따라 달라집니다.
화학적 분류
실시간 PCR 기술은 PCR 제품, 특정 또는 비특이적 플루오로크롬 검출에 사용되는 화학작용으로 분류할 수 있다.
비특이적 검출: 리포터로 이중사슬 DNA 결합 염료를 사용한 실시간 PCR
DNA결합염료는 PCR의 모든 이중가닥(ds) DNA에 결합하여 염료의 형광양자수율을 증가시킨다.따라서 PCR 중 DNA 생성물의 증가는 각 사이클에서 측정된 형광 강도의 증가로 이어진다.그러나 SYBR Green과 같은 dsDNA 염료는 비특이 PCR 제품(프라이머 이합체 등)을 포함한 모든 dsDNA PCR 제품에 결합한다.이로 인해 대상 시퀀스의 정확한 모니터링이 방해되거나 방해될 수 있습니다.
dsDNA 염료를 사용하는 실시간 PCR에서는 형광 dsDNA 염료를 첨가하여 평상시와 같이 반응을 준비한다.그런 다음 반응을 실시간 PCR 기기로 실행하고, 각 사이클 후에 검출기를 사용하여 형광 강도를 측정한다. 염료는 dsDNA(즉 PCR 제품)에 결합할 때만 형광을 발생시킨다.이 방법은 증폭을 수행하기 위해 한 쌍의 프라이머만 있으면 된다는 장점이 있어 비용을 낮출 수 있습니다. 다양한 유형의 염료를 사용하여 튜브에서 여러 개의 표적 시퀀스를 모니터링할 수 있습니다.
특이 검출 : 형광 리포터 프로브법
형광 리포터 프로브는 프로브에 상보적인 배열을 포함한 DNA만을 검출하기 때문에 리포터 프로브를 사용하면 특이성이 크게 높아져 다른 dsDNA가 존재하는 경우에도 이 기술을 실행할 수 있다.형광 프로브는 다른 색상의 라벨을 사용하여 멀티플렉스 어세이에서 동일한 튜브에서 여러 대상 시퀀스를 모니터링하는 데 사용할 수 있습니다.형광 리포터 프로브의 특수성은 또한 PCR에서 바람직하지 않은 부산물인 프라이머 이합체에 의한 측정 간섭을 방지합니다.그러나 형광 리포터 프로브는 프라이머 이합체의 억제 효과를 방지하지 않으며, 반응 시 원하는 생성물의 축적을 억제할 수 있습니다.
이 방법은 한쪽 끝에 형광 리포터가 있고 반대쪽 끝에 형광 담금질이 있는 DNA 기반 탐침에 의존합니다.리포터는 담금질에 근접하여 형광검출을 방해하며, Taq 중합효소인 5'~3' 엑소핵산가수분해효소 활성에 의해 프로브가 분해되어 리포터와 담금질되지 않은 형광방출이 가능하여 레이저로 들뜬 후 검출할 수 있다.따라서 PCR 사이클마다 Reporter Probe가 목표로 하는 제품이 증가하면 Probe의 고장과 Reporter의 방출에 따라 형광이 비례적으로 증가한다.
- PCR은 평소와 같이 준비되고(PCR 참조), Reporter Probe가 추가됩니다.
- 반응이 시작되면 PCR의 아닐 단계에서 DNA 타겟에 대한 프로브와 프라이머 모두 아닐한다.
- 프라이머로부터 새로운 DNA 가닥의 중합이 개시되어 중합효소가 프로브에 도달하면 그 5'-3'-엑소핵산가수분해효소가 프로브를 분해하여 형광 리포터를 퀀처로부터 물리적으로 분리하여 형광을 증가시킨다.
- 실시간 PCR 기계에서 형광을 검출하여 측정하고, 제품의 지수 증가에 따른 기하학적 증가를 이용해 각 반응에서의 정량 사이클(Cq)을 결정한다.
융접 온도 분석

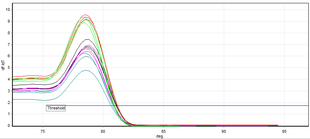
실시간 PCR은 용해 온도 분석(용융 온도에서 T 값이라고도m 함)을 사용하여 증폭된 특정 DNA 단편을 식별할 수 있도록 합니다.사용되는 방법은 보통 이중 가닥 DNA 결합 염료를 리포터로 사용하는 PCR이며 사용되는 염료는 보통 SYBR Green이다.DNA 용융 온도는 증폭된 조각에 고유합니다.이 기술의 결과는 분석된 DNA [11]샘플의 해리 곡선을 비교함으로써 얻어진다.
기존 PCR과 달리 이 방법은 모든 검체의 결과를 보여주기 위해 전기영동 기법을 사용하지 않습니다.이것은 운동 기술임에도 불구하고, 정량적 PCR은 보통 별개의 끝점에서 평가되기 때문이다.따라서 이 기술은 일반적으로 전기영동보다 더 빠른 결과를 제공하며/또는 더 적은 반응물을 사용합니다.후속 전기영동이 필요한 경우 실시간 PCR이 의심스러운 것으로 나타난 샘플을 테스트하거나 특정 결정 인자에 대해 양성으로 테스트된 샘플에 대한 결과를 승인하는 것이 필요합니다.
모델링.
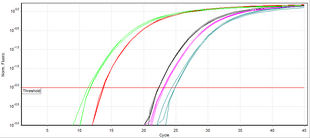
엔드포인트 PCR(기존 PCR)과는 달리 실시간 PCR은 형광을 측정함으로써 증폭 프로세스의 임의의 지점에서 원하는 제품을 감시할 수 있습니다(실시간 프레임에서는 주어진 역치 이상의 레벨로 측정됩니다).일반적으로 사용되는 실시간 PCR에 의한 DNA 정량 방법은 로그 척도의 주기 수에 대한 형광 플롯에 의존합니다.DNA 기반 형광 검출 역치는 신호 노이즈의 표준 편차의 3~5배로 백그라운드보다 높게 설정된다.형광이 임계값을 초과하는 사이클 수를 임계값 사이클(Ct) 또는 MIQE 지침에 따라 정량 사이클(Cq)[1]이라고 합니다.
지수 증폭 단계 동안 타깃 DNA 템플릿(앰프 아이콘)의 양은 사이클마다 두 배로 증가합니다.예를 들어, C가 다른q 검체보다 3주기 앞서 있는 DNA 검체는 2 = 8배 더 많은 템플릿을 포함했다3.그러나 증폭 효율은 프라이머와 템플릿에 따라 변동하는 경우가 많습니다.따라서 프라이머-템플릿 조합의 효율을 DNA 템플릿의 연속 희석을 이용한 적정 실험에서 평가하여 희석할 때마다 (Cq)의 변화의 표준 곡선을 작성한다.그런 다음 선형 회귀의 기울기를 사용하여 증폭의 효율성을 결정합니다. 증폭 효율은 1:2의 희석으로q (C) 차이가 1이 되는 경우 100%입니다.사이클 임계값 방법은 반응 메커니즘에 대한 몇 가지 가정을 하며 데이터 분석 [12]중에 상당한 분산을 초래할 수 있는 증폭 프로파일의 낮은 신호 대 잡음 영역의 데이터에 의존합니다.
유전자 발현을 정량화하기 위해 같은 시료 내의 하우스키핑 유전자에서q RNA/DNA의 (Cq)에서 RNA 또는 DNA의 (C)를 감산하여 서로 다른 시료 간의 RNA량 및 품질의 변동을 정규화한다.이 정규화 절차는 일반적으로 δC-법이라고t[13] 불리며, 다양한 샘플 간에 관심 있는 유전자의 발현을 비교할 수 있습니다.그러나 이러한 비교를 위해 정규화 기준 유전자의 발현은 모든 시료에서 매우 유사해야 한다.따라서 이 기준을 충족하는 기준 유전자를 선택하는 것은 매우 중요하며, 종종 어려운 일입니다. 왜냐하면 오직 극소수의 유전자만이 다양한 조건이나 조직에 [14][15]걸쳐 동일한 수준의 발현을 보이기 때문입니다.주기 임계값 분석이 많은 상용 소프트웨어 시스템과 통합되어 있지만 재현성이 [12]우려되는 경우 고려해야 할 증폭 프로파일 데이터를 분석하는 더 정확하고 신뢰할 수 있는 방법이 있다.
메커니즘 기반의 qPCR 정량 방법도 제안되었으며, 정량화에 표준 곡선이 필요하지 않다는 장점이 있다.MAK2와[16] 같은 방법은 표준 곡선 방법과 같거나 더 나은 정량적 성능을 가진 것으로 나타났습니다.이러한 메커니즘 기반 방법은 중합효소 증폭 과정에 대한 지식을 사용하여 원래 샘플 농도의 추정치를 생성합니다.이 접근방식의 확장은 전체 PCR 반응 프로파일의 정확한 모델을 포함하며,[12] 이를 통해 높은 신호 대 잡음 데이터를 사용할 수 있고 분석 전에 데이터 품질을 검증할 수 있다.
Ruijter [17]등의 연구에 따르면MAK2는 PCR 반응 중에 일정한 증폭 효율을 가정합니다.그러나 MAK2가 파생된 중합효소 연쇄반응의 이론적 분석 결과 증폭효율이 PCR 전체에서 일정하지 않은 것으로 밝혀졌다.MAK2 정량화는 정상적인 qPCR 조건에서 샘플의 표적 DNA 농도를 신뢰할 수 있는 추정치를 제공하지만, MAK2는 qPCR 측정의 표적 농도를 신뢰할 수 있게 정량화하지 않는다.
적용들
실험실에서 정량적 중합효소 연쇄반응을 위한 수많은 응용이 있다.이것은 일반적으로 진단 및 기초 연구에 모두 사용됩니다.식품이나 식물성 물질에 대한 미생물 부하 정량화, 유전자 변형 생물(GMO) 검출, 인간 바이러스 병원체의 정량화 및 유전자형화 등이 산업에서의 기술 활용에 포함된다.
유전자 발현 정량화
전통적인 DNA 검출 방법으로 유전자 발현을 정량화하는 것은 신뢰할 수 없다.노던 블롯 또는 겔 또는 서던 블롯의 PCR 제품에서 mRNA를 검출하는 것은 정확한 [18]정량화를 허용하지 않습니다.예를 들어, 일반적인 PCR의 20-40 사이클 동안, DNA 생성물의 양은 초기 [19]PCR의 표적 DNA의 양과 직접적으로 상관되지 않는 고원에 도달한다.
실시간 PCR은 상대 정량화와 절대 [20]정량화의 두 가지 일반적인 방법으로 핵산을 정량화하는 데 사용될 수 있습니다.절대 정량화는 검정곡선을 이용한 DNA 표준과 비교하여 정확한 대상 DNA 분자의 수를 알려준다.따라서 샘플의 PCR과 표준이 동일한 증폭 [21]효율을 갖는 것이 중요합니다.상대정량은 표적 유전자의 발현에 있어서의 접힘 차이를 결정하기 위해 내부 참조 유전자에 기초한다.정량화는 보완 DNA(cDNA, mRNA의 역전사에 의해 생성된)로 해석되는 mRNA의 발현 수준의 변화로 표현된다.상대정량화는 대조기준유전자량과 비교하기 때문에 검정곡선이 필요 없기 때문에 하기 쉽다.
상대적인 정량화의 결과를 표현하는 데 사용되는 단위는 중요하지 않기 때문에 결과는 여러 RTqPCR에 걸쳐 비교할 수 있다.하나 이상의 하우스키핑 유전자를 사용하는 이유는 사용된 RNA의 양과 품질의 차이와 같은 비특이적 변이를 교정하기 위해서이며, 이는 역전사의 효율성과 따라서 전체 PCR 과정의 효율에 영향을 미칠 수 있다.그러나 이 과정의 가장 중요한 측면은 기준 유전자가 [22]안정적이어야 한다는 것이다.
이러한 기준 유전자의 선택은 전통적으로 RNA 겔의 육안 검사, 노던 블롯 농도 측정 또는 반정량 PCR(PCR 모방)과 같은 질적 또는 반정량적 연구를 사용하여 분자 생물학에서 수행되었다.현재 게놈 시대에는 전사체 [23]기술을 사용하여 많은 생물에 대해 보다 상세한 추정이 가능하다.그러나 mRNA 발현을 정량화하는 데 사용되는 대다수의 기준 유전자의 증폭은 실험 [24][25][26]조건에 따라 다르다는 연구결과가 나왔다.따라서 가장 적합한 기준 유전자를 선택하기 위해서는 통계적으로 건전한 초기 방법론적 연구를 수행할 필요가 있다.
주어진 조건에서 어떤 유전자가 사용하기에 가장 적합한지를 검출할 수 있는 많은 통계 알고리즘이 개발되었습니다.geNORM이나 BestKeeper와 같은 이들은 서로 다른 기준 유전자와 [4][6]조직의 매트릭스에 대해 쌍 또는 기하학적 평균을 비교할 수 있다.
진단 용도
진단 정성 PCR은 예를 들어 전염병, 암, 유전적 이상을 진단하는 핵산을 신속하게 검출하기 위해 적용된다.임상 미생물학 연구소에 정성적 PCR 어세이 도입으로 감염성 [27]질환의 진단이 대폭 개선되었으며, [29][30]진단 테스트에서 신종 플루, [28]코로나 바이러스 등 새롭게 출현하는 질병을 검출하는 도구로 활용되고 있다.
미생물학적 용도
정량 PCR은 식품 안전, 식품 부패 및 발효 분야, 수질(음용수 및 레크리에이션 워터)의 미생물 위험 평가 및 공중 보건 [31]보호 분야에서도 사용됩니다.
또한 qPCR은 환경 [32]샘플에서 채취한 DNA 내 유전자의 분류학적 또는 기능적 마커를 증폭하는 데 사용될 수 있다.마커는 DNA 또는 상보적인 [32]DNA의 유전자 조각으로 나타납니다.특정 유전원소를 증폭함으로써 증폭 [32]전에 시료 중 원소의 양을 정량화할 수 있다.분류학적 마커(리보솜 유전자)와 qPCR을 사용하면 샘플 내 미생물의 양을 결정하는 데 도움이 될 수 있으며,[32] 마커의 특이성에 따라 다른 과, 속 또는 종을 식별할 수 있습니다.기능성 마커(단백질 코드화 유전자)를 사용하는 것은 지역사회 내에서 유전자 발현을 보여줄 수 있으며,[32] 이는 환경에 대한 정보를 드러낼 수 있다.
식물성 병원체 검출
농업은 경제적 손실을 방지하고 건강을 지키기 위해 병원균이 없는 식물 전파나 묘목을 생산하기 위해 끊임없이 노력하고 있다.떡갈나무와 다른 종들을 죽이는 난균류인 피토포라 라모룸의 DNA를 숙주 식물의 DNA와 혼합하여 소량의 DNA를 검출할 수 있는 시스템이 개발되었습니다.병원체와 식물의 DNA 판별은 각 [33]분류군에 특징적인 리보솜 RNA 유전자의 코드 영역에 위치한 스페이서인 ITS 배열의 증폭에 기초한다.이 기술의 필드 기반 버전도 동일한 [34]병원체를 식별하기 위해 개발되었습니다.
유전자 조작 생물 검출
리버스 트랜스크립션(RT-qPCR)을 사용한qPCR은 DNA 검출의 감도 및 동적 범위를 고려하여 GMO 검출에 사용할 수 있습니다.DNA나 단백질 분석과 같은 대안은 보통 덜 민감하다.트랜스유전자가 아닌 프로모터, 터미네이터 또는 벡터를 엔지니어링하는 과정에서 사용되는 중간 배열을 증폭하는 특정 프라이머가 사용됩니다.트랜스제닉 식물을 만드는 과정은 보통 트랜스제닉의 두 개 이상의 복사본 삽입으로 이어지기 때문에 트랜스제닉의 양도 일반적으로 평가된다.이것은 종종 단일 [35][36]복사본으로만 존재하는 처리된 종의 대조 유전자를 사용하여 상대적인 정량화에 의해 수행된다.
임상 정량화 및 유전자형성
바이러스는 직접적인 감염이나 공동 감염으로 인해 사람에게 존재할 수 있으며, 이는 고전적인 기술을 사용하여 진단하기 어렵고 잘못된 예후와 치료를 초래할 수 있습니다.qPCR을 사용하면 B형 간염 [37]바이러스와 같은 바이러스의 정량화와 유전자형성(용융 곡선을 사용하여 수행되는 균주의 특성화)을 모두 수행할 수 있습니다.환자 조직의 단위당 바이러스 게놈 복사본으로 수량화된 감염 정도는 많은 경우에 관련이 있습니다. 예를 들어, 1형 단순 헤르페스 바이러스가 재활성화할 확률은 신경절 [38]내 감염된 뉴런의 수와 관련이 있습니다.HPV(인간 유두종 바이러스)[39]의 경우처럼 바이러스가 인간 게놈에 통합될 경우 발생하는 것과 같이, 역전사를 사용하거나 사용하지 않고 수행됩니다.실시간 PCR은 고형 장기나 골수이식 [40]후 면역억제 환자에게 나타나는 인간 사이토메갈로바이러스(CMV)의 정량화도 가져왔다.
레퍼런스
- ^ a b Bustin SA, Benes V, Garson JA, Hellemans J, Huggett J, Kubista M, Mueller R, Nolan T, Pfaffl MW, Shipley GL, Vandesompele J, Wittwer CT (2009). "The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments". Clinical Chemistry. 55 (4): 611–622. doi:10.1373/clinchem.2008.112797. PMID 19246619.
- ^ Logan, Julie; Edwards, Kirstin & Saunders, Nick, eds. (2009). Real-Time PCR: Current Technology and Applications. Caister Academic Press. ISBN 978-1-904455-39-4.
- ^ Watson, J D; Baker, T A; Bell, S P; Gann, A; Levine, M; Losick, R (2004). Molecular Biology of the Gene (Fifth ed.). San Francisco: Benjamin Cummings. ISBN 978-0-321-22368-5.
- ^ a b c Pfaffl, MW; Tichopad, A; Prgomet, C; Neuvians, TP (March 2004). "Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper--Excel-based tool using pair-wise correlations". Biotechnology Letters. 26 (6): 509–15. doi:10.1023/b:bile.0000019559.84305.47. PMID 15127793. S2CID 977404.
- ^ Pfaffl, MW; Horgan, GW; Dempfle, L (2002). "Relative Expression Software Tool (REST©) for group wise comparison and statistical analysis of relative expression results in real-time PCR". Nucleic Acids Res. 30 (9): e36. doi:10.1093/nar/30.9.e36. PMC 113859. PMID 11972351.
- ^ a b Vandesompele, J; De Preter, K; Pattyn, F; Poppe, B; Van Roy, N; De Paepe, A; Speleman, F (2002). "Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes". Genome Biology. 3 (7): 1–12. doi:10.1186/gb-2002-3-7-research0034. PMC 126239. PMID 12184808.
- ^ Rychlik W, Spencer WJ, Rhoads RE (1990). "Optimization of the annealing temperature for DNA amplification in vitro". Nucleic Acids Res. 18 (21): 6409–6412. doi:10.1093/nar/18.21.6409. PMC 332522. PMID 2243783.
- ^ Pfaffl, Michael (2000). "Development and Validation of an Externally Standardized Quantitative Insulin-like Growth Factor-1 RT-PCR Using LightCycler SYBR Green I Technology" (PDF). Biochemica (3) – via gene-quantification.org.
- ^ Sambrook, Joseph; Russel, David W. (2001). Molecular Cloning: A Laboratory Manual (3rd ed.). Cold Spring Harbor, N.Y.: Cold Spring Harbor Laboratory Press. ISBN 978-0-87969-576-7.
- ^ Ponchel F; Toomes C; Bransfield K; Leong F.T; Douglas S.H; Field S.L; Bell S.M; Combaret V; Puisieux A; Mighell A.J (2003). "Real-time PCR based on SYBR-Green I fluorescence: An alternative to the TaqMan assay for a relative quantification of gene rearrangements, gene amplifications and micro gene deletions". BMC Biotechnol. 3: 18. doi:10.1186/1472-6750-3-18. PMC 270040. PMID 14552656.
- ^ Ririe K.M; Rasmussen R.P; Wittwer C.T. (1997). "Product Differentiation by Analysis of DNA Melting Curves during the Polymerase Chain Reaction" (PDF). Analytical Biochemistry. 245 (2): 154–160. doi:10.1006/abio.1996.9916. PMID 9056205.
- ^ a b c Carr, A. C.; Moore, S. D. (2012). Lucia, Alejandro (ed.). "Robust Quantification of Polymerase Chain Reactions Using Global Fitting". PLOS ONE. 7 (5): e37640. Bibcode:2012PLoSO...737640C. doi:10.1371/journal.pone.0037640. PMC 3365123. PMID 22701526.
- ^ Schefe JH, Lehmann KE, Buschmann IR, Unger T, Funke-Kaiser H (2006). "Quantitative real-time RT-PCR data analysis: current concepts and the novel "gene expression's CT difference" formula". J Mol Med. 84 (11): 901–910. doi:10.1007/s00109-006-0097-6. PMID 16972087. S2CID 10427299.
- ^ Nailis H, Coenye T, Van Nieuwerburgh F, Deforce D, Nelis HJ (2006). "Development and evaluation of different normalization strategies for gene expression studies in Candida albicans biofilms by real-time PCR". BMC Mol. Biol. 7 (1): 25. doi:10.1186/1471-2199-7-25. PMC 1557526. PMID 16889665.
- ^ Nolan T, Hands RE, Bustin SA (2006). "Quantification of mRNA using real-time RT-PCR". Nat. Protoc. 1 (3): 1559–1582. doi:10.1038/nprot.2006.236. PMID 17406449. S2CID 10108148.
- ^ Boggy G, Woolf PJ (2010). Ravasi T (ed.). "A Mechanistic Model of PCR for Accurate Quantification of Quantitative PCR Data". PLOS ONE. 5 (8): e12355. Bibcode:2010PLoSO...512355B. doi:10.1371/journal.pone.0012355. PMC 2930010. PMID 20814578.
- ^ Ruijter JM, Pfaffl MW, Zhao S, Spiess AN, Boggy G, Blom J, Rutledge RG, Sisti D, Lievens A, De Preter K, Derveaux S, Hellemans J, Vandesompele J (2012). "Evaluation of qPCR curve analysis methods for reliable biomarker discovery: bias, resolution, precision, and implications". Methods. 59 (1): 32–46. doi:10.1016/j.ymeth.2012.08.011. PMID 22975077.
- ^ Bruce Gelerter. "PEMF For Treatment Of Corneal Disorders". lemuriatechnologies.com. Archived from the original on 2014-06-09.
- ^ Overbergh, L.; Giulietti, A.; Valckx, D.; Decallonne, R.; Bouillon, R.; Mathieu, C. (2003). "The use of real-time reverse transcriptase PCR for the quantification of cytokine gene expression". Journal of Biomolecular Techniques. 14 (1): 33–43. PMC 2279895. PMID 12901609.
- ^ S. Dhanasekaran; T. Mark Doherty; John Kenneth; TB Trials Study Group (March 2010). "Comparison of different standards for real-time PCR-based absolute quantification". Journal of Immunological Methods. 354 (1–2): 34–39. doi:10.1016/j.jim.2010.01.004. PMID 20109462.
- ^ Bar, Tzachi; Kubista, Mikael; Tichopad, Ales (2011-10-19). "Validation of kinetics similarity in qPCR". Nucleic Acids Research. 40 (4): 1395–1406. doi:10.1093/nar/gkr778. ISSN 0305-1048. PMC 3287174. PMID 22013160.
- ^ Brunner, AM; Yakovlev, IA; Strauss, SH (2004). "Validating internal controls for quantitative plant gene expression studies". BMC Plant Biol. 4: 14. doi:10.1186/1471-2229-4-14. PMC 515301. PMID 15317655. Archived from the original on 2013-08-02.
- ^ McGettigan, Paul A (2013). "Transcriptomics in the RNA-seq era". Current Opinion in Chemical Biology. 17 (1): 4–11. doi:10.1016/j.cbpa.2012.12.008. PMID 23290152.
- ^ Thellin, O; Zorzi, W; Lakaye, B; De Borman, B; Coumans, B; Henne, G; Grisar, T; Igout, A; Heinen, E (1999). "Housekeeping genes as internal standards: use and limits". J Biotechnol. 75 (2–3): 197–200. doi:10.1016/s0168-1656(99)00163-7. PMID 10617337.
- ^ Radonic, A; Thulke, S; Mackay, IM; Landt, O; Siegert, W; Nitsche, A (2004). "Guideline for reference gene selection for quantitative real-time PCR". Biochem Biophys Res Commun. 313 (4): 856–862. doi:10.1016/j.bbrc.2003.11.177. PMID 14706621. Archived from the original on 2013-08-02.
- ^ Dheda, K; Huggett, JF; Bustin, SA; Johnson, MA; Rook, G; Zumla, A (2004). "Validation of housekeeping genes for normalizing RNA expression in real-time PCR". BioTechniques. 37 (1): 112–119. doi:10.2144/04371RR03. PMID 15283208.
- ^ Espy, M.J. (January 2006). "Real-Time PCR in Clinical Microbiology: Applications for Routine Laboratory Testing". Clinical Microbiology Reviews. 19 (3): 165–256. doi:10.1128/CMR.19.1.165-256.2006. PMC 1360278. PMID 16418529.
- ^ Dhamad, AE; Abdal Rhida, MA (2020). "COVID-19: molecular and serological detection methods". PeerJ. 8: e10180. doi:10.7717/peerj.10180. PMC 7547594. PMID 33083156.
- ^ "FDA-cleared RT-PCR Assays and Other Molecular Assays for Influenza Viruses" (PDF). cdc.gov.
- ^ "rRT-PCR, a method to confirm Wuhan coronavirus case – Artificial Intelligence for Chemistry". Retrieved 2020-01-26.
- ^ Filion, M, ed. (2012). Quantitative Real-time PCR in Applied Microbiology. Caister Academic Press. ISBN 978-1-908230-01-0.
- ^ a b c d e Bouchez, Blieux, Dequiedt, Domaizon, Dufresne, Ferreira, Godon, Hellal, Joulian, Quaiser, Martin-Laurent, Mauffret, Monier, Peyret, Schmitt-Koplin, Sibourg, D’oiron, Bispo, Deportes, Grand, Cuny, Maron, Ranjard (September 2016). "Molecular Microbiology Methods For Environmental Diagnosis". Environmental Chemistry Letters. 14 (4): 423–441. doi:10.1007/s10311-016-0581-3. S2CID 88827291. Retrieved 11 May 2020.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Baldwin, B.G. (1992). "Phylogenetic utility of the internal transcribed spacers of nuclear ribosomal DNA in plants: An example from the Compositaogy". Molecular Phylogenetics and Evolution. 1 (1): 3–16. doi:10.1016/1055-7903(92)90030-K. PMID 1342921.
- ^ Tomlinson, J. A.; Barker, I.; Boonham, N. (2007). "Faster, Simpler, More-Specific Methods for Improved Molecular Detection of Phytophthora ramorum in the Field". Applied and Environmental Microbiology. 73 (12): 4040–4047. Bibcode:2007ApEnM..73.4040T. doi:10.1128/AEM.00161-07. PMC 1932743. PMID 17449689.
- ^ Holst-Jensen, Arne; Rønning, Sissel B.; Løvseth, Astrid; Berdal, Knut G. (2003). "PCR technology for screening and quantification of genetically modified organisms (GMOs)". Analytical and Bioanalytical Chemistry. 375 (8): 985–993. doi:10.1007/s00216-003-1767-7. PMID 12733008. S2CID 43681211.
- ^ Brodmann P.D; Ilg E.C; Berthoud H; Herrmann A. (2002). "… -Time Quantitative Polymerase Chain Reaction Methods for Four Genetically Modified Maize Varieties …". Journal of AOAC International. 85 (3): 646–653. doi:10.1093/jaoac/85.3.646. PMID 12083257.
- ^ Yeh S.H. Tsai C.Y. Kao J.H. Liu C.J. Kuo T.J. Lin M.W. Huang W.L. Lu S.F. Jih J. Chen D.S. Others (2004). "Quantification and genotyping of hepatitis B virus in a single reaction by real-time PCR and melting …". Journal of Hepatology. 41 (4): 659–666. doi:10.1016/j.jhep.2004.06.031. PMID 15464248.
- ^ Sawtell N.M. (1998). "The Probability of in Vivo Reactivation of Herpes Simplex Virus Type 1 Increases with the Number of Latently Infected Neurons in the Ganglia". Journal of Virology. 72 (8): 6888–6892. doi:10.1128/JVI.72.8.6888-6892.1998. PMC 109900. PMID 9658140.
- ^ Peter M. Rosty C. Couturier J. Radvanyi F. Teshima H. Sastre-garau X. (2006). "MYC activation associated with the integration of HPV DNA at the MYC locus in genital tumours". Oncogene. 25 (44): 5985–5993. doi:10.1038/sj.onc.1209625. PMID 16682952.
- ^ Mackay, Ian M.; Arden, Katherine E.; Nitsche, Andreas (2002-03-15). "Real-time PCR in virology". Nucleic Acids Research. 30 (6): 1292–1305. doi:10.1093/nar/30.6.1292. ISSN 0305-1048. PMC 101343. PMID 11884626.
참고 문헌
- Elyse; Houde, Alain (2002). "La PCR en temps réel: principes et applications" (PDF). Reviews in Biology and Biotechnology. 2 (2): 2–11. Archived from the original (PDF) on 2009-06-12.
- Bustin, SA (2000). "Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays". J Mol Endocrinol. 25 (2): 169–193. doi:10.1677/jme.0.0250169. PMID 11013345.
- Higuchi, R.; Dollinger, G.; Walsh, P.S.; Griffith, R. (1992). "Simultaneous amplification and detection of specific DNA-sequences". Bio-Technology. 10 (4): 413–417. doi:10.1038/nbt0492-413. PMID 1368485. S2CID 1684150.
- Holland, P.M.; Abramson, R.D.; Watson, R.; Gelfand, D.H. (1991). "Detection of specific polymerase chain reaction product by utilizing the 50 !30 exonuclease activity of Thermus aquaticus DNA polymerase". Proc. Natl. Acad. Sci. USA. 88 (16): 7276–7280. Bibcode:1991PNAS...88.7276H. doi:10.1073/pnas.88.16.7276. JSTOR 2357665. PMC 52277. PMID 1871133.
- Kubista, M; Andrade, JM; Bengtsson, M; Forootan, A; Jonak, J; Lind, K; Sindelka, R; Sjoback, R; Sjogreen, B; Strombom, L; Stahlberg, A; Zoric, N (2006). "The real-time polymerase chain reaction". Mol. Aspects Med. 27 (2–3): 95–125. doi:10.1016/j.mam.2005.12.007. PMID 16460794.
- Higuchi, R.; Fockler, C.; Dollinger, G.; Watson, R. (1993). "Kinetic PCR: Real time monitoring of DNA amplification reactions". Biotechnology. 11 (9): 1026–1030. doi:10.1038/nbt0993-1026. PMID 7764001. S2CID 5714001.
- Filion, M. (2012). Quantitative Real-time PCR in Applied Microbiology. Caister Academic Press. ISBN 978-1-908230-01-0.
- Wawrik, B; Paul, JH; Tabita, FR (2002). "Real-time PCR quantification of rbcL (ribulose-1,5-bisphosphate carboxylase/oxygenase) mRNA in diatoms and pelagophytes". Appl. Environ. Microbiol. 68 (8): 3771–3779. Bibcode:2002ApEnM..68.3771W. doi:10.1128/aem.68.8.3771-3779.2002. PMC 123995. PMID 12147471.
- Logan J; Edwards K; Saunders N, eds. (2009). Real-Time PCR: Current Technology and Applications. Caister Academic Press. ISBN 978-1-904455-39-4.