생합성
Biosynthesis생합성은 기질이 살아있는 유기체에서 보다 복잡한 산물로 변환되는 다단계 효소 촉매 과정이다.생합성에서는 간단한 화합물이 수정되거나 다른 화합물로 전환되거나 결합되어 고분자가 형성된다.이 과정은 종종 대사 경로로 구성됩니다.이러한 생합성 경로 중 일부는 단일 세포 소기관 내에 위치하는 반면, 다른 일부는 여러 세포 소기관 내에 위치한 효소를 포함한다.이러한 생합성 경로의 예는 지질막 성분 및 뉴클레오티드의 생산을 포함한다.생합성은 보통 동화작용과 동의어이다.
생합성을 위한 필수 요소에는 전구체 화합물, 화학 에너지(예: ATP) 및 코엔자임(예: 코엔자임)을 필요로 할 수 있는 촉매 효소(예: 코엔자임)가 포함된다.NADH, NADPH)이러한 원소는 고분자의 구성 요소인 단량체를 생성합니다.몇몇 중요한 생물학적 고분자는 펩타이드 결합을 통해 결합된 아미노산 단량체로 구성된 단백질과 포스포디에스터 결합을 통해 결합된 뉴클레오티드로 구성된 DNA 분자를 포함한다.
화학반응의 특성
생합성은 일련의 화학 반응에 의해 일어난다.이러한 반응이 일어나려면 다음 요소가 필요합니다.[1]
- 전구체 화합물: 이 화합물은 반응의 시작 분자 또는 기질입니다.이것들은 또한 주어진 화학 과정에서의 반응물질로 볼 수 있다.
- 화학 에너지: 화학 에너지는 높은 에너지 분자의 형태로 발견될 수 있습니다.이 분자들은 에너지적으로 좋지 않은 반응에 필요하다.또한 이들 화합물의 가수분해는 반응을 촉진한다.ATP와 같은 고에너지 분자는 3개의 인산염이 있다.종종 말단 인산염은 가수분해 중에 분리되어 다른 분자로 옮겨진다.
- 촉매 효소: 이 분자들은 반응 속도를 높이고 활성화 에너지를 낮춤으로써 반응을 촉매하는 특별한 단백질입니다.
- 코엔자임 또는 보조인자: 보조인자는 화학반응을 돕는 분자입니다.이것은 금속 이온, NADH 및 아세틸 CoA와 같은 비타민 유도체 또는 ATP와 같은 비타민이 아닌 유도체일 수 있습니다.NADH의 경우, 분자는 수소를 전달하고, 아세틸 CoA는 아세틸기를 전달하며, ATP는 인산염을 전달한다.
가장 간단한 의미에서 생합성에서 발생하는 반응은 다음과 같은 형식을 [2]갖는다.
이 기본 방정식의 일부 변형은 나중에 자세히 설명하겠습니다.[3]
- 보통 다단계 반응 경로의 일부로 다른 화합물로 변환되는 단순한 화합물.이러한 유형의 반응의 두 가지 예는 핵산 생성과 번역 전 tRNA 충전 중에 발생합니다.이러한 단계 중 일부에서는 화학 에너지가 필요합니다.
- 보조 인자의 도움을 받아 다른 화합물로 변환되는 단순한 화합물.예를 들어 인지질 합성은 아세틸 CoA를 필요로 하는 반면, 다른 막 성분인 스핑고지질 합성은 스핑고신 골격을 형성하기 위해 NADH와 FADH를 필요로 한다.이 예제의 일반적인 방정식은 다음과 같습니다.
- 고분자를 만들기 위해 결합하는 단순한 화합물들.예를 들어, 지방산은 결합해서 인지질을 형성한다.이어서 인지질과 콜레스테롤이 비공유적으로 상호작용하여 지질 이중층을 형성한다.이 반응은 다음과 같이 나타낼 수 있습니다.
지질
많은 복잡한 고분자들이 단순하고 반복적인 [4]구조의 패턴으로 합성됩니다.예를 들어, 가장 간단한 지질 구조는 지방산이다.지방산은 탄화수소 유도체이며, 카르복실기 "머리"와 탄화수소 사슬 "꼬리"[4]를 포함한다.이러한 지방산은 더 큰 구성 요소를 만들고, 이는 다시 지질 [4]이중층을 형성하기 위해 비공유 상호작용을 통합합니다.지방산 사슬은 막지질의 두 가지 주요 성분인 인지질과 스핑고지질에서 발견됩니다.세 번째 주요 막 성분인 콜레스테롤은 이러한 지방산 [5]단위를 포함하지 않습니다.
인지질
모든 생체막의 기초는 인지질의 [6]이중층 구조로 구성되어 있다.인지질 분자는 양성이다; 그것은 친수성 극성 머리와 소수성 비극성 [4]꼬리를 포함한다.인지질 머리는 서로 및 수성 매체와 상호작용하는 반면 탄화수소 꼬리는 [7]물에서 멀리 떨어진 중심에서 방향을 잡습니다.이러한 후자의 상호작용은 이온과 [8]분자의 장벽으로 작용하는 양층 구조를 구동합니다.
인지질에는 다양한 종류가 있다. 따라서, 그들의 합성 경로는 다르다.그러나 인지질 합성의 첫 번째 단계는 소포체 및 미토콘드리아 [7]외막에서 인산염 또는 디아실글리세롤 3-인산의 형성을 포함한다.합성 경로는 다음과 같습니다.
이 경로는 글리세롤 3-인산으로 시작되며, 글리세롤 3-인산은 아실 코엔자임 [9]A에 의해 제공되는 지방산 사슬의 첨가를 통해 리소포스파티드로 전환된다.다음으로, 리소포스파티드는 제2의 아실 CoA에 의해 기여되는 다른 지방산 사슬의 첨가를 통해 포스파티드로 전환된다.이 모든 단계는 글리세롤 인산 아실전달효소 [9]효소에 의해 촉매된다.인지질 합성은 소포체 내에서 지속되며, 생합성 경로는 [9]특정 인지질 성분에 따라 분리된다.
스핑고지질
인지질처럼, 이 지방산 유도체들은 양극성 머리와 비극성 [5]꼬리를 가지고 있다.인지질과는 달리 스핑고지질은 스핑고신 [10]골격을 가지고 있다.스핑고지질은 진핵세포에 존재하며 특히 중추신경계에 [7]풍부하다.예를 들어, 스핑고미엘린은 [11]신경섬유의 미엘린 칼집의 일부입니다.
스핑고신 골격의 아미노기에 부착된 지방산 사슬로 이루어진 세라마이드로부터 스핑고지질을 형성한다.이 세라마이드는 스핑고신의 [11]아실화로부터 합성된다.스핑고신의 생합성 경로는 다음과 같다.
그림과 같이 스핑고신 합성 중에 팔미토일 CoA와 세린이 축합반응을 일으켜 데히드로스핑고신을 [7]형성한다.이 생성물은 환원되어 디히드로스핑고신을 형성하고,[7] 디히드로스핑고신은 FAD에 의한 산화 반응을 통해 스핑고신으로 변환된다.
콜레스테롤
이 지질은 [5]스테롤이라고 불리는 분자의 종류에 속합니다.스테롤은 4개의 융합 고리와 1개의 수산기를 [5]가지고 있다.콜레스테롤은 특히 중요한 분자입니다.그것은 지질막의 구성요소 역할을 할 뿐만 아니라, 코르티솔, 테스토스테론, 그리고 [12]에스트로겐을 포함한 몇몇 스테로이드 호르몬의 전구체이기도 하다.
콜레스테롤은 아세틸 [12]CoA에서 합성된다.경로는 다음과 같습니다.
보다 일반적으로, 이 합성은 3단계로 이루어지며, 1단계는 세포질, 2단계와 3단계는 소포질에서 [9]일어난다.단계는 다음과 같습니다.[12]
- 1. 콜레스테롤의 '구성요소'인 이소펜테닐피로인산염의 합성
- (이) 인산 이소펜테닐 6분자의 축합에 의한 스쿠알렌의 생성
- (3) 스쿠알렌이 몇 가지 효소 반응을 통해 콜레스테롤로 변환되는 것
뉴클레오티드
뉴클레오티드의 생합성은 기질을 보다 복잡한 [1]산물로 변환하는 효소 촉매 반응을 포함한다.뉴클레오티드는 DNA와 RNA의 구성 요소이다.뉴클레오티드는 RNA의 리보스 당과 DNA의 디옥시리보스 당으로 이루어진 5원환으로 구성되어 있다.이러한 당은 [13]당분의 5' 위치에 글리코시드 결합과 인산기가 있는 퓨린 또는 피리미딘 염기와 연결되어 있다.
퓨린뉴클레오티드
DNA 뉴클레오티드 아데노신과 구아노신은 리보스 당에 글리코시드 결합으로 결합된 퓨린 염기로 구성됩니다.RNA 뉴클레오티드 디옥시아노신 및 디옥시구아노신에서는 푸린 염기가 글리코시드 결합에 의해 디옥시리보스 당에 부착되어 있다.DNA와 RNA 뉴클레오티드의 푸린 염기는 대부분의 단세포 유기체에 존재하는 12단계 반응 메커니즘으로 합성된다.고등 진핵생물들은 10개의 반응 단계에서 유사한 반응 메커니즘을 사용한다.푸린염기는 포스포리보실피로인산(PRPP)을 이노신일인산(IMP)으로 변환함으로써 합성되며, 이는 푸린염기 [14]생합성 최초의 키 중간체이다.IMP의 추가적인 효소적 수식은 뉴클레오티드의 아데노신 및 구아노신 염기를 생성한다.
- 푸린 생합성의 첫 단계는 글루타민-PRPP 아미도전달효소에 의해 수행되는 축합 반응이다.이 효소는 아미노기를 글루타민에서 PRPP로 전달하여 5-포스포리보실아민을 형성합니다.다음 단계는 ATP로부터 인산기를 추가하여 글리신을 활성화해야 한다.
- GAR 합성효소는[15] 활성 글리신을 PRPP에 응축시켜 글리신아미드 리보뉴클레오티드(GAR)를 형성한다.
- GAR 트랜스폼라아제는 GAR의 아미노기에 포밀기를 첨가하여 포밀글리신아미드 리보뉴클레오티드(FGAR)를 형성한다.
- FGAR아미드전달효소는[16] FGAR에 질소기를 첨가하는 것을 촉매하여 포름글리신아미딘 리보뉴클레오티드(FGAM)를 형성한다.
- FGAM 시클라아제는 물 분자의 제거를 수반하는 고리 폐쇄를 촉매하여 5원 이미다졸 고리 5-아미노이미다졸 리보뉴클레오티드(AIR)를 형성한다.
- N5-CAIR 합성효소는 카르복실기를 전달하여 중간 N5-카르복시아미노이미다졸 리보뉴클레오티드(N5-CAIR)[17]를 형성한다.
- N5-CAIR 뮤타아제는 카르복실 관능기를 재배열하여 이미다졸 고리상에 전달하여 카르복시아미노-이미다졸 리보뉴클레오티드(CAIR)를 형성한다.AIR에서 CAIR 형성의 2단계 메커니즘은 대부분 단세포 생물에서 발견된다.고등 진핵생물은 AIR 카르복실화효소를 [18]포함하고 있는데, AIR 카르복실기는 AIR 이미다졸 고리로 직접 전달되어 CAIR를 형성한다.
- SAICAR 합성효소는 아스파르트산과 이미다졸 고리의 부가 카르복실기 사이에 펩타이드 결합을 형성하여 N-수치닐-5-아미노이미다졸-4-카르복사미드 리보뉴클레오티드(SAICAR)를 형성한다.
- SAICAR 리아제는 첨가된 아스파르트산의 탄소 골격을 제거하여 아미노기를 떠나 5-아미노이미다졸-4-카르복사미드 리보뉴클레오티드(AICAR)를 형성한다.
- AICAR 트랜스폼실라아제는 카르보닐기를 AICAR로 전달하여 N-포름아미노이미다졸-4-카르복사미드 리보뉴클레오티드(FAICAR)를 형성한다.
- 마지막 단계는 퓨린 고리 폐쇄를 수행하고 이노신 일인산 [5]중간체를 형성하는 효소 IMP 합성효소를 포함한다.
피리미딘뉴클레오티드
글리코시드 결합을 통해 리보스 당과 연결된 다른 DNA 및 RNA 뉴클레오티드 염기는 티민, 시토신, 우라실이다.우리딘1인산생합성은 미토콘드리아 내막에 위치한 효소와 세포에 [19]위치한 다기능성 효소를 포함한다.
- 첫 번째 단계는 ATP 의존성 반응에서 글루타민과2 CO를 결합하여 카르바모일 인산합성효소이다.
- 아스파르트산카르바모일전달효소는 아스파르트산카르바모일인산과 아스파르트산을 축합하여 우리도숙신산을 형성한다.
- 디히드로오로타아제(Dihydroorotase)는 물을 잃는 반응인 고리 폐쇄를 통해 디히드로오로테이트를 형성합니다.
- 미토콘드리아 내막 [19]내에 위치한 디히드로오로테이트 탈수소효소는 디히드로트로테이트를 산화시켜 오로테이트로 만든다.
- 오로틴산포스포리보실히드로라아제(OMP pyrophosphorylase)는 오로틴산염과 PRPP를 응축하여 오로티딘-5'-인산을 형성한다.
- OMP 탈탄산화효소는 오로티딘-5'-인산의 [20]UMP로의 전환을 촉매한다.
우리딘뉴클레오티드염기를 합성한 후 다른 염기인 시토신 및 티민을 합성한다.시토신 생합성은 UMP에서 UTP로 변환되는 2단계 반응이다.UMP에 대한 인산염 첨가는 키나제 효소에 의해 촉매된다.CTP 합성효소는 다음 반응 단계인 글루타민에서 우리딘으로 아미노기를 전달하여 UTP를 CTP로 변환하는 것을 촉매하며, 이는 CTP의 [21]시토신 염기를 형성한다.UTP + ATP + 글루타민 δ CTP + ADP + 글루탐산염의 반응을 나타내는 메커니즘은 다음과 같습니다.
시토신은 DNA와 RNA에 모두 존재하는 뉴클레오티드이다.그러나 우라실은 RNA에서만 발견된다.따라서 UTP가 합성된 후에는 디옥시 형태로 변환되어 DNA에 통합되어야 한다.이 전환은 리보뉴클레오시드 삼인산 환원효소를 포함한다.디옥시리보스를 생성하기 위해 리보스의 2'-OH를 제거하는 이 반응은 설탕에 부착된 염기의 영향을 받지 않습니다.이러한 비특이성은 리보뉴클레오시드 삼인산 환원효소가 [21]유사한 메커니즘에 의해 모든 뉴클레오티드 삼인산을 디옥시리보뉴클레오티드로 변환할 수 있게 한다.
유라실과 대조적으로, 티민 염기는 RNA가 아닌 DNA에서 주로 발견된다. 세포는 보통 RNA의 리보스 당과 연결된 티민 염기를 포함하지 않기 때문에, 세포는 디옥시리보스와 연결된 티민만을 합성한다는 것을 나타낸다.티미딜레이트 합성효소는 dUMP에서 dTMP로 티민 잔기를 합성하는 역할을 하며, 이 반응은 dUMP의 유라실 염기로 메틸기를 전달하여 dTMP를 [21]생성한다.티미딜산합성효소 반응인 dUMP + 5,10-메틸렌테트라히드로폴레이트 d dTMP + 디히드로폴레이트이다.
DNA
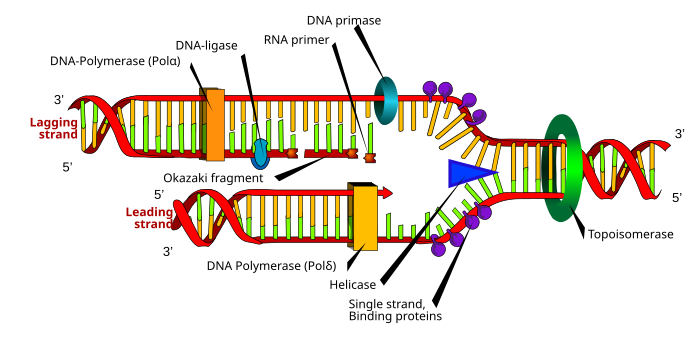
진핵생물과 원핵생물의 DNA 합성에 차이가 있지만, 다음 절은 두 유기체가 공유하는 DNA 복제의 주요 특징을 나타낸다.
DNA는 포스포디에스터 [4]결합에 의해 결합되는 뉴클레오티드로 구성되어 있다.핵에서 일어나는 DNA 합성은 반보전적인 과정으로, 이것은 결과 DNA 분자가 모구조에서 원래의 가닥과 새로운 [22]가닥을 포함하고 있다는 것을 의미한다.DNA 합성은 4개의 디옥시뉴클레오시드 삼인산염, 템플릿 가닥 및 프리 3'을 가진 프라이머를 필요로 하는 DNA 중합효소 계열에 의해 촉매된다.뉴클레오티드를 [23]포함하는 OH.
DNA 복제가 일어나기 위해 복제 포크는 DNA [23]나선을 풀어주는 헬리케이스라고 불리는 효소에 의해 만들어집니다.복제 포크의 토포이소머라아제는 DNA 풀림에 의한 슈퍼코일을 제거하고, 외가닥 DNA 결합 단백질은 복제 전에 [13]안정화된 두 개의 외가닥 DNA 템플릿을 유지한다.
DNA 합성은 RNA 중합효소 프리마아제에 의해 시작되며, 프리마아제는 3'[23]OH의 RNA 프라이머를 만든다.이 프라이머는 단일 가닥 DNA 템플릿에 부착되며 DNA 중합효소는 뉴클레오티드를 포함시킴으로써 사슬을 연장시킨다. DNA 중합효소는 또한 새로 합성된 DNA [23]가닥을 교정한다.
DNA 중합효소에 의해 촉매되는 중합 반응 중에 3'에 의해 친핵성 공격이 발생한다.디옥시뉴클레오시드 삼인산염의 가장 안쪽 인 원자에 있는 성장 사슬의 OH; 이것은 새로운 뉴클레오티드를 부착하고 피로인산을 [9]방출하는 포스포디에스테르 브릿지를 형성합니다.
리플리케이션 중에 연속적으로 합성되어 리플리케이션 포크를 향해 성장하는 선두 스트랜드와 오카자키 단편으로 불연속적으로 만들어져 리플리케이션 [22]포크와 멀어지는 후행 스트랜드 두 종류가 동시에 작성된다.오카자키 단편을 DNA 리가아제에 의해 공유 결합해 연속 [22]가닥을 형성한다.그 후 DNA 복제를 완료하기 위해 RNA 프라이머를 제거하고 DNA로 치환하여 DNA 연결효소를 [22]통해 결합한다.
아미노산
단백질은 펩타이드 결합에 의해 연결된 아미노산으로 구성된 중합체이다.자연에서 발견된 300개 이상의 아미노산이 있으며, 그 중 표준 아미노산으로 알려진 20개만이 [24]단백질의 구성 요소이다.오직 녹색 식물과 대부분의 미생물만이 모든 살아있는 종들이 필요로 하는 20개의 표준 아미노산을 모두 합성할 수 있다.포유류는 표준 아미노산 20개 중 10개만 합성할 수 있다.다른 아미노산인 발린, 메티오닌, 류신, 이소류신, 페닐알라닌, 리신, 트레오닌, 트립토판, 아기용 아르기닌은 [25]식이요법으로 얻을 수 있다.
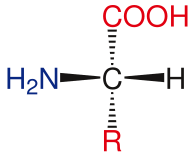
아미노산 염기성 구조
표준 아미노산의 일반적인 구조는 1차 아미노기, 카르복실기 및 α-탄소에 부착된 관능기를 포함한다.다른 아미노산은 관능기에 의해 식별된다.α-탄소에 붙어 있는 3개의 서로 다른 군으로 인해 아미노산은 비대칭 분자이다.글리신을 제외한 모든 표준 아미노산에 대해 α-탄소는 키랄 중심이다.글리신의 경우 α-탄소에는 수소 원자가 2개 있어 대칭성을 더한다.프롤린을 제외한 모든 아미노산은 L-아이소폼 구조를 가지고 있다.프롤린은 아미노기와 [24]고리를 형성하는 α-탄소 위에 관능기를 가지고 있다.
질소원
아미노산 생합성의 주요 단계 중 하나는 α-탄소 위에 질소기를 통합하는 것이다.세포에는 질소 그룹을 통합하는 두 가지 주요 경로가 있습니다.하나의 경로는 글루타민 옥소글루타르산 아미노기를 제거하고 그것을 2-옥소글루타르산염으로 전달하여 두 개의 글루탐산 분자를 생성하는 효소 GOGAT를 포함한다.이 촉매 반응에서 글루타민은 질소원으로 작용한다.이 반응을 나타내는 이미지는 오른쪽에 있습니다.
아미노산의 α-탄소에 질소를 결합하는 다른 경로는 글루탐산탈수소효소(GDH)를 포함한다.GDH는 암모니아를 2-옥소글루타르산염으로 전달하여 글루탐산을 형성할 수 있다.또한 글루타민 합성효소(GS)는 글루탐산염에 암모니아를 전달하여 글루타민을 합성하여 글루타민을 [26]보충할 수 있다.
글루탐산 아미노산 패밀리
글루타메이트 계열의 아미노산은 아미노산 글루타메이트에서 유래한 아미노산을 포함한다.이 과는 글루탐산염, 글루타민, 프롤린 및 아르기닌을 포함한다.이 과는 또한 α-케토글루타르산으로부터 파생된 아미노산 [27]리신을 포함한다.
글루타메이트와 글루타민의 생합성은 위에서 논의한 질소 동화의 핵심 단계이다.GOGAT와 GDH 효소는 질소 동화 반응을 촉매한다.
박테리아에서 글루탐산 5-키나아제 효소는 ATP에서 글루탐산염으로 인산기를 전달함으로써 프롤린의 생합성을 개시한다.다음 반응은 L-글루탐산 5-인산의 δ-카르복실기 환원을 촉매하는 효소인 파이로린-5-카르복실산합성효소(P5CS)에 의해 촉매된다.이로 인해 글루탐산세미알데히드가 생성되고, 이 세미알데히드는 자발적으로 피롤린-5-카르본산염으로 순환한다.또한 피롤린-5-카르본산염 환원효소(P5CR)에 의해 피롤린-5-카르본산염이 환원되어 프롤린 아미노산이 [28]된다.
세균 내 아르기닌 생합성 1단계에서는 아세틸기를 아세틸-CoA에서 N-α 위치로 전달함으로써 글루타메이트를 아세틸화함으로써 자발적인 환화를 방지한다.N-아세틸글루탐산합성효소(글루탐산N-아세틸전달효소)는 아세틸화 단계를 촉매화하는 역할을 한다.후속 공정은 N-아세틸글루탐산인산화효소, N-아세틸감마-글루타밀-인산환원효소 및 아세틸오르니틴/수치닐디아미노피멜산아미노전달효소에 의해 촉매되어 N-아세틸-L-오르니틴을 생성한다.아세틸오르니틴의 아세틸기는 아세틸오르니티나아제(AO) 또는 오르니틴아세틸전달효소(OAT)에 의해 제거되고 오르니틴을 생성한다.그 후 시트룰린과 아르기노숙신산염 효소는 오르니틴을 아르기닌으로 [29]변환한다.
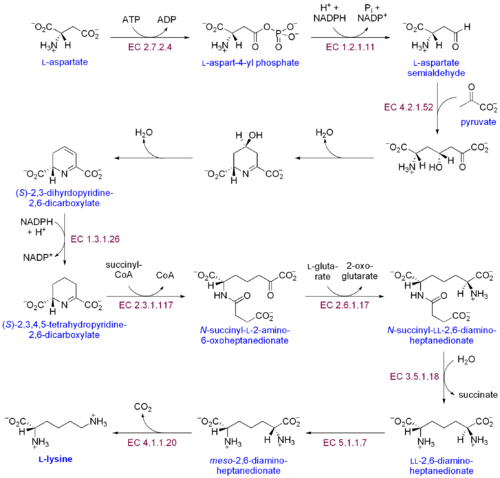
디아미노피멜산 경로와 α-아미노아디페이트 경로의 두 가지 뚜렷한 리신 생합성 경로가 있습니다.두 가지 합성 경로 중 가장 일반적인 경로는 디아미노피멜산 경로이며, 아스파르트산염에 탄소기를 [30]추가하여 리신을 생성하는 몇 가지 효소 반응으로 구성됩니다.
- 아스파르트산인산화효소는 아스파르트산을 인산화하여 인산아스파틸을 생성함으로써 디아미노피멜산 경로를 개시한다.
- 아스파르트산세미알데히드탈수소효소는 인산아스파르트산의 NADPH의존성 환원을 촉매하여 아스파르트산세미알데히드를 생성한다.
- β-아스파틸-4-세미알데히드에 피루브산기를 첨가하여 물분자를 제거한다.이는 환화를 일으키고 (2S,4S)-4-히드록시-2,3,4,5-테트라히드로디피콜리네이트를 일으킨다.
- 4-히드록시-테트라히드로디피콜린산 환원효소는 NADPH에 의해 (2S,4S)-4-히드록시-2,3,4,5-테트라히드로디피콜린산염의 환원을 촉매하여 δ'-피페리딘-2,6-디카르복실레이트(2,3,4,5-테트라히드로디카르복시콜린산염2)를 생성한다.
- 테트라히드로디피콜린산아실전달효소(Tetrahydrodipicolinate acyl transferase)는 고리개방을 초래하는 아세틸화 반응을 촉매하여 N-아세틸α-아미노-γ-케토피멜레이트를 생성한다.
- N-수시닐α-아미노-γ-케토피멜레이트-글루타미노트랜스아미노-글루타미나아제 N-아세틸α-아미노-γ-케토피멜레이트의 케토기를 제거하는 아미노기를 촉매하여 N-수시닐-L-디아미노필레이트를 [31]생성한다.
- N-아실디아미노필레이트탈아실라아제는 N-수치닐-L-디아미노필레이트의 탈아실화를 촉매하여 L,L-디아미노필레이트를 [32]생성한다.
- DAP 에피머라아제는 L,L-디아미노피멜레이트를 L,L-디아미노피멜레이트의 [33]메소 형태로 변환하는 것을 촉매한다.
- DAP 탈탄산화효소는 카르복실기의 제거를 촉매하여 L-리신을 생성한다.
아미노산의 세린족
아미노산의 세린족은 세린, 시스테인 및 글리신을 포함한다.대부분의 미생물 및 식물은 아미노산 시스테인으로부터 메티오닌을 합성하기 위한 황을 얻는다.또한 세린의 글리신으로의 변환은 메티오닌과 히스티딘의 [27]생합성에 필요한 탄소를 제공한다.
세린생합성시 [34]포스포글리세린산탈수소효소는 3-포스포-D-글리세린산을 산화시키는 초기반응을 촉매하여 3-포스포녹시피루브산을 [35]생성한다.다음 반응은 글루탐산 아미노기를 3-포스포노옥시피루브산으로 전달하여 L-포스포세린을 [36]생성하는 포스포세린 아미노전달효소에 의해 촉매된다.마지막 단계는 포스포세린 포스파타아제에 의해 촉매되며, 포스포세린은 L-포스포세린을 탈인산하여 L-세린을 [37]생성한다.
글리신의 생합성을 위한 두 가지 알려진 경로가 있다.주요 탄소원으로 에탄올과 아세테이트를 사용하는 유기체는 글리신 생성 경로를 이용하여 글리신을 합성한다.글리신 생합성의 다른 경로는 해당과정 경로로 알려져 있다.이 경로는 해당과정의 중간체로부터 합성된 세린을 글리신으로 변환한다.세린히드록시메틸전달효소는 세린의 분해를 촉매하여 글리신을 생성하고, 분해된 세린의 탄소기를 테트라히드로폴산염에 전사하여 5,10-메틸렌테트라히드로폴산을 [38]형성한다.
시스테인 생합성은 무기황의 혼합을 수반하는 2단계 반응이다.미생물 및 식물에서 세린아세틸전달효소는 아세틸기가 아세틸-CoA에서 L-세린으로의 전달을 촉매하여 O-아세틸-L-세린을 [39]생성한다.O-아세틸세린(티올) 분해효소에 의해 촉매되는 다음 반응 단계는 O-아세틸-L-세린의 아세틸기를 황화물로 대체하여 시스테인을 [40]생성한다.
아스파르트산 아미노산 패밀리
아스파르트산군은 트레오닌, 리신, 메티오닌, 이소류신 및 아스파르트산을 포함한다.리신과 이소류신은 비록 그들의 탄소 골격의 일부가 피루브산염으로부터 파생되었지만 아스파르트산염 계열의 일부로 간주된다.메티오닌의 경우 메틸카본은 세린과 황기로부터 유도되지만 대부분의 생물에서는 [27]시스테인으로부터 유도된다.
아스파르트산염의 생합성은 단일 효소에 의해 촉매되는 1단계 반응이다.아스파르트산 아미노전달효소는 아스파르트산염에서 α-케토글루타르산염으로의 아미노기 전달을 촉매하여 글루탐산염과 옥살아세트산을 [41]생성한다.아스파라긴은 아스파라긴에 대한 아미노기의 ATP 의존적 첨가에 의해 합성되며, 아스파라긴 합성효소는 글루타민 또는 가용성 암모니아로부터 아스파라긴에 질소의 첨가를 [42]촉매하여 아스파라긴을 생성한다.
리신의 디아미노피멜산 생합성 경로는 아스파르트산 아미노산에 속한다.이 경로는 아스파르트산을 [43]리신으로 변환하는 9가지 효소 촉매 반응을 포함한다.
- 아스파르트산인산화효소는 ATP에서 아스파르트산의 [44]카르본산기로 포스포릴을 전달함으로써 디아미노피멜산 경로의 초기 단계를 촉매한다.
- 아스파르트산-세미알데히드탈수소효소는 아스파르트산β-인산의 탈인산화로 환원반응을 촉매하여 아스파르트산β-세미알데히드를 [45]생성한다.
- 디히드로디피콜린산합성효소는 아스파르트산β-세미알데히드와 피루브산의 축합반응을 촉매하여 디히드로디피콜린산을 [46]생성한다.
- 4-히드록시-테트라히드로디피콜린산 환원효소는 디히드로디피콜린산의 환원을 촉매하여 테트라히드로디피콜린산을 [47]생성한다.
- 숙시닐기를 숙시닐-CoA에서 테트라히드로디피콜린산으로 전이시키는 것을 촉매하여 N-숙시닐-L-2,6-디아미노헵탄디오에이트를 [48]얻는다.
- N-수키닐디아미노필레이트아미노전달효소(N-sucinyldiaminopimelate aminotransferase)는 아미노기를 글루탐산으로부터 N-수키닐-L-2,6-디아미노헵탄디오산으로의 전달을 촉매하여 N-수키닐-L, L-디아미노필산을 [49]생성한다.
- 숙시닐-디아미노피멜레이트 데수치닐라아제는 N-수시닐-L,L-디아미노피멜산으로부터의 아실기 제거를 촉매하여 L,L-디아미노피멜산을 [50]생성한다.
- 디아미노피멜레이트 에피머라아제는 L,L-디아미노피멜산의 α-탄소 반전을 촉매하여 메소디아미노피멜산을 [51]생성한다.
- 시아미노피멜산탈탄산화효소는 리신생합성 마지막 단계를 촉매하여 메소디아미노피멜산으로부터 이산화탄소기를 제거하여 L-리신을 [52]생성한다.
단백질
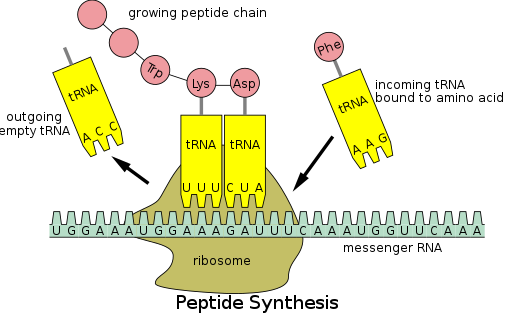
단백질 합성은 [53]번역이라고 불리는 과정을 통해 일어난다.번역하는 동안, mRNA라고 불리는 유전 물질은 단백질 폴리펩타이드 [53]사슬을 생성하기 위해 리보솜에 의해 읽힌다.이 과정은 한쪽 끝에서 아미노산을 결합하고 다른 한쪽 끝에서 mRNA와 상호작용함으로써 어댑터 역할을 하는 전달 RNA(tRNA)를 필요로 한다. tRNA와 mRNA 사이의 후자 쌍은 올바른 아미노산이 [53]사슬에 추가되도록 보장한다.단백질 합성은 시작, 신장, [13]종료의 세 단계로 이루어진다.원핵생물(아카에알과 박테리아) 번역은 진핵생물 번역과 다르다. 그러나 이 절에서는 대부분 두 생물 사이의 공통점에 초점을 맞출 것이다.
기타 배경
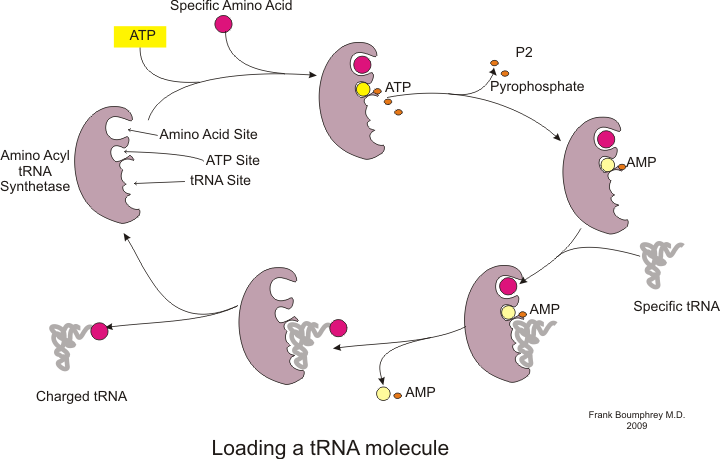
번역을 시작하기 전에 특정 아미노산을 해당 tRNA에 결합하는 과정이 일어나야 합니다.tRNA 충전이라고 불리는 이 반응은 아미노아실 tRNA 합성효소에 [54]의해 촉매된다.특정 tRNA 합성효소는 특정 아미노산을 [54]인식하고 충전하는 역할을 한다.또한 tRNA와 그 동족 아미노산과의 [54]올바른 결합을 확보하기 위한 특별한 식별자 영역을 가진다.아미노산을 해당 tRNA에 결합하는 첫 번째 단계는 아미노아실-AMP의 [54]생성이다.
이어서 아미노아실-AMP에서 tRNA 분자로 아미노아실기가 전달된다.생성된 분자는 아미노아실-tRNA입니다.[54]
아미노아실 tRNA 합성효소에 의해 촉매되는 이 두 단계의 조합은 성장하는 폴리펩타이드 사슬에 아미노산을 첨가할 준비가 된 대전된 tRNA를 생성한다.
아미노산과 결합하는 것 외에도, tRNA는 코돈이라고 불리는 mRNA에 특정 뉴클레오티드 세쌍과 염기쌍을 이루는 안티코돈이라고 불리는 세 개의 뉴클레오티드 단위를 가지고 있습니다; 코돈은 특정 아미노산을 [55]암호화합니다.이러한 상호작용은 단백질 합성의 장소 역할을 하는 리보솜 덕분에 가능하다.리보솜은 아미노아실 부위(A 부위), 펩티딜 부위(P 부위), 출구 부위(E 부위)[56]의 3가지 tRNA 결합 부위를 가지고 있다.
mRNA 전사체에는 수많은 코돈이 있으며 아미노산이 하나 이상의 코돈에 의해 특정되는 것은 매우 흔하다. 이러한 현상을 [57]퇴행성이라고 한다.20개의 아미노산 중 하나에 대해 각 코드 61개씩 총 64개의 코돈이 있으며, 나머지 코돈은 사슬 [57]종단을 규정한다.
단계별 번역
앞에서 설명한 바와 같이 변환은 개시, 신장, 종료의 3단계로 이루어집니다.
순서 1: 시작
개시 단계의 완료는, 다음의 3개의 [13]이벤트에 의해서 결정됩니다.
1. mRNA로의 리보솜 채용
(2) 하전 개시제 tRNA가 리보솜의 P 부위에 결합하는 것
3. 리보솜과 mRNA의 시작 코돈의 적절한 정렬
2단계: 연장
개시 후, 폴리펩타이드 사슬은 안티코돈:코돈 상호작용을 통해 연장되며, 리보솜은 폴리펩타이드 사슬에 아미노산을 한 번에 하나씩 첨가한다.아미노산을 [58]올바르게 첨가하려면 다음 단계를 수행해야 합니다.
(1) 리보솜의 A 부위에 대한 올바른 tRNA의 결합
(2) A부위의 tRNA와 P부위의 tRNA에 부착된 폴리펩타이드 사슬의 펩타이드 결합 형성
(3) tRNA-mRNA 복합체의 3뉴클레오티드에 의한 전위 또는 진행
전위는 E 부위에서 tRNA를 "차단"하고 TRNA를 A 부위에서 P 부위로 이동시켜 들어오는 tRNA가 다른 아미노산을 추가할 수 있도록 A 부위가 자유롭게 남습니다.
순서 3: 종료
변환의 마지막 단계는 정지 코돈이 A [1]사이트에 들어갈 때 발생합니다.그 후, 다음의 순서가 실행됩니다.
(1) P부위에 있는[1] tRNA에서 폴리펩타이드 사슬을 가수분해시키는 방출인자에 의한 코돈 인식
(2) 폴리펩타이드[57] 사슬의 방출
3. 리보솜의 해리와 향후 번역[57] 프로세스에서의 '재활용'
번역의 주요 담당자를 정리한 표는 다음과 같습니다.
| 번역의 주요 담당자 | 번역 단계 | 목적 |
|---|---|---|
| tRNA합성효소 | 개시전에 | tRNA 충전 담당 |
| mRNA | 개시, 신장, 종료 | 단백질 합성을 위한 템플릿. 아미노산을 코드하는 코돈이라는 이름의 영역을 포함한다. |
| tRNA | 개시, 신장, 종료 | 리보솜 부위 A, P, E; 올바른 아미노산이 성장하는 폴리펩타이드 사슬에 통합되도록 보장하기 위해 mRNA 코돈과 항코돈 염기쌍을 결합한다. |
| 리보솜 | 개시, 신장, 종료 | 단백질 합성을 지시하고 펩타이드 결합 형성을 촉매한다. |
고분자 결핍과 관련된 질병
생합성 경로의 오류는 고분자의 기형이나 기능성 분자의 저생산 등 유해한 결과를 초래할 수 있다.다음은 이러한 비효율성으로 인해 발생하는 중단을 보여주는 예입니다.
- 가족성 고콜레스테롤혈증: 이 장애는 LDL에 [59]대한 기능적 수용체의 부재로 특징지어진다. LDL 수용체의 형성이 부족하면 LDL의 간 및 다른 [59]세포로의 진입을 저해하는 잘못된 수용체를 야기할 수 있다.이것은 혈장에서 LDL의 축적을 유발하며 동맥을 좁히고 심장마비의 [59]위험을 증가시키는 아테롬성 동맥경화증을 일으킨다.
- Lesch-Nyhan 증후군: 이 유전병은 자해, 정신 결핍, [60]통풍으로 특징지어진다.푸린뉴클레오티드 [60]형성에 필요한 효소인 하이포산틴-구아닌 포스포리보실전달효소 부재로 인해 발생한다.효소의 결핍은 필요한 뉴클레오티드의 수준을 감소시키고 생합성 중간체의 축적을 유발하며, 이는 앞서 언급한 특이한 [60]행동을 야기한다.
- 중증복합면역결핍증(SCID):SCID는 T세포의 [61]손실을 특징으로 한다.이러한 면역 체계 구성 요소의 부족은 영향을 받는 사람들이 면역학적 [61]기억을 발달시킬 수 없기 때문에 감염원에 대한 민감성을 증가시킨다.이 면역학적 장애는 dATP의 축적을 유발하는 아데노신 디니마아제 활성의 결핍에서 비롯된다.이러한 dATP 분자는 DNA [61]합성을 방해하는 리보뉴클레오티드 환원효소를 억제한다.
- 헌팅턴병: 이 신경학적 질환은 DNA [62]합성 중에 발생하는 오류로 인해 발생합니다.이러한 오류 또는 돌연변이는 돌연변이 헌팅틴 단백질의 발현으로 이어지며,[62] 이 단백질은 유전자에서 CAG 트리뉴클레오티드 반복을 확장함으로써 코드화된 반복 글루타민 잔류물을 포함한다.헌팅턴병은 신경손실과 신경교화증을 특징으로 한다.그 병의 증상에는 운동 장애, 인지력 저하, 행동 [63]장애 등이 포함된다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b c d Alberts, Bruce (2008). Molecular Biology of the Cell (5th ed.). New York: Garland Science. ISBN 978-0815341055.
- ^ Zumdahl, Steven S. Zumdahl, Susan A. (2008). Chemistry (8th ed.). CA: Cengage Learning. ISBN 978-0547125329.
- ^ Voet, Donald; Voet, Judith G.; Pratt, Charlotte W. (2013). Fundamentals of Biochemistry: Life at the Molecular Level (4th ed.). Hoboken, NJ: Wiley. ISBN 978-0470547847.
- ^ a b c d e Lodish, Harvey; et al. (2007). Molecular cell biology (6th ed.). New York: W.H. Freeman. ISBN 978-0716743668.
- ^ a b c d e Cox, David L. Nelson, Michael M. (2008). Lehninger principles of biochemistry (5th ed.). New York: W.H. Freeman. ISBN 9780716771081.
- ^ Hanin, Israel (2013). Phospholipids: Biochemical, Pharmaceutical, and Analytical Considerations. Springer. ISBN 978-1475713664.
- ^ a b c d e Vance, Dennis E.; Vance, Jean E. (2008). Biochemistry of lipids, lipoproteins and membranes (5th ed.). Amsterdam: Elsevier. ISBN 978-0444532190.
- ^ Katsaras, J.; et al. (2001). Lipid bilayers : structure and interactions ; with 6 tables. Berlin [u.a.]: Springer. ISBN 978-3540675556.
- ^ a b c d e Stryer, Jeremy M. Berg; John L. Tymoczko; Lubert (2007). Biochemistry (6. ed., 3. print. ed.). New York: Freeman. ISBN 978-0716787242.
- ^ Gault, CR; LM Obeid; YA Hannun (2010). An Overview of sphingolipid metabolism: from synthesis to breakdown. Adv Exp Med Biol. Advances in Experimental Medicine and Biology. Vol. 688. pp. 1–23. doi:10.1007/978-1-4419-6741-1_1. ISBN 978-1-4419-6740-4. PMC 3069696. PMID 20919643.
- ^ a b Siegel, George J. (1999). Basic neurochemistry : molecular, cellular and medical aspects (6. ed.). Philadelphia, Pa. [u.a.]: Lippincott Williams & Wilkins. ISBN 978-0397518203.
- ^ a b c Harris, J. Robin (2010). Cholesterol binding and cholesterol transport proteins : structure and function in health and disease. Dordrecht: Springer. ISBN 978-9048186211.
- ^ a b c d Watson, James D.; et al. (2007). Molecular biology of the gene (6th ed.). San Francisco, Calif.: Benjamin Cummings. ISBN 978-0805395921.
- ^ Kappock, TJ; Ealick, SE; Stubbe, J (October 2000). "Modular evolution of the purine biosynthetic pathway". Current Opinion in Chemical Biology. 4 (5): 567–72. doi:10.1016/s1367-5931(00)00133-2. PMID 11006546.
- ^ Sampei, G; Baba, S; Kanagawa, M; Yanai, H; Ishii, T; Kawai, H; Fukai, Y; Ebihara, A; Nakagawa, N; Kawai, G (October 2010). "Crystal structures of glycinamide ribonucleotide synthetase, PurD, from thermophilic eubacteria". Journal of Biochemistry. 148 (4): 429–38. doi:10.1093/jb/mvq088. PMID 20716513.
- ^ Hoskins, AA; Anand, R; Ealick, SE; Stubbe, J (Aug 17, 2004). "The formylglycinamide ribonucleotide amidotransferase complex from Bacillus subtilis: metabolite-mediated complex formation". Biochemistry. 43 (32): 10314–27. doi:10.1021/bi049127h. PMID 15301530.
- ^ Mueller, EJ; Meyer, E; Rudolph, J; Davisson, VJ; Stubbe, J (Mar 1, 1994). "N5-carboxyaminoimidazole ribonucleotide: evidence for a new intermediate and two new enzymatic activities in the de novo purine biosynthetic pathway of Escherichia coli". Biochemistry. 33 (8): 2269–78. doi:10.1021/bi00174a038. PMID 8117684.
- ^ Firestine, SM; Poon, SW; Mueller, EJ; Stubbe, J; Davisson, VJ (Oct 4, 1994). "Reactions catalyzed by 5-aminoimidazole ribonucleotide carboxylases from Escherichia coli and Gallus gallus: a case for divergent catalytic mechanisms". Biochemistry. 33 (39): 11927–34. doi:10.1021/bi00205a031. PMID 7918411.
- ^ a b Srere, PA (1987). "Complexes of sequential metabolic enzymes". Annual Review of Biochemistry. 56 (1): 89–124. doi:10.1146/annurev.bi.56.070187.000513. PMID 2441660.
- ^ Broach, edited by Jeffrey N. Strathern, Elizabeth W. Jones, James R. (1981). The Molecular biology of the yeast Saccharomyces. Cold Spring Harbor, N.Y.: Cold Spring Harbor Laboratory. ISBN 978-0879691394.
{{cite book}}:first=범용명(도움말)이 있습니다. - ^ a b c O'Donovan, GA; Neuhard, J (September 1970). "Pyrimidine metabolism in microorganisms". Bacteriological Reviews. 34 (3): 278–343. doi:10.1128/MMBR.34.3.278-343.1970. PMC 378357. PMID 4919542.
- ^ a b c d Geer, Gerald Karp ; responsible for the revision of chapter 15 Peter van der (2004). Cell and molecular biology : concepts and experiments (4th ed., Wiley International ed.). New York: J. Wiley & Sons. ISBN 978-0471656654.
- ^ a b c d Griffiths, Anthony J. F. (1999). Modern genetic analysis (2. print. ed.). New York: Freeman. ISBN 978-0716731184.
- ^ a b Wu, G (May 2009). "Amino acids: metabolism, functions, and nutrition". Amino Acids. 37 (1): 1–17. doi:10.1007/s00726-009-0269-0. PMID 19301095. S2CID 1870305.
- ^ Mousdale, D. M.; Coggins, J. R. (1991). Amino Acid Synthesis. Target Sites for Herbicide Action. pp. 29–56. doi:10.1007/978-1-4899-2433-9_2. ISBN 978-1-4899-2435-3.
- ^ Miflin, B. J.; Lea, P. J. (1977). "Amino Acid Metabolism". Annual Review of Plant Physiology. 28: 299–329. doi:10.1146/annurev.pp.28.060177.001503.
- ^ a b c Umbarger, HE (1978). "Amino acid biosynthesis and its regulation". Annual Review of Biochemistry. 47 (1): 532–606. doi:10.1146/annurev.bi.47.070178.002533. PMID 354503.
- ^ Pérez-Arellano, I; Carmona-Alvarez, F; Martínez, AI; Rodríguez-Díaz, J; Cervera, J (March 2010). "Pyrroline-5-carboxylate synthase and proline biosynthesis: from osmotolerance to rare metabolic disease". Protein Science. 19 (3): 372–82. doi:10.1002/pro.340. PMC 2866264. PMID 20091669.
- ^ Xu, Y; Labedan, B; Glansdorff, N (March 2007). "Surprising arginine biosynthesis: a reappraisal of the enzymology and evolution of the pathway in microorganisms". Microbiology and Molecular Biology Reviews. 71 (1): 36–47. doi:10.1128/MMBR.00032-06. PMC 1847373. PMID 17347518.
- ^ "MetaCyc: L-lysine biosynthesis I".
- ^ PETERKOFSKY, B; GILVARG, C (May 1961). "N-Succinyl-L-diaminopimelic-glutamic transaminase". The Journal of Biological Chemistry. 236 (5): 1432–8. doi:10.1016/S0021-9258(18)64192-4. PMID 13734750.
- ^ KINDLER, SH; GILVARG, C (December 1960). "N-Succinyl-L-2,6-diaminopimelic acid deacylase". The Journal of Biological Chemistry. 235: 3532–5. doi:10.1016/S0021-9258(18)64502-8. PMID 13756049.
- ^ Born, TL; Blanchard, JS (October 1999). "Structure/function studies on enzymes in the diaminopimelate pathway of bacterial cell wall biosynthesis". Current Opinion in Chemical Biology. 3 (5): 607–13. doi:10.1016/s1367-5931(99)00016-2. PMID 10508663.
- ^ "Escherichia coli K-12 substr. MG1655". serine biosynthesis. SRI International. Retrieved 12 December 2013.
- ^ Bell, JK; Grant, GA; Banaszak, LJ (Mar 30, 2004). "Multiconformational states in phosphoglycerate dehydrogenase". Biochemistry. 43 (12): 3450–8. doi:10.1021/bi035462e. PMID 15035616.
- ^ Dubnovitsky, AP; Kapetaniou, EG; Papageorgiou, AC (January 2005). "Enzyme adaptation to alkaline pH: atomic resolution (1.08 A) structure of phosphoserine aminotransferase from Bacillus alcalophilus". Protein Science. 14 (1): 97–110. doi:10.1110/ps.041029805. PMC 2253317. PMID 15608117.
- ^ Wang, W; Kim, R; Jancarik, J; Yokota, H; Kim, SH (Jan 10, 2001). "Crystal structure of phosphoserine phosphatase from Methanococcus jannaschii, a hyperthermophile, at 1.8 A resolution". Structure. 9 (1): 65–71. doi:10.1016/s0969-2126(00)00558-x. PMID 11342136.
- ^ Monschau, N; Stahmann, KP; Sahm, H; McNeil, JB; Bognar, AL (May 1, 1997). "Identification of Saccharomyces cerevisiae GLY1 as a threonine aldolase: a key enzyme in glycine biosynthesis". FEMS Microbiology Letters. 150 (1): 55–60. doi:10.1111/j.1574-6968.1997.tb10349.x. PMID 9163906.
- ^ Pye, VE; Tingey, AP; Robson, RL; Moody, PC (Sep 24, 2004). "The structure and mechanism of serine acetyltransferase from Escherichia coli". The Journal of Biological Chemistry. 279 (39): 40729–36. doi:10.1074/jbc.M403751200. PMID 15231846.
- ^ Huang, B; Vetting, MW; Roderick, SL (May 2005). "The active site of O-acetylserine sulfhydrylase is the anchor point for bienzyme complex formation with serine acetyltransferase". Journal of Bacteriology. 187 (9): 3201–5. doi:10.1128/JB.187.9.3201-3205.2005. PMC 1082839. PMID 15838047.
- ^ McPhalen, CA; Vincent, MG; Picot, D; Jansonius, JN; Lesk, AM; Chothia, C (Sep 5, 1992). "Domain closure in mitochondrial aspartate aminotransferase". Journal of Molecular Biology. 227 (1): 197–213. doi:10.1016/0022-2836(92)90691-C. PMID 1522585.
- ^ Larsen, TM; Boehlein, SK; Schuster, SM; Richards, NG; Thoden, JB; Holden, HM; Rayment, I (Dec 7, 1999). "Three-dimensional structure of Escherichia coli asparagine synthetase B: a short journey from substrate to product". Biochemistry. 38 (49): 16146–57. CiteSeerX 10.1.1.453.5998. doi:10.1021/bi9915768. PMID 10587437.
- ^ Velasco, AM; Leguina, JI; Lazcano, A (October 2002). "Molecular evolution of the lysine biosynthetic pathways". Journal of Molecular Evolution. 55 (4): 445–59. Bibcode:2002JMolE..55..445V. doi:10.1007/s00239-002-2340-2. PMID 12355264. S2CID 19460256.
- ^ Kotaka, M; Ren, J; Lockyer, M; Hawkins, AR; Stammers, DK (Oct 20, 2006). "Structures of R- and T-state Escherichia coli aspartokinase III. Mechanisms of the allosteric transition and inhibition by lysine". The Journal of Biological Chemistry. 281 (42): 31544–52. doi:10.1074/jbc.M605886200. PMID 16905770.
- ^ Hadfield, A; Kryger, G; Ouyang, J; Petsko, GA; Ringe, D; Viola, R (Jun 18, 1999). "Structure of aspartate-beta-semialdehyde dehydrogenase from Escherichia coli, a key enzyme in the aspartate family of amino acid biosynthesis". Journal of Molecular Biology. 289 (4): 991–1002. doi:10.1006/jmbi.1999.2828. PMID 10369777.
- ^ Mirwaldt, C; Korndörfer, I; Huber, R (Feb 10, 1995). "The crystal structure of dihydrodipicolinate synthase from Escherichia coli at 2.5 A resolution". Journal of Molecular Biology. 246 (1): 227–39. doi:10.1006/jmbi.1994.0078. PMID 7853400.
- ^ Cirilli, M; Zheng, R; Scapin, G; Blanchard, JS (Sep 16, 2003). "The three-dimensional structures of the Mycobacterium tuberculosis dihydrodipicolinate reductase-NADH-2,6-PDC and -NADPH-2,6-PDC complexes. Structural and mutagenic analysis of relaxed nucleotide specificity". Biochemistry. 42 (36): 10644–50. doi:10.1021/bi030044v. PMID 12962488.
- ^ Beaman, TW; Binder, DA; Blanchard, JS; Roderick, SL (Jan 21, 1997). "Three-dimensional structure of tetrahydrodipicolinate N-succinyltransferase". Biochemistry. 36 (3): 489–94. doi:10.1021/bi962522q. PMID 9012664.
- ^ Weyand, S; Kefala, G; Weiss, MS (Mar 30, 2007). "The three-dimensional structure of N-succinyldiaminopimelate aminotransferase from Mycobacterium tuberculosis". Journal of Molecular Biology. 367 (3): 825–38. doi:10.1016/j.jmb.2007.01.023. PMID 17292400.
- ^ Nocek, BP; Gillner, DM; Fan, Y; Holz, RC; Joachimiak, A (Apr 2, 2010). "Structural basis for catalysis by the mono- and dimetalated forms of the dapE-encoded N-succinyl-L,L-diaminopimelic acid desuccinylase". Journal of Molecular Biology. 397 (3): 617–26. doi:10.1016/j.jmb.2010.01.062. PMC 2885003. PMID 20138056.
- ^ Pillai, B; Cherney, M; Diaper, CM; Sutherland, A; Blanchard, JS; Vederas, JC; James, MN (Nov 23, 2007). "Dynamics of catalysis revealed from the crystal structures of mutants of diaminopimelate epimerase". Biochemical and Biophysical Research Communications. 363 (3): 547–53. doi:10.1016/j.bbrc.2007.09.012. PMID 17889830.
- ^ Gokulan, K; Rupp, B; Pavelka MS, Jr; Jacobs WR, Jr; Sacchettini, JC (May 16, 2003). "Crystal structure of Mycobacterium tuberculosis diaminopimelate decarboxylase, an essential enzyme in bacterial lysine biosynthesis". The Journal of Biological Chemistry. 278 (20): 18588–96. doi:10.1074/jbc.M301549200. PMID 12637582.
- ^ a b c Weaver, Robert F. (2005). Molecular biology (3rd ed.). Boston: McGraw-Hill Higher Education. ISBN 978-0-07-284611-9.
- ^ a b c d e Cooper, Geoffrey M. (2000). The Cell: A Molecular Approach (2nd ed.). Washington (DC): ASM Press. ISBN 978-0878931064.
- ^ Jackson, R.J.; et al. (February 2010). "The mechanism of eukaryotic translation initiation and principles of its regulation". Molecular Cell Biology. 10 (2): 113–127. doi:10.1038/nrm2838. PMC 4461372. PMID 20094052.
- ^ Green, Rachel; Harry F. Noller; et al. (1997). "Ribosomes and Translation". Annu. Rev. Biochem. 66: 679–716. doi:10.1146/annurev.biochem.66.1.679. PMID 9242921.
- ^ a b c d Weissbach, Herbert; Pestka, Sidney (1977). Molecular Mechanisms of Protein Biosynthesis. New York: Academic Press. ISBN 978-0127442501.
- ^ Frank, J; Haixiao Gao; et al. (September 2007). "The process of mRNA–tRNA translocation". PNAS. 104 (50): 19671–19678. doi:10.1073/pnas.0708517104. PMC 2148355. PMID 18003906.
- ^ a b c Bandeali, Salman J.; Daye, Jad; Virani, Salim S. (30 November 2013). "Novel Therapies for Treating Familial Hypercholesterolemia". Current Atherosclerosis Reports. 16 (1): 382. doi:10.1007/s11883-013-0382-0. PMID 24293346. S2CID 8903481.
- ^ a b c Kang, Tae Hyuk; Park, Yongjin; Bader, Joel S.; Friedmann, Theodore; Cooney, Austin John (9 October 2013). "The Housekeeping Gene Hypoxanthine Guanine Phosphoribosyltransferase (HPRT) Regulates Multiple Developmental and Metabolic Pathways of Murine Embryonic Stem Cell Neuronal Differentiation". PLOS ONE. 8 (10): e74967. Bibcode:2013PLoSO...874967K. doi:10.1371/journal.pone.0074967. PMC 3794013. PMID 24130677.
- ^ a b c Walport, Ken Murphy, Paul Travers, Mark (2011). Janeway's Immunobiology (8. ed.). Oxford: Taylor & Francis. ISBN 978-0815342434.
- ^ a b Hughes, edited by Donald C. Lo, Robert E. (2010). Neurobiology of Huntington's disease : applications to drug discovery (2nd ed.). Boca Raton: CRC Press/Taylor & Francis Group. ISBN 978-0849390005.
{{cite book}}:first=범용명(도움말)이 있습니다. - ^ Biglan, Kevin M.; Ross, Christopher A.; Langbehn, Douglas R.; Aylward, Elizabeth H.; Stout, Julie C.; Queller, Sarah; Carlozzi, Noelle E.; Duff, Kevin; Beglinger, Leigh J.; Paulsen, Jane S. (26 June 2009). "Motor abnormalities in premanifest persons with Huntington's disease: The PREDICT-HD study". Movement Disorders. 24 (12): 1763–1772. doi:10.1002/mds.22601. PMC 3048804. PMID 19562761.
![{\displaystyle {\ce {Reactant ->[][enzyme] Product}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/d97fbef58f64fb0b7d05d90fa0a73b05f5919fc8)

![{\displaystyle {\ce {{Precursor~molecule}+Cofactor->[][enzyme]macromolecule}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/b192f25eec9ceebed179bfeb43ae27795f3a91be)