DNA복제
DNA replication
분자생물학에서, DNA 복제는 하나의 원래 DNA [1]분자로부터 두 개의 동일한 DNA 복제를 생성하는 생물학적 과정이다.DNA 복제는 생물학적 유전에서 가장 중요한 역할을 하는 모든 살아있는 유기체에서 일어난다.이것은 또한 각각의 새로운 세포들이 [2]DNA의 복사본을 받는 것을 보장하는 반면, 손상된 조직의 성장과 복구 동안 세포 분열을 위해 필수적이다.세포는 DNA의 복제를 필수적으로 만드는 분열이라는 독특한 특성을 가지고 있다.
DNA는 상보적인 두 가닥의 이중나선으로 구성되어 있다.이중 나선은 이중 가닥 DNA의 외관을 나타내며, 따라서 서로 반대 방향으로 달리고 함께 꼬여 [3]형성되는 두 개의 선형 가닥으로 구성됩니다.복제하는 동안 이러한 가닥이 분리됩니다.원래의 DNA 분자의 각 가닥은 반보수적 복제라고 불리는 과정인 상대 DNA 분자의 생산을 위한 템플릿 역할을 한다.반보존적 복제의 결과로, 새로운 나선은 새로 합성된 [4]가닥뿐만 아니라 원래의 DNA 가닥으로 구성될 것이다.세포 교정 및 오류 검사 메커니즘은 DNA [5][6]복제를 위한 거의 완벽한 무결성을 보장합니다.
세포에서, DNA 복제는 [8]유기체의 유전 물질을 포함하는 게놈의[7] 특정한 위치 또는 복제의 기원에서 시작된다.헬리케이스로 알려진 효소에 의해 수용되는 새로운 가닥의 원점에서 DNA를 풀고 합성하는 것은 원점에서 양방향으로 성장하는 복제 포크를 초래한다.DNA 합성의 시작과 지속을 돕기 위해 많은 단백질이 복제 포크와 관련되어 있다.가장 두드러지게, DNA 중합효소는 각각의 (템플릿) 가닥을 보완하는 뉴클레오티드를 첨가함으로써 새로운 가닥을 합성한다.DNA 복제는 간상 S단계에서 일어난다.
DNA 복제(DNA 증폭)는 체외(인공적으로 세포 외부)에서도 수행될 수 있다.세포와 인공 DNA 프라이머에서 분리된 DNA 중합효소는 템플릿 DNA 분자의 알려진 배열에서 DNA 합성을 시작하기 위해 사용될 수 있다.중합효소 연쇄반응(PCR), 리가아제 연쇄반응(LCR) 및 전사 매개 증폭(TMA)이 그 예이다.2021년 3월, 연구원들은 번역의 필수 요소인 전달 RNA의 예비 형태, 유전자 코드에 따른 새로운 단백질의 생물학적 합성, 즉 생명체의 매우 초기 발달, 즉 [9][10]생물의 발생에서 복제자 분자 그 자체일 수 있다는 증거를 보고했다.
DNA구조
DNA는 특징적인 이중나선을 형성하기 위해 두 가닥이 함께 감겨 있는 이중가닥 구조로 존재한다.각각의 DNA 가닥은 네 가지 종류의 뉴클레오티드의 사슬이다.DNA의 뉴클레오티드는 디옥시리보스 당, 인산염 및 핵산염기를 포함한다.네 가지 유형의 뉴클레오티드는 일반적으로 A, C, G, T로 약칭되는 네 가지 핵염기 아데닌, 시토신, 구아닌, 티민에 해당한다.아데닌과 구아닌은 퓨린 염기이고, 시토신과 티민은 피리미딘이다.이러한 뉴클레오티드는 포스포디에스터 결합을 형성하여 핵염기가 안쪽으로 향하는 DNA 이중나선의 인산염-디옥시리보스 골격을 형성한다(즉, 반대쪽 가닥을 향함).핵산염기들은 수소 결합을 통해 가닥 사이에 매칭되어 염기쌍을 형성한다.아데닌은 티민과, 구아닌은 시토신과 짝을 이룬다.[citation needed]
DNA 가닥은 방향성을 가지며, 단일 가닥의 서로 다른 끝을 "3µ (3-prime)"와 "5-prime (5-prime)"라고 부릅니다.관례상 단일 DNA 가닥의 염기서열이 주어지면 배열의 왼쪽 끝은 5º 말단이고, 배열의 오른쪽 끝은 3º 말단이다.이중나선의 가닥은 반평행이며 하나는 5µ~3µ, 반대쪽 가닥은 3µ~5㎜이다.이러한 용어는 사슬에서 다음 인산염이 결합되는 디옥시리보스의 탄소 원자를 나타냅니다.DNA 중합효소는 DNA 가닥의 3' 말단에 뉴클레오티드를 첨가함으로써 한 방향으로만 DNA를 합성할 수 있기 때문에 방향성은 DNA 합성에 영향을 미친다.
(수소 결합을 통해) DNA에서 상보적 염기의 쌍은 각 가닥에 포함된 정보가 중복됨을 의미합니다.포스포디에스터(가닥 내) 결합은 수소(가닥 간) 결합보다 강하다.포스포디에스터 결합의 실제 역할은 DNA 폴리머에서 한 뉴클레오티드의 5' 탄소 원자와 다른 뉴클레오티드의 3' 탄소 원자를 연결하는 것이며, 수소 결합은 나선 축을 가로질러 DNA 이중 나선을 안정화하지만 축 [11]1의 방향은 안정화하지 않는다.이를 통해 가닥을 서로 분리할 수 있습니다.따라서 단일 가닥의 뉴클레오티드는 새로 합성된 파트너 [12]가닥의 뉴클레오티드를 재구성하는 데 사용될 수 있다.
DNA중합효소

DNA 중합효소는 모든 형태의 DNA [14]복제를 수행하는 효소의 한 종류이다.일반적으로 DNA 중합효소는 새로운 가닥의 합성을 시작할 수 없지만, 템플릿 가닥과 쌍을 이루는 기존 DNA 또는 RNA 가닥만 확장할 수 있습니다.합성을 시작하기 위해, 프라이머라고 불리는 RNA의 짧은 조각이 생성되어야 하고 템플릿 DNA 가닥과 쌍을 이루어야 합니다.
DNA 중합효소는 기존 뉴클레오티드 사슬의 3' 말단을 연장함으로써 새로운 DNA 가닥을 첨가하고, 포스포디에스테르 결합의 생성을 통해 템플릿 가닥에 매치된 새로운 뉴클레오티드를 한 번에 하나씩 첨가한다.이 DNA 중합 과정의 에너지는 각 비법인 염기에 부착된 세 개의 인산염 사이의 고에너지 인산염(인산수소) 결합의 가수분해에서 나온다.인산기가 부착된 유리 염기는 뉴클레오티드라고 불리며, 특히 세 개의 인산기가 부착된 염기는 뉴클레오사이드 트리포스페이트라고 불린다.성장 중인 DNA 가닥에 뉴클레오티드가 첨가될 때 뉴클레오티드의 근위 인산염과 성장 사슬 사이의 포스포디에스테르 결합의 형성은 피로인산염 원위 인산기의 방출과 함께 고에너지 인산염 결합의 가수분해를 수반한다.생성된 피로인산염의 무기인산염으로의 효소적 가수분해는 제2의 고에너지 인산염 결합을 소비하여 효과적으로 되돌릴 [Note 1]수 없는 반응을 일으킨다.
일반적으로 DNA 중합효소는 매우 정확하며, 10개의 뉴클레오티드가 [15]첨가될 때마다7 고유 오류율이 1개 미만이다.게다가, 일부 DNA 중합효소들은 또한 교정 능력을 가지고 있다; 그들은 불일치 염기를 교정하기 위해 성장하는 가닥의 끝에서 뉴클레오티드를 제거할 수 있다.마지막으로 복제 후 불일치 복구 기구는 DNA의 오류를 감시하고 새로 합성된 DNA 가닥의 불일치를 원래의 가닥 배열과 구별할 수 있다.이 세 가지 식별 단계를 함께 사용하면 10개의 뉴클레오티드가 [15]추가될 때마다9 1개 미만의 오류가 복제됩니다.
살아있는 세포에서 DNA 복제 속도는 먼저 파지에 감염된 [16]대장균에서 파지 T4 DNA 신장 속도로 측정되었다.37°C에서 DNA가 기하급수적으로 증가하는 동안, 속도는 초당 749개의 뉴클레오티드였다.파지 T4 DNA 합성 중 복제당 염기쌍당 돌연변이율은 [17]10당8 1.7이다.
레플리케이션 프로세스
DNA 복제는 모든 생물학적 중합 과정과 마찬가지로 효소적으로 촉매되고 조정된 세 가지 단계, 즉 시작, 신장 및 종료로 진행됩니다.
개시
세포가 분열하려면 먼저 DNA를 [18]복제해야 합니다. DNA 복제는 모두 아니면 전혀 하지 않는 과정입니다. 복제가 시작되면 완료됩니다.복제가 완료되면 동일한 셀 사이클에서 다시 발생하지 않습니다.이것은, 프리 레플리케이션 복합체의 개시 분할에 의해서 가능하게 됩니다.
프리 리플리케이션의 복잡성
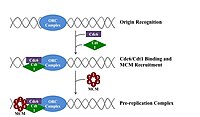
후기 유사분열과 초기 G1 단계에서, "원래원"[7]으로 알려진 DNA의 특정 지점에서 많은 이니시에이터 단백질 복합체가 사전 복제 복합체로 조립된다.대장균에서 1차 개시 단백질은 DnaA이고, 효모에서는 이것이 원산지 [19]인식 복합체이다.개시자 단백질에 의해 사용되는 배열은 AT가 풍부한 경향이 있는데, 이는 A-T 염기쌍이 (C-G 쌍에 형성된 세 개의 수소 결합보다) 두 개의 수소 결합을 가지고 있기 때문에 가닥 [20]분리가 더 쉽기 때문이다.진핵생물에서 원점인식복합체는 개시단백질의 조립을 전복제복합체로 촉매한다.또한 최근 보고에 따르면 발아 효모 ORC는 라이선스를 [21][22]제어하기 위해 세포 주기 의존적인 방식으로 이합체화된다.ORC와 Noc3p는 세포주기 [23]전체에 걸쳐 염색질에 연속적으로 결합되어 있다.다음으로 Cdc6와 Cdt1은 DNA에 Mcm 복합체를 로드하는 데 필요한 더 큰 복합체를 형성하기 위해 원점에서 결합된 기원 인식 복합체와 관련된다.Mcm 콤플렉스는 진핵생물의 복제 기원과 복제 분기점에서 DNA 나선을 풀어주는 헬리케이스입니다.Mcm 복합체는 G1 후기 단계에서 모집되며, ORC-Cdc6-Cdt1 복합체에 의해 ATP 의존성 단백질 리모델링을 통해 DNA에 적재된다.원점 DNA에 대한 Mcm 복합체의 로딩은 복제 전 복합체 [24]형성의 완료를 나타낸다.
환경조건이 G1 후반기에 적합하면 G1 및 G1/S 사이클린-Cdk 복합체가 활성화되어 DNA 합성기계의 성분을 코드하는 유전자의 발현을 촉진하고, 또한 G1/S-Cdk 활성화는 복제기원에 따라 활성화될 수 있는 S-Cdk 복합체의 발현과 활성화를 촉진한다.n종과 세포 유형.이러한 CDK의 제어는 세포 유형과 발달 단계에 따라 달라집니다.이 조절은 S 사이클린 Clb5와 Clb6가 주로 DNA [25]복제를 담당하는 싹이 트는 효모에서 가장 잘 이해된다.Clb5,6-Cdk1 복합체는 레플리케이션 원점의 활성화를 직접 트리거하므로 S단계 [24]전체에서 각 원점을 직접 활성화해야 한다.
마찬가지로 복제 원점을 활성화하기 위해 S단계에서 Cdc7이 필요합니다.Cdc7은 세포주기 내내 활성화되지 않으며, 그 활성화는 DNA 복제의 조기 개시를 피하기 위해 엄격하게 타이밍을 맞춘다.후기 G1에서 Cdc7은 조절 서브유닛 DBF4와의 연관성의 결과로 급격히 상승하며, DBF4는 Cdc7을 직접 결합하고 단백질 키나아제 활성을 촉진한다.Cdc7은 원산지 활성의 속도 제한 조절기로 판명되었습니다.G1/S-Cdks 및/또는 S-Cdks 및/또는 Cdc7은 함께 협력하여 복제원점을 직접 활성화하고 DNA [24]합성을 개시한다.
사전 개시 복합체
초기 S단계에서 S-Cdk 및 Cdc7 활성화는 원점에서 형성된 거대 단백질 복합체인 프리 개시 복합체의 집합으로 이어진다.프리 개시 복합체의 형성에 의해, 오리지날 리플리케이션 복합체로부터 Cdc6 및 Cdt1이 치환되어 프리 레플리케이션 복합체가 비활성화 및 분해된다.원점에 사전 개시 복합체를 적재하면 Mcm 헬리케이스가 활성화되어 DNA 나선의 풀림을 일으킨다.사전 개시 복합체는 또한 [24]α-프리마아제 및 다른 DNA 중합효소를 DNA에 적재한다.
α-프리마아제가 제1프라이머를 합성한 후 프라이머-템플릿 접합부는 클램프 로더와 상호작용하고 클램프 로더는 슬라이딩 클램프를 DNA에 로드하여 DNA 합성을 시작한다.사전 시작 복합체의 구성 요소는 [24]원래 위치에서 이동할 때 복제 포크와 연결된 상태로 유지됩니다.
신장
DNA 중합효소는 5µ–3µ 활성을 가진다.알려진 모든 DNA 복제 시스템은 합성을 시작하기 전에 자유로운 3' 하이드록실기를 필요로 한다(참고: DNA 템플릿은 3' ~ 5' 방향으로 읽히는 반면, 새로운 가닥은 5' ~ 3' 방향으로 합성되므로 종종 혼동된다).DNA 합성을 위한 네 가지 메커니즘이 인식된다.
- 모든 세포 생명체 및 많은 DNA 바이러스, 위상 및 플라스미드는 프리마아제를 사용하여 짧은 RNA 프라이머를 DNA 중합효소에 의해 연장되는 유리 3µ OH기와 합성한다.
- 레트로바이러스(retrovirus)는 역전사효소에 의한 연장에 사용되는 자유 3µ OH를 제공함으로써 DNA 복제를 촉진하는 전달 RNA를 사용한다.
- DNA 중합효소에 의해 뉴클레오티드가 첨가되어 새로운 가닥을 형성하는 게놈 부착단백질(말단단백질)의 아미노산 측쇄에 의해 제공되는 아데노바이러스 및 29개 이상의 박테리오파지족.
- 단일 가닥 DNA 바이러스(서클 바이러스, 쌍둥이자리 바이러스, 파르보 바이러스 등을 포함한 그룹) 및 롤링 서클 복제(RCR) 메커니즘을 사용하는 많은 페이징과 플라스미드에서 RCR 엔도핵산가수분해효소는 게놈 가닥(단일 가닥 바이러스) 또는 DNA 가닥(플라스미드) 중 하나에 흠집을 일으킨다.절단된 가닥의 5' 끝은 뉴클레아제 상의 티로신 잔기로 전달되고, 그 후 DNA 중합효소에 의해 유리 3'OH기가 새로운 가닥을 합성하기 위해 사용된다.
첫 번째는 이러한 메커니즘 중 가장 잘 알려져 있으며 세포 유기체에 의해 사용된다.이 메커니즘에서, 두 가닥이 분리되면, 프리마아제는 템플릿 가닥에 RNA 프라이머를 추가합니다.선행 가닥에는 하나의 RNA 프라이머가 공급되고 지연 가닥에는 여러 개의 RNA 프라이머가 공급됩니다.선두 가닥은 높은 처리성을 가진 DNA 중합효소에 의해 프라이머에서 연속적으로 연장되고, 후행 가닥은 오카자키 단편을 형성하는 각 프라이머에서 불연속적으로 연장된다.RNase는 프라이머 RNA 단편을 제거하고 복제 중합효소와는 다른 저프로세서성 DNA 중합효소가 들어가 그 틈새를 메운다.이것이 완료되면 리딩 스트랜드에 1개의 칼집이 있고, 래깅 스트랜드에 여러 개의 칼집이 있습니다.리가아제는 이러한 흠집을 메우기 위해 작용하여 새로 복제된 DNA 분자를 완성합니다.
이 과정에서 사용되는 프리마아제는 박테리아와 고세균/유핵생물 사이에 현저하게 다르다.박테리아는 TPRIM [26]폴드형의 촉매 도메인을 포함하는 DnaG 단백질 슈퍼패밀리에 속하는 프리마아제를 사용한다.TOPRIM 접힘은 로스만 유사 위상에 보존된 4개의 가닥을 가진 α/β 코어를 포함한다.이러한 구조는 또한 토포이소머라아제 Ia, 토포이소머라아제 II, OLD 패밀리 핵산분해효소 및 RecR 단백질과 관련된 DNA 복구 단백질의 촉매 도메인에서도 발견된다.
이와는 대조적으로, 고세균과 진핵생물에 의해 사용되는 프리마아제는 RNA 인식 모티브(RRM)의 고도로 파생된 버전을 포함하고 있습니다.이 프리마아제는 DNA 복제 및 복구에 관여하는 A/B/Y 계열의 많은 바이러스 RNA 의존성 RNA 중합효소, 역전사효소, 고리형 뉴클레오티드 생성 사이클라아제 및 DNA 중합효소 등과 구조적으로 유사하다.진핵생물 복제에서 프리마아제는 폴α와 [27]복합체를 형성한다.
여러 DNA 중합효소는 DNA 복제 과정에서 서로 다른 역할을 합니다.대장균에서 DNA Pol III는 주로 DNA 복제를 담당하는 중합효소이다.레플리케이션의 분기점에 있는 레플리케이션 컴플렉스로 조립되어, 레플리케이션 사이클 전체에 걸쳐서 지극히 높은 처리 능력을 발휘합니다.반면, DNA Pol I은 RNA 프라이머를 DNA로 대체하는 효소이며, DNA Pol I은 중합효소 활성 외에 5μ~3μ 엑소핵산가수분해효소 활성을 가지고 있으며, 닉 번역이라고 불리는 프로세스에서 엑소핵산가수분해효소 활성을 이용하여 RNA 프라이머를 앞에서 분해한다.폴리 I은 폴리 III보다 훨씬 덜 생산적이다. 왜냐하면 폴리 I의 주된 기능은 매우 긴 몇 개의 영역보다는 짧은 많은 DNA 영역을 만드는 것이기 때문이다.
진핵생물에서 저프로세서 효소인 [28]폴α는 프리마아제와의 복합체를 형성하기 때문에 복제를 시작하는데 도움을 준다.진핵생물에서는 선도 가닥 합성이 Pol ε에 의해 이루어지는 것으로 생각되지만, 최근 이러한 관점에 대한 의문이 제기되어 [29]Pol[30] for에 대한 역할이 제시되고 있다. Primer 제거는 완료되고, 복제 중 DNA 복구는 Pol pol에 의해 완료된다.
DNA 합성이 계속됨에 따라 원래 DNA 가닥은 기포의 각 측면에서 풀리면서 두 갈래로 복제 포크를 형성합니다.원형 염색체에 복제의 단일 기원을 가진 박테리아에서, 이 과정은 "세타 구조"를 형성한다.반대로, 진핵 생물은 더 긴 선형 염색체를 가지고 있고 [31]이것들 안에서 여러 기원에서 복제를 시작합니다.
레플리케이션 포크
복제 포크는 DNA 복제 중에 긴 나선형 DNA 내에 형성되는 구조입니다.그것은 헬리케이스에 의해 생성되는데, 헬리케이스는 두 DNA 가닥을 나선에서 함께 고정시키는 수소 결합을 파괴합니다.그 결과 만들어진 구조는 두 개의 분기된 "가지"를 가지며, 각각은 하나의 DNA 가닥으로 구성됩니다.이 두 가닥은 DNA 중합효소가 상보적인 뉴클레오티드를 템플릿에 일치시킬 때 생성되는 선행 및 지연 가닥 템플릿의 역할을 합니다. 템플릿은 선행 가닥 템플릿 및 지연 가닥 템플릿으로 적절하게 언급될 수 있습니다.
DNA 중합효소에 의해 3~5㎜ 방향으로 DNA가 읽히므로 새로운 가닥이 5'~3' 방향으로 합성된다.리딩 스트랜드 템플릿과 래깅 스트랜드 템플릿은 레플리케이션 포크의 방향과 반대 방향인 새로운 래깅 스트랜드 DNA의 합성을 실현하는 것이 주요 문제입니다.
선두 가닥
선두 가닥은 성장하는 복제 포크와 같은 방향으로 합성되는 새로운 DNA 가닥입니다.이런 종류의 DNA 복제는 계속된다.
래깅 스트랜드
지연된 가닥은 합성 방향이 복제 포크의 방향과 반대인 새로운 DNA 가닥입니다.그 배향 때문에, 선두의 스트랜드에 비해, 후행하는 스트랜드의 복제가 복잡하다.결과적으로, 이 가닥의 DNA 중합효소는 다른 가닥의 뒤에 "뒤처지는" 것으로 보입니다.
지연된 가닥은 짧은 분리된 세그먼트로 합성됩니다.지연성 가닥 템플릿에서 프리마아제는 템플릿 DNA를 "읽고" 짧은 상보적 RNA 프라이머의 합성을 시작한다.DNA 중합효소는 프라이밍 세그먼트를 연장하여 오카자키 단편을 형성한다.RNA 프라이머는 제거되고 DNA로 대체되며, DNA 조각은 DNA 연결 효소에 의해 결합됩니다.
레플리케이션 포크에서의 다이내믹스
모든 경우에 헬리케이스는 복제되는 DNA의 한 가닥만을 감싸는 6개의 폴리펩타이드로 구성됩니다.두 중합효소는 헬리케이스 헥시머에 결합되어 있다.진핵생물에서는 헬리케이스가 선두 가닥을 감싸고, 원핵생물에서는 뒤처진 [32]가닥을 감싸고 있다.
헬리케이스가 복제 포크에서 DNA를 풀면 전방의 DNA가 강제로 회전합니다.이 과정은 앞에 [33]있는 DNA의 뒤틀림을 초래한다.이 축적은 결국 복제 포크의 진행을 중단시키는 비틀림 저항을 형성합니다.토포이소머라아제(topoisomerase)는 DNA 나선의 두 가닥을 풀면서 일시적으로 DNA의 가닥을 끊는 효소이다. 토포이소머라아제(DNA gyrase 포함)는 DNA [34]나선에 음의 슈퍼코일을 추가함으로써 이를 달성한다.
맨 단가닥 DNA는 2차 구조를 형성하면서 스스로 접히는 경향이 있다; 이러한 구조는 DNA 중합효소의 움직임을 방해할 수 있다.이를 방지하기 위해 단일 가닥 결합 단백질은 두 번째 가닥이 합성될 때까지 DNA에 결합해 2차 구조 [35]형성을 방해한다.
이중 가닥 DNA는 유전자 발현을 조절하는 데 중요한 역할을 하는 히스톤 주위에 감겨져 있기 때문에 복제된 DNA는 원래 DNA와 같은 장소에서 히스톤 주위에 감겨져야 한다.이를 위해 히스톤 샤페론은 염색질이 복제되기 전에 분해하고 히스톤을 올바른 위치에 치환한다.이 재조립의 일부 단계는 다소 [36]추측적입니다.
클램프 단백질은 DNA 주위에 슬라이딩 클램프를 형성하여 DNA 중합효소가 그 템플릿과 접촉하는 것을 돕고, 그 결과 프로세스성을 돕는다.클램프의 내부 표면은 클램프를 통해 DNA를 나사산할 수 있도록 합니다.중합효소가 템플릿의 끝에 도달하거나 이중 가닥 DNA를 검출하면 슬라이딩 클램프는 DNA 중합효소를 방출하는 구조 변화를 겪습니다.클램프 로드 단백질은 템플릿과 RNA 프라이머 사이의 [6]:274-5접합을 인식하여 클램프를 최초로 로드하는 데 사용됩니다.
DNA복제단백질
복제 갈림길에서, 많은 복제 효소는 DNA에서 레피좀이라고 불리는 복잡한 분자 기계로 모입니다.다음은 [37]리플리솜에 관여하는 주요 DNA 복제 효소의 목록입니다.
| 효소 | DNA 복제 기능 |
|---|---|
| DNA헬리카아제 | 나선 불안정 효소라고도 알려져 있습니다.헬리케이스는 토포이소머라아제 뒤의 복제 포크에 있는 두 가닥의 DNA를 분리한다. |
| DNA중합효소 | DNA 복제 중 5µ~3µ 방향으로 DNA에 대한 뉴클레오티드 기질의 첨가를 촉매하는 효소.또한 교정 및 오류 수정도 수행합니다.다양한 종류의 DNA 중합효소가 존재하며, 각각의 DNA 중합효소는 다른 종류의 세포에서 서로 다른 기능을 수행합니다. |
| DNA클램프 | 늘어나는 DNA 중합효소가 DNA 모선으로부터 해리되는 것을 방지하는 단백질. |
| 단일사슬DNA결합단백질 | ssDNA에 결합하고 DNA 헬리케이스가 풀린 후 DNA 이중나선이 재결합하는 것을 방지하여 가닥 분리를 유지하고 새로운 가닥의 합성을 촉진한다. |
| 토포이소머라아제 | DNA의 초코일성을 완화시켜 줍니다. |
| DNA자이라아제 | DNA 헬리케이스에 의한 풀림 긴장을 완화합니다.이것은 토포이소머라아제의 특정 유형입니다. |
| DNA리가아제 | 반보존 가닥을 다시 제거하여 지연 가닥의 Okazaki fragments를 결합합니다. |
| 프라이마아제 | DNA 중합효소가 새로운 DNA 가닥의 합성을 시작하기 위한 RNA(또는 DNA)의 시작점을 제공합니다. |
| 텔로머라아제 | 진핵생물 염색체 끝에 반복적인 뉴클레오티드 배열을 추가함으로써 텔로머 DNA를 늘린다.이것은 생식 세포와 줄기세포가 세포 [38]분열에 대한 헤이플릭 제한을 피할 수 있게 해준다. |
레플리케이션 머신
복제 기계는 DNA 복제 및 템플릿 ssDNA에 나타나는 인자로 구성됩니다.복제 기계에는 복제 효소인 프리모터(primosotor)가 포함된다.DNA 중합효소, DNA 헬리케이스, DNA 클램프와 DNA 토포이소머라아제 및 복제 단백질(예를 들어 단일 가닥 DNA 결합 단백질(SSB)이다.레플리케이션 머신에서는, 이러한 컴포넌트가 조정됩니다.대부분의 박테리아에서, DNA 복제에 관련된 모든 요소들은 복제 포크에 위치해 있고 복합체들은 DNA 복제 동안 포크에 머물러 있다.이 복제 기계들은 레피솜 또는 DNA 복제효소 시스템이라고 불립니다.이러한 용어는 복제 포크에 위치한 단백질의 총칭입니다.진핵세포와 일부 박테리아 세포에서는 레피좀이 형성되지 않는다.
복제 기계는 공장 등의 템플릿 DNA에 상대적으로 이동하지 않기 때문에 복제 [39]팩토리라고 불립니다.또 다른 그림에서, DNA 공장은 프로젝터와 비슷하고 DNA는 영화 필름이 프로젝터로 끊임없이 전달되는 것과 같습니다.리플리케이션 팩토리 모델에서는 선두 스트랜드와 지연 스트랜드를 위한 양쪽 DNA 헬리케이스가 템플릿 DNA에 로드된 후 헬리케이스가 DNA를 따라 서로 내부로 이동한다.헬리케이스는 복제 과정의 나머지 기간 동안 연관된 상태로 유지됩니다.Peter Meister 등은 녹색 형광 단백질(GFP) 태그 부착 DNA 중합효소α를 모니터링하여 발아 효모에서 직접 복제 부위를 관찰했다.그들은 복제 원점에서 대칭적으로 떨어져 있는 태그 부착 위치 쌍의 DNA 복제를 감지했고,[40] 시간이 지날수록 쌍 사이의 거리가 현저하게 줄어든다는 것을 발견했다.이 연구 결과는 DNA 복제의 메커니즘이 DNA 공장과 일치함을 시사한다.즉, 레플리케이션 팩토리 쌍은 레플리케이션의 발신기지와 서로 관련되어 있는 팩토리에 로딩됩니다.또한 템플릿 DNA가 공장 안으로 이동하여 템플릿 ssDNA와 새로운 DNA를 압출합니다.마이스터의 발견은 복제 공장 모델의 첫 번째 직접 증거입니다.후속 연구는 DNA 헬리케이스가 많은 진핵 세포에서 이합체를 형성하고 박테리아 복제 기계는 DNA [39]합성 동안 단일 핵 내 위치에 머무른다는 것을 보여주었다.
복제 공장에서는 자매 염색체의 분해를 수행합니다.분리는 DNA 복제 후 염색분체를 딸세포로 분배하는데 필수적이다.DNA 복제 후 자매 염색체는 코메틴 고리에 의해 서로 고정되기 때문에, DNA 복제에서 분해를 일으킬 수 있는 유일한 기회가 있다.복제 기계를 복제 팩토리로 수정하면 DNA 복제 성공률을 높일 수 있습니다.복제 포크가 염색체 내에서 자유롭게 움직이면 핵의 응집작용이 심해져 유사분열을 [40]방해한다.
종료
진핵생물은 염색체의 여러 지점에서 DNA 복제를 시작하므로, 복제 포크는 염색체의 여러 지점에서 만나고 끝납니다.진핵생물들은 선형 염색체를 가지고 있기 때문에, DNA 복제가 염색체의 맨 끝에 도달할 수 없다.이 문제로 인해 염색체 끝에서 각각의 복제 사이클에서 DNA가 손실된다.텔로미어는 말단 가까이에 있는 반복적인 DNA의 영역이며 이러한 단축으로 인한 유전자 손실을 막는데 도움을 준다.텔로미어의 단축은 체세포의 정상적인 과정이다.이것은 딸 DNA 염색체의 텔로미어를 줄여준다.결과적으로, 세포는 DNA 손실이 더 이상의 분열을 막을 때까지 일정 횟수만 분열할 수 있다.(이것은 Hayflick 한계라고 불립니다).다음 세대에 DNA를 전달하는 생식세포주 내에서 텔로머라아제는 텔로미어 영역의 반복 배열을 확장하여 열화를 방지한다.텔로머라아제는 실수로 체세포에서 활성화되어 때로는 암 형성을 초래할 수 있다.텔로머라아제 활성 증가는 암의 특징 중 하나이다.
종료하려면 DNA 복제 포크의 진행을 중지하거나 차단해야 합니다.특정 궤적에서의 종단은 발생 시 (1) DNA 내의 종단 부위 배열과 (2) DNA 복제를 물리적으로 정지시키기 위해 이 배열에 결합하는 단백질의 두 성분 간의 상호작용을 포함한다.다양한 박테리아 종에서, 이것은 DNA 복제 말단 부위 결합 단백질 또는 Ter 단백질로 불린다.
박테리아는 원형 염색체를 가지고 있기 때문에 두 복제 포크가 부모 염색체의 반대쪽 끝에서 서로 만나면 복제 종료가 일어난다.대장균은 Tus 단백질에 의해 결합되었을 때 복제 포크의 한 방향만 통과할 수 있는 종단 시퀀스를 사용하여 이 과정을 조절합니다.그 결과 복제 포크는 염색체의 [41]종단 영역 내에서 항상 만나도록 구속된다.
규정
진핵생물
진핵생물 내에서 DNA 복제는 세포주기의 맥락에서 제어된다.세포가 성장하고 분열하면서 세포 주기의 단계를 거친다; DNA 복제는 S 단계(합성 단계) 동안 이루어진다.진핵세포가 순환하는 과정은 세포주기 체크포인트에 의해 제어된다.체크포인트를 통한 진행은 사이클린과 사이클린 의존성 [42]키나제를 포함한 다양한 단백질 사이의 복잡한 상호작용을 통해 제어된다.박테리아와 달리, 진핵 DNA는 [43]핵의 경계에서 복제된다.
G1/S 체크포인트(또는 제한 체크포인트)는 진핵세포가 DNA 복제 및 후속 분열 과정에 진입하는지 여부를 조절합니다.이 체크포인트를 통과하지 못한 세포는 G0 단계에 머물러 DNA를 복제하지 않는다.
G1/S 체크포인트를 통과한 후 DNA는 각 세포 사이클에서 한 번만 복제해야 합니다.Mcm 단지가 원점에서 멀어지면 프리 리플리케이션 단지가 해체된다.사전 레플리케이션서브유닛이 다시 활성화될 때까지 새로운 Mcm 컴플렉스를 오리진에 로드할 수 없기 때문에 복제의 1개의 오리진을 같은 셀사이클로 [24]2회 사용할 수 없습니다.
S 초기 단계에서 S-Cdks의 활성화는 개별 사전 복제 복합 성분의 파괴 또는 억제를 촉진하여 즉각적인 재조립을 방지한다.S 및 M-Cdks는 S 단계가 완료된 후에도 복제 전 복합 어셈블리를 계속 차단하여 후기 [24]유사분열에서 모든 Cdk 활동이 감소될 때까지 조립이 다시 발생하지 않도록 합니다.
발아 효모에서 조립 억제는 복제 전 복합성분의 Cdk의존성 인산화에 의해 일어난다.S상 개시 시에 Cdk1에 의한 Cdc6의 인산화로 Cdc6의 SCF 유비퀴틴 단백질 리가아제와의 결합이 일어나 Cdc6의 단백질 분해 파괴를 일으킨다.Cdk 의존성 Mcm 단백질의 인산화(인산화)는 S 단계 동안 Cdt1과 함께 핵 밖으로의 수출을 촉진하여 단일 세포 주기 동안 원점에서 새로운 Mcm 복합체의 부하를 방지한다.원점복제복합체의 Cdk인산화도 복제전복합체조립을 저해한다.이들 3가지 메커니즘 중 하나의 개별 존재만으로도 복제 전 복잡한 어셈블리를 억제하기에 충분합니다.그러나, 같은 세포에 있는 세 단백질의 돌연변이는 한 세포 [24][44]주기 내의 많은 복제 기원에서 재재개를 촉발한다.
동물 세포에서 단백질 제미닌은 복제 전 복합체 조립의 주요 억제제이다.제미닌은 Cdt1을 결합시켜 원점 인식 복합체에 결합하는 것을 방지한다.G1에서는 제미닌의 농도는 APC에 의해 낮게 유지되며, APC는 제미닌을 유비쿼터스화하여 분해 대상으로 한다.제미닌이 파괴되면 Cdt1이 해방되어 레플리케이션 전의 복잡한 어셈블리에서 기능합니다.G1의 마지막에 APC가 비활성화되어 제미닌이 축적되어 Cdt1과 [24]결합할 수 있다.
엽록체와 미토콘드리아 게놈의 복제는 D-루프 복제 과정을 통해 세포 주기와 독립적으로 일어난다.
레플리케이션의 초점
척추동물 세포에서 복제 부위는 복제 [40]포치라고 불리는 위치에 집중됩니다.복제 부위는 딸 가닥과 복제 효소를 면역화하고 GFP 태그 부착 복제 인자를 감시함으로써 검출할 수 있다.이러한 방법에 의해 다양한 크기와 위치의 복제 포치가 세포 분열의 S상에 나타나며 핵당 개수가 게놈 복제 포크 수보다 훨씬 적은 것을 알 수 있다.
P. Heun 외 [40]연구진(2001)은 발아 효모세포에서 GFP 태그 부착 복제 포시를 추적하여 복제 기원이 G1 및 S 단계에서 지속적으로 이동하고 [40]S 단계에서 역학은 현저하게 감소함을 밝혀냈다.전통적으로 복제 부위는 핵 매트릭스 또는 라민에 의해 염색체의 공간 구조에 고정되었다.Heun의 결과는 전통적인 개념을 부정하고, 싹트기 효모에는 라민이 없으며, 레플리케이션의 근원이 되는 자가 조립 및 레플리케이션 포치를 형성할 수 있도록 지원합니다.
공간적, 시간적으로 제어된 복제 원점의 발사에 의해 복제 포치의 형성을 규제한다.D. A. 잭슨 외 연구진(1998)은 인접한 기원이 포유류의 [40]세포에서 동시에 불을 낸다고 밝혔다.레플리케이션 사이트의 공간 병치는 레플리케이션 포크의 클러스터화를 가져옵니다.클러스터링은 정지된 복제를 복구하며 복제 포크의 정상적인 진행 상태를 선호합니다.복제 포크의 진행은 단백질 또는 DNA에 강하게 결합하는 복합체와의 충돌, dNTP의 결핍, 템플릿 DNA의 흠집 등 많은 요인에 의해 억제된다.복제 포크가 중지되고 정지된 포크의 나머지 시퀀스가 복제되지 않으면 도터 스트랜드가 복제되지 않은 사이트를 얻은 것입니다.한쪽 부모 가닥에 있는 복제되지 않은 부위는 다른 쪽 가닥을 서로 붙들어 매고 있지만 딸 가닥은 그렇지 않아요따라서 자매 염색체는 서로 분리되지 않고 두 개의 딸 세포로 분할되지 않는다.인접 발신기지에서 화재가 발생하여 한 발신기지에서 포크가 정지된 경우 정지된 포크의 반대 방향으로 다른 발신기지에서 접근하여 복제되지 않은 사이트를 복제합니다.구조의 다른 메커니즘으로는 정상적인 DNA 복제에서 과도한 기원이 발생하지 않는 휴면 복제 기원의 적용이 있다.
박테리아
대부분의 박테리아는 잘 정의된 세포 주기를 거치지 않고 그들의 DNA를 지속적으로 복제한다; 빠른 성장 동안, 이것은 여러 번의 복제의 [45]동시 발생을 초래할 수 있다.가장 특징적인 박테리아인 대장균에서 DNA 복제는 원산지 배열의 반메틸화 및 시퀀싱, 아데노신 삼인산(ADP) 대 아데노신 이인산(ADP)의 비율 및 단백질 DnaA 수준을 포함한 여러 메커니즘을 통해 조절된다.이 모든 것들이 개시자 단백질의 원점 배열에 대한 결합을 조절한다.
대장균은 GATC DNA 배열을 메틸화시키기 때문에 DNA 합성은 반메틸화 배열을 낳는다.이 반메틸화 DNA는 단백질 SeqA에 의해 인식되며, 단백질 SeqA는 원서열을 결합하고 염기서열을 해독한다. 또한, (복제 개시에 필요한) DnaA는 반메틸화 DNA에 덜 잘 결합한다.그 결과, 새롭게 복제된 기원은 곧바로 또 다른 DNA [46]복제를 개시하는 것을 방지한다.
ATP는 세포가 풍부한 배지에 있을 때 축적되어 세포가 특정 크기에 도달하면 DNA 복제를 일으킨다.ATP는 DnaA와 결합하기 위해 ADP와 경쟁하고, DnaA-ATP 복합체는 복제를 시작할 수 있습니다.DNA 복제에는 일정 수의 DnaA 단백질도 필요합니다.원점을 복사할 때마다 DnaA 결합 부위의 수가 두 배로 증가하며, 더 많은 DnaA 합성이 복제의 또 다른 시작을 가능하게 합니다.
대장균과 같이 빠르게 성장하는 박테리아에서는 염색체 복제가 세포를 분열시키는 것보다 더 많은 시간이 걸린다.이 박테리아는 이전 복제판이 [47]종료되기 전에 새로운 복제판을 시작하여 이 문제를 해결합니다.새로운 복제는 분열 세포로부터 두 세대 후에 태어난 세포의 염색체를 형성할 것이다.이 메커니즘은 중복되는 복제 사이클을 생성합니다.
DNA 복제 문제
이 섹션은 확장해야 합니다.추가해서 도움을 드릴 수 있습니다. (2020년 5월) |
레플리케이션의 부하에는,[48] 다음과 같은 많은 이벤트가 있습니다.
- 리보뉴클레오티드의 오동작
- 특이한 DNA 구조
- 복제와 문자 변환 간의 충돌
- 필수 레플리케이션 팩터 부족
- 일반적인 취약 사이트
- 발암유전자의 과잉 발현 또는 구성 활성화
- 염색질 불접근성
중합효소 연쇄반응
연구자들은 일반적으로 중합효소 연쇄반응(PCR)을 이용하여 DNA를 체외로 복제한다.PCR은 한 쌍의 프라이머를 사용하여 템플릿 DNA의 표적 영역을 넘나들며, 그런 다음 내열성 DNA 중합효소를 사용하여 이들 프라이머에서 각 방향으로 파트너 가닥을 중합합니다.이 과정을 여러 사이클에 걸쳐 반복하면 표적 DNA 영역이 증폭됩니다.각 사이클 시작 시 템플릿과 프라이머의 혼합물을 가열하여 새롭게 합성된 분자와 템플릿을 분리한다.그 후 혼합물이 식으면 둘 다 새로운 프라이머를 아닐하기 위한 템플릿이 되고 중합효소는 이들로부터 연장된다.그 결과 대상 영역의 복사본 수는 라운드마다 두 배로 증가하여 [49]기하급수적으로 증가합니다.
「 」를 참조해 주세요.
메모들
- ^ 이 과정의 에너지학은 또한 합성의 방향성을 설명하는 데 도움을 줄 수 있다. DNA가 3µ ~ 5µ 방향으로 합성된다면, 그 과정의 에너지는 자유 뉴클레오티드가 아닌 성장 가닥의 5µ 끝에서 올 것이다.문제는 고에너지 트리포스페이트가 유리 뉴클레오티드가 아닌 성장 가닥에 있을 경우, 일치하지 않는 말단 뉴클레오티드를 제거하여 교정하는 것이 문제가 될 수 있다는 것입니다.일단 뉴클레오티드가 첨가되면, 삼인산은 손실되고 단일 인산염은 새로운 뉴클레오티드와 가닥의 나머지 사이의 골격에 남는다.만약 첨가된 뉴클레오티드가 일치하지 않는다면, 제거는 높은 에너지 삼인산이 아닌 "성장하는 가닥"의 끝에서 일인산에 의해 종결되는 DNA 가닥을 야기할 것이다.그래서 가닥이 막혀서 더 이상 자랄 수 없을 거야.실제로 각 단계에서 가수분해되는 고에너지 트리인산염은 중합된 가닥이 아닌 유리뉴클레오티드에서 발생하므로 이 문제는 존재하지 않는다.
레퍼런스
- ^ "DNA replication why we have to study DNA replication?". Microb Life. 2020-05-25. Retrieved 2020-05-29.
- ^ "GENETICS / DNA REPLICATION (BASIC) – Pathwayz". pathwayz.org. Retrieved 2020-12-10.
- ^ "double helix Learn Science at Scitable". Nature. Retrieved 2020-12-10.
- ^ Pray LA. "Semi-Conservative DNA Replication; Meselson and Stahl".
- ^ 불완전한 DNA 복제가 돌연변이를 일으킨다.Berg JM, Tymoczko JL, Stryer L, Clarke ND (2002). "Chapter 27: DNA Replication, Recombination, and Repair". Biochemistry. W.H. Freeman and Company. ISBN 0-7167-3051-0. Archived from the original on 2020-03-26. Retrieved 2019-08-09.
- ^ a b Lodish H, Berk A, Zipursky SL, et al. (2000). "DNA Replication, Repair, and Recombination". Molecular Cell Biology (4th ed.). WH Freeman. ISBN 0-7167-3136-3.
- ^ a b Berg JM, Tymoczko JL, Stryer L, Clarke ND (2002). "Chapter 27, Section 4: DNA Replication of Both Strands Proceeds Rapidly from Specific Start Sites". Biochemistry. W.H. Freeman and Company. ISBN 0-7167-3051-0. Archived from the original on 2020-03-26. Retrieved 2019-08-09.
- ^ "What is a genome?". yourgenome. Retrieved 2020-12-10.
- ^ Kühnlein, Alexandra; Lanzmich, Simon A.; Brun, Dieter (2 March 2021). "tRNA sequences can assemble into a replicator". eLife. 10: e63431. doi:10.7554/eLife.63431. PMC 7924937. PMID 33648631.
- ^ Maximilian, Ludwig (3 April 2021). "Solving the Chicken-and-the-Egg Problem – "A Step Closer to the Reconstruction of the Origin of Life"". SciTechDaily. Retrieved 3 April 2021.
- ^ "DNA function & structure (with diagram) (article)". Khan Academy. Retrieved 2020-12-10.
- ^ Alberts B, et al. (2002). Molecular Biology of the Cell (4th ed.). Garland Science. pp. 238–240. ISBN 0-8153-3218-1.
- ^ Allison LA (2007). Fundamental Molecular Biology. Blackwell Publishing. p. 112. ISBN 978-1-4051-0379-4.
- ^ Berg JM, Tymoczko JL, Stryer L, Clarke ND (2002). Biochemistry. W.H. Freeman and Company. ISBN 0-7167-3051-0. 27장 섹션 2: DNA 중합효소에는 템플릿과 프라이머가 필요합니다.
- ^ a b McCulloch SD, Kunkel TA (January 2008). "The fidelity of DNA synthesis by eukaryotic replicative and translesion synthesis polymerases". Cell Research. 18 (1): 148–61. doi:10.1038/cr.2008.4. PMC 3639319. PMID 18166979.
- ^ McCarthy D, Minner C, Bernstein H, Bernstein C (October 1976). "DNA elongation rates and growing point distributions of wild-type phage T4 and a DNA-delay amber mutant". Journal of Molecular Biology. 106 (4): 963–81. doi:10.1016/0022-2836(76)90346-6. PMID 789903.
- ^ Drake JW(1970) 돌연변이의 분자 기반.홀든데이, 샌프란시스코 ISBN 0816224501 ISBN 978-0816224500
- ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2002). Molecular Biology of the Cell. Garland Science. ISBN 0-8153-3218-1. 5장: DNA 복제 메커니즘
- ^ Weigel C, Schmidt A, Rückert B, Lurz R, Messer W (November 1997). "DnaA protein binding to individual DnaA boxes in the Escherichia coli replication origin, oriC". The EMBO Journal. 16 (21): 6574–83. doi:10.1093/emboj/16.21.6574. PMC 1170261. PMID 9351837.
- ^ Lodish H, Berk A, Zipursky LS, Matsudaira P, Baltimore D, Darnell J (2000). Molecular Cell Biology. W. H. Freeman and Company. ISBN 0-7167-3136-3.12.1. 염색체 복제의 일반적인 특징:레플리케이션의 3가지 공통 기능
- ^ Yin YC, Prasanth SG (July 2021). "Replication initiation: Implications in genome integrity". DNA Repair. 103: 103131. doi:10.1016/j.dnarep.2021.103131. PMC 8296962. PMID 33992866.
- ^ Amin A, Wu, R, Cheung MH, Scott JF, Wang Z, Zhou Z, Liu C, Zhu G, Wong KC, Yu Z, Liang C (March 2020). "An Essential and Cell-Cycle-Dependent ORC Dimerization Cycle Regulates Eukaryotic Chromosomal DNA Replication". Cell Reports. 30 (10): 3323–3338.e6. doi:10.1016/j.celrep.2020.02.046. PMID 32160540.
- ^ Zhang Y, Yu Z, Fu X, Liang C (June 2002). "Noc3p, a bHLH Protein, Plays an Integral Role in the Initiation of DNA Replication in Budding Yeast". Cell. 109 (7): 849–860. doi:10.1016/s0092-8674(02)00805-x. PMID 12110182.
- ^ a b c d e f g h i Morgan DO (2007). The cell cycle : principles of control. London: New Science Press. pp. 64–75. ISBN 978-0-19-920610-0. OCLC 70173205.
- ^ Donaldson AD, Raghuraman MK, Friedman KL, Cross FR, Brewer BJ, Fangman WL (August 1998). "CLB5-dependent activation of late replication origins in S. cerevisiae". Molecular Cell. 2 (2): 173–82. doi:10.1016/s1097-2765(00)80127-6. PMID 9734354.
- ^ Aravind L, Leipe DD, Koonin EV (September 1998). "Toprim--a conserved catalytic domain in type IA and II topoisomerases, DnaG-type primases, OLD family nucleases and RecR proteins". Nucleic Acids Research. 26 (18): 4205–13. doi:10.1093/nar/26.18.4205. PMC 147817. PMID 9722641.
- ^ Frick DN, Richardson CC (July 2001). "DNA primases". Annual Review of Biochemistry. 70: 39–80. doi:10.1146/annurev.biochem.70.1.39. PMID 11395402. S2CID 33197061.
- ^ Barry ER, Bell SD (December 2006). "DNA replication in the archaea". Microbiology and Molecular Biology Reviews. 70 (4): 876–87. doi:10.1128/MMBR.00029-06. PMC 1698513. PMID 17158702.
- ^ Stillman B (July 2015). "Reconsidering DNA Polymerases at the Replication Fork in Eukaryotes". Molecular Cell. 59 (2): 139–41. doi:10.1016/j.molcel.2015.07.004. PMC 4636199. PMID 26186286.
- ^ Rossi ML (February 2009). Distinguishing the pathways of primer removal during Eukaryotic Okazaki fragment maturation (Ph.D. thesis). School of Medicine and Dentistry, University of Rochester. hdl:1802/6537.
- ^ Huberman JA, Riggs AD (March 1968). "On the mechanism of DNA replication in mammalian chromosomes". Journal of Molecular Biology. 32 (2): 327–41. doi:10.1016/0022-2836(68)90013-2. PMID 5689363.
- ^ Gao Y, Cui Y, Fox T, Lin S, Wang H, de Val N, et al. (February 2019). "Structures and operating principles of the replisome". Science. 363 (6429): 835. doi:10.1126/science.aav7003. PMC 6681829. PMID 30679383.
- ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2002). Molecular Biology of the Cell. Garland Science. ISBN 0-8153-3218-1. DNA 복제 메커니즘: DNA 토포이소머라아제 복제 중 DNA 엉킴 방지
- ^ Reece RJ, Maxwell A (26 September 2008). "DNA gyrase: structure and function". Critical Reviews in Biochemistry and Molecular Biology. 26 (3–4): 335–75. doi:10.3109/10409239109114072. PMID 1657531.
- ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2002). Molecular Biology of the Cell. Garland Science. ISBN 0-8153-3218-1. DNA 복제 메커니즘:복제 포크 앞에서 DNA 이중나선을 여는 데 도움을 주는 특수 단백질
- ^ Ransom M, Dennehey B, Tyler JK (22 January 2010). "Chaperoning histones during DNA replication and repair". Cell. 140 (2): 183–195. doi:10.1016/j.cell.2010.01.004. PMC 3433953. PMID 20141833.
- ^ Griffiths AJ, Wessler SR, Lewontin RC, Carroll SB (2008). Introduction to Genetic Analysis. W. H. Freeman and Company. ISBN 978-0-7167-6887-6.[7장: DNA: 구조와 복제]페이지 283–180]
- ^ "Will the Hayflick limit keep us from living forever?". Howstuffworks. 2009-05-11. Retrieved January 20, 2015.
- ^ a b 제임스 D.왓슨 등(2008), "유전자 분자생물학", 피어슨 교육: 237
- ^ a b c d e f Peter Meister, Angela Taddei1, Susan M. Gasser (2006년 6월), "복제공장 출입", 셀 125(7): 1233~1235
- ^ Brown TA (2002). Genomes. BIOS Scientific Publishers. ISBN 1-85996-228-9.13.2.3 복제의 종료
- ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2002). Molecular Biology of the Cell. Garland Science. ISBN 0-8153-3218-1. 세포주기 이벤트 세포내 제어: S상 사이클린-Cdk 복합체(S-Cdks)는 사이클당 1회 DNA 복제를 시작한다.
- ^ Brown TA (2002). "13". Genomes (2nd ed.). Oxford: Wiley-Liss.
- ^ Nguyen VQ, Co C, Li JJ (June 2001). "Cyclin-dependent kinases prevent DNA re-replication through multiple mechanisms". Nature. 411 (6841): 1068–73. Bibcode:2001Natur.411.1068N. doi:10.1038/35082600. PMID 11429609. S2CID 4393812.
- ^ Tobiason DM, Seifert HS (June 2006). "The obligate human pathogen, Neisseria gonorrhoeae, is polyploid". PLOS Biology. 4 (6): e185. doi:10.1371/journal.pbio.0040185. PMC 1470461. PMID 16719561.
- ^ Slater S, Wold S, Lu M, Boye E, Skarstad K, Kleckner N (September 1995). "E. coli SeqA protein binds oriC in two different methyl-modulated reactions appropriate to its roles in DNA replication initiation and origin sequestration". Cell. 82 (6): 927–36. doi:10.1016/0092-8674(95)90272-4. PMID 7553853. S2CID 14652024.
- ^ Cooper S, Helmstetter CE (February 1968). "Chromosome replication and the division cycle of Escherichia coli B/r". Journal of Molecular Biology. 31 (3): 519–40. doi:10.1016/0022-2836(68)90425-7. PMID 4866337.
- ^ Zeman MK, Cimprich KA (January 2014). "Causes and consequences of replication stress". Nature Cell Biology. 16 (1): 2–9. doi:10.1038/ncb2897. PMC 4354890. PMID 24366029.
- ^ Saiki RK, Gelfand DH, Stoffel S, Scharf SJ, Higuchi R, Horn GT, et al. (January 1988). "Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase". Science. 239 (4839): 487–91. Bibcode:1988Sci...239..487S. doi:10.1126/science.239.4839.487. PMID 2448875.