리보뉴클레오티드
Ribonucleotide생화학에서 리보뉴클레오티드(libonucleotide)는 리보스를 펜토스 성분으로 함유한 뉴클레오티드다. 그것은 핵산의 분자 전구체로 여겨진다. 뉴클레오티드는 DNA와 RNA의 기본 구성블록이다. 리보뉴클레오티드는 그 자체가 RNA의 기본 단핵구성블록이다. 리보뉴클레오티드는 리보뉴클레오티드 환원효소(RNR)로 리보뉴클레오티드를 감소시켜 형성된 디옥시리보뉴클레오티드는 DNA의 필수 구성블록이다.[1] DNA 디옥시리보뉴클레오티드와 RNA 리보뉴클레오티드 사이에는 몇 가지 차이점이 있다. 연속 뉴클레오티드는 인광 결합을 통해 서로 연결된다.
리보뉴클레오티드는 다른 세포 기능에도 활용된다. 이러한 특별한 모노머는 아데노신-모노인산염(AMP)에서 볼 수 있는 세포 조절과 세포 신호 모두에 활용된다. 게다가 리보뉴클레오티드는 유기체의 에너지 통화인 아데노신 3인산염(ATP)으로 전환될 수 있다. 리보뉴클레오티드는 유기체의 호르몬을 조절하기 위해 순환 아데노신 단인산염(순환 AMP)으로 변환될 수 있다.[1] 살아있는 유기체에서 리보뉴클레오티드의 가장 흔한 근거는 아데닌(A), 구아닌(G), 시토신(C), 우라실(U)이다. 질소 베이스는 두 개의 모화합물인 퓨린과 피리미딘으로 분류된다.
구조
일반구조
리보뉴클레오타이드의 일반적인 구조는 인산염 그룹, 리보스 당 그룹, 뉴클레오바아제로 구성되어 있는데, 이 뉴클레오바아제는 아데닌, 구아닌, 시토신 또는 우라실일 수 있다. 인산염군이 없으면 뉴클레오바제와 설탕의 구성을 뉴클레오사이드라고 한다. 교환 가능한 질소 뉴클레오바제는 두 개의 모화합물인 퓨린과 피리미딘에서 추출된다. 뉴클레오티드는 이성교배 화합물로서, 즉 그들은 고리의 구성원으로서 적어도 두 개의 다른 화학 원소를 포함하고 있다.
RNA와 DNA 모두 아데닌(A)과 구아닌(G)이라는 두 가지 주요 푸린 베이스와 두 가지 주요 피리미딘을 포함하고 있다. DNA와 RNA 모두에서 피리미딘 중 하나는 시토신(C)이다. 그러나 DNA와 RNA는 제2대 피리미딘에서 차이가 있다. DNA는 티민(T)을, RNA는 우라실(U)을 함유하고 있다. RNA에서 티민, DNA에서 우라실 등에서 티민 현상이 나타나는 드문 사례도 있다.[1]
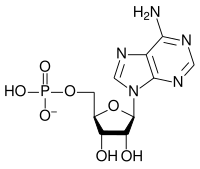
여기 RNA의 구조 단위인 4대 리보뉴클레오티드(리보뉴클레오시드 5'-모노포스페이트)가 있다.
| 뉴클레오티드 | 기호 | 뉴클레오사이드 |
|---|---|---|
| 아데닐산염(아데노신 5'-모노인산염) | A, AMP | 아데노신 |
| 구아닐산염(과노신 5'-모노인산염) | G, GMP | 구아노신 |
| 우리딜산염(우리딜산 5'-모노인산염) | U, UMP | 우리딘 |
| 시티딜레이트(사이티딘 5'-모노포스페이트) | C, CMP | 키티딘 |
디옥시리보뉴클레오티드 대 RNA 리보뉴클레오티드
리보뉴클레오티드에서 당 성분은 리보스, 디옥시리보뉴클레오티드에서 당 성분은 디옥시리보스다. 리보스 링의 두 번째 탄소에 있는 히드록실 그룹 대신 수소 원자로 대체된다.[2]
DNA와 RNA에 있는 두 종류의 펜토스는 모두 β-푸라노오스(폐쇄된 5-membed ring) 형태로 핵산의 정체성을 규정한다. DNA는 2'-데옥시-리보스 핵산을 함유하여 정의되는 반면 RNA는 리보스 핵산을 함유하여 정의된다.[1]
어떤 경우에, DNA와 RNA는 약간의 작은 염기들을 포함할 수 있다. 주요 염기들의 메틸화된 형태는 DNA에서 가장 흔하다. 바이러스성 DNA에서 일부 염기는 히드록시메틸화 또는 글루코실화일 수 있다. RNA에서는 경미하거나 변형된 염기가 더 자주 발생한다. 일부 예로는 저산산산틴, 이수소수라실, 우라실, 시토신, 구아닌의 메틸화 형태와 변형된 뉴클레오사이드 유사우르디딘 등이 있다.[3] 5' 탄소가 아닌 위치에 인산염 그룹이 있는 뉴클레오티드도 관찰되었다. 예를 들면, 분리 가능한 중간체인 리보뉴클레오시드 2' ,3'사이클릭 단인산염, 특정 리보뉴클레아제에 의한 RNA의 가수분해 최종 산물인 리보뉴클레오시드 3'-모노인산염 등이 있다. 다른 변화로는 아데노신 3', 5'-순환 모노인산염(cAMP) 및 구아노신 3', 5'-순환 모노인산염(cGMP)이 있다.[4]
연속 뉴클레오티드 연결
리보뉴클레오티드는 인광 결합을 통해 RNA 가닥을 형성하기 위해 서로 연결되어 있다. 하나의 뉴클레오티드로 이루어진 5'-인산염 그룹은 다음 뉴클레오티드의 3'-히드로옥실 그룹에 연결되어 인산염과 펜토오스 잔류물의 교대 백본을 생성한다. 폴리뉴클레오타이드의 양쪽 끝에는 인산염 결합이 없다.[5] RNA 중합효소에 의해 리보뉴클레오티드 사이에 인산염 결합이 형성된다. RNA 체인은 체인의 마지막 리보뉴클레오티드의 3'-hydroxyl 그룹이 뉴클레오피드의 역할을 하고 들어오는 리보뉴클레오티드의 5'-삼인산염에 대한 친수 공격을 개시하여 피로인산염을 바이[6] 제품으로 방출하면서 5' 끝에서 3'끝까지 합성된다. 뉴클레오티드의 물리적 특성 때문에 RNA의 등뼈는 매우 친수성과 극성을 띤다. 중성 pH에서는 각 인산염군이 음전하를 띠기 때문에 핵산이 고도로 충전된다.[7]
DNA와 RNA는 모두 아미노산보다 열역학적으로 결합 가능성이 적은 단핵산 단핵생물로도 알려져 있다. 인광 결합은 가수 분해 시 상당한 양의 자유 에너지를 방출한다. 그러므로 핵산은 자연적으로 수산화하여 단핵화물로 만드는 경향이 있다. RNA의 전구체는 GTP, CTP, UTP, ATP로 그룹 전달 반응에서 주요 에너지원이다.[8]
함수
디옥시리보뉴클레오티드 전구체
과학자들은 RNA가 DNA보다 먼저 개발되었다고 믿는다.[9]
디옥시리보뉴클레오티드에 대한 리보뉴클레오티드의 감소는 리보뉴클레오티드 환원효소에 의해 촉매로 작용한다. 리보뉴클레오티드 환원효소(RNR)는 DNA 복제와 수리에 필요한 4개의 디옥시리보뉴클레오티드(dNTP) 합성의 마지막 단계를 담당하기 때문에 모든 살아있는 유기체에 필수적인 효소다.[10] 그 반응에는 두 개의 다른 단백질인 티오레독신과 티오레독신 환원효소도 필요하다. 리보뉴클레오시드 디포스포산염(NDP)은 티오레독신(tioredoxin)에 의해 디옥시리보뉴클레오시드 디포스포산염(dNTP)으로 감소한다.
일반적인 반응은: 리보뉴클레오시드 디포스포산 + NADPH+ + H -> 디옥시리보뉴클레오시드 디포스포산 + NADP+ + HO이다2.
이 방정식을 설명하기 위해 dATP와 dGTP는 각각 ADP와 GDP에서 합성된다. 그것들은 먼저 RNR에 의해 감소된 다음 뉴클레오사이드 디인산염 키나아제에 의해 dATP와 dGTP에 인산염화된다. 리보뉴클레오티드 환원효소는 알로스테릭 상호작용에 의해 제어된다. DATP가 리보뉴클레오티드 환원효소에 결합되면 효소의 전반적인 촉매 활성도가 감소하는데, 이는 디옥시리보뉴클레오티드가 풍부함을 의미하기 때문이다. 이 피드백 억제는 ATP가 결합되면 역전된다.[12]
리보뉴클레오티드차별
DNA 합성 동안, DNA 중합체는 디옥시리보뉴클레오티드에 비해 훨씬 높은 수준으로 존재하는 리보뉴클레오티드에 대해 선택해야 한다. DNA 복제는 그 유기체의 게놈을 유지하기 위해 정확해야 하기 때문에 선택성이 있다는 것이 중요하다. Y 계열 DNA 중합체의 활성 부위는 리보뉴클레오티드 대비 높은 선택성을 유지하는 것으로 나타났다.[13] 대부분의 DNA 중합체는 또한 리보스 링의 2'-히드로옥실 그룹을 강직하게 차단할 수 있는 부피가 큰 사이드 체인 잔류물을 통해 리보뉴클레오티드를 활성 부위에서 제외할 수 있도록 설비되어 있다. 그러나 많은 핵복제 및 수리 DNA 중합체는 리보뉴클레오티드를 DNA에 통합하여 배제 메커니즘이 완벽하지 않음을 시사한다.[14][15][16]
합성
리보뉴클레오티드합성
리보뉴클레오티드는 드노보 경로를 통해 작은 분자로부터 나온 유기체에서 합성되거나 인양 경로를 통해 재활용될 수 있다. de novo 경로의 경우, 청어와 피리미딘은 모두 아미노산, 리보스-5-인산염, CO2, NH3의 전구체로부터 파생된 성분으로부터 합성된다.[17][18]
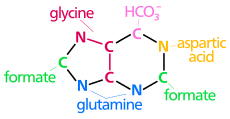 | 퓨린 고리 원자의 생합성 기원 N은1 아민 그룹인 Asp에서 발생한다. C와2 C는8 형태에서 비롯된다. N과3 N은9 Gln의 아미드 그룹에 의해 기여된다. C4, C, N은57 글리에서 파생된 것이다. C는6 HCO3−(CO2)에서 나온다. |
퓨린 뉴클레오티드의 디노보 생합성은 상당히 복잡하며 여러 효소 반응으로 구성된다. 5링의 당 구조를 베이스로 삼아 퓨린 링은 11단계 공정으로 한번에 몇 개의 원자를 만들어 인포시네이트(IMP)의 형성을 이끈다. 본질적으로, IMM은 핵산 합성에 필요한 퓨린 뉴클레오티드로 변환된다.[17]
이 경로는 효소 리보스-인산염 디프인산염(PRPS1)에 의해 리보스-5-인산염(R5P)이 인포리보실 피로인산염(PRPP)으로 전환되면서 시작된다. 그런 다음 글루타민이 아미노 그룹을 PRPP의 C-1에 기증함에 따라 PRPP는 5-인산소립아민(5-PRA)으로 변환된다. 응축 반응에서 효소 GAR 합성효소는 글리신, ATP와 함께 5-PRA의 글리신 카르복실화효소 그룹을 활성화시켜 글리시나미드 리보뉴클레오티드(GAR)를 형성한다. Co-enzyme N10-formyl-THF는 효소 GAR transformylase와 함께 아미노 그룹에 1탄소 단위를 GAR의 글리신 위에 기부한 다음, FGAR 아미도트란스페라아제에 의한 글루타민 첨가로 포밀글리시나미딘 리보뉴클레오티드(FGAM)의 형성으로 이어진다. 효소 FGAM 사이클라아제에 의해 FGAM이 탈수되면 5-아미노미다졸 리보뉴클레오티드(AIR)로 이미다졸 링이 폐쇄된다. 카복실 그룹은 N5-CAIR 합성효소에 의해 AIR에 부착되어 N5-Carboxyaminoimidazole 리보뉴클레오티드(N5-CAIR)를 형성하고, N5-CAIR 뮤타아제를 가지고 카복시아미노-이미다졸 리보뉴클레오티드(CAIR)로 전환된다. 효소 SAICAR 합성효소는 아스파르타이트의 아미노그룹과 함께 아미드 결합을 형성하여 N-수치닐-5-아미노미다잘레-4-카박스아미드 리보뉴클레오타이드(SAICAR)를 생성한다. 경로를 계속 내려가면 SAICAR 리야아제에 의해 아스파르타이트의 탄소 골격을 제거하면 5-아미노이미다졸-4-카복사미드 리보뉴클레오타이드(AICAR)가 발생한다. 효소 AICAR transformylase는 N10-formylaminoimidazole-4-carboxamide 리보뉴클레오타이드(FAICAR)를 형성하면서 N10-formyltetrahydrofolate로부터의 최종 탄소 전달을 돕는다. 마지막으로, 두 번째 링 구조의 폐쇄는 IMM synthase에 의해 수행되어 IMM을 형성하며, 여기서 IMM 운명은 퓨린 뉴클레오티드의 형성으로 이어진다.[17]
피리미딘 뉴클레오티드의 합성은 훨씬 간단한 과정이다. 피리미딘 링의 형성은 카바모일 인산염으로 응축반응을 일으켜 아스파르테이트가 N-카르바모일라스파테이트로 변환되면서 시작된다. 디히드로오타아제와 디히드로오타아제 탈수소효소는 그 후 N-Carbamoylaspartate를 오로테이트로 변환한다. 오로테이트(Orotate phosipysol-transferase)에 의한 오로티보시솔-전달효소(OMP)에 의한 오로티딜산 데카르복실화에 따라 우리딜산(UMP) 리보뉴클레오타이드 구조가 형성된다. UMP는 이후 두 개의 키나제 반응에 의해 우리딘-5'-트리스포네이트(UTP)로 변환될 수 있다. UTP로부터 Cytidine-5'-Trisphosphate(CTP)의 형성은 아킬인산염 중간재에 의한 Cytidylate synthetase에 의해 달성될 수 있다.[17]
리보뉴클레오티드 사전생물합성
생명체가 어떻게 생겨났는지 이해하기 위해서는 그럴듯한 사전 생물학적 조건 하에서 생명체의 주요 구성 요소를 형성할 수 있는 화학적 경로에 대한 지식이 필요하다. RNA 세계 가설에 따르면 원시 수프에는 자유 유동 리보뉴클레오티드(free-floating libonucleotide)가 존재했다. 이것들은 RNA를 형성하기 위해 직렬로 결합되는 기본 분자였다. RNA처럼 복잡한 분자들은 물리적 화학적 과정에 의해 반응성이 지배되는 작은 분자들로부터 생겨났음에 틀림없다. RNA는 퓨린과 피리미딘 뉴클레오티드로 구성되는데, 둘 다 믿을 만한 정보 전달에 필요한 것으로 다윈의 자연 선택과 진화에 필요한 것이다. 남 [19]외 연구진은 리보핵산을 수용성 마이크로 드랍에 투여하기 위해 리보핵산을 포함한 뉴클레오바아제의 직접 응축 현상을 시연했는데, 이는 RNA 형성을 이끄는 핵심 단계다. 또한 베커 외 연구진은 습식 건조 주기를 이용해 피리미딘과 퓨린 리보뉴클레오티드 합성을 위한 그럴듯한 프리바이오틱 공정을 제시했다. [20]
역사
로잘린드 프랭클린의 X선 결정 이미지에서 DNA의 구조를 상세히 기술한 제임스 왓슨과 프랜시스 크릭의 랜드마크 논문 이전에, 그 발견에 기여한 몇몇 역사적 과학자들도 있었다.[21] 1869년에 처음으로 백혈구의 핵에서 핵물질을 분리하고 식별한 스위스의 의사 프리드리히 미셰르는 DNA 발견의 길을 열었다.[22] 미에세서스의 작업에 이어 독일의 생화학자인 알브레히트 코셀이 1878년 '뉴클레인'의 비단백 성분을 분리해 핵산에서 존재하는 아데닌, 시토신, 구아닌, 티민, 우라실 등 다섯 가지 뉴클레오바제를 발견했다.[23] 이러한 초기 발견으로 인해 핵산에 대한 몇몇 근본적인 사실이 알려졌지만, 핵산의 구조와 기능은 미스터리로 남아 있었다.
1919년 러시아-리투아니아 생화학자인 피버스 레베네에 의해 뉴클레오티드가 발견되기 전까지는 DNA 발견의 관문을 다시 열었다. 레베네는 처음에 효모 RNA에 존재하는 탄수화물 성분이 사실 리보스라는 것을 확인했다. 그러나 흉선핵산의 탄수화물 성분도 당분이지만 데옥시리보스라고 불리는 산소원자가 한 개 부족하다는 사실을 발견한 후에야 그의 발견은 과학계로부터 널리 인정받았다. 결국 레베네는 인산염-당분자 기반 장치인 RNA와 DNA의 성분이 합치는 정확한 순서를 확인할 수 있었고, 그 결과 후에 뉴클레오티드라고 불렀다. 뉴클레오티드 성분의 순서는 레베네에 의해 잘 이해되었지만, 우주에서의 뉴클레오티드 배열의 구조와 그 유전 코드는 그의 경력 초기에도 여전히 미스터리로 남아 있었다.[24]
참고 항목
- 리보뉴클레오시드 또는 리보시드
참조
- ^ a b c d Nelson, David (2008). Lehninger Principles of Biochemistry. W H Freeman and Co. pp. 272–273.
- ^ Newsholme, Eric A.; Leech, Anthony R.; Board, Mary (2008). Functional biochemistry in health & disease: metabolic regulation in health and disease (2nd ed.). Hoboken, N.J.: Wiley. ISBN 978-0-471-98820-5.
- ^ Das, Debajyoti (2010). Biochemistry. Bimal Kumar Dhur of Academic Publishers.
- ^ Cox, Michael M.; Nelson, David L. (2008). Principles of Biochemistry. W H Freeman & Co. ISBN 978-1-4292-2263-1.
- ^ Raymond, Kenneth W. (2010). General, organic, and biological chemistry: an integrated approach (3rd ed.). Hoboken, NJ: Wiley. ISBN 978-0-470-55124-0.
- ^ Schaechter, Moselio; Lederberg, Joshua, eds. (2004). The Desk Encyclopedia of Microbiology (1st ed.). Amsterdam: Elsevier Acad. Press. ISBN 0-12-621361-5.
- ^ Turner, Phil; et al. (2005). Molecular Biology. Instant Notes (3rd ed.). Boca Raton, FL: CRC, Taylor & Francis. ISBN 0-415-35167-7.
- ^ Nelson, David (2008). Lehninger Principles of Biochemistry. W H Freeman and Co. pp. 274–275.
- ^ Chauhan, Ashok K.; Varma, Ajit, eds. (2009). A textbook of molecular biotechnology. New Delhi: I.K. International Pub. House. ISBN 978-93-80026-37-4.
- ^ Cendra Mdel, M; Juárez, A; Torrents, E (2012). "Biofilm modifies expression of ribonucleotide reductase genes in Escherichia coli". PLOS ONE. 7 (9): e46350. Bibcode:2012PLoSO...746350C. doi:10.1371/journal.pone.0046350. PMC 3458845. PMID 23050019.
- ^ Campbell, Mary K.; Farrell, Shawn O. (2009). Biochemistry (7th ed.). Belmont, CA: Brooks/Cole Cengage Learning. ISBN 978-0-8400-6858-3.
- ^ Berg, Jeremy M.; Tymoczko, John L.; Stryer, Lubert (2007). Biochemistry (6th ed., 3rd print ed.). New York: Freeman. ISBN 978-0-7167-8724-2.
- ^ Kevin N. Kirouac, Zucai Suo, Hong Ling, Kevin N.; Suo, Zucai; Ling, Hong (1 April 2011). "Structural Mechanism of Ribonucleotide Discrimination by a Y-Family DNA Polymerase". Journal of Molecular Biology. 407 (3): 382–390. doi:10.1016/j.jmb.2011.01.037. PMID 21295588.
{{cite journal}}: CS1 maint : 복수이름 : 작성자 목록(링크) - ^ Nick McElhinny, SA; Kumar, D; Clark, AB; Watt, DL; Watts, BE; Lundström, EB; Johansson, E; Chabes, A; Kunkel, TA (October 2010). "Genome instability due to ribonucleotide incorporation into DNA". Nature Chemical Biology. 6 (10): 774–81. doi:10.1038/nchembio.424. PMC 2942972. PMID 20729855.
- ^ Nick McElhinny, SA; Watts, BE; Kumar, D; Watt, DL; Lundström, EB; Burgers, PM; Johansson, E; Chabes, A; Kunkel, TA (16 March 2010). "Abundant ribonucleotide incorporation into DNA by yeast replicative polymerases". Proceedings of the National Academy of Sciences of the United States of America. 107 (11): 4949–54. Bibcode:2010PNAS..107.4949N. doi:10.1073/pnas.0914857107. PMC 2841928. PMID 20194773.
- ^ Kasiviswanathan, R; Copeland, WC (Sep 9, 2011). "Ribonucleotide discrimination and reverse transcription by the human mitochondrial DNA polymerase". The Journal of Biological Chemistry. 286 (36): 31490–500. doi:10.1074/jbc.M111.252460. PMC 3173122. PMID 21778232.
- ^ a b c d Nelson, David (2008). Lehninger Principles of Biochemistry. W H Freeman and Co. pp. 881–894.
- ^ Berg, JM (2002). Biochemistry. Purine Bases can be Synthesized by de Novo or Recycled by Salvage Pathways. New York: W H Freeman. pp. Sec. 25.2.
- ^ Nam I, Nam HG, Zare RN. 수성 마이크로 드롭렛에 푸린과 피리미딘 리보뉴클레오시드의 아바이오틱 합성. Proc Natl Acad Sci 2018년 1월 2일:115(1):36-40. doi: 10.1073/pnas.1718559115. Epub 2017년 12월 18일. PMID: 29255025, PMCID: PMC5776833
- ^ 베커 S, 펠드만 J, 위데만 S, 오카무라 H, 슈나이더 C, 이완 K, 크리스프 A, 로사 M, 아마토프 T, 카렐 T. 피리미딘과 푸린 RNA 리보뉴클레오티드의 사전 생물학적으로 그럴싸한 합성을 통합했다. 과학 2019년 10월 4일 (6461년):76-82.도이:10.1126/과학.aax2747.ax2747. PMID: 31604305.
- ^ WATSON, JD; CRICK, FH (Apr 25, 1953). "Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid". Nature. 171 (4356): 737–8. Bibcode:1953Natur.171..737W. doi:10.1038/171737a0. PMID 13054692. S2CID 4253007.
- ^ Dahm, R (January 2008). "Discovering DNA: Friedrich Miescher and the early years of nucleic acid research". Human Genetics. 122 (6): 565–81. doi:10.1007/s00439-007-0433-0. PMID 17901982. S2CID 915930.
- ^ JONES, ME (September 1953). "Albrecht Kossel, a biographical sketch". The Yale Journal of Biology and Medicine. 26 (1): 80–97. PMC 2599350. PMID 13103145.
- ^ Levene, Phoebus (1919). The structure of yeast nucleic acid. Journal of Biological Chemistry 40(2). pp. 415–24.