유전자 코드
GENCODE| 내용 | |
|---|---|
| 묘사 | 유전자 및 유전자 변형 백과사전 |
| 데이터형 발동. | 인간과 생쥐 게놈의 모든 유전자 특징 |
| 연락 | |
| 연구소 | 웰컴 트러스트 생어 연구소 |
| 작가들 | 해로우 J 외 |
| 주요 인용문 | PMID 22955987 |
| 발매일 | 2012년 9월() |
| 접근 | |
| 웹 사이트 | 웹사이트 Gencode |
| 도구들 | |
| 웹 | UCSC Genome Browser: http://genome.cse.ucsc.edu/encode/ |
| 여러가지 종류의 | |
| 면허증. | 오픈 액세스 |
| 데이터 릴리즈 빈도수. | 휴먼 - 분기 마우스 - 반기 |
| 버전 | 인간 - 릴리스 37 (2021년 2월) 마우스 - 릴리즈 M26 (2021년 2월) |
GENCODE는 게놈 연구에 관한 과학적 프로젝트이며 ENCYclopedia Of DNA Elements(ENCYclopedia Of DNA Elements) 스케일업 프로젝트의 일부입니다.
GENCODE 컨소시엄은 처음에 ENCODE 영역(인간 [2]게놈의 약 1%) 내의 모든 단백질 코드 유전자를 식별하고 매핑하기 위한 ENCODE 프로젝트의 파일럿 단계의 일부로 구성되었다.프로젝트의 초기 성공을 감안할 때, GENCODE는 이제 "유전자 및 유전자 [2]변형 백과사전"을 구축하는 것을 목표로 하고 있다.
결과는 대체적으로 전사된 [3]변형을 가진 모든 단백질 코드화 위치, 전사 증거를 가진 비 코드화 위치[4] 및 [5]유사 유전자를 포함하는 주석 집합이 될 것이다.
현재 진행 상황
GENCODE는 현재 프로젝트 [6]단계 2에서 목표를 향해 나아가고 있습니다.
인간 유전자 집합 주석의 가장 최근 공개는 Gencode 36이며 동결일은 2020년 12월이다.이 릴리스는 최신 GRCh38 인간 참조 게놈 [7]어셈블리를 사용합니다.
마우스 유전자 세트 주석의 최신 릴리스는 Gencode M25이며,[7] 동결 날짜도 2020년 12월이다.
2009년 9월부터 GENCODE는 Ensembl 프로젝트에서 사용되는 인간 유전자 세트이며, 각각의 새로운 GENCODE 릴리스는 Ensembl [8]릴리스에 대응하고 있다.
역사
2003년 9월
이 프로젝트는 파일럿 단계, 테크놀로지 개발 단계 [9]및 프로덕션 단계의 3가지 단계로 설계되었습니다.ENCODE 프로젝트의 파일럿 단계는 인간 게놈의 약 1%에 해당하는 총 30Mb의 44개 영역을 계산 및 실험적으로 매우 깊이 조사하는 것을 목표로 했다.이 단계의 일부로, ENCODE 영역 [2]내의 모든 단백질 코드 유전자를 식별하고 매핑하기 위해 GENCODE 컨소시엄이 구성되었다.처음 두 단계의 결과는 비용 효율적이고 포괄적인 생산 [9]단계에서 인간 게놈의 나머지 99%를 분석하기 위한 최선의 경로를 결정하는 데 사용될 것으로 예상되었다.
2005년 4월
44개의 ENCODE 영역의 주석의 첫 번째 릴리스는 2005년 4월 29일에 동결되었으며, 첫 번째 ENCODE 게놈 주석 평가 프로젝트(E-GASP)[2] 워크숍에서 사용되었다.GENCODE Release 1은 416개의 알려진 위치, 26개의 신규(코드 DNA 배열) CDS 위치, 82개의 신규 전사 위치, 78개의 추정 위치, 104개의 처리된 의사 유전자 및 66개의 미처리 의사 유전자를 포함했다.
2005년 10월
두 번째 버전(릴리스 02)은 2005년 10월 14일에 동결되었으며, RACE 및 RT-PCR [2]기술을 사용한 실험 검증에서 발견한 이후 업데이트가 포함되어 있다.GENCODE 릴리스 2에는 411개의 알려진 위치, 30개의 새로운 CDS 위치, 81개의 새로운 전사 위치, 83개의 추정 위치, 104개의 처리된 의사 유전자 및 66개의 미처리 의사 유전자가 포함되어 있었다.
2007년 6월
파일럿 프로젝트의 결론은 2007년 [10]6월에 발표되었습니다.연구 결과는 인간 게놈의 기능적 요소를 특징짓기 위한 실현 가능한 플랫폼과 새로운 기술을 만드는 파일럿 프로젝트의 성공을 강조하여 게놈 전체에 걸친 연구에 대한 연구의 길을 열었다.
2007년 10월
새로운 자금은 추가 파일럿 규모의 연구와 함께 전체 게놈의 생산 단계로 ENCODE 프로젝트를 확대하려는 NHGRI의 노력의 일부였다.
2012년 9월
2012년 9월, GENCODE 컨소시엄은 2011년 [11]12월에 동결된 GENCODE Release 7이라는 메이저 릴리스의 결과를 설명하는 주요 논문을 발표했습니다.
2018
2018년에 GENCODE 프로젝트에 가장 최근에 추가된 것 중 하나는 인간과 모델 유기체 어셈블리의 CRISPR/Cas9 트랙이었습니다.CRISPR은 높은 특이성으로 편집된 영역에 성공적으로 결합하는 RNA의 염기서열을 사용하는 게놈 편집 기법이다.새로운 트랙은 전사된 영역 옆에 있거나 200bp 이내에 있는 CRISPR/Cas9 복합체에 대한 잠재적 바인딩 사이트를 나열함으로써 적절한 가이드 문장을 찾는 데 도움을 주기 위해 설계되었습니다.각 현장에 대해 트랙은 가능한 가이드 시퀀스를 해당 가이드 시퀀스에 대한 예측 효율성 및 특이성 점수와 함께 제공합니다.또한, 오프타겟과 [11]가이드 사이의 미스매치 수로 그룹화된 잠재적인 오프타겟에 대한 정보도 제공합니다.
2020
다른 성과 중 마우스 참조 게놈의 첫 번째 패스 수동 주석을 완료했고, 주석 수렴 달성을 위해 RefSeq 및 Uniprot 참조 주석 데이터베이스와의 협력을 시작했으며, lncRNA의 주석은 기존 위치에 있는 새로운 위치 및 새로운 스크립트를 발견함으로써 개선되었다.g loci. 또한 2020년 COVID-19 대유행으로 인해 상황에 대응하는 연구를 지원하고 싶은 충동이 있어 GENCODE는 SARSCoV-2 [12]감염과 관련된 단백질 코드 유전자 세트의 주석을 검토하고 개선했다.
주요 참가자
GENCODE 프로젝트의 주요 참가자는 다양한 단계에서 비교적 일관성을 유지하고 있으며, 현재 Wellcome Trust Sanger Institute가 프로젝트 전반을 주도하고 있습니다.
각 단계의 주요 참여 기관의 개요를 [6][13]아래에 나타냅니다.
| GENCODE 단계 2(현재) | GENCODE 스케일업 단계 | GENCODE 파일럿 단계 | |
|---|---|---|---|
| Wellcome Trust Sanger Institute, 영국 케임브리지 | Wellcome Trust Sanger Institute, 영국 케임브리지 | Wellcome Trust Sanger Institute, 영국 케임브리지
| |
| Centre de Regulacio Genmmica(CRG), 바르셀로나, 카탈로니아, 스페인 | Centre de Regulacio Genmmica(CRG), 바르셀로나, 카탈로니아, 스페인 | 스페인 바르셀로나, 카탈로니아, IMIM(Investigacio Médica) 시립 기관 | |
| 스위스 로잔 대학교 | 스위스 로잔 대학교 | 스위스 제네바 대학교 | |
| 캘리포니아 대학교 산타크루즈(UCSC), 캘리포니아, 미국 | 캘리포니아 대학교(UCSC), 미국 산타크루즈 | 워싱턴 대학교(WashU), 미국 세인트루이스 | |
| 매사추세츠 공과대학(MIT), 미국 보스턴 | 매사추세츠 공과대학(MIT), 미국 보스턴 | 캘리포니아 대학교 버클리, 미국 | |
| 미국 뉴헤이븐의 예일 대학교(예일) | 미국 뉴헤이븐의 예일 대학교(예일) | 영국 힌스턴 유럽생물정보학연구소 | |
| 스페인 국립암연구센터(CNIO), 스페인 마드리드 | 스페인 국립암연구센터(CNIO), 스페인 마드리드 | ||
| 워싱턴 대학교(WashU), 미국 세인트루이스 |
참가자, PI 및 CO-PI
- Paul Flicek (리드PI), EMBL 유럽생물정보학연구소, 영국 케임브리지
- 스페인 바르셀로나, 카탈로니아, CRG(Centre de Regulacio Genicamica), Roderic Guigo(PI), Centre de Regulacio Genmmica(CRG)
- Massachusetts Institute of Technology(MIT), 미국 보스턴, Manolis Kellis(PI)
- 마크 거스타인(PI), 미국 뉴헤이븐 예일대
- 베네딕트 패튼(PI), 캘리포니아 산타크루즈 캘리포니아 대학, 미국
- 스페인 마드리드 국립암연구센터(CNIO), Michael Tress씨
- Jyoti Choudhary, 암연구소(ICR), 영국 런던
주요 통계 정보
GENCODE는 초기부터 36가지 버전의 인간 유전자 세트 주석을 출시했다(소소한 업데이트 제외).
최신 GENCODE 인간 유전자 세트 주석의 주요 요약 통계(공개 36, 2020년 12월 동결)는 [14]다음과 같다.
| 분류 | 총 | 분류 | 총 |
|---|---|---|---|
| 유전자 총수 | 60,660 | 총 성적표 수 | 232,117 |
| 단백질 코드화 유전자 | 19,962 | 단백질 코드 트랜스크립트 | 85,269 |
| 긴 비코드 RNA 유전자 | 17,958 | - 전체 길이 단백질 분해: | 59,269 |
| 코드화되지 않은 작은 RNA 유전자 | 7,569 | - 부분 길이 단백질 매개체: | 26,000 |
| 유사 유전자 | 14,761 | 넌센스 매개 붕괴 기록 | 17,378 |
| - 처리된 의사 유전자: | 10,669 | 긴 비코드 RNA 위치 전사 | 48,734 |
| - 처리되지 않은 의사 발생: | 3,554 | ||
| - 유니터리 의사 유전자: | 236 | ||
| - 다형성 유사유전자: | 48 | ||
| - 의사 발생: | 18 | ||
| 면역글로불린/T세포수용체 유전자 세그먼트 | 645 | 개별 번역 합계 수 | 63,058 |
| - 단백질 코딩 세그먼트: | 409 | 하나 이상의 다른 번역이 있는 유전자 | 13,685 |
| - 의사 발생: | 236 |
시퀀스 기술(RT-PCR-seq 등)의 진보, 수동 주석(HAVANA 그룹)의 적용 범위 증가, Ensembl을 사용한 자동 주석 알고리즘의 개선을 통해 GENCODE 주석의 정확성과 완성도는 릴리스 반복을 통해 지속적으로 개선되었다.
2014년까지의 3대 GENCODE 릴리스의 주요 통계 비교는 [14]다음과 같습니다.발견된 총 유전자 수에서 커버리지가 꾸준히 증가하고 있지만, 실제로 단백질 코드화 유전자의 수는 감소하고 있는 것이 분명하다.이는 주로 캡 분석 유전자 발현(CAGE) 클러스터, 주석이 달린 PolyA 부위 및 펩타이드 [11]적중 등을 사용하여 얻은 새로운 실험 증거에 기인한다.
- 버전 7 (2010년 12월 동결, GRCh37) - Ensembl
- 버전 10 (2011년 7월 동결, GRCh37) - 앙상블 65
- 버전 20(2014년 4월 동결, GRCh38) - 앙상블 76
방법론
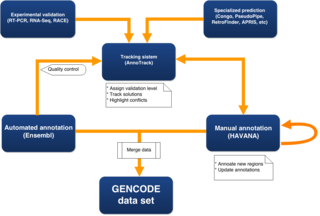
추정 궤적은 습식 실험실 실험에 의해 검증될 수 있으며 계산 예측은 수동으로 [15]분석된다.현재 주석 세트가 수동으로 주석을 붙인 영역뿐만 아니라 전체 게놈을 커버하도록 하기 위해 합병된 데이터 세트가 자동으로 주석을 붙인 Ensembl 유전자 세트의 자동 주석과 함께 아바나의 수동 주석을 사용하여 생성된다.이 과정은 또한 Ensembl 단백질 코딩 세트의 고유한 전체 길이 CDS 예측을 수동으로 주석이 달린 유전자에 추가하여 [16]게놈의 가장 완전하고 최신 주석을 제공한다.
자동 주석(Ensubl)
앙상블 전사는 앙상블 유전자 구축이라고 불리는 앙상블 자동 유전자 주석 시스템(유전자 주석 파이프라인의 집합)의 산물이다.모든 앙상블 성명은 실험 증거를 기반으로 하므로 자동화된 파이프라인은 과학계의 공공 데이터베이스에 [17]축적된 mRNA와 단백질 시퀀스에 의존한다.
수동 주석(HAVANA 그룹)
GENCODE 컨소시엄에는 수동 주석자가 비주석 영역에서 모델을 생성하는 데 도움이 되는 파이프라인을 운영하고, 완전히 누락된 위치, 누락된 대체 등형식, 잘못된 접합 부위 및 잘못된 생체 유형을 포함하여 잠재적으로 누락되거나 잘못된 수동 주석을 식별하는 여러 분석 그룹이 있다.이러한 정보는 AnnoTrack 추적 [18]시스템을 사용하여 수동 주석으로 피드백됩니다.이러한 파이프라인 중 일부는 RNASeq 데이터, 히스톤 수정, CAGE 및 Ditag 데이터 등 다른 ENCODE 하위 그룹의 데이터를 사용합니다.RNAseq 데이터는 새로운 증거의 중요한 원천이지만, 그것으로부터 완전한 유전자 모델을 생성하는 것은 어려운 문제이다.GENCODE의 일환으로 다양한 RNAseq 예측 파이프라인에서 생성되는 예측의 품질을 평가하기 위한 경합이 실시되었다(아래의 RGASP 참조).불확실한 모델을 확인하기 위해 GENCODE에는 RNA 시퀀싱 및 [16]RACE를 사용하는 실험 검증 파이프라인이 있습니다.
품질 평가
GENCODE 7의 경우,[2] 트랜스크립트 모델은 트랜스크립트의 품질을 평가하기 위해 개발된 새로운 방법에 따라 높은 수준 또는 낮은 수준의 지원이 할당됩니다.
사용/접근
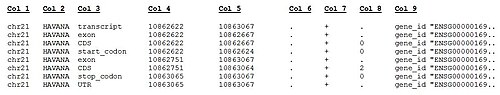
현재 GENCODE 인간 유전자 세트 버전(GENCODE Release 20)은 주석 파일(GTF 및 GFF3 형식), FASTA 파일 및 METADA를 포함한다.모든 게놈 영역의 GENCODE 주석과 관련된 TA 파일(참조 염색체/패치/스카폴드/하플로타입).주석 데이터는 참조 염색체에 참조되며 다음을 포함하는 분리된 파일에 저장됩니다.유전자 주석, PolyA는 HAVANA에 의해 주석이 붙여진 (Retroposed) 유사유전자가 예일 및 UCSC 파이프라인에 의해 예측되지만 HAVAN, 긴 비코드 RNA 및 tRNA-Scan에 의해 예측된 tRNA 구조에 의해 예측되지 않는다.GTF 형식의 행의 예를 다음에 나타냅니다.
GENCODE GTF 파일 형식 내의 컬럼은 다음과 같습니다.
GENCODE GTF 파일의 형식 설명.탭으로 구분된 표준 GTF 열
| 열번호 | 내용 | 값/형식 |
|---|---|---|
| 1 | 염색체명 | chr{1,2,3,4,6,7,8,9,10,11,12,13,14,15,16,17,18,19,20,21,22,X,Y,M} |
| 2 | 주석원 | {ENSAMBL,HAVANA} |
| 3 | 기능형 | {gene, transcript, exon, CDS, UTR, start_codon, stop_codon, Selenocysteine} |
| 4 | 게놈 시작 위치 | 정수값(1 베이스) |
| 5 | 게놈 말단 위치 | 정수값 |
| 6 | 점수(미사용) | . |
| 7 | 게놈사슬 | {+,-} |
| 8 | 게놈 단계(CDS 기능의 경우) | {0,1,2,.} |
| 9 | 키와 값의 쌍으로서의 추가 정보 | 아래 표의 설명을 참조하십시오. |
GENCODE GTF 파일의 9번째 열에 있는 키와 값의 쌍에 대한 설명(형식: 키 "값")
| 키명 | 값 형식 |
|---|---|
| 유전자_id | ENSGXXXXXXXXXXXXXXXXXX |
| transcript_id | enstXXXXXXXXXXXXXXXXXX |
| 유전자형 | 생물형 목록 |
| 유전자 상태 | {알고 있는 것, 노벨, 추정} |
| 유전자명 | 스트링 |
| transcript_type | 생물형 목록 |
| transcript_status | {알고 있는 것, 노벨, 추정} |
| transcript_name | 스트링 |
| exon_number | 성적표에서 엑손의 생물학적 위치를 나타내는 |
| exon_id | 엔섹스XXXXXXXXXXXXXXXXXX |
| 수평 |
|
바이오 얼라이언스 게놈 브라우저
또한 GENCODE 웹 사이트에는 인간과 마우스용 Genome Browser(게놈 브라우저)가 포함되어 있어 염색체 번호와 시작 위치(예: 22:30,700,000)를 지정하면 모든 게놈 영역에 도달할 수 있습니다.30,900,000), ENS 전사 ID(버전 있음/없음), ENS 유전자 ID(버전 있음/없음) 및 유전자 이름.브라우저는 바이오 얼라이언스 [19]기능을 탑재하고 있습니다.
과제들
'유전자'의 정의
"유전자"의 정의는 인간 게놈이 발견된 이후 몇 년 동안 수많은 정의와 개념들이 제안되면서 결코 사소한 문제가 아니었다.우선 유전자는 1900년대에 유전의 별개의 단위로 생각되어 단백질 합성의 청사진으로 생각되었고, 최근에는 RNA로 변환되는 유전자 코드로 정의되고 있다.비록 유전자의 정의는 지난 세기에 크게 발전했지만, 많은 연구자들에게는 도전적이고 논쟁적인 주제로 남아있었다.ENCODE/GENCODE 프로젝트의 등장으로 대체 스플라이싱(일련의 엑손이 인트론에 의해 분리됨), 유전자간 전사 및 분산조절의 복잡한 패턴, 비유전자 보존 및 비부호화 RNA g의 풍부함을 포함한 정의의 훨씬 더 문제가 있는 측면이 밝혀졌다.유전자와 유전자 변형에 대한 백과사전을 구축하기 위해 GENCODE가 노력함에 따라,[20] 이러한 문제들은 유전자의 업데이트된 개념을 생각해내는데 GENCODE 프로젝트에 대한 도전의 강도를 높였다.
인간 게놈 프로젝트
인간 게놈 프로젝트는 인간 게놈의 염기서열을 결정하고 그 안에 포함된 유전자를 식별하기 위한 국제적인 연구였다.이 프로젝트는 미국 국립보건원과 미국 에너지부에 의해 조정되었습니다.미국 전역의 대학과 영국, 프랑스, 독일, 일본 및 중국의 국제 파트너도 추가 기여했습니다.인간 게놈 프로젝트는 1990년에 공식적으로 시작되었고 원래 [21]일정보다 2년 앞당겨진 2003년에 완성되었다.
서브프로젝트
앙상블
lncRNA Expression 마이크로어레이 설계
GENCODE 프로젝트의 핵심 연구 분야는 긴 비코드 RNA(lncRNA)의 생물학적 의미를 조사하는 것이었다.인간의 lncRNA 표현을 더 잘 이해하기 위해 GENCODE에 의해 하위 프로젝트가 생성되어 GENCODE lncRNA [4]주석의 스크립트를 수량화할 수 있는 커스텀 마이크로 어레이 플랫폼을 개발했습니다.애질런트테크놀로지스 eArray 시스템을 사용하여 여러 가지 설계가 생성되었으며, 이러한 설계는 표준 사용자 정의 애질런트 [4]형식으로 제공됩니다.
RGASP
RNA-seq Genome Annotation Assessment Project(RGASP) 프로젝트는 고품질 RNA-시퀀스 데이터 분석을 위한 다양한 계산 방법의 효과를 평가하기 위해 설계되었습니다.RGASP의 주요 목표는 RNA-seq 정렬, 전사 특성화(발견, 재구성 및 정량화) 소프트웨어에 대한 편견 없는 평가를 제공하고 전사체 [23]염기서열 분석을 기반으로 자동화된 게놈 주석의 실현 가능성을 결정하는 것이다.
RGASP는 EGASP(Encode Genome Annotation Assessment Project) 유전자 예측 워크숍 이후 모델링된 컨소시엄 프레임워크로 조직되며, RNA-seq 분석의 다른 측면과 시퀀스 기술 및 형식의 변화를 다루기 위해 두 차례의 워크숍이 수행되었다.프로젝트의 1차 및 2차에서 발견한 주요 사항 중 하나는 생성된 유전자 예측 품질에 대한 읽기 정렬의 중요성입니다.따라서,[23] 게놈에 대한 읽기 매핑에 주로 초점을 맞춘 3차 RGASP 워크숍이 현재(2014년) 진행 중이다.
「 」를 참조해 주세요.
레퍼런스
- ^ Harrow J, Frankish A, Gonzalez JM, Tapanari E, Diekhans M, Kokocinski F, et al. (September 2012). "GENCODE: the reference human genome annotation for The ENCODE Project" (PDF). Genome Research. 22 (9): 1760–74. doi:10.1101/gr.135350.111. PMC 3431492. PMID 22955987.
- ^ a b c d e f Harrow J, Denoeud F, Frankish A, Reymond A, Chen CK, Chrast J, et al. (2006). "GENCODE: producing a reference annotation for ENCODE". Genome Biology. 7 (Suppl 1): S4.1–9. doi:10.1186/gb-2006-7-s1-s4. PMC 1810553. PMID 16925838.
- ^ Frankish A, Mudge JM, Thomas M, Harrow J (2012). "The importance of identifying alternative splicing in vertebrate genome annotation". Database. 2012: bas014. doi:10.1093/database/bas014. PMC 3308168. PMID 22434846.
- ^ a b c Derrien T, Johnson R, Bussotti G, Tanzer A, Djebali S, Tilgner H, et al. (September 2012). "The GENCODE v7 catalog of human long noncoding RNAs: analysis of their gene structure, evolution, and expression". Genome Research. 22 (9): 1775–89. doi:10.1101/gr.132159.111. PMC 3431493. PMID 22955988.
- ^ Pei B, Sisu C, Frankish A, Howald C, Habegger L, Mu XJ, et al. (September 2012). "The GENCODE pseudogene resource". Genome Biology. 13 (9): R51. doi:10.1186/gb-2012-13-9-r51. PMC 3491395. PMID 22951037.
- ^ a b "GENCODE - Homepage". 20 December 2020.
- ^ a b "GENCODE – Data". GENCODE. Wellcome Trust Sanger Institute. September 2019. Retrieved 14 October 2019.
- ^ a b "GENCODE". Wellcome Trust Sanger Institute. p. The GENCODE Project: Encyclopædia of genes and gene variants. Retrieved 20 December 2020.
{{cite web}}: CS1 maint :url-status (링크) - ^ a b The ENCODE Project Consortium (October 2004). "The ENCODE (ENCyclopedia Of DNA Elements) Project". Science. 306 (5696): 636–40. Bibcode:2004Sci...306..636E. doi:10.1126/science.1105136. PMID 15499007. S2CID 22837649.
- ^ Birney E, Stamatoyannopoulos JA, Dutta A, Guigó R, Gingeras TR, Margulies EH, et al. (June 2007). "Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project". Nature. 447 (7146): 799–816. Bibcode:2007Natur.447..799B. doi:10.1038/nature05874. PMC 2212820. PMID 17571346.
- ^ a b c Casper J, Zweig AS, Villarreal C, Tyner C, Speir ML, Rosenbloom KR, et al. (January 2018). "The UCSC Genome Browser database: 2018 update". Nucleic Acids Research. 46 (D1): D762–D769. doi:10.1093/nar/gkx1020. PMC 5753355. PMID 29106570.
- ^ Frankish A, Diekhans M, Jungreis I, Lagarde J, Loveland JE, Mudge JM, et al. (December 2020). "GENCODE 2021". Nucleic Acids Research. 49 (D1): D916–D923. doi:10.1093/nar/gkaa1087. PMC 7778937. PMID 33270111. S2CID 227260109.
- ^ "GENCODE Project Participants". Genome BioInformatics Research Lab. Genome BioInformatics Research Lab. c. 2005. Retrieved 8 September 2014.
- ^ a b "GENCODE – Statistics". GENCODE. Wellcome Trust Sanger Institute. c. 2014. Archived from the original on 20 December 2020. Retrieved 20 December 2020.
- ^ "GENCODE – Goals". GENCODE. Wellcome Trust Sanger Institute. c. 2013. Retrieved 5 September 2014.
- ^ a b Searle S, Frankish A, Bignell A, Aken B, Derrien T, Diekhans M, et al. (2010). "The GENCODE human gene set". Genome Biology. 11 (Suppl 1): 36. doi:10.1186/gb-2010-11-S1-P36. PMC 3026266.
- ^ "Ensembl - Homepage". Ensembl. August 2014. Retrieved 6 September 2014.
- ^ Kokocinski F, Harrow J, Hubbard T (October 2010). "AnnoTrack--a tracking system for genome annotation". BMC Genomics. 11: 538. doi:10.1186/1471-2164-11-538. PMC 3091687. PMID 20923551.
- ^ "Biodalliance - Homepage". 20 December 2020.
- ^ Gerstein MB, Bruce C, Rozowsky JS, Zheng D, Du J, Korbel JO, et al. (June 2007). "What is a gene, post-ENCODE? History and updated definition". Genome Research. 17 (6): 669–81. doi:10.1101/gr.6339607. PMID 17567988.
- ^ "Human Genome Project - Homepage". 20 December 2020.
- ^ "ENCODE data in Ensembl". Ensembl. August 2014. Retrieved 7 September 2014.
- ^ a b Steijger T, Abril JF, Engström PG, Kokocinski F, Hubbard TJ, Guigó R, et al. (December 2013). "Assessment of transcript reconstruction methods for RNA-seq". Nature Methods. 10 (12): 1177–84. doi:10.1038/nmeth.2714. PMC 3851240. PMID 24185837.