프로테아솜
Proteasome프로테아솜은 펩타이드 결합을 깨는 화학 반응인 프로테롤리시스(Proteolyis)에 의해 불필요하거나 손상된 단백질을 분해하는 단백질 복합체다.그러한 반응을 돕는 효소를 프로테아제라고 부른다.
프로테아솜은 세포가 특정 단백질의 농도를 조절하고 잘못 접힌 단백질을 분해하는 주요 메커니즘의 일부다.단백질은 유비퀴틴이라고 불리는 작은 단백질로 분해의 꼬리표가 붙는다.태깅 반응은 유비퀴틴 리깅스라고 불리는 효소에 의해 촉매된다.일단 단백질이 하나의 유비퀴틴 분자로 태그가 붙으면, 이것은 추가 유비퀴틴 분자를 붙이기 위한 다른 묶음들에 대한 신호다.그 결과 프로테아솜에 묶인 폴리우비퀴틴 체인으로 태그가 붙은 단백질을 분해할 수 있게 된다.[1]분해 과정은 약 7~8개의 아미노산 펩타이드의 길이를 산출하는데, 이것은 더 짧은 아미노산 염기서열로 분해되어 새로운 단백질을 합성하는데 사용될 수 있다.[1]
프로테아솜은 모든 진핵생물들과 고고학자들 안에서 그리고 몇몇 박테리아에서 발견된다.진핵생물에서 프로테아솜은 핵과 세포질 양쪽에 위치한다.[2]
구조상 프로테아솜은 중심공공을 형성하는 4개의 쌓인 고리의 "핵심"이 들어 있는 원통형 복합체다.각각의 고리는 7개의 개별 단백질로 구성되어 있다.내부 2개의 링은 3-7개의 프로테아제 활성 부위가 포함된 7개의 β 서브유닛으로 제작된다.이러한 부위는 링의 내부 표면에 있으므로 대상 단백질이 분해되기 전에 중심 공극으로 들어가야 한다.바깥쪽 두 개의 고리는 각각 7 α 서브유닛을 포함하고 있는데, 그 기능은 단백질이 통으로 들어가는 "게이트"를 유지하는 것이다.이러한 α 서브유닛은 단백질 기판에 부착된 폴리우비퀴틴 태그를 인식하고 분해 과정을 시작하는 구조 또는 규제 입자에 "캡"하여 제어된다.편재와 단백질 분해의 전반적인 시스템은 편재-단백질 계통이라고 알려져 있다.[3]
단백질 분해 경로는 세포 주기, 유전자 발현 조절, 산화 스트레스에 대한 반응 등 많은 세포 과정에 필수적이다.세포 내 단백질 분해의 중요성과 단백질 분해 경로에서 유비퀴틴의 역할은 아론 시차노버, 에이브람 허쉬코, 어윈 로즈에게 수여된 2004년 노벨 화학상 시상식에서 인정되었다.[4]
디스커버리
유비퀴틴-단백질 체계가 발견되기 전에는 세포의 단백질 분해는 주로 리소솜, 산성과 단백질 분해효소가 가득한 내부를 가진 막 결합형 유기체에 의존하여 외부 단백질과 노화되거나 손상된 유기체를 분해하여 재활용할 수 있다고 생각되었다.[1]그러나 1977년 조셉 에틀링거와 알프레드 골드버그가 리소솜이 부족한 망막세포의 ATP 의존성 단백질 분해에 관한 연구에서는 제2 세포내 분해 메커니즘의 존재를 시사했다.[5]이것은 1978년에 여러 개의 뚜렷한 단백질 체인으로 구성되어 있다는 것을 보여주었는데, 이것은 당시 프로테아제들 사이에서 신기했다.[6]히스톤의 변경에 대한 이후의 연구는 히스톤의 리신 측 사슬과 알려진 기능이 없는 단백질인 유비퀴틴의 C-단자 글리신 잔류물 사이의 결합에 의해 히스톤 단백질의 예기치 않은 공동 가치 변형을 확인하게 되었다.[7]그 후 이전에 확인된 단백질 분해 인자 1(ATP 의존적 단백질 분해 인자 1 (APF-1)로 알려진 단백질 분해 단백질 분해 단백질은 유비퀴틴과 동일한 단백질이라는 것이 밝혀졌다.[8]이 체계의 단백질 분해 활동은 셔윈 윌크와 마리온 오를로우스키에 의해 원래 다단백질 단백질 분해효소 복합체라고 불리던 다단백질 복합체로서 격리되었다.[9]이후 유비퀴틴 의존성 단백질 분해의 원인이 되는 ATP 의존 단백질 분해 복합체가 발견되어 26S 단백질 분해 물질로 불리게 되었다.[10][11]
유비퀴틴 프로테아솜 시스템의 발견에 이르는 초기 작업의 대부분은 1970년대 후반과 1980년대 초반에 아론 시차노버가 대학원생으로 일했던 아브람 헤르쉬코 연구소의 테크니온에서 일어났다.폭스 체이스 암 센터의 어윈 로즈 연구실에서 1년 동안 진행된 허쉬코의 안식년차는 중요한 개념적 통찰력을 제공했지만, 로즈는 나중에 이 발견에서 자신의 역할을 경시했다.[12]이 세 사람은 이 시스템을 발견한 공로로 2004년 노벨 화학상을 공동 수상했다.[4]
프로테아좀의 적층 고리 구조를 보여주는 전자 현미경 데이터가 1980년대 중반에 입수되었지만 프로테아솜 코어 입자의 첫 번째 구조는 1994년까지 X선 결정학으로 해결되지 않았다.[13][14]2018년에는 다극성 단백질 기질이 있는 복합체에서 인간 26S 프로테아솜 홀로엔자임(Holoenozyme)의 첫 원자구조를 극저온 전자현미경 검사로 해결하여 기질이 인간 26S 프로테아솜에 의해 인식, 디비큐레이션, 펼쳐지고 분해되는 메커니즘을 밝혀냈다.[15]
구조 및 조직
프로테아솜 하위 성분은 흔히 스베드베르크 침전 계수(S로 표시됨)로 언급된다.포유류에게 가장 독점적으로 사용되는 프로테아솜은 세포질 26S 프로테아솜으로 20S 단백질 서브유닛 1개와 19S 규제 캡 서브유닛 2개를 포함하는 분자질량 약 2000킬로달톤(kDa)이다.코어는 속이 비어서 단백질이 분해되는 밀폐된 공동체를 제공한다. 코어의 양쪽 끝에서 열린 구멍은 대상 단백질이 들어갈 수 있게 한다.코어 입자의 각 끝은 여러 개의 ATPase 활성 사이트와 유비퀴틴 결합 사이트를 포함하는 19S 규제 하위 장치와 연결된다. 이 구조는 다극성 단백질을 인식하여 촉매 코어로 전달하는 것이다.[15]11S 입자라고 불리는 다른 형태의 규제 하위 장치는 19S 입자와 본질적으로 같은 방식으로 노심과 연관될 수 있다; 11S는 바이러스에 의해 감염된 후 생성된 것과 같은 외국 펩타이드의 분해에 역할을 할 수 있다.[16]
20S 코어 입자
20S 코어 입자에 포함된 서브유닛의 수와 다양성은 유기체에 따라 달라진다; 구별되고 특화된 서브유닛의 수는 단세포 유기체보다 다세포적으로, 원핵생물보다 진핵생물에서 더 크다.모든 20S 입자는 서로 다른 두 가지 유형의 서브유닛으로 구성된 4개의 스택형 헵탄 링 구조로 구성된다. α 서브유닛은 본질적으로 구조적인 반면 β 서브유닛은 주로 촉매적인 구조로 구성된다.α 서브유닛은 β 서브유닛에 동질성이 있는 유사효소다.그것들은 β 서브유닛에 인접한 N-termini와 조립된다.[17]스택의 외부 2개 링은 각각 7 α 서브유닛으로 구성되며, 이는 규제 입자의 도킹 영역 역할을 하며 알파 서브유닛 N-termini(Pfam PF10584)는 내부 공동에 대한 비규제되지 않은 기판의 접근을 차단하는 게이트를 형성한다.[18]내측 2개의 링은 각각 7개의 β 서브유닛으로 구성되며, 이들의 N-termini에는 프로테롤리시스 반응을 수행하는 프로테아제 활성 부위가 포함되어 있다.[19]정제 복합체에서 세 가지 뚜렷한 촉매 활성(Chymotrypsin 유사, 트립신 유사, 펩티딜글루타밀-펩타이드 가수 분해)이 확인되었다.[20]프로테아솜의 크기는 비교적 보존되어 있으며 115 å까지 약 150 앵스트롬( ()이다.내부 챔버는 입구가 13 å 정도로 좁을 수 있지만, 최대 53 wide의 넓이로 기질 단백질이 들어가려면 최소한 부분적으로 펼쳐져야 함을 시사한다.[21]
테르모플라즈마산소필룸과 같은 고고학에서는 모든 α와 모든 β 서브유닛이 동일한 반면 효모에 들어 있는 것과 같은 진핵 프로테아솜은 각 서브유닛의 7가지 뚜렷한 유형을 포함하고 있다.포유류에서 β1, β2 및 β5 서브유닛은 촉매제가 된다. 공통 메커니즘을 공유하지만, 키모트립신 유사, 트립신 유사, 펩티딜-글루타밀 펩타미드-하이드로라이징(PHGH)으로 간주되는 세 가지 뚜렷한 기질 특이성이 있다.[22]β1i, β2i, β5i로 표시된 대체 β형태는 특히 인터페론 감마선과 같은 친염증 신호에 대한 노출에 대응하여 조혈세포로 표현할 수 있다.이러한 대체 서브유닛과 함께 조립된 프로테아솜은 면역단백질이라고 알려져 있는데, 이 면역단백질들의 기질특성은 정상 프로테아솜에 비해 변화된다.[21]최근 α3 코어 부위가 없는 인간 세포에서 대체 단백질 물질이 확인되었다.[23]이러한 프로테아솜(α4-α4 프로테아솜으로 알려져 있음)은 대신에 누락된 α3 서브 유닛 대신 추가 α4 서브 유닛을 포함하는 20S 코어 입자를 형성한다.이러한 대체 'α4-α4' 프로테아솜은 이전에 효모 속에 존재하는 것으로 알려져 있다.[24]비록 이러한 프로테아솜의 정확한 기능은 아직 대부분 알려져 있지 않지만, 이러한 프로테아솜을 발현하는 세포들은 카드뮴과 같은 금속 이온에 의해 유발되는 독성에 대한 내성을 강화했다.[23][25]
19S 규제 입자
진핵생물의 19S 입자는 19개의 개별 단백질로 구성되며, 2개의 횡단구성요소, 20S 코어 입자의 α 링에 직접 결합하는 9개 서브 유닛 베이스, 10개 서브 유닛 리드로 분할된다.9가지 기본 단백질 중 6개는 AAA 계열의 ATPase 하위 단백질이며, 이러한 ATPases의 진화적 호몰로로그는 PAN(단백질-활성화 뉴클레오티아제)이라고 불리는 고고학계에 존재한다.[26]19S와 20S 입자의 연결은 19S ATPase 하위 단위에 ATP의 결합이 필요하며, 조립된 복합체가 접혀지고 보편화된 단백질을 분해하기 위해서는 ATP 가수분해가 필요하다.기질 전개 단계만 ATP 가수분해로부터 에너지를 필요로 하는 반면, ATP 결합만으로 단백질 분해에 필요한 다른 모든 단계(예: 복잡한 조립, 게이트 개방, 변환, 프로톨리시스)를 지원할 수 있다는 점에 유의한다.[27][28]실제로 ATP 바인딩 자체가 ATPases에 대한 급속한 단백질 분해에 도움을 준다.단, ATP 가수 분해는 전개에만 필요하지만, 이 에너지가 이러한 일부 단계의 결합에 사용될 수 있는지는 아직 명확하지 않다.[28][29]
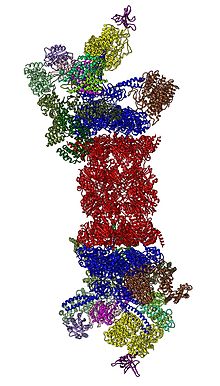
2012년, 두 개의 독립적인 노력이 단일 입자 전자 현미경 검사에 의해 26S 프로테아좀의 분자 구조를 설명하였다.[31][32]2016년 3가지 독자적인 노력으로 극저온 전자파 기판이 없는 상태에서 인간 26S 프로테아솜의 첫 번째 근원자 분해능 구조를 결정했다.[33][34][35]2018년, 기질결합 26S 프로테아솜의 7개 원자구조를 동시에 결정함으로써 기질분해, 변환의 개시, 기질체 공정화의 세부 메커니즘을 규명하는 데 큰 노력이 있었다.[15]20S에 직접 인접한 19S의 중심에는 Rpt1/Rpt2/Rpt6/Rpt3/Rpt4/Rpt5의 헤테로헥사메리카 링에 조립되는 AAA-ATPases(AAA 단백질)가 있다.이 링은 디머로 구성된 트리머: Rpt1/Rpt2, Rpt6/Rpt3 및 Rpt4/Rpt5 디머라이즈(N-단자 코일 코일을 통한 디머라이즈).이 코일들은 육각형 고리로부터 돌출되어 있다.가장 큰 규제 입자 비 ATPASes Rpn1과 Rpn2는 각각 Rpt1/2와 Rpt6/3의 끝에 결합된다.유비퀴틴 수용체 Rpn13은 Rpn2에 결합하여 베이스 큐 콤플렉스를 완성한다.뚜껑은 AAA-ATPase 헥사머(Rpt6/Rpt3/Rpt4)의 1/2을 덮으며, 예기치 않게 Rpn6를 통해 20S에 직접 접촉하고 Rpn5는 더 작은 범위로 한다.COP9 콤플렉스와 eIF3(PCI subunits)의 서브유닛과 구조적으로 관련이 있는 서브유닛 Rpn9, Rpn5, Rpn6, Rpn7, Rpn3 및 Rpn12는 Rpn8/Rpn11 헤테로디머를 둘러싸는 말발굽 모양의 구조물에 모인다.Rpn11은 AAA-ATPase 헥사머의 입구에 위치하며, 기판이 20S로 변환되기 직전에 유비퀴틴 모이에티를 제거하기 위해 이상적인 위치에 있다.현재까지 확인된 두 번째 유비퀴틴 수용체 Rpn10은 뚜껑의 주변부에 위치하며, Rpn8과 Rpn9에 가깝다.
19S의 정합성 보장성
26S proteasome holoenozyme 내의 19S 규제 입자는 현재까지 기판이 없는 상태에서 강하게 다른 6개 순응 상태에서 관찰되었다.[36][37]이 주요 저에너지 상태에서 AAA-ATPase 구성의 특징은 AAA-돔의 계단식 또는 잠금 와셔식 배열이다.[30][31]ATP가 존재하지만 기질 3 대안이 없는 경우, AAA-ATPase 모듈에 관한 뚜껑 위치의 차이에서 19S의 덜 풍부한 순응이 주로 채택된다.[33][37]ATP-traceS 또는 기질이 있는 경우, AAA-ATPase 모듈의 극적인 구조적 변화를 보여주는 훨씬 더 많은 순응이 관찰되었다.[15][36][38][39]기판 결합형 적합성 중 일부는 기판 없는 것과 높은 유사성을 가지고 있지만, 특히 AAA-ATPase 모듈에서 완전히 동일하지는 않다.[15][36]26S 조립에 앞서, 자유 형태의 19S 규제 입자도 7개의 일치 상태에서 관찰되었다.[40]특히 이들 컨포머는 모두 다소 다르고 뚜렷한 특징이 있다.따라서 19S 규제 입자는 다른 생리적 조건에서 적어도 20개의 일치 상태를 샘플링할 수 있다.
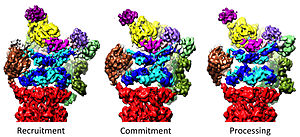
19S에 의한 20S 규정
19S 규제 입자는 20S를 자극해 단백질을 분해하는 역할을 한다.19S 규제 ATPases의 일차적인 기능은 20S에서 기판이 열화실로 들어가는 것을 차단하는 게이트를 여는 것이다.[41]프로테아소말 ATPase가 이 게이트를 여는 메커니즘이 최근에 설명되었다.[18]20S 게이트 개방, 즉 기질 열화에는 프로테아소말 ATPases의 C-termini가 필요하며, 여기에는 특정 모티브(예: HbYX 모티브)가 포함되어 있다.ATPases C-termini는 20S 상단의 포켓에 결합하고, ATPase 콤플렉스를 20S 프로테롤리틱 콤플렉스에 테더링하여 20S 분해 기계와 기질 전개 장비를 결합한다.이러한 C-termini를 이 20S 포켓에 스스로 결합하는 것은 "키인어락"이 문을 여는 것과 같은 방식으로 20S의 게이트의 열기를 자극한다.[18]20S 게이트를 완전히 열기 위해서는 ATPase subunits Rpt1/2/3/5/6의 C-termini 5개를 20S 표면 포켓에 삽입해야 함을 제안하면서 이 "키인어 잠금" 메커니즘이 거의 원자 분해능에서 인간 26S 프로테아솜의 맥락에서 구조적으로 설명되었다.[36][15][33]
기타 규제입자
또한 20S 프로테아솜은 ATPases를 포함하지 않고 짧은 펩타이드의 열화를 촉진할 수 있지만 완전한 단백질은 아닌 두 번째 유형의 규제 입자인 11S 규제 입자와 연관될 수 있다.단지가 더 큰 기판을 펼 수 없기 때문으로 추정된다.이 구조는 PA28, REG 또는 PA26이라고도 알려져 있다.[17]서브유닛의 C-단자 꼬리를 통해 코어 입자에 결합하고 α-링 순응적 변화를 유도하여 20S 게이트를 여는 메커니즘은 19S 입자와 유사한 메커니즘을 제안한다.[42]11S 입자의 표현은 인터페론 감마선에 의해 유도되며, 면역단백질 β 서브유닛과 함께 주요 조직적합성 복합체에 결합되는 펩타이드의 생성을 책임진다.[16]
그러나 또 다른 형태의 ATPase 규제 입자는 Blm10 (yeast) 또는 PA200/PSME4 (인간)이다.20S 게이트에서 1 α 서브 유닛만 열고 그 자체가 그 위에 아주 작은 모공이 있는 돔으로 접힌다.[17]
조립
프로테아솜의 조립은 활성 콤플렉스를 형성하기 위해 연결해야 하는 서브유닛의 수 때문에 복잡한 과정이다.β 서브유닛은 20S 입자 조립 중 변환 후 수정된 N-단자 "프로펩타이드"와 합성되어 프로톨리틱 활성 부지를 노출시킨다.20S 입자는 반단백질 2개로 조립되는데, 각각 7mb α 링에 부착된 7mb pro-β 링으로 구성된다.두 개의 반단백질 β 링의 연관성은 활성 부지를 노출시키기 위해 프로펠러이드의 트레오닌 의존적 자동분해를 촉발한다.이러한 β 상호작용은 주로 염교와 돌연변이에 의한 붕괴가 프로테아솜의 조립 능력을 손상시키는 보존된 알파 나선형 사이의 소수성 상호작용에 의해 매개된다.[43]반단백질 조립은 헥타미 링에 α 서브유닛을 조립하여 해당 프로-β 링의 연결을 위한 템플릿을 형성함으로써 시작된다.α 서브유닛의 조립은 특성화되지 않았다.[44]
최근에서야 19S 규제입자의 조립과정이 상당 부분 해명됐다.19S 규제 입자는 두 개의 뚜렷한 하위 구성 요소인 베이스와 뚜껑으로 조립된다.기본 단지의 조립은 HSM3/S5b, Nas2/p27, Rpn14/PAF1 및 Nas6/gankyrin(이스트/매몰의 이름)의 4개 조립체 보호판으로 촉진된다.[45]이러한 어셈블리 보호판은 AAA-ATPase 하위 단위에 결합되며, 이들의 주요 기능은 헤테로헥사메리카 AAA-ATPase 링의 적절한 조립을 보장하는 것으로 보인다.현재까지 기지 복합체가 별도로 조립되는지, 20S 핵심 입자에 의해 조립되는지, 또는 대체 조립 경로가 존재하는지는 여전히 논쟁 중이다.4개 조립체 보호판 외에 디우비큐레이션 효소 Ubp6/Usp14도 염기조립을 촉진시키지만 반드시 필요한 것은 아니다.[46]뚜껑은 특정 순서에 따라 별도로 조립되며 조립 샤프론이 필요하지 않다.[47]
단백질 분해 과정
유비쿼터스화 및 타겟팅
단백질은 프로테아솜이 세 효소의 조정된 반응을 필요로 하는 리신 잔여물의 공동 수정으로 분해하는 것을 목표로 한다.첫 번째 단계에서 유비퀴틴 활성 효소(E1)는 ATP를 가수분해하고 아데닐릴린 분자를 유비퀴틴 분자로 한다.그리고 나서 이것은 두 번째 유비퀴틴의 아데닐리화와 함께 E1의 활성 사이트 시스테인 잔류물로 전달된다.[48]이 아데닐릴레이티드 유비퀴틴은 그 후 두 번째 효소인 유비퀴틴-콘쥬깅 효소(E2)의 시스테인으로 전달된다.마지막 단계에서, 유비퀴틴 리게스(E3)라고 알려진 매우 다양한 종류의 효소의 구성원은 특정 단백질이 유비퀴티화된다는 것을 인식하고, 유비퀴틴이 E2에서 이 표적 단백질로 전달되는 것을 촉매한다.대상 단백질은 프로테아솜 뚜껑에 의해 인식되기 전에 최소한 4개의 유비퀴틴 단량체(폴리유비퀴틴 체인 형태)로 라벨을 부착해야 한다.[49]따라서 기질 특수성을 이 시스템에 전달하는 것은 E3이다.[50]표현된 E1, E2, E3 단백질의 수는 유기체와 세포 유형에 따라 다르지만 인간에는 다양한 E3 효소가 많이 존재하여 유비퀴틴 프로테아솜 계통의 표적이 엄청나게 많다는 것을 알 수 있다.
다극성 단백질이 프로테아솜을 대상으로 하는 메커니즘은 완전히 이해되지 않는다.다극성 단백질에 결합된 프로테아솜의 고해상도 스냅샷 몇 개를 보면 유비퀴틴 수용체가 초기 기질 타겟팅 및 결합을 위해 Deubiquitinase Rpn11과 조정될 수 있다는 것을 알 수 있다.[15]유비퀴틴 수용체 단백질은 UBL(N-terminal ubasitin-like) 도메인과 하나 이상의 유비퀴틴 연관(UBA) 도메인을 가지고 있다.UBL 도메인은 19S proteasome caps에 의해 인식되고 UBA 도메인은 3헬릭스 번들을 통해 유비쿼티틴을 바인딩한다.이러한 수용체 단백질은 다극성 단백질을 프로테아솜으로 호송할 수 있지만, 이러한 상호작용과 그 규제는 명확하지 않다.[51]
유비퀴틴 단백질 자체는 76개의 아미노산이며, 보존성이 높고 알려진 진핵생물에서 모두 발견되기 때문에 유비쿼터스 성격 때문에 이름이 붙여졌다.[52]진핵생물에서 유비퀴틴을 부호화하는 유전자들은 두 개의 반복으로 배열되는데, 아마도 세포에 충분한 유비퀴틴을 생산하기 위해 이들 유전자에 대한 과도한 전사 요구 때문일 것이다.유비퀴틴은 현재까지 확인된 가장 느리게 진화하는 단백질이라고 제안되었다.[53]유비퀴틴은 다른 유비퀴틴을 묶을 수 있는 7개의 리신 잔류물을 함유하고 있어 여러 종류의 폴리우비퀴틴 사슬이 형성된다.[54]각각의 추가적인 유비퀴틴이 이전 유비퀴틴의 리신 48과 연결되는 체인은 프로테아솜 타겟팅의 역할을 하는 반면, 다른 유형의 체인은 다른 프로세스에 관여할 수 있다.[55][56]

펼치기 및 번역
단백질이 편재된 후, ATP 의존적 결합 단계에서 19S 규제 입자에 의해 인식된다.[15][28]그런 다음 기질 단백질은 20S 입자의 내부로 들어가 프로톨리틱 활성 부위와 접촉해야 한다.20S 입자의 중심 채널은 좁고 α 링 서브유닛의 N단자 꼬리에 의해 게이트되기 때문에 기판은 코어에 들어가기 전에 최소한 부분적으로 펼쳐져야 한다.[15]펴진 기질이 핵으로 들어가는 통로를 번역이라고 하며 반드시 디큐비티화 후에 발생한다.[15][28]그러나 기판이 디비큐레이션되고 펼쳐지는 순서는 아직 명확하지 않다.[57]이러한 과정 중 어느 것이 전체 단백질 분해 반응에서 속도 제한 단계인가? 특정 기질에 따라 달라진다. 일부 단백질의 경우, 발현 과정은 속도 제한인 반면, 분비는 다른 단백질의 경우 가장 느린 단계다.[27]전에 전좌가 약 20아미노산 잔류물은substrate-engaged 26S은deubiquitylation-compatible state,[15]지만 실질적인 삼차 구조에 단백질 분해 효소 복합체의 원자 구조, 그리고 2황화물 채권과 같은 특정 비국부적 상호 작용에서로 제안된 범위에 기질, inh에 충분하다 전개되어야 한다.ibit [58]분해단백질 종단부 또는 내부적으로 충분한 크기의 본질적으로 질서 정연한 단백질 세그먼트의 존재도 효율적인 분해 시작을 촉진하기 위해 제안되었다.[59][60]
α 서브유닛에 의해 형성된 게이트는 약 4개의 잔류물보다 긴 펩타이드들이 20S 입자 내부로 유입되는 것을 방지한다.초기 인식 단계 이전에 결합한 ATP 분자는 변환하기 전에 가수 분해된다.기질 전개에는 에너지가 필요하지만, 번역에는 필요하지 않다.[27][28]조립된 26S 프로테아솜은 비수체성 ATP 아날로그가 있는 상태에서 펼쳐진 단백질을 분해할 수 있지만 접힌 단백질을 분해할 수는 없으므로 ATP 가수분해로부터의 에너지가 기질 전개를 위해 사용됨을 알 수 있다.[27]19S 캡이 ATP 바인딩 상태일 경우 열린 게이트를 통해 펼쳐지는 기질이 쉽게 확산되어 통과된다.[61]
구상 단백질의 발현 메커니즘은 반드시 일반적이지만 아미노산 수열에 다소 의존한다.긴 글리신과 알라닌 교대 순서는 기질 전개를 억제하여 단백질 분해의 효율성을 감소시키는 것으로 나타났다. 이는 부분적으로 분해된 부산물이 방출되는 결과를 낳는데, 이는 ATP 가수 분해와 전개 단계의 디커플링 때문일 수 있다.[62]그러한 글리신 알라닌 반복은 실크로인과 같은 자연에서도 발견된다. 특히 이 염기서열을 가진 특정 엡스타인-바르 바이러스 유전자 제품은 프로테아솜을 지연시켜 주요 조직적합성 복합체에 대한 항원 발현을 방지함으로써 바이러스가 전파되는 것을 도울 수 있다.[63]
프로테롤리시스
프로테아솜은 내단백질 역할을 한다.[64][65][66][67]20S 코어 입자의 β 서브유닛에 의한 프로톨리시스 메커니즘은 트레오닌 의존성 핵포함 공격을 통해 이루어진다.이 메커니즘은 반응성 세레오닌 수산화기의 감응을 위해 관련 물 분자에 의존할 수 있다.분해는 두 개의 β 링의 연결에 의해 형성된 중앙 챔버 내에서 발생하며 일반적으로 부분적으로 분해된 제품을 방출하지 않고, 대신 유기체와 기질에 따라 4개에서 25개까지 잔류물이 있을 수 있지만 일반적으로 기질을 7–9개의 짧은 폴리펩타이드로 감소시킨다.제품 길이를 결정하는 생화학적 메커니즘은 완전히 특징지어지지 않는다.[68]3개의 촉매 β 서브유닛은 공통 메커니즘을 가지고 있지만, 기질 특이성이 약간 다르며, 이는 키모트립신 유사, 트립신 유사, 펩티딜-글루타밀 펩타미드-수체화(PHGH) 유사 물질로 간주된다.이러한 특수성의 변화는 각 서브 유닛의 활성 현장 근처에 있는 국소 잔류물과의 원자 간 접촉의 결과물이다.또한 각 촉매 β 하위 장치는 프로테롤리시스에 필요한 보존된 라이신 잔류물을 가지고 있다.[22]
비록 프로테아솜은 보통 매우 짧은 펩타이드 조각을 생산하지만, 어떤 경우에는 이러한 제품들 자체가 생물학적으로 활성적이고 기능적인 분자들이다.포유류 복합체 NF-164B의 한 성분을 포함하여 특정 유전자의 발현을 조절하는 특정 전사 인자는 비활동적인 전구체로 합성되며, 편재화와 후속 단백질 분해로 인해 전구체가 활성 형태로 전환된다.그러한 활동에는 프로테아솜이 하나의 종단으로부터 기질 단백질을 프로세스적으로 분해하는 것이 아니라 내부적으로 분해할 것을 요구한다.이들 단백질 표면의 긴 루프는 단백질의 기판 역할을 하며, 단백질의 대다수가 바깥에 남아 있는 동안 중심강으로 들어간다는 것이 제안되었다.[69]효모 단백질에서도 유사한 효과가 관찰되었다; 선택적 분해의 이 메커니즘은 규제된 유비퀴틴/단백질 의존적 처리(RUP)라고 알려져 있다.[70]
유비퀴틴 독립 분해
대부분의 프로테아소마 기판은 분해되기 전에 보편화되어야 하지만, 특히 프로테아솜이 단백질의 변환 후 처리에서 정상적인 역할을 할 때, 이 일반적인 규칙에는 몇 가지 예외가 있다.내부 단백질분해를 통해 p105를 p50으로 처리하여 NF-164B의 단백질 활성화가 대표적인 예다.[69]본질적으로 구조화되지 않은 영역으로 인해 불안정하다고 가정된 일부 단백질은 유비쿼터스 독립 방식으로 분해된다.[71]유비퀴틴 독립 프로테아솜 기질 중 가장 잘 알려진 예는 오르니틴 데카르복실라아제 효소다.[72]p53 역시 유비퀴틴 의존적 열화의 대상이지만 p53과 같은 키 셀 사이클 규제자를 대상으로 하는 유비퀴틴 독립 메커니즘도 보고되었다.[73]마지막으로, 구조적으로 비정상적이거나 잘못 접히거나 산화성이 높은 단백질도 세포 스트레스 조건에서 유비퀴틴 독립적이고 19S 독립적으로 분해된다.[74]
진화
20S 프로테아솜은 어디에나 있고 진핵생물이나 고고학에서 필수적이다.박테리아 주문인 Actinomycetales는 또한 20S 프로테아솜의 호몰로로그를 공유하지만, 대부분의 박테리아는 열충격 유전자 hslV와 hslU를 가지고 있는데, 그들의 유전자 생산물은 2층 링과 ATPase로 배열된 복합 단백질 효소다.[75]hslV 단백질은 20S 프로테아솜의 가능한 조상과 유사하다고 가정되어 왔다.[76]일반적으로 HslV는 박테리아에서 필수적인 것은 아니며 모든 박테리아가 그것을 소유하는 것은 아니지만, 일부 양성자들은 20S와 hslV 시스템을 모두 소유하고 있다.[75]많은 박테리아는 또한 프로테아솜과 관련된 ATPase의 다른 호몰로로그를 가지고 있는데, 가장 두드러지게 ClpP와 ClpX가 있다.이러한 중복성은 HslUV 시스템이 필수적이지 않은 이유를 설명한다.
시퀀스 분석 결과 촉매 β 서브유닛이 주로 구조 α 서브유닛보다 진화 초기에 분화되었다고 한다.20S 프로테아솜을 표현하는 박테리아에서 β 서브유닛은 고고학 및 진핵생물 β 서브유닛에 대한 높은 시퀀스 아이덴티티를 갖는 반면 α 시퀀스 아이덴티티는 훨씬 낮다.박테리아에 20S 프로테아솜이 존재하는 것은 측면 유전자 전달에 의한 것일 수 있는 반면, 진핵생물들 사이의 서브유닛의 다양화는 다중 유전자 복제 사건에 기인한다.[75]
셀 사이클 제어
셀 사이클 진행은 셀 사이클의 단계를 구분하는 특정 사이클린에 의해 활성화되는 사이클린 의존 키나제(CDK)의 순서에 따라 제어된다.세포에서 단 몇 분 동안만 지속되는 미토틱 사이클린은 세포 내 모든 단백질 중 가장 짧은 수명을 가지고 있다.[1]CDK-사이클로인 콤플렉스가 그 기능을 수행한 후, 관련 사이클린은 프로테아솜에 의해 폴리우비티화 및 파괴되어 세포 사이클의 방향성을 제공한다.특히 유사분열에서 벗어나려면 유사분열 촉진인자 복합체에서 규제요소인 사이클린 B의 단백질 의존적 분리가 필요하다.[77]척추동물 세포에서는 이 출구가 스핀들 체크포인트에 의해 지연됨에도 불구하고 조기 M상 출구로 이어지는 미토틱 체크포인트를 통한 '슬립피지'가 발생할 수 있다.[78]
G1 위상과 S 위상 사이의 제한 후 지점 점검과 같은 초기 세포 주기 체크포인트는 유사하게 E3 유비퀴틴 리게아제인 아나파제 촉진 복합체(APC)에 의해 편재화가 촉진되는 사이클린 A의 단백질 분해와 관련이 있다.[79]APC와 Skp1/Cul1/F-box 단백질 복합체(SCF 복합체)는 사이클린 분해와 체크포인트 제어의 두 가지 핵심 규제 기관으로, SCF 자체는 어댑터 단백질인 Skp2의 편재화를 통해 APC에 의해 규제되며 G1-S 전환 이전에 SCF 활동을 차단한다.[80]
19S 입자의 개별 구성 요소에는 자체적인 규제 역할이 있다.Gankyrin, a recently identified oncoprotein, is one of the 19S subcomponents that also tightly binds the cyclin-dependent kinase CDK4 and plays a key role in recognizing ubiquitinated p53, via its affinity for the ubiquitin ligase MDM2. Gankyrin is anti-apoptotic and has been shown to be overexpressed in some tumor cell types such as hepatocellula암종[81]
진핵생물처럼 일부 고고학자는 세포 주기를 제어하기 위해 프로테아솜을 사용하기도 하는데, 특히 ESCRT-III 매개 세포 분열을 제어함으로써 그렇다.[82]
식물성장의 규제
식물에서, 식물 성장의 방향과 열대성을 명령하는 보조기(Aux/IAA) 또는 피토호르몬에 의한 신호는 단백질 분해에 대한 Aux/IAA 단백질로 알려진 일종의 전사 인자 억제기의 표적을 유도한다.이러한 단백질은 보조 수용체 TIR1과 복합적으로 SCF 또는 SCF에 의해 보편화된다.AUX/IAA 단백질 분해는 ARF(Auxin-response factor) 계열의 전사 인자를 억제하고 ARF 유도 유전자 발현을 유도한다.[83]ARF 활성화의 세포 결과는 식물의 종류와 발달 단계에 따라 다르지만 뿌리와 잎맥의 성장을 이끄는 데 관여한다.ARF 감압에 대한 구체적인 반응은 개별 ARF와 Aux/IAA 단백질의 결합에서 특수성에 의해 매개되는 것으로 생각된다.[84]
사멸
내부 신호와 외부 신호 모두 세포 사멸 또는 프로그램된 세포 사멸의 유도를 초래할 수 있다.결과적으로 세포 구성 요소의 탈구축은 주로 캐스파제라고 알려진 전문적 보호에 의해 수행되지만 프로테아솜은 또한 세포사멸 과정에서 중요하고 다양한 역할을 한다.이 과정에서 프로테아솜의 관여는 단백질 편재 증가와 세포사멸 전에 잘 관찰되는 E1, E2, E3 효소의 증가로 나타난다.[85][86][87]세포사멸 동안, 핵에 국부화된 단백질은 세포사멸의 특징인 외부 막 표백으로 번역되는 것이 관찰되었다.[88]
프로테아솜 억제는 세포 유형에 따라 세포 사멸 유도에 다른 영향을 미친다.일반적으로 프로테아솜은 연구된 대부분의 세포유형에서 프로테아솜을 억제하는 것은 친중독성이지만 세포유형에 대해서는 필요하지 않다.세포사멸은 세포주기 단백질의 조절된 열화를 방해하여 매개된다.[89]그러나 일부 세포 선들, 특히 흉선세포와 뉴런과 같은 정지하고 분화된 세포의 1차 배양액들은 프로테아솜 억제제에 노출되었을 때 세포사멸을 겪지 못하게 된다.이 효과의 메커니즘은 명확하지 않지만 대기 상태의 셀에 특정하거나 친중독성 키나제 JNK의 차등 활성에서 기인하도록 가정한다.[90]빠르게 분열하는 세포에서 세포사멸을 유도하는 프로테아솜 억제제의 능력은 최근 개발된 보르테조미브와 살리노스포라미드 A와 같은 여러 화학요법에 이용되어 왔다.
세포응력에 대한 반응
감염, 열 쇼크 또는 산화 손상과 같은 세포 스트레스에 대응하여 잘못 접히거나 펴진 단백질을 식별하고 단백질 분해의 대상을 지정하는 열 쇼크 단백질이 표현된다.Hsp27과 Hsp90 - 모두 체세포 단백질은 비록 그 과정에 직접 참여하지는 않지만 유비퀴틴-단백질 시스템의 활동을 증가시키는데 관여해왔다.[91]반면 Hsp70은 잘못 접힌 단백질 표면에 노출된 소수성 패치를 묶고 칩과 같은 E3 유비쿼티틴 리게이즈를 채용해 단백질의 단백질 분해에 태그를 달게 한다.[92]CHIP 단백질(Hsp70-상호작용 단백질의 카르복실 종단)은 E3 효소 CHIP와 E2 결합 파트너 사이의 상호작용을 억제하여 스스로 조절된다.[93]
프로테아솜 시스템을 통해 산화적으로 손상된 단백질의 저하를 촉진하기 위한 유사한 메커니즘이 존재한다.특히 핵에 국부화된 프로테아솜은 PARP에 의해 조절되어 부적절하게 산화된 히스톤을 적극적으로 저하시킨다.[94]종종 세포에서 큰 아모르퍼스 골재를 형성하는 산화 단백질은 19S 규제 상한 없이 20S 코어 입자에 의해 직접 분해될 수 있으며, ATP 가수분해나 유비퀴틴으로 태깅을 할 필요가 없다.[74]그러나 높은 수준의 산화 손상은 단백질 조각들 사이의 교차 링크의 정도를 증가시켜 단백질 분해에 내성이 있는 골재를 만든다.이렇게 산화성이 높은 골재의 수와 크기는 노화와 관련이 있다.[95]
유비퀴틴 프로테아솜 시스템의 조절이 잘못되면 몇 가지 신경 질환에 기여할 수 있다.그것은 아스트로사이토마스와 같은 뇌종양으로 이어질 수 있다.[96]파킨슨병이나 알츠하이머병처럼 잘못 접힌 단백질의 집단을 공통적인 특징으로 공유하는 일부 후기 신경퇴행성질환에서는 잘못 접힌 단백질의 불용성 큰 집적합물이 형성되어 신경독성을 유발할 수 있는데, 이는 아직 잘 이해되지 않은 메커니즘을 통해서이다.단백질 활동 감소는 파킨슨병에서 집적과 루이소체 형성의 원인으로 제시되어 왔다.[97]이 가설은 파킨슨병 효모모델이 낮은 프로테아솜 활성 조건 하에서 루이스 신체의 주요 단백질 성분인 α-시뉴클레인의 독성에 더 취약하다는 관측에 의해 뒷받침된다.[98]손상된 단백질 활동은 자폐 스펙트럼 장애와 같은 인지 장애와 체내 근병증과 같은 근육 및 신경 질환의 근본 원인이 될 수 있다.[96]
면역체계에서의 역할
프로테아솜은 적응성 면역체계의 기능에서 직설적이지만 결정적인 역할을 한다.펩타이드 항원은 항원을 나타내는 세포의 표면에 주요 조직적합성 복합체 I(MHC) 단백질이 표시한다.이 펩타이드들은 침입한 병원체에 의해 유발된 단백질의 단백질 분해의 산물이다.구성성 표현 프로테아솜은 이 과정에 참여할 수 있지만, 인터페론 감마선에 의해 발현이 유도되는 단백질로 구성된 특화된 복합체는 MHC 결합을 위해 크기와 구성이 최적인 펩타이드의 1차 생산물이다.면역 반응 동안 발현이 증가하는 이러한 단백질은 MHC 리간드의 생성을 조절하는 것으로 알려진 11S 규제 입자와 기질 특이성을 변화시킨 β1i, β2i, β5i라고 불리는 전문화된 β 서브유닛을 포함한다.전문화된 β 서브유닛으로 형성된 콤플렉스는 면역항암제로 알려져 있다.[16]또 다른 β5i 변종 서브 유닛인 β5t는 흉선에 표현되어, 아직 기능이 불분명하지만 흉선 특유의 "시모프로테아솜"으로 이어진다.[99]
MHC 등급 I 리간드 결합의 강도는 수소 결합과 MHC 표면의 "B 포켓"이라고 불리는 부위와의 밀접 접촉에 의해 결합되기 때문에 리간드 C-terminus의 구성에 따라 달라진다.많은 MHC 등급 I은 소수성 C-단말 잔류물을 선호하며 면역단백질 복합체가 소수성 C-단말기를 발생시킬 가능성이 더 높다고 주장한다.[100]
시토카인 발현에 대한 항중독성 및 친염증 조절기인 NF-κB의 활성화 형태를 만들어 내는 역할 때문에 프로테아소마 활동은 염증성 및 자가면역질환과 연계되어 왔다.프로테아솜 활동의 증가된 수준은 질병 활동과 관련이 있으며 전신 루푸스 홍반성 관절염과 류마티스성 관절염을 포함한 자가면역 질환에 관련되어 있다.[16]
프로테아솜은 또한 항체 결합형 바이러스의 세포내 항체 매개 단백질 분해에도 관여한다.이 중화 경로에서 TRIM21(삼중 모티브 계열의 단백질)은 면역글로불린 G와 결합하여 처녀성이 분해되는 프로테아솜으로 유도한다.[101]
프로테아솜 억제제
프로테아솜 억제제는 세포 배양에서 효과적인 항투석 활동을 가지고 있으며, 친성장 세포 순환 단백질의 조절된 열화를 방해하여 사멸을 유도한다.[89]종양 세포의 세포사멸을 선택적으로 유도하는 이러한 접근법은 동물 모델과 인간의 실험에 효과적이라는 것이 입증되었다.
스트렙토미세스 박테리아에 의해 합성된 천연물인 락타시스틴은 비펩티드 프로테아솜 억제제로 최초로 발견되어[102] 생화학 및 세포생물학의 연구 도구로 널리 사용되고 있다.락타시스틴은 마이오제닉스/프로스크립트에 허가를 받았으며, 마이오제닉/프로스크립트는 현재 다케다제약의 일부분인 밀레니엄제약에 인수되었다.락타시스틴은 프로테아솜의 촉매 β 서브유닛의 아미노-단자 세레오닌을 공밸런싱하며, 특히 프로테아솜의 키모트리핀 유사 활동을 담당하는 β5 서브유닛을 수정한다.이 발견은 프로테아솜을 기계적으로 새로운 종류의 프로테아제로 확립하는데 도움을 주었다: 아미노-단자-트레오닌 프로테아제.
밀레니엄제약에서 개발해 벨케이드로 시판된 분자 보르테조미브(Bortezomib, Boronated MG132)는 항암화학요법제로서 임상적 용도에 도달한 최초의 단백질 억제제다.[103]보르테조미브는 다발성 골수종 치료에 쓰인다.[104]특히 다발성 골수종은 혈청 내 단백질 유도 펩타이드 수치가 증가하여 항암화학요법에 따라 정상 수준으로 감소하는 것으로 관찰되었다.[105]동물에 대한 연구는 보르테조미브가 췌장암에도 임상적으로 중요한 영향을 미칠 수 있다는 것을 보여주었다.[106][107]다른 B세포 관련 암,[108] 특히 일부 비호지킨 림프종의 치료에서 보르테조미브의 효과를 검사하기 위해 임상 전 및 초기 임상 연구가 시작되었다.[109]임상 결과는 또한 B세포 급성 림프절성 백혈병 프로테아솜 억제제는 글루코르티코이드에 내성이 있는 배양 백혈병 세포의 일부 유형을 죽일 수 있기 때문에 화학요법과 결합된 프로테아솜 억제제의 사용을 정당화하는 것으로 보인다.[111]
노르비르로 시판되는 이 분자 리토나비르는 프로테아제 억제제로 개발돼 HIV 감염을 목표로 삼았다.그러나, 그것은 프로테아솜뿐만 아니라 무료 프로테아솜을 억제하는 것으로 보여진다; 구체적으로는 프로테아솜의 키모트립신 같은 활동은 리토나비르에 의해 억제되는 반면 트립신 같은 활동은 다소 강화된다.[112]동물 모델에 대한 연구는 리토나비르가 교모세포의 성장에 억제 효과를 줄 수 있다는 것을 시사한다.[113]
프로테아솜 억제제 역시 동물 모델에서 자가면역질환 치료의 가능성을 보여주었다.예를 들어, 인간 피부 이식을 가진 쥐에 대한 연구는 프로테아솜 억제제로 치료한 후 건선으로 인한 병변 크기가 감소한다는 것을 발견했다.[114]억제제는 천식의 설치류 모델에서도 긍정적인 효과를 나타낸다.[115]
프로테아솜의 라벨링과 억제도 세포 내 프로테아솜 활성의 체외 및 생체내 연구 모두에 대한 실험실 설정에서 관심 있다.가장 일반적으로 사용되는 실험실 억제제는 락타시스틴과 골드버그 연구소에서 초기에 개발한 펩타이드 알데히드 MG132이다.형광 억제제는 또한 조립된 프로테아솜의 활성 부지에 특별히 라벨을 붙이도록 개발되었다.[116]
임상적 유의성
프로테아솜과 그 서브유닛은 적어도 두 가지 이유로 임상적으로 중요한데, (1) 손상된 복합체 결합 또는 기능장애 프로테아솜은 특정 질병의 근본적인 병태생리와 관련될 수 있으며, (2) 치료적 개입의 약물 대상으로 악용될 수 있다.보다 최근에는 새로운 진단 마커와 전략의 개발을 위한 프로테아솜을 고려하는 노력이 더 많이 이루어지고 있다.프로테아좀의 병태생리학에 대한 개선되고 포괄적인 이해는 향후 임상적 응용으로 이어져야 한다.
프로테아솜은 유비퀴틴-단백질 시스템(UPS)과 해당 세포 단백질 품질 관리(PQC)의 중추적 구성 요소를 형성한다.단백질 편재와 그에 따른 프로테아솜에 의한 단백질 분해와 분해는 세포 주기, 세포 성장과 분화, 유전자 전사, 신호 전달 및 세포 사멸의 조절에 있어 중요한 메커니즘이다.[118]그 후 단백질 복합체 조립 및 기능이 손상되면 단백질 분해 활동이 감소하고 단백질 종들이 손상되거나 잘못 접히는 현상이 발생한다.이러한 단백질이 축적되면 신경퇴행성질환,[119][120] 심혈관질환,[121][122][123] 염증반응 및 자가면역질환,[124] 전신 DNA손상반응에서 병생성과 표현특성에 기여하여 악성종양으로 이어질 수 있다.[125]
몇몇 임상 실험 연구 성격의 UPS수차와 규제 완화 등 몇몇이고myodegenerative 신경 퇴행성 질환의 발병에 기여하고의 disease,[128]인 근 위축성 측색 경화증(근 위축성 측색 경화증)[128]헌팅턴 disease,[127]C다 알츠하이머 disease,[126]파킨슨 병 disease[127]등을 표시했습니다reutzfeldt–Jakob disease,[129]그리고 운동 뉴런 질환, 폴리글루타민(PolyQ) 질환, 근위축성[130] 근육 및 치매와 관련된 몇 가지 희귀한 형태의 신경퇴행성 질환이 있다.[131]유비퀴틴-단백질계통(UPS)의 일부로서 프로테아솜은 심장 단백질 동점증을 유지하여 심장 허혈성 부상,[132] 심실 비대증[133], 심부전 등에 큰 역할을 한다.[134]게다가, UPS가 악성 변형에 필수적인 역할을 한다는 증거가 축적되고 있다.UPS 단백질 분해는 암의 발병에 중요한 자극 신호에 대한 암세포의 반응에 중요한 역할을 한다.따라서 p53, c-준, c-Fos, NF-bB, c-Myc, HIF-1α, MATα2, STAT3, 스테롤 조절 요소 결합 단백질 및 안드로겐 수용체와 같은 전사 인자의 저하로 인한 유전자 발현이 모두 UPS에 의해 제어되어 다양한 악성종양 개발에 관여한다.[135]Moreover, the UPS regulates the degradation of tumor suppressor gene products such as adenomatous polyposis coli (APC) in colorectal cancer, retinoblastoma (Rb). and von Hippel–Lindau tumor suppressor (VHL), as well as a number of proto-oncogenes (Raf, Myc, Myb, Rel, Src, Mos, ABL).UPS도 염증반응 규제에 관여하고 있다.이러한 활동은 대개 TNF-α, IL-β, IL-8, 접착분자(ICAM-1, VCAM-1, P-selectin)와 프로스타글란딘과 질산화물(NO)과 같은 프로테아그란딘의 발현을 더욱 조절하는 NF-164B의 활성화에 있어서 프로테아솜의 역할에 기인한다.[124]또한 UPS는 주로 사이클린의 단백질 분해와 CDK 억제제의 저하를 통해 백혈구 증식의 조절자로서 염증 반응에도 역할을 한다.[136]마지막으로, SLE, Sögren 증후군, 류마티스 관절염(RA)을 앓고 있는 자가면역질환자들은 임상 바이오마커로 응용할 수 있는 순환 프로테아솜을 주로 나타낸다.[137]
참고 항목
참조
- ^ a b c d Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J (2004). "3". Molecular cell biology (5th ed.). New York: W.H. Freeman and CO. pp. 66–72. ISBN 978-0-7167-4366-8.
- ^ Peters JM, Franke WW, Kleinschmidt JA (March 1994). "Distinct 19 S and 20 S subcomplexes of the 26 S proteasome and their distribution in the nucleus and the cytoplasm". The Journal of Biological Chemistry. 269 (10): 7709–18. doi:10.1016/S0021-9258(17)37345-3. PMID 8125997.
- ^ Nassif, Nicholas D.; Cambray, Samantha E.; Kraut, Daniel A. (May 2014). "Slipping up: Partial substrate degradation by ATP-dependent proteases". IUBMB Life. 66 (5): 309–317. doi:10.1002/iub.1271. PMID 24823973. S2CID 29860298.
- ^ a b Nobel Prize Committee (2004). "Nobel Prize Awardees in Chemistry, 2004". Retrieved 11 December 2006.
- ^ Etlinger JD, Goldberg AL (January 1977). "A soluble ATP-dependent proteolytic system responsible for the degradation of abnormal proteins in reticulocytes". Proceedings of the National Academy of Sciences of the United States of America. 74 (1): 54–8. Bibcode:1977PNAS...74...54E. doi:10.1073/pnas.74.1.54. PMC 393195. PMID 264694.
- ^ Ciehanover A, Hod Y, Hershko A (April 1978). "A heat-stable polypeptide component of an ATP-dependent proteolytic system from reticulocytes". Biochemical and Biophysical Research Communications. 81 (4): 1100–5. doi:10.1016/0006-291X(78)91249-4. PMID 666810.
- ^ Goldknopf IL, Busch H (March 1977). "Isopeptide linkage between nonhistone and histone 2A polypeptides of chromosomal conjugate-protein A24". Proceedings of the National Academy of Sciences of the United States of America. 74 (3): 864–8. Bibcode:1977PNAS...74..864G. doi:10.1073/pnas.74.3.864. PMC 430507. PMID 265581.
- ^ Ciechanover A (September 2005). "Early work on the ubiquitin proteasome system, an interview with Aaron Ciechanover. Interview by CDD". Cell Death and Differentiation. 12 (9): 1167–77. doi:10.1038/sj.cdd.4401691. PMID 16094393.
- ^ Wilk S, Orlowski M (November 1980). "Cation-sensitive neutral endopeptidase: isolation and specificity of the bovine pituitary enzyme". Journal of Neurochemistry. 35 (5): 1172–82. doi:10.1111/j.1471-4159.1980.tb07873.x. PMID 6778972. S2CID 9028201.
- ^ Arrigo AP, Tanaka, K, Goldberg F, Welch WJ (1988). "Identity of 19S prosome particle with the large multifunctional protease complex of mammalian cells". Nature. 331 (6152): 192–94. doi:10.1038/331192a0. PMID 3277060. S2CID 97688.Tanaka K, Waxman L, Goldberg AL (June 1983). "ATP serves two distinct roles in protein degradation in reticulocytes, one requiring and one independent of ubiquitin". The Journal of Cell Biology. 96 (6): 1580–5. doi:10.1083/jcb.96.6.1580. PMC 2112434. PMID 6304111.
- ^ Hough R, Pratt G, Rechsteiner M (June 1987). "Purification of two high molecular weight proteases from rabbit reticulocyte lysate". The Journal of Biological Chemistry. 262 (17): 8303–13. doi:10.1016/S0021-9258(18)47564-3. PMID 3298229.
- ^ Hershko A (September 2005). "Early work on the ubiquitin proteasome system, an interview with Avram Hershko. Interview by CDD". Cell Death and Differentiation. 12 (9): 1158–61. doi:10.1038/sj.cdd.4401709. PMID 16094391.
- ^ Kopp F, Steiner R, Dahlmann B, Kuehn L, Reinauer H (August 1986). "Size and shape of the multicatalytic proteinase from rat skeletal muscle". Biochimica et Biophysica Acta (BBA) - Protein Structure and Molecular Enzymology. 872 (3): 253–60. doi:10.1016/0167-4838(86)90278-5. PMID 3524688.
- ^ Löwe J, Stock D, Jap B, Zwickl P, Baumeister W, Huber R (April 1995). "Crystal structure of the 20S proteasome from the archaeon T. acidophilum at 3.4 A resolution". Science. 268 (5210): 533–9. doi:10.1126/science.7725097. PMID 7725097.
- ^ a b c d e f g h i j k Dong Y, Zhang S, Wu Z, Li X, Wang WL, Zhu Y, Stoilova-McPhie S, Lu Y, Finley D, Mao Y (November 2018). "Cryo-EM structures and dynamics of substrate-engaged human 26S proteasome". Nature. 565 (7737): 49–55. doi:10.1038/s41586-018-0736-4. PMC 6370054. PMID 30479383.
- ^ a b c d Wang J, Maldonado MA (August 2006). "The ubiquitin-proteasome system and its role in inflammatory and autoimmune diseases". Cellular & Molecular Immunology. 3 (4): 255–61. PMID 16978533.
- ^ a b c Stadtmueller, BM; Hill, CP (7 January 2011). "Proteasome activators". Molecular Cell. 41 (1): 8–19. doi:10.1016/j.molcel.2010.12.020. PMC 3040445. PMID 21211719.
- ^ a b c Smith DM, Chang SC, Park S, Finley D, Cheng Y, Goldberg AL (September 2007). "Docking of the proteasomal ATPases' carboxyl termini in the 20S proteasome's alpha ring opens the gate for substrate entry". Molecular Cell. 27 (5): 731–44. doi:10.1016/j.molcel.2007.06.033. PMC 2083707. PMID 17803938.
- ^ "MEROPS Family T1". EMBL-EBI. Retrieved 16 February 2019.
- ^ Wilk S, Orlowski M (March 1983). "Evidence that pituitary cation-sensitive neutral endopeptidase is a multicatalytic protease complex". Journal of Neurochemistry. 40 (3): 842–9. doi:10.1111/j.1471-4159.1983.tb08056.x. PMID 6338156. S2CID 23508675.
- ^ a b Nandi D, Tahiliani P, Kumar A, Chandu D (March 2006). "The ubiquitin-proteasome system" (PDF). Journal of Biosciences. 31 (1): 137–55. doi:10.1007/BF02705243. PMID 16595883. S2CID 21603835.
- ^ a b Heinemeyer W, Fischer M, Krimmer T, Stachon U, Wolf DH (October 1997). "The active sites of the eukaryotic 20 S proteasome and their involvement in subunit precursor processing". The Journal of Biological Chemistry. 272 (40): 25200–9. doi:10.1074/jbc.272.40.25200. PMID 9312134.
- ^ a b Padmanabhan A, Vuong SA, Hochstrasser M (March 2016). "Assembly of an Evolutionarily Conserved Alternative Proteasome Isoform in Human Cells". Cell Reports. 14 (12): 2962–74. doi:10.1016/j.celrep.2016.02.068. PMC 4828729. PMID 26997268.
- ^ Velichutina I, Connerly PL, Arendt CS, Li X, Hochstrasser M (February 2004). "Plasticity in eucaryotic 20S proteasome ring assembly revealed by a subunit deletion in yeast". The EMBO Journal. 23 (3): 500–10. doi:10.1038/sj.emboj.7600059. PMC 1271798. PMID 14739934.
- ^ Kusmierczyk AR, Kunjappu MJ, Funakoshi M, Hochstrasser M (March 2008). "A multimeric assembly factor controls the formation of alternative 20S proteasomes". Nature Structural & Molecular Biology. 15 (3): 237–44. doi:10.1038/nsmb.1389. PMID 18278055. S2CID 21181637.
- ^ Zwickl P, Ng D, Woo KM, Klenk HP, Goldberg AL (September 1999). "An archaebacterial ATPase, homologous to ATPases in the eukaryotic 26 S proteasome, activates protein breakdown by 20 S proteasomes". The Journal of Biological Chemistry. 274 (37): 26008–14. doi:10.1074/jbc.274.37.26008. PMID 10473546.
- ^ a b c d Smith DM, Kafri G, Cheng Y, Ng D, Walz T, Goldberg AL (December 2005). "ATP binding to PAN or the 26S ATPases causes association with the 20S proteasome, gate opening, and translocation of unfolded proteins". Molecular Cell. 20 (5): 687–98. doi:10.1016/j.molcel.2005.10.019. PMID 16337593.
- ^ a b c d e Liu CW, Li X, Thompson D, Wooding K, Chang TL, Tang Z, Yu H, Thomas PJ, DeMartino GN (October 2006). "ATP binding and ATP hydrolysis play distinct roles in the function of 26S proteasome". Molecular Cell. 24 (1): 39–50. doi:10.1016/j.molcel.2006.08.025. PMC 3951175. PMID 17018291.
- ^ Lam YA, Lawson TG, Velayutham M, Zweier JL, Pickart CM (April 2002). "A proteasomal ATPase subunit recognizes the polyubiquitin degradation signal". Nature. 416 (6882): 763–7. Bibcode:2002Natur.416..763L. doi:10.1038/416763a. PMID 11961560. S2CID 4421764.
- ^ a b Beck F, Unverdorben P, Bohn S, Schweitzer A, Pfeifer G, Sakata E, Nickell S, Plitzko JM, Villa E, Baumeister W, Förster F (September 2012). "Near-atomic resolution structural model of the yeast 26S proteasome". Proceedings of the National Academy of Sciences of the United States of America. 109 (37): 14870–5. Bibcode:2012PNAS..10914870B. doi:10.1073/pnas.1213333109. PMC 3443124. PMID 22927375.
- ^ a b Lander GC, Estrin E, Matyskiela ME, Bashore C, Nogales E, Martin A (February 2012). "Complete subunit architecture of the proteasome regulatory particle". Nature. 482 (7384): 186–91. Bibcode:2012Natur.482..186L. doi:10.1038/nature10774. PMC 3285539. PMID 22237024.
- ^ Lasker K, Förster F, Bohn S, Walzthoeni T, Villa E, Unverdorben P, Beck F, Aebersold R, Sali A, Baumeister W (January 2012). "Molecular architecture of the 26S proteasome holocomplex determined by an integrative approach". Proceedings of the National Academy of Sciences of the United States of America. 109 (5): 1380–7. doi:10.1073/pnas.1120559109. PMC 3277140. PMID 22307589.
- ^ a b c Chen S, Wu J, Lu Y, Ma YB, Lee BH, Yu Z, Ouyang Q, Finley DJ, Kirschner MW, Mao Y (November 2016). "Structural basis for dynamic regulation of the human 26S proteasome". Proceedings of the National Academy of Sciences of the United States of America. 113 (46): 12991–12996. doi:10.1073/pnas.1614614113. PMC 5135334. PMID 27791164.
- ^ Huang X, Luan B, Wu J, Shi Y (September 2016). "An atomic structure of the human 26S proteasome". Nature Structural & Molecular Biology. 23 (9): 778–785. doi:10.1038/nsmb.3273. PMID 27428775. S2CID 21909333.
- ^ Schweitzer A, Aufderheide A, Rudack T, Beck F, Pfeifer G, Plitzko JM, Sakata E, Schulten K, Förster F, Baumeister W (July 2016). "Structure of the human 26S proteasome at a resolution of 3.9 Å". Proceedings of the National Academy of Sciences of the United States of America. 113 (28): 7816–7821. doi:10.1073/pnas.1614614113. PMC 5135334. PMID 27791164.
- ^ a b c d Zhu Y, Wang WL, Yu D, Ouyang Q, Lu Y, Mao Y (April 2018). "Structural mechanism for nucleotide-driven remodeling of the AAA-ATPase unfoldase in the activated human 26S proteasome". Nature Communications. 9 (1): 1360. Bibcode:2018NatCo...9.1360Z. doi:10.1038/s41467-018-03785-w. PMC 5893597. PMID 29636472.
- ^ a b c Unverdorben P, Beck F, Śledź P, Schweitzer A, Pfeifer G, Plitzko JM, Baumeister W, Förster F (April 2014). "Deep classification of a large cryo-EM dataset defines the conformational landscape of the 26S proteasome". Proceedings of the National Academy of Sciences of the United States of America. 111 (15): 5544–9. Bibcode:2014PNAS..111.5544U. doi:10.1073/pnas.1403409111. PMC 3992697. PMID 24706844.
- ^ Śledź P, Unverdorben P, Beck F, Pfeifer G, Schweitzer A, Förster F, Baumeister W (April 2013). "Structure of the 26S proteasome with ATP-γS bound provides insights into the mechanism of nucleotide-dependent substrate translocation". Proceedings of the National Academy of Sciences of the United States of America. 110 (18): 7264–7269. Bibcode:2013PNAS..110.7264S. doi:10.1073/pnas.1305782110. PMC 3645540. PMID 23589842.
- ^ Matyskiela ME, Lander GC, Martin A (July 2013). "Conformational switching of the 26S proteasome enables substrate degradation". Nature Structural & Molecular Biology. 20 (7): 781–788. doi:10.1038/nsmb.2616. PMC 3712289. PMID 23770819.
- ^ Lu Y, Wu J, Dong Y, Chen S, Sun S, Ma YB, Ouyang Q, Finley D, Kirschner MW, Mao Y (July 2017). "Conformational Landscape of the p28-Bound Human Proteasome Regulatory Particle". Molecular Cell. 67 (2): 322–333.e6. doi:10.1016/j.molcel.2017.06.007. PMC 5580496. PMID 28689658.
- ^ Köhler A, Cascio P, Leggett DS, Woo KM, Goldberg AL, Finley D (June 2001). "The axial channel of the proteasome core particle is gated by the Rpt2 ATPase and controls both substrate entry and product release". Molecular Cell. 7 (6): 1143–52. doi:10.1016/S1097-2765(01)00274-X. PMID 11430818.
- ^ Förster A, Masters EI, Whitby FG, Robinson H, Hill CP (May 2005). "The 1.9 A structure of a proteasome-11S activator complex and implications for proteasome-PAN/PA700 interactions". Molecular Cell. 18 (5): 589–99. doi:10.1016/j.molcel.2005.04.016. PMID 15916965.
- ^ Witt S, Kwon YD, Sharon M, Felderer K, Beuttler M, Robinson CV, Baumeister W, Jap BK (July 2006). "Proteasome assembly triggers a switch required for active-site maturation". Structure. 14 (7): 1179–88. doi:10.1016/j.str.2006.05.019. PMID 16843899.
- ^ Krüger E, Kloetzel PM, Enenkel C (2001). "20S proteasome biogenesis". Biochimie. 83 (3–4): 289–93. doi:10.1016/S0300-9084(01)01241-X. PMID 11295488.
- ^ Murata S, Yashiroda H, Tanaka K (February 2009). "Molecular mechanisms of proteasome assembly". Nature Reviews Molecular Cell Biology. 10 (2): 104–115. doi:10.1038/nrm2630. PMID 19165213. S2CID 21263837.
- ^ Sakata E, Stengel F, Fukunaga K, Zhou M, Saeki Y, Förster F, Baumeister W, Tanaka K, Robinson CV (June 2011). "The catalytic activity of Ubp6 enhances maturation of the proteasomal regulatory particle". Molecular Cell. 42 (5): 637–649. doi:10.1016/j.molcel.2011.04.021. PMID 21658604.
- ^ Fukunaga K, Kudo T, Toh-e A, Tanaka K, Saeki Y (June 2010). "Dissection of the assembly pathway of the proteasome lid in Saccharomyces cerevisiae". Biochemical and Biophysical Research Communications. 396 (4): 1048–1053. doi:10.1016/j.bbrc.2010.05.061. PMID 20471955.
- ^ Haas AL, Warms JV, Hershko A, Rose IA (March 1982). "Ubiquitin-activating enzyme. Mechanism and role in protein-ubiquitin conjugation". The Journal of Biological Chemistry. 257 (5): 2543–8. doi:10.1016/S0021-9258(18)34958-5. PMID 6277905.
- ^ Thrower JS, Hoffman L, Rechsteiner M, Pickart CM (January 2000). "Recognition of the polyubiquitin proteolytic signal". The EMBO Journal. 19 (1): 94–102. doi:10.1093/emboj/19.1.94. PMC 1171781. PMID 10619848.
- ^ Risseeuw EP, Daskalchuk TE, Banks TW, Liu E, Cotelesage J, Hellmann H, Estelle M, Somers DE, Crosby WL (June 2003). "Protein interaction analysis of SCF ubiquitin E3 ligase subunits from Arabidopsis". The Plant Journal. 34 (6): 753–67. doi:10.1046/j.1365-313X.2003.01768.x. PMID 12795696.
- ^ Elsasser S, Finley D (August 2005). "Delivery of ubiquitinated substrates to protein-unfolding machines". Nature Cell Biology. 7 (8): 742–9. doi:10.1038/ncb0805-742. PMID 16056265. S2CID 21069699.
- ^ Sadanandom A, Bailey M, Ewan R, Lee J, Nelis S (October 2012). "The ubiquitin-proteasome system: central modifier of plant signalling". The New Phytologist. 196 (1): 13–28. doi:10.1111/j.1469-8137.2012.04266.x. PMID 22897362.
- ^ Sharp PM, Li WH (1987). "Ubiquitin genes as a paradigm of concerted evolution of tandem repeats". Journal of Molecular Evolution. 25 (1): 58–64. Bibcode:1987JMolE..25...58S. doi:10.1007/BF02100041. PMID 3041010. S2CID 7929162.
- ^ Pickart CM, Fushman D (December 2004). "Polyubiquitin chains: polymeric protein signals". Current Opinion in Chemical Biology. 8 (6): 610–16. doi:10.1016/j.cbpa.2004.09.009. PMID 15556404.
- ^ Xu P, Duong DM, Seyfried NT, Cheng D, Xie Y, Robert J, Rush J, Hochstrasser M, Finley D, Peng J (April 2009). "Quantitative proteomics reveals the function of unconventional ubiquitin chains in proteasomal degradation". Cell. 137 (1): 133–45. doi:10.1016/j.cell.2009.01.041. PMC 2668214. PMID 19345192.
- ^ Pickart CM (November 2000). "Ubiquitin in chains". Trends in Biochemical Sciences. 25 (11): 544–8. doi:10.1016/S0968-0004(00)01681-9. PMID 11084366.
- ^ Zhu Q, Wani G, Wang QE, El-mahdy M, Snapka RM, Wani AA (July 2005). "Deubiquitination by proteasome is coordinated with substrate translocation for proteolysis in vivo". Experimental Cell Research. 307 (2): 436–51. doi:10.1016/j.yexcr.2005.03.031. PMID 15950624.
- ^ Wenzel T, Baumeister W (March 1995). "Conformational constraints in protein degradation by the 20S proteasome". Nature Structural Biology. 2 (3): 199–204. doi:10.1038/nsb0395-199. PMID 7773788. S2CID 41599619.
- ^ Inobe T, Fishbain S, Prakash S, Matouschek A (March 2011). "Defining the geometry of the two-component proteasome degron". Nature Chemical Biology. 7 (3): 161–7. doi:10.1038/nchembio.521. PMC 3129032. PMID 21278740.
- ^ van der Lee R, Lang B, Kruse K, Gsponer J, Sánchez de Groot N, Huynen MA, Matouschek A, Fuxreiter M, Babu MM (September 2014). "Intrinsically disordered segments affect protein half-life in the cell and during evolution". Cell Reports. 8 (6): 1832–44. doi:10.1016/j.celrep.2014.07.055. PMC 4358326. PMID 25220455.
- ^ Smith DM, Benaroudj N, Goldberg A (October 2006). "Proteasomes and their associated ATPases: a destructive combination". Journal of Structural Biology. 156 (1): 72–83. doi:10.1016/j.jsb.2006.04.012. PMID 16919475.
- ^ Hoyt MA, Zich J, Takeuchi J, Zhang M, Govaerts C, Coffino P (April 2006). "Glycine-alanine repeats impair proper substrate unfolding by the proteasome". The EMBO Journal. 25 (8): 1720–9. doi:10.1038/sj.emboj.7601058. PMC 1440830. PMID 16601692.
- ^ Zhang M, Coffino P (March 2004). "Repeat sequence of Epstein–Barr virus-encoded nuclear antigen 1 protein interrupts proteasome substrate processing". The Journal of Biological Chemistry. 279 (10): 8635–41. doi:10.1074/jbc.M310449200. PMID 14688254.
- ^ Seemüller E, Lupas A, Stock D, Löwe J, Huber R, Baumeister W (April 1995). "Proteasome from Thermoplasma acidophilum: a threonine protease". Science. 268 (5210): 579–82. Bibcode:1995Sci...268..579S. doi:10.1126/science.7725107. PMID 7725107.
- ^ Coux O, Tanaka K, Goldberg AL (1996). "Structure and functions of the 20S and 26S proteasomes". Annual Review of Biochemistry. 65: 801–47. doi:10.1146/annurev.bi.65.070196.004101. PMID 8811196.
- ^ Groll M, Ditzel L, Löwe J, Stock D, Bochtler M, Bartunik HD, Huber R (April 1997). "Structure of 20S proteasome from yeast at 2.4 A resolution". Nature. 386 (6624): 463–71. doi:10.1038/386463a0. PMID 9087403. S2CID 4261663.
- ^ Dick TP, Nussbaum AK, Deeg M, Heinemeyer W, Groll M, Schirle M, Keilholz W, Stevanović S, Wolf DH, Huber R, Rammensee HG, Schild H (October 1998). "Contribution of proteasomal beta-subunits to the cleavage of peptide substrates analyzed with yeast mutants". The Journal of Biological Chemistry. 273 (40): 25637–46. doi:10.1074/jbc.273.40.25637. PMID 9748229.
- ^ Voges D, Zwickl P, Baumeister W (1999). "The 26S proteasome: a molecular machine designed for controlled proteolysis". Annual Review of Biochemistry. 68 (1): 1015–68. doi:10.1146/annurev.biochem.68.1.1015. PMID 10872471.
- ^ a b Rape M, Jentsch S (May 2002). "Taking a bite: proteasomal protein processing". Nature Cell Biology. 4 (5): E113–6. doi:10.1038/ncb0502-e113. PMID 11988749. S2CID 7126477.
- ^ Rape M, Jentsch S (November 2004). "Productive RUPture: activation of transcription factors by proteasomal processing". Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1695 (1–3): 209–13. doi:10.1016/j.bbamcr.2004.09.022. PMID 15571816.
- ^ Asher G, Reuven N, Shaul Y (August 2006). "20S proteasomes and protein degradation "by default"". BioEssays. 28 (8): 844–9. doi:10.1002/bies.20447. PMID 16927316.
- ^ Zhang M, Pickart CM, Coffino P (April 2003). "Determinants of proteasome recognition of ornithine decarboxylase, a ubiquitin-independent substrate". The EMBO Journal. 22 (7): 1488–96. doi:10.1093/emboj/cdg158. PMC 152902. PMID 12660156.
- ^ Asher G, Shaul Y (August 2005). "p53 proteasomal degradation: poly-ubiquitination is not the whole story". Cell Cycle. 4 (8): 1015–8. doi:10.4161/cc.4.8.1900. PMID 16082197.
- ^ a b Shringarpure R, Grune T, Mehlhase J, Davies KJ (January 2003). "Ubiquitin conjugation is not required for the degradation of oxidized proteins by proteasome". The Journal of Biological Chemistry. 278 (1): 311–8. doi:10.1074/jbc.M206279200. PMID 12401807.
- ^ a b c Gille C, Goede A, Schlöetelburg C, Preissner R, Kloetzel PM, Göbel UB, Frömmel C (March 2003). "A comprehensive view on proteasomal sequences: implications for the evolution of the proteasome". Journal of Molecular Biology. 326 (5): 1437–48. doi:10.1016/S0022-2836(02)01470-5. PMID 12595256.
- ^ Bochtler M, Ditzel L, Groll M, Hartmann C, Huber R (1999). "The proteasome". Annual Review of Biophysics and Biomolecular Structure. 28 (1): 295–317. doi:10.1146/annurev.biophys.28.1.295. PMID 10410804.
- ^ Chesnel F, Bazile F, Pascal A, Kubiak JZ (August 2006). "Cyclin B dissociation from CDK1 precedes its degradation upon MPF inactivation in mitotic extracts of Xenopus laevis embryos". Cell Cycle. 5 (15): 1687–98. doi:10.4161/cc.5.15.3123. PMID 16921258.
- ^ Brito DA, Rieder CL (June 2006). "Mitotic checkpoint slippage in humans occurs via cyclin B destruction in the presence of an active checkpoint". Current Biology. 16 (12): 1194–200. doi:10.1016/j.cub.2006.04.043. PMC 2749311. PMID 16782009.
- ^ Havens CG, Ho A, Yoshioka N, Dowdy SF (June 2006). "Regulation of late G1/S phase transition and APC Cdh1 by reactive oxygen species". Molecular and Cellular Biology. 26 (12): 4701–11. doi:10.1128/MCB.00303-06. PMC 1489138. PMID 16738333.
- ^ Bashir T, Dorrello NV, Amador V, Guardavaccaro D, Pagano M (March 2004). "Control of the SCF(Skp2-Cks1) ubiquitin ligase by the APC/C(Cdh1) ubiquitin ligase". Nature. 428 (6979): 190–3. doi:10.1038/nature02330. PMID 15014502. S2CID 4401971.
- ^ Higashitsuji H, Liu Y, Mayer RJ, Fujita J (October 2005). "The oncoprotein gankyrin negatively regulates both p53 and RB by enhancing proteasomal degradation". Cell Cycle. 4 (10): 1335–7. doi:10.4161/cc.4.10.2107. PMID 16177571.
- ^ Tarrason Risa, Gabriel; Hurtig, Fredrik; Bray, Sian; Hafner, Anne E.; Harker-Kirschneck, Lena; Faull, Peter; Davis, Colin; Papatziamou, Dimitra; Mutavchiev, Delyan R.; Fan, Catherine; Meneguello, Leticia; Arashiro Pulschen, Andre; Dey, Gautam; Culley, Siân; Kilkenny, Mairi; Souza, Diorge P.; Pellegrini, Luca; de Bruin, Robertus A. M.; Henriques, Ricardo; Snijders, Ambrosius P.; Šarić, Anđela; Lindås, Ann-Christin; Robinson, Nicholas P.; Baum, Buzz (7 August 2020). "The proteasome controls ESCRT-III–mediated cell division in an archaeon". Science. 369 (6504): eaaz2532. doi:10.1126/science.aaz2532. PMC 7116001. PMID 32764038.
- ^ Dharmasiri S, Estelle M (2002). "The role of regulated protein degradation in auxin response". Plant Molecular Biology. 49 (3–4): 401–9. doi:10.1023/A:1015203013208. PMID 12036263. S2CID 7669386.
- ^ Weijers D, Benkova E, Jäger KE, Schlereth A, Hamann T, Kientz M, Wilmoth JC, Reed JW, Jürgens G (May 2005). "Developmental specificity of auxin response by pairs of ARF and Aux/IAA transcriptional regulators". The EMBO Journal. 24 (10): 1874–85. doi:10.1038/sj.emboj.7600659. PMC 1142592. PMID 15889151.
- ^ Haas AL, Baboshina O, Williams B, Schwartz LM (April 1995). "Coordinated induction of the ubiquitin conjugation pathway accompanies the developmentally programmed death of insect skeletal muscle". The Journal of Biological Chemistry. 270 (16): 9407–12. doi:10.1074/jbc.270.16.9407. PMID 7721865.
- ^ Schwartz LM, Myer A, Kosz L, Engelstein M, Maier C (October 1990). "Activation of polyubiquitin gene expression during developmentally programmed cell death". Neuron. 5 (4): 411–9. doi:10.1016/0896-6273(90)90080-Y. PMID 2169771. S2CID 33829749.
- ^ Löw P, Bussell K, Dawson SP, Billett MA, Mayer RJ, Reynolds SE (January 1997). "Expression of a 26S proteasome ATPase subunit, MS73, in muscles that undergo developmentally programmed cell death, and its control by ecdysteroid hormones in the insect Manduca sexta". FEBS Letters. 400 (3): 345–9. doi:10.1016/S0014-5793(96)01413-5. PMID 9009228. S2CID 10873052.
- ^ Pitzer F, Dantes A, Fuchs T, Baumeister W, Amsterdam A (September 1996). "Removal of proteasomes from the nucleus and their accumulation in apoptotic blebs during programmed cell death". FEBS Letters. 394 (1): 47–50. doi:10.1016/0014-5793(96)00920-9. PMID 8925925. S2CID 29256092.
- ^ a b Adams J, Palombella VJ, Sausville EA, Johnson J, Destree A, Lazarus DD, Maas J, Pien CS, Prakash S, Elliott PJ (June 1999). "Proteasome inhibitors: a novel class of potent and effective antitumor agents". Cancer Research. 59 (11): 2615–22. PMID 10363983.
- ^ Orlowski RZ (April 1999). "The role of the ubiquitin-proteasome pathway in apoptosis". Cell Death and Differentiation. 6 (4): 303–13. doi:10.1038/sj.cdd.4400505. PMID 10381632.
- ^ Garrido C, Brunet M, Didelot C, Zermati Y, Schmitt E, Kroemer G (November 2006). "Heat shock proteins 27 and 70: anti-apoptotic proteins with tumorigenic properties". Cell Cycle. 5 (22): 2592–601. doi:10.4161/cc.5.22.3448. PMID 17106261.
- ^ Park SH, Bolender N, Eisele F, Kostova Z, Takeuchi J, Coffino P, Wolf DH (January 2007). "The cytoplasmic Hsp70 chaperone machinery subjects misfolded and endoplasmic reticulum import-incompetent proteins to degradation via the ubiquitin-proteasome system". Molecular Biology of the Cell. 18 (1): 153–65. doi:10.1091/mbc.E06-04-0338. PMC 1751312. PMID 17065559.
- ^ Dai Q, Qian SB, Li HH, McDonough H, Borchers C, Huang D, Takayama S, Younger JM, Ren HY, Cyr DM, Patterson C (November 2005). "Regulation of the cytoplasmic quality control protein degradation pathway by BAG2". The Journal of Biological Chemistry. 280 (46): 38673–81. doi:10.1074/jbc.M507986200. PMID 16169850.
- ^ Bader N, Grune T (2006). "Protein oxidation and proteolysis". Biological Chemistry. 387 (10–11): 1351–5. doi:10.1515/BC.2006.169. PMID 17081106. S2CID 30385354.
- ^ Davies KJ (2003). "Degradation of oxidized proteins by the 20S proteasome". Biochimie. 83 (3–4): 301–10. doi:10.1016/S0300-9084(01)01250-0. PMID 11295490.
- ^ a b Lehman NL (September 2009). "The ubiquitin proteasome system in neuropathology". Acta Neuropathologica. 118 (3): 329–47. doi:10.1007/s00401-009-0560-x. PMC 2716447. PMID 19597829.
- ^ McNaught KS, Jackson T, JnoBaptiste R, Kapustin A, Olanow CW (May 2006). "Proteasomal dysfunction in sporadic Parkinson's disease". Neurology. 66 (10 Suppl 4): S37–49. doi:10.1212/01.wnl.0000221745.58886.2e. PMID 16717251.
- ^ Sharma N, Brandis KA, Herrera SK, Johnson BE, Vaidya T, Shrestha R, Debburman SK (2006). "alpha-Synuclein budding yeast model: toxicity enhanced by impaired proteasome and oxidative stress". Journal of Molecular Neuroscience. 28 (2): 161–78. doi:10.1385/JMN:28:2:161. PMID 16679556.
- ^ Murata S, Sasaki K, Kishimoto T, Niwa S, Hayashi H, Takahama Y, Tanaka K (June 2007). "Regulation of CD8+ T cell development by thymus-specific proteasomes". Science. 316 (5829): 1349–53. Bibcode:2007Sci...316.1349M. doi:10.1126/science.1141915. PMID 17540904. S2CID 37185716.
- ^ Cascio P, Hilton C, Kisselev AF, Rock KL, Goldberg AL (May 2001). "26S proteasomes and immunoproteasomes produce mainly N-extended versions of an antigenic peptide". The EMBO Journal. 20 (10): 2357–66. doi:10.1093/emboj/20.10.2357. PMC 125470. PMID 11350924.
- ^ Mallery DL, McEwan WA, Bidgood SR, Towers GJ, Johnson CM, James LC (November 2010). "Antibodies mediate intracellular immunity through tripartite motif-containing 21 (TRIM21)". Proceedings of the National Academy of Sciences of the United States of America. 107 (46): 19985–19990. Bibcode:2010PNAS..10719985M. doi:10.1073/pnas.1014074107. PMC 2993423. PMID 21045130.
- ^ Fenteany G, Standaert RF, Lane WS, Choi S, Corey EJ, Schreiber SL (May 1995). "Inhibition of proteasome activities and subunit-specific amino-terminal threonine modification by lactacystin". Science. 268 (5211): 726–31. Bibcode:1995Sci...268..726F. doi:10.1126/science.7732382. PMID 7732382. S2CID 37779687.
- ^ 미국 식품의약국(FDA)의 보도 자료는 2003년 5월 13일 웨이백 머신에 2007년 2월 19일 보관되었다.접속 날짜 2006년 12월 29일.FDA Velcade 정보 페이지를 참조하십시오.
- ^ Fisher RI, Bernstein SH, Kahl BS, Djulbegovic B, Robertson MJ, de Vos S, Epner E, Krishnan A, Leonard JP, Lonial S, Stadtmauer EA, O'Connor OA, Shi H, Boral AL, Goy A (October 2006). "Multicenter phase II study of bortezomib in patients with relapsed or refractory mantle cell lymphoma". Journal of Clinical Oncology. 24 (30): 4867–74. doi:10.1200/JCO.2006.07.9665. PMID 17001068.
- ^ Jakob C, Egerer K, Liebisch P, Türkmen S, Zavrski I, Kuckelkorn U, Heider U, Kaiser M, Fleissner C, Sterz J, Kleeberg L, Feist E, Burmester GR, Kloetzel PM, Sezer O (March 2007). "Circulating proteasome levels are an independent prognostic factor for survival in multiple myeloma". Blood. 109 (5): 2100–5. doi:10.1182/blood-2006-04-016360. PMID 17095627.
- ^ Shah SA, Potter MW, McDade TP, Ricciardi R, Perugini RA, Elliott PJ, Adams J, Callery MP (2001). "26S proteasome inhibition induces apoptosis and limits growth of human pancreatic cancer". Journal of Cellular Biochemistry. 82 (1): 110–22. doi:10.1002/jcb.1150. PMID 11400168. S2CID 21223980.
- ^ Nawrocki ST, Sweeney-Gotsch B, Takamori R, McConkey DJ (January 2004). "The proteasome inhibitor bortezomib enhances the activity of docetaxel in orthotopic human pancreatic tumor xenografts". Molecular Cancer Therapeutics. 3 (1): 59–70. PMID 14749476.
- ^ Schenkein D (June 2002). "Proteasome inhibitors in the treatment of B-cell malignancies". Clinical Lymphoma. 3 (1): 49–55. doi:10.3816/CLM.2002.n.011. PMID 12141956.
- ^ O'Connor OA, Wright J, Moskowitz C, Muzzy J, MacGregor-Cortelli B, Stubblefield M, Straus D, Portlock C, Hamlin P, Choi E, Dumetrescu O, Esseltine D, Trehu E, Adams J, Schenkein D, Zelenetz AD (February 2005). "Phase II clinical experience with the novel proteasome inhibitor bortezomib in patients with indolent non-Hodgkin's lymphoma and mantle cell lymphoma". Journal of Clinical Oncology. 23 (4): 676–84. doi:10.1200/JCO.2005.02.050. PMID 15613699.
- ^ Messinger YH, Gaynon PS, Sposto R, van der Giessen J, Eckroth E, Malvar J, Bostrom BC (July 2012). "Bortezomib with chemotherapy is highly active in advanced B-precursor acute lymphoblastic leukemia: Therapeutic Advances in Childhood Leukemia & Lymphoma (TACL) Study". Blood. 120 (2): 285–90. doi:10.1182/blood-2012-04-418640. PMID 22653976.
- ^ Lambrou GI, Papadimitriou L, Chrousos GP, Vlahopoulos SA (April 2012). "Glucocorticoid and proteasome inhibitor impact on the leukemic lymphoblast: multiple, diverse signals converging on a few key downstream regulators". Molecular and Cellular Endocrinology. 351 (2): 142–51. doi:10.1016/j.mce.2012.01.003. PMID 22273806. S2CID 28749125.
- ^ Schmidtke G, Holzhütter HG, Bogyo M, Kairies N, Groll M, de Giuli R, Emch S, Groettrup M (December 1999). "How an inhibitor of the HIV-I protease modulates proteasome activity". The Journal of Biological Chemistry. 274 (50): 35734–40. doi:10.1074/jbc.274.50.35734. PMID 10585454.
- ^ Laurent N, de Boüard S, Guillamo JS, Christov C, Zini R, Jouault H, Andre P, Lotteau V, Peschanski M (February 2004). "Effects of the proteasome inhibitor ritonavir on glioma growth in vitro and in vivo". Molecular Cancer Therapeutics. 3 (2): 129–36. PMID 14985453.
- ^ Zollner TM, Podda M, Pien C, Elliott PJ, Kaufmann R, Boehncke WH (March 2002). "Proteasome inhibition reduces superantigen-mediated T cell activation and the severity of psoriasis in a SCID-hu model". The Journal of Clinical Investigation. 109 (5): 671–9. doi:10.1172/JCI12736. PMC 150886. PMID 11877475.
- ^ Elliott PJ, Pien CS, McCormack TA, Chapman ID, Adams J (August 1999). "Proteasome inhibition: A novel mechanism to combat asthma". The Journal of Allergy and Clinical Immunology. 104 (2 Pt 1): 294–300. doi:10.1016/S0091-6749(99)70369-6. PMID 10452747.
- ^ Verdoes M, Florea BI, Menendez-Benito V, Maynard CJ, Witte MD, van der Linden WA, van den Nieuwendijk AM, Hofmann T, Berkers CR, van Leeuwen FW, Groothuis TA, Leeuwenburgh MA, Ovaa H, Neefjes JJ, Filippov DV, van der Marel GA, Dantuma NP, Overkleeft HS (November 2006). "A fluorescent broad-spectrum proteasome inhibitor for labeling proteasomes in vitro and in vivo". Chemistry & Biology. 13 (11): 1217–26. doi:10.1016/j.chembiol.2006.09.013. PMID 17114003.
- ^ Kleiger G, Mayor T (June 2014). "Perilous journey: a tour of the ubiquitin-proteasome system". Trends in Cell Biology. 24 (6): 352–9. doi:10.1016/j.tcb.2013.12.003. PMC 4037451. PMID 24457024.
- ^ Goldberg AL, Stein R, Adams J (August 1995). "New insights into proteasome function: from archaebacteria to drug development". Chemistry & Biology. 2 (8): 503–8. doi:10.1016/1074-5521(95)90182-5. PMID 9383453.
- ^ Sulistio YA, Heese K (January 2015). "The Ubiquitin–Proteasome System and Molecular Chaperone Deregulation in Alzheimer's Disease". Molecular Neurobiology. 53 (2): 905–31. doi:10.1007/s12035-014-9063-4. PMID 25561438. S2CID 14103185.
- ^ Ortega Z, Lucas JJ (2014). "Ubiquitin-proteasome system involvement in Huntington's disease". Frontiers in Molecular Neuroscience. 7: 77. doi:10.3389/fnmol.2014.00077. PMC 4179678. PMID 25324717.
- ^ Sandri M, Robbins J (June 2014). "Proteotoxicity: an underappreciated pathology in cardiac disease". Journal of Molecular and Cellular Cardiology. 71: 3–10. doi:10.1016/j.yjmcc.2013.12.015. PMC 4011959. PMID 24380730.
- ^ Drews O, Taegtmeyer H (December 2014). "Targeting the ubiquitin–proteasome system in heart disease: the basis for new therapeutic strategies". Antioxidants & Redox Signaling. 21 (17): 2322–43. doi:10.1089/ars.2013.5823. PMC 4241867. PMID 25133688.
- ^ Wang ZV, Hill JA (February 2015). "Protein quality control and metabolism: bidirectional control in the heart". Cell Metabolism. 21 (2): 215–26. doi:10.1016/j.cmet.2015.01.016. PMC 4317573. PMID 25651176.
- ^ a b Karin M, Delhase M (February 2000). "The I kappa B kinase (IKK) and NF-kappa B: key elements of proinflammatory signalling". Seminars in Immunology. 12 (1): 85–98. doi:10.1006/smim.2000.0210. PMID 10723801.
- ^ Ermolaeva MA, Dakhovnik A, Schumacher B (January 2015). "Quality control mechanisms in cellular and systemic DNA damage responses". Ageing Research Reviews. 23 (Pt A): 3–11. doi:10.1016/j.arr.2014.12.009. PMC 4886828. PMID 25560147.
- ^ Checler F, da Costa CA, Ancolio K, Chevallier N, Lopez-Perez E, Marambaud P (July 2000). "Role of the proteasome in Alzheimer's disease". Biochimica et Biophysica Acta (BBA) - Molecular Basis of Disease. 1502 (1): 133–8. doi:10.1016/s0925-4439(00)00039-9. PMID 10899438.
- ^ a b Chung KK, Dawson VL, Dawson TM (November 2001). "The role of the ubiquitin-proteasomal pathway in Parkinson's disease and other neurodegenerative disorders". Trends in Neurosciences. 24 (11 Suppl): S7–14. doi:10.1016/s0166-2236(00)01998-6. PMID 11881748. S2CID 2211658.
- ^ a b Ikeda K, Akiyama H, Arai T, Ueno H, Tsuchiya K, Kosaka K (July 2002). "Morphometrical reappraisal of motor neuron system of Pick's disease and amyotrophic lateral sclerosis with dementia". Acta Neuropathologica. 104 (1): 21–8. doi:10.1007/s00401-001-0513-5. PMID 12070660. S2CID 22396490.
- ^ Manaka H, Kato T, Kurita K, Katagiri T, Shikama Y, Kujirai K, Kawanami T, Suzuki Y, Nihei K, Sasaki H (May 1992). "Marked increase in cerebrospinal fluid ubiquitin in Creutzfeldt–Jakob disease". Neuroscience Letters. 139 (1): 47–9. doi:10.1016/0304-3940(92)90854-z. PMID 1328965. S2CID 28190967.
- ^ Mathews KD, Moore SA (January 2003). "Limb-girdle muscular dystrophy". Current Neurology and Neuroscience Reports. 3 (1): 78–85. doi:10.1007/s11910-003-0042-9. PMID 12507416. S2CID 5780576.
- ^ Mayer RJ (March 2003). "From neurodegeneration to neurohomeostasis: the role of ubiquitin". Drug News & Perspectives. 16 (2): 103–8. doi:10.1358/dnp.2003.16.2.829327. PMID 12792671.
- ^ Calise J, Powell SR (February 2013). "The ubiquitin proteasome system and myocardial ischemia". American Journal of Physiology. Heart and Circulatory Physiology. 304 (3): H337–49. doi:10.1152/ajpheart.00604.2012. PMC 3774499. PMID 23220331.
- ^ Predmore JM, Wang P, Davis F, Bartolone S, Westfall MV, Dyke DB, Pagani F, Powell SR, Day SM (March 2010). "Ubiquitin proteasome dysfunction in human hypertrophic and dilated cardiomyopathies". Circulation. 121 (8): 997–1004. doi:10.1161/CIRCULATIONAHA.109.904557. PMC 2857348. PMID 20159828.
- ^ Powell SR (July 2006). "The ubiquitin-proteasome system in cardiac physiology and pathology". American Journal of Physiology. Heart and Circulatory Physiology. 291 (1): H1–H19. doi:10.1152/ajpheart.00062.2006. PMID 16501026.
- ^ Adams J (April 2003). "Potential for proteasome inhibition in the treatment of cancer". Drug Discovery Today. 8 (7): 307–15. doi:10.1016/s1359-6446(03)02647-3. PMID 12654543.
- ^ Ben-Neriah Y (January 2002). "Regulatory functions of ubiquitination in the immune system". Nature Immunology. 3 (1): 20–6. doi:10.1038/ni0102-20. PMID 11753406. S2CID 26973319.
- ^ Egerer K, Kuckelkorn U, Rudolph PE, Rückert JC, Dörner T, Burmester GR, Kloetzel PM, Feist E (October 2002). "Circulating proteasomes are markers of cell damage and immunologic activity in autoimmune diseases". The Journal of Rheumatology. 29 (10): 2045–52. PMID 12375310.
추가 읽기
- Glickman MH, Adir N (January 2004). "The proteasome and the delicate balance between destruction and rescue". PLOS Biology. 2 (1): e13. doi:10.1371/journal.pbio.0020013. PMC 314468. PMID 14737189.
- 서브유닛과 그림 목록이 있는 이스트 26S 프로테아솜
- Ciechanover A (September 2005). "Early work on the ubiquitin proteasome system, an interview with Aaron Ciechanover. Interview by CDD". Cell Death and Differentiation. 12 (9): 1167–77. doi:10.1038/sj.cdd.4401691. PMID 16094393.
- Hershko A (September 2005). "Early work on the ubiquitin proteasome system, an interview with Avram Hershko. Interview by CDD". Cell Death and Differentiation. 12 (9): 1158–61. doi:10.1038/sj.cdd.4401709. PMID 16094391.
- Rose I (September 2005). "Early work on the ubiquitin proteasome system, an interview with Irwin Rose. Interview by CDD". Cell Death and Differentiation. 12 (9): 1162–6. doi:10.1038/sj.cdd.4401700. PMID 16094392.
- Cvek B, Dvorak Z (2007). "Targeting of nuclear factor-kappaB and proteasome by dithiocarbamate complexes with metals". Current Pharmaceutical Design. 13 (30): 3155–67. doi:10.2174/138161207782110390. PMID 17979756. Archived from the original on 29 July 2012.
외부 링크
| 위키미디어 커먼즈에는 프로테아솜과 관련된 미디어가 있다. |