보존된 시퀀스
Conserved sequence진화생물학에서 보존배열은 핵산(DNA와 RNA) 또는 단백질(정통배열), 게놈(패럴로그 배열) 내 또는 기증자와 수용체 분류군(제놀로그 배열) 사이의 동일하거나 유사한 배열이다.보존은 자연 선택에 의해 시퀀스가 유지되었음을 나타냅니다.
고도로 보존된 염기서열은 계통수 위쪽으로, 따라서 지질학적으로 훨씬 이전까지 비교적 변하지 않은 염기서열이다.고도로 보존된 배열의 예로는 생명체의 모든 영역에 존재하는 리보솜의 RNA 성분, 진핵생물 사이에 널리 퍼져 있는 호메오박스 배열, 박테리아 내 tmRNA 등이 있다.염기서열 보존 연구는 유전체학, 단백질학, 진화생물학, 계통학, 생물정보학, 수학 분야와 겹친다.
역사
유전에서의 DNA의 역할의 발견과 1949년 [2]프레드릭 생어의 동물 인슐린 사이의 변이에 대한 관찰은 초기 분자 생물학자들이 분자 관점에서 [3][4]분류학을 연구하도록 자극했다.1960년대 연구는 헤모글로빈과[5] 시토크롬 [6]c와 같은 알려진 직교 단백질 사이의 유사성을 측정하기 위해 DNA 교배와 단백질 교차 반응 기술을 사용했다.1965년, 에밀 주커칸들과 라이너스 폴링은 분자 [7]시계의 개념을 도입했고, 아미노산 치환의 꾸준한 비율이 두 유기체가 분리된 이후 시간을 추정하는데 사용될 수 있다고 제안했다.초기 계통 발생이 화석 기록과 밀접하게 일치했지만, 일부 유전자가 다른 속도로 진화하는 것으로 보인다는 관찰은 분자 진화 [3][4]이론의 발전을 이끌었다.마가렛 데이호프의 1966년 페로독신 염기서열 비교는 자연 선택이 생명에 필수적인 단백질 [8]염기서열을 보존하고 최적화하는 역할을 한다는 것을 보여주었다.
메커니즘
여러 세대에 걸쳐, 진화 계통의 게놈에 있는 핵산 배열은 무작위 돌연변이와 [9][10]결실로 인해 시간이 지남에 따라 점차 변화할 수 있다.염기서열은 염색체 재배열로 인해 재결합되거나 삭제될 수도 있다.보존된 염기서열은 그러한 힘에도 불구하고 게놈에 계속 존재하는 염기서열로,[11] 백그라운드 돌연변이율보다 느린 돌연변이율을 가진다.
보존은 코드화 및 코드화되지 않은 핵산 배열에서 발생할 수 있다.고도로 보존된 DNA 염기서열은 기능적 가치가 있다고 생각되지만, 많은 고도로 보존되지 않은 DNA 염기서열의 역할은 잘 알려져 [12][13]있지 않다.배열이 보존되는 정도는 다양한 선택 압력, 돌연변이에 대한 견고성, 모집단 크기 및 유전적 표류에 의해 영향을 받을 수 있다.많은 기능적 배열은 또한 [14]단백질 도메인과 같이 독립적인 선택 압력의 영향을 받을 수 있는 영역을 포함하는 모듈러형이다.
부호화 시퀀스
코드 배열에서 유전자 코드의 축퇴는 코드 배열의 동의어 돌연변이가 그 단백질 [15]생성물의 아미노산 배열에 영향을 주지 않는 것을 의미하기 때문에 핵산과 아미노산 배열을 다른 범위로 보존할 수 있다.
단백질 또는 도메인의 구조 또는 기능을 유지하기 위해 아미노산 배열을 보존할 수 있다.보존된 단백질은 아미노산 치환량이 적거나 비슷한 생화학적 [16]성질을 가진 아미노산을 대체할 가능성이 높다.배열 내에서, 접힘, 구조적 안정성에 중요하거나 결합 부위를 형성하는 아미노산은 더 많이 [17][18]보존될 수 있다.
단백질 코드 유전자의 핵산 배열은 또한 다른 선택 압력에 의해 보존될 수 있다.일부 유기체의 코돈 사용 편향은 일련의 동의어 돌연변이의 유형을 제한할 수 있다.코드화 유전자의 mRNA에서 2차 구조를 일으키는 핵산 배열은 일부 구조가 번역에 부정적으로 영향을 미칠 수 있으므로 선택되거나 mRNA가 기능적 비코드 [19][20]RNA로서도 작용하는 경우에 보존될 수 있다.
비코딩
유전자 조절에 중요한 비코드 배열, 예를 들어 리보솜과 전사 인자의 결합 또는 인식 부위는 게놈 내에 보존될 수 있다.예를 들어 보존된 유전자 또는 오퍼론의 프로모터도 보존할 수 있다.단백질과 마찬가지로 비코드 RNA(ncRNA)의 구조와 기능에 중요한 핵산도 보존될 수 있다.그러나, ncRNA의 배열 보존은 단백질 코드 배열에 비해 일반적으로 열악하며,[21][22] 구조나 기능에 기여하는 염기쌍이 대신 보존되는 경우가 많다.
신분증
보존된 염기서열은 일반적으로 염기서열 정렬에 기초한 생체정보학 접근법에 의해 식별된다.2000년대 [23][24]초부터 높은 처리량 DNA 염기서열 분석과 단백질 질량 분석의 진보로 단백질 염기서열과 전체 게놈의 비교 가용성이 크게 향상되었다.
호몰로지 검색
보존된 시퀀스는 BLAST, HMMER, OrthologR [25]및 Infernal과 [26]같은 도구를 사용하여 호몰로지 검색을 통해 식별할 수 있습니다.호몰로지 검색 도구는 개별 핵산 또는 단백질 염기서열을 입력으로 사용하거나 알려진 관련 염기서열의 다중 염기서열 정렬에서 생성된 통계 모델을 사용할 수 있습니다.프로필-HM과 같은 통계 모델 및 구조 [27]정보를 포함하는 RNA 공분산 모델은 보다 멀리 관련된 시퀀스를 검색할 때 유용합니다.입력 시퀀스는 관련된 개인 또는 다른 종으로부터의 시퀀스의 데이터베이스에 대해 정렬됩니다.그런 다음 일치하는 아미노산 또는 염기의 수와 정렬에 의해 생성된 간격 또는 결손의 수를 기준으로 정렬을 채점합니다.PAM 및 BLOSUM과 같은 치환 매트릭스를 사용하여 허용 가능한 보존 치환을 식별할 수 있다.점수가 높은 정렬은 상동 시퀀스에서 나온 것으로 가정한다.배열의 보존은 광범위한 계통 발생 [28]범위에 걸쳐 매우 유사한 상동성을 검출함으로써 추론할 수 있다.
다중 시퀀스 얼라인먼트
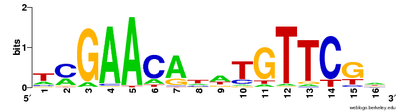
여러 시퀀스 정렬을 사용하여 보존된 시퀀스를 시각화할 수 있습니다.그 CLUSTAL 형식은 정렬의 주석으로 기둥에 일반 텍스트, 보수적인 돌연변이들semi-conservative 돌연변이(.)(:)conserved 순서(*)을 나타내는 표시, 그리고non-conservative 돌연변이()[30]순서 로고 또한 각 지점에서 맞춤에 나오는 인물들의 비율을 대표하는 등으로 순서를 보여 준다.타고높이[29]
게놈 정렬
전체 게놈 정렬(WGAs)은 또한 종에 걸쳐 고도로 보존된 영역을 식별하기 위해 사용될 수 있다.현재 WGA 툴의 정확성과 확장성은 재배치, 반복 영역 및 [32]많은 진핵생물 게놈의 큰 크기를 다루는 계산 복잡성으로 인해 제한적이다.그러나, 30개 이상의 밀접하게 관련된 박테리아(원핵생물)의 WGA는 현재 점점 [33][34]더 실현가능해지고 있다.
스코어링 시스템
다른 접근방식은 예상되는 배경(중립) 돌연변이율에 따라 다르게 변이되는 시퀀스를 식별하려는 통계 테스트에 기초한 보존 측정을 사용한다.
GERP(Genomic Evolutionary Rate Profiling) 프레임워크는 종에 걸친 유전자 배열의 보존 점수를 매긴다.이 접근법은 다중 배열 정렬에서 종의 집합에서 중성 돌연변이 비율을 추정하여 예상보다 적은 돌연변이를 보이는 염기서열 영역을 식별한다.그런 다음 이러한 영역에는 관찰된 돌연변이율과 예상 백그라운드 돌연변이율 사이의 차이에 따라 점수가 할당된다.GERP 점수가 높으면 보존율이 높은 [35][36]시퀀스를 나타냅니다.
LIST(Local Identity and Shared Taxa)는[37] 사람과 가까운 관계인 종에서 관찰된 변동이 먼 친척 종에 비해 보존을 평가할 때 더 유의하다는 가정에 기초한다.따라서 LIST는 각 위치 주변의 국소 정렬 정체성을 이용하여 다중 시퀀스 정렬(MSA)에서 관련 시퀀스를 식별한 다음 인간에 대한 이들 시퀀스의 분류학적 거리에 기초하여 보존을 추정한다.다른 도구와 달리 LIST는 MSA의 변동 개수/빈도를 무시합니다.
아미노드는[39] 여러 배열과 계통학적 분석을 결합하여 상동성 단백질의 변화를 분석하고 진화적 변화의 국소 속도를 나타내는 플롯을 생성합니다.이 접근방식은 단백질에서 진화적으로 제약된 영역을 식별하는데, 이는 정제 선택 대상이 되는 세그먼트이며 일반적으로 정상적인 단백질 기능에 중요하다.
PhyloP 및 PhyloHM과 같은 다른 접근법은 치환율의 확률분포를 비교하기 위해 통계적 계통유전학 방법을 포함하며, 이는 보존 및 가속 돌연변이를 모두 검출할 수 있게 한다.우선 계통수에 근거해 복수 배열 배열의 컬럼에 대해 발생할 것으로 예상되는 치환수의 백그라운드 확률 분포를 생성한다.관심 종 사이의 추정된 진화적 관계는 치환의 유의성을 계산하는데 사용된다(즉, 밀접하게 관련된 두 종 사이의 치환이 원거리 관련 종보다 발생할 가능성이 낮기 때문에 더 유의하다).보존을 검출하기 위해 다중 시퀀스 정렬의 서브셋에 대해 확률 분포를 계산하고 우도비 검정 또는 점수 검정과 같은 통계적 테스트를 사용하여 배경 분포와 비교합니다.그런 다음 두 분포를 비교하여 생성된 P-값을 사용하여 보존된 영역을 식별합니다.PhyloHM은 숨겨진 마르코프 모델을 사용하여 확률 분포를 생성합니다.PhyloP 소프트웨어 패키지는 우도비 테스트 또는 점수 테스트를 사용하여 확률 분포를 비교하고 GERP와 유사한 스코어링 [40][41][42]시스템을 사용합니다.
극도의 보존
초절약 요소
UCE(Ultra-Conserved Elements)는 여러 분류학적 그룹 간에 매우 유사하거나 동일한 시퀀스입니다.이것들은 척추동물에서 [43]처음 발견되었고, 이후 널리 퍼지는 분류군 내에서 확인되었다.[44]UCE의 기원과 기능은 [45]잘 알려져 있지 않지만, 양수,[46][47] 곤충, 그리고 동물과 [48]식물 사이의 깊은 시간 차이를 조사하는데 사용되어 왔다.
보편적으로 보존된 유전자
가장 잘 보존된 유전자는 모든 유기체에서 찾을 수 있는 유전자들이다.이들은 주로 전사와 번역에 필요한 ncRNA와 단백질로 구성되며, 이는 모든 [49]생물의 마지막 보편적인 공통 조상으로부터 보존된 것으로 추정된다.
보편적으로 보존되는 것으로 밝혀진 유전자 또는 유전자 패밀리에는 GTP 결합 신장 인자, 메티오닌 아미노펩티다아제 2, 세린 히드록시메틸전달효소 및 ATP 전달체가 [50]포함된다.RNA 중합효소나 헬리케이스 등의 전사기계 성분과 리보솜 RNA, tRNA 및 리보솜 단백질 등의 번역기계 성분도 보편적으로 [51]보존된다.
적용들
계통학 및 분류학
보존된 염기서열 세트는 유사한 염기서열을 가진 유기체가 밀접하게 [52]관련되어 있다고 가정할 수 있기 때문에 계통수 생성에 종종 사용된다.시퀀스의 선택은 연구의 분류학적 범위에 따라 달라질 수 있다.예를 들어 16S RNA 및 기타 리보솜 배열과 같이 가장 보존성이 높은 유전자는 메타게노믹스 [53][54]연구에서 깊은 계통발생적 관계를 재구축하고 세균의 필라를 동정하는 데 유용하다.분지 내에 보존되어 있지만 하우스키핑 유전자와 같은 돌연변이를 겪는 배열은 종의 [55][56][57]관계를 연구하는데 사용될 수 있다.보존된 rRNA 유전자 간격에 필요하지만 빠른 진화를 겪는 내부 전사 스페이서(ITS) 영역은 빠르게 진화하는 균류와 [58][59][60][61]균주를 분류하는 데 일반적으로 사용된다.
의학 연구
고도로 보존된 배열은 종종 중요한 생물학적 기능을 가지고 있기 때문에, 그것들은 유전적 질병의 원인을 규명하는 데 유용한 출발점이 될 수 있다.많은 선천성 대사 장애와 리소좀 저장 질환은 개별 보존된 유전자의 변화로 인해 발생하며, 질병의 증상의 근본 원인이 되는 효소가 누락되거나 결함이 발생한다.유전병은 인간과 쥐, 초파리 [63]등 실험실[62] 유기체 사이에 보존된 염기서열을 확인하고 이들 [64]유전자의 녹아웃 효과를 연구함으로써 예측할 수 있다.게놈 전체의 연관 연구는 질병이나 건강 결과와 관련된 보존된 배열의 변화를 식별하기 위해 사용될 수도 있다.알제히머 병에서는 24개 이상의 새로운 잠재적 감수성 위치가 발견되었다[65][66].
기능 주석
보존된 배열을 식별하는 것은 [67]유전자와 같은 기능적 배열을 발견하고 예측하는 데 사용될 수 있다.단백질 도메인과 같이 알려진 기능을 가진 보존된 배열은 배열의 기능을 예측하는 데 사용될 수도 있다.Pfam 및 Conserved Domain Database와 같은 보존 단백질 도메인의 데이터베이스는 예측 단백질 코딩 [68]유전자의 기능 도메인에 주석을 달기 위해 사용될 수 있다.
「 」를 참조해 주세요.
레퍼런스
- ^ "Clustal FAQ #Symbols". Clustal. Archived from the original on 24 October 2016. Retrieved 8 December 2014.
- ^ Sanger, F. (24 September 1949). "Species Differences in Insulins". Nature. 164 (4169): 529. Bibcode:1949Natur.164..529S. doi:10.1038/164529a0. PMID 18141620. S2CID 4067991.
- ^ a b Marmur, J; Falkow, S; Mandel, M (October 1963). "New Approaches to Bacterial Taxonomy". Annual Review of Microbiology. 17 (1): 329–372. doi:10.1146/annurev.mi.17.100163.001553. PMID 14147455.
- ^ a b Pace, N. R.; Sapp, J.; Goldenfeld, N. (17 January 2012). "Phylogeny and beyond: Scientific, historical, and conceptual significance of the first tree of life". Proceedings of the National Academy of Sciences. 109 (4): 1011–1018. Bibcode:2012PNAS..109.1011P. doi:10.1073/pnas.1109716109. PMC 3268332. PMID 22308526.
- ^ Zuckerlandl, Emile; Pauling, Linus B. (1962). "Molecular disease, evolution, and genetic heterogeneity". Horizons in Biochemistry: 189–225.
- ^ Margoliash, E (October 1963). "Primary Structure and Evolution of Cytochrome C". Proc Natl Acad Sci U S A. 50 (4): 672–679. Bibcode:1963PNAS...50..672M. doi:10.1073/pnas.50.4.672. PMC 221244. PMID 14077496.
- ^ Zuckerkandl, E; Pauling, LB (1965). Evolutionary Divergence and Convergence in Proteins. Evolving Genes and Proteins. pp. 96–166. doi:10.1016/B978-1-4832-2734-4.50017-6. ISBN 9781483227344.
- ^ Eck, R. V.; Dayhoff, M. O. (15 April 1966). "Evolution of the Structure of Ferredoxin Based on Living Relics of Primitive Amino Acid Sequences". Science. 152 (3720): 363–366. Bibcode:1966Sci...152..363E. doi:10.1126/science.152.3720.363. PMID 17775169. S2CID 23208558.
- ^ Kimura, M (17 February 1968). "Evolutionary Rate at the Molecular Level". Nature. 217 (5129): 624–626. Bibcode:1968Natur.217..624K. doi:10.1038/217624a0. PMID 5637732. S2CID 4161261.
- ^ King, J. L.; Jukes, T. H. (16 May 1969). "Non-Darwinian Evolution". Science. 164 (3881): 788–798. Bibcode:1969Sci...164..788L. doi:10.1126/science.164.3881.788. PMID 5767777.
- ^ Kimura, M; Ohta, T (1974). "On Some Principles Governing Molecular Evolution". Proc Natl Acad Sci USA. 71 (7): 2848–2852. Bibcode:1974PNAS...71.2848K. doi:10.1073/pnas.71.7.2848. PMC 388569. PMID 4527913.
- ^ Asthana, Saurabh; Roytberg, Mikhail; Stamatoyannopoulos, John; Sunyaev, Shamil (28 December 2007). Brudno, Michael (ed.). "Analysis of Sequence Conservation at Nucleotide Resolution". PLOS Computational Biology. 3 (12): e254. Bibcode:2007PLSCB...3..254A. doi:10.1371/journal.pcbi.0030254. ISSN 1553-7358. PMC 2230682. PMID 18166073.
- ^ Cooper, G. M.; Brown, C. D. (1 February 2008). "Qualifying the relationship between sequence conservation and molecular function". Genome Research. 18 (2): 201–205. doi:10.1101/gr.7205808. ISSN 1088-9051. PMID 18245453.
- ^ Gilson, Amy I.; Marshall-Christensen, Ahmee; Choi, Jeong-Mo; Shakhnovich, Eugene I. (2017). "The Role of Evolutionary Selection in the Dynamics of Protein Structure Evolution". Biophysical Journal. 112 (7): 1350–1365. arXiv:1606.05802. Bibcode:2017BpJ...112.1350G. doi:10.1016/j.bpj.2017.02.029. PMC 5390048. PMID 28402878.
- ^ Hunt, Ryan C.; Simhadri, Vijaya L.; Iandoli, Matthew; Sauna, Zuben E.; Kimchi-Sarfaty, Chava (2014). "Exposing synonymous mutations". Trends in Genetics. 30 (7): 308–321. doi:10.1016/j.tig.2014.04.006. PMID 24954581.
- ^ Zhang, Jianzhi (2000). "Rates of Conservative and Radical Nonsynonymous Nucleotide Substitutions in Mammalian Nuclear Genes". Journal of Molecular Evolution. 50 (1): 56–68. Bibcode:2000JMolE..50...56Z. doi:10.1007/s002399910007. ISSN 0022-2844. PMID 10654260. S2CID 15248867.
- ^ Sousounis, Konstantinos; Haney, Carl E; Cao, Jin; Sunchu, Bharath; Tsonis, Panagiotis A (2012). "Conservation of the three-dimensional structure in non-homologous or unrelated proteins". Human Genomics. 6 (1): 10. doi:10.1186/1479-7364-6-10. ISSN 1479-7364. PMC 3500211. PMID 23244440.
- ^ Kairys, Visvaldas; Fernandes, Miguel X. (2007). "SitCon: Binding site residue conservation visualization and protein sequence-to-function tool". International Journal of Quantum Chemistry. 107 (11): 2100–2110. Bibcode:2007IJQC..107.2100K. doi:10.1002/qua.21396. ISSN 0020-7608.
- ^ Chamary, JV; Hurst, Laurence D (2005). "Evidence for selection on synonymous mutations affecting stability of mRNA secondary structure in mammals". Genome Biology. 6 (9): R75. doi:10.1186/gb-2005-6-9-r75. PMC 1242210. PMID 16168082.
- ^ Wadler, C. S.; Vanderpool, C. K. (27 November 2007). "A dual function for a bacterial small RNA: SgrS performs base pairing-dependent regulation and encodes a functional polypeptide". Proceedings of the National Academy of Sciences. 104 (51): 20454–20459. Bibcode:2007PNAS..10420454W. doi:10.1073/pnas.0708102104. PMC 2154452. PMID 18042713.
- ^ Johnsson, Per; Lipovich, Leonard; Grandér, Dan; Morris, Kevin V. (March 2014). "Evolutionary conservation of long non-coding RNAs; sequence, structure, function". Biochimica et Biophysica Acta (BBA) - General Subjects. 1840 (3): 1063–1071. doi:10.1016/j.bbagen.2013.10.035. PMC 3909678. PMID 24184936.
- ^ Freyhult, E. K.; Bollback, J. P.; Gardner, P. P. (6 December 2006). "Exploring genomic dark matter: A critical assessment of the performance of homology search methods on noncoding RNA". Genome Research. 17 (1): 117–125. doi:10.1101/gr.5890907. PMC 1716261. PMID 17151342.
- ^ Margulies, E. H. (1 December 2003). "Identification and Characterization of Multi-Species Conserved Sequences". Genome Research. 13 (12): 2507–2518. doi:10.1101/gr.1602203. ISSN 1088-9051. PMC 403793. PMID 14656959.
- ^ Edwards, John R.; Ruparel, Hameer; Ju, Jingyue (2005). "Mass-spectrometry DNA sequencing". Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 573 (1–2): 3–12. doi:10.1016/j.mrfmmm.2004.07.021. PMID 15829234.
- ^ Drost, Hajk-Georg; Gabel, Alexander; Grosse, Ivo; Quint, Marcel (1 May 2015). "Evidence for Active Maintenance of Phylotranscriptomic Hourglass Patterns in Animal and Plant Embryogenesis". Molecular Biology and Evolution. 32 (5): 1221–1231. doi:10.1093/molbev/msv012. ISSN 0737-4038. PMC 4408408. PMID 25631928.
- ^ Nawrocki, E. P.; Eddy, S. R. (4 September 2013). "Infernal 1.1: 100-fold faster RNA homology searches". Bioinformatics. 29 (22): 2933–2935. doi:10.1093/bioinformatics/btt509. PMC 3810854. PMID 24008419.
- ^ Eddy, SR; Durbin, R (11 June 1994). "RNA sequence analysis using covariance models". Nucleic Acids Research. 22 (11): 2079–88. doi:10.1093/nar/22.11.2079. PMC 308124. PMID 8029015.
- ^ Trivedi, Rakesh; Nagarajaram, Hampapathalu Adimurthy (2020). "Substitution scoring matrices for proteins ‐ An overview". Protein Science. 29 (11): 2150–2163. doi:10.1002/pro.3954. ISSN 0961-8368. PMC 7586916. PMID 32954566.
- ^ a b "Weblogo". UC Berkeley. Retrieved 30 December 2017.
- ^ "Clustal FAQ #Symbols". Clustal. Archived from the original on 24 October 2016. Retrieved 8 December 2014.
- ^ "ECR Browser". ECR Browser. Retrieved 9 January 2018.
- ^ Earl, Dent; Nguyen, Ngan; Hickey, Glenn; Harris, Robert S.; Fitzgerald, Stephen; Beal, Kathryn; Seledtsov, Igor; Molodtsov, Vladimir; Raney, Brian J.; Clawson, Hiram; Kim, Jaebum; Kemena, Carsten; Chang, Jia-Ming; Erb, Ionas; Poliakov, Alexander; Hou, Minmei; Herrero, Javier; Kent, William James; Solovyev, Victor; Darling, Aaron E.; Ma, Jian; Notredame, Cedric; Brudno, Michael; Dubchak, Inna; Haussler, David; Paten, Benedict (December 2014). "Alignathon: a competitive assessment of whole-genome alignment methods". Genome Research. 24 (12): 2077–2089. doi:10.1101/gr.174920.114. PMC 4248324. PMID 25273068.
- ^ Rouli, L.; Merhej, V.; Fournier, P.-E.; Raoult, D. (September 2015). "The bacterial pangenome as a new tool for analysing pathogenic bacteria". New Microbes and New Infections. 7: 72–85. doi:10.1016/j.nmni.2015.06.005. PMC 4552756. PMID 26442149.
- ^ Méric, Guillaume; Yahara, Koji; Mageiros, Leonardos; Pascoe, Ben; Maiden, Martin C. J.; Jolley, Keith A.; Sheppard, Samuel K.; Bereswill, Stefan (27 March 2014). "A Reference Pan-Genome Approach to Comparative Bacterial Genomics: Identification of Novel Epidemiological Markers in Pathogenic Campylobacter". PLOS ONE. 9 (3): e92798. Bibcode:2014PLoSO...992798M. doi:10.1371/journal.pone.0092798. PMC 3968026. PMID 24676150.
- ^ Cooper, G. M. (17 June 2005). "Distribution and intensity of constraint in mammalian genomic sequence". Genome Research. 15 (7): 901–913. doi:10.1101/gr.3577405. PMC 1172034. PMID 15965027.
- ^ "Sidow Lab - GERP".
- ^ Nawar Malhis; Steven J. M. Jones; Jörg Gsponer (2019). "Improved measures for evolutionary conservation that exploit taxonomy distances". Nature Communications. 10 (1): 1556. Bibcode:2019NatCo..10.1556M. doi:10.1038/s41467-019-09583-2. PMC 6450959. PMID 30952844.
- ^ Nawar Malhis; Matthew Jacobson; Steven J. M. Jones; Jörg Gsponer (2020). "LIST-S2: Taxonomy Based Sorting of Deleterious Missense Mutations Across Species". Nucleic Acids Research. 48 (W1): W154–W161. doi:10.1093/nar/gkaa288. PMC 7319545. PMID 32352516.
- ^ Chang KT, Guo J, di Ronza A, Sardiello M (January 2018). "Aminode: Identification of Evolutionary Constraints in the Human Proteome". Sci. Rep. 8 (1): 1357. Bibcode:2018NatSR...8.1357C. doi:10.1038/s41598-018-19744-w. PMC 5778061. PMID 29358731.
- ^ Pollard, K. S.; Hubisz, M. J.; Rosenbloom, K. R.; Siepel, A. (26 October 2009). "Detection of nonneutral substitution rates on mammalian phylogenies". Genome Research. 20 (1): 110–121. doi:10.1101/gr.097857.109. PMC 2798823. PMID 19858363.
- ^ "PHAST: Home".
- ^ Fan, Xiaodan; Zhu, Jun; Schadt, Eric E; Liu, Jun S (2007). "Statistical power of phylo-HMM for evolutionarily conserved element detection". BMC Bioinformatics. 8 (1): 374. doi:10.1186/1471-2105-8-374. PMC 2194792. PMID 17919331.
- ^ Bejerano, G. (28 May 2004). "Ultraconserved Elements in the Human Genome". Science. 304 (5675): 1321–1325. Bibcode:2004Sci...304.1321B. CiteSeerX 10.1.1.380.9305. doi:10.1126/science.1098119. PMID 15131266. S2CID 2790337.
- ^ Siepel, A. (1 August 2005). "Evolutionarily conserved elements in vertebrate, insect, worm, and yeast genomes". Genome Research. 15 (8): 1034–1050. doi:10.1101/gr.3715005. PMC 1182216. PMID 16024819.
- ^ Harmston, N.; Baresic, A.; Lenhard, B. (11 November 2013). "The mystery of extreme non-coding conservation". Philosophical Transactions of the Royal Society B: Biological Sciences. 368 (1632): 20130021. doi:10.1098/rstb.2013.0021. PMC 3826495. PMID 24218634.
- ^ Faircloth, B. C.; McCormack, J. E.; Crawford, N. G.; Harvey, M. G.; Brumfield, R. T.; Glenn, T. C. (9 January 2012). "Ultraconserved Elements Anchor Thousands of Genetic Markers Spanning Multiple Evolutionary Timescales". Systematic Biology. 61 (5): 717–726. doi:10.1093/sysbio/sys004. PMID 22232343.
- ^ Faircloth, Brant C.; Branstetter, Michael G.; White, Noor D.; Brady, Seán G. (May 2015). "Target enrichment of ultraconserved elements from arthropods provides a genomic perspective on relationships among Hymenoptera". Molecular Ecology Resources. 15 (3): 489–501. doi:10.1111/1755-0998.12328. PMC 4407909. PMID 25207863.
- ^ Reneker, J.; Lyons, E.; Conant, G. C.; Pires, J. C.; Freeling, M.; Shyu, C.-R.; Korkin, D. (10 April 2012). "Long identical multispecies elements in plant and animal genomes". Proceedings of the National Academy of Sciences. 109 (19): E1183–E1191. doi:10.1073/pnas.1121356109. PMC 3358895. PMID 22496592.
- ^ Isenbarger, Thomas A.; Carr, Christopher E.; Johnson, Sarah Stewart; Finney, Michael; Church, George M.; Gilbert, Walter; Zuber, Maria T.; Ruvkun, Gary (14 October 2008). "The Most Conserved Genome Segments for Life Detection on Earth and Other Planets". Origins of Life and Evolution of Biospheres. 38 (6): 517–533. Bibcode:2008OLEB...38..517I. doi:10.1007/s11084-008-9148-z. PMID 18853276. S2CID 15707806.
- ^ Harris, J. K. (12 February 2003). "The Genetic Core of the Universal Ancestor". Genome Research. 13 (3): 407–412. doi:10.1101/gr.652803. PMC 430263. PMID 12618371.
- ^ Ban, Nenad; Beckmann, Roland; Cate, Jamie HD; Dinman, Jonathan D; Dragon, François; Ellis, Steven R; Lafontaine, Denis LJ; Lindahl, Lasse; Liljas, Anders; Lipton, Jeffrey M; McAlear, Michael A; Moore, Peter B; Noller, Harry F; Ortega, Joaquin; Panse, Vikram Govind; Ramakrishnan, V; Spahn, Christian MT; Steitz, Thomas A; Tchorzewski, Marek; Tollervey, David; Warren, Alan J; Williamson, James R; Wilson, Daniel; Yonath, Ada; Yusupov, Marat (February 2014). "A new system for naming ribosomal proteins". Current Opinion in Structural Biology. 24: 165–169. doi:10.1016/j.sbi.2014.01.002. PMC 4358319. PMID 24524803.
- ^ Gadagkar, Sudhindra R.; Rosenberg, Michael S.; Kumar, Sudhir (15 January 2005). "Inferring species phylogenies from multiple genes: Concatenated sequence tree versus consensus gene tree". Journal of Experimental Zoology Part B: Molecular and Developmental Evolution. 304B (1): 64–74. doi:10.1002/jez.b.21026. PMID 15593277.
- ^ Ludwig, W; Schleifer, KH (October 1994). "Bacterial phylogeny based on 16S and 23S rRNA sequence analysis". FEMS Microbiology Reviews. 15 (2–3): 155–73. doi:10.1111/j.1574-6976.1994.tb00132.x. PMID 7524576.
- ^ Hug, Laura A.; Baker, Brett J.; Anantharaman, Karthik; Brown, Christopher T.; Probst, Alexander J.; Castelle, Cindy J.; Butterfield, Cristina N.; Hernsdorf, Alex W.; Amano, Yuki; Ise, Kotaro; Suzuki, Yohey; Dudek, Natasha; Relman, David A.; Finstad, Kari M.; Amundson, Ronald; Thomas, Brian C.; Banfield, Jillian F. (11 April 2016). "A new view of the tree of life". Nature Microbiology. 1 (5): 16048. doi:10.1038/nmicrobiol.2016.48. PMID 27572647.
- ^ Zhang, Liqing; Li, Wen-Hsiung (February 2004). "Mammalian Housekeeping Genes Evolve More Slowly than Tissue-Specific Genes". Molecular Biology and Evolution. 21 (2): 236–239. doi:10.1093/molbev/msh010. PMID 14595094.
- ^ Clermont, O.; Bonacorsi, S.; Bingen, E. (1 October 2000). "Rapid and Simple Determination of the Escherichia coli Phylogenetic Group". Applied and Environmental Microbiology. 66 (10): 4555–4558. Bibcode:2000ApEnM..66.4555C. doi:10.1128/AEM.66.10.4555-4558.2000. PMC 92342. PMID 11010916.
- ^ Kullberg, Morgan; Nilsson, Maria A.; Arnason, Ulfur; Harley, Eric H.; Janke, Axel (August 2006). "Housekeeping Genes for Phylogenetic Analysis of Eutherian Relationships". Molecular Biology and Evolution. 23 (8): 1493–1503. doi:10.1093/molbev/msl027. PMID 16751257.
- ^ Schoch, C. L.; Seifert, K. A.; Huhndorf, S.; Robert, V.; Spouge, J. L.; Levesque, C. A.; Chen, W.; Bolchacova, E.; Voigt, K.; Crous, P. W.; Miller, A. N.; Wingfield, M. J.; Aime, M. C.; An, K.-D.; Bai, F.-Y.; Barreto, R. W.; Begerow, D.; Bergeron, M.-J.; Blackwell, M.; Boekhout, T.; Bogale, M.; Boonyuen, N.; Burgaz, A. R.; Buyck, B.; Cai, L.; Cai, Q.; Cardinali, G.; Chaverri, P.; Coppins, B. J.; Crespo, A.; Cubas, P.; Cummings, C.; Damm, U.; de Beer, Z. W.; de Hoog, G. S.; Del-Prado, R.; Dentinger, B.; Dieguez-Uribeondo, J.; Divakar, P. K.; Douglas, B.; Duenas, M.; Duong, T. A.; Eberhardt, U.; Edwards, J. E.; Elshahed, M. S.; Fliegerova, K.; Furtado, M.; Garcia, M. A.; Ge, Z.-W.; Griffith, G. W.; Griffiths, K.; Groenewald, J. Z.; Groenewald, M.; Grube, M.; Gryzenhout, M.; Guo, L.-D.; Hagen, F.; Hambleton, S.; Hamelin, R. C.; Hansen, K.; Harrold, P.; Heller, G.; Herrera, C.; Hirayama, K.; Hirooka, Y.; Ho, H.-M.; Hoffmann, K.; Hofstetter, V.; Hognabba, F.; Hollingsworth, P. M.; Hong, S.-B.; Hosaka, K.; Houbraken, J.; Hughes, K.; Huhtinen, S.; Hyde, K. D.; James, T.; Johnson, E. M.; Johnson, J. E.; Johnston, P. R.; Jones, E. B. G.; Kelly, L. J.; Kirk, P. M.; Knapp, D. G.; Koljalg, U.; Kovacs, G. M.; Kurtzman, C. P.; Landvik, S.; Leavitt, S. D.; Liggenstoffer, A. S.; Liimatainen, K.; Lombard, L.; Luangsa-ard, J. J.; Lumbsch, H. T.; Maganti, H.; Maharachchikumbura, S. S. N.; Martin, M. P.; May, T. W.; McTaggart, A. R.; Methven, A. S.; Meyer, W.; Moncalvo, J.-M.; Mongkolsamrit, S.; Nagy, L. G.; Nilsson, R. H.; Niskanen, T.; Nyilasi, I.; Okada, G.; Okane, I.; Olariaga, I.; Otte, J.; Papp, T.; Park, D.; Petkovits, T.; Pino-Bodas, R.; Quaedvlieg, W.; Raja, H. A.; Redecker, D.; Rintoul, T. L.; Ruibal, C.; Sarmiento-Ramirez, J. M.; Schmitt, I.; Schussler, A.; Shearer, C.; Sotome, K.; Stefani, F. O. P.; Stenroos, S.; Stielow, B.; Stockinger, H.; Suetrong, S.; Suh, S.-O.; Sung, G.-H.; Suzuki, M.; Tanaka, K.; Tedersoo, L.; Telleria, M. T.; Tretter, E.; Untereiner, W. A.; Urbina, H.; Vagvolgyi, C.; Vialle, A.; Vu, T. D.; Walther, G.; Wang, Q.-M.; Wang, Y.; Weir, B. S.; Weiss, M.; White, M. M.; Xu, J.; Yahr, R.; Yang, Z. L.; Yurkov, A.; Zamora, J.-C.; Zhang, N.; Zhuang, W.-Y.; Schindel, D. (27 March 2012). "Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi". Proceedings of the National Academy of Sciences. 109 (16): 6241–6246. doi:10.1073/pnas.1117018109. PMC 3341068. PMID 22454494.
- ^ Man, S. M.; Kaakoush, N. O.; Octavia, S.; Mitchell, H. (26 March 2010). "The Internal Transcribed Spacer Region, a New Tool for Use in Species Differentiation and Delineation of Systematic Relationships within the Campylobacter Genus". Applied and Environmental Microbiology. 76 (10): 3071–3081. Bibcode:2010ApEnM..76.3071M. doi:10.1128/AEM.02551-09. PMC 2869123. PMID 20348308.
- ^ Ranjard, L.; Poly, F.; Lata, J.-C.; Mougel, C.; Thioulouse, J.; Nazaret, S. (1 October 2001). "Characterization of Bacterial and Fungal Soil Communities by Automated Ribosomal Intergenic Spacer Analysis Fingerprints: Biological and Methodological Variability". Applied and Environmental Microbiology. 67 (10): 4479–4487. Bibcode:2001ApEnM..67.4479R. doi:10.1128/AEM.67.10.4479-4487.2001. PMC 93193. PMID 11571146.
- ^ Bidet, Philippe; Barbut, Frédéric; Lalande, Valérie; Burghoffer, Béatrice; Petit, Jean-Claude (June 1999). "Development of a new PCR-ribotyping method for based on ribosomal RNA gene sequencing". FEMS Microbiology Letters. 175 (2): 261–266. doi:10.1111/j.1574-6968.1999.tb13629.x. PMID 10386377.
- ^ Ala, Ugo; Piro, Rosario Michael; Grassi, Elena; Damasco, Christian; Silengo, Lorenzo; Oti, Martin; Provero, Paolo; Di Cunto, Ferdinando; Tucker-Kellogg, Greg (28 March 2008). "Prediction of Human Disease Genes by Human-Mouse Conserved Coexpression Analysis". PLOS Computational Biology. 4 (3): e1000043. Bibcode:2008PLSCB...4E0043A. doi:10.1371/journal.pcbi.1000043. PMC 2268251. PMID 18369433.
- ^ Pandey, U. B.; Nichols, C. D. (17 March 2011). "Human Disease Models in Drosophila melanogaster and the Role of the Fly in Therapeutic Drug Discovery". Pharmacological Reviews. 63 (2): 411–436. doi:10.1124/pr.110.003293. PMC 3082451. PMID 21415126.
- ^ Huang, Hui; Winter, Eitan E; Wang, Huajun; Weinstock, Keith G; Xing, Heming; Goodstadt, Leo; Stenson, Peter D; Cooper, David N; Smith, Douglas; Albà, M Mar; Ponting, Chris P; Fechtel, Kim (2004). "Evolutionary conservation and selection of human disease gene orthologs in the rat and mouse genomes". Genome Biology. 5 (7): R47. doi:10.1186/gb-2004-5-7-r47. PMC 463309. PMID 15239832.
- ^ Ge, Dongliang; Fellay, Jacques; Thompson, Alexander J.; Simon, Jason S.; Shianna, Kevin V.; Urban, Thomas J.; Heinzen, Erin L.; Qiu, Ping; Bertelsen, Arthur H.; Muir, Andrew J.; Sulkowski, Mark; McHutchison, John G.; Goldstein, David B. (16 August 2009). "Genetic variation in IL28B predicts hepatitis C treatment-induced viral clearance". Nature. 461 (7262): 399–401. Bibcode:2009Natur.461..399G. doi:10.1038/nature08309. PMID 19684573. S2CID 1707096.
- ^ Bertram, L. (2009). "Genome-wide association studies in Alzheimer's disease". Human Molecular Genetics. 18 (R2): R137–R145. doi:10.1093/hmg/ddp406. PMC 2758713. PMID 19808789.
- ^ Kellis, Manolis; Patterson, Nick; Endrizzi, Matthew; Birren, Bruce; Lander, Eric S. (15 May 2003). "Sequencing and comparison of yeast species to identify genes and regulatory elements". Nature. 423 (6937): 241–254. Bibcode:2003Natur.423..241K. doi:10.1038/nature01644. PMID 12748633. S2CID 1530261.
- ^ Marchler-Bauer, A.; Lu, S.; Anderson, J. B.; Chitsaz, F.; Derbyshire, M. K.; DeWeese-Scott, C.; Fong, J. H.; Geer, L. Y.; Geer, R. C.; Gonzales, N. R.; Gwadz, M.; Hurwitz, D. I.; Jackson, J. D.; Ke, Z.; Lanczycki, C. J.; Lu, F.; Marchler, G. H.; Mullokandov, M.; Omelchenko, M. V.; Robertson, C. L.; Song, J. S.; Thanki, N.; Yamashita, R. A.; Zhang, D.; Zhang, N.; Zheng, C.; Bryant, S. H. (24 November 2010). "CDD: a Conserved Domain Database for the functional annotation of proteins". Nucleic Acids Research. 39 (Database): D225–D229. doi:10.1093/nar/gkq1189. PMC 3013737. PMID 21109532.