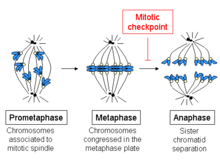
스핀들 체크포인트
Spindle checkpoint스핀들 체크포인트(SPINDLE-to-Anapase transition), 스핀들 조립체 체크포인트(SAC), 은유체 체크포인트, 또는 유사체 체크포인트라고도 하는 스핀들 체크포인트는 각 염색체가 스핀들에 적절하게 부착될 때까지 중복된 염색체(아나파제)의 분리를 막는 세포 주기 체크포인트다.적절한 분리를 위해 자매 크로마티드의 두 키네토코어를 반대편 스핀들 폴(양극 방향)에 부착해야 한다.[1]오직 이러한 부착 패턴만이 각 딸 세포가 염색체의 복사본을 받을 수 있도록 보장할 것이다.이 검문소의 생화학적 특징을 정의하는 것은 M상 사이클린-CDK 콤플렉스에 의한 아나파제 촉진 콤플렉스의 자극으로, 이는 차례로 사이클린과 자매 크로마티드를 함께 잡아주는 단백질의 프로테롤 파괴를 일으킨다.[2]
개요 및 중요도
은유효소의 시작은 미세관절이 염색체의 키네토코르에 연결되어 있을 뿐만 아니라 세포 중간에 있는 염색체들이 정렬되어 있는 것이 특징이다.각 크로마티드는 고유의 키네토코르를 가지고 있으며, 자매 크로마티드의 키네토코르에 묶인 모든 미세관들은 세포의 반대편 극지방에서 방사된다.이 미세관들은 세포의 반대쪽 끝을 향해 염색체에 당김력을 발휘하는 반면, 염색체 자매간의 응집력은 이 힘에 반대한다.
아나파아제 전환에 대한 은유효소에서는 자매 크로마티드의 이 응집력이 용해되고, 분리된 크로마티드는 스핀들 마이크로튜브에 의해 셀의 반대편으로 당겨진다.크로마티드는 스핀들 폴 자체의 물리적 움직임에 의해 더욱 분리된다.크로마티드의 조기 분리는 딸 세포에서 염색체 분리와 무극성으로 이어질 수 있다.따라서 스핀들 체크포인트의 일은 염색체가 제대로 부착될 때까지, 자매 크로마티드가 분리되기 전까지 이러한 아나파제로의 전환을 막는 것이다.
세포의 정체성과 적절한 기능을 보존하기 위해서는 각 세포분열 후 적절한 수의 염색체를 유지할 필요가 있다.예상보다 적은 수의 염색체를 가진 딸 세포를 생성하는 오류(아뉴플로이드라고 불리는 상황), 최상의 경우 세포 사망으로 이어질 수 있으며, 또는 대안으로 치명적인 표현형 세포를 생성할 수 있다.[3][4]예를 들면 다음과 같다.
- 암세포에서 양극화는 빈번한 현상이며, 이러한 세포들이 분리가 올바르게 수행되도록 보장하는 메커니즘뿐만 아니라 염색체 분리에 관련된 기계에 결함을 나타낸다.
- 인간에서 다운증후군은 세포에 염색체 분리의 결함으로 인해 세포에 21번 염색체를 한 장 더 넣고 다니는 아이들에게 나타난다.이 결함은 21번 염색체를 추가로 가진 생식세포(Spermatozoide 또는 난모세포)를 생성한다.수정 후에, 이 생식세포는 21번 염색체의 세 개의 복사본을 가진 배아를 생성하게 될 것이다.
SAC(Spindle Assembly Checkpoint) 검색

지르켈레(1970년)는 한 염색체만 지연되어 은유체 판에 도달하면 아나파제 발병이 도착 후 몇 분 내로 연기된다는 것을 관찰한 최초의 연구자 중 한 명이었다.[5]이러한 관찰은 유사한 관찰과 함께 은유-대-아나파제 전환 시 제어 메커니즘이 존재함을 시사했다.노코다졸, 콜치약 등의 약물을 사용하면 유사분열에서 아나파제로의 전환에서 유사분열 스핀들 분해 및 세포주기가 차단된다.이러한 약물을 사용하여(1992년[6] Reder 및 Palazzo의 검토 참조) 투입 제어 메커니즘을 SAC(Spindle Assembly Checkpoint)라고 명명했다.이 규제 메커니즘은 그 이후로 집중적으로 연구되어 왔다.[7]
유전학은 다른 종류의를 사용하는 결함을 다양한 종류의 예술의 전당은 되살리는 것이 가능:스핀들 이동 원체 염색체(두 centromeres과)[10]centromeres 스핀들 장대에 몸 속에 있는 kineto에 cerevisiae,[12]결함의 도리를 벗어난 way,[11]결함이 분리의 존재 depolymerization,[8][9]이 확립되어 졌다.잡일 proteins,[13]뮤체세포 DNA의[14] 침전 또는 유사시 활성 분자 모터의 결함.[8]이러한 관찰의 요약은 1999년 하드윅과 협력자들의 글에서 찾을 수 있다.[15]
자체 관측 결과를 사용해, Zirkle[5]은"일부(…)물질의 세포 anaphase로 진행하기 위해서 필요한, C(마지막 염색체의 중기판 도착의 순간)몇분, 또는 세포질 상태에 급격한 변화 후에 C또는 C직후에 나타난다"를 제안하도록 이 쿵푸에 시사하는 처음이었다.nct이온은 미토틱 스핀들에 부착되지 않은 키네토코어에 위치한다.맥인토쉬는 이 제안을 확장하여, 두 자매 키네토코르스가 양극성 장력을 받지 않을 때 중앙부에 위치한 장력에 민감한 효소 하나가 아나파제 발병에 대한 억제제를 생성한다고 제안했다.[16]실제로 이용 가능한 데이터는 "아나파제로 들어가기 위해 기다림" 신호가 대부분 부착되지 않은 키네토코르에서 생성되거나 근처에서 생성된다는 것을 시사했다.[17]미소관의 동원체(로 리더와 협력자들에 의해 1995[17]에 제안한)그러나 1차 행사는 스핀들로 중기 정지가 억제성 신호 비활성화시킬 수 있는 동원체 부착물과 관련된, 있을 획득, 또는 장력으로 미소관의 기점화 안정화. 그키네토코레스(Nicklas의 연구실에서[18] 실현된 실험에서 제안된 바와 같이).단 하나의 세포질 내에 두 개의 독립된 유사분열 스핀들을 포함하는 세포에 대한 후속 연구는 은유-대-아나파제 전환의 억제제가 미첨가 키네토코르에 의해 생성되며 세포질에서 자유롭게 확산되지 않는다는 것을 보여주었다.[19]그러나 동일한 연구에서 세포의 한 부분에서 은유효소로부터 아나파제로의 전환이 시작되면 이 정보는 세포질 전체를 따라 확장되며, 연결되지 않은 키네토코르를 포함하는 두 번째 스핀들과 연관된 "아나파아제로의 진입 대기" 신호를 극복할 수 있다는 것이 밝혀졌다.
자매 염색분체 복제, 응집 및 분리의 배경
세포분열 : 물질복제 및 딸세포로의 분포
세포가 분열할 준비가 되었을 때 세포 크기가 충분히 크거나 적절한 자극을 받기 때문에 세포 주기에 진입하는 메커니즘을 활성화하고, 세포 중심체를 포함한 S(합성) 단계 동안 대부분의 유기체를 복제한다.[20]따라서 세포분열 과정이 종료되면 각 딸세포는 완전한 세포세트를 받게 된다.동시에, S단계 동안 모든 세포는 DNA 복제라고 불리는 과정을 매우 정확하게 복제해야 한다.일단 DNA 복제가 완료되면 진핵생물에서는 DNA 분자가 압축되고 응축되어 유사 염색체를 형성하는데, 각각은 그들 사이의 응집력의 확립에 의해 함께 유지되는 두 자매 크로마티드에 의해 구성된다; 각각의 크로마티드는 마이크로튜브를 통해 2 센트로놈 중 하나에 부착된 완전한 DNA 분자다.그는 세포의 반대편 극에 위치한 세포 분리를 한다.센트로놈과 마이크로튜브에 의해 형성된 구조는 두 개의 센트로놈 사이에 염색체를 고정시키는 특징적인 형태 때문에 미토콘 스핀들이라고 불린다.두 자매 크로마티드는 아나파제가 될 때까지 함께 있다; 이 순간 그들은 서로 분리되어 그들이 붙어 있는 중심부를 향해 이동한다.이렇게 해서 분할 과정이 끝날 때 두 딸세포가 분리되면 각각의 딸세포는 완전한 염색체 세트를 받게 된다.세포분열 중 자매 크로마티드의 정확한 분포를 담당하는 메커니즘은 염색체 분리라고 불린다.
염색체 분리가 올바르게 이루어지도록 하기 위해, 세포는 정확하고 복잡한 메커니즘을 개발했다.우선 세포는 중심 복제를 DNA 복제와 함께 조정해야 하며, 이 조정에서 실패하면 독과점이나 다극성 유사분열 스핀들을 생성하게 되는데, 이는 일반적으로 비정상적인 염색체 분리를 생성하게 되는데,[21] 이 경우 염색체 분배가 균형 있게 이루어지지 않기 때문이다.
유사분열: 스핀들에 염색체 고정 및 염색체 분리
S 페이즈 동안 센트로솜은 복제되기 시작한다.유사분열 초기 두 중심부는 최대 길이를 달성하고 추가 물질을 모집하며 미세관 핵화 능력이 증가한다.유사분열이 진행되면 두 센트로놈은 분리되어 유사분열 스핀들을 생성한다.[22]이렇게 해서 유사분열 스핀들에는 미세관(microtubule)을 내뿜는 두 개의 극이 있다.마이크로튜브(MTs)는 세포의 중심을 탐색하며 염색체를 탐색하는 세포의 중심을 탐구하며 비대칭적인 극단성을 가진 긴 단백질 필라멘트로, 한쪽 끝은 "마이너스(-) 끝"이라고 하며 비교적 안정적이고 센트로솜에 가까운 끝, 끝은 "플러스"라고 한다.각 크로마티드에는 센트롬이라는 특수한 영역이 있으며, 그 위에 키네토코레라고 불리는 단백질 구조가 조립되어 미세관 플러스 엔드를 안정시킬 수 있다.따라서 우연히 세포의 중심을 탐사하는 미세 관이 키네토코레와 마주치면 키네토코레가 이를 포착해 염색체가 자매 크로마티드의 키네토코레(kinetochore)를 통해 스핀들에 부착될 수도 있다.염색체는 스핀들에 키네토코르를 부착하는 데 적극적인 역할을 한다.염색체에 묶인 것은 염색체 근처의 세포질 란(Ran guanine nucleotide actor, GEF)을 자극하여 GDP 대신 GTP를 묶게 하는 Ran guanine nucleotide actor (GEF)활성화된 GTP 결합 형태의 란(Ran)은 사이토솔 내 단백질 복합체로부터 TPX2와 같은 미세관 안정화 단백질을 방출하여 염색체 주위의 미세관 핵화와 중합화를 유도한다.[23]이러한 키네토코어 유래 마이크로튜브는 외부 키네토코어 내 키네신 모터 단백질과 함께 스핀들 폴 유래 마이크로튜브의 측면 표면과의 상호작용을 용이하게 한다.그러나 이러한 측면 부착물은 불안정하므로 종단 부착물로 변환해야 한다.측면 부착에서 엔드온 부착으로 변환하면 미세관 플러스 엔드의 성장과 수축이 염색체를 밀고 당기는 힘으로 전환되어 적절한 생체지향을 달성할 수 있다.공교롭게도 자매 크로마토르(chromatids)가 함께 붙어 있고 양쪽 키네토코르(kinetochores)가 양쪽 크로마토르(chromatids)에 백 투 백으로 위치하게 되면, 하나의 키네토르(kinetochore)가 반대편극에 위치한 센트로놈에 노출되게 된다. 이러한 이유로 대부분의 경우 제2의 키네토르(kinetochore)는 서로 연관되게 된다.반대 극의 센트로솜은 마이크로튜브를 통해 염색체가 "비 지향적"이 되도록 하는데,[24] 이는 세포가 분열할 때 염색체 분리가 올바르게 이루어지도록 하는 근본적인 구성(암피텔릭이라고도 함)이다.[25][26]때때로, 두 자매 키네토코르 중 한 마리가 양쪽 극에서 생성된 MT에 동시에 부착될 수 있는데, 메로텔릭이라는 구성은 스핀들 체크포인트에 의해 검출되지 않지만, 아나페아제 동안 뒤떨어지는 염색체를 생성하여 결과적으로 무우플로이드를 발생시킬 수 있다.메로텔릭 방향(키네토코르 자매 사이의 긴장 부재로 특징지어짐)은 유사 초기에 빈번하게 발생하지만, 단백질 오로라 B(효모에서 척추동물로 보존된 키나아제)는 이러한 유형의 닻을 검출하여 제거한다.([27]주:오로라 B는 다양한 종류의 종양에서 자주 과다압박되며 현재 항암제 개발의 대상이다.)[28]
체세포 분열 중 염색질 화합물
응집력: SMC 단백질
앞서 언급한 바와 같이, 자매 크로마티드는 S상(DNA가 복제되어 두 개의 동일한 복사본, 두 개의 크로마티드를 생성하는 경우)에서 아나파제까지 연관성을 유지한다.이때 두 자매 크로마티드가 분리되어 분열 세포의 반대 극지방으로 이동한다.효모와 제노푸스 레비스의 달걀 추출물에 대한 유전적, 생화학 연구에서는 다단백질 복합체가 자매 크로마티스 응집작용의 필수적 주체로 확인되었다(2000년[29] 히라노의 리뷰 참조).이 콤플렉스는 응집성 복합체로 알려져 있으며, 사카로마이오스 세레비시아에는 적어도 4개의 하위 유니트인 Smc1p, Smc3p, Scc1p(또는 Mcd1p), Scc3p로 구성되어 있다.smc1p와 smc3p는 모두 염색체 ATPas의 그룹을 구성하고 이질화체(Smc1p/Smc3p)를 형성하는 염색체 구조 유지(Smc)용 단백질군에 속한다.Scc1p는 Rad21의 S.cerevisiae에 있는 호몰로그로, S. pombe에서 DNA 수리에 관여하는 단백질로 처음 확인되었다.이 네 가지 단백질은 효모에 필수적이며, 그 중 어느 하나라도 돌연변이가 일어나면 조산 자매 크로마티드가 분리될 것이다.효모에서 응집력은 염색체 팔을 따라 선호 부위에 결합하며, 염색체 면역복귀를 이용한 연구에서 보여지듯이 중심부에 매우 풍부하다.[30]
헤테로크로마틴의 역할
고전적인 세포질 관찰은 자매 염색체가 이염색체 영역에 더 강하게 부착되어 있음을 시사했고,[31] 이는 이염색체의 특수한 구조나 구성이 응집력 모집을 선호할 수 있음을 시사했다.[32]실제로 Swi6(S. pombe에서 HP-1의 호몰로어)는 히스톤 H3의 메틸화 리스 9에 결합하고 S. pombe에서 센트롬 반복에 응집력의 결합을 촉진하는 것으로 나타났다.[33][34]보다 최근의 연구는 RNAi 기계가 이질 색소 침착을 조절한다는 것을 보여주는데, 이는 결국[35] S. 폼베와 척추동물 세포 모두에서 이 지역에 응집력을 모집한다.[36]단, S. 세레비시아에는 센트롬 옆에 이질색화합물이 없기 때문에, 센트롬에서 강화응집력을 보장하기 위해 헤테로크롬화 이외의 다른 메커니즘이 있어야 하지만, 기능적인 중심화의 존재는 20-50kb에 이르는 인접 지역에서 응집력 연관성의 증가를 유도한다.[37]
이 방향에서, Orc2(한개의 단백질은 원산지 인정 단지, ORC, DNA복제를 시작한 S단계 동안에 연루되어에 포함되)또한 인간 세포에서 유사 분열 동안 kinetochores에;[38]이 지역화에 합의하여 경험이 Orc2 효모 세포 안에서 언니 염색 분체 응집력에 연루되고 있다고 지적한다 위치해 있다.아무도 sMoval은 SAC 활성화를 유도한다.[39]ORC 복합체의 다른 구성요소(S. pompbe의 orc5 등)가 응집작용에 관여한다는 사실도 관찰되었다.[40]그러나 ORC 단백질을 포함하는 분자경로는 응집체의 경로에 첨가된 것으로 보이며 대부분 알려져 있지 않다.
응집기능과 응집해체

중심 응집력은 스핀들 마이크로튜브가 극을 향해 발휘하는 힘에 저항하며, 이는 자매 키네토코르 사이에서 긴장을 발생시킨다.결과적으로, 이러한 장력은 단백질인 오로라 B를 포함하는 메커니즘을 통해 부착 미세관-키네토코어를 안정화시킨다(이 문제에 대한 검토: Hauf와 와타나베 2004[41]).
실제로 응집체 세포수치의 감소는 자매 크로마티드의 조기 분리와 더불어 오로라 B 단백질을 함유하고 있는 염색체 여객복합체 내 염색체 결합의 결함과 단백질의 탈초점화를 일으킨다.[42][43]응집 복합체를 위한 제안된 구조는 이 콤플렉스가 두 자매 크로마티드를 직접 연결한다는 것을 시사한다.[44]이 제안된 구조에서 SMC 결합의 SMC 구성요소는 구조적인 역할을 하므로 SMC 이질결합기는 ATP에 의해 순응이 규제되는 DNA 결합 단백질로 기능할 수 있다.[45]그러나 Scc1p와 Scc3p는 규제적 역할을 할 것이다.[29]
S. 세레비시아에서 Pds1p(일명 증권)는 프로테아제 Esp1p(분리 또는 분리효소)를 결합하고 억제하기 때문에 자매 크로마티스 응집력을 조절한다.아나파제 발생이 트리거되면 아나파제 촉진 복합체(APC/C 또는 사이클로솜)는 증권화를 저하시킨다.APC/C는 유비퀴틴이 탑재된 E2유비퀴틴 콘쥬깅 효소를 채용하는 링 E3유비퀴틴 리기아제다.Securin은 활성제 서브유닛인 Cdc20이 APC/C 코어에 바인딩된 경우에만 인정된다.증권화, Cdc20, E2가 모두 APC/C E2 유비쿼터스화에 묶여 있을 때 증권화를 선택적으로 떨어뜨린다.Securin 분해효소는 프로테아제 Esp1p/분리효소를 방출하여 두 자매 크로마티드를 연결하는 응집력 고리를 분해하여 자매 크로마티드의 분리를 촉진한다.[46]또한 Scc1 절삭부 옆에 Polo/Cdc5 kinase phosphorylates serine 잔류물도 확인되었으며, 이러한 인산화 작용은 절삭 활동을 용이하게 할 것이다.[47]
비록 이 기계는 진화를 통해 보존되지만 척추동물에서 대부분의 응집 분자는 APC/C의 존재와는 독립적으로 Polo 유사 1(PLK1)과 Ourora B에 의존하는 과정에서 프로 페이즈로 방출된다.[48][49][50]그러나 소량의 Scc1은 은유화되기 전까지 인간 세포의 센트롬과 연관되어 있으며, 이와 유사한 양이 센트롬에서 사라질 때 아나파아제로 절단된다.[51]한편, 몇몇 실험에서는 자매결핵이 분리되고 난 후, 자매결핵이 세포의 반대 극을 향해 움직인다는 것을 보여준다.[52][53]
일부 관측에 따르면 염색체 팔의 응집력과 중심 응집체의 일부분은 슈고신(Sgo1) 단백질에 의해 보호되어 프로 페이즈 중에 분비되는 것을 피한다.[54][55]중추 응집력의 보호자 역할을 할 수 있으려면 Sgo1은 Pds1p뿐만 아니라 아나파제 초기에 비활성화되어야 한다.실제로 Pds1p와 Sgo1은 모두 척추동물에서 APC/C의 기질이다.[56]
스핀들 어셈블리 체크포인트 개요
스핀들 조립체 체크포인트(SAC)는 키네토코어가 잘못 부착되어 생성되는 활성 신호로, 모든 진핵생물에 보존되어 있다.SAC는 CDC20을 부정적으로 규제함으로써 세포 주기를 정지시켜, 아나파제 촉진 복합체(APC)의 다극성 활동의 활성화를 방해한다.SAC신호를 담당하는 단백질은 SAC 단백질, MAD2/MAD3(Mitotic Attack complex, MCC), BUB3(Benzimidazole에 의해 억제되지 않은 버딩), CDC20을 포함한다.[57]SAC에 관여하는 다른 단백질로는 MAD1, BUB1, MPS1, 오로라 B가 있다.상위 진핵생물의 경우, SAC의 추가 규제자는 ROD-ZW10 복합체, p31comet, MAPK, CDK1-사이클로인-B, NEK2 및 PLK1의 구성요소를 포함한다.[58]
체크포인트 활성화
SAC는 부적절하게 연결된 키네토코어와 스핀들 마이크로튜브 사이의 상호작용을 모니터링하며, 키네토코어가 스핀들에 올바르게 부착될 때까지 유지된다.프로메타파아제 동안 CDC20과 SAC 단백질은 스핀들 조립체에 부착하기 전에 키네토코어에 농축된다.이러한 단백질은 제거되고 올바른 키네토코레-마이크로튜브 부착이 이루어질 때까지 SAC를 활성화시킨다.부착되지 않은 키네토코어 하나만 있어도 스핀들 체크포인트를 유지할 수 있다.[57]미세관 플러스 엔드 부착 및 키네토코어 미세관 형성 후 MAD1과 MAD2는 키네토코어 조립체에서 고갈된다.체크포인트 활성화의 또 다른 조절기는 키네토코어 장력이다.자매 키네토코어가 반대편 스핀들 폴에 적절히 부착되면 유사체 스핀들 내의 힘은 키네토코어에 장력을 발생시킨다.바이 지향적인 자매 키네토코레스는 키네토코레-마이크로튜브 어셈블리를 안정화시키는 반면, 약한 장력은 불안정한 효과를 가진다.양쪽 키네토코어가 하나의 스핀들 폴에 부착되는 싱텔릭 부착물과 같은 부정확한 키네토코어 부착물에 대응하여, 약한 장력으로 인해 잘못된 부착이 불안정해지고 키네토코어가 스핀들 본체에 올바르게 다시 부착될 수 있게 된다.이 과정에서 유사분열 스핀들에 부착되지만 장력이 걸리지 않는 키네토코어가 스핀들 체크포인트를 트리거한다.염색체 승객 복합체의 Aurora-B/Ipl1 kinase는 부적절한 키네토코어 부착물에서 장력 센서로 기능한다.마이크로튜브-키네토코어 인터페이스에서 마이크로튜브-세버링 KINI kinesin MCAK, DASH 콤플렉스, Ndc80/Hec1 콤플렉스의[59] 제어를 통해 부정확한 부착물을 검출·안정화한다.[58]또한 Aurora-B/Ipl1 kinase는 한 개의 키네토코어가 양쪽 스핀들 폴에 동시에 부착되는 메로텔릭 부착물을 교정하는데 중요하다.Merotelic 부착물은 충분한 장력을 발생시키고 SAC에 의해 감지되지 않으며, 보정하지 않으면 염색체 분리가 느린 크로마티드 이동 속도로 인해 잘못 이루어질 수 있다.SAC 활성화를 위해서는 미세관 부착이 독립적으로 필요하지만, 장력과 함께 서로 다른 규제 행위가 발생하는 것은 분명하지만, 장력이 SAC의 독립적 조절기인지 여부는 불분명하다.
스핀들 체크포인트가 활성화되면 유사체 검문소 활동규제를 통해 아나파제 추진단지를 억제하여 아나파제 진입을 차단한다.MCC가 BUBR1의 KEN-box 모티브를 사용하여 APC에 가성비로서 결합한다는 가설은 있지만, MCC에 의한 APC 억제 메커니즘은 잘 이해되지 않는다.미토틱 체크포인트 콤플렉스가 활성화되는 것과 동시에 센트로메르 단백질 CENP-E가 BUBR1을 활성화시켜, 아나파제를 차단하기도 한다.[58]
미토틱 체크포인트 복합형성
유사체 검문소는 BUB3와 함께 MAD2와 Cdc20으로 묶인 MAD3로 구성된다.MAD2와 MAD3는 CDC20에 뚜렷한 바인딩 사이트를 가지고 있으며, APC/C를 억제하기 위해 시너지 작용을 한다.MAD3 콤플렉스는 GLEBS 모티브로 알려진 짧은 선형 모티브를 통해 MAD3와 BUB1B에 바인딩되는 BUB3로 구성되어 있다.MCC를 형성하기 위해 발생해야 하는 첨부 파일의 정확한 순서는 알려지지 않았다.BUBR1-BUB3-Cdc20이 또 다른 콤플렉스를 형성하는 것과 동시에 Mad2-Cdc20이 콤플렉스를 형성할 가능성이 있으며, 결과적으로 이 두 하위 콤플렉스가 결합되어 유사체 체크포인트 콤플렉스를 형성하게 된다.[57]인간 세포에서 BUBR1과 CDC20의 결합은 MAD2와 CDC20의 사전 결합을 필요로 하므로 MAD2-CDC20 하위 복합체가 MCC 형성을 위한 개시자 역할을 할 가능성이 있다.BUBR1 고갈은 Mad2-Cdc20 수준의 경미한 감소로 이어지는 반면 BubR1-Bub3와 Cdc20의 결합에 Mad2가 필요하다.그럼에도 불구하고, BUBR1은 여전히 체크포인트 활성화를 위해 필요하다.[58]
MCC의 형성 메커니즘은 불분명하며 키네토코어 의존성 및 키네토코어 독립성 형성에 대한 경쟁 이론이 있다.키네토코어 독립 이론을 지지하여, MCC는 코어 키네토코어 조립단백질이 변이된 S. 세레비시아아 세포와 SAC가 비활성화된 세포에서 검출할 수 있는데, 이는 키네토코어 국산화 없이 유사시 MCC를 조립할 수 있음을 시사한다.하나의 모델에서, 비첨부 프로메타파제 키네토코레스는 기능하는 SAC를 통해 APC를 키네토코레로 모집함으로써 MCC의 억제를 '감지'할 수 있다.게다가, 다양한 SAC 단백질의 감퇴는 MAD2와 BUBR1 감퇴가 키네토코르와는 독립적으로 유사분열 시기에 영향을 미치는 반면, 다른 SAC 단백질의 감퇴는 유사분열 기간을 변경하지 않고 기능불능 SAC를 초래한다는 것을 밝혀냈다.따라서 SAC는 1단계에서 MAD2와 BUBR1이 유사분열 지속시간을 제어하는 2단계 타이머를 통해 기능할 수 있으며, 다른 SAC 단백질뿐만 아니라 미연결 키네토코어가 있는 경우 2단계에서도 연장될 수 있다.[58]그러나 키네토코어 독립 어셈블리에 불리한 증거가 있다.MCC는 스핀들 조립을 방지하기 위해 핵 및 nocodazole의 정자를 첨가하지 않고서는 X. laevis meiosis II 추출물의 성분으로부터 형성되지 않는 반면, MCC는 아직 상간에서 발견되지 않았다.
MCC 형성의 선두 모델은 "MAD2-템플릿 모델"로 MAD2의 키네토코어 역학에 의존하여 MCC를 생성하며, MAD1은 MAD2에 강하게 결합하면서 미부착 키네토코어로 국소화한다.키네토코어에 대한 MAD2와 BubR1의 국산화도 오로라 B 키나제에 의존할 수 있다.[60]오로라 B가 결핍된 세포들은 염색체에 미세관 부착이 부족해도 은유법으로 체포되지 않는다.[61]연결되지 않은 키네토코레스는 먼저 MAD1-C-MAD2-p31comet 콤플렉스에 바인딩하고 알 수 없는 메커니즘을 통해 p31을comet 방출한다.그 결과 MAD-C-MAD2 콤플렉스는 Mad2(O-Mad2)의 오픈 컨포머를 키네토코레스에 채용한다.이 O-Mad2는 닫힌 Mad2(C-Mad2)로 순응을 변경하여 Mad1을 묶는다.이 Mad1/C-Mad2 콤플렉스는 키네토코르에 O-Mad2를 더 많이 채용할 책임이 있는데, 이 콤플렉스는 C-Mad2에 대한 순응을 변경하고 자동증화 반응으로 Cdc20을 바인딩한다.MAD1과 CDC20 모두 유사한 MAD2 바인딩 모티브를 포함하므로, 빈 O-MAD2 순응은 CDC20에 바인딩하는 동안 C-MAD2로 변경된다.이 긍정적 피드백 루프는 p31에comet 의해 부정적으로 규제되며, p31은 MAD1 또는 CDC20에 바인딩된 C-MAD2에 경쟁적으로 바인딩되며, C-MAD2에 바인딩되는 O-MAD2를 더 감소시킨다.p31이comet 하위 eukaryotes에 존재하지 않는다는 점을 고려할 때 추가적인 제어 메커니즘이 존재할 수도 있다.따라서 '템플릿 모델' 명명법은 MAD1-C-MAD2가 C-MAD2-CDC20 복사본의 형성을 위한 템플릿 역할을 하는 프로세스에서 도출된다.이러한 Cdc20의 격리조치는 스핀들 체크포인트를 유지하는데 필수적이다.[57]
체크포인트 비활성화
자매 크로마티드의 정확한 양방향화 후 SAC를 비활성화하기 위한 몇 가지 메커니즘이 존재한다.마이크로튜브-키네토코어 부착 시, 다이네인-다이닌 모터 콤플렉스를 통한 박리 메커니즘이 스핀들 체크포인트 단백질을 키네토코어로부터 멀리 운반한다.[58]그런 다음 MAD1, MAD2, MPS1, CENP-F를 포함한 벗겨진 단백질을 스핀들 폴에 재분배한다.박리과정은 미세관을 따라 다인 운동성뿐만 아니라 손상되지 않은 미세관 구조물에 대한 의존도가 높다.P31은comet C-MAD2 양성 피드백 루프의 조절기 역할뿐만 아니라 SAC의 비활성화기 역할도 할 수 있다.부착되지 않은 키네토코르스는 p31을comet 일시적으로 비활성화하지만, 부착은 단백질을 재활성화시키고 MAD2 활성화를 억제하며, 아마도 억제 인산화 작용에 의해 억제된다.SAC 불활성화의 또 다른 가능한 메커니즘은 CDC20의 비분해적 편재성을 통한 MAD2-CDC20 복합체의 에너지 의존적 분리에서 비롯된다.반대로 SAC를 유지하기 위해서는 탈유효소 프로텍틴이 필요하다.따라서 연결되지 않은 키네토코어는 MAD2-CDC20 하위 복합체를 구성 요소에서 지속적으로 재생성하여 체크포인트를 유지한다.또한 SAC는 APC 활성화 유도 단백질 분해에 의해 비활성화될 수 있다.아나파제 중 자매-염색체 응집력 상실에 의해 SAC가 재활성화되지 않기 때문에, 사이클린 B의 프로테롤리시스와 CDK1-사이클로닌-B키나제의 불활성화 또한 SAC 활동을 억제한다.아나파제 중 MPS1의 분해는 자매-염색체 응집체 제거 후 SAC의 재활성화를 방지한다.체크포인트 비활성화 후, 그리고 셀 사이클의 정상적인 아나파제 동안, 아나파제 촉진 콤플렉스는 MCC 활동 감소를 통해 활성화된다.이렇게 되면 효소 복합체 폴리우비티트는 아나파제 억제제 증권화를 촉진한다.은유효소 끝의 유비쿼터스화와 증권화의 파괴는 분리효소라 불리는 활성 프로테아제를 방출한다.분리효소는 자매 크로마티드를 함께 지탱하는 응집 분자를 쪼개어 아나파제를 활성화시킨다.[23]
S. 세레비시아의 새 SAC 비활성화 모델: 기계 스위치
키네토코어의 엔드온 미세관 부착이 SAC 신호의 특정 단계를 방해할 수 있는 방법을 설명하는 새로운 메커니즘이 제안되었다.비첨부 키네토코르에서 MCC 형성의 첫 번째 단계는 키나제 Mps1에 의한 Spc105의 인산화다.인산염 Spc105는 그 후 하류 신호 단백질인 Bub1과 3, Mad 1,2,3과 Cdc20을 모집할 수 있다.연결되지 않은 키네토코르에서 Mad1과 연관되면 Mad2가 개방형(O-Mad2)에서 폐쇄형(C-Mad2)으로 변환하는 순응적 변화를 겪게 된다.Mad1에 묶인 C-Mad2는 두 번째 O-Mad2로 약화되고 Cdc20을 중심으로 폐쇄를 촉진한다.이 C-Mad2와 Cdc20 콤플렉스인 MCC는 Mad1과 C-Mad2를 키네토코어에 남겨두고 또 다른 MCC를 형성한다.MCC는 각각 APC/C와의 상호작용을 방지하기 위해 두 개의 Cdc20 분자를 분리하여 SAC를 유지한다.[23]Mps1의 Spc105의 인산화 작용은 SAC 신호 경로를 개시하기에 필요하면서도 충분하지만, 이 단계는 키네토코어에 미세관절 부착이 없는 경우에만 발생할 수 있다.내생성 Mps1은 Ndc80의 칼포닌-호몰로지(CH) 영역과 연관되어 있는 것으로 보여지는데, Ndc80은 염색체로부터 멀리 떨어져 있는 외부 키네토코레 영역에 위치한다.Mps1은 외부 키네토코레에 도킹되어 있지만, Ndc80의 유연한 힌지 영역 때문에 내부 키네토코레와 인산염 Spc105 내에서 국소화가 가능하다.그러나 기계식 스위치 모델은 키네토코어에 마이크로튜브를 엔드온으로 부착하면 두 가지 메커니즘을 통해 SAC가 비활성화된다는 것을 제안한다.부착된 마이크로튜브의 존재는 Ndc80 CH 영역과 Spc105 사이의 거리를 증가시킨다.또한 부착된 미세관 주위에 고리를 형성하는 160개의 단백질로 구성된 대규모 복합체 댐1/DASH는 두 단백질 사이의 장벽 역할을 한다.분리는 Mps1과 Spc105 사이의 상호작용을 방지하므로 SAC 신호 경로를 억제한다.[62]
동물을 포함한 상위 유기체에서는 이 모델이 SAC 규제에 적용되지 않는다는 점에 유의해야 한다.기계식 스위치 메커니즘의 주요 측면은 키네토코어의 구조가 하나의 미세관만 부착할 수 있다는 것이다.반면에 동물들의 키네토코르들은 다수의 미세관들을 위한 결합 부지를 포함하고 있는 훨씬 더 복잡한 메쉬웍스다.[63]모든 키네토코어 결합 부위의 미세관 부착은 SAC 비활성화 및 아나파아제 진행에 필요하지 않다.따라서 SAC가 억제되는 동안 동물 키네토코어에는 마이크로튜브 부착 및 마이크로튜브 미 부착 상태가 공존한다.이 모델에는 부착된 키네토코어와 연관된 Mps1이 인접하지 않은 키네토코어에서 Spc105를 인산화하지 못하도록 하는 장벽을 포함하지 않는다.더욱이 동물세포에는 효모댐1/DASH 복합체가 존재하지 않는다.
스핀들 체크포인트 결함 및 암
스핀들 체크포인트가 잘못 기능할 경우, 이는 염색체 분리의 오류, 무섬유화, 심지어 종양화까지 초래할 수 있다.[58]특히 전체 염색체 또는 그 중 많은 부분에서 게놈의 총체적인 수준에서 유전적 무결성의 유지보수가 분해될 때 변형이 일어나고 가속된다.사실 무연화증은 인간 고형종양의 가장 일반적인 특징이며 따라서 스핀들 조립체 검사점은 투석방지 치료의 가능한 대상으로 간주될 수 있다.[64]이는 온코겐이나 종양 억제기로 알려진 특정 유전자의 돌연변이가 주로 유전적 불안정과 종양억제제의 배후에 있다고 생각되기 때문에 잘 인식되지 않는 사실이다.보통 세포 주기의 다양한 체크포인트는 세포의 동점선을 유지하고 종양성(tomigenesis)을 예방하는 데 중요한 보존성이 높은 중복 메커니즘을 통해 게놈의 무결성을 관리한다.몇몇 스핀들 조립체 검사점 단백질은 게놈 불안정이라고도 알려진 염색체 불안정성(CIN)을 방지하는 각 세포 주기에서 적절한 염색체 분리를 보장하기 위해 양성 및 음성 조절기 역할을 한다.
유전적 무결성은 현재 일부 종양에서 기저 대체, 삽입 및 삭제로 나타나는 불안정성을 보이는 반면, 대다수는 전체 염색체의 손익을 나타내는 몇 가지 수준에서 인정받고 있다.[65]
초기에는 유사 조절 단백질의 변화가 무극성으로 이어질 수 있고 암에서 자주 발생하는 현상이라는 점 때문에 이러한 유전자가 암 조직에서 변이될 수 있다고 생각되었다.[66][67]
암의 돌연변이 유전자
일부 암에서는 변형을 초래하는 결함의 기초가 되는 유전자가 잘 특징지어진다.다발성 골수종 세포유전적 이상과 같은 혈액학적 암에서는 면역글로불린 유전자 재배치에 필요한 DNA파열의 본질적 특성 때문에 매우 흔하다.그러나 SAC에서 주로 기능하는 MAD2와 같은 단백질의 결함도 다발성 골수종에서 특징지어진다.[68]대부분의 고형종양도 주로 무극성 종양이다.대장암의 경우, BUB1과 BUBR1과 STK15의 증폭은 암을 유발하는 유전학적 불안정성에 관련되어 있는 핵심 규제 기관이다.[69]유방암에서 BRCA-1 유전자로 특징지어지는 유전적 형태는 산발적인 형태보다 유전학적 불안정성의 정도가 더 높다.실험 결과 BRCA-1 null 생쥐는 주축 체크포인트 단백질 MAD2의 발현이 감소했으며,[70] 다른 암의 경우 무연화증의 원인을 규명하기 위해 더 많은 연구가 필요하다.
암에서 전통적으로 SAC와 관련이 없는 다른 유전자
이러한 단백질(Mad2 또는 BubR1)의 생리학적 수준의 분명한 변화는 무우플로이드와 종양성(tominigenesis)[71][72]과 연관되어 있으며, 이는 동물 모델을 사용하여 증명되었다.그러나, 최근의 연구는, 발생되는 것으로 보이는 것이 더 복잡한 시나리오라는 것을 보여준다: 무우플로이디는 조직에서 특정한 유사체 검문소 성분의 수준(감소 또는 과다압박)의 변화가 종양을 유발할 수 있는 다른 결함을 유발할 때에만 높은 종양 발생을 초래할 것이다.[73]즉, DNA 손상 증가, 염색체 재배열 및/또는 세포사망 발생률 감소와 같은 결함이다.일부 유사체 검문소 구성요소의 경우, 핵 수입(Mad1), 전사 억제(Bub3) 및 세포 사망, DNA 손상 반응, 노화, BubR1용 메가카리오피시스 등 유사체외 기능에 관여하는 것으로 알려져 있다.이 모든 것은 종양이 증가한다는 것이 무신경증 이외의 결함과 연관되어 있다는 결론을 뒷받침한다.[73]
BUB1이나 BUBR1과 같이 알려진 체크포인트 유전자에 영향을 미치는 암 관련 돌연변이는 실제로 드물다.그러나 암과 관련된 몇몇 단백질은 스핀들 조립 네트워크와 교차한다.p53과 같은 주요 종양 억제기도 스핀들 체크포인트에서 역할을 한다.인간암에서 가장 흔하게 변이된 유전자 p53의 부재는 세포주기 체크포인트 규제기관에 큰 영향을 미치며 과거 G1 체크포인트에서 활동하는 것으로 나타났지만 이제는 스핀들 체크포인트를 규제하는 데도 중요한 것으로 보인다.[74]암의 또 다른 중요한 측면은 세포사멸이나 세포사멸의 억제다.세포사멸 억제제(IAP) 계열의 멤버인 리바이벌린은 센트로솜 근처에 있는 미생물 스핀들의 미세관 및 은유효소 염색체의 키네토코르에 있는 풀에 국부화된다.생존인은 종양기세증을 촉진하기 위해 세포사멸을 억제할 뿐만 아니라, 염색체 분리의 중요한 조절기로서 (실험용 녹아웃 생쥐를 통해) 관여되어 왔고, 보다 원시적인 유기체에서 그것의 역할과 유사한 말기 유사분열증이다.[75]
기타 스핀들 조립체 체크포인트의 키네토코어 부착, 마이크로튜브 기능, 자매 크로마티드 응집력 등은 결점이 발생하여 음극성을 유발할 수 있다.암세포는 스핀들 조립체 검문소를 피하여 다극성 경화증을 유발함으로써 다방향으로 분열되는 것이 관찰되었다.[76]다극성 은유효소-아나파아제 전환은 불완전한 분리효소 주기를 통해 발생하며, 암세포의 음극성을 증폭시키는 비절연 사건이 빈번하게 발생한다.
SAC 암 치료법
이 분야의 발전으로 스핀들 조립품 결함을 목표로 하는 일부 치료법의 개발이 도입되었다.vinca 종류의 알칼로이드와 taxanes 같은 나이 든 치료 목표는 예술의 전당은, 결국 그들의 죽음으로 이어지는 세포를 체포하는 미소관 역학의 붕괴를 수반하다 유사 분열방추 형성 미소관.[77]택솔과 Docetaxel은 아직 유방 암, 난소암, 그리고 epithelial로 다른 종류의 치료에 사용된다.암의그러나 이러한 치료법은 부작용과 약물에 대한 내성이 높은 경우가 많다.
SAC에 영향을 미치는 규제기관 네트워크 내의 다른 목표물들 또한 추구되고 있다; 강한 관심이 오로라 키나아제 단백질 쪽으로 이동했다.[78] 암 유전자 예술의 전당은 제3기와 후속 aneuploidy의 비정상적인 제안 및 또한 TAXOL에 대한 저항에를 재정의할 때만큼 증폭되는 인산화 효소 유전자 오로라 A행위 .[79]Excitingly, 오로라 A의 작은 분자 억제제에서 vivo 모델 추가 임상 dev에 사람 만한 좋은 타겟을 제안하는 것에 항종양 효과 보여 주었다.elopment.[80] 또한 임상적 발달에 있는 오로라 B 억제제는 미세관 부착에 비정상적인 키네토코르로 이어지며, 또한 미토틱 체크포인트를 폐지한다.[78]서바이벌린은 또한 다수의 경로에서 주요 노드 역할을 하기 때문에 임상 치료 개발의 매력적인 분자 대상이며, 그 중 하나가 스핀들 형성과 체크포인트 제어다.[81]더 나아가서는 KSP와 같은 유사 운동 단백질의 억제에 대한 고찰이 포함되었다.최근 임상시험에 들어간 이러한 억제제는 스핀들 조립체 검문소를 결합시켜 유사체포증을 유발한다.[82][3]
참조
- ^ Santaguida S, Musacchio A (September 2009). "The life and miracles of kinetochores". The EMBO Journal. 28 (17): 2511–31. doi:10.1038/emboj.2009.173. PMC 2722247. PMID 19629042.
- ^ Morgan, David Owen, 1958- (2007). The cell cycle : principles of control. London: New Science Press. ISBN 978-0-19-920610-0. OCLC 70173205.
{{cite book}}: CS1 maint : 복수이름 : 작성자 목록(링크) - ^ a b Sinha, D.; Duijf, P.H.G.; Khanna, K.K. (2019), "Mitotic slippage: an old tale with a new twist", Cell Cycle, 18 (1): 7–15, doi:10.1080/15384101.2018.1559557, PMC 6343733, PMID 30601084
- ^ Santaguida S, Amon A (August 2015). "Short- and long-term effects of chromosome mis-segregation and aneuploidy". Nature Reviews Molecular Cell Biology. 16 (8): 473–85. doi:10.1038/nrm4025. hdl:1721.1/117201. PMID 26204159. S2CID 205495880.
- ^ a b Zirkle RE (March 1970). "Ultraviolet-microbeam irradiation of newt-cell cytoplasm: spindle destruction, false anaphase, and delay of true anaphase". Radiation Research. 41 (3): 516–37. Bibcode:1970RadR...41..516Z. doi:10.2307/3572841. JSTOR 3572841. PMID 5438206.
- ^ Rieder CL, Palazzo RE (July 1992). "Colcemid and the mitotic cycle". Journal of Cell Science. 102 ( Pt 3) (3): 387–92. doi:10.1242/jcs.102.3.387. PMID 1506421.
- ^ Burke DJ, Stukenberg PT (April 2008). "Linking kinetochore-microtubule binding to the spindle checkpoint". Developmental Cell. 14 (4): 474–9. doi:10.1016/j.devcel.2008.03.015. PMC 2696048. PMID 18410725.
- ^ a b Li R, Murray AW (August 1991). "Feedback control of mitosis in budding yeast". Cell. 66 (3): 519–31. doi:10.1016/0092-8674(81)90015-5. PMID 1651172. S2CID 11306198.
- ^ Hoyt MA, Totis L, Roberts BT (August 1991). "S. cerevisiae genes required for cell cycle arrest in response to loss of microtubule function". Cell. 66 (3): 507–17. doi:10.1016/0092-8674(81)90014-3. PMID 1651171. S2CID 10832842.
- ^ Neff MW, Burke DJ (September 1992). "A delay in the Saccharomyces cerevisiae cell cycle that is induced by a dicentric chromosome and dependent upon mitotic checkpoints". Molecular and Cellular Biology. 12 (9): 3857–64. doi:10.1128/MCB.12.9.3857. PMC 360258. PMID 1324407.
- ^ Wells WA, Murray AW (April 1996). "Aberrantly segregating centromeres activate the spindle assembly checkpoint in budding yeast". The Journal of Cell Biology. 133 (1): 75–84. doi:10.1083/jcb.133.1.75. PMC 2120768. PMID 8601615.
- ^ Hardwick KG, Weiss E, Luca FC, Winey M, Murray AW (August 1996). "Activation of the budding yeast spindle assembly checkpoint without mitotic spindle disruption". Science. 273 (5277): 953–6. Bibcode:1996Sci...273..953H. doi:10.1126/science.273.5277.953. PMID 8688079. S2CID 37404757.
- ^ Wang Y, Burke DJ (December 1995). "Checkpoint genes required to delay cell division in response to nocodazole respond to impaired kinetochore function in the yeast Saccharomyces cerevisiae". Molecular and Cellular Biology. 15 (12): 6838–44. doi:10.1128/MCB.15.12.6838. PMC 230938. PMID 8524250.
- ^ Spencer F, Hieter P (October 1992). "Centromere DNA mutations induce a mitotic delay in Saccharomyces cerevisiae". Proceedings of the National Academy of Sciences of the United States of America. 89 (19): 8908–12. Bibcode:1992PNAS...89.8908S. doi:10.1073/pnas.89.19.8908. JSTOR 2360300. PMC 50033. PMID 1409584.
- ^ Hardwick KG, Li R, Mistrot C, Chen RH, Dann P, Rudner A, Murray AW (June 1999). "Lesions in many different spindle components activate the spindle checkpoint in the budding yeast Saccharomyces cerevisiae". Genetics. 152 (2): 509–18. doi:10.1093/genetics/152.2.509. PMC 1460633. PMID 10353895.
- ^ McIntosh JR (1991). "Structural and mechanical control of mitotic progression". Cold Spring Harbor Symposia on Quantitative Biology. 56: 613–9. doi:10.1101/sqb.1991.056.01.070. PMID 1819511.
- ^ a b Rieder CL, Cole RW, Khodjakov A, Sluder G (August 1995). "The checkpoint delaying anaphase in response to chromosome monoorientation is mediated by an inhibitory signal produced by unattached kinetochores". The Journal of Cell Biology. 130 (4): 941–8. doi:10.1083/jcb.130.4.941. PMC 2199954. PMID 7642709.
- ^ Li X, Nicklas RB (March 1997). "Tension-sensitive kinetochore phosphorylation and the chromosome distribution checkpoint in praying mantid spermatocytes". Journal of Cell Science. 110 ( Pt 5) (5): 537–45. doi:10.1242/jcs.110.5.537. PMID 9092936.
- ^ Rieder CL, Khodjakov A, Paliulis LV, Fortier TM, Cole RW, Sluder G (May 1997). "Mitosis in vertebrate somatic cells with two spindles: implications for the metaphase/anaphase transition checkpoint and cleavage". Proceedings of the National Academy of Sciences of the United States of America. 94 (10): 5107–12. Bibcode:1997PNAS...94.5107R. doi:10.1073/pnas.94.10.5107. PMC 24639. PMID 9144198.
- ^ Conlon I, Raff M (January 1999). "Size control in animal development". Cell. 96 (2): 235–44. doi:10.1016/S0092-8674(00)80563-2. PMID 9988218.
- ^ Meraldi P, Lukas J, Fry AM, Bartek J, Nigg EA (June 1999). "Centrosome duplication in mammalian somatic cells requires E2F and Cdk2-cyclin A". Nature Cell Biology. 1 (2): 88–93. doi:10.1038/10054. PMID 10559879. S2CID 24795991.
- ^ Mayor T, Meraldi P, Stierhof YD, Nigg EA, Fry AM (June 1999). "Protein kinases in control of the centrosome cycle". FEBS Letters. 452 (1–2): 92–5. doi:10.1016/S0014-5793(99)00534-7. PMID 10376685. S2CID 22671038.
- ^ a b c Morgan, David O. (2006-09-06). The Cell Cycle: Principles of Control (Primers in Biology) (1 ed.). New Science Press, Ltd. ISBN 978-0-87893-508-6.
- ^ Nicklas RB (January 1997). "How cells get the right chromosomes". Science. 275 (5300): 632–7. doi:10.1126/science.275.5300.632. PMID 9005842. S2CID 30090031.
- ^ Loncarek J, Kisurina-Evgenieva O, Vinogradova T, Hergert P, La Terra S, Kapoor TM, Khodjakov A (November 2007). "The centromere geometry essential for keeping mitosis error free is controlled by spindle forces". Nature. 450 (7170): 745–9. Bibcode:2007Natur.450..745L. doi:10.1038/nature06344. PMC 2586812. PMID 18046416.
- ^ Dewar H, Tanaka K, Nasmyth K, Tanaka TU (March 2004). "Tension between two kinetochores suffices for their bi-orientation on the mitotic spindle". Nature. 428 (6978): 93–7. Bibcode:2004Natur.428...93D. doi:10.1038/nature02328. PMID 14961024. S2CID 4418232.
- ^ Cimini D, Wan X, Hirel CB, Salmon ED (September 2006). "Aurora kinase promotes turnover of kinetochore microtubules to reduce chromosome segregation errors". Current Biology. 16 (17): 1711–8. doi:10.1016/j.cub.2006.07.022. PMID 16950108. S2CID 18117282.
- ^ Gautschi O, Heighway J, Mack PC, Purnell PR, Lara PN, Gandara DR (March 2008). "Aurora kinases as anticancer drug targets". Clinical Cancer Research. 14 (6): 1639–48. doi:10.1158/1078-0432.CCR-07-2179. PMID 18347165.
- ^ a b Hirano T (2000). "Chromosome cohesion, condensation, and separation". Annual Review of Biochemistry. 69: 115–44. doi:10.1146/annurev.biochem.69.1.115. PMID 10966455.
- ^ Tanaka K, Hao Z, Kai M, Okayama H (October 2001). "Establishment and maintenance of sister chromatid cohesion in fission yeast by a unique mechanism". The EMBO Journal. 20 (20): 5779–90. doi:10.1093/emboj/20.20.5779. PMC 125673. PMID 11598020.
- ^ Gonzalez C, Casal Jimenez J, Ripoll P, Sunkel CE (January 1991). "The spindle is required for the process of sister chromatid separation in Drosophila neuroblasts". Experimental Cell Research. 192 (1): 10–5. doi:10.1016/0014-4827(91)90150-S. PMID 1898588.
- ^ Losada A, Hirano T (October 2001). "Shaping the metaphase chromosome: coordination of cohesion and condensation". BioEssays. 23 (10): 924–35. doi:10.1002/bies.1133. PMID 11598959. S2CID 31210810.
- ^ Bernard P, Maure JF, Partridge JF, Genier S, Javerzat JP, Allshire RC (December 2001). "Requirement of heterochromatin for cohesion at centromeres". Science. 294 (5551): 2539–42. Bibcode:2001Sci...294.2539B. doi:10.1126/science.1064027. PMID 11598266. S2CID 31166180.
- ^ Nonaka N, Kitajima T, Yokobayashi S, Xiao G, Yamamoto M, Grewal SI, Watanabe Y (January 2002). "Recruitment of cohesin to heterochromatic regions by Swi6/HP1 in fission yeast". Nature Cell Biology. 4 (1): 89–93. doi:10.1038/ncb739. PMID 11780129. S2CID 23036084.
- ^ Hall IM, Noma K, Grewal SI (January 2003). "RNA interference machinery regulates chromosome dynamics during mitosis and meiosis in fission yeast". Proceedings of the National Academy of Sciences of the United States of America. 100 (1): 193–8. Bibcode:2003PNAS..100..193H. doi:10.1073/pnas.232688099. PMC 140924. PMID 12509501.
- ^ Fukagawa T, Nogami M, Yoshikawa M, Ikeno M, Okazaki T, Takami Y, Nakayama T, Oshimura M (August 2004). "Dicer is essential for formation of the heterochromatin structure in vertebrate cells". Nature Cell Biology. 6 (8): 784–91. doi:10.1038/ncb1155. PMID 15247924. S2CID 24798145.
- ^ Weber SA, Gerton JL, Polancic JE, DeRisi JL, Koshland D, Megee PC (September 2004). "The kinetochore is an enhancer of pericentric cohesin binding". PLOS Biology. 2 (9): E260. doi:10.1371/journal.pbio.0020260. PMC 490027. PMID 15309047.

- ^ Prasanth SG, Prasanth KV, Siddiqui K, Spector DL, Stillman B (July 2004). "Human Orc2 localizes to centrosomes, centromeres and heterochromatin during chromosome inheritance". The EMBO Journal. 23 (13): 2651–63. doi:10.1038/sj.emboj.7600255. PMC 449767. PMID 15215892.
- ^ Shimada K, Gasser SM (January 2007). "The origin recognition complex functions in sister-chromatid cohesion in Saccharomyces cerevisiae". Cell. 128 (1): 85–99. doi:10.1016/j.cell.2006.11.045. PMID 17218257.
- ^ Kato H, Matsunaga F, Miyazaki S, Yin L, D'Urso G, Tanaka K, Murakami Y (April 2008). "Schizosaccharomyces pombe Orc5 plays multiple roles in the maintenance of genome stability throughout the cell cycle". Cell Cycle. 7 (8): 1085–96. doi:10.4161/cc.7.8.5710. PMID 18414064.
- ^ Hauf S, Watanabe Y (October 2004). "Kinetochore orientation in mitosis and meiosis". Cell. 119 (3): 317–27. doi:10.1016/j.cell.2004.10.014. PMID 15507205.
- ^ Sonoda E, Matsusaka T, Morrison C, Vagnarelli P, Hoshi O, Ushiki T, Nojima K, Fukagawa T, Waizenegger IC, Peters JM, Earnshaw WC, Takeda S (December 2001). "Scc1/Rad21/Mcd1 is required for sister chromatid cohesion and kinetochore function in vertebrate cells". Developmental Cell. 1 (6): 759–70. doi:10.1016/S1534-5807(01)00088-0. PMID 11740938.
- ^ Vass S, Cotterill S, Valdeolmillos AM, Barbero JL, Lin E, Warren WD, Heck MM (February 2003). "Depletion of Drad21/Scc1 in Drosophila cells leads to instability of the cohesin complex and disruption of mitotic progression" (PDF). Current Biology. 13 (3): 208–18. doi:10.1016/S0960-9822(03)00047-2. hdl:20.500.11820/b75b5706-3f21-4cfe-85be-466268afc918. PMID 12573216. S2CID 16037196.
- ^ Haering CH, Löwe J, Hochwagen A, Nasmyth K (April 2002). "Molecular architecture of SMC proteins and the yeast cohesin complex". Molecular Cell. 9 (4): 773–88. doi:10.1016/S1097-2765(02)00515-4. PMID 11983169.
- ^ Hirano T (January 1999). "SMC-mediated chromosome mechanics: a conserved scheme from bacteria to vertebrates?". Genes & Development. 13 (1): 11–9. doi:10.1101/gad.13.1.11. PMID 9887095.
- ^ Ciosk R, Zachariae W, Michaelis C, Shevchenko A, Mann M, Nasmyth K (June 1998). "An ESP1/PDS1 complex regulates loss of sister chromatid cohesion at the metaphase to anaphase transition in yeast". Cell. 93 (6): 1067–76. doi:10.1016/S0092-8674(00)81211-8. PMID 9635435.
- ^ Alexandru G, Uhlmann F, Mechtler K, Poupart MA, Nasmyth K (May 2001). "Phosphorylation of the cohesin subunit Scc1 by Polo/Cdc5 kinase regulates sister chromatid separation in yeast". Cell. 105 (4): 459–72. doi:10.1016/S0092-8674(01)00362-2. PMID 11371343.
- ^ Leismann O, Herzig A, Heidmann S, Lehner CF (September 2000). "Degradation of Drosophila PIM regulates sister chromatid separation during mitosis". Genes & Development. 14 (17): 2192–205. doi:10.1101/gad.176700. PMC 316890. PMID 10970883.
- ^ Zur A, Brandeis M (February 2001). "Securin degradation is mediated by fzy and fzr, and is required for complete chromatid separation but not for cytokinesis". The EMBO Journal. 20 (4): 792–801. doi:10.1093/emboj/20.4.792. PMC 145417. PMID 11179223.
- ^ Sumara I, Vorlaufer E, Gieffers C, Peters BH, Peters JM (November 2000). "Characterization of vertebrate cohesin complexes and their regulation in prophase". The Journal of Cell Biology. 151 (4): 749–62. doi:10.1083/jcb.151.4.749. PMC 2169443. PMID 11076961.
- ^ Losada A, Yokochi T, Kobayashi R, Hirano T (August 2000). "Identification and characterization of SA/Scc3p subunits in the Xenopus and human cohesin complexes". The Journal of Cell Biology. 150 (3): 405–16. doi:10.1083/jcb.150.3.405. PMC 2175199. PMID 10931856.
- ^ Giménez-Abián JF, Sumara I, Hirota T, Hauf S, Gerlich D, de la Torre C, Ellenberg J, Peters JM (July 2004). "Regulation of sister chromatid cohesion between chromosome arms". Current Biology. 14 (13): 1187–93. doi:10.1016/j.cub.2004.06.052. PMID 15242616.
- ^ Paliulis LV, Nicklas RB (December 2004). "Micromanipulation of chromosomes reveals that cohesion release during cell division is gradual and does not require tension". Current Biology. 14 (23): 2124–9. doi:10.1016/j.cub.2004.11.052. PMID 15589155.
- ^ Nakajima M, Kumada K, Hatakeyama K, Noda T, Peters JM, Hirota T (December 2007). "The complete removal of cohesin from chromosome arms depends on separase". Journal of Cell Science. 120 (Pt 23): 4188–96. doi:10.1242/jcs.011528. PMID 18003702.
- ^ McGuinness BE, Hirota T, Kudo NR, Peters JM, Nasmyth K (March 2005). "Shugoshin prevents dissociation of cohesin from centromeres during mitosis in vertebrate cells". PLOS Biology. 3 (3): e86. doi:10.1371/journal.pbio.0030086. PMC 1054882. PMID 15737064.

- ^ Salic A, Waters JC, Mitchison TJ (September 2004). "Vertebrate shugoshin links sister centromere cohesion and kinetochore microtubule stability in mitosis". Cell. 118 (5): 567–78. doi:10.1016/j.cell.2004.08.016. PMID 15339662.
- ^ a b c d De Antoni A, Pearson CG, Cimini D, Canman JC, Sala V, Nezi L, Mapelli M, Sironi L, Faretta M, Salmon ED, Musacchio A (February 2005). "The Mad1/Mad2 complex as a template for Mad2 activation in the spindle assembly checkpoint". Current Biology. 15 (3): 214–25. doi:10.1016/j.cub.2005.01.038. PMID 15694304. S2CID 3224122.
- ^ a b c d e f g Musacchio A, Salmon ED (May 2007). "The spindle-assembly checkpoint in space and time". Nature Reviews. Molecular Cell Biology. 8 (5): 379–93. doi:10.1038/nrm2163. PMID 17426725. S2CID 205494124.
- ^ Martin-Lluesma S, Stucke VM, Nigg EA (September 2002). "Role of Hec1 in spindle checkpoint signaling and kinetochore recruitment of Mad1/Mad2". Science. 297 (5590): 2267–70. Bibcode:2002Sci...297.2267M. doi:10.1126/science.1075596. PMID 12351790. S2CID 7879023.
- ^ Lens SM, Wolthuis RM, Klompmaker R, Kauw J, Agami R, Brummelkamp T, Kops G, Medema RH (June 2003). "Survivin is required for a sustained spindle checkpoint arrest in response to lack of tension". The EMBO Journal. 22 (12): 2934–47. doi:10.1093/emboj/cdg307. PMC 162159. PMID 12805209.
- ^ Hauf S, Cole RW, LaTerra S, Zimmer C, Schnapp G, Walter R, Heckel A, van Meel J, Rieder CL, Peters JM (April 2003). "The small molecule Hesperadin reveals a role for Aurora B in correcting kinetochore-microtubule attachment and in maintaining the spindle assembly checkpoint". The Journal of Cell Biology. 161 (2): 281–94. doi:10.1083/jcb.200208092. PMC 2172906. PMID 12707311.
- ^ Aravamudhan P, Goldfarb AA, Joglekar AP (July 2015). "The kinetochore encodes a mechanical switch to disrupt spindle assembly checkpoint signalling". Nature Cell Biology. 17 (7): 868–79. doi:10.1038/ncb3179. PMC 4630029. PMID 26053220.
- ^ Alberts B, Johnson A, Lewis J, Morgan D, Raff M, Roberts K, Walter P (2015). Molecular Biology of The Cell (6th ed.). New York, NY: Garland Science, Taylor & Francis Group. p. 988. ISBN 978-0-8153-4432-2.
- ^ Kops GJ, Weaver BA, Cleveland DW (October 2005). "On the road to cancer: aneuploidy and the mitotic checkpoint". Nature Reviews. Cancer. 5 (10): 773–85. doi:10.1038/nrc1714. PMID 16195750. S2CID 2515388.
- ^ Lengauer C, Kinzler KW, Vogelstein B (December 1998). "Genetic instabilities in human cancers". Nature. 396 (6712): 643–9. Bibcode:1998Natur.396..643L. doi:10.1038/25292. PMID 9872311. S2CID 204996480.
- ^ Weaver BA, Cleveland DW (December 2006). "Does aneuploidy cause cancer?". Current Opinion in Cell Biology. 18 (6): 658–67. doi:10.1016/j.ceb.2006.10.002. PMID 17046232.
- ^ Cahill DP, Lengauer C, Yu J, Riggins GJ, Willson JK, Markowitz SD, Kinzler KW, Vogelstein B (March 1998). "Mutations of mitotic checkpoint genes in human cancers". Nature. 392 (6673): 300–3. Bibcode:1998Natur.392..300C. doi:10.1038/32688. PMID 9521327. S2CID 4416376.
- ^ Díaz-Rodríguez E, Álvarez-Fernández S, Chen X, Paiva B, López-Pérez R, García-Hernández JL, San Miguel JF, Pandiella A (2011). "Deficient spindle assembly checkpoint in multiple myeloma". PLOS ONE. 6 (11): e27583. Bibcode:2011PLoSO...627583D. doi:10.1371/journal.pone.0027583. PMC 3223182. PMID 22132115.

- ^ Grady, William M. (2004). "Genomic instability and colon cancer". Cancer and Metastasis Reviews. 23 (1–2): 11–27. doi:10.1023/A:1025861527711. PMID 15000146. S2CID 1177511.
- ^ Wang RH, Yu H, Deng CX (December 2004). "A requirement for breast-cancer-associated gene 1 (BRCA1) in the spindle checkpoint". Proceedings of the National Academy of Sciences of the United States of America. 101 (49): 17108–13. Bibcode:2004PNAS..10117108W. doi:10.1073/pnas.0407585101. PMC 535394. PMID 15563594.
- ^ Sotillo R, Hernando E, Díaz-Rodríguez E, Teruya-Feldstein J, Cordón-Cardo C, Lowe SW, Benezra R (January 2007). "Mad2 overexpression promotes aneuploidy and tumorigenesis in mice". Cancer Cell. 11 (1): 9–23. doi:10.1016/j.ccr.2006.10.019. PMC 1850996. PMID 17189715.
- ^ Yamamoto Y, Matsuyama H, Chochi Y, Okuda M, Kawauchi S, Inoue R, Furuya T, Oga A, Naito K, Sasaki K (April 2007). "Overexpression of BUBR1 is associated with chromosomal instability in bladder cancer". Cancer Genetics and Cytogenetics. 174 (1): 42–7. doi:10.1016/j.cancergencyto.2006.11.012. PMID 17350465.
- ^ a b Weaver BA, Cleveland DW (June 2009). "The role of aneuploidy in promoting and suppressing tumors". The Journal of Cell Biology. 185 (6): 935–7. doi:10.1083/jcb.200905098. PMC 2711620. PMID 19528293.
- ^ Cross, Shawn M.; Sanchez, Carissa A; Morgan, Catherine A.; Schimke, Melana K.; Reid, Brian J. (1995). "A p53-dependant mouse spindle checkpoint". Science. 3 (5202): 1353–1356. Bibcode:1995Sci...267.1353C. doi:10.1126/science.7871434. PMID 7871434. S2CID 38128370.
- ^ Altieri DC (December 2001). "The molecular basis and potential role of survivin in cancer diagnosis and therapy". Trends in Molecular Medicine. 7 (12): 542–7. doi:10.1016/S1471-4914(01)02243-2. PMID 11733216.
- ^ Gisselsson D, Håkanson U, Stoller P, Marti D, Jin Y, Rosengren AH, Stewénius Y, Kahl F, Panagopoulos I (April 2008). "When the genome plays dice: circumvention of the spindle assembly checkpoint and near-random chromosome segregation in multipolar cancer cell mitoses". PLOS ONE. 3 (4): e1871. Bibcode:2008PLoSO...3.1871G. doi:10.1371/journal.pone.0001871. PMC 2289843. PMID 18392149.

- ^ Zhou J, Giannakakou P (January 2005). "Targeting microtubules for cancer chemotherapy". Current Medicinal Chemistry. Anti-Cancer Agents. 5 (1): 65–71. doi:10.2174/1568011053352569. PMID 15720262.
- ^ a b Carvajal RD, Tse A, Schwartz GK (December 2006). "Aurora kinases: new targets for cancer therapy". Clinical Cancer Research. 12 (23): 6869–75. doi:10.1158/1078-0432.CCR-06-1405. PMID 17145803.
- ^ Anand S, Penrhyn-Lowe S, Venkitaraman AR (January 2003). "AURORA-A amplification overrides the mitotic spindle assembly checkpoint, inducing resistance to Taxol". Cancer Cell. 3 (1): 51–62. doi:10.1016/S1535-6108(02)00235-0. PMID 12559175.
- ^ Harrington EA, Bebbington D, Moore J, Rasmussen RK, Ajose-Adeogun AO, Nakayama T, Graham JA, Demur C, Hercend T, Diu-Hercend A, Su M, Golec JM, Miller KM (March 2004). "VX-680, a potent and selective small-molecule inhibitor of the Aurora kinases, suppresses tumor growth in vivo". Nature Medicine. 10 (3): 262–7. doi:10.1038/nm1003. PMID 14981513. S2CID 12918452.
- ^ Altieri DC (January 2008). "Survivin, cancer networks and pathway-directed drug discovery". Nature Reviews. Cancer. 8 (1): 61–70. doi:10.1038/nrc2293. PMID 18075512. S2CID 25597711.
- ^ Tao W, South VJ, Zhang Y, Davide JP, Farrell L, Kohl NE, Sepp-Lorenzino L, Lobell RB (July 2005). "Induction of apoptosis by an inhibitor of the mitotic kinesin KSP requires both activation of the spindle assembly checkpoint and mitotic slippage". Cancer Cell. 8 (1): 49–59. doi:10.1016/j.ccr.2005.06.003. PMID 16023598.
추가 읽기
- Larsen NA, Al-Bassam J, Wei RR, Harrison SC (January 2007). "Structural analysis of Bub3 interactions in the mitotic spindle checkpoint". Proceedings of the National Academy of Sciences of the United States of America. 104 (4): 1201–6. Bibcode:2007PNAS..104.1201L. doi:10.1073/pnas.0610358104. PMC 1770893. PMID 17227844.
- Wang X, Babu JR, Harden JM, Jablonski SA, Gazi MH, Lingle WL, de Groen PC, Yen TJ, van Deursen JM (July 2001). "The mitotic checkpoint protein hBUB3 and the mRNA export factor hRAE1 interact with GLE2p-binding sequence (GLEBS)-containing proteins". The Journal of Biological Chemistry. 276 (28): 26559–67. doi:10.1074/jbc.M101083200. PMID 11352911.
- Kitagawa R, Rose AM (December 1999). "Components of the spindle-assembly checkpoint are essential in Caenorhabditis elegans". Nature Cell Biology. 1 (8): 514–21. doi:10.1038/70309. PMID 10587648. S2CID 25953096.
외부 링크
- Ted Salmon의 연구실: 세포 영화를 나누는 것.[1]
- 안드레아 무사키오의 연구실: 스핀들 체크포인트 계획.[2]
- http://www.uniprot.org/uniprot/O60566