엑소좀 복합체
Exosome complex
엑소좀 복합체(또는 PM/Scl 복합체, 종종 엑소좀이라고도 함)는 다양한 종류의 RNA(리보핵산) 분자를 분해할 수 있는 다중 단백질 세포내 복합체입니다. 엑소좀 복합체는 진핵세포와 고세균 모두에서 발견되는 반면, 박테리아에서는 분해소체라고 불리는 더 단순한 복합체가 유사한 기능을 수행합니다.
엑소좀의 핵심은 다른 단백질이 붙어 있는 6원 고리 구조를 포함하고 있습니다. 진핵 세포에서 엑소좀 복합체는 세포질, 핵, 특히 핵소체에 존재하지만, 다른 단백질은 이러한 구획에서 엑소좀 복합체와 상호작용하여 이들 세포 구획에 특이적인 기질에 대한 복합체의 RNA 분해 활성을 조절합니다. 엑소좀의 기질에는 전령 RNA, 리보솜 RNA 및 많은 종의 작은 RNA가 포함됩니다. 엑소좀은 엑소리보핵 분해 기능을 가지고 있는데, 이는 한쪽 끝에서 시작하여 RNA를 분해하는 것을 의미하며, 진핵생물에서도 분자 내의 위치에서 RNA를 절단하는 엔도리보핵 분해 기능을 가지고 있습니다.
엑소좀의 여러 단백질은 특정 자가면역질환(특히 PM/Scl 중복 증후군) 환자의 자가항체의 표적이 되며, 엑소좀의 활성을 차단하여 암 기능을 위한 일부 항대사 화학요법의 표적이 됩니다. 또한 엑소좀 성분 3의 돌연변이는 소뇌 저형성증과 척추 운동 뉴런증을 유발합니다.
디스커버리
엑소좀은 1997년 흔히 사용되는 모델 유기체인 신진 효모 Saccharomyces cerevisiae에서 RNase로 처음 발견되었습니다.[1] 얼마 지나지 않아 1999년, 엑소좀이 실제로 인간 세포에서 이미 설명된 PM/Scl 복합체와 동등한 효모라는 것을 알게 되었고, 이는 몇 년 전에 특정 자가면역질환 환자에서 자가항원으로 확인되었습니다(아래 참조).[2] 이 "PM/Scl 복합체"의 정제를 통해 더 많은 인간 엑소좀 단백질을 확인할 수 있었고 결국 복합체의 모든 구성 요소를 특성화할 수 있었습니다.[3][4] 2001년, 이용 가능해진 게놈 데이터의 양이 증가함에 따라 고세균의 엑소좀 단백질을 예측할 수 있게 되었지만, 고세균 유기체의 첫 번째 엑소좀 복합체가 정제되기까지는 2년이 더 걸릴 것입니다.[5][6]
구조.
핵심단백질

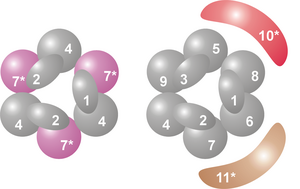
복합체의 핵심은 RNase PH 유사 단백질인 RNase의 동일한 클래스에 속하는 6개의 단백질로 구성된 고리 구조를 가지고 있습니다.[7] 고세균에는 두 개의 다른 PH 유사 단백질(Rrp41 및 Rrp42)이 있으며, 각각 세 번씩 교대로 존재합니다. 진핵생물 엑소좀 복합체는 고리 구조를 형성하는 6가지 다른 단백질을 가지고 있습니다.[8][9] 이 6가지 진핵 단백질 중 3가지는 고세균 Rrp41 단백질과 비슷하고 나머지 3가지 단백질은 고세균 Rrp42 단백질과 더 유사합니다.[10]

이 고리 위에는 S1 RNA 결합 도메인(RBD)을 가진 세 개의 단백질이 있습니다. 또한 두 개의 단백질은 K-호몰로지(KH) 도메인을 가지고 있습니다.[7] 진핵생물에서는 서로 다른 세 개의 S1 단백질이 고리에 결합되어 있는 반면, 고세균에서는 하나 또는 두 개의 서로 다른 S1 단백질이 엑소좀의 일부가 될 수 있습니다(단, 복합체에는 항상 세 개의 S1 소단위가 부착되어 있습니다).[11]
이 고리 구조는 단백질 RNase PH 및 PNPase와 매우 유사합니다. 박테리아에서 tRNA 처리에 관여하는 단백질 RNase PH는 6개의 동일한 RNase PH 단백질로 구성된 6량체 고리를 형성합니다.[12][13] 박테리아와 일부 진핵생물의 엽록체 및 미토콘드리아에서 발견되는 인산분해 RNA 분해 단백질인 PNPase의 경우, 2개의 RNase PH 도메인과 S1 및 KH RNA 결합 도메인이 모두 단일 단백질의 일부로서, 엑소좀과 거의 동일한 구조를 갖는 삼량체 복합체를 형성합니다.[14] 이러한 단백질 도메인과 구조 모두에서 유사성이 높기 때문에 이들 복합체는 진화적으로 연관되어 있으며 공통적인 조상을 가지고 있는 것으로 생각됩니다.[15] RNase PH 유사 엑소좀 단백질, PNPase 및 RNase PH는 모두 RNase PH 계열에 속하며, RNA 분자의 3' 말단에서 뉴클레오티드를 제거하기 위해 무기 인산염을 사용하는 인산분해 엑소리보뉴클레아제입니다.[7]
관련 단백질
이 9개의 핵심 엑소좀 단백질 외에 다른 2개의 단백질은 종종 진핵생물의 복합체와 결합합니다. 그 중 하나는 가수분해성 RNase R 계열의 가수분해성 엑소리보뉴클레아제(물을 이용하여 뉴클레오티드 결합을 절단하는 뉴클레오티드)인 Rrp44입니다. Rrp44는 엑소리보핵 분해 효소일 뿐만 아니라 단백질의 별도 영역에 존재하는 엔도리보핵 분해 활성을 가지고 있습니다.[16][17] 효모에서 Rrp44는 모든 엑소좀 복합체와 연관되어 있으며 효모 엑소좀 복합체의 활성에 중요한 역할을 합니다.[18] 단백질의 인간 상동체가 존재하지만, 인간 상동체가 인간 엑소좀 복합체와 연관되어 있다는 증거는 오랫동안 발견되지 않았습니다.[7] 그러나 2010년 인간은 3개의 Rrp44 상동체를 가지고 있으며 이 중 2개는 엑소좀 복합체와 연관될 수 있다는 것이 밝혀졌습니다. 이 두 단백질은 다른 세포 위치로 인해 다른 RNA 기질을 분해할 가능성이 가장 높으며, 하나는 세포질(DIS3L1)에 국한되고 다른 하나는 핵(DIS3)에 국한됩니다.[19][20]

두 번째로 일반적인 관련 단백질은 Rrp6 (효모에서) 또는 PM/Scl-100 (인간에서)이라고 불립니다. 이 단백질은 Rrp44와 마찬가지로 가수분해성 엑소리보뉴클레아제이지만, 이 경우 RNase D 단백질 계열입니다.[21] 단백질 PM/Scl-100은 세포의 핵에 있는 엑소좀 복합체의 가장 일반적인 부분이지만 세포질 엑소좀 복합체의 일부를 형성할 수도 있습니다.[22]
조절단백질
이 두 개의 단단히 결합된 단백질 소단위체 외에도 많은 단백질이 세포의 세포질과 핵 모두에서 엑소좀 복합체와 상호작용합니다. 느슨하게 결합된 이러한 단백질은 엑소좀 복합체의 활성과 특이성을 조절할 수 있습니다. 세포질에서 엑소좀은 mRNA의 분해를 촉진하거나 방지할 수 있는 AU-풍부 요소(ARE) 결합 단백질(예: KRSP 및 TTP)과 상호작용합니다. 핵 엑소좀은 특정 기질을 처리하는 데 필요한 RNA 결합 단백질(예: 인간/효모의 MPP6/Mpp6 및 C1D/Rrp47)과 결합합니다.[7]
단일 단백질 외에도 다른 단백질 복합체가 엑소좀과 상호 작용합니다. 그 중 하나는 RNA 헬리카제(Ski2)를 포함하고 mRNA 분해에 관여하는 세포질 스키 복합체입니다.[23] 핵에서, 엑소좀에 의한 rRNA 및 snoRNA의 처리는 RNA 헬리카제(Mtr4) 및 폴리아데닐화(Trf4) 활성을 모두 포함하는 TRAMP 복합체에 의해 매개됩니다.[24]
기능.
효소기능
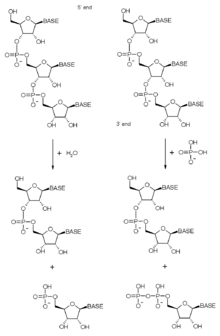
위에서 언급한 바와 같이, 엑소좀 복합체는 리보뉴클레아제 도메인을 갖는 많은 단백질을 포함합니다. 이러한 리보뉴클레아제 도메인의 정확한 특성은 다양한 활동이 얻고 없어짐에 따라 박테리아에서 고세균에서 진핵 복합체로 진화 전반에 걸쳐 변화했습니다. 엑소좀은 주로 3'-5' 엑소리보뉴클레아제인데, 이는 3' 말단의 RNA 분자를 분해한다는 것을 의미합니다. 엑소좀 복합체에 포함된 엑소리보뉴클레아제는 인분해성(RNase PH 유사 단백질) 또는 진핵생물에서 가수분해성(RNase R 및 RNase D 도메인 단백질)입니다. 인산 분해 효소는 무기 인산염을 사용하여 포스포다이에스테르 결합을 절단하여 뉴클레오티드 이인산을 방출합니다. 가수분해 효소는 물을 사용하여 이러한 결합을 가수분해하여 뉴클레오티드 모노인산염을 방출합니다.
고세균에서 복합체의 Rrp41 서브유닛은 인산분해성 엑소리보뉴클레아제입니다. 이 단백질의 3개 사본이 고리에 존재하며 복합체의 활동을 담당합니다.[9] 진핵생물에서 RNase PH 소단위체 중 어느 것도 이 촉매 활성을 유지하지 못했는데, 이는 인간 엑소좀의 핵심 고리 구조에 효소 활성 단백질이 없다는 것을 의미합니다.[25] 이러한 촉매 활성의 손실에도 불구하고 코어 엑소좀의 구조는 고세균에서 인간에 이르기까지 고도로 보존되어 있어 복합체가 중요한 세포 기능을 수행함을 시사합니다. 진핵생물에서 인산분해 활성의 부재는 이러한 유기체에서 엑소좀의 리보뉴클레아제 활성을 담당하는 가수분해 효소의 존재에 의해 보상됩니다.[26][27][28]
위에서 언급한 바와 같이, 가수분해 단백질 Rrp6 및 Rrp44는 효모의 엑소좀과 연관되어 있으며, 인간의 경우 Rrp6 외에도 두 가지 다른 단백질인 Dis3 및 Dis3L1이 효모 Rrp44 단백질의 위치에 연관될 수 있습니다.[19][20] 원래 S1 도메인 단백질도 3'-5' 가수분해성 엑소리보뉴클레아제 활성을 갖는 것으로 생각되었지만, 이 활성의 존재에 대한 의문이 최근 제기되었으며 이 단백질은 복합체에 의해 분해되기 전에 기질에 결합하는 역할을 할 수 있습니다.[26]
기판
엑소좀은 다양한 RNA 종의 분해 및 처리에 관여합니다. 세포의 세포질에서 전령RNA(mRNA) 분자의 전환에 관여합니다. 복합체는 넌센스 매개 붕괴 또는 논스톱 붕괴 경로의 단백질과의 상호작용을 통해 오류를 포함하기 때문에 분해를 위해 태그된 mRNA 분자를 분해할 수 있습니다. 대체적인 방식으로 mRNA는 정상적인 회전율의 일부로 분해됩니다. mRNA의 3' 비번역 영역에 있는 AU가 풍부한 요소와의 결합을 통해 mRNA 분자를 안정화시키거나 불안정하게 만드는 여러 단백질이 엑소좀 복합체와 상호작용합니다.[29][30][31] 핵에서 엑소좀은 몇몇 작은 핵 RNA 분자의 정확한 처리를 위해 필요합니다.[32] 마지막으로, 핵소체는 대부분의 엑소좀 복합체가 발견되는 구획입니다. 거기서 그것은 5.8S 리보솜 RNA(exosome의 최초로 확인된 기능)와 몇몇 작은 핵 RNA의 처리에 역할을 합니다.[1][32][33]
대부분의 세포는 RNA의 3' 말단이나 5' 말단에서 RNA를 분해할 수 있는 다른 효소를 가지고 있지만, 엑소좀 복합체는 세포 생존에 필수적입니다. 예를 들어 RNA의 간섭에 의해 엑소좀 단백질의 발현이 인위적으로 줄어들거나 멈추면 성장이 멈추고 세포는 결국 죽게 됩니다. 엑소좀 복합체의 핵심 단백질과 두 가지 주요 결합 단백질은 모두 필수 단백질입니다.[34] 박테리아는 엑소좀 복합체를 가지고 있지 않지만, 유사한 기능은 디데오좀이라고 불리는 단백질 PNPase를 포함하는 더 단순한 복합체에 의해 수행됩니다.[35]

엑소좀은 세포 RNA 품질 관리의 핵심 복합체입니다. 원핵생물과 달리, 진핵생물은 핵에서 빠져나오기 전에 미처리 및 오처리된 RNA-단백질 복합체(리보솜과 같은)를 인식하는 매우 활동적인 RNA 감시 시스템을 가지고 있습니다. 이 시스템이 비정상적인 복합체가 단백질 합성과 같은 중요한 세포 과정을 방해하는 것을 방지하는 것으로 추정됩니다.[36]
RNA 처리, 회전율 및 감시 활동 외에도 엑소좀은 효모 유전체 내 수천 개의 유전자좌에서 생성되는 소위 암호화 불안정 전사체(CUT)의 분해에 중요합니다.[37][38] 이러한 불안정한 RNA의 중요성과 분해는 아직 명확하지 않지만, 인간 세포에서도 유사한 RNA 종이 검출되었습니다.[39]
질병
자가면역
엑소좀 복합체는 다양한 자가면역질환 환자의 자가항체 표적입니다. 이 자가항체들은 주로 PM/Scl 중복 증후군을 가진 사람들에게서 발견되는데, 이는 환자들이 공막피증과 다발성근염 혹은 피부근염의 증상을 모두 가지고 있는 자가면역질환입니다.[40] 자가항체는 다양한 분석을 통해 환자의 혈청에서 검출될 수 있습니다. 과거에 가장 많이 사용된 방법은 송아지 흉선 추출물을 이용한 이중 면역확산법, HEp-2 세포에 대한 면역형광법 또는 인간 세포 추출물의 면역침전법이었습니다. 항-엑소좀 양성 혈청의 혈청을 사용한 면역침전 분석에서는 독특한 단백질 세트가 침전됩니다. 엑소좀 복합체가 확인되기 몇 년 전에 이 패턴을 PM/Scl 복합체라고 불렀습니다.[41] 이 환자들의 혈청을 이용한 면역형광은 일반적으로 세포의 핵소체의 전형적인 염색을 보여주는데, 이는 자가항체에 의해 인식되는 항원이 리보솜 합성에 중요할 수 있다는 제안을 촉발시켰습니다.[42] 보다 최근에는 재조합 엑소좀 단백질을 이용할 수 있게 되었으며 이러한 항체를 검출하기 위한 라인 면역분석(LIA) 및 효소연계 면역흡착분석(ELISA)을 개발하는 데 사용되었습니다.[7]
이러한 질병에서 항체는 주로 PM/Scl-100(단백질과 같은 RNase D) 및 PM/Scl-75(고리에서 나온 단백질과 같은 RNase PH 중 하나)라고 불리는 복합체의 단백질 중 두 가지에 대해 지시되며, 이러한 단백질을 인식하는 항체는 PM/Scl 오버랩 증후군 환자의 약 30%에서 발견됩니다.[43] 이 두 단백질이 자가항체의 주요 표적이지만, 다른 엑소좀 소단위체 및 관련 단백질(C1D와 같은)이 이러한 환자에서 표적이 될 수 있습니다.[44][45] 현재 이러한 항체를 검출하는 가장 민감한 방법은 ELISA에서 완전한 단백질 대신 PM/Scl-100 단백질에서 유래한 펩타이드를 항원으로 사용하는 것입니다. 이 방법으로 자가항체는 PM/Scl 중복증후군 환자의 최대 55%에서 발견되지만 공막진피증이나 다발근염, 피부근염 단독 환자에서도 검출이 가능합니다.[46]
자가면역질환의 특징을 가진 환자들에게서 주로 자가체가 발견되기 때문에, 이 환자들의 임상 증상은 매우 다양할 수 있습니다. 가장 많이 보이는 증상은 개별 자가면역질환의 전형적인 증상으로 레이노 현상, 관절염, 근염, 공막종 등이 있습니다.[47] 이 환자들의 치료는 증상이 있으며 종종 면역 억제제 또는 면역 조절제를 포함하는 개별 자가 면역 질환에 대한 치료와 유사합니다.[48]
암치료
엑소좀은 암의 화학요법에 사용되는 약물인 항대사물질 플루오로우라실에 의해 억제되는 것으로 나타났습니다. 고형 종양 치료에 가장 성공적인 약물 중 하나입니다. 플루오로우라실을 처리한 효모세포에서는 분자생물학적 전략에 의해 엑소좀의 활성이 차단되었을 때 보이는 것과 동일한 리보솜 RNA의 처리에서 결함이 발견되었습니다. 정확한 리보솜 RNA 처리가 부족하면 세포에 치명적이어서 약물의 항대사 효과를 설명합니다.[49]
신경학적 장애
엑소좀 성분 3의 돌연변이는 신생아 척추 운동 뉴런증, 소뇌 위축, 진행성 소두증 및 심각한 전지구적 발달 지연을 유발하며, 이는 췌장 소뇌 저형성 1B형(PCH1B; MIM 614678)과 일치합니다.[50]
소단위 목록
| 전설 | 일반명 | 도메인 | 인간 | 효모 (S. cerevisiae) | 고세균 | MW(kD) | 인간유전자 | 효모유전자 |
|---|---|---|---|---|---|---|---|---|
| 1 | Csl4 | S1 RBD | hCsl4 | Csl4p/Ski4p | Csl4 | 21–32 | EXOSC1 | YNL232W |
| 2 | Rrp4 | 에스원/케이에이치알비디 | hRrp4 | Rrp4p | Rrp4 | 28–39 | EXOSC2 | YHR069C |
| 3 | Rrp40 | 에스원/케이에이치알비디 | hRrp40 | Rrp40p | (Rrp4)A | 27–32 | EXOSC3 | YOL142W |
| 4 | Rrp41 | RNase PH | hRrp41 | Rrp41p/Ski6p | Rrp41C | 26–28 | EXOSC4 | YGR195W |
| 5 | Rrp46 | RNase PH | hRrp46 | Rrp46p | (Rrp41)A,C | 25–28 | EXOSC5 | YGR095C |
| 6 | Mtr3 | RNase PH | hMtr3 | Mtr3p | (Rrp41)A,C | 24–37 | EXOSC6 | YGR158C |
| 7 | Rrp42 | RNase PH | hRrp42 | Rrp42p | Rrp42 | 29–32 | EXOSC7 | YDL111C |
| 8 | Rrp43 | RNase PH | OIP2 | Rrp43p | (Rrp42)A | 30–44 | EXOSC8 | YCR035C |
| 9 | Rrp45 | RNase PH | PM/Scl-75 | Rrp45p | (Rrp42)A | 34–49 | EXOSC9 | YDR280W |
| 10 | Rrp6 | RNase D | PM/Scl-100C | Rrp6pC | n/a | 84–100 | EXOSC10 | YOR001W |
| 11 | Rrp44 | RNase R | 디스3B,C | Rrp44p/Dis3pC | n/a | 105–113 | DIS3 | YOL021C |
- A 고세균에서 여러 엑소좀 단백질이 여러 사본으로 존재하여 엑소좀 복합체의 전체 코어를 형성합니다.
- B 인간의 경우 두 가지 다른 단백질이 이 위치에서 연관될 수 있습니다. 세포의 세포질에서 Diss3L1은 엑소좀과 결합하는 반면 핵에서 Diss3는 코어 복합체와 결합할 수 있습니다.
- C 복합체의 리보핵 분해 활성에 기여합니다.
참고 항목
참고문헌
- ^ a b Mitchell, P; Petfalski, E; Shevchenko, A; Mann, M; Tollervey, D (1997). "The Exosome: A Conserved Eukaryotic RNA Processing Complex Containing Multiple 3′→5′ Exoribonucleases". Cell. 91 (4): 457–466. doi:10.1016/S0092-8674(00)80432-8. PMID 9390555. S2CID 16035676.
- ^ Allmang, C; Petfalski, E; Podtelejnikov, A; Mann, M; Tollervey, D; Mitchell, P (1999). "The yeast exosome and human PM-Scl are related complexes of 3' --> 5' exonucleases". Genes & Development. 13 (16): 2148–58. doi:10.1101/gad.13.16.2148. PMC 316947. PMID 10465791.
- ^ Brouwer, R; Allmang, C; Raijmakers, R; Van Aarssen, Y; Egberts, WV; Petfalski, E; Van Venrooij, WJ; Tollervey, D; Pruijn, GJ (2001). "Three novel components of the human exosome". Journal of Biological Chemistry. 276 (9): 6177–84. doi:10.1074/jbc.M007603200. hdl:2066/186951. PMID 11110791.
- ^ Chen, CY; Gherzi, R; Ong, SE; Chan, EL; Raijmakers, R; Pruijn, GJ; Stoecklin, G; Moroni, C; et al. (2001). "AU binding proteins recruit the exosome to degrade ARE-containing mRNAs". Cell. 107 (4): 451–64. doi:10.1016/S0092-8674(01)00578-5. PMID 11719186. S2CID 14817671.
- ^ Koonin, EV; Wolf, YI; Aravind, L (2001). "Prediction of the archaeal exosome and its connections with the proteasome and the translation and transcription machineries by a comparative-genomic approach". Genome Research. 11 (2): 240–52. doi:10.1101/gr.162001. PMC 311015. PMID 11157787.
- ^ Evguenieva-Hackenberg, E; Walter, P; Hochleitner, E; Lottspeich, F; Klug, G (2003). "An exosome-like complex in Sulfolobus solfataricus". EMBO Reports. 4 (9): 889–93. doi:10.1038/sj.embor.embor929. PMC 1326366. PMID 12947419.
- ^ a b c d e f Schilders, G; Van Dijk, E; Raijmakers, R; Pruijn, GJ (2006). Cell and molecular biology of the exosome: how to make or break an RNA. International Review of Cytology. Vol. 251. pp. 159–208. doi:10.1016/S0074-7696(06)51005-8. ISBN 9780123646552. PMID 16939780.
- ^ Lorentzen, E; Walter, P; Fribourg, S; Evguenieva-Hackenberg, E; Klug, G; Conti, E (2005). "The archaeal exosome core is a hexameric ring structure with three catalytic subunits". Nature Structural & Molecular Biology. 12 (7): 575–81. doi:10.1038/nsmb952. PMID 15951817. S2CID 2003922.
- ^ a b Shen, V; Kiledjian, M (2006). "A view to a kill: structure of the RNA exosome". Cell. 127 (6): 1093–5. doi:10.1016/j.cell.2006.11.035. PMC 1986773. PMID 17174886.
- ^ Raijmakers, R; Egberts, WV; Van Venrooij, WJ; Pruijn, GJ (2002). "Protein-protein interactions between human exosome components support the assembly of RNase PH-type subunits into a six-membered PNPase-like ring". Journal of Molecular Biology. 323 (4): 653–63. doi:10.1016/S0022-2836(02)00947-6. PMID 12419256.
- ^ Walter, P; Klein, F; Lorentzen, E; Ilchmann, A; Klug, G; Evguenieva-Hackenberg, E (2006). "Characterization of native and reconstituted exosome complexes from the hyperthermophilic archaeon Sulfolobus solfataricus". Molecular Microbiology. 62 (4): 1076–89. doi:10.1111/j.1365-2958.2006.05393.x. PMID 17078816. S2CID 27114625.
- ^ Ishii, R; Nureki, O; Yokoyama, S (2003). "Crystal structure of the tRNA processing enzyme RNase PH from Aquifex aeolicus". Journal of Biological Chemistry. 278 (34): 32397–404. doi:10.1074/jbc.M300639200. PMID 12746447.
- ^ Harlow, LS; Kadziola, A; Jensen, KF; Larsen, S (2004). "Crystal structure of the phosphorolytic exoribonuclease RNase PH from Bacillus subtilis and implications for its quaternary structure and tRNA binding". Protein Science. 13 (3): 668–77. doi:10.1110/ps.03477004. PMC 2286726. PMID 14767080.
- ^ Symmons, MF; Jones, GH; Luisi, BF (2000). "A duplicated fold is the structural basis for polynucleotide phosphorylase catalytic activity, processivity, and regulation". Structure. 8 (11): 1215–26. doi:10.1016/S0969-2126(00)00521-9. PMID 11080643.
- ^ Lin-Chao, S; Chiou, NT; Schuster, G (2007). "The PNPase, exosome and RNA helicases as the building components of evolutionarily-conserved RNA degradation machines". Journal of Biomedical Science. 14 (4): 523–32. doi:10.1007/s11373-007-9178-y. PMID 17514363.
- ^ Lebreton, A; Tomecki, R; Dziembowski, A; Séraphin, B (2008). "Endonucleolytic RNA cleavage by a eukaryotic exosome" (PDF). Nature. 456 (7224): 993–6. Bibcode:2008Natur.456..993L. doi:10.1038/nature07480. PMID 19060886. S2CID 1808371.
- ^ Schneider, C; Leung, E; Brown, J; Tollervey, D (2009). "The N-terminal PIN domain of the exosome subunit Rrp44 harbors endonuclease activity and tethers Rrp44 to the yeast core exosome". Nucleic Acids Research. 37 (4): 1127–40. doi:10.1093/nar/gkn1020. PMC 2651783. PMID 19129231.
- ^ Schneider, C; Anderson, JT; Tollervey, D (2007). "The exosome subunit Rrp44 plays a direct role in RNA substrate recognition". Molecular Cell. 27 (2): 324–31. doi:10.1016/j.molcel.2007.06.006. PMC 7610968. PMID 17643380.
- ^ a b Staals, RH; Bronkhorst, AW; Schilders, G; Slomovic, S; Schuster, G; Heck, AJ; Raijmakers, R; Pruijn, GJ (2010). "Dis3-like 1: a novel exoribonuclease associated with the human exosome". The EMBO Journal. 29 (14): 2358–67. doi:10.1038/emboj.2010.122. PMC 2910272. PMID 20531389.
- ^ a b Tomecki, R; Kristiansen, MS; Lykke-Andersen, S; Chlebowski, A; Larsen, KM; Szczesny, RJ; Drazkowska, K; Pastula, A; et al. (2010). "The human core exosome interacts with differentially localized processive RNases: hDIS3 and hDIS3L". The EMBO Journal. 29 (14): 2342–57. doi:10.1038/emboj.2010.121. PMC 2910271. PMID 20531386.
- ^ Mian, IS (1997). "Comparative sequence analysis of ribonucleases HII, III, II PH and D". Nucleic Acids Research. 25 (16): 3187–3195. doi:10.1093/nar/25.16.3187. PMC 146874. PMID 9241229.
- ^ Raijmakers, R; Schilders, G; Pruijn, GJ (2004). "The exosome, a molecular machine for controlled RNA degradation in both nucleus and cytoplasm". European Journal of Cell Biology. 83 (5): 175–83. doi:10.1078/0171-9335-00385. PMID 15346807.
- ^ Wang, L; Lewis, MS; Johnson, AW (2005). "Domain interactions within the Ski2/3/8 complex and between the Ski complex and Ski7p". RNA. 11 (8): 1291–302. doi:10.1261/rna.2060405. PMC 1370812. PMID 16043509.
- ^ LaCava, J; Houseley, J; Saveanu, C; Petfalski, E; Thompson, E; Jacquier, A; Tollervey, D (2005). "RNA degradation by the exosome is promoted by a nuclear polyadenylation complex". Cell. 121 (5): 713–24. doi:10.1016/j.cell.2005.04.029. PMID 15935758. S2CID 14898055.
- ^ Liu, Q; Greimann, JC; Lima, CD (2007). "Erratum: Reconstitution, activities, and structure of the eukaryotic RNA exosome". Cell. 131 (1): 188–189. doi:10.1016/j.cell.2007.09.019.
- ^ a b Dziembowski, A; Lorentzen, E; Conti, E; Séraphin, B (2007). "A single subunit, Dis3, is in essence responsible for yeast exosome core activity". Nature Structural & Molecular Biology. 14 (1): 15–22. doi:10.1038/nsmb1184. PMID 17173052. S2CID 24691764.
- ^ Liu, Q; Greimann, JC; Lima, CD (2006). "Reconstitution, activities, and structure of the eukaryotic RNA exosome". Cell. 127 (6): 1223–37. doi:10.1016/j.cell.2006.10.037. PMID 17174896. S2CID 62785677.
- ^ Lorentzen, E; Conti, E (2005). "Structural basis of 3' end RNA recognition and exoribonucleolytic cleavage by an exosome RNase PH core". Molecular Cell. 20 (3): 473–81. doi:10.1016/j.molcel.2005.10.020. PMID 16285928.
- ^ LeJeune, F; Li, X; Maquat, LE (2003). "Nonsense-mediated mRNA decay in mammalian cells involves decapping, deadenylating, and exonucleolytic activities". Molecular Cell. 12 (3): 675–87. doi:10.1016/S1097-2765(03)00349-6. PMID 14527413.
- ^ Wilson, MA; Meaux, S; Van Hoof, A (2007). "A genomic screen in yeast reveals novel aspects of nonstop mRNA metabolism". Genetics. 177 (2): 773–84. doi:10.1534/genetics.107.073205. PMC 2034642. PMID 17660569.
- ^ Lin, WJ; Duffy, A; Chen, CY (2007). "Localization of AU-rich element-containing mRNA in cytoplasmic granules containing exosome subunits". Journal of Biological Chemistry. 282 (27): 19958–68. doi:10.1074/jbc.M702281200. PMID 17470429.
- ^ a b Allmang, C; Kufel, J; Chanfreau, G; Mitchell, P; Petfalski, E; Tollervey, D (1999). "Functions of the exosome in rRNA, snoRNA and snRNA synthesis". EMBO Journal. 18 (19): 5399–410. doi:10.1093/emboj/18.19.5399. PMC 1171609. PMID 10508172.
- ^ Schilders, G; Raijmakers, R; Raats, JM; Pruijn, GJ (2005). "MPP6 is an exosome-associated RNA-binding protein involved in 5.8S rRNA maturation". Nucleic Acids Research. 33 (21): 6795–804. doi:10.1093/nar/gki982. PMC 1310903. PMID 16396833.
- ^ van Dijk, EL; Schilders, G; Pruijn, GJ (2007). "Human cell growth requires a functional cytoplasmic exosome, which is involved in various mRNA decay pathways". RNA. 13 (7): 1027–35. doi:10.1261/rna.575107. PMC 1894934. PMID 17545563.
- ^ Carpousis AJ, AJ (2002). "The Escherichia coli RNA degradosome: structure, function and relationship in other ribonucleolytic multienzyme complexes". Biochem. Soc. Trans. 30 (2): 150–5. doi:10.1042/BST0300150. PMID 12035760.
- ^ Houseley J, LaCava J, Tollervey D (July 2006). "RNA-quality control by the exosome". Nat. Rev. Mol. Cell Biol. 7 (7): 529–39. doi:10.1038/nrm1964. PMID 16829983. S2CID 22499032.
- ^ Wyers F, F; Rougemaille, M; Badis, G; et al. (June 2005). "Cryptic pol II transcripts are degraded by a nuclear quality control pathway involving a new poly(A) polymerase". Cell. 121 (5): 725–37. doi:10.1016/j.cell.2005.04.030. PMID 15935759. S2CID 1390706.
- ^ Neil H, Malabat C, d'Aubenton-Carafa Y, Xu Z, Steinmetz LM, Jacquier A (February 2009). "Widespread bidirectional promoters are the major source of cryptic transcripts in yeast". Nature. 457 (7232): 1038–42. Bibcode:2009Natur.457.1038N. doi:10.1038/nature07747. PMID 19169244. S2CID 4329373.
- ^ Preker P, P; Nielsen, J; Kammler, S; et al. (December 2008). "RNA exosome depletion reveals transcription upstream of active human promoters". Science. 322 (5909): 1851–4. Bibcode:2008Sci...322.1851P. doi:10.1126/science.1164096. PMID 19056938.
- ^ J.E. Pope, JE (2002). "Scleroderma overlap syndromes". Current Opinion in Rheumatology. 14 (6): 704–10. doi:10.1097/00002281-200211000-00013. PMID 12410095.
- ^ Gelpi, C; Algueró, A; Angeles Martinez, M; Vidal, S; Juarez, C; Rodriguez-Sanchez, JL (1991). "Identification of protein components reactive with anti-PM/Scl autoantibodies". Clinical and Experimental Immunology. 81 (1): 59–64. doi:10.1111/j.1365-2249.1990.tb05291.x. PMC 1535032. PMID 2199097.
- ^ Targoff, IN; Reichlin, M (1985). "Nucleolar localization of the PM-Scl antigen". Arthritis & Rheumatism. 28 (2): 226–30. doi:10.1002/art.1780280221. PMID 3918546.
- ^ Raijmakers, R; Renz, M; Wiemann, C; Egberts, WV; Seelig, HP; Van Venrooij, WJ; Pruijn, GJ (2004). "PM-Scl-75 is the main autoantigen in patients with the polymyositis/scleroderma overlap syndrome". Arthritis & Rheumatism. 50 (2): 565–9. doi:10.1002/art.20056. PMID 14872500.
- ^ Brouwer, R; Vree Egberts, WT; Hengstman, GJ; Raijmakers, R; Van Engelen, BG; Seelig, HP; Renz, M; Mierau, R; et al. (2002). "Autoantibodies directed to novel components of the PM/Scl complex, the human exosome". Arthritis Research & Therapy. 4 (2): 134–8. doi:10.1186/ar389. PMC 83843. PMID 11879549.
- ^ Schilders, G; Egberts, WV; Raijmakers, R; Pruijn, GJ (2007). "C1D is a major autoantibody target in patients with the polymyositis-scleroderma overlap syndrome". Arthritis & Rheumatism. 56 (7): 2449–54. doi:10.1002/art.22710. PMID 17599775.
- ^ Mahler, M; Raijmakers, R; Dähnrich, C; Blüthner, M; Fritzler, MJ (2005). "Clinical evaluation of autoantibodies to a novel PM/Scl peptide antigen". Arthritis Research & Therapy. 7 (3): R704–13. doi:10.1186/ar1729. PMC 1174964. PMID 15899056.
- ^ Mahler, M; Raijmakers, R (2007). "Novel aspects of autoantibodies to the PM/Scl complex: Clinical, genetic and diagnostic insights". Autoimmunity Reviews. 6 (7): 432–7. doi:10.1016/j.autrev.2007.01.013. PMID 17643929.
- ^ Jablonska, S; Blaszczyk, M (1998). "Scleromyositis: a scleroderma/polymyositis overlap syndrome". Clinical Rheumatology. 17 (6): 465–7. doi:10.1007/BF01451281. PMID 9890673. S2CID 39237322.
- ^ Lum, PY; Armour, CD; Stepaniants, SB; Cavet, G; Wolf, MK; Butler, JS; Hinshaw, JC; Garnier, P; et al. (2004). "Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes". Cell. 116 (1): 121–37. doi:10.1016/S0092-8674(03)01035-3. PMID 14718172. S2CID 11922219.
- ^ Wan, J.; Yourshaw, M.; Mamsa, H.; Rudnik-Schöneborn, S.; Menezes, M. P.; Hong, J. E.; Leong, D. W.; Senderek, J.; Salman, M. S.; Chitayat, D.; Seeman, P.; Von Moers, A.; Graul-Neumann, L.; Kornberg, A. J.; Castro-Gago, M.; Sobrido, M. A. J. S.; Sanefuji, M.; Shieh, P. B.; Salamon, N.; Kim, R. C.; Vinters, H. V.; Chen, Z.; Zerres, K.; Ryan, M. M.; Nelson, S. F.; Jen, J. C. (2012). "Mutations in the RNA exosome component gene EXOSC3 cause pontocerebellar hypoplasia and spinal motor neuron degeneration". Nature Genetics. 44 (6): 704–708. doi:10.1038/ng.2254. PMC 3366034. PMID 22544365.
더보기
- Schilders, G; Pruijn, GJ (2008). "Chapter 11 Biochemical Studies of the Mammalian Exosome with Intact Cells". RNA Turnover in Eukaryotes: Nucleases, Pathways and Analysis of mRNA Decay. Methods in Enzymology. Vol. 448. pp. 211–226. doi:10.1016/S0076-6879(08)02611-6. ISBN 9780123743787. PMID 19111178.
{{cite book}}:journal=무시됨(도움말) - Houseley, J; Tollervey, D (2008). "The nuclear RNA surveillance machinery: the link between ncRNAs and genome structure in budding yeast?". Biochim Biophys Acta. 1779 (4): 239–246. doi:10.1016/j.bbagrm.2007.12.008. PMID 18211833.
- Vanacova, S; Stefl, R (2007). "The exosome and RNA quality control in the nucleus". EMBO Reports. 8 (7): 651–657. doi:10.1038/sj.embor.7401005. PMC 1905902. PMID 17603538.
- Büttner, K; Wenig, K; Hopfner, KP (2006). "The exosome: a macromolecular cage for controlled RNA degradation". Molecular Microbiology. 61 (6): 1372–1379. CiteSeerX 10.1.1.232.6756. doi:10.1111/j.1365-2958.2006.05331.x. PMID 16968219. S2CID 6872855.
- Lorentzen, E; Conti, E (2006). "The Exosome and the Proteasome: Nano-Compartments for Degradation". Cell. 125 (4): 651–654. doi:10.1016/j.cell.2006.05.002. PMID 16713559.
- Pruijn, GJ (2005). "Doughnuts dealing with RNA". Nature Structural & Molecular Biology. 12 (7): 562–564. doi:10.1038/nsmb0705-562. PMID 15999107. S2CID 43218090.


