약제유전체학
Pharmacogenomics| 시리즈의 일부 |
| 유전학 |
|---|
 |
약제유전체학은 약물반응에서 게놈의 역할에 대한 연구이다.그 이름(약리-+게노믹스)은 약리학과 유전체학의 결합을 반영한다.약제유전체학은 개인의 유전자 구성이 [1]약물에 대한 반응에 어떤 영향을 미치는지 분석한다.유전자 발현이나 단핵 다형성을 약물역학(약물 흡수, 분포, 대사 및 제거) 및 약역학(약물의 생물학적 [2][3][4]표적을 통해 매개되는 효과)과 연관시킴으로써 환자의 약물 반응에 대한 후천적 유전적 변이의 영향을 다룬다.
약제유전체학은 환자의 유전자형과 관련하여 약물치료를 최적화하는 합리적인 방법을 개발하여 부작용을 [5]최소화하면서 최대의 효율성을 보장하는 것을 목표로 한다.약물유전학의 활용을 통해, 의약품 치료법이 소위 말하는 "일률적인" 접근법에서 벗어날 수 있기를 바란다.약리유전체학은 또한 처방 시행착오 방법을 제거하려고 시도하며, 의사가 환자의 유전자, 이러한 유전자의 기능, 그리고 이것이 환자의 현재 또는 미래 치료의 효과에 어떻게 영향을 미칠 수 있는지를 고려하도록 한다(그리고 해당되는 경우, 과거 치료의 실패에 대한 설명을 제공한다).그러한 [6][7]접근법은 정밀 의약품과 심지어 개인화된 의약품의 등장을 약속한다.약물과 약물의 조합은 환자의 좁은 부분 집합이나 개인의 고유한 유전자 [8][9]구성에 최적화된다.
치료에 대한 환자의 반응 또는 그 결여를 설명하는 데 사용하든, 예측 도구로 사용하든, 더 나은 치료 결과, 더 큰 효과, 약물 독성 발생의 최소화 및 약물 부작용(ADR)을 달성하기를 희망한다.치료에 대한 치료 반응이 부족한 환자에게는 그들의 요구에 가장 적합한 대체 치료법이 처방될 수 있다.특정 약물에 대한 약제유전학적 권장사항을 제공하기 위해 유전자형,[10] 엑솜 또는 전체 게놈 염기서열 분석의 두 가지 가능한 입력 유형을 사용할 수 있습니다.시퀀싱은 합성 단백질(조기 정지 코돈)[10]을 조기에 종료하는 돌연변이 검출을 포함하여 더 많은 데이터 포인트를 제공합니다.
역사
약제유전체학은 기원전 510년경에 피타고라스가 용혈성 빈혈과 산화적 스트레스와의 파바 콩 섭취의 위험성을 연관시켰을 때 처음 인식되었다.이 식별은 나중에 검증되었고 1950년대에 G6PD의 결핍에 기인하여 [11][12]favism이라고 불렸다.최초의 공식 출판물은 [13]1961년으로 거슬러 올라가지만, 1950년대 경에 이 과학의 비공식적인 시작을 알렸다.마취 중 숙시닐콜린 주사 투여 후 부틸콜린에스테라아제('pseudocholinesterase')가 결핍된 환자의 장기 마비 및 유전자 변형과 관련된 치명적 반응에 대한 보고는 [2][14]1956년에 처음 보고되었다.약물유전학이라는 용어는 1959년 독일 하이델베르크의 프리드리히 보겔에 의해 처음 만들어졌다.[15]1960년대 후반, 쌍둥이 연구는 일란성 쌍둥이가 이란성 [16]쌍둥이에 비해 약물 반응과 현저한 유사성을 공유하면서 약물 대사에 유전적인 관여의 추론을 뒷받침했다.약물유전학이라는 용어는 1990년대 [11]무렵 처음 등장하기 시작했다.
FDA의 약물유전학적 테스트에 대한 첫 승인은 2005년이었다[9](CYP2D6 및 CYP2C19의 대립 유전자에 대한).
약제유전학과 약제유전학
약제유전체학이라는 용어는 종종 약제유전학과 호환되게 사용된다.비록 두 용어 모두 유전적 영향에 기초한 약물 반응과 관련이 있지만.약제유전학은 유전체학 및 후생유전학을 통합하면서 약물 [6][17][18]반응에 대한 여러 유전자 또는 심지어 염색체의 영향을 다루는 게놈 전체의 연관 접근법을 포함한다.약리유전학은 치료 효과뿐만 아니라 [19]부작용 측면에서 약물에 대한 개별 반응에 영향을 미칠 수 있는 약물 대사 경로(및 효소, 메신저, 수용체 등 다른 약리학적 원리)에서 유전되는 유전적 차이를 연구한다.반면에, 약리유전학은 단일 환자나 집단에서 약물의 더 나은 사용을 이해하기 위해 대립 유전자, 우성 및 유전자 다형성을 카운트하는 단일 약물-유전자 상호작용에 초점을 맞춘다.
약물대사효소
약물의 신진대사와 반응의 변화에 크게 영향을 미치는 몇 가지 알려진 유전자가 있다.이 기사의 초점은 간결함을 위해 임상적으로 더 널리 받아들여지고 사용되는 유전자에 머물 것이다.
- 시토크롬 P450
- VKORC1
- TPMT
시토크롬 P450
가장 일반적인 약물 대사 효소(DME)는 시토크롬 P450(CYP) 효소입니다.이 효소들은 반응성 또는 극성 그룹을 약물과 같은 이생균제에 도입한다.시토크롬 P450이라는 용어는 1962년 오무라(Omura)와 사토(Sato)에 의해 [20]일산화탄소와 복합될 때 450nm 스펙트럼 피크를 특징으로 하는 막 결합 헴 함유 단백질을 설명하기 위해 만들어졌다.인간 CYP 패밀리는 57개의 유전자로 구성되어 있으며, 18개의 패밀리와 44개의 아패밀리로 구성되어 있습니다.CYP 단백질은 아미노산 배열 사이에 식별된 유사성에 기초하여 이들 패밀리와 아패밀리로 편리하게 배열된다.35~40%의 정체성을 공유하는 효소는 아라비아 숫자로 같은 과에 할당되며 55~70%를 공유하는 효소는 지정된 [21]문자로 특정 아군을 구성한다.예를 들어 CYP2D6은 패밀리 2, 서브 패밀리 D 및 유전자 번호 6을 나타냅니다.
임상적 관점에서 가장 일반적으로 테스트되는 CYP는 CYP2D6, CYP2C19, CYP2C9, CYP3A4 및 CYP3A5를 포함한다.이들 유전자는 현재 이용 가능한 처방약물의 [22][23]약 70-90%를 차지한다.아래 표에는 이러한 경로를 사용하는 일부 약물의 요약이 나와 있습니다.
| 주요[24][25] CYP의 약물 대사 | |||||
|---|---|---|---|---|---|
| 효소 | 약물 대사 비율(%) | 약의 예시 | |||
| CYP2C9 | 10 | 톨부타미드, 이부프로펜, 메페남산, 테트라히드로카나비놀, 로사탄, 디클로페낙 | |||
| CYP2C19 | 5 | S-메페니토인, 아미트리프틸린, 디아제팜, 오메프라졸, 프로구아닐, 헥소바르비탈, 프로프라놀롤, 이미프라민 | |||
| CYP2D6 | 20-30 | 데블소킨, 메토프로롤, 스파르테인, 프로프라놀롤, 엔카이나이드, 코데인, 덱스트로메토르판, 클로자핀, 데시프라민, 할로페리돌, 아미트리프틸린, 이미프라민 | |||
| CYP3A4 | 40-45 | 에리트로마이신, 에티닐에스트라디올, 니페디핀, 트리아졸람, 시클로스포린, 아미트리푸틸린, 이미프라민 | |||
| CYP3A5 | 1 미만 | 에리트로마이신, 에티닐에스트라디올, 니페디핀, 트리아졸람, 시클로스포린, 아미트리푸틸린, 알도스테론 | |||
CYP2B6
CYP2B6는 항HIV제 에파비렌즈, 항말라리아 아르테미시닌, 항우울제 부프로피온 및 케타민, 항암제 시클로포스파미드 및 오피오이드 메타돈을 포함한 약물의 [26]대사에 중요한 역할을 한다.이것은 RNA 처리 오류와 효소 수준 저하로 이어지는 변종 CYP2B6*6을 가진 고다형 효소이다.두 번째 중요한 변종 CYP2B6*18도 기능성 단백질을 생성하지 못한다.CYP2B6*6 변종은 전 세계 다양한 집단에서 15~60%의 유병률로 발생하는 반면, CYP2B68*18은 주로 아프리카인에게서 발견된다.에파비렌츠로 치료된 미국과 유럽 환자에 비해 아프리카에서 중추신경계 부작용의 유병률이 높은 것은 사하라 이남 아프리카 [27]인구에서 CYP2B6 느린 대사제 표현형의 빈도가 높기 때문이다.
CYP2D6
파편소킨 하이드록실라아제라고도 알려진 CYP2D6는 가장 잘 알려져 있고 광범위하게 연구되고 있는 CYP [28]유전자이다.다형성이 매우 높고 약물 대사의 수가 많기 때문에(주경로 및 부경로로서) 매우 흥미로운 유전자이기도 하다.100개 이상의 CYP2D6 유전자 변형이 확인되었다.[25]CYP2D6 유전자의 다형성(다른 수준의 대사 활성을 가진 효소 버전으로 유도)과 복사 번호 변이 모두 알려져 있다.CYP2D6에 의해 주로 대사되는 특정 약물의 경우, 이러한 변이는 혈청에서 약물 농도가 비정상적으로 높거나 낮아질 수 있으며(각각 저대사물 및 초대사물 표현형이라고 함) 부작용 증가 또는 효과 감소로 이어질 수 있다.일반적으로 영향을 받는 약으로는 트라마돌, 벤라팍신, 모르핀, 미르타자핀, 메토프로롤 [29]등이 있다.CYP2D6의 빈도는 지리적으로 다양하며, 동아시아에서 발견된 느린 대사제의 유병률은 가장 높고,[30] 아메리카 대륙에서 가장 낮다.
CYP2C19
1980년대 초에 발견된 CYP2C19는 약리유전체학에서 [24]두 번째로 널리 연구되고 잘 알려진 유전자이다.항우울제 및 [31]양성자 펌프 억제제와 [32]같은 여러 등급의 약물의 신진대사에 영향을 미치는 CYP2C19에 대해 28개 이상의 유전자 변형이 확인되었다.
CYP2C9
CYP2C9는 CYP2C 서브패밀리의 대부분을 구성하며, 간 함량의 약 20%를 차지한다.와파린과 톨부타미드와 [32][33]같이 치료창이 좁은 약물을 포함하는 약물의 약 10%의 신진대사에 관여한다.CYP2C9와 [31]관련된 약 57개의 유전자 변형이 있다.
CYP3A4 및 CYP3A5
CYP3A 계열은 간에서 가장 많이 발견되며, CYP3A4는 간 [24]함량의 29%를 차지한다.이러한 효소들은 또한 현재 처방 약물의 40-50%를 차지하며, CYP3A4는 이러한 [12]약물의 40-45%를 차지한다.CYP3A5는 본 [31]출판물 당시 확인된 11개 이상의 유전자 변형을 가지고 있다.
VKORC1
비타민 K 에폭시드 환원효소 복합체 서브유닛 1(VKORC1)은 와파린의 [34]약리역학을 담당합니다.VKORC1은 CYP2C9와 함께 와파린 투여 시 출혈 위험을 확인하는 데 유용합니다.와파린은 VKORC1 유전자에 의해 암호화된 VKOR를 억제함으로써 작용한다.다형성을 가진 사람들은 와파린 [35]치료에 영향을 받는다.
TPMT
티오푸린메틸전달효소(TPMT)는 티오푸린의 S-메틸화를 촉매하여 조혈세포 [36]중 세포독성 티오구아닌뉴클레오티드와 불활성대사물 간의 균형을 조절한다.TPMT는 6MP 대사 및 TSMT 활성에 매우 관여하며 TPMT 유전자형은 독성 위험에 영향을 미치는 것으로 알려져 있습니다.6-MP의 과도한 수치는 골수억제 [37]및 골수독성을 유발할 수 있습니다.관련 특허 소송은 Mayo Collaborative Services 대 Prometheus Laboratories, Inc.에서 일어났으며, 미국 대법원은 이 약의 용량 측정에 관한 특허가 특허 자격이 있다고 판결했다.
코데인, 클로피도그렐, 타목시펜 및 와파린은 상기 대사 경로를 따르는 약물의 몇 가지 예입니다.
약제학 패널
약물유전학적 패널은 약물대사와 관련된 여러 유전자의 변화를 분석 및 검출하기 위해 사용되며, 이러한 패널을 제공하는 회사 중 임상의는 다음과 같이 명명할 수 있습니다.
예측 처방
환자 유전자형은 일반적으로 다음과 같은 예측 표현형으로 분류됩니다.
- UM: 초급속 대사제: 대사 활동이 상당히 증가한 환자;
- 앨런 머스크 : 광범위한 대사제 : 정상적인 대사 활동;
- 중간대사제: 대사활동이 저하된 환자
- PM: 불충분한 대사제: 기능 대사 활동이 거의 없거나 전혀 없는 환자들.
이 스펙트럼의 양 극단은 저조한 대사물과 초급속 대사물이다.약물의 효능은 위의 대사 상태뿐만 아니라 소비되는 약물의 종류에도 기초한다.약물은 크게 두 가지 그룹으로 분류될 수 있다: 활성 약물과 프로드러그.활성 약물은 대사 중에 불활성화된 약물을 말하며, 프로드럭은 대사될 때까지 불활성화된다.

예를 들어, 통증 완화를 위해 코데인을 복용하는 두 명의 환자가 있습니다.코데인은 프로드러그이므로 비활성형에서 활성형으로의 전환이 필요합니다.코데인의 활성 형태는 모르핀으로, 통증 완화의 치료 효과를 제공합니다.A가 CYP2D6 유전자를 코드하기 위해 어머니와 아버지로부터 각각 1개의 *1개의 대립 유전자를 받는 경우, 해당 대립 유전자 *1은 정상적인 기능을 가진 것으로 간주되므로 광범위한 대사제(EM) 표현형을 가진 것으로 간주됩니다(이는 CYP2D6 *1/1로 표시됨).반면 B가 어머니로부터 *1 대립 유전자, 아버지로부터 *4 대립 유전자를 받았다면, 그 개체는 중간 대사제(IM)가 될 것이다(유전자형은 CYP2D6 *1/*4).비록 두 사람이 같은 양의 코데인을 복용하고 있지만, B 사람은 코데인의 활성 상대 모르핀으로의 전환 속도가 감소하기 때문에 잠재적으로 코데인의 치료적 이점이 부족할 수 있다.
각 표현형은 개별 유전자형 내의 대립 유전자 변이에 기초한다.그러나 여러 유전적 사건이 동일한 표현형 특성에 영향을 미칠 수 있으며 유전자형 대 표현형 관계를 확립하는 것은 많은 효소적 패턴과 합의된 것과는 거리가 멀 수 있다.예를 들어, CYP2D6*1/*4 대립 유전자가 타목시펜으로 치료된 환자의 임상 결과에 미치는 영향은 오늘날에도 여전히 논의되고 있다.종양학에서는 5-FU/capcitabine, 이리노테칸, 6-메르캅토푸린 및 젬시타빈/시타라빈의 약동학에 관여하는 DPD, UGT1A1, TPMT, CDA를 코드하는 유전자가 모두 고다형인 것으로 기술되어 있다.이러한 유전적 다형성의 영향을 받는 환자는 약물 섭취 시 심각한/치사적 독성을 경험하게 되며, 사전 치료 검사는 적응 투여 [39]전략을 통해 치료 관련 독성의 위험을 줄이는 데 도움이 된다는 강력한 증거가 있다.
적용들
아래 목록은 일반적으로 알려진 약리유전체학 [40]응용 프로그램을 몇 가지 더 제공합니다.
- 약물 안전성 향상 및 ADR 감소
- 최적의 투여량을 식별하여 환자 고유의 유전자 사전 배치에 부합하도록 치료법을 맞춤화한다.
- 인간 질병을 대상으로 한 약물 발견 개선
- 유효성 시험을 위한 원칙 증명을 개선합니다.
약제유전체학은 통증관리, 심장학, 종양학, 정신의학을 포함한 여러 의학 분야에 적용될 수 있다.법의학 병리학 분야에서도 약제유전체학을 사용하여 [citation needed]부검을 통한 발견이 없는 약물 관련 사망의 사인을 결정할 수 있는 장소가 존재할 수 있다.
암 치료에서 약물유전학 검사는 어떤 환자가 특정 암 약물에 반응할 가능성이 가장 높은지를 확인하기 위해 사용된다.행동 건강에서, 약제 유전학 테스트는 의사와 간병인이 의약품 선택과 부작용 개선을 더 잘 관리할 수 있는 도구를 제공합니다.약제유전체학은 동반 진단으로도 알려져 있는데, 이는 약물과 함께 제공되는 테스트를 의미합니다.예를 들어 cetuximab을 사용한 KRAS 테스트와 gefitinib를 사용한EGFR 테스트가 있습니다.효력 외에도 생식선 약리유전학은 표준 [41]5-FU와 같은 유전적 다형성과 관련하여 해독장애를 보이는 세포독소가 주어졌을 때 심각한 독성을 겪을 가능성이 있는 환자를 식별하는 데 도움을 줄 수 있다.특히 DPD, UGT1A1, TPMT, CDA 및 CYP2D6을 코드하는 유전자에 영향을 미치는 유전자 규제 해제는 현재 5-FU/capecitabine, 이리노테칸, mercopurine/azitioprine, gemcitabine/capecitabine/citara/citabine으로 치료된 환자에게 중요한 문제로 간주되고 있다.
심혈관 질환에서 주요 관심사는 와파린, 클로피도그렐, 베타 차단제, 스타틴을 [10]포함한 약물에 대한 반응이다.클로피도그렐을 복용하는 CYP2C19 환자에서는 심혈관 위험이 증가하여 [43]규제당국에 의한 의약품 패키지 삽입 업데이트로 이어진다.제2형 당뇨병 환자의 경우, 합토글로빈(Hp) 유전자형은 심혈관 질환에 영향을 미치며, Hp2-2는 더 높은 위험성이 있고 보충 비타민 E는 HDL에 영향을 [44]미쳐 위험을 감소시킨다.
2010년 현재 정신의학에서는 특히 5-HTTPR과 DRD2에 [45]초점을 맞추고 있다.
임상적 구현
임상의의 채용을 촉진하기 위한 이니셔티브에는 유럽의 유비쿼터스 약리유전학 프로그램과 미국의 [46]임상약리유전학 실장 컨소시엄(CPIC)이 포함됩니다.2017년 유럽 임상의들을 대상으로 한 조사에서, 전년도의 2/3는 약리학적 [47]검사를 지시하지 않았다.
2010년 Valderbilt 대학 메디컬 센터는 치료 및 치료를 위한 강화된 결정을 위한 약리유전학적 자원(PREDICENT)[48]을 시작했습니다.2015년 조사에서는 임상의 3분의 2가 약리유전학적 테스트를 [49]의뢰했습니다.
미국에서는 FDA가 게놈 [50]증거를 기반으로 의약품 패키지 삽입물을 업데이트했습니다.
2019년 최대 민영 의료 보험사인 United Healthcare는 정신과 약물에 대한 반응을 예측하기 위해 유전자 검사 비용을 지불할 것이라고 발표했다. 2019년 현재, 이 보험사는 그러한 [51]보험을 제공하는 유일한 민영 보험사이다.
2020년 캐나다의 4대 건강 치과 보험사인 그린 실드 캐나다(Green Shield Canada)는 정신 건강 [52]처방을 최적화하고 개인화하기 위한 약리학적 테스트와 관련 임상 의사결정 지원 소프트웨어를 지불할 것이라고 발표했다.
다약성 저감
약물유전학이 할 수 있는 잠재적인 역할은 다약성의 발생을 줄이는 것이다.맞춤형 약물 치료를 통해, 환자들은 같은 상태를 치료하기 위해 여러 가지 약을 복용할 필요가 없을 것이라는 이론이 있다.이를 통해 ADR 발생을 최소화하고 치료 결과를 개선하며 외부 의약품 구입을 피함으로써 비용을 절감할 수 있다.이것의 예는 정신의학에서 발견될 수 있는데, 정신의학에서 환자들은 심지어 나이와 일치하는 정신의학이 아닌 환자들보다 더 많은 약을 받는 경향이 있다.이는 부적절한 [53]처방의 위험 증가와 관련이 있다.
1998년 2월부터 2007년 4월까지 보스턴 대학 슬론 역학 센터가 실시한 조사에서 약물유전학 맞춤형 치료제의 필요성이 가장 뚜렷하게 드러날 수 있다.연구에 따르면 미국 성인의 평균 82%가 최소 한 가지 약물(처방약 또는 비처방약, 비타민/미네랄, 허브/천연 보충제)을 복용하고 있으며 29%는 5가지 이상의 약을 복용하고 있다.이 연구는 65세 이상이 계속해서 가장 많은 약물을 소비하고 있으며, 이 연령대의 17-19%가 일주일에 적어도 10개의 약을 복용하고 있다고 시사했다.다약제도 2000년 이후 23%에서 29%[54]로 증가한 것으로 나타났다.
도입 사례
사례 A – 항정신병 약물 부작용[55]
환자 A는 정신분열증이 있습니다.그들의 치료에는 지프라시돈, 올란자핀, 트라조돈, 벤츠트로핀의 조합이 포함되었다.환자는 어지럼증과 진정제를 경험했고 지프라시돈과 올란자핀을 잘라낸 후 케티아핀으로 전환했다.트라조돈은 단종되었다.그 후, 환자는 과도한 땀, 빈맥, 목 통증을 경험했고, 체중이 상당히 증가했으며 환각을 경험했다.5개월 후 케티아핀은 테이퍼화되어 중단되었으며 과도한 체중 증가로 인해 지프라시돈이 치료에 재도입되었다.환자는 체중이 지나치게 줄었지만 근육 경직, 톱니바퀴, 떨림, 식은땀이 났다.벤츠트로핀이 첨가되었을 때 그들은 시야가 흐릿해졌다.추가 5개월 후, 환자는 지프라시돈에서 아리피프라졸로 전환되었다.8개월 동안 환자 A는 점점 더 많은 체중 증가, 진정, 보행, 경직, 톱니바퀴 운동 및 운동장애를 경험했다.이후 약제유전체학 테스트에서 환자가 IM의 예측 표현형과 IM의 예측 표현형을 가진 CYP2D6 *1/*41을 가지고 있음을 증명했다.
사례 B – 과제 관리 [56]
환자 B는 제왕절개로 출산한 여성입니다.그녀의 주치의는 발작 후 통증에 코데인을 처방했다.그녀는 표준 처방량을 복용했지만 코데인을 복용하는 동안 메스꺼움과 어지럼증을 경험했다.그녀는 또한 그녀의 모유 수유 아기가 무기력하고 제대로 먹지 못한다는 것을 알아챘다.환자가 의사에게 이러한 증상을 말했을 때, 그들은 그녀에게 코데인의 사용을 중단하라고 권고했다.며칠 안에 환자와 아기의 증상은 더 이상 나타나지 않았다.약제유전학 검사를 받았다면 코드인 사용에 대한 ADR을 설명하면서 초급속대사제(UM) 범주에 들어가는 CYP2D6 유전자의 복제가 밝혀졌을 것으로 추정된다.
사례 C – 유아의 코데인 과다 복용에 대한 FDA 경고[57]
2013년 2월 20일, FDA는 CYP2D6 UM으로 알려진 어린이 사이의 연결과 편도선 절제 및/또는 아데노이드 제거 수술 후 코데인에 대한 치명적인 반응에 관한 심각한 우려를 다루는 성명을 발표했다.그들은 코데인을 소비하는 CYP2D6 UM의 위험을 설명하기 위해 가장 강력한 박스형 경고를 발표했습니다.코데인은 CYP2D6에 의해 모르핀으로 전환되며, UM 표현형을 가진 사람은 유전자의 기능 증가로 인해 다량의 모르핀이 생성될 위험이 있다.모르핀은 2012년 8월 세 명의 어린이 사망으로 명백해진 것처럼 생명을 위협하거나 치명적인 양으로 증가할 수 있다.
의약품 라벨링
미국 식품의약국(FDA)은 약제유전체 바이오마커를 [59]라벨에 포함하는 120개 이상의 FDA 승인 약물을 통해 입증되었듯이 약제유전체학[58] 과학에 매우 많은 투자를 하고 있는 것으로 보인다.이 증가량은 [60]해마다 다릅니다.2014년 6월 20일 현재 FDA가 승인한 의약품의 라벨에 대한 연구에 따르면 약제유전체 바이오마커가 [61]라벨에 포함된 140개의 다른 약이 있는 것으로 밝혀졌다.약물은 서로 다른 바이오마커를 가질 수 있기 때문에, 이는 158개의 약물-바이오마커 [61]쌍에 해당한다.
29%만이 유전자 바이오마커 검사에 대한 요구 사항이나 권고 사항을 언급했지만, 이는 종양학 약물의 경우(62%)[61]가 더 높았다.2005년 5월 22일, FDA는 FDA에 제출해야 하는 약제유전체 데이터의 유형과 [62]시기를 명확히 한 첫 번째 산업지침: 약제유전체 데이터 제출을 발표했다.전문가들은 약리유전체학 실험이 부정적인 규제 [63]결과를 초래하지 않을 것이라는 FDA의 인정의 중요성을 인식했다.FDA는 2013년 1월에 최신 가이드 임상 약리유전체학(PGX: Pre-Market Evaluation in Early-Phase Clinical Studies and Recommends for Labeling)을 발표했습니다.이 가이드는 의약품 개발 및 규제 검토 과정에서 게놈 정보의 사용을 다루기 위한 것이다.
과제들
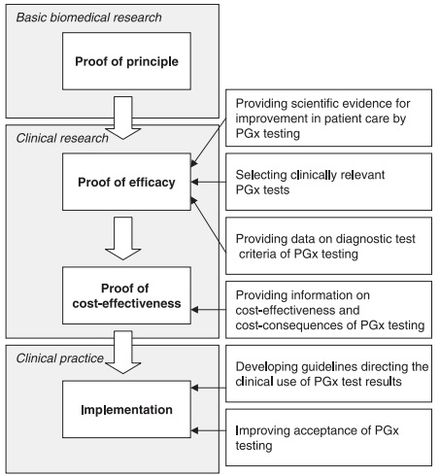
의사와 의료 [65]전문가들 사이에서 약제유전체학의 기본 원칙을 일반적으로 수용하는 것처럼 보이지만, 약제유전체학의 이해, 구현 및 표준화를 늦추는 몇 가지 과제가 존재한다.의사가 제기한 우려 사항은 다음과 같습니다.[18][65][66]
- 임상 실무 및 치료에 테스트를 적용하는 방법에 대한 제한
- 테스트의 가용성 결여에 대한 일반적인 느낌
- 증거 기반 연구의 이해 및 해석
- 처방전 최적화를 위해 검사 결과를 다른 환자 데이터와 결합한다.
- 윤리적, 법적, 사회적 문제
테스트의 가용성과 관련된 문제는 다음과 같습니다.[64]
- 과학적 데이터의 가용성 결여:약물의 대사 경로에는 상당한 수의 DME가 포함되지만, 임상 환경에서 약물의 사용을 검증하기에 충분한 과학적 데이터를 보유한 것은 극히 일부에 불과하다.
- 약제유전체학의 비용효과의 실증: 약제유전체학의 약리경제학에 관한 간행물은 거의 없기 때문에, 이 시점에서, 테스트의 비용효과의 검증과 비용결과의 검증에 충분한 증거가 존재하지 않는다.
약제유전체학(임상사용 가이드라인 개발 등)의 느린 진행에 기여하는 다른 요인도 있지만, 위의 요인이 가장 일반적인 것으로 보인다.약제유전학의 임상적 사용에 대한 실질적인 증거와 산업단체의 가이드라인은 정밀의학에 대한 인구 전반의 접근을 가능하게 했다.비용, 상환, 교육 및 치료 시점에서의 손쉬운 사용은 광범위한 도입에 있어 여전히 큰 장벽으로 남아 있습니다.
논쟁
특정 집단 간에 빈도가 다른 일부 대립 유전자는 특정 약물에 대한 차이 반응과 관련이 있는 것으로 나타났다.베타 차단제인 아테놀롤은 미국의 [citation needed]흑인 환자보다 백인 환자의 혈압을 더 크게 낮추는 것으로 나타난 항고혈압제이다.이러한 관찰은 백인 집단과 아프리카계 미국인 집단이 [67]아테놀롤과 다르게 반응하는 올레산 생화학을 지배하는 다른 대립 유전자를 가지고 있다는 것을 암시합니다.마찬가지로, 항레트로바이러스제 아바카비르에 대한 과민성은 [68]모집단 간에 빈도가 다른 단핵 다형증과 강하게 관련되어 있다.
비록 BiDil의 레이블 어떤 유전적 변이를 밝히지 않은 그 약 BiDil(isosorbide dinitrate/hydralazine)의 레이블에 울혈성 심부전 환자 아프리카계 미국인들을 지정하여는 FDA승인지만 인종 self-identificatio을 바탕으로 한 때 인종 문제가 약과 유전적 stereotyping,[69]에 대한 두려움에 대한 파장의 폭풍을 만들었다.n.[70][71]
미래.
계산의 진보에 의해,[72] 보다 저렴하고 고속의 시퀀싱이 가능하게 되었습니다.연구는 조합 화학,[73] 게놈 마이닝, 오믹 테크놀로지 및 높은 스루풋 스크리닝에 초점을 맞추고 있습니다.
유전자 검사당 비용이 감소함에 따라 맞춤형 약물 치료법의 개발도 [74]증가할 것이다.기술은 이제 약물의 신진대사와 반응에 관여하는 수백 개의 표적 유전자를 1,000달러 이하로 24시간 이내에 유전자 분석할 수 있게 해준다.이것은 매일의 의학적인 결정에 약리학적 기술을 도입하기 위한 큰 진전입니다.마찬가지로 deCODE genetics, MD Labs Pharmacgenetics, Navigenics 및 23andMe와 같은 기업은 게놈 스캔을 제공합니다.이 회사들은 GWAS 연구에 사용된 것과 동일한 유전자형 칩을 사용하여 고객에게 다양한 특성 및 질병에 대한 개별 위험 기록과 50만 개의 알려진 SNP에 대한 테스트를 제공합니다.비용은 $995 ~ $2500이며, 새로운 연구 데이터를 입수할 수 있는 시점에서 업데이트를 포함합니다.더 비싼 패키지에는 결과를 [75]논의하기 위해 유전학 상담사와의 전화 상담도 포함되어 있었다.
윤리
약제유전학은 생명윤리학의 분야에서 논란이 되고 있다.사생활과 기밀유지가 주요 [76]관심사입니다.유전자 테스트의 유익성 또는 위해성의 증거는 암시적일 수 있으며,[76]: 145 이는 공급자에게 딜레마를 야기할 수 있다.약물 개발은 영향을 받을 수 있으며, 희귀한 유전자 변형은 연구를 [76]덜 받을 수 있다.접근과 환자의 자율성 또한 [77]: 680 논의의 여지가 있다.
웹 기반 리소스
| 약리유전체학[78][79] 웹 리소스 | |||||
|---|---|---|---|---|---|
| 데이터 원본 | 주요 용도 | 인용문 | |||
| PharmGKB | 인간 유전자 변이가 약물 반응에 미치는 영향에 관한 지식의 집계, 큐레이션, 통합 및 전파를 담당하는 공개 가능한 온라인 지식 기반. | [80] | |||
| PharmVar | 하플로타입 구조와 대립 유전자 변이에 초점을 맞춘 약 유전자 변이의 중앙 저장소 | [81] | |||
| SuperCYP 바이오 인포매틱스 툴 | 3800개 이상의 상호작용을 가진 1170개의 약물과 약 2000개의 알려진 SNP를 함유하고 있다.이러한 SNP는 표현 및/또는 활동에 미치는 영향에 따라 나열 및 정렬됩니다. | [82] | |||
| PharmGKB | PharmGenomics Knowledge Base(PharmGKB)는 유전자 변이가 약물 반응에 어떻게 영향을 미치는지 조사하는 연구자들을 위한 대화형 도구이다. | [83] | |||
| dbSNP 데이터베이스 | 검출, 컴파일, 정식명칭 후 보고되는 SNP 및 기타 변종 저장소.이것들은 전체적으로 SNP입니다. | [84][85] | |||
| FINDBase | 다양한 모집단의 약리학적 마커 대립 유전자 빈도 저장소 | [86] | |||
| 약물 라벨 표시에 있어서의 약제유전체학 바이오마커 | 약제유전체학 관련 경고 라벨이 부착된 FDA 승인 의약품 식별표 | [87] | |||
| SNPedia | SNP의 Wiki 기반 바이오 인포매틱스 데이터베이스 | [88][89] | |||
| 약리유전체학연구네트워크(PGRN) | PGRN은 약제유전체학 및 정밀의학 분야의 공동 연구를 촉진하기 위한 자원과 정보를 제공한다. | [90] | |||
「 」를 참조해 주세요.
레퍼런스
- ^ Ermak G (2015). Emerging Medical Technologies. World Scientific. ISBN 978-981-4675-80-2.
- ^ a b Johnson JA (November 2003). "Pharmacogenetics: potential for individualized drug therapy through genetics". Trends in Genetics. 19 (11): 660–6. doi:10.1016/j.tig.2003.09.008. PMID 14585618.
- ^ "Center for Pharmacogenomics and Individualized Therapy". Unc Eshelman School of Pharmacy. Retrieved 2014-06-25.
- ^ "overview of pharmacogenomics". Up-to-Date. May 16, 2014. Retrieved 2014-06-25.
- ^ Becquemont L (June 2009). "Pharmacogenomics of adverse drug reactions: practical applications and perspectives". Pharmacogenomics. 10 (6): 961–9. doi:10.2217/pgs.09.37. PMID 19530963.
- ^ a b Sheffield LJ, Phillimore HE (May 2009). "Clinical use of pharmacogenomic tests in 2009". The Clinical Biochemist. Reviews. 30 (2): 55–65. PMC 2702214. PMID 19565025.
- ^ Hauser AS, Chavali S, Masuho I, Jahn LJ, Martemyanov KA, Gloriam DE, Babu MM (January 2018). "Pharmacogenomics of GPCR Drug Targets". Cell. 172 (1–2): 41–54.e19. doi:10.1016/j.cell.2017.11.033. PMC 5766829. PMID 29249361.
- ^ "Guidance for Industry Pharmacogenomic Data Submissions" (PDF). U.S. Food and Drug Administration. March 2005. Retrieved 2008-08-27.
- ^ a b Squassina A, Manchia M, Manolopoulos VG, Artac M, Lappa-Manakou C, Karkabouna S, et al. (August 2010). "Realities and expectations of pharmacogenomics and personalized medicine: impact of translating genetic knowledge into clinical practice". Pharmacogenomics. 11 (8): 1149–67. doi:10.2217/pgs.10.97. PMID 20712531.
- ^ a b c Huser V, Cimino JJ (2013). "Providing pharmacogenomics clinical decision support using whole genome sequencing data as input". AMIA Joint Summits on Translational Science Proceedings. AMIA Joint Summits on Translational Science. 2013: 81. PMID 24303303.
- ^ a b Pirmohamed M (October 2001). "Pharmacogenetics and pharmacogenomics". British Journal of Clinical Pharmacology. 52 (4): 345–7. doi:10.1046/j.0306-5251.2001.01498.x. PMC 2014592. PMID 11678777.
- ^ a b Prasad K (January 2009). "Role of regulatory agencies in translating pharmacogenetics to the clinics". Clinical Cases in Mineral and Bone Metabolism. 6 (1): 29–34. PMC 2781218. PMID 22461095.
- ^ Evans DA, Clarke CA (September 1961). "Pharmacogenetics". British Medical Bulletin. 17 (3): 234–40. doi:10.1093/oxfordjournals.bmb.a069915. PMID 13697554.
- ^ Kalow W (2006). "Pharmacogenetics and pharmacogenomics: origin, status, and the hope for personalized medicine". The Pharmacogenomics Journal. 6 (3): 162–5. doi:10.1038/sj.tpj.6500361. PMID 16415920.
- ^ Vogel F (1959). "Moderne probleme der humangenetik" [Modern human genetics problems]. Ergebnisse der Inneren Medizin und Kinderheilkunde [Results of internal medicine and pediatrics] (in German). Berlin, Heidelberg: Springer: 52–125.
- ^ Motulsky AG, Qi M (February 2006). "Pharmacogenetics, pharmacogenomics and ecogenetics". Journal of Zhejiang University. Science. B. 7 (2): 169–70. doi:10.1631/jzus.2006.B0169. PMC 1363768. PMID 16421980.
- ^ Shin J, Kayser SR, Langaee TY (April 2009). "Pharmacogenetics: from discovery to patient care". American Journal of Health-System Pharmacy. 66 (7): 625–37. doi:10.2146/ajhp080170. PMID 19299369.
- ^ a b "Center for Genetics Education".[영구 데드링크]
- ^ Klotz U (2007). "The role of pharmacogenetics in the metabolism of antiepileptic drugs: pharmacokinetic and therapeutic implications". Clinical Pharmacokinetics. 46 (4): 271–279. doi:10.2165/00003088-200746040-00001. PMID 17375979. S2CID 30702170.
- ^ Debose-Boyd RA (February 2007). "A helping hand for cytochrome p450 enzymes". Cell Metabolism. 5 (2): 81–3. doi:10.1016/j.cmet.2007.01.007. PMID 17276348.
- ^ Nebert DW, Russell DW (October 2002). "Clinical importance of the cytochromes P450". Lancet. 360 (9340): 1155–62. doi:10.1016/s0140-6736(02)11203-7. PMID 12387968. S2CID 13577054.
- ^ Hart SN, Wang S, Nakamoto K, Wesselman C, Li Y, Zhong XB (January 2008). "Genetic polymorphisms in cytochrome P450 oxidoreductase influence microsomal P450-catalyzed drug metabolism". Pharmacogenetics and Genomics. 18 (1): 11–24. doi:10.1097/FPC.0b013e3282f2f121. PMID 18216718. S2CID 38880360.
- ^ Gomes AM, Winter S, Klein K, Turpeinen M, Schaeffeler E, Schwab M, Zanger UM (April 2009). "Pharmacogenomics of human liver cytochrome P450 oxidoreductase: multifactorial analysis and impact on microsomal drug oxidation". Pharmacogenomics. 10 (4): 579–99. doi:10.2217/pgs.09.7. PMID 19374516.
- ^ a b c Hasler JA (February 1999). "Pharmacogenetics of cytochromes P450". Molecular Aspects of Medicine. 20 (1–2): 12–24, 25–137. doi:10.1016/s0098-2997(99)00005-9. PMID 10575648.
- ^ a b Ingelman-Sundberg M (April 2004). "Pharmacogenetics of cytochrome P450 and its applications in drug therapy: the past, present and future". Trends in Pharmacological Sciences. 25 (4): 193–200. doi:10.1016/j.tips.2004.02.007. PMID 15063083.
- ^ Zanger UM, Klein K (2013). "Pharmacogenetics of cytochrome P450 2B6 (CYP2B6): advances on polymorphisms, mechanisms, and clinical relevance". Frontiers in Genetics. 4: 24. doi:10.3389/fgene.2013.00024. PMC 3588594. PMID 23467454.
- ^ Masimirembwa C, Dandara C, Leutscher PD (October 2016). "Rolling out Efavirenz for HIV Precision Medicine in Africa: Are We Ready for Pharmacovigilance and Tackling Neuropsychiatric Adverse Effects?". Omics. 20 (10): 575–580. doi:10.1089/omi.2016.0120. PMID 27627692.
- ^ Badyal DK, Dadhich AP (October 2001). "Cytochrome P450 and drug interactions" (PDF). Indian Journal of Pharmacology. 33: 248–259.
- ^ Zhou SF (2009). "Polymorphism of human cytochrome P450 2D6 and its clinical significance: Part I". Clinical Pharmacokinetics. 48 (11): 689–723. doi:10.2165/11318030-000000000-00000. PMID 19817501. S2CID 5936561.
- ^ Zhou Y, Ingelman-Sundberg M, Lauschke VM (October 2017). "Worldwide Distribution of Cytochrome P450 Alleles: A Meta-analysis of Population-scale Sequencing Projects". Clinical Pharmacology and Therapeutics. 102 (4): 688–700. doi:10.1002/cpt.690. PMC 5600063. PMID 28378927.
- ^ a b c Ingelman-Sundberg M, Nebert DW, Sim SC. "The Human Cytochrome P450 (CYP) Allele Nomenclature Database". Retrieved 2014-09-03.
- ^ a b Ingelman-Sundberg M, Sim SC, Gomez A, Rodriguez-Antona C (December 2007). "Influence of cytochrome P450 polymorphisms on drug therapies: pharmacogenetic, pharmacoepigenetic and clinical aspects". Pharmacology & Therapeutics. 116 (3): 496–526. doi:10.1016/j.pharmthera.2007.09.004. PMID 18001838.
- ^ Sikka R, Magauran B, Ulrich A, Shannon M (December 2005). "Bench to bedside: Pharmacogenomics, adverse drug interactions, and the cytochrome P450 system". Academic Emergency Medicine. 12 (12): 1227–35. doi:10.1111/j.1553-2712.2005.tb01503.x. PMID 16282513.
- ^ Teh LK, Langmia IM, Fazleen Haslinda MH, Ngow HA, Roziah MJ, Harun R, et al. (April 2012). "Clinical relevance of VKORC1 (G-1639A and C1173T) and CYP2C9*3 among patients on warfarin". Journal of Clinical Pharmacy and Therapeutics. 37 (2): 232–6. doi:10.1111/j.1365-2710.2011.01262.x. PMID 21507031. S2CID 22671298.
- ^ U.S. Food and Drug Administration (FDA). "Table of Pharmacogenomic Biomarkers in Drug Labels". Food and Drug Administration. Retrieved 2014-09-03.
- ^ a b Crews KR, Hicks JK, Pui CH, Relling MV, Evans WE (October 2012). "Pharmacogenomics and individualized medicine: translating science into practice". Clinical Pharmacology and Therapeutics. 92 (4): 467–75. doi:10.1038/clpt.2012.120. PMC 3589526. PMID 22948889.
- ^ Sim SC, Kacevska M, Ingelman-Sundberg M (February 2013). "Pharmacogenomics of drug-metabolizing enzymes: a recent update on clinical implications and endogenous effects". The Pharmacogenomics Journal. 13 (1): 1–11. doi:10.1038/tpj.2012.45. PMID 23089672.
- ^ Matey ET, Ragan AK, Oyen LJ, Vitek CR, Aoudia SL, Ragab AK, et al. (February 2022). "Nine-gene pharmacogenomics profile service: The Mayo Clinic experience". The Pharmacogenomics Journal. 22 (1): 69–74. doi:10.1038/s41397-021-00258-0. PMID 34671112. S2CID 239050972.
- ^ Lee SY, McLeod HL (January 2011). "Pharmacogenetic tests in cancer chemotherapy: what physicians should know for clinical application". The Journal of Pathology. 223 (1): 15–27. doi:10.1002/path.2766. PMID 20818641. S2CID 6330198.
- ^ Cohen N (November 2008). Pharmacogenomics and Personalized Medicine (Methods in Pharmacology and Toxicology). Totowa, NJ: Humana Press. p. 6. ISBN 978-1934115046.
- ^ Ciccolini J, Gross E, Dahan L, Lacarelle B, Mercier C (October 2010). "Routine dihydropyrimidine dehydrogenase testing for anticipating 5-fluorouracil-related severe toxicities: hype or hope?". Clinical Colorectal Cancer. 9 (4): 224–8. doi:10.3816/CCC.2010.n.033. PMID 20920994.
- ^ Yang CG, Ciccolini J, Blesius A, Dahan L, Bagarry-Liegey D, Brunet C, et al. (January 2011). "DPD-based adaptive dosing of 5-FU in patients with head and neck cancer: impact on treatment efficacy and toxicity". Cancer Chemotherapy and Pharmacology. 67 (1): 49–56. doi:10.1007/s00280-010-1282-4. PMID 20204365. S2CID 25362813.
- ^ Dean L (2012). "Clopidogrel Therapy and CYP2C19 Genotype". In Pratt VM, McLeod HL, Rubinstein WS, et al. (eds.). Medical Genetics Summaries. National Center for Biotechnology Information (NCBI). PMID 28520346. Bookshelf ID: NBK84114.
- ^ Bale BF, Doneen AL, Vigerust DJ (2018). "Precision Healthcare of Type 2 Diabetic Patients Through Implementation of Haptoglobin Genotyping". Frontiers in Cardiovascular Medicine. 5: 141. doi:10.3389/fcvm.2018.00141. PMC 6198642. PMID 30386783.
- ^ Malhotra AK (2010). "The state of pharmacogenetics". Psychiatr Times. 27 (4): 38–41, 62.
- ^ Williams MS (August 2019). "Early Lessons from the Implementation of Genomic Medicine Programs". Annual Review of Genomics and Human Genetics. 20 (1): 389–411. doi:10.1146/annurev-genom-083118-014924. PMID 30811224. S2CID 73460688.
- ^ Just KS, Steffens M, Swen JJ, Patrinos GP, Guchelaar HJ, Stingl JC (October 2017). "Medical education in pharmacogenomics-results from a survey on pharmacogenetic knowledge in healthcare professionals within the European pharmacogenomics clinical implementation project Ubiquitous Pharmacogenomics (U-PGx)". European Journal of Clinical Pharmacology. 73 (10): 1247–1252. doi:10.1007/s00228-017-2292-5. PMC 5599468. PMID 28669097.
- ^ Carlson B (2012). "Vanderbilt pioneers bedside genetics". Biotechnology Healthcare. 9 (2): 31–2. PMC 3411230. PMID 22876213.
- ^ Peterson JF, Field JR, Shi Y, Schildcrout JS, Denny JC, McGregor TL, et al. (August 2016). "Attitudes of clinicians following large-scale pharmacogenomics implementation". The Pharmacogenomics Journal. 16 (4): 393–8. doi:10.1038/tpj.2015.57. PMC 4751074. PMID 26261062.
- ^ Drozda K, Pacanowski MA, Grimstein C, Zineh I (August 2018). "Pharmacogenetic Labeling of FDA-Approved Drugs: A Regulatory Retrospective". JACC. Basic to Translational Science. 3 (4): 545–549. doi:10.1016/j.jacbts.2018.06.001. PMC 6115648. PMID 30175278.
- ^ "Pharmacogenetic test makers cheer UnitedHealth coverage. Other payers aren't there yet". MedTech Dive. Retrieved 2019-12-29.
- ^ "Personalized drug treatment through pharmacogenetics".
- ^ Ritsner M (2013). Polypharmacy in Psychiatry Practice, Volume I. Multiple Medication Strategies. Dordrecht: Springer Science and Business Media. ISBN 978-94-007-5804-9.
- ^ "Patterns of Medication Use in the United States". Boston University, Slone Epidemiology Center. 2006.
- ^ Foster A, Wang Z, Usman M, Stirewalt E, Buckley P (December 2007). "Pharmacogenetics of antipsychotic adverse effects: Case studies and a literature review for clinicians". Neuropsychiatric Disease and Treatment. 3 (6): 965–73. doi:10.2147/ndt.s1752. PMC 2656342. PMID 19300635.
- ^ "Pharmacogenetics: increasing the safety and effectiveness of drug therapy [Brochure]" (PDF). American Medical Association. 2011.
- ^ "FDA Drug Safety Communication: Safety review update of codeine use in children; new Boxed Warning and Contraindication on use after tonsillectomy and/or adenoidectomy". United States Food and Drug Administration. 2013-02-20.
- ^ "Pharmacogenetics and Pharmacogenomics: State-of-the-art and potential socio-economic impacts in the EU". European Commission, Joint Research Centre, Institute for Prospective Technological Studies. 2006-04-01.
- ^ "Table of Pharmacogenomic Biomarkers in Drug Labels". United States Food and Drug Administration. 2013-06-19.
- ^ Abrahams E, Silver M (July 2009). "The case for personalized medicine". Journal of Diabetes Science and Technology. 3 (4): 680–4. doi:10.1177/193229680900300411. PMC 2769975. PMID 20144313.
- ^ a b c Vivot A, Boutron I, Ravaud P, Porcher R (September 2015). "Guidance for pharmacogenomic biomarker testing in labels of FDA-approved drugs". Genetics in Medicine. 17 (9): 733–8. doi:10.1038/gim.2014.181. PMID 25521333.
- ^ Xie HG, Frueh FW (November 2005). "Pharmacogenomics steps toward personalized medicine". Personalized Medicine. 2 (4): 325–337. doi:10.2217/17410541.2.4.325. PMID 29788578.
- ^ Katsnelson A (May 2005). "Cautious welcome for FDA pharmacogenomics guidance". Nature Biotechnology. 23 (5): 510. doi:10.1038/nbt0505-510. PMID 15877053. S2CID 28473989.
- ^ a b Swen JJ, Huizinga TW, Gelderblom H, de Vries EG, Assendelft WJ, Kirchheiner J, Guchelaar HJ (August 2007). "Translating pharmacogenomics: challenges on the road to the clinic". PLOS Medicine. 4 (8): e209. doi:10.1371/journal.pmed.0040209. PMC 1945038. PMID 17696640.
- ^ a b Stanek EJ, Sanders CL, Taber KA, Khalid M, Patel A, Verbrugge RR, et al. (March 2012). "Adoption of pharmacogenomic testing by US physicians: results of a nationwide survey". Clinical Pharmacology and Therapeutics. 91 (3): 450–8. doi:10.1038/clpt.2011.306. PMID 22278335. S2CID 21366195.
- ^ Ma JD, Lee KC, Kuo GM (August 2012). "Clinical application of pharmacogenomics". Journal of Pharmacy Practice. 25 (4): 417–27. doi:10.1177/0897190012448309. PMID 22689709. S2CID 1212666.
- ^ Wikoff WR, Frye RF, Zhu H, Gong Y, Boyle S, Churchill E, et al. (2013). "Pharmacometabolomics reveals racial differences in response to atenolol treatment". PLOS ONE. 8 (3): e57639. Bibcode:2013PLoSO...857639W. doi:10.1371/journal.pone.0057639. PMC 3594230. PMID 23536766.
- ^ Rotimi CN, Jorde LB (October 2010). "Ancestry and disease in the age of genomic medicine". The New England Journal of Medicine. 363 (16): 1551–8. doi:10.1056/NEJMra0911564. PMID 20942671. S2CID 21293807.
- ^ Bloche MG (November 2004). "Race-based therapeutics". The New England Journal of Medicine. 351 (20): 2035–7. doi:10.1056/NEJMp048271. PMID 15533852. S2CID 1467851.
- ^ Frank R (March 30 – April 1, 2006). "Back with a Vengeance: the Reemergence of a Biological Conceptualization of Race in Research on Race/Ethnic Disparities in Health". Annual Meeting of the Population Association of America. Los Angeles, California. Archived from the original on December 1, 2008. Retrieved 2008-11-20.
- ^ Crawley L (July 2007). "The paradox of race in the Bidil debate". Journal of the National Medical Association. 99 (7): 821–2. PMC 2574363. PMID 17668653.
- ^ Kalow W (2005). Pharmacogenomics. New York: Taylor & Francis. pp. 552–3. ISBN 978-1-57444-878-8.
- ^ Thorpe DS (2001). "Combinatorial chemistry: starting the second decade". The Pharmacogenomics Journal. 1 (4): 229–32. doi:10.1038/sj.tpj.6500045. PMID 11908762.
- ^ Paul NW, Fangerau H (December 2006). "Why should we bother? Ethical and social issues in individualized medicine". Current Drug Targets. 7 (12): 1721–7. doi:10.2174/138945006779025428. PMID 17168846.
- ^ Topol E (2012). The Creative Destruction of Medicine: How the Digital Revolution Will Create Better Health Care. New York: Basic Books. ISBN 978-0-465-02550-3.
- ^ a b c Corrigan OP (March 2005). "Pharmacogenetics, ethical issues: review of the Nuffield Council on Bioethics Report". Journal of Medical Ethics. 31 (3): 144–8. doi:10.1136/jme.2004.007229. PMC 1734105. PMID 15738433.
- ^ Breckenridge A, Lindpaintner K, Lipton P, McLeod H, Rothstein M, Wallace H (September 2004). "Pharmacogenetics: ethical problems and solutions". Nature Reviews. Genetics. 5 (9): 676–80. doi:10.1038/nrg1431. PMID 15372090. S2CID 6149591.
- ^ Barh D, Dhawan D, Ganguly NK (2013). Barh D, Dhawan D, Ganguly NK (eds.). Omics for Personalized Medicine. India: Springer Media. doi:10.1007/978-81-322-1184-6. ISBN 978-81-322-1183-9. S2CID 46120003.
- ^ Stram D (2014). "Post-GWAS Analyses". Design, Analysis, and Interpretation of Genome-Wide Association Scans. Statistics for Biology and Health. Los Angeles: Springer Science and Business Media. pp. 285–327. doi:10.1007/978-1-4614-9443-0_8. ISBN 978-1-4614-9442-3.
- ^ "PharmGKB".
- ^ Gaedigk A, Ingelman-Sundberg M, Miller NA, Leeder JS, Whirl-Carrillo M, Klein TE (March 2018). "The Pharmacogene Variation (PharmVar) Consortium: Incorporation of the Human Cytochrome P450 (CYP) Allele Nomenclature Database". Clinical Pharmacology and Therapeutics. 103 (3): 399–401. doi:10.1002/cpt.910. PMC 5836850. PMID 29134625.
- ^ Preissner S, Kroll K, Dunkel M, Senger C, Goldsobel G, Kuzman D, et al. (January 2010). "SuperCYP: a comprehensive database on Cytochrome P450 enzymes including a tool for analysis of CYP-drug interactions". Nucleic Acids Research. 38 (Database issue): D237-43. doi:10.1093/nar/gkp970. PMC 2808967. PMID 19934256.
- ^ Thorn CF, Klein TE, Altman RB (2013). "PharmGKB: the Pharmacogenomics Knowledge Base". Pharmacogenomics. Methods in Molecular Biology. Vol. 1015. Clifton, N.J. pp. 311–20. doi:10.1007/978-1-62703-435-7_20. ISBN 978-1-62703-434-0. PMC 4084821. PMID 23824865.
- ^ "DBSNP Home Page". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ Smigielski EM, Sirotkin K, Ward M, Sherry ST (January 2000). "dbSNP: a database of single nucleotide polymorphisms". Nucleic Acids Research. 28 (1): 352–5. doi:10.1093/nar/28.1.352. PMC 102496. PMID 10592272.
- ^ Papadopoulos P, Viennas E, Gkantouna V, Pavlidis C, Bartsakoulia M, Ioannou ZM, et al. (January 2014). "Developments in FINDbase worldwide database for clinically relevant genomic variation allele frequencies". Nucleic Acids Research. 42 (Database issue): D1020-6. doi:10.1093/nar/gkt1125. PMC 3964978. PMID 24234438.
- ^ "Table of Pharmacogenomic Biomarkers in Drug Labeling". FDA. 18 August 2021.
- ^ "SNPedia".
- ^ Cariaso M, Lennon G (January 2012). "SNPedia: a wiki supporting personal genome annotation, interpretation and analysis". Nucleic Acids Research. 40 (Database issue): D1308–12. doi:10.1093/nar/gkr798. PMC 3245045. PMID 22140107.
- ^ "Pharmacogenomics Research Network PGRN".
추가 정보
- Katsnelson A (August 2005). "A Drug to Call One's Own: Will medicine finally get personal?". Scientific American.
- Karczewski KJ, Daneshjou R, Altman RB (2012). "Chapter 7: Pharmacogenomics". PLOS Computational Biology. 8 (12): e1002817. Bibcode:2012PLSCB...8E2817K. doi:10.1371/journal.pcbi.1002817. PMC 3531317. PMID 23300409.
외부 링크
- "Pharmacogenomics Factsheet". National Center for Biotechnology Information (NCBI), U.S. National Library of Medicine. Retrieved 2011-07-11.
a quick introduction to customised drugs
- "Pharmacogenomics Education Initiatives". U.S. Food and Drug Administration. 2010-09-24. Retrieved 2011-07-11.
- "Personalized Medicine (Pharmacogenetics)". University of Utah's Genetic Science Learning Center. Archived from the original on 2011-05-19. Retrieved 2011-07-11.
- "Center for Pharmacogenomics and Individualized Therapy". University of North Carolina at Chapel Hill Center for Pharmacogenomics and Individualized Therapy. Archived from the original on 2014-08-06. Retrieved 2014-06-25.
저널:
- "Pharmacogenomics". Future Medicine Ltd.
- "Pharmacogenetics and Genomics". Lippincott Williams & Wilkins. ISSN 1744-6872.
- "The Pharmacogenomics Journal". The Pharmacogenomics Journal. Nature Publishing Group. ISSN 1470-269X.


