계통수
Phylogenetic tree| 시리즈의 일부 |
| 진화생물학 |
|---|
 |

계통수(phylogenetic tree, 계통수 또는 진화수)는 다양한 생물학적 종이나 다른 개체 간의 진화적 관계를 나타내는 분기도 또는 나무이다.지구상의 모든 생명체는 공통의 조상을 나타내는 단일 계통수의 일부이다.
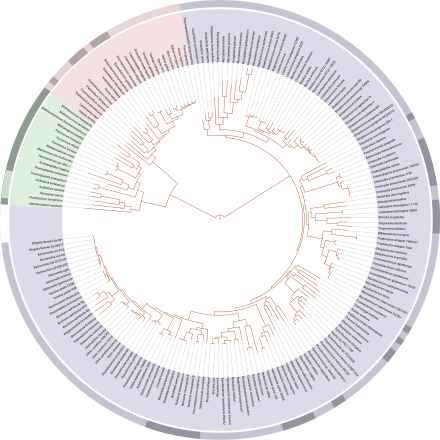
뿌리 계통수에서 후손을 가진 각 노드는 이들 [citation needed]후손의 추정된 가장 최근의 공통 조상을 나타내며, 일부 나무의 가장자리 길이는 시간 추정치로 해석될 수 있다.각 노드를 분류 단위라고 합니다.내부 노드는 직접 관찰할 수 없기 때문에 일반적으로 가상의 분류 단위라고 불립니다.나무는 생물정보학, 계통학, 계통학 그리고 계통학 같은 생물학 분야에서 유용하다.뿌리가 없는 나무는 잎 노드의 관련성만을 나타내며, 조상들의 뿌리를 알거나 추론할 필요가 없습니다.
역사
"생명의 나무"에 대한 생각은 사다리처럼 낮은 곳에서 더 높은 형태의 삶으로 나아간다는 고대 개념에서 비롯되었습니다."가지치기" 계통수의 초기 표현은 Edward Hitchcock의 저서 Elementary Geology (초판: 1840)에 식물과 동물 사이의 지질학적 관계를 보여주는 "고대학 차트"를 포함합니다.
찰스 다윈은 또한 최초의 삽화들 중 하나를 만들었고 그의 정설 책 종의 기원에서 진화적인 "나무"의 개념을 결정적으로 대중화했다.한 세기가 지난 지금도 진화생물학자들은 진화를 묘사하기 위해 나무 도표를 사용한다. 왜냐하면 그러한 도표는 계통들의 적응적 분할과 반난수적 분할을 통해 분화가 일어난다는 개념을 효과적으로 전달하기 때문이다.시간이 지남에 따라 종 분류는 덜 정적이고 더 역동적이 되었다.
The term phylogenetic, or phylogeny, derives from the two ancient greek words φῦλον (phûlon), meaning "race, lineage", and γένεσις (génesis), meaning "origin, source".[4][5]
특성.
루트 트리

루트 계통수(위의 2개의 그래픽 참조)는 고유 노드(루트)를 가진 방향성 트리입니다.이 노드는 트리의 잎에 있는 모든 엔티티의 최신 공통 조상(통상 귀속)에 대응합니다.루트 노드에는 부모 노드가 없지만 트리 내의 다른 모든 노드의 부모 역할을 합니다.따라서 루트는 degree 2 노드이지만 다른 내부 노드는 최소 degree 3입니다(여기서 "degree"는 착신 및 발신 에지의 총수를 나타냅니다).
나무를 뿌리는 가장 일반적인 방법은 논란의 여지가 없는 아웃그룹을 사용하는 것이다. 즉, 특성 데이터나 분자 시퀀싱에서 추론할 수 있을 만큼 가깝지만, 명확한 아웃그룹으로 만들 수 있을 만큼 충분히 멀리 떨어져 있다.또 다른 방법은 중간점 루팅 또는 비정상 대체 [6]모델을 사용하여 트리를 루팅할 수도 있습니다.
뿌리 없는 나무
뿌리가 없는 나무는 조상에 대한 가정을 하지 않고 잎절의 연관성을 보여준다.그들은 조상들의 뿌리를 알거나 [8]추론할 필요가 없다.뿌리가 없는 나무는 뿌리가 빠지는 것만으로 뿌리에서 항상 생성될 수 있다.반대로, 뿌리가 없는 나무의 뿌리를 추론하는 것은 조상을 식별하는 수단을 필요로 한다.이것은 보통 입력 데이터에 아웃그룹을 포함하여 루트가 반드시 아웃그룹과 트리 내의 나머지 분류군 사이에 오도록 하거나 분자 시계 [9]가설의 적용과 같은 각 분기의 상대적 진화 속도에 대한 추가 가정을 도입함으로써 이루어진다.
분압 대 멀티분압
뿌리와 뿌리가 없는 트리는 둘 다 분기 또는 다중 분기 중 하나입니다.루트 분기 트리는 각 내부 노드(즉, 바이너리 트리를 형성함)에서 발생하는 정확히 2개의 후손을 가지며, 루트가 없는 분기 트리는 루트되지 않은 바이너리 트리, 즉 각 내부 노드에 정확히 3개의 네이버가 있는 자유 트리 형태를 취한다.이와는 대조적으로 루트 멀티퍼레이터 트리는 일부 노드에서 3개 이상의 자식을 가질 수 있으며, 루트되지 않은 멀티퍼레이터 트리는 일부 노드에서 3개 이상의 네이버를 가질 수 있습니다.
라벨 부착 대 라벨 부착 안 함
뿌리와 뿌리가 없는 트리는 둘 다 라벨을 붙이거나 라벨을 붙이지 않을 수 있습니다.레이블이 지정된 트리는 해당 리프에 특정 값이 할당되지만 레이블이 지정되지 않은 트리(트리 모양이라고도 함)는 토폴로지만 정의합니다.Phylotree와 [10]같은 작은 게놈 궤적에서 만들어진 일부 배열 기반 나무들은 추정된 조상 하플로타입으로 표시된 내부 노드를 특징으로 합니다.
트리 열거

특정 리프 노드의 수에 대해 가능한 트리의 수는 특정 유형의 트리에 따라 다르지만, 항상 라벨이 붙어 있지 않은 트리보다 라벨이 붙어 있고, 분기하는 트리보다 멀티퍼시티가 많고, 뿌리가 없는 트리보다 뿌리가 깊습니다.마지막 구별은 생물학적으로 가장 관련이 있다; 그것은 뿌리를 내릴 수 있는 뿌리가 없는 나무 위에 많은 장소가 있기 때문에 발생한다.레이블이 지정된 트리를 분기하는 경우 루트 트리의 총 수는 다음과 같습니다.
- 은는) n 2({style n2에 대해 n({displaystyle n }은 [11]리프 노드의 수를 나타냅니다.
라벨이 붙은 트리를 분기하는 경우 루트되지 않은 트리의 총 수는 다음과 같습니다.[11]
- ( 의 경우) (\n\3 ) 。
라벨이 붙은 분기수 중 잎을 뿌리없는 나무의 수는 n-의 [3]을 가진 뿌리깊은 나무의 수와 같다
뿌리나무의 수는 팁의 수에 따라 빠르게 증가한다.10가지 팁에 대해 × 6 ({ 스타일 34\6}) 의 분기 트리가 있으며, 멀티퍼팅 트리의 수는 전자의 약 7배 정도로 더 빠르게 증가하고 있습니다.
| 라벨 부착 나뭇잎들 | 바이너리 뿌리째 뽑히지 않은 나무 | 바이너리 뿌리가 있는 나무 | 멀티퍼레이팅 뿌리가 있는 나무 | 가능한 모든 것 뿌리가 있는 나무 |
|---|---|---|---|---|
| 1 | 1 | 1 | 0 | 1 |
| 2 | 1 | 1 | 0 | 1 |
| 3 | 1 | 3 | 1 | 4 |
| 4 | 3 | 15 | 11 | 26 |
| 5 | 15 | 105 | 131 | 236 |
| 6 | 105 | 945 | 1,807 | 2,752 |
| 7 | 945 | 10,395 | 28,813 | 39,208 |
| 8 | 10,395 | 135,135 | 524,897 | 660,032 |
| 9 | 135,135 | 2,027,025 | 10,791,887 | 12,818,912 |
| 10 | 2,027,025 | 34,459,425 | 247,678,399 | 282,137,824 |
특수 트리 유형
덴드로그램
덴드로그램은 계통 발생 여부에 관계없이 나무의 총칭이며, 따라서 계통 [12]발생 나무의 도식적 표현도 포함한다.
분해도
분기도는 분기 패턴만을 나타냅니다.즉, 분기도는 문자 변화의 시간이나 상대적인 양을 나타내지 않으며 내부 노드는 [13]조상을 나타내지 않습니다.
필로그램
계통도는 성격 [15]변화의 양에 비례하는 가지 길이를 가진 계통수입니다.
크로노그램은 분기 [16]길이를 통해 시간을 명시적으로 나타내는 계통수입니다.
달그레노그램
달그레노그램은 계통수의 단면을 나타내는 다이어그램이다.
계통망
계통발생 네트워크는 엄밀하게 말하면 트리가 아니라 보다 일반적인 그래프 또는 루트 네트워크의 경우 방향 비순환 그래프입니다.그것들은 나무에 내재된 몇 가지 한계를 극복하기 위해 사용된다.
스핀들 다이어그램
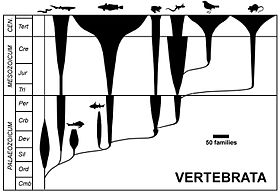
스핀들 다이어그램 또는 버블 다이어그램은 미국의 고생물학자 알프레드 로머에 [17]의해 대중화된 이후 로메로그램으로 종종 불린다.다양한 분류군의 시간적 풍요의 변화를 반영하기 위해 지질학적 시간(수직 축)에 대한 분류학적 다양성(수평 폭)을 나타낸다.그러나 스핀들 다이어그램은 진화적인 [18]트리가 아니다: 분류학적 스핀들은 딸 분류군에 대한 부모[17] 분류군의 실제 관계를 모호하게 하고 부모 [19]그룹의 병언과 관련된 단점을 가지고 있다.이 유형의 다이어그램은 원래 제안된 [19]형식에서 더 이상 사용되지 않습니다.
생명의 산호
다윈은[20] 또한 산호가 나무보다 더 적절한 비유일 수도 있다고 언급했다.실제로, 계통발생적 산호는 과거와 현재의 삶을 묘사하는 데 유용하며 나무보다 몇 가지 장점이 있다(아노스토마스가 허용됨 등).[19]
건설
중요하지 않은 수의 입력 시퀀스로 구성된 계통수를 계산 계통학 방법을 사용하여 구축한다.다중 배열 정렬에서 유전적 거리를 계산하는 인접 결합 또는 UPGMA와 같은 거리 매트릭스 방법은 구현하기 가장 간단하지만 진화 모델을 호출하지는 않는다.또한 ClusteralW와 같은 많은 시퀀스 정렬 방법은 트리 구성의 단순한 알고리즘(즉, 거리에 기반한 알고리즘)을 사용하여 트리를 생성한다.최대 절약성은 계통수 추정의 또 다른 간단한 방법이지만, 암묵적인 진화 모델(즉 절약)을 암시한다.보다 진보된 방법은 종종 베이지안 프레임워크 내에서 최대 우도의 최적성 기준을 사용하고 계통수 [3]추정에 진화의 명시적 모델을 적용한다.이러한 많은 기술을 사용하여 최적의 트리를 식별하는 것은 NP-hard이기 때문에 [3]데이터에 적합한 상당히 양호한 트리를 식별하기 위해 경험적 검색 및 최적화 방법을 트리 스코어링 함수와 함께 사용한다.
나무 구축 방법은 다음과 같은 [21]몇 가지 기준에 따라 평가할 수 있다.
- 효율성(답안을 계산하는 데 얼마나 걸립니까, 필요한 메모리는 어느 정도입니까?)
- power(데이터를 잘 사용하고 있는지, 정보가 낭비되고 있는지)
- 일관성(같은 모델 문제에 대해 매번 다른 데이터가 주어진 경우 동일한 답변에 반복적으로 수렴됩니까?)
- 견고성(기본 모델의 가정 위반에 잘 대처합니까?)
- 반증 가능성(사용이 좋지 않을 때, 즉 가정이 위반되었을 때 경고합니까?)
나무 만드는 기술 또한 수학자들의 관심을 끌었다.트리는 또한 [22]T이론을 사용하여 만들 수 있다.
파일 형식
이 섹션은 어떠한 출처도 인용하지 않습니다.2020년 6월 (이를 에 대해 설명합니다) |
트리는 다양한 형식으로 인코딩할 수 있으며, 모두 트리의 중첩된 구조를 나타내야 합니다.분기 길이 및 기타 기능을 인코딩하거나 인코딩하지 않을 수 있습니다.표준화된 포맷은 기존 소프트웨어에 Import하기 어려운 그래픽 출력에 의존하지 않고 트리를 배포 및 공유하기 위해 매우 중요합니다.일반적으로 사용되는 형식은 다음과 같습니다.
계통발생학적 분석의 한계
비록 다른 종의 염기서열 유전자나 게놈 데이터를 기반으로 생성된 계통수가 진화적 통찰력을 제공할 수 있지만, 이러한 분석들은 중요한 한계를 가지고 있다.가장 중요한 것은, 나무가 생성하는 나무가 반드시 올바른 것은 아니며, 포함된 분류군의 진화 역사를 정확하게 나타내는 것은 아니다.다른 과학적 결과와 마찬가지로, 추가 연구(예: 추가 데이터 수집, 개선된 방법으로 기존 데이터 분석)에 의해 위조될 수 있다.이들의 기초가 되는 데이터는 [23]노이즈가 있을 수 있다. 분석은 유전자 재조합,[24] 수평 유전자 이동,[25] 교배가 일어나기 전에 나무에서 가장 가까운 이웃이 아니었던 종들 간의 교배, 수렴 진화 및 보존된 염기서열로 혼동될 수 있다.
또한 단일 유전자나 단백질과 같은 단일 유형의 특성을 분석하거나 형태학적 분석에만 기초하는 데 문제가 있는데, 이는 다른 관련 데이터 소스로부터 구성되는 수목이 최초와 다른 경우가 많기 때문에 종 간의 계통학적 관계를 추론하는 데 있어 매우 주의가 요구되기 때문이다.이것은 다른 하플로타입 블록이 다른 이력을 가질 수 있는 측면 유전자 전달과 재조합의 대상이 되는 유전 물질에 가장 해당된다.이러한 유형의 분석에서 단일 유전자의 계통발생학적 분석의 출력 트리는 유전자의 계통발생(즉 유전자 나무) 추정치이며, 이러한 특성이 샘플링된 분류군(즉, 종 나무)의 계통발생은 아니지만 이상적으로는 둘 다 매우 가까워야 한다.이러한 이유로, 심각한 계통발생학 연구는 일반적으로 서로 다른 게놈 소스(예: 미토콘드리아 또는 플라스티드 대 핵 게놈)[26]에서 유래한 유전자 또는 서로 다른 선택 체제에서 진화할 것으로 예상되는 유전자의 조합을 사용한다. 따라서 호모플라스(거짓 호몰로지)는 자연선택에서 발생할 가능성이 낮다.
멸종된 종이 분석에 말단 노드로 포함되는 경우(예를 들어 내부 노드를 제한하는 것이 아니라), 그것들은 현존하는 종의 직접적인 조상을 나타내지 않는 것으로 간주된다.멸종된 종들은 일반적으로 고품질의 DNA를 가지고 있지 않다.
유용한 DNA 재료의 범위는 추출과 염기서열 분석 기술의 발달로 확대되었다.더 작은 조각이나 DNA 분해 생성물의 공간적 패턴으로부터 시퀀스를 추론할 수 있는 기술의 개발은 유용하다고 여겨지는 DNA의 범위를 더욱 넓힐 것이다.
계통수는 형태학, 특정 유형의 유전자의 유무, 삽입 및 삭제 이벤트, 그리고 진화적 신호를 포함하는 것으로 생각되는 다른 관찰 등 다양한 데이터 유형에서 추론할 수 있다.
계통발생학적 네트워크는 나무를 분리하는 것이 적절하지 않을 때 사용된다. 이러한 복잡성 때문에 표본으로 추출된 유기체의 진화 이력이 더 정확하다는 것을 암시한다.
「 」를 참조해 주세요.
- 클레이드
- 클래디스트리스
- 계산 계통학
- 진화생물학
- 진화분류학
- 범용 트리 얼라인먼트
- 계통학 소프트웨어 목록
- 계통수 시각화 소프트웨어 목록
- 단백질 도메인을 포함하는 생물학적 데이터베이스인 PANDIT
- 계통학적 비교법
레퍼런스
- ^ Letunic, Ivica; Bork, Peer (1 January 2007). "Interactive Tree Of Life (iTOL): an online tool for phylogenetic tree display and annotation" (PDF). Bioinformatics. 23 (1): 127–128. doi:10.1093/bioinformatics/btl529. ISSN 1367-4803. PMID 17050570. Archived (PDF) from the original on November 29, 2015. Retrieved 2015-07-21.
- ^ Ciccarelli, F. D.; Doerks, T.; Von Mering, C.; Creevey, C. J.; Snel, B.; Bork, P. (2006). "Toward automatic reconstruction of a highly resolved tree of life" (PDF). Science. 311 (5765): 1283–1287. Bibcode:2006Sci...311.1283C. CiteSeerX 10.1.1.381.9514. doi:10.1126/science.1123061. PMID 16513982. S2CID 1615592.
- ^ a b c d 펠센슈타인 J. (2004)Phylogenies Sinauer Associates 추론:선덜랜드, 매사추세츠 주
- ^ Bailly, Anatole (1981-01-01). Abrégé du dictionnaire grec français. Paris: Hachette. ISBN 978-2010035289. OCLC 461974285.
- ^ Bailly, Anatole. "Greek-french dictionary online". www.tabularium.be. Archived from the original on April 21, 2014. Retrieved March 2, 2018.
- ^ Dang, Cuong Cao; Minh, Bui Quang; McShea, Hanon; Masel, Joanna; James, Jennifer Eleanor; Vinh, Le Sy; Lanfear, Robert (9 February 2022). "nQMaker: Estimating Time Nonreversible Amino Acid Substitution Models". Systematic Biology: syac007. doi:10.1093/sysbio/syac007.
- ^ Hodge T, Cope M (1 October 2000). "A myosin family tree". J Cell Sci. 113 (19): 3353–4. doi:10.1242/jcs.113.19.3353. PMID 10984423. Archived from the original on 30 September 2007.
- ^ ""Tree" Facts: Rooted versus Unrooted Trees". Archived from the original on 2014-04-14. Retrieved 2014-05-26.
- ^ Maher BA (2002). "Uprooting the Tree of Life". The Scientist. 16 (2): 90–95. Bibcode:2000SciAm.282b..90D. doi:10.1038/scientificamerican0200-90. PMID 10710791. Archived from the original on 2003-10-02.
- ^ van Oven, Mannis; Kayser, Manfred (2009). "Updated comprehensive phylogenetic tree of global human mitochondrial DNA variation". Human Mutation. 30 (2): E386–E394. doi:10.1002/humu.20921. PMID 18853457. S2CID 27566749.
- ^ a b c Felsenstein, Joseph (1978-03-01). "The Number of Evolutionary Trees". Systematic Biology. 27 (1): 27–33. doi:10.2307/2412810. ISSN 1063-5157. JSTOR 2412810.
- ^ Fox, Emily. "The dendrogram". coursea. coursea. Archived from the original on 28 September 2017. Retrieved 28 September 2017.
- ^ Mayr, Ernst(1974) "클래디스트 분석 또는 클래디스트 분류?"동물계통학 및 진화연구 저널 12:94~128.doi:10.1111/j.1439-0469.1974.tb00160.x.
- ^ Labandeira, C. C.; Dilcher, D. L.; Davis, D. R.; Wagner, D. L. (1994-12-06). "Ninety-seven million years of angiosperm-insect association: paleobiological insights into the meaning of coevolution". Proceedings of the National Academy of Sciences. 91 (25): 12278–12282. Bibcode:1994PNAS...9112278L. doi:10.1073/pnas.91.25.12278. ISSN 0027-8424. PMC 45420. PMID 11607501.
- ^ Soares, Antonio; Râbelo, Ricardo; Delbem, Alexandre (2017). "Optimization based on phylogram analysis". Expert Systems with Applications. 78: 32–50. doi:10.1016/j.eswa.2017.02.012. ISSN 0957-4174.
- ^ Santamaria, R.; Theron, R. (2009-05-26). "Treevolution: visual analysis of phylogenetic trees". Bioinformatics. 25 (15): 1970–1971. doi:10.1093/bioinformatics/btp333. PMID 19470585.
- ^ a b "Evolutionary systematics: Spindle Diagrams". Palaeos.com. 2014-11-10. Retrieved 2019-11-07.
- ^ "Trees, Bubbles, and Hooves". A Three-Pound Monkey Brain — Biology, programming, linguistics, phylogeny, systematics …. 2007-11-21. Retrieved 2019-11-07.
- ^ a b c Podani, János (2019-06-01). "The Coral of Life". Evolutionary Biology. 46 (2): 123–144. doi:10.1007/s11692-019-09474-w. ISSN 1934-2845.
- ^ Darwin, Charles (1837). Notebook B. p. 25.
- ^ Penny, D.; Hendy, M. D.; Steel, M. A. (1992). "Progress with methods for constructing evolutionary trees". Trends in Ecology and Evolution. 7 (3): 73–79. doi:10.1016/0169-5347(92)90244-6. PMID 21235960.
- ^ A. 드레스, K.T. 휴버, 뷔.몰튼.2001. 순수 및 응용 수학의 미터법 공간.Documenta Mathematica LSU 2001: 121-139
- ^ Townsend JP, Su Z, Tekle Y (2012). "Phylogenetic Signal and Noise: Predicting the Power of a Data Set to Resolve Phylogeny". Genetics. 61 (5): 835–849. doi:10.1093/sysbio/sys036. PMID 22389443.
- ^ Arenas M, Posada D (2010). "The effect of recombination on the reconstruction of ancestral sequences". Genetics. 184 (4): 1133–1139. doi:10.1534/genetics.109.113423. PMC 2865913. PMID 20124027.
- ^ Woese C (2002). "On the evolution of cells". Proc Natl Acad Sci USA. 99 (13): 8742–7. Bibcode:2002PNAS...99.8742W. doi:10.1073/pnas.132266999. PMC 124369. PMID 12077305.
- ^ Parhi, J.; Tripathy, P.S.; Priyadarshi, H.; Mandal, S.C.; Pandey, P.K. (2019). "Diagnosis of mitogenome for robust phylogeny: A case of Cypriniformes fish group". Gene. 713: 143967. doi:10.1016/j.gene.2019.143967. PMID 31279710. S2CID 195828782.
추가 정보
- 슈, R.T., A.V. Z. 브라우어2009. 생물학적 체계학: 원칙과 응용(2차 교육)ISBN 978-0-8014-4799-0
- Manuel Lima, The Book of Trees: Visualizing Branchs of Knowledge, 2014, 뉴욕 프린스턴 건축 프레스.
- MEGA, 계통수를 그리는 무료 소프트웨어.
- 곤티에, N. 2011생명의 나무 퇴치: 진화 나무 다이어그램의 철학적, 역사적 뿌리.진화, 교육, 아웃리치 4: 515~538.
외부 링크
이미지들
일반
- 다양한 트리 시각화 방법에 대한 개요는 다음 웹 사이트에서 볼 수 있습니다.
- OneZoom:Tree of Life – 직관적이고 확대 가능한 프랙탈 탐색기(응답형 설계)로 모든 생물종
- 미국 국립과학재단의 생명의 나무 조립 프로젝트를 기반으로 한 인터랙티브 트리 발견
- Phylo Code(필로코드)
- 139개의 미오신 배열과 계통수의 다중 정렬
- 트리 오브 라이프 웹 프로젝트
- T-REX 서버에서의 계통학적 추론
- NCBI 분류 데이터베이스[1]
- ETE: 나무 탐사를 위한 파이썬 환경 이것은 계통수 분석, 조작 및 시각화를 위한 프로그래밍 라이브러리입니다.레퍼런스
- 매일 갱신되는 라이프 트리(시퀀스 완료) Fang, H.; Oates, M. E.; Pethica, R. B.; Greenwood, J. M.; Sardar, A. J.; Rackham, O. J. L.; Donoghue, P. C. J.; Stamatakis, A.; De Lima Morais, D. A.; Gough, J. (2013). "A daily-updated tree of (sequenced) life as a reference for genome research". Scientific Reports. 3: 2015. Bibcode:2013NatSR...3E2015F. doi:10.1038/srep02015. PMC 6504836. PMID 23778980.