비교유전체학
Comparative genomics
비교유전체학(Comparative Genomics)은 생물학적 연구의 한 분야로 서로 다른 생물의 유전적 특징을 비교하는 학문입니다.[2][3]유전적 특징은 DNA 서열, 유전자, 유전자 순서, 조절 서열 및 기타 유전적 구조적 랜드마크를 포함할 수 있습니다.[3]유전체학의 이 분야에서, 게놈 프로젝트로 인한 게놈의 전체 또는 큰 부분은 유기체들 사이의 진화적 관계뿐만 아니라 기본적인 생물학적 유사성과 차이점을 연구하기 위해 비교됩니다.[2][4][5]비교유전체학의 주요 원리는 두 유기체의 공통적인 특징이 종종 그들 사이에서 진화적으로 보존되는 DNA 안에 암호화된다는 것입니다.[6]따라서 비교 게놈 접근법은 어떤 형태로든 게놈 서열을 정렬하고 정렬된 게놈에서 직교 시퀀스(같은 조상을 공유하는 시퀀스)를 찾고 그 시퀀스가 어느 정도까지 보존되는지 확인하는 것에서 시작합니다.이것들을 바탕으로 게놈과 분자 진화를 추론하고 이것은 다시 표현형 진화 또는 집단 유전학의 맥락에 놓일 수 있습니다.[7]
1995년에 두 유기체의 전체 게놈(즉, 박테리아 해모필루스 인플루엔자와 마이코플라즈마 생식기의 게놈)을 이용할 수 있게 되자마자 사실상 시작된 비교 게놈은 이제 모든 새로운 게놈 서열 분석의 표준 구성 요소입니다.[2][8]2000년대 후반 DNA 염기서열 분석 기술의 발전, 특히 차세대 염기서열 분석 방법의 발전으로 유전체 프로젝트의 수가 급증하면서 이 분야는 더욱 정교해져 한 번의 연구로 많은 유전체를 다룰 수 있게 되었습니다.[9]비교 유전체학은 인간과 침팬지와 같은 밀접한 관련이 있는 유기체들 간의 높은 유사성을 드러냈고, 더 놀라운 것은 인간과 효모 사카로마이세스 세레비시아와 같은 멀리 관련이 있는 것처럼 보이는 유기체들 간의 유사성을 드러냈습니다.[4]그것은 또한 다른 진화 계통에서 유전자 구성의 극도의 다양성을 보여주었습니다.[8]
역사
참고 항목:게놈학의 역사
비교유전체학은 1980년대 초 바이러스 유전체 비교에 뿌리를 두고 있습니다.[8]예를 들어, 동물을 감염시키는 작은 RNA 바이러스(피코르나바이러스)와 감염시키는 식물(소 완두콩 모자이크 바이러스)을 비교하여 상당한 서열 유사성과 부분적으로 유전자의 순서를 공유하는 것으로 밝혀졌습니다.[10]1986년, 각각 100개 이상의 유전자를 포함하는 바리셀라-조스터 바이러스와 엡스타인-바 바이러스의 게놈을 비교한 더 큰 규모의 첫 비교 게놈 연구가 발표되었습니다.[11]
세포 유기체의 최초의 완전한 게놈 서열인 Haemophilus influenzae Rd는 1995년에 발표되었습니다.[12]두 번째 게놈 시퀀싱 논문은 같은 해에 발표된 작은 기생 박테리아 마이코플라즈마 생식기에 관한 것이었습니다.[13]본 논문을 기점으로 새로운 유전체에 대한 보고는 필연적으로 비교유전체학적 연구가 되었습니다.[8]
미생물 유전체.10-15kbp의 미생물 유전체의 최초의 고해상도 전체 유전체 비교 시스템은 1998년 아트 델처, 사이먼 카시프 및 스티븐 샐즈버그에 의해 개발되었으며 게놈 연구소(TIGR)의 협력자와 관련성이 높은 전체 미생물의 비교에 적용되었습니다.이 시스템은 MUMMER라고 불리며 1999년 핵산 연구지에 기술되었습니다.이 시스템은 연구자들이 큰 재배열, 단일 염기 변이, 반전, 탠덤 반복 확장 및 기타 다형성을 식별하는 데 도움이 됩니다.박테리아에서 MUMMER는 독성, 병원성, 항생물학적 내성을 담당하는 다형성의 식별을 가능하게 합니다.이 시스템은 또한 TIGR의 최소 유기체 프로젝트와 그 후 많은 다른 비교 유전체학 프로젝트에도 적용되었습니다.
진핵생물 유전체.제빵사의 효모인 Saccharomyces cerevisiae는 1996년 완전한 게놈 서열을 가진 최초의 진핵생물이었습니다.[14]1998년[15] 회충 Caenorhabditis elegans 게놈이 발표되고 2000년 초파리 Drosophila melanogaster 게놈과 함께 Gerald M.[16] 루빈과 그의 팀은 "진핵생물의 유전체 비교"라는 제목의 논문을 발표했는데, 이 논문에서 그들은 원핵생물 H. 인플루엔자와 함께 진핵생물 D. melanogaster, C. elegans, S. cerevisiae의 게놈을 비교했습니다.[17]동시에 보니 버거(Bonnie Berger), 에릭 랜더(Eric Lander), 그리고 그들의 팀은 인간과 쥐의 전체 게놈 비교에 관한 논문을 발표했습니다.[18]
2000년대에 인간, 복어 타키푸구 루브립스, 그리고 쥐를 포함한 척추동물의 큰 유전체가 발표되면서, 미리 계산된 큰 유전체 비교 결과가 다운로드를 위해 또는 게놈 브라우저에서 시각화를 위해 발표되었습니다.대부분의 생물학자들은 자체적인 분석을 수행하는 대신 이러한 대규모 종간 비교에 접근할 수 있으며 게놈의 크기로 인한 비현실성을 피할 수 있습니다.[19]
2007년에 처음 소개된 차세대 염기서열 분석 방법은 엄청난 양의 유전체 데이터를 만들어냈고, 연구자들이 한 번에 여러 개의 (원핵) 드래프트 유전체 서열을 생성할 수 있게 했습니다.이러한 방법은 또한 잘 주석이 달린 참조 게놈에 조립되지 않은 판독치를 매핑하여 단일 뉴클레오티드 다형성, 삽입 및 삭제를 신속하게 발견할 수 있으며, 따라서 균주 간의 기능적 변이의 기초가 될 수 있는 유전자 차이의 목록을 제공합니다.[9]
진화 원리
생물학의 한 가지 특징은 진화론이고, 진화론은 비교유전체학의 이론적 토대이기도 하며, 동시에 비교유전체학의 결과는 전례 없이 진화론을 풍부하고 발전시켰습니다.두 개 이상의 게놈 서열을 비교하면 계통발생학적 트리에서 서열의 진화적 관계를 추론할 수 있습니다.다양한 생물 유전체 데이터와 수직적, 수평적 진화 과정에 대한 연구를 바탕으로 유전자 구조의 중요한 부분과 그 조절 기능을 이해할 수 있습니다.
관련 유전체의 유사성은 비교유전체학의 기초가 됩니다.만약 두 생물체가 최근 공통 조상을 가지고 있다면, 두 종의 게놈의 차이점은 조상의 게놈에서 진화된 것입니다.두 유기체 간의 관계가 가까울수록, 그들의 게놈 간의 유사성이 높아집니다.만약 그들 사이에 밀접한 관계가 있다면, 그들의 게놈은 선형적인 행동(synteny), 즉 유전자 서열의 일부 또는 전부가 보존될 것입니다.따라서 게놈 서열은 알려진 기능의 유전자와 상동성(서열 유사성)을 분석하여 유전자 기능을 식별하는 데 사용될 수 있습니다.

직교 배열은 다른 종에서 관련된 서열입니다: 유전자가 원래의 종에 존재하고, 두 종으로 나뉘어서, 새로운 종의 유전자는 원래의 종의 서열에 직교합니다.유전자 복제(유전자 복제)를 통해 유사한 서열이 분리됩니다. 게놈의 특정 유전자가 복제되면 두 서열의 복제는 원래 유전자와 유사합니다.직교 시퀀스 쌍을 직교 쌍(orthologous pairs)이라고 하고, 병렬 시퀀스 쌍을 부수 쌍(paralogs)이라고 합니다.직교 쌍은 일반적으로 동일하거나 유사한 기능을 갖지만, 반드시 측부 쌍의 경우에는 그렇지 않습니다.부수적인 쌍에서, 시퀀스는 다른 기능을 갖도록 진화하는 경향이 있습니다.
비교 유전체학은 선택이 이러한 요소들에 어떻게 작용했는지를 추론하기 위해 다른 유기체의 단백질, RNA, 그리고 조절 영역의 유사성과 차이점을 이용합니다.다른 종들 간의 유사성에 책임이 있는 요소들은 시간을 통해 보존되어야 하고(선택 안정화), 종들 간의 차이에 책임이 있는 요소들은 발산적이어야 합니다(선택적 선택).마지막으로, 유기체의 진화적 성공에 중요하지 않은 요소들은 비보존될 것입니다(선택은 중립적입니다).
이 분야의 중요한 목표 중 하나는 진핵생물 유전체 진화의 메커니즘을 규명하는 것입니다.그러나 그것은 종종 개별 계통의 역사를 통해 일어난 사건들의 다중성으로 인해 복잡해지고, 각 살아있는 유기체의 게놈에 왜곡되고 중첩된 흔적만 남깁니다.이러한 이유로 작은 모델 유기체(예: Caenorhabditis elegans 모델 및 밀접하게 관련된 Caenorhabditis brigsae 모델)에 대한 비교 게놈 연구는 일반적인 진화 메커니즘에 대한 이해를 증진시키는 데 매우 중요합니다.[20][21]
방법들
게놈에서 인코딩된 많은 양의 데이터를 고려할 때, 게놈 비교를 위해서는 계산 접근법이 필요합니다.전체 게놈 비교부터 유전자 발현 분석에 이르기까지 많은 도구들이 현재 공개적으로 이용 가능합니다.[22]여기에는 시스템 및 제어, 정보 이론, 문자열 분석 및 데이터 마이닝 등의 접근 방식이 포함됩니다.[23]특히 정보 과학과 게놈 생물학이 함께 교육될 때 컴퓨터 접근 방식은 연구와 교육에 매우 중요할 것입니다.[24]

비교유전체학은 유전체 크기와 유전자 밀도의 기본적인 비교부터 시작합니다.예를 들어, 유전체 크기는 코딩 용량에 중요하고 아마도 규제적인 이유로 중요합니다.높은 유전자 밀도는 게놈 주석, 환경 선택 분석을 용이하게 합니다.대조적으로, 낮은 유전자 밀도는 인간 게놈에서와 같이 유전 질환 지도 작성을 방해합니다.
순서 정렬
선형은 조상, 공통 진화 혈통 또는 공통 구조 및 기능과 같은 유사한 시퀀스에 대한 정보를 캡처하는 데 사용됩니다.정렬은 유전자 배열과 단백질 배열 모두에 대해 수행될 수 있습니다.[26][27]선형은 로컬 또는 전역 쌍방향 선형과 다중 시퀀스 선형으로 구성됩니다.전역 정렬을 찾는 한 가지 방법은 Needleman-Wunsch 알고리즘으로 알려진 동적 프로그래밍 알고리즘을 사용하는 것입니다.이 알고리즘을 수정하여 로컬 선형을 찾는 데 사용할 수 있습니다.

계통발생학적 재구성
비교유전체학의 또 다른 계산 방법은 계통발생학적 재구성입니다.그것은 공통 조상의 관점에서 진화적 관계를 설명하는 데 사용됩니다.이 관계들은 보통 계통발생 나무라고 불리는 나무로 표현됩니다.마찬가지로, 연합 이론은 모집단의 유전자의 모든 유전자를 모집단 구성원이 공유하는 단일 조상 사본으로 추적하는 회고적 모델입니다.이것은 가장 최근의 일반적인 조상으로도 알려져 있습니다.연합 이론에 근거한 분석은 돌연변이의 도입과 모집단의 특정 대립유전자 또는 유전자 분포 사이의 시간을 예측하려고 시도합니다.이 기간은 가장 최근의 공통 조상이 얼마나 오래 전에 존재했는지와 같습니다.상속 관계는 계통발생 트리와 유사한 형태로 시각화됩니다.합체(또는 유전자 계보)는 덴드로그램을 사용하여 시각화될 수 있습니다.[28]
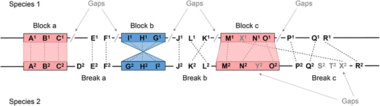
게놈지도
비교유전체학에서 추가적인 방법은 유전자 지도 작성입니다.유전자 지도 제작에서 합성을 시각화하는 것은 염색체에 유전자가 보존된 순서를 볼 수 있는 한 가지 방법입니다.그것은 보통 공통 조상으로부터 비롯되는 관련 종의 염색체에 사용됩니다.[30]이것과 다른 방법들은 진화의 역사를 밝힐 수 있습니다.최근의 한 연구는 포유류 계통발생학 전반에 걸쳐 16개의 조상 핵형을 재구성하기 위해 비교 유전체학을 사용했습니다.컴퓨터 재구성은 포유류 진화 동안 염색체들이 어떻게 자신들을 재배열하는지를 보여주었습니다.개발 프로세스의 제어와 관련된 일부 지역의 보존에 대한 통찰력을 제공했습니다.게다가, 그것은 염색체의 진화와 DNA 재배열과 관련된 유전 질환에 대한 이해를 제공하는 데 도움을 주었습니다.[citation needed]

도구들
대량의 게놈 데이터를 이용할 수 있기 때문에 서열 분석과 완전한 게놈 분석을 위한 계산 도구가 빠르게 발전하고 있습니다.이와 동시에 비교 분석 도구를 진행하고 개선합니다.이러한 분석에 대한 과제에서는 비교 결과를 시각화하는 것이 매우 중요합니다.[32]
서열 보존의 시각화는 비교 서열 분석의 어려운 작업입니다.우리가 알고 있는 바와 같이 긴 게놈 영역의 정렬을 수동으로 조사하는 것은 매우 비효율적입니다.인터넷 기반 게놈 브라우저는 게놈 영역에 대한 모든 서열 기반 생물학적 정보를 통합하기 때문에 게놈 서열을 조사하는 데 유용한 많은 도구를 제공합니다.많은 양의 관련 생물학적 데이터를 추출하면 매우 쉽게 사용할 수 있고 시간 소모도 적을 수 있습니다.[32]
- UCSC 브라우저:이 사이트에는 대량의 게놈 모음을 위한 참조 시퀀스 및 작업 초안 어셈블리가 포함되어 있습니다.[33]
- 앙상블:Ensembl 프로젝트는 척추동물과 다른 진핵생물 종들을 위한 게놈 데이터베이스를 생산하고, 이 정보를 온라인에서 자유롭게 이용할 수 있도록 합니다.[34]
- 지도 보기:맵 뷰어는 다양한 게놈 매핑 및 염기서열 분석 데이터를 제공합니다.[35]
- VISTA는 게놈 서열 비교 분석을 위한 포괄적인 프로그램 및 데이터베이스 제품군입니다.DNA배열을 기반으로 비교 분석한 결과를 시각화하기 위해 제작되었습니다.VISTA에서 생성된 비교 데이터는 소규모 데이터와 대규모 데이터 모두에 쉽게 적합합니다.[36]
- BlueJay Genome Browser: 주석이 달린 유전체 및 기타 유전체 요소의 다중 스케일 보기를 위한 독립형 시각화 도구.[37]
온라인 도구를 사용하는 장점은 이러한 웹 사이트가 지속적으로 개발되고 업데이트된다는 것입니다.효율성을 높이기 위해 많은 새로운 설정과 콘텐츠를 온라인에서 사용할 수 있습니다.[32]
선택된 어플리케이션
농업
농업은 비교유전체학의 혜택을 받는 분야입니다.유리한 유전자의 위치를 확인하는 것은 수확량, 비용 효율성, 품질 및 질병 저항성 향상에 최적화된 작물을 사육하는 데 있어 중요한 단계입니다.예를 들어, 517개의 논에 대해 수행된 게놈 전체 연관 연구에서 곡물 중량, 아밀로스 함량 및 가뭄 내성과 같은 여러 범주의 농학적 성능과 관련된 80개의 장소가 밝혀졌습니다.많은 로키들이 이전에는 특징이 없었습니다.[38]이 방법론은 강력할 뿐만 아니라 빠릅니다.농학적 성과와 관련된 위치를 식별하는 이전의 방법은 몇 세대에 걸쳐 주의 깊게 관찰된 모균주 번식을 필요로 했는데, 이는 비교 유전학 연구에 불필요한 시간 소모적인 노력입니다.[39]
약
백신개발
의학 분야도 비교유전체학 연구의 혜택을 받습니다.역백신학(reverse vaccinology)으로 알려진 방법으로 연구자들은 병원체 또는 병원체 계열의 유전체를 분석함으로써 백신 개발을 위한 후보 항원을 발견할 수 있습니다.[40]여러 관련 병원체의 유전체를 분석해 비교유전체학적 접근을 적용하면 다중보호 백신 개발로 이어질 수 있습니다.한 연구팀은 심각한 신생아 감염의 원인이 되는 박테리아 그룹인 B 그룹 스트렙토코커스의 보편적인 백신을 만들기 위해 이와 같은 방법을 사용했습니다.[41]비교 유전체학은 또한 상식적인 미생물과 밀접한 관련이 있는 병원균에 대한 백신의 특이성을 생성하는 데 사용될 수 있습니다.예를 들어, 연구자들은 병원성 균주에 대한 면역 반응을 초래하는 항원을 발견하기 위한 근거로서 병원성 균주와 병원성 균주에 대한 유전자 비교 분석을 사용했습니다.[42]2019년 5월, 영국과 호주의 한 팀은 글로벌 게놈 세트를 사용하여 전 세계적으로 수집된 수천 개의 A 그룹 스트렙토코커스 격리물을 염기서열 분석하여 S. 피오게네스라고도 알려진 병원체에 대항하는 백신을 개발하기 위한 잠재적인 표적을 제공했습니다.[43]

면역학의 마우스 모델
T 세포는 골수의 줄기세포로부터 자라는 면역세포입니다.그들은 몸을 감염으로부터 방어하는 데 도움을 주고 암과의 싸움에 도움을 줄 수도 있습니다.인간과 형태적, 생리적, 유전적 유사성 때문에, 쥐와 쥐는 오랫동안 생물 의학 연구 동물 모델로 선호되어 왔습니다.비교 의학 연구는 한 종의 정보를 사용하여 다른 종의 동일한 과정을 이해하는 능력을 기반으로 합니다.우리는 비교유전체학을 이용하여 사람과 쥐의 T세포와 면역체계에 미치는 영향을 비교함으로써 분자 경로에 대한 새로운 통찰력을 얻을 수 있습니다.글루스만은 TCRs와 그 유전자를 이해하기 위해 인간과 쥐 T세포 수용체 위치의 염기서열 분석 연구를 진행했습니다.TCR 유전자는 잘 알려져 있으며, 기능성 유전체학을 지원하고 유전체의 유전자와 유전체 간 영역이 생물학적 과정에 어떻게 기여하는지를 이해하는 데 중요한 자료로 사용됩니다.[44]
T 세포 면역 수용체는 세포 면역계에서 병원체의 세계를 보는 데 중요합니다.인간과 생쥐 TCR loci를 염기서열 분석한 이유 중 하나는 비교유전체학을 이용하여 직교 유전자 계열 서열을 일치시키고 보존된 영역을 발견하기 위함이었습니다.이것들은 두 종류의 생물학적 정보, 즉 (1) 엑손과 (2) 조절 서열을 반영할 것으로 생각되었습니다.실제로 이 방법에서는 V, D, J, Cexon의 대부분을 확인할 수 있었습니다.가변 영역은 T 세포(TCR) 분화 동안 재배열되고 연결되는 다수의 고유한 DNA 요소에 의해 인코딩됩니다: 및 폴리펩티드를 위한 변수(V), 다양성(D) 및 결합(J) 요소; 및 및 및 폴리펩티드를 위한 V 및 J 요소.[도 1] 그러나, 유전체의 짧은 비코딩 보존 블록들이 몇 개 보여졌습니다.사람과 마우스 모티프 모두 200bp [그림 2]에서 크게 군집화되어 있으며, TCR/에서 알려진 3' 강화제가 확인되었으며, 마우스 Jintron에서 100bp의 보존 영역이 조절 기능을 갖는 것으로 나타났습니다.

염색체 상의 특정 유전자의 각 물리적 부위 또는 위치와 종 간의 게놈 서열 비교는 다른 메커니즘 및 기타 조절 신호에 대한 연구를 가능하게 합니다.어떤 사람들은 다른 척추동물 종의 TCR 유전자 보완과 비교하여 TCR의 진화에 대한 새로운 가설을 제시합니다.인간과 쥐의 유전체 비교 조사는 분명히 다른 많은 유전자의 발견과 주석을 허용할 뿐만 아니라 조절 서열을 위해 다른 종에서 확인할 수 있습니다.[44]
조사.
비교유전체학은 또한 다른 연구 분야에서도 새로운 길을 열어줍니다.DNA 염기서열 분석 기술이 더욱 접근하기 쉬워지면서, 염기서열 분석된 유전체의 수가 증가했습니다.이용 가능한 게놈 데이터의 저장량이 증가함에 따라 비교 게놈 추론의 가능성도 커졌습니다.
이러한 효능 증가의 주목할 만한 사례는 최근 영장류 연구에서 발견됩니다.비교 유전학적 방법은 연구자들이 이전의 데이터와 방법을 사용하여 구별할 수 없었던 영장류의 유전적 변이, 유전자의 분화, 그리고 진화 역학에 대한 정보를 모을 수 있게 했습니다.[45]
유인원 게놈 프로젝트
유인원 게놈 프로젝트는 6종의 유인원 종과 관련하여 유전적 변이를 조사하기 위해 비교 유전학적 방법을 사용하여 개체수의 감소에도 불구하고 유전자 풀에서 건강한 수준의 변이를 발견했습니다.[46]또 다른 연구는 유전자 발현에 대한 알려진 조절 메커니즘인 DNA 메틸화의 패턴이 인간 대 침팬지의 전전두엽 피질에서 다르다는 것을 보여주었고, 두 종의 진화적 차이를 암시했습니다.[47]
참고 항목
참고문헌
- ^ Darling AE, Miklós I, Ragan MA (July 2008). "Dynamics of genome rearrangement in bacterial populations". PLOS Genetics. 4 (7): e1000128. doi:10.1371/journal.pgen.1000128. PMC 2483231. PMID 18650965.

- ^ a b c Touchman J (2010). "Comparative Genomics". Nature Education Knowledge. 3 (10): 13.
- ^ a b Xia X (2013). Comparative Genomics. SpringerBriefs in Genetics. Heidelberg: Springer. doi:10.1007/978-3-642-37146-2. ISBN 978-3-642-37145-5. S2CID 5491782.
- ^ a b Russel PJ, Hertz PE, McMillan B (2011). Biology: The Dynamic Science (2nd ed.). Belmont, CA: Brooks/Cole. pp. 409–410.
- ^ Primrose SB, Twyman RM (2003). Principles of Genome Analysis and Genomics (3rd ed.). Malden, MA: Blackwell Publishing. ISBN 9781405101202.
- ^ Hardison RC (November 2003). "Comparative genomics". PLOS Biology. 1 (2): E58. doi:10.1371/journal.pbio.0000058. PMC 261895. PMID 14624258.

- ^ Ellegren H (November 2008). "Comparative genomics and the study of evolution by natural selection". Molecular Ecology. 17 (21): 4586–4596. doi:10.1111/j.1365-294X.2008.03954.x. PMID 19140982. S2CID 43171654.
- ^ a b c d Koonin EV, Galperin MY (2003). Sequence - Evolution - Function: Computational approaches in comparative genomics. Dordrecht: Springer Science+Business Media.
- ^ a b Hu B, Xie G, Lo CC, Starkenburg SR, Chain PS (November 2011). "Pathogen comparative genomics in the next-generation sequencing era: genome alignments, pangenomics and metagenomics". Briefings in Functional Genomics. 10 (6): 322–333. doi:10.1093/bfgp/elr042. PMID 22199376.
- ^ Argos P, Kamer G, Nicklin MJ, Wimmer E (September 1984). "Similarity in gene organization and homology between proteins of animal picornaviruses and a plant comovirus suggest common ancestry of these virus families". Nucleic Acids Research. 12 (18): 7251–7267. doi:10.1093/nar/12.18.7251. PMC 320155. PMID 6384934.
- ^ McGeoch DJ, Davison AJ (May 1986). "DNA sequence of the herpes simplex virus type 1 gene encoding glycoprotein gH, and identification of homologues in the genomes of varicella-zoster virus and Epstein-Barr virus". Nucleic Acids Research. 14 (10): 4281–4292. doi:10.1093/nar/14.10.4281. PMC 339861. PMID 3012465.
- ^ Fleischmann RD, Adams MD, White O, Clayton RA, Kirkness EF, Kerlavage AR, et al. (July 1995). "Whole-genome random sequencing and assembly of Haemophilus influenzae Rd". Science. 269 (5223): 496–512. Bibcode:1995Sci...269..496F. doi:10.1126/science.7542800. PMID 7542800.
- ^ Fraser CM, Gocayne JD, White O, Adams MD, Clayton RA, Fleischmann RD, et al. (October 1995). "The minimal gene complement of Mycoplasma genitalium". Science. 270 (5235): 397–403. Bibcode:1995Sci...270..397F. doi:10.1126/science.270.5235.397. PMID 7569993. S2CID 29825758.
- ^ Goffeau A, Barrell BG, Bussey H, Davis RW, Dujon B, Feldmann H, et al. (October 1996). "Life with 6000 genes". Science. 274 (5287): 546, 563–546, 567. Bibcode:1996Sci...274..546G. doi:10.1126/science.274.5287.546. PMID 8849441. S2CID 16763139.
- ^ The C. elegans Sequencing Consortium (December 1998). "Genome sequence of the nematode C. elegans: a platform for investigating biology". Science. 282 (5396): 2012–2018. Bibcode:1998Sci...282.2012.. doi:10.1126/science.282.5396.2012. PMID 9851916.
- ^ Adams MD, Celniker SE, Holt RA, Evans CA, Gocayne JD, Amanatides PG, et al. (March 2000). "The genome sequence of Drosophila melanogaster". Science. 287 (5461): 2185–2195. Bibcode:2000Sci...287.2185.. CiteSeerX 10.1.1.549.8639. doi:10.1126/science.287.5461.2185. PMID 10731132.
- ^ Rubin GM, Yandell MD, Wortman JR, Gabor Miklos GL, Nelson CR, Hariharan IK, et al. (March 2000). "Comparative genomics of the eukaryotes". Science. 287 (5461): 2204–2215. Bibcode:2000Sci...287.2204.. doi:10.1126/science.287.5461.2204. PMC 2754258. PMID 10731134.
- ^ Batzoglou S, Pachter L, Mesirov JP, Berger B, Lander ES (July 2000). "Human and mouse gene structure: comparative analysis and application to exon prediction". Genome Research. 10 (7): 950–958. doi:10.1101/gr.10.7.950. PMC 310911. PMID 10899144.

- ^ Ureta-Vidal A, Ettwiller L, Birney E (April 2003). "Comparative genomics: genome-wide analysis in metazoan eukaryotes". Nature Reviews. Genetics. 4 (4): 251–262. doi:10.1038/nrg1043. PMID 12671656. S2CID 2037634.
- ^ Stein LD, Bao Z, Blasiar D, Blumenthal T, Brent MR, Chen N, et al. (November 2003). "The genome sequence of Caenorhabditis briggsae: a platform for comparative genomics". PLOS Biology. 1 (2): E45. doi:10.1371/journal.pbio.0000045. PMC 261899. PMID 14624247.

- ^ "Newly Sequenced Worm a Boon for Worm Biologists". PLOS Biology. 1 (2): e4. 2003. doi:10.1371/journal.pbio.0000044.

- ^ Cristianini N, Hahn M (2006). Introduction to Computational Genomics. Cambridge University Press. ISBN 978-0-521-67191-0.
- ^ Pratas D, Silva RM, Pinho AJ, Ferreira PJ (May 2015). "An alignment-free method to find and visualise rearrangements between pairs of DNA sequences". Scientific Reports. 5: 10203. Bibcode:2015NatSR...510203P. doi:10.1038/srep10203. PMC 4434998. PMID 25984837.
- ^ Via A, De Las Rivas J, Attwood TK, Landsman D, Brazas MD, Leunissen JA, et al. (October 2011). "Ten simple rules for developing a short bioinformatics training course". PLOS Computational Biology. 7 (10): e1002245. Bibcode:2011PLSCB...7E2245V. doi:10.1371/journal.pcbi.1002245. PMC 3203054. PMID 22046119.

- ^ Damas J, Corbo M, Kim J, Turner-Maier J, Farré M, Larkin DM, et al. (October 2022). "Evolution of the ancestral mammalian karyotype and syntenic regions". Proceedings of the National Academy of Sciences of the United States of America. 119 (40): e2209139119. doi:10.1073/pnas.2209139119. PMC 9550189. PMID 36161960.
- ^ Altschul SF, Pop M (2017). "Sequence Alignment". In Rosen KH, Shier DR, Goddard W (eds.). Handbook of Discrete and Combinatorial Mathematics (2nd ed.). Boca Raton (FL): CRC Press/Taylor & Francis. ISBN 978-1-58488-780-5. PMID 29206392. Retrieved 2022-12-18.
- ^ Prjibelski AD, Korobeynikov AI, Lapidus AL (2019-01-01). "Sequence Analysis". In Ranganathan S, Gribskov M, Nakai K, Schönbach C (eds.). Encyclopedia of Bioinformatics and Computational Biology. Oxford: Academic Press. pp. 292–322. doi:10.1016/b978-0-12-809633-8.20106-4. ISBN 978-0-12-811432-2. S2CID 226247797.
- ^ Haubold B, Wiehe T (September 2004). "Comparative genomics: methods and applications". Die Naturwissenschaften. 91 (9): 405–421. doi:10.1007/s00114-004-0542-8. PMID 15278216. S2CID 2041895.
- ^ Liu D, Hunt M, Tsai IJ (January 2018). "Inferring synteny between genome assemblies: a systematic evaluation". BMC Bioinformatics. 19 (1): 26. doi:10.1186/s12859-018-2026-4. PMC 5791376. PMID 29382321.
- ^ Duran C, Edwards D, Batley J (2009). "Genetic maps and the use of synteny". Plant Genomics. Methods in Molecular Biology. Vol. 513. pp. 41–55. doi:10.1007/978-1-59745-427-8_3. ISBN 978-1-58829-997-0. PMID 19347649.
- ^ Damas J, Corbo M, Kim J, Turner-Maier J, Farré M, Larkin DM, et al. (October 2022). "Evolution of the ancestral mammalian karyotype and syntenic regions". Proceedings of the National Academy of Sciences of the United States of America. 119 (40): e2209139119. doi:10.1073/pnas.2209139119. PMC 9550189. PMID 36161960.
- ^ a b c Bergman NH (2007). Bergman NH (ed.). Comparative Genomics: Volumes 1 and 2. Totowa, New Jersey: Humana Press. ISBN 978-193411-537-4. PMID 21250292.
- ^ "UCSC Browser".
- ^ "Ensembl Genome Browser". Archived from the original on 2013-10-21.
- ^ "Map Viewer".
- ^ "VISTA tools".
- ^ Soh J, Gordon PM, Sensen CW (March 2012). "The Bluejay genome browser". Current Protocols in Bioinformatics. John Wiley & Sons, Inc. 37: Unit10.9. doi:10.1002/0471250953.bi1009s37. ISBN 9780471250951. PMID 22389011. S2CID 34553139.
- ^ Huang X, Wei X, Sang T, Zhao Q, Feng Q, Zhao Y, et al. (November 2010). "Genome-wide association studies of 14 agronomic traits in rice landraces". Nature Genetics. 42 (11): 961–967. doi:10.1038/ng.695. PMID 20972439. S2CID 439442.

- ^ Morrell PL, Buckler ES, Ross-Ibarra J (December 2011). "Crop genomics: advances and applications". Nature Reviews. Genetics. 13 (2): 85–96. doi:10.1038/nrg3097. PMID 22207165. S2CID 13358998.

- ^ Seib KL, Zhao X, Rappuoli R (October 2012). "Developing vaccines in the era of genomics: a decade of reverse vaccinology". Clinical Microbiology and Infection. 18 Suppl 5 (SI): 109–116. doi:10.1111/j.1469-0691.2012.03939.x. PMID 22882709.

- ^ Maione D, Margarit I, Rinaudo CD, Masignani V, Mora M, Scarselli M, et al. (July 2005). "Identification of a universal Group B streptococcus vaccine by multiple genome screen". Science. 309 (5731): 148–150. Bibcode:2005Sci...309..148M. doi:10.1126/science.1109869. PMC 1351092. PMID 15994562.

- ^ Rasko DA, Rosovitz MJ, Myers GS, Mongodin EF, Fricke WF, Gajer P, et al. (October 2008). "The pangenome structure of Escherichia coli: comparative genomic analysis of E. coli commensal and pathogenic isolates". Journal of Bacteriology. 190 (20): 6881–6893. doi:10.1128/JB.00619-08. PMC 2566221. PMID 18676672.

- ^ "Group a Streptococcus Vaccine Target Candidates Identified from Global Genome Set". 28 May 2019.
- ^ a b c d Glusman G, Rowen L, Lee I, Boysen C, Roach JC, Smit AF, et al. (September 2001). "Comparative genomics of the human and mouse T cell receptor loci". Immunity. 15 (3): 337–349. doi:10.1016/s1074-7613(01)00200-x. PMID 11567625.
- ^ Rogers J, Gibbs RA (May 2014). "Comparative primate genomics: emerging patterns of genome content and dynamics". Nature Reviews. Genetics. 15 (5): 347–359. doi:10.1038/nrg3707. PMC 4113315. PMID 24709753.

- ^ Prado-Martinez J, Sudmant PH, Kidd JM, Li H, Kelley JL, Lorente-Galdos B, et al. (July 2013). "Great ape genetic diversity and population history". Nature. 499 (7459): 471–475. Bibcode:2013Natur.499..471P. doi:10.1038/nature12228. PMC 3822165. PMID 23823723.

- ^ Zeng J, Konopka G, Hunt BG, Preuss TM, Geschwind D, Yi SV (September 2012). "Divergent whole-genome methylation maps of human and chimpanzee brains reveal epigenetic basis of human regulatory evolution". American Journal of Human Genetics. 91 (3): 455–465. doi:10.1016/j.ajhg.2012.07.024. PMC 3511995. PMID 22922032.

추가열람
- Kellis M, Patterson N, Endrizzi M, Birren B, Lander ES (May 2003). "Sequencing and comparison of yeast species to identify genes and regulatory elements". Nature. 423 (6937): 241–254. Bibcode:2003Natur.423..241K. doi:10.1038/nature01644. PMID 12748633. S2CID 1530261.
- Cliften P, Sudarsanam P, Desikan A, Fulton L, Fulton B, Majors J, et al. (July 2003). "Finding functional features in Saccharomyces genomes by phylogenetic footprinting". Science. 301 (5629): 71–76. Bibcode:2003Sci...301...71C. doi:10.1126/science.1084337. PMID 12775844. S2CID 1305166.
- Boffelli D, McAuliffe J, Ovcharenko D, Lewis KD, Ovcharenko I, Pachter L, Rubin EM (February 2003). "Phylogenetic shadowing of primate sequences to find functional regions of the human genome". Science. 299 (5611): 1391–1394. doi:10.1126/science.1081331. PMID 12610304. S2CID 17217612.
- Dujon B, Sherman D, Fischer G, Durrens P, Casaregola S, Lafontaine I, et al. (July 2004). "Genome evolution in yeasts". Nature. 430 (6995): 35–44. Bibcode:2004Natur.430...35D. doi:10.1038/nature02579. PMID 15229592. S2CID 4399964.
- Filipski A, Kumar S (2005). "Comparative genomics in eukaryotes". In Gregory TR (ed.). The Evolution of the Genome. San Diego: Elsevier. pp. 521–583.
- Gregory TR, DeSalle R (2005). "Comparative genomics in prokaryotes". In Gregory TR (ed.). The Evolution of the Genome. San Diego: Elsevier. pp. 585–675.
- Xie X, Lu J, Kulbokas EJ, Golub TR, Mootha V, Lindblad-Toh K, et al. (March 2005). "Systematic discovery of regulatory motifs in human promoters and 3' UTRs by comparison of several mammals". Nature. 434 (7031): 338–345. Bibcode:2005Natur.434..338X. doi:10.1038/nature03441. PMC 2923337. PMID 15735639.
- Champ PC, Binnewies TT, Nielsen N, Zinman G, Kiil K, Wu H, et al. (March 2006). "Genome update: purine strand bias in 280 bacterial genomes". Microbiology. 152 (Pt 3): 579–583. doi:10.1099/mic.0.28637-0. PMID 16514138.
- Kumar L, Breakspear A, Kistler C, Ma LJ, Xie X (March 2010). "Systematic discovery of regulatory motifs in Fusarium graminearum by comparing four Fusarium genomes". BMC Genomics. 11: 208. doi:10.1186/1471-2164-11-208. PMC 2853525. PMID 20346147.

- Batzoglou S, Pachter L, Mesirov JP, Berger B, Lander ES (July 2000). "Human and mouse gene structure: comparative analysis and application to exon prediction". Genome Research. 10 (7): 950–958. doi:10.1101/gr.10.7.950. PMC 310911. PMID 10899144.

외부 링크
이 글의 외부 링크 사용은 위키백과의 정책이나 지침을 따르지 않을 수 있습니다.하거나 한 유용한 링크를 로 하여 본 해 주시기 (2017년 2월)( 메시지 방법 및 ) |
- 게놈 온라인 데이터베이스(GOLD)
- 지놈뉴스네트워크
- JCVI 종합 미생물 자원
- Pathema: Clade Specific Bioinformatics Resource Center
- CBS 게놈 아틀라스 데이터베이스
- UCSC 게놈 브라우저
- 미국 국립 인간 게놈 연구소
- Ensemble The Ensemble Genome Browser(유전체 탐색기 조립)
- 헤미아스코메테투스 효모의 유전체학, 비교유전체학
- 최근에 개발된 방법인 계통발생 추론 그룹(PhIGs)은 비교 유전체학에 사용하기 위해 유전자 클러스터를 구축하는 데 계통발생학적 신호를 통합합니다.
- 메타조메 2006-08-10 메타조안 유전자 패밀리의 계통발생학적 탐색 및 분석을 위한 자원인 웨이백 머신(Wayback Machine)에서 보관.
- IMG DOE-JGI에 의한 유전체 비교 분석을 위한 통합 미생물 유전체 시스템.
- Dcode.org Dcode.org Dcode.org 비교유전체학 센터
- 모든 완전 서열화된 유기체에 대한 SUPERFAMILY 단백질 주석
- 비교유전체학
- 블라스톨로지 및 오픈 소스:요구와 행동
- 정렬이 없는 비교 유전체학 도구


