효소억제제
Enzyme inhibitor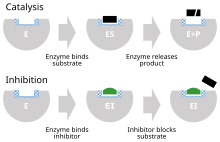
효소 억제제는 효소에 결합하고 그 활성을 차단하는 분자입니다.효소는 기질 분자가 생성물로 변환되는 생명에 필요한 화학 반응을 빠르게 하는 단백질입니다.[1]효소는 반응의 가장 어려운 단계를 가속화하는 효소의 전문 영역인 활성 부위에 기질을 결합시킴으로써 특정 화학 반응을 촉진합니다.
효소 억제제는 효소의 활성 부위에 결합함으로써(따라서 기질 자체가 결합하는 것을 막음으로써) 또는 효소의 촉매 작용이 차단되도록 효소의 다른 부위에 결합함으로써 이 과정을 중단("억제")시킵니다.효소 억제제는 가역적 또는 비가역적으로 결합할 수 있습니다.비가역적 억제제는 효소와 화학적 결합을 형성하여 화학적 결합이 깨질 때까지 효소가 억제됩니다.반대로, 가역적 억제제는 비공유적으로 결합하고 자발적으로 효소를 이탈하여 효소가 기능을 재개할 수 있습니다.가역적 억제제는 효소, 효소-기질 복합체 또는 둘 다와 결합하는지에 따라 다양한 유형의 억제를 생성합니다.
효소 억제제는 일반적으로 각각 하나의 효소에 특이적이고 그 효소의 활성을 조절하는 역할을 하기 때문에 모든 세포에서 중요한 역할을 합니다.예를 들어, 대사 경로의 효소는 경로의 나중에 생성되는 분자에 의해 억제되어 더 이상 필요하지 않은 분자의 생성을 줄일 수 있습니다.이런 부정적인 피드백은 세포의 균형을 유지하는 중요한 방법입니다.[2]효소 억제제는 또한 프로테아제나 뉴클레아제와 같은 필수적인 효소들을 조절하는데, 이는 만약 방치한다면 세포를 손상시킬 수도 있습니다.동물이나 식물이 만들어내는 많은 독들은 먹잇감이나 포식자의 중요한 효소의 활동을 막는 효소 억제제입니다.
많은 약물 분자들은 비정상적인 인간 효소나 바이러스, 박테리아, 기생충과 같은 병원체의 생존에 중요한 효소를 억제하는 효소 억제제입니다.예를 들면 메토트렉세이트(화학요법 및 류마티스 관절염 치료에 사용됨)와 HIV/AIDS 치료에 사용되는 프로테아제 억제제가 있습니다. 항병원성 억제제는 일반적으로 한 가지 효소만을 대상으로 하기 때문에, 유사한 효소가 인간에서 발견되지 않는 한, 그러한 약물은 매우 특이적이고 일반적으로 인간에게 거의 부작용을 일으키지 않습니다.(이러한 병원체와 사람은 유전적으로 멀리 떨어져 있기 때문에 종종 이런 경우가 있습니다.약용 효소 억제제는 종종 해리 상수가 낮은 경우가 있는데, 이는 효소를 억제하는 데 필요한 억제제의 양이 아주 적음을 의미합니다.효소 억제제의 농도가 낮으면 사람의 간과 신장 손상 및 기타 약물 부작용의 위험이 줄어듭니다.따라서 효소 억제제의 발견과 정제는 생화학과 약리학의 활발한 연구 분야입니다.
구조 클래스
효소 억제제는 유기 소분자에서 고분자 단백질에 이르기까지 크기가 다양한 화학적으로 다양한 물질들의 집합입니다.
소분자 억제제는 이러한 대사물질을 생성하는 상류 효소를 억제하는 필수적인 1차 대사물질을 포함합니다.이것은 대사 물질의 과잉 생산을 방지하고 따라서 세포 항상성을 유지하는 음의 피드백 루프를 제공합니다.[3][2]소분자 효소 억제제는 2차 대사산물도 포함하는데, 2차 대사산물은 그것을 생산하는 유기체에 필수적이지는 않지만 포식자나 경쟁하는 유기체를 물리치거나 먹이를 고정시키는 데 사용될 수 있다는 점에서 유기체에 진화적 이점을 제공합니다.[4]또한, 많은 약물들은 환자의[1]: 5 질병 변형 효소 또는 병원체의 성장과 번식에 필요한 병원체의 효소를 표적으로 하는 소분자 효소 억제제입니다.[5]
작은 분자 외에도, 몇몇 단백질들은 효소 억제제 역할을 합니다.가장 대표적인 예는 부적절한 효소의 활성화를 보호하기 위해 동물이 생산하고 포식을 방지하기 위해 식물이 생산하는 서핀(세린 프로테아제 억제제)입니다.[6]억제제 단백질의 또 다른 종류는 리보뉴클레아제 억제제인데, 리보뉴클레아제는 가장 엄격한 단백질-단백질 상호작용 중 하나에서 리보뉴클레아제에 결합합니다.[7]단백질 효소 억제제의 특별한 경우는 조절되지 않는 촉매 작용에 대한 보호 메커니즘으로서 활성을 분자 내로 차단하는 효소의 활성 부위에 결합하는 자동 억제 N 말단 펩타이드를 포함하는 지모겐입니다.N 말단 펩타이드는 지모겐 효소 전구체로부터 다른 효소에 의해 분해(분열)되어 활성 효소를 방출합니다.[8]
효소에 대한 억제제의 결합 부위는 가장 일반적으로 효소의 기질을 결합하는 부위와 동일합니다.이러한 활성 부위 억제제는 오르토스테릭("정규" 방향) 억제제로 알려져 있습니다.[9]오르토스테릭 억제의 메커니즘은 단순히 직접적인 경쟁을 통해 효소에 기질이 결합하는 것을 막는 것이며, 이것은 다시 효소가 기질을 생성물로 전환시키는 것을 촉매하는 것을 막습니다.또는 억제제는 효소 활성 부위에서 멀리 떨어진 부위에 결합할 수 있습니다.이들은 알로스테릭("대체" 방향") 억제제로 알려져 있습니다.[9]알로스테릭 억제의 메커니즘은 다양하며, 효소가 더 이상 기질을 결합할 수 없도록 효소의 형태(형태)를 변경하거나(경쟁 오르토스테릭 억제와 역학적으로 구별할 수 없도록),[10] 또는 대안적으로 효소에 기질의 결합을 안정화하지만 더 이상 카타가 아닌 형태로 효소를 가두는 것을 포함합니다.유리질의[11]
가역억제제
가역적 억제제는 수소 결합, 소수성 상호 작용 및 이온 결합과 같은 비공유 상호 작용을 갖는 효소에 부착됩니다.[12]억제제와 효소 활성 부위 사이의 다수의 약한 결합이 결합하여 강하고 특이적인 결합을 생성합니다.
비가역적 억제제와 대조적으로 가역적 억제제는 일반적으로 효소에 결합될 때 화학 반응을 겪지 않으며 희석 또는 투석에 의해 쉽게 제거될 수 있습니다.특별한 경우는 효소와 화학적 결합을 형성하는 공유 가역 억제제이지만, 결합은 분해될 수 있으므로 억제는 완전히 가역적입니다.[13]
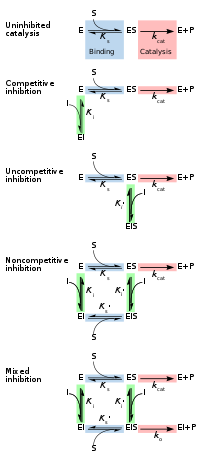
가역적 억제제는 일반적으로 4가지 유형으로 분류되는데, 1963년 Cleland에 의해 도입되었습니다.[14]이들은 효소의 기질 농도가 다양함에 따라 저해제가max V(효소에 의해 촉매되는 최대 반응 속도m)와 K(기질의 농도가 반 최대 효소 활성을 나타내는)에 미치는 영향에 따라 분류됩니다.[15][16]
경쟁적인
경쟁적 억제에서 기질과 억제제는 효소에 동시에 결합할 수 없습니다.[17]: 134 이는 일반적으로 기질이 결합하는 효소의 활성 부위에 대한 친화력을 갖는 억제제에서 비롯됩니다. 기질과 억제제는 효소의 활성 부위에 대한 접근을 위해 경쟁합니다.이러한 유형의 억제는 충분히 높은 농도의 기질(V는max 일정하게 유지됨), 즉 억제제와 경쟁함으로써 극복될 수 있습니다.[17]: 134–135 그러나 K 지점m, 즉 V의max 절반에 도달하기 위해서는 기판의 농도가 높아지기 때문에 겉보기 K는m 증가할 것입니다.경쟁 억제제는 종종 실제 기질과 구조가 유사합니다(예: "약물" 섹션의 "메토트렉세이트 대 엽산" 그림 참조).[17]: 134
경쟁력 없음
비경쟁적 억제에서 억제제는 효소-기질 복합체에만 결합합니다.[17]: 139 이러한 유형의 억제는 V를max 감소시키고 (활성화된 복합체를 제거한 결과로 최대 속도가 감소함) K를m 감소시킵니다 (르 샤틀리에 원리의 결과로 결합 효율이 향상되고 ES 복합체의 효과적인 제거로 인해 결합 친화도가 더 높은 K가 감소함m).[18]비경쟁적인 억제는 거의 없습니다.[17]: 139 [19]
비경쟁
비경쟁적 억제에서 효소에 대한 억제제의 결합은 그 활성을 감소시키지만 기질의 결합에는 영향을 미치지 않습니다.[16]결과적으로 억제의 정도는 억제제의 농도에 따라서만 달라집니다.V는max 반응이 효율적으로 진행될 수 없기 때문에 감소하겠지만, K는m 정의상 기판의 실제 결합과 동일하게 유지될 것입니다.[20]
혼합된
혼합 억제에서 억제제는 기질이 이미 결합했든 안했든 간에 효소에 결합할 수 있습니다.따라서 혼합 억제는 경쟁적 억제와 비경쟁적 억제의 조합입니다.[16]또한, 유리 효소 및 효소-기질 복합체에 대한 저해제의 친화도는 상이할 수 있습니다.[17]: 136–139 기판 [S]의 농도를 증가시킴으로써, 이러한 유형의 억제는 (경쟁적 기여로 인해) 감소될 수 있지만 (비경쟁적 구성요소로 인해) 완전히 극복되지는 않습니다.[21]: 381–382 비록 혼합형 억제제가 활성 부위에서 결합하는 것은 가능하지만, 이러한 유형의 억제는 일반적으로 억제제가 효소의 다른 부위에 결합하는 알로스테릭 효과에서 비롯됩니다.이 알로스테릭 부위에 결합하는 억제제는 활성 부위에 대한 기질의 친화도가 감소하도록 효소의 입체 구조(즉, 3차 구조 또는 3차원 형상)를 변화시킵니다.[22]
이들 4가지 유형의 억제는 또한 주어진 양의 억제제에 의해 야기되는 억제 정도에 대한 기질 농도 [S]의 증가 효과에 의해 구별될 수 있습니다.경쟁적 억제의 경우에는 [S]를 증가시킴으로써 억제 정도가 감소하고, 비경쟁적 억제의 경우에는 억제 정도가 변하지 않으며, 비경쟁적(반경쟁적) 억제의 경우에는 [S]에 따라 억제 정도가 증가합니다.[23]
정량적 기술
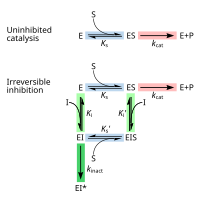
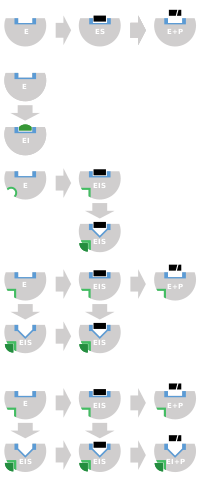
가역적 억제는 효소와 효소-기질 복합체에 대한 억제제의 결합 및 효소의 운동 상수에 대한 효과 측면에서 정량적으로 설명될 수 있습니다.[24]: 6 고전적인 Michaelis-Menten 방식("금지 메커니즘 도식" 다이어그램에 나타남)에서 효소(E)는 기질(S)에 결합하여 효소-기질 복합체 ES를 형성합니다.촉매 작용을 하면 이 복합체는 분해되어 생성물 P와 유리효소를 방출합니다.[24]: 55 저해제(I)는 각각 해리 상수 Ki 또는 Ki'로 E 또는 ES 중 하나에 결합할 수 있습니다.[24]: 87
- 경쟁 억제제는 E에는 결합할 수 있지만 ES에는 결합할 수 없습니다.경쟁적 억제는 K를m 증가시키지만(즉, 억제제는 기질 결합을 방해하지만) V에는max 영향을 미치지 않습니다(억제제는 ES에 결합할 수 없기 때문에 ES의 촉매 작용을 방해하지 않습니다).[24]: 102
- 경쟁력이 없는 억제제는 ES에 결합합니다.비경쟁적 억제는 K와m V를max 모두 감소시킵니다.저해제는 기질에 대한 효소 친화도를 증가(K 감소m)시키고 촉매 작용을 방해(Vmax 감소)함으로써 기질 결합에 영향을 미칩니다.[24]: 106
- 비경쟁 억제제는 E와 ES에 대해 동일한 친화도를 갖습니다(K = K').비경쟁적 억제는 K를m 변화시키지 않고(즉, 기질 결합에 영향을 미치지 않음) V를max 감소시킵니다(즉, 억제제 결합 장애물 촉매 작용).[24]: 97
- 혼합형 억제제는 E와 ES에 모두 결합하지만, 이 두 형태의 효소에 대한 친화도는 다릅니다(K ≠ K').따라서, 혼합형 억제제는 기질 결합(증감 또는 감소 Km)에 영향을 미치고, ES 복합체에서 촉매작용(감소 Vmax)을 방해합니다.[25]: 63–64
효소가 여러 개의 기질을 가지고 있을 때, 억제제는 어떤 기질을 고려하느냐에 따라 다른 종류의 억제를 보일 수 있습니다.이는 활성 부위 내에 두 개의 서로 다른 결합 부위를 포함하는 활성 부위에서 기인하며, 각 기질마다 하나씩 있습니다.예를 들어, 억제제는 첫 번째 결합 부위를 위해 기질 A와 경쟁할 수 있지만, 두 번째 결합 부위에서 기질 B와 관련하여 비경쟁적인 억제제가 될 수 있습니다.[26]
전통적으로 가역적 효소 억제제는 K와m V에max 미치는 영향에 따라 경쟁적, 비경쟁적 또는 비경쟁적으로 분류되어 왔습니다.[14]이 세 가지 유형의 억제는 각각 기질 S가 없는 상태에서 효소 E에만 결합하는 억제제, 효소-기질 복합체 ES 또는 둘 모두에서 발생합니다.이러한 클래스의 구분은 파생 문제에서 발생하며 하나의 결합 이벤트에 대해 두 개의 서로 다른 결합 상수를 사용해야 하는 결과를 초래합니다.[27]또한 효소에 대한 억제제의 결합은 100% 억제를 초래하고 부분적인 억제의 가능성을 고려하지 못하는 것으로 추정됩니다.[27]억제 용어의 일반적인 형태는 또한 효소에 결합하는 억제제와 리간드 수용체 결합과 관련된 미카엘리스-멘텐 방정식 또는 용량 반응 곡선과 같은 다른 결합 용어와의 관계를 모호하게 합니다.이 관계를 입증하기 위해 다음과 같이 재정렬할 수 있습니다.[28]
이 재배열은 미카엘리스-멘텐 방정식과 유사하게 최대 반응 속도는 기질과 상호 작용하는 효소 개체군의 비율에 따라 달라진다는 것을 보여줍니다.
기질에 의해 결합된 효소군의 분율
억제제에 의해 결합된 효소군의 분율
억제제의 효과는 억제제와 상호작용하는 효소 인구의 백분율의 결과입니다.현재 형태에서 이 방정식의 유일한 문제는 억제제 결합을 가진 효소의 절대적인 억제를 가정한다는 것입니다. 실제로 기질 전환의 100% 억제에서 무 억제까지 광범위한 효과가 있을 수 있습니다.이를 설명하기 위해 방정식을max 델타 V 항을 포함하여 다른 정도의 억제를 허용하도록 쉽게 수정할 수 있습니다.[29]: 361
아니면
이 용어는 억제제가 모집단의 개별 효소와 상호 작용할 때 존재하는 잔류 효소 활성을 정의할 수 있습니다.그러나 이 항을 포함하는 것은 보조max V 항이 초기 항보다 높은 것으로 판명될 경우 활성화 가능성을 허용하는 부가가치가 있습니다.활성화 가능성을 설명하기 위해, 여기서 "X"로 표시되는 수식어 용어(자극기 또는 억제기)로 억제기 "I"를 대체하여 표기를 다시 쓸 수 있습니다.[28]: eq 13
이 용어는 미카엘리스-멘텐 방정식의 최대 속도와 관련된 운동 효과를 다루는 단순화된 방식을 초래하지만, K와m 관련된 효과를 설명하는 데 사용되는 용어로 잠재적인 문제를 강조합니다.기질에 대한 효소의 친화도와 관련된 K는m 대부분의 경우 효소 억제제 상호작용으로 직접적으로 발생하는 효소의 결합 부위의 잠재적인 변화와 관련이 있어야 합니다.V를max 변조하기 위해 위에서 제안한 델타 Vmax 항과 유사한 용어는 대부분의 상황에서 적절해야 합니다.[28]: eq 14
해리 상수
![2D plots of 1/[S] concentration (x-axis) and 1/V (y-axis) demonstrating that as inhibitor concentration is changed, competitive inhibitor lines intersect at a single point on the y-axis, non-competitive inhibitors intersect at the x-axis, and mixed inhibitors intersect a point that is on neither axis](http://upload.wikimedia.org/wikipedia/commons/thumb/b/ba/Inhibition_diagrams-1-.png/220px-Inhibition_diagrams-1-.png)
효소 억제제는 억제제의 절반이 효소를 차지하는 농도인 해리 상수 K를i 특징으로 합니다.비경쟁적 저해에서 저해제는 또한 효소-기질 복합체에 결합할 수 있고, 결합된 기질의 존재는 효소에 대한 저해제의 친화도를 변화시켜 두 번째 해리 상수 Ki'를 생성할 수 있습니다.따라서 K와i Ki'는 각각 효소에 대한 억제제와 효소-기질 복합체에 대한 해리 상수입니다.[30]: Glossary 효소-억제제 상수 K는i 다양한 방법으로 직접 측정될 수 있는데, 특히 정확한 방법은 효소 용액에 억제제를 적정하고 방출되거나 흡수된 열을 측정하는 등온 적정 열량 측정법입니다.[31]그러나 다른 하나의 해리i 상수 K'는 효소-기질 복합체가 수명이 짧고 생성물을 형성하기 위한 화학 반응을 거치기 때문에 직접 측정하기가 어렵습니다.따라서 K'는i 다양한 기질과 억제제 농도 하에서 효소 활성을 관찰하고 비선형[32] 회귀를 통해 데이터를 수정된 미카엘리스-멘텐 방정식에 맞추어 간접적으로 측정됩니다.[21]
여기서 변형인자 α와 α'는 억제제 농도와 그것의 두 해리 상수에 의해 정의됩니다.
따라서, 저해제가 존재하는 경우, 효소의 유효 K와m V는max 각각 (α/α')K와m (1/α')V가max 됩니다.그러나 수정된 Michaelis-Menten 방정식은 효소에 대한 억제제의 결합이 평형에 도달했다고 가정하는데, 이는 아나노몰 해리 상수를 갖는 억제제에 대해 매우 느린 과정일 수 있습니다.이러한 경우에는 억제가 효과적으로 비가역적이 되므로, 이러한 엄격한 결합 억제제를 비가역적으로 취급하는 것이 더 실용적입니다(아래 참조).
효소 활성에 대한 다양한 유형의 가역적 효소 억제제의 효과는 라인위버-버크, 이디-호프스티 또는 하네스-울프 플롯과 같은 마이클리스-멘텐 방정식의 그래픽 표현을 사용하여 시각화할 수 있습니다.[17]: 140–144 그림은 라인위버-버크 다이어그램 그림에 표시된 세 개의 라인위버-버크 그림에 의해 제공됩니다.위 도표에서 경쟁 억제선은 y축에서 교차하며, 이러한 억제선은 V에max 영향을 미치지 않음을 나타냅니다.아래 도표에서 비경쟁 억제선은 x축에서 교차하며, 이 억제선들이 K에m 영향을 미치지 않음을 보여줍니다.그러나 이러한 그림에서 K와i Ki'를 정확하게 추정하기 어려울 수 있으므로 보다 신뢰할 수 있는 비선형 회귀 분석 방법을 사용하여 이러한 상수를 추정하는 것이 좋습니다.[33][33]
특수한 경우
부분 경쟁력
부분 경쟁 억제 메커니즘은 EIS 복합체가 효소-기질(ES) 복합체보다 낮거나 심지어 더 높을 수 있는 촉매 활성을 가지고 있다는 점을 제외하고는 비경쟁적 억제 메커니즘과 유사합니다.이 억제는 일반적으로 V는max 낮지만 K 값에는m 영향을 주지 않습니다.[18]
기판 또는 제품
기질 또는 생성물 억제는 효소 기질 또는 생성물이 또한 억제제 역할을 하는 곳입니다.이러한 억제는 경쟁적이거나 비경쟁적이거나 혼합적인 패턴을 따를 수 있습니다.기질 억제에서 잠재적으로 두 개의 경쟁 기질 결합 부위를 가진 효소로부터 높은 기질 농도에서 점진적인 활성 감소가 있습니다.저기판에서는 고친화성 부위가 점유되고 정상적인 동역학이 따릅니다.그러나 고농도에서는 두 번째 억제 부위가 점유되어 효소를 억제합니다.[34]생성물 억제(효소 자체 생성물 또는 대사 경로의 하류에 있는 효소로의 생성물)는 종종 대사의 조절 특징이며 부정적인 피드백의 한 형태가 될 수 있습니다.[2]
슬로우-타이트
느린 엄격한 억제는 초기 효소-억제제 복합체 EI가 두 번째로 더 엄격하게 유지되는 복합체 EI*로 입체 이성질화(모양의 변화)를 겪지만 전체적인 억제 과정은 가역적일 때 발생합니다.이것은 효소 억제를 천천히 증가시키는 것으로 나타납니다.이러한 조건에서 전통적인 미카엘리스-멘텐 역학은 시간에 의존적인 K에i 대해 잘못된 값을 부여합니다.K의i 참값은 비가역적 억제와 유사한 운동학과의 억제제 연관성에 대한 온(kon) 및 오프(k) 속도off 상수의 보다 복잡한 분석을 통해 얻을 수 있습니다.[17]: 168
다중기판 아날로그
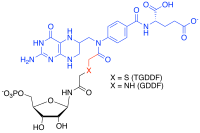
다중-기질 유사체 억제제는 하나의 분자 내에 각각의 기질의 결합 에너지를 포획함으로써 하나 이상의 기질과의 반응을 촉매하는 효소에 대해 제조될 수 있는 고친화성 선택적 억제제.[35][36]예를 들어, 퓨린 생합성의 포밀 전달 반응에서, GAR 기질과 N-10-포밀 테트라하이드로폴산 보조 인자의 유사체를 함께 연결하여 티오글리신아마이드 리보뉴클레오티드 디아자폴레이트(TGDDF)를 생성함으로써, GAR(Glycinamide Libonucleotide)에 대한 강력한 다중-기질 부가물 억제제(MAI) TFase가 합성적으로 제조되었다,[37]또는 천연 GAR 기질로부터 효소적으로 GDDF를 생성합니다.[38]여기서 TGDDF의 서브나노몰 해리 상수(KD)는 구성 요소를 연결하는 원자를 통해 얻은 엔트로픽 이점 및/또는 양의 상호 작용으로 인해 예상보다 컸습니다.MAI는 또한 이소니아지드[39](isoniazid) 또는 효소 억제제 리간드([40]예를 들어, PTC124)와 같은 프로-약물과 각각 니코틴아마이드 아데닌 다이뉴클레오티드(NADH) 및 아데노신 삼인산(ATP)과 같은 세포 보조 인자의 반응에 의해 세포에서 생성되는 것으로 관찰되었습니다.[41]
예
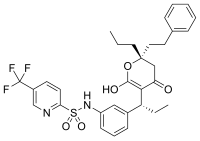
효소들이 그들의 기질을 단단히 결합하도록 진화하고, 대부분의 가역적인 억제제들이 효소의 활성 부위에서 결합하기 때문에, 이러한 억제제들 중 일부가 그들의 표적의 기질과 구조가 현저하게 유사하다는 것은 놀라운 일이 아닙니다.디하이드로폴산 환원효소(dihydrofolate reductase, DHFR)의 억제제가 대표적인 예입니다.[42]이러한 기질 모방의 다른 예로는 HIV/AIDS 치료에 사용되는 치료적으로 효과적인 항레트로바이러스제 부류인 프로테아제 억제제가 있습니다.[43][44]3개의 펩타이드 결합을 포함하는 펩티도미메틱(펩타이드 모방) 프로테아제 억제제인 리토나비르의 구조는, 상기 "경쟁 억제" 그림에 나타낸 바와 같습니다.이 약은 HIV 프로테아제의 기질인 펩타이드와 유사하기 때문에 효소의 활성 부위에서 기질과 경쟁하게 됩니다.[45]
효소 억제제는 종종 효소 촉매 반응의 전이 상태 또는 중간체를 모방하도록 설계됩니다.[46]이것은 억제제가 효소의 전이 상태 안정화 효과를 이용하여 기질 기반 설계보다 더 나은 결합 친화도(낮은 K)를 갖도록i 보장합니다.그러한 전이 상태 억제제의 예는 항바이러스제인 오셀타미비르입니다; 이 약은 바이러스 효소인 뉴라미니다제의 반응에서 고리 옥소늄 이온의 평면적 성질을 모방합니다.[47]
그러나 모든 억제제가 기질의 구조에 기초하는 것은 아닙니다.예를 들어, 또 다른 HIV 프로테아제 억제제 티프라나비르의 구조는 펩타이드를 기반으로 하지 않으며 단백질 기질과 뚜렷한 구조적 유사성이 없습니다.펩티다아제의 기질이 아닐 것이고 분해될 가능성이 적기 때문에 이러한 비펩티다아제 억제제는 펩티다아제를 포함하는 억제제보다 더 안정적일 수 있습니다.[48]
약물 설계에서는 표적 효소가 노출되는 기질의 농도를 고려하는 것이 중요합니다.예를 들어, 일부 단백질 키나아제 억제제는 이러한 효소의 기질 중 하나인 ATP와 유사한 화학적 구조를 가지고 있습니다.[49]하지만 단순한 경쟁 억제제인 약물은 세포 내의 높은 농도의 ATP와 경쟁해야 할 것입니다.단백질 키나아제는 또한 그들의 기질 단백질과 상호작용하는 결합 부위에서 경쟁에 의해 억제될 수 있고, 대부분의 단백질은 ATP의 농도보다 훨씬 낮은 농도로 세포 내부에 존재합니다.결과적으로 두 단백질 키나아제 억제제가 유사한 친화성을 가지고 활성 부위에서 결합하지만, 단 하나만 ATP와 경쟁하면 된다면, 단백질 결합 부위의 경쟁 억제제는 효소를 더 효과적으로 억제할 것입니다.[50]
비가역억제제
종류들
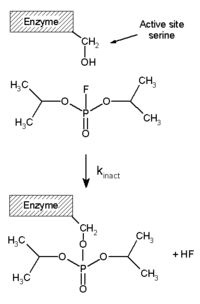
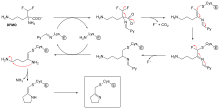
비가역적 억제제는 효소에 공유 결합하고, 따라서 이러한 종류의 억제는 쉽게 되돌릴 수 없습니다.[51]비가역적 억제제는 종종 질소 머스타드, 알데하이드, 할로알칸, 알켄, 마이클 억셉터, 페닐 설포네이트 또는 플루오로포스폰네이트와 같은 반응성 작용기를 포함합니다.[52]이러한 친전자성 그룹은 아미노산 곁사슬과 반응하여 공유 부가물을 형성합니다.[51]변형된 잔기는 히드록실 또는 설프히드릴기와 같은 친핵체를 포함하는 측쇄를 갖는 잔기이며, 이러한 잔기는 세린(DFP와 반응하는 아미노산 세린, "DFP 반응" 다이어그램 참조) 및 시스테인, 트레오닌 또는 티로신을 포함합니다.[53]
비가역적 억제는 비가역적 효소의 불활성화와는 다릅니다.[54]비가역적 억제제는 일반적으로 한 종류의 효소에 특이적이며 모든 단백질을 불활성화하지는 않습니다; 그들은 단백질 구조를 파괴함으로써 기능하지 않고 그들의 표적의 활성 부위를 특별히 변경시킴으로써 기능합니다.예를 들어, pH나 온도의 극단은 일반적으로 모든 단백질 구조의 변성을 일으키지만, 이것은 비특이적인 효과입니다.마찬가지로, 일부 비특이적인 화학적 처리는 단백질 구조를 파괴합니다: 예를 들어, 농축된 염산에서 가열하면 단백질을 결합하는 펩타이드 결합이 가수분해되어 유리 아미노산이 방출됩니다.[55]
비가역적 억제제는 시간 의존적 억제를 나타내므로 IC50 값으로 그 효력을 나타낼 수 없습니다.주어진 농도의 비가역억제제에서 활성효소의 양은 억제제가 효소와 함께 얼마나 오랫동안 사전 배양되느냐에 따라 다를 것이기 때문입니다.대신 kobs/[I] 값이 사용되며,[56] 여기서 k는obs 관측된 의사 1차 비활성화 속도(시간 대비 % 활성의 로그를 표시하여 얻은 값)이고 [I]는 억제제의 농도입니다.k/[I] 파라미터는 억제제가 효소와의 결합을 포화시키지 않는 한(이 경우 k = k), 여기서 k는 비활성화 속도입니다.
측정
비가역적 억제제는 먼저 효소(EI 또는 ESI)와 가역적 비공유 복합체를 형성합니다.이어서 효소와 억제제 사이에 화학 반응이 일어나 공유결합으로 변형된 "막다른 복합체" EI*(불역 공유결합 복합체)를 생성합니다.EI*가 형성되는 속도를 비활성화 속도 또는 k라고inact 합니다.[13]EI의 형성은 ES와 경쟁할 수 있기 때문에, 비가역적 억제제의 결합은 기질과의 경쟁 또는 두 번째 가역적 억제제와의 경쟁에 의해 방지될 수 있습니다.이러한 보호 효과는 비가역적 억제제가 활성 부위와 특정한 반응을 보이는 좋은 증거입니다.
이 반응의 결합과 비활성화 단계는 효소를 억제제와 함께 배양하고 시간이 지남에 따라 남아있는 활성의 양을 분석함으로써 조사됩니다.활동량은 시간에 따라 감소하며, 보통 지수적으로 감소합니다.이러한 데이터를 속도 방정식에 적합시키면 억제제 농도에서 비활성화되는 속도를 얻을 수 있습니다.이 작업은 여러 가지 다른 농도의 억제제에서 수행됩니다.가역적 EI 복합체가 관련된 경우 비활성화 속도가 포화할 수 있고 이 곡선에 적합하면 k와inact K가i 됩니다.[57]
이러한 분석에 널리 사용되는 또 다른 방법은 질량 분석법입니다.여기서 변형되지 않은 천연 효소와 불활성화 효소의 질량을 정확하게 측정하면 억제제와의 반응에 의한 질량 증가를 얻을 수 있고 반응의 화학양론을 보여줍니다.[58]이 작업은 일반적으로 MALDI-TOF 질량분석기를 사용합니다.[59]보완적인 기술에서, 펩타이드 매스 핑거프린팅은 트립신과 같은 단백질 분해효소로 고유 단백질과 변형 단백질을 소화시키는 것을 포함합니다.이것은 질량 분석계를 사용하여 분석할 수 있는 펩티드 세트를 만들어 낼 것입니다.억제제와 반응한 후 질량이 변하는 펩타이드는 변형 부위를 포함하는 펩타이드일 것입니다.[60]
느린 바인딩
모든 비가역적 억제제들이 그들의 효소 표적과 공유 부가물들을 형성하는 것은 아닙니다.일부 가역적 억제제는 그들의 표적 효소에 매우 단단히 결합하여 본질적으로 비가역적입니다.이러한 엄격한 결합 억제제는 공유 비가역 억제제와 유사한 운동학을 보일 수 있습니다.이러한 경우 이러한 억제제 중 일부는 저친화성 EI 복합체에서 효소에 빠르게 결합하고 이후 매우 단단히 결합된 EI* 복합체로 더 느리게 재배치됩니다("돌이킬 수 없는 억제 메커니즘" 다이어그램 참조).이러한 운동 동작을 느린 결합이라고 합니다.[62]결합 후의 이 느린 재배열은 종종 효소가 억제제 분자 주위에 "밀어 붙으면서" 입체구조의 변화를 수반합니다.느린 결합 억제제의 예로는 메토트렉세이트,[63] 알로푸리놀 [64]및 활성화된 형태의 아시클로비르와 같은 일부 중요한 약물이 있습니다.[65]
몇가지 예시들
다이소프로필플루오로포스페이트(DFP)는 비가역적 프로테아제 저해제의 한 예입니다("DFP 반응" 다이어그램 참조).효소는 인-불소 결합을 가수분해하지만, 인산 잔류물은 활성 부위의 세린에 결합된 상태로 남아 있어 비활성화됩니다.[67]유사하게, DFP는 또한 뉴런의 시냅스에서 아세틸콜린 에스터레이스의 활성 부위와 반응하고, 결과적으로 치명적인 용량이 100 mg 미만인 강력한 신경독소입니다.[68]
자살 억제는 효소가 활성 부위에서 억제제를 반응성 형태로 바꾸는 비정상적인 비가역적 억제입니다.[69]폴리아민 생합성 억제제인 α-디플루오로메틸오르니틴(DFMO)은 아미노산 오르니틴과 유사하며 아프리카 트리파노소미아증(수면 질환)을 치료하는 데 사용됩니다.오르니틴 디카르복실화효소는 오르니틴 대신 DFMO의 디카르복실화를 촉매할 수 있습니다("DFMO 억제 메커니즘" 다이어그램 참조).그러나, 이 탈카복실화 반응은 불소 원자의 제거에 의해 뒤따르고, 이는 이 촉매 중간체를 매우 친전자성이 높은 종인 공액 이민으로 전환시킵니다.DFMO의 이러한 반응성 형태는 활성 부위의 시스테인 또는 라이신 잔기와 반응하여 효소를 비가역적으로 불활성화시킵니다.[61]
비가역적 억제는 종종 비공유 효소 억제제(EI) 복합체의 초기 형성을 수반하기 때문에,[13] 때때로 억제제가 한 가지 이상의 방법으로 효소에 결합하는 것이 가능합니다.예를 들어, 인간 원생동물 기생충 트리파노소마 크루지(Trypanosoma cruzi)로부터의 트리파노티온 환원효소를 보여주는 그림에서, 퀴나크린 머스타드(quinacrine mustard)라고 불리는 억제제의 두 분자가 그 활성 부위에 결합되어 있습니다.위쪽 분자는 가역적으로 결합되어 있지만, 아래쪽 분자는 질소 머스타드 그룹을 통해 아미노산 잔기와 반응하여 공유 결합되어 있습니다.[70]
적용들
효소 억제제는 자연에서[71] 발견되며 실험실에서 인공적으로 생산되기도 합니다.[72]자연적으로 발생하는 효소 억제제는 많은 대사 과정을 조절하고 삶에 필수적입니다.[3][1]또한, 자연적으로 생성된 독은 포식자, 먹이 및 경쟁 생물에 대한 독성 작용제로 사용하기 위해 진화한 효소 억제제입니다.[4]이러한 천연 독소에는 알려진 가장 독성이 강한 물질들이 포함되어 있습니다.[73]인공 억제제는 종종 약물로 사용되지만 말라티온과 같은 살충제, 글리포세이트와 같은 제초제 [74]또는 트리콜산과 같은 소독제가 될 수도 있습니다.다른 인공 효소 억제제는 아세틸콜린을 분해하는 효소인 아세틸콜린에스테라아제를 차단하고 화학전에서 신경작용제로 사용됩니다.[75]
대사조절
효소 억제는 세포에서 대사 경로 조절의 공통적인 특징입니다.[3]경로를 통한 대사 흐름은 종종 동일한 경로의 효소에 대한 억제제 및 증진제 역할을 하는 경로의 대사 물질에 의해 조절됩니다.당분해 경로는 전형적인 예입니다.[76]이 이화작용 경로는 포도당을 소비하고 ATP, NADH 및 피루브산을 생성합니다.해당과정의 조절을 위한 핵심 단계는 포스포프럭토키네이스-1(PFK1)에 의해 촉매되는 경로에서의 초기 반응입니다.ATP 수치가 상승하면 ATP는 PFK1의 알로스테릭 부위에 결합하여 효소 반응 속도를 감소시키고, 해당과정은 억제되어 ATP 생성이 떨어집니다.이러한 음성 피드백 조절은 세포 내 ATP의 일정한 농도를 유지하도록 돕습니다.그러나 효소의 활성화도 마찬가지로 중요하기 때문에 대사경로는 단지 억제를 통해 조절되지 않습니다.PFK1과 관련하여 과당 2,6-이중인산 및 ADP는 알로스테릭 활성화제인 대사산물의 예입니다.[77]
생리적 효소 억제는 특정 단백질 억제제에 의해서도 생성될 수 있습니다.이 메커니즘은 지모겐으로 알려진 많은 소화 전구 효소를 합성하는 췌장에서 발생합니다.이 중 많은 부분이 트립신 프로테아제에 의해 활성화되므로 장기 자체가 소화되지 않도록 췌장에서 트립신의 활동을 억제하는 것이 중요합니다.트립신의 활성이 조절되는 한 가지 방법은 췌장에서 특이적이고 강력한 트립신 억제 단백질의 생성입니다.이 억제제는 트립신에 단단히 결합하여 그렇지 않으면 장기에 해로울 수 있는 트립신의 활성을 막습니다.[78]트립신 억제제는 단백질이지만, 트립신의 활성 부위에서 물을 배제하고 전이 상태를 불안정하게 하여 단백질 분해효소에 의해 기질로 가수분해되는 것을 방지합니다.[79]생리적 효소 억제제 단백질의 다른 예로는 박테리아 리보뉴클레아제 바르나아제의 바스타 억제제가 있습니다.[80]
천연독

동물과 식물은 2차 대사산물,[81] 펩타이드 및 억제제 역할을 할 수 있는 단백질을[82] 포함한 광범위한 독성 제품을 합성하도록 진화해 왔습니다.천연 독소는 보통 작은 유기 분자이고 매우 다양해서 아마도 대부분의 대사 과정에 천연 억제제가 있을 것입니다.[83]자연독이 목표로 하는 대사 과정은 대사 경로의 효소 이상을 포함하며 세포에서 수용체, 채널 및 구조 단백질 기능의 억제도 포함할 수 있습니다.예를 들어, 태평양 주목 나무에서 발견되는 유기 분자인 paclitaxel (taxol)은 tubulin 이량체에 단단히 결합하고 세포골격에서 미세한 튜브로 그것들이 조립되는 것을 억제합니다.[84]
많은 천연 독들은 마비를 일으켜 죽음에 이르게 할 수 있는 신경독의 역할을 하며 포식자로부터 방어하거나 사냥과 사냥에서 기능을 합니다.이러한 자연 억제제 중 일부는 [85]독성 특성에도 불구하고 낮은 용량으로 치료에 유용합니다.[86]신경독의 예는 아세틸콜린에스테라아제 억제제인 솔라나과(감자, 토마토, 가지 포함) 식물종의 글리코알칼로이드입니다.이 효소의 억제는 아세틸콜린 신경전달물질의 조절되지 않는 증가, 근육 마비, 그리고 나서 사망을 야기합니다.신경독성은 수용체의 억제로 인해 발생할 수도 있습니다. 예를 들어 무스카린 아세틸콜린 수용체의 경쟁적 길항제 역할을 하는 치명적인 밤 그늘(아트로파 벨라도나)의 아트로핀입니다.[87]
비록 많은 천연 독소들이 2차 대사산물이지만, 이러한 독들은 펩티드와 단백질도 포함합니다.독성이 있는 펩타이드의 예로는 알파-아마니틴이 있는데, 이것은 죽음의 모자 버섯의 친척에게서 발견됩니다.이것은 RNA 중합효소 II 효소가 DNA를 전사하는 것을 막는 강력한 효소 억제제입니다.[88]알갈 독소 마이크로시스틴은 또한 펩타이드이며 단백질 포스파타아제의 억제제입니다.[89]이 독소는 녹조 발생 후 상수도를 오염시킬 수 있으며, 또한 급성 간출혈과 고용량 사망을 유발할 수 있는 알려진 발암물질입니다.[90]
단백질은 일부 콩류에서 발견되는 트립신 억제제(위의 "대사 조절" 부분에서 설명)와 같은 천연 독 또는 항영양제일 수도 있습니다.[91]덜 흔한 종류의 독소는 독성 효소입니다: 이것들은 그들의 표적 효소의 비가역적 억제제로서 작용하고 그들의 기질 효소를 화학적으로 변형시킴으로써 작용합니다.한 예로 피마자 기름 콩에서 발견되는 매우 강력한 단백질 독소인 리신이 있습니다.[92]이 효소는 리보솜을 불활성화시키는 글리코시다제입니다.[93]리신은 촉매 비가역 억제제이기 때문에, 이것은 단지 한 분자의 리신이 세포를 죽이는 것을 가능하게 합니다.[94]
마약

효소 억제제의 가장 일반적인 용도는 질병을 치료하기 위한 약물입니다.이러한 억제제들 중 많은 것들이 인간 효소를 목표로 하며 병적인 상태를 교정하는 것을 목표로 합니다.예를 들어, 아스피린은 사이클로옥시게나제 효소의 자살 억제제 역할을 하는 널리 사용되는 약물입니다.[95]이 억제는 차례로 소염증성 프로스타글란딘의 생성을 억제하고 따라서 아스피린은 통증, 발열, 염증을 줄이는데 사용될 수 있습니다.[95]
2017년 기준으로 [update]승인된 약물의 약 29%가 효소 억제제이며[96], 이 중 약 1/5이 키나아제 억제제입니다.[96]키나아제 약물 표적의 주목할 만한 종류는 세포 성장을 조절하는 필수 효소인 수용체 티로신 키나아제입니다; 그들의 과도한 활성화는 암을 초래할 수 있습니다.따라서 이미티닙과 같은 키나아제 억제제는 악성 종양을 치료하는 데 자주 사용됩니다.[97]야누스 키나아제는 약물 효소 표적의 또 다른 주목할 만한 예입니다.야누스 키나아제의 억제제는 염증성 사이토카인의 생성을 차단하여 관절염, 천식, 크론병 등의 다양한 염증성 질환을 치료하는 데 사용됩니다.[98]
일부 억제제의 구조적 유사성의 예는 약물 메토트렉세이트와 엽산을 비교하는 그림에서 볼 수 있습니다.엽산은 메토트렉세이트에 의해 잠재적으로 억제되는 효소인 디하이드로폴산 환원효소의 기질의 산화된 형태입니다.메토트렉세이트는 디하이드로폴산 환원효소의 작용을 차단하여 티미딘 생합성을 막습니다.[42]이 뉴클레오티드 생합성 블록은 빠르게 성장하는 세포에 선택적으로 독성을 나타내므로 메토트렉세이트는 종종 암 화학 요법에 사용됩니다.[99]
발기부전의 일반적인 치료법은 실데나필(Viagra)입니다.[100]이 화합물은 신호전달 분자인 고리형 구아노신 일인산을 분해하는 효소인 cGMP 특이적 포스포다이에스테라제 타입 5의 강력한 억제제입니다.[101]이 신호 전달 분자는 부드러운 근육 이완을 유발하고 발기를 일으키는 코퍼스 캐버노섬으로 혈액이 흐를 수 있게 합니다.그 약은 신호를 멈추게 하는 효소의 활동을 감소시키기 때문에, 그 신호를 더 오랜 시간 지속하게 만듭니다.
항생제
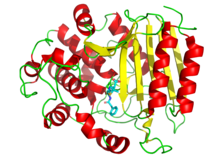
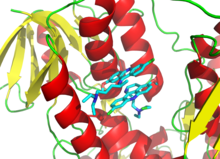
약은 병원체의 생존에 필요한 효소를 억제하기 위해 사용되기도 합니다.예를 들면, 박테리아는 펩티도글리칸이라고 불리는 그물 모양의 중합체로 만들어진 두꺼운 세포벽으로 둘러싸여 있습니다.페니실린과 반코마이신과 같은 많은 항생제들은 이 중합체의 가닥들을 생성하고 그 다음에 교차 연결하는 효소들을 억제합니다.[102][103]이것은 세포벽이 힘을 잃고 박테리아가 터지게 만듭니다.그림에서 페니실린 분자(볼 앤 스틱 형태로 표시됨)는 박테리아 스트렙토마이세스 R61(단백질은 리본 다이어그램으로 표시됨)의 트랜스펩타이드인 표적에 결합되어 있습니다.
항생제 약물 설계는 병원체의 생존에 필수적인 효소가 사람에게 없거나 매우 다를 때 촉진됩니다.[104]인간은 펩티도글리칸을 만들지 않기 때문에 이 과정을 억제하는 항생제는 박테리아에 선택적으로 독성이 있습니다.[105]선택적 독성은 박테리아의 리보솜의 구조의 차이 또는 [106]그것들이 지방산을 만드는 방법을 이용하여 항생제에서도 생성됩니다.[107]
항바이러스제
바이러스 복제에 필요한 효소를 억제하는 약은 바이러스 감염 치료에 효과적입니다.[108]항바이러스제는 HIV/AIDS[109] 및 C형 간염 치료에 사용되는 단백질분해효소 억제제,[110] HIV/AIDS를 표적으로 하는 역전사효소 억제제,[111] 인플루엔자를 표적으로 하는 뉴라미니다제 억제제 [112]및 인간 세포메갈로바이러스를 표적으로 하는 터마제 억제제를 포함합니다.[113]
살충제
많은 살충제들이 효소 억제제입니다.[114]아세틸콜린에스테라아제는 곤충에서 사람에 이르기까지 동물에서 발견되는 효소입니다.신경전달물질인 아세틸콜린을 그 구성성분인 아세테이트와 콜린으로 분해하는 메커니즘을 통해 신경세포의 기능에 필수적입니다.[115]세로토닌, 도파민, 노르에피네프린 등 대부분이 시냅스의 갈라진 틈에서 흡수되기 때문에 신경전달물질 중에서는 다소 이례적인 일입니다.많은 수의 AChE 억제제는 의학과 농업에 모두 사용됩니다.[116]에드로포늄, 피지스티그민, 네오스티그민과 같은 가역적 경쟁 억제제는 근막증의[117] 치료와 근육 차단을 되돌리기 위한 마취에 사용됩니다.[118]카바메이트 살충제는 또한 가역적 AChE 억제제의 예입니다.말라티온, 파라티온, 클로르피리포스와 같은 유기인산 살충제는 아세틸콜린에스테라아제를 비가역적으로 억제합니다.[119]
제초제
제초제 글리포세이트는 3-포스포시카메이트 1-카복시비닐트랜스퍼레이스의 억제제이며,[120] 술포닐우레아스는 아세톨락테이트 합성효소를 억제합니다.[121]두 효소 모두 식물이 분지쇄 아미노산을 만드는 데 필요합니다.지질과 카로티노이드의 생합성에 필요한 효소와 광합성과 산화적 인산화 과정을 포함한 많은 다른 효소들이 제초제에 의해 억제됩니다.[122]
디스커버리 및 디자인

신약은 오랜 약물 개발 과정의 산물이며, 그 첫 단계는 종종 새로운 효소 억제제의 발견입니다.[123]이러한 억제제를 발견하는 데에는 두 가지 원칙적인 접근법이 있습니다.[124]
첫 번째 일반적인 방법은 효소에 의해 촉매되는 화학 반응의 전이 상태를 모방하는 것에 기초한 합리적인 약물 설계입니다.[125]설계된 억제제는 화학 반응을 일으키는 기질의 부분이 전이 상태와 유사한 화학적으로 안정한 작용기로 대체되는 것을 제외하고는 종종 기질과 유사합니다.효소가 전이 상태를 안정화하도록 진화했기 때문에, 전이 상태 유사체는 일반적으로 기질에 비해 효소에 대해 더 높은 친화력을 가지므로 효과적인 억제제입니다.[46]
새로운 효소 억제제를 발견하는 두 번째 방법은 구조적으로 다양한 화합물의 큰 라이브러리에서 효소에 결합하는 히트 분자를 식별하는 높은 처리량의 스크리닝입니다.이 방법은 컴퓨터를 사용하여 다양한 분자의 데이터베이스를 가상 스크리닝하고 이어서 가상 스크리닝 히트의 결합을 실험적으로 확인하는 [126][127]것을 포함하도록 확장되었습니다.[128]억제제에 대한 새로운 출발점을 제공할 수 있는 보완적인 접근법으로는 단편 기반 납 발견[129] 및 DNA 인코딩 화학 라이브러리(DEL)가 있습니다.[130]
위의 접근법들 중 임의의 것으로부터의 히트는 효소를 효율적으로 억제하는 고친화성 바인더로 최적화될 수 있습니다.[131]분자 도킹[132] 및 분자 역학과 같은 효소에 대한 억제제의 결합 방향 및 친화도를 예측하는 컴퓨터 기반 방법을 사용하여 최적화 프로세스를 지원할 수 있습니다.[133]새로운 억제제는 억제제/효소 복합체에서 효소의 결정학적 구조를 얻는 데 사용되어 분자가 활성 부위에 어떻게 결합하는지를 보여주고, 구조 기반 약물 설계로 알려진 과정에서 결합을 최적화하기 위해 억제제에 대한 변경을 허용합니다.[1]: 66 이 테스트 및 개선 주기는 충분히 강력한 억제제가 생성될 때까지 반복됩니다.
참고 항목
- 활성 기반 프로테오믹스 – 공유 효소 억제제를 리포터로 사용하여 효소 활성을 모니터링하는 프로테오믹스의 한 분야입니다.
- 항메타볼라이트 – 세포의 성장과 분열을 방해하는 효소 억제제
- 전이 상태 유사체 – 효소에 의해 촉매되는 화학 반응의 전이 상태를 모방하는 효소 억제제의 일종
참고문헌
- ^ a b c d Copeland RA (March 2013). "Why Enzymes as Drug Targets? Enzyme are Essential for Life". Evaluation of Enzyme Inhibitors in Drug Discovery: A Guide for Medicinal Chemists and Pharmacologists (Second ed.). John Wiley & Sons, Inc. pp. 1–23. doi:10.1002/9781118540398.ch1. ISBN 978-1-118-48813-3.
- ^ a b c Sauro HM (February 2017). "Control and regulation of pathways via negative feedback". Journal of the Royal Society, Interface. 14 (127): 1–13. doi:10.1098/rsif.2016.0848. PMC 5332569. PMID 28202588.
- ^ a b c Plaxton WC (2004). "Principles of Metabolic Control". In Storey KB (ed.). Functional Metabolism: Regulation and Adaptation. Hoboken, N.J.: John Wiley & Sons. pp. 1–24 (12). ISBN 978-0-471-67557-0. Archived from the original on 28 March 2023. Retrieved 14 April 2022.
- ^ a b Haefner B (June 2003). "Drugs from the deep: marine natural products as drug candidates". Drug Discovery Today. 8 (12): 536–44. doi:10.1016/s1359-6446(03)02713-2. PMID 12821301.
- ^ Gualerzi CO, Brandi L, Fabbretti A, Pon CL (2013). Antibiotics: Targets, Mechanisms and Resistance. Hoboken: John Wiley & Sons. ISBN 978-3-527-65970-8.
- ^ Sanrattana W, Maas C, de Maat S (2019). "SERPINs-From Trap to Treatment". Frontiers in Medicine. 6: 25. doi:10.3389/fmed.2019.00025. PMC 6379291. PMID 30809526.
- ^ Shapiro R, Vallee BL (February 1991). "Interaction of human placental ribonuclease with placental ribonuclease inhibitor". Biochemistry. 30 (8): 2246–2255. doi:10.1021/bi00222a030. PMID 1998683.
- ^ Boon L, Ugarte-Berzal E, Vandooren J, Opdenakker G (April 2020). "Protease propeptide structures, mechanisms of activation, and functions". Critical Reviews in Biochemistry and Molecular Biology. 55 (2): 111–165. doi:10.1080/10409238.2020.1742090. PMID 32290726. S2CID 215772580.
- ^ a b Rydzewski RM (2010). "Chapter 7.2.1: Competition and Allostery". Real World Drug Discovery: A Chemist's Guide to Biotech and Pharmaceutical Research (1st ed.). Amsterdam: Elsevier. pp. 281–285. ISBN 978-0-08-091488-6. Archived from the original on 28 March 2023. Retrieved 20 July 2022.
- ^ Jakubík J, Randáková A, El-Fakahany EE, Doležal V (2019). "Analysis of equilibrium binding of an orthosteric tracer and two allosteric modulators". PLOS ONE. 14 (3): e0214255. Bibcode:2019PLoSO..1414255J. doi:10.1371/journal.pone.0214255. PMC 6436737. PMID 30917186.
- ^ Patrick GL (2013). "Chapter 7: Enzymes as Drug Targets". An Introduction to Medicinal Chemistry (Fifth ed.). Oxford, UK: Oxford University Press. p. 90. ISBN 978-0-19-969739-7. Archived from the original on 20 July 2022. Retrieved 20 July 2022.
- ^ Kuriyan J, Konforti B, Wemmer D (2012). "Molecular Recognition: The Thermodynamics of Binding". The Molecules of Life : Physical and Chemical Principles (First ed.). Boca Raton, FL: Garland Science. pp. 531–580. ISBN 978-1-135-08892-7. Archived from the original on 7 June 2022. Retrieved 7 June 2022.
- ^ a b c Tuley A, Fast W (June 2018). "The Taxonomy of Covalent Inhibitors". Biochemistry. 57 (24): 3326–3337. doi:10.1021/acs.biochem.8b00315. PMC 6016374. PMID 29689165.
- ^ a b Cleland WW (February 1963). "The kinetics of enzyme-catalysed reactions with two or more substrates or products. II. Inhibition: nomenclature and theory". Biochimica et Biophysica Acta (BBA) - Specialized Section on Enzymological Subjects. 67: 173–187. doi:10.1016/0926-6569(63)90226-8. PMID 14021668.
- ^ Berg J, Tymoczko J, Stryer L (2002). Biochemistry. W. H. Freeman and Company. ISBN 978-0-7167-4955-4. Archived from the original on 26 September 2009. Retrieved 31 August 2017.
- ^ a b c "Types of Inhibition". NIH Center for Translational Therapeutics. Archived from the original on 8 September 2011. Retrieved 2 April 2012.
- ^ a b c d e f g h Cornish-Bowden A (2012). Fundamentals of Enzyme Kinetics (4th ed.). Weinheim: Wiley-VCH. ISBN 978-3-527-66549-5.
- ^ a b Segel IH (1993). Enzyme Kinetics: Behavior and Analysis of Rapid Equilibrium and Steady-State Enzyme Systems (New ed.). Wiley-Interscience. ISBN 978-0-471-30309-1.
- ^ Palmer T, Bonner PL (2007). "Enzyme inhibition.". Enzymes: Biochemistry, Biotechnology, Clinical Chemistry (2nd ed.). Woodhead Publishing. pp. 126–152 (135). doi:10.1533/9780857099921.2.126. ISBN 978-1-904275-27-5.
- ^ Delaune KP, Alsayouri K (September 2021). "Physiology: Noncompetitive Inhibitor". StatPearls. Treasure Island (FL): StatPearls Publishing. p. 31424826. PMID 31424826. Archived from the original on 28 November 2021. Retrieved 3 April 2022.
- ^ a b Voet D, Voet JG, Pratt CW (2016). "Chapter 12: Enzyme Kinetics, Inhibition, and Control". Fundamentals of Biochemistry: Life at the Molecular Level (Fifth ed.). Hoboken, NJ: Wiley. pp. 361–401. ISBN 978-1-118-91840-1.
- ^ Buker SM, Boriack-Sjodin PA, Copeland RA (June 2019). "Enzyme-Inhibitor Interactions and a Simple, Rapid Method for Determining Inhibition Modality". SLAS Discovery: Advancing Life Sciences R & D. 24 (5): 515–522 (516). doi:10.1177/2472555219829898. PMID 30811960. S2CID 73480979.
In some cases, the inhibitor may bind to a distinct site on the enzyme that is in allosteric communication with the substrate binding pocket. In many cases, allosteric, substrate competitive compounds result in conformational changes to the enzyme that change the ability of the enzyme to bind substrate.
- ^ Laidler KJ (1978). Physical Chemistry with Biological Applications. Benjamin/Cummings. p. 437. ISBN 978-0-8053-5680-9.
- ^ a b c d e f Bisswanger H (2017). Enzyme Kinetics: Principles and Methods (3rd ed.). Newark: John Wiley & Sons, Incorporated. ISBN 978-3-527-80647-8.
- ^ Marangoni AG (2003). "Reversible Enzyme Inhibition". Enzyme Kinetics: A Modern Approach. Hoboken, N.J.: Wiley-Interscience. pp. 61–69. ISBN 978-0-471-15985-8.
- ^ Segel IH (1993). Enzyme Kinetics: Behavior and Analysis of Rapid Equilibrium and Steady-State Enzyme Systems (New ed.). Wiley–Interscience. ISBN 978-0-471-30309-1.
- ^ a b Walsh R, Martin E, Darvesh S (December 2011). "Limitations of conventional inhibitor classifications". Integrative Biology. 3 (12): 1197–1201. doi:10.1039/c1ib00053e. PMID 22038120.
- ^ a b c Walsh R, Martin E, Darvesh S (May 2007). "A versatile equation to describe reversible enzyme inhibition and activation kinetics: modeling beta-galactosidase and butyrylcholinesterase". Biochimica et Biophysica Acta (BBA) - General Subjects. 1770 (5): 733–746. doi:10.1016/j.bbagen.2007.01.001. PMID 17307293.
- ^ Walsh R (May 2012). "Alternative perspectives of enzyme kinetic modeling". Medicinal Chemistry and Drug Design. InTech (16): 357–372.
- ^ Strelow J, Dewe W, Iversen PW, Brooks PB, Radding JA, McGee J, Weidner J (October 2012). "Mechanism of Action Assays for Enzymes". In Markossian S, Grossman A, Brimacombe K, et al. (eds.). Assay Guidance Manual. Eli Lilly & Company and the National Center for Advancing Translational Sciences. PMID 22553872. Archived from the original on 15 June 2022. Retrieved 9 April 2022.
- ^ Holdgate GA (July 2001). "Making cool drugs hot: isothermal titration calorimetry as a tool to study binding energetics". BioTechniques. 31 (1): 164–6, 168, 170 passim. PMID 11464510.
- ^ Leatherbarrow RJ (December 1990). "Using linear and non-linear regression to fit biochemical data". Trends in Biochemical Sciences. 15 (12): 455–458. doi:10.1016/0968-0004(90)90295-M. PMID 2077683.
- ^ a b Tseng SJ, Hsu JP (August 1990). "A comparison of the parameter estimating procedures for the Michaelis-Menten model". Journal of Theoretical Biology. 145 (4): 457–464. Bibcode:1990JThBi.145..457T. doi:10.1016/S0022-5193(05)80481-3. PMID 2246896.
- ^ Dixon M, Webb EC, Thorne CJ, Tipton KF (1979). Enzymes (3rd ed.). London: Longman. p. 126. ISBN 978-0-470-20745-1.
- ^ Radzicka A, Wolfenden R (1995). Transition state and multisubstrate analog inhibitors. Methods in Enzymology. Vol. 249. pp. 284–312. doi:10.1016/0076-6879(95)49039-6. ISBN 9780121821500. PMID 7791615.
- ^ Schiffer CF, Burke JF, Besarab A, Lasker N, Simenhoff ML (January 1977). "Amylase/creatinine clearance fraction in patients on chronic hemodialysis". Annals of Internal Medicine. 86 (1): 65–66. doi:10.7326/0003-4819-86-1-65. PMID 319722.
- ^ Inglese J, Blatchly RA, Benkovic SJ (May 1989). "A multisubstrate adduct inhibitor of a purine biosynthetic enzyme with a picomolar dissociation constant". Journal of Medicinal Chemistry. 32 (5): 937–940. doi:10.1021/jm00125a002. PMID 2709379.
- ^ Inglese J, Benkovic SJ (1991). "Multisubstrate Adduct Inhibitors of Glycinamide Ribonucleotide Transformylase: Synthetic and Enzyme Generated". Tetrahedron. 47 (14–15): 2351–2364. doi:10.1016/S0040-4020(01)81773-7.
- ^ Rozwarski DA, Grant GA, Barton DH, Jacobs WR, Sacchettini JC (January 1998). "Modification of the NADH of the isoniazid target (InhA) from Mycobacterium tuberculosis". Science. 279 (5347): 98–102. Bibcode:1998Sci...279...98R. doi:10.1126/science.279.5347.98. PMID 9417034.
- ^ Auld DS, Lovell S, Thorne N, Lea WA, Maloney DJ, Shen M, et al. (March 2010). "Molecular basis for the high-affinity binding and stabilization of firefly luciferase by PTC124". Proceedings of the National Academy of Sciences of the United States of America. 107 (11): 4878–4883. Bibcode:2010PNAS..107.4878A. doi:10.1073/pnas.0909141107. PMC 2841876. PMID 20194791.
- ^ Le Calvez PB, Scott CJ, Migaud ME (December 2009). "Multisubstrate adduct inhibitors: drug design and biological tools". Journal of Enzyme Inhibition and Medicinal Chemistry. 24 (6): 1291–318. doi:10.3109/14756360902843809. PMID 19912064. S2CID 21808708.
- ^ a b Avendano C, Menendez JC (June 2015). "Chapter 2.5: Inhibitors of Dihydrofolate Reductase". Medicinal Chemistry of Anticancer Drugs. Elsevier. pp. 54–58. ISBN 978-0-444-62667-7.
- ^ Hsu JT, Wang HC, Chen GW, Shih SR (2006). "Antiviral drug discovery targeting to viral proteases". Current Pharmaceutical Design. 12 (11): 1301–1314. doi:10.2174/138161206776361110. PMID 16611117.
- ^ Agbowuro AA, Huston WM, Gamble AB, Tyndall JD (July 2018). "Proteases and protease inhibitors in infectious diseases". Medicinal Research Reviews. 38 (4): 1295–1331. doi:10.1002/med.21475. PMID 29149530. S2CID 25269012.
- ^ Qiu X, Liu ZP (2011). "Recent developments of peptidomimetic HIV-1 protease inhibitors". Current Medicinal Chemistry. 18 (29): 4513–37. doi:10.2174/092986711797287566. PMID 21864279.
- ^ a b Schramm VL (November 2018). "Enzymatic Transition States and Drug Design". Chemical Reviews. 118 (22): 11194–11258. doi:10.1021/acs.chemrev.8b00369. PMC 6615489. PMID 30335982.
- ^ Lew W, Chen X, Kim CU (June 2000). "Discovery and development of GS 4104 (oseltamivir): an orally active influenza neuraminidase inhibitor". Current Medicinal Chemistry. 7 (6): 663–672. doi:10.2174/0929867003374886. PMID 10702632.
- ^ Fischer PM (October 2003). "The design, synthesis and application of stereochemical and directional peptide isomers: a critical review". Current Protein & Peptide Science. 4 (5): 339–356. doi:10.2174/1389203033487054. PMID 14529528.
- ^ Breen ME, Soellner MB (January 2015). "Small molecule substrate phosphorylation site inhibitors of protein kinases: approaches and challenges". ACS Chemical Biology. 10 (1): 175–89. doi:10.1021/cb5008376. PMC 4301090. PMID 25494294.
- ^ Bogoyevitch MA, Barr RK, Ketterman AJ (December 2005). "Peptide inhibitors of protein kinases-discovery, characterisation and use". Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1754 (1–2): 79–99. doi:10.1016/j.bbapap.2005.07.025. PMID 16182621.
- ^ a b Patrick GL (2017). "Enzymes as Drug Targets". An Introduction to Medicinal Chemistry (Sixth ed.). Oxford, United Kingdom: Oxford University Press. p. 95. ISBN 978-0-19-874969-1. Archived from the original on 28 March 2023. Retrieved 3 June 2022.
- ^ Gehringer M, Laufer SA (June 2019). "Emerging and Re-Emerging Warheads for Targeted Covalent Inhibitors: Applications in Medicinal Chemistry and Chemical Biology". Journal of Medicinal Chemistry. 62 (12): 5673–5724. doi:10.1021/acs.jmedchem.8b01153. PMID 30565923. S2CID 56480231.
- ^ Lundblad RL (2004). Chemical reagents for protein modification (3rd ed.). CRC Press. ISBN 978-0-8493-1983-9.
- ^ Polakovič M, Vrabel P, Báleš V (January 1998). "Approaches for improved identification of mechanisms of enzyme inactivation". Progress in Biotechnology. Elsevier. 15: 77–82. doi:10.1016/S0921-0423(98)80013-0. ISBN 978-0-444-82970-2.
Enzyme inactivation is generally explained as a chemical process involving several phenomena like aggregation, dissociation into subunits, or denaturation (conformational changes), which occur simultaneously during the inactivation of a specific enzyme.
- ^ Price N, Hames B, Rickwood D (1996). Proteins LabFax. BIOS Scientific Publishers. ISBN 978-0-12-564710-6.
- ^ Adam GC, Cravatt BF, Sorensen EJ (January 2001). "Profiling the specific reactivity of the proteome with non-directed activity-based probes". Chemistry & Biology. 8 (1): 81–95. doi:10.1016/S1074-5521(00)90060-7. PMID 11182321.
- ^ Maurer T, Fung HL (2000). "Comparison of methods for analyzing kinetic data from mechanism-based enzyme inactivation: application to nitric oxide synthase". AAPS PharmSci. 2 (1): 68–77. doi:10.1208/ps020108. PMC 2751003. PMID 11741224.
- ^ Loo JA, DeJohn DE, Du P, Stevenson TI, Ogorzalek Loo RR (July 1999). "Application of mass spectrometry for target identification and characterization". Medicinal Research Reviews. 19 (4): 307–319. doi:10.1002/(SICI)1098-1128(199907)19:4<307::AID-MED4>3.0.CO;2-2. PMID 10398927. S2CID 11766917.
- ^ Purich DL (2010). "Irreversible Enzyme Inhibition by Affinity Labelling Agents". Enzyme Kinetics: Catalysis and Control: A Reference of Theory and Best-Practice Methods. San Diego, Calif.: Elsevier Academic. p. 542. ISBN 978-0-12-380925-4. Archived from the original on 20 July 2022. Retrieved 20 July 2022.
- ^ Sibille E, Bana E, Chaouni W, Diederich M, Bagrel D, Chaimbault P (November 2012). "Development of a matrix-assisted laser desorption/ionization-mass spectrometry screening test to evidence reversible and irreversible inhibitors of CDC25 phosphatases". Analytical Biochemistry. 430 (1): 83–91. doi:10.1016/j.ab.2012.08.006. PMID 22902804.
- ^ a b Poulin R, Lu L, Ackermann B, Bey P, Pegg AE (January 1992). "Mechanism of the irreversible inactivation of mouse ornithine decarboxylase by alpha-difluoromethylornithine. Characterization of sequences at the inhibitor and coenzyme binding sites". The Journal of Biological Chemistry. 267 (1): 150–158. doi:10.1016/S0021-9258(18)48472-4. PMID 1730582.
- ^ Szedlacsek SE, Duggleby RG (1995). "[6] Kinetics of slow and tight-binding inhibitors". Kinetics of slow and tight-binding inhibitors. Methods in Enzymology. Vol. 249. pp. 144–80. doi:10.1016/0076-6879(95)49034-5. ISBN 978-0-12-182150-0. PMID 7791610.
- ^ Stone SR, Morrison JF (February 1986). "Mechanism of inhibition of dihydrofolate reductases from bacterial and vertebrate sources by various classes of folate analogs". Biochimica et Biophysica Acta (BBA) - Protein Structure and Molecular Enzymology. 869 (3): 275–285. doi:10.1016/0167-4838(86)90067-1. PMID 3511964.
- ^ Pick FM, McGartoll MA, Bray RC (January 1971). "Reaction of formaldehyde and of methanol with xanthine oxidase". European Journal of Biochemistry. 18 (1): 65–72. doi:10.1111/j.1432-1033.1971.tb01215.x. PMID 4322209.
- ^ Reardon JE (November 1989). "Herpes simplex virus type 1 and human DNA polymerase interactions with 2'-deoxyguanosine 5'-triphosphate analogs. Kinetics of incorporation into DNA and induction of inhibition". The Journal of Biological Chemistry. 264 (32): 19039–19044. doi:10.1016/S0021-9258(19)47263-3. PMID 2553730.
- ^ PDB: 1GXF; Saravanamuthu A, Vickers TJ, Bond CS, Peterson MR, Hunter WN, Fairlamb AH (July 2004). "Two interacting binding sites for quinacrine derivatives in the active site of trypanothione reductase: a template for drug design". The Journal of Biological Chemistry. 279 (28): 29493–500. doi:10.1074/jbc.M403187200. PMC 3491871. PMID 15102853.
- ^ Cohen JA, Oosterbaan RA, Berends F (1967). "[81] Organophosphorus compounds". Enzyme Structure. Methods in Enzymology. Vol. 11. pp. 686–702. doi:10.1016/S0076-6879(67)11085-9. ISBN 978-0-12-181860-9. Archived from the original on 28 February 2018.
- ^ Brenner GM (2000). Pharmacology (1st ed.). Philadelphia, PA: W.B. Saunders. ISBN 978-0-7216-7757-6.
- ^ Walsh CT (1984). "Suicide substrates, mechanism-based enzyme inactivators: recent developments". Annual Review of Biochemistry. 53: 493–535. doi:10.1146/annurev.bi.53.070184.002425. PMID 6433782.
- ^ Saravanamuthu A, Vickers TJ, Bond CS, Peterson MR, Hunter WN, Fairlamb AH (July 2004). "Two interacting binding sites for quinacrine derivatives in the active site of trypanothione reductase: a template for drug design". The Journal of Biological Chemistry. 279 (28): 29493–29500. doi:10.1074/jbc.M403187200. PMC 3491871. PMID 15102853.
- ^ Pereira DM, Andrade C, Valentão P, Andrade PB (October 2017). "Natural products as enzyme inhibitors." (PDF). Natural Products Targeting Clinically Relevant Enzymes (First ed.). Wiley-VCH Verlag GmbH & Co. KGaA. ISBN 978-3-527-34205-1. Archived (PDF) from the original on 5 December 2022. Retrieved 3 April 2022.
- ^ Hiratake J (2005). "Enzyme inhibitors as chemical tools to study enzyme catalysis: rational design, synthesis, and applications". Chemical Record. New York, N.Y. 5 (4): 209–28. doi:10.1002/tcr.20045. PMID 16041744.
- ^ Page C, Pitchford S (2021). "Venoms, toxins, poisons and herbs". Dale's Pharmacology Condensed E-Book (Third ed.). Philadelphia, Pa.: Elsevier Health Sciences. pp. 153–155. ISBN 978-0-7020-7819-4. Archived from the original on 25 April 2013. Retrieved 3 June 2022.
- ^ Stenersen J (2004). "Chapter 5: Specific Enzyme Inhibitors". Chemical Pesticides Mode of Action and Toxicology. Boca Raton: CRC Press. pp. 73–114. ISBN 978-0-203-64683-0. Archived from the original on 28 March 2023. Retrieved 14 April 2022.
- ^ Greathouse B, Zahra F, Brady MF (September 2021). "Acetylcholinesterase Inhibitors Toxicity". StatPearls [Internet]. Treasure Island (FL): StatPearls Publishing. PMID 30571049. Archived from the original on 17 January 2023. Retrieved 14 April 2022.
- ^ Orencio-Trejo M, Utrilla J, Fernández-Sandoval MT, Huerta-Beristain G, Gosset G, Martinez A (2010). "Engineering the Escherichia coli Fermentative Metabolism". In Cordes M, Wittmann C, Krull R (eds.). Biosystems Engineering II: Linking Cellular Networks and Bioprocesses. Berlin: Springer Science & Business Media. pp. 77–78. ISBN 978-3-642-13865-2.
- ^ Okar DA, Lange AJ (1999). "Fructose-2,6-bisphosphate and control of carbohydrate metabolism in eukaryotes". BioFactors. 10 (1): 1–14. doi:10.1002/biof.5520100101. PMID 10475585. S2CID 24586866.
- ^ Price NC, Stevens L (1999). Fundamentals of enzymology : the cell and molecular biology of catalytic proteins (3rd ed.). Oxford University Press. ISBN 978-0-19-850229-6.
- ^ Smyth TP (August 2004). "Substrate variants versus transition state analogues as noncovalent reversible enzyme inhibitors". Bioorganic & Medicinal Chemistry. 12 (15): 4081–4088. doi:10.1016/j.bmc.2004.05.041. PMID 15246086. Archived from the original on 28 March 2023. Retrieved 14 April 2022.
- ^ Hartley RW (November 1989). "Barnase and barstar: two small proteins to fold and fit together". Trends in Biochemical Sciences. 14 (11): 450–454. doi:10.1016/0968-0004(89)90104-7. PMID 2696173.
- ^ Maheshwari VL (2022). Natural Products as Enzyme Inhibitors. Singapore: Springer. ISBN 978-981-19-0932-0.
- ^ Birk Y (2003). Plant Protease Inhibitors: Significance in Nutrition, Plant Protection, Cancer Prevention and Genetic Engineering. Berlin: Springer. ISBN 978-3-540-00118-8.
- ^ Tan G, Gyllenhaal C, Soejarto DD (March 2006). "Biodiversity as a source of anticancer drugs". Current Drug Targets. 7 (3): 265–277. doi:10.2174/138945006776054942. PMID 16515527.
- ^ Abal M, Andreu JM, Barasoain I (June 2003). "Taxanes: microtubule and centrosome targets, and cell cycle dependent mechanisms of action". Current Cancer Drug Targets. 3 (3): 193–203. doi:10.2174/1568009033481967. PMID 12769688.
- ^ Hostettmann K, Borloz A, Urbain A, Marston A (2006). "Natural Product Inhibitors of Acetylcholinesterase". Current Organic Chemistry. 10 (8): 825–847. doi:10.2174/138527206776894410.
- ^ Knight R, Khondoker M, Magill N, Stewart R, Landau S (2018). "A Systematic Review and Meta-Analysis of the Effectiveness of Acetylcholinesterase Inhibitors and Memantine in Treating the Cognitive Symptoms of Dementia". Dementia and Geriatric Cognitive Disorders. 45 (3–4): 131–151. doi:10.1159/000486546. PMID 29734182. S2CID 13701908.
- ^ DeFrates LJ, Hoehns JD, Sakornbut EL, Glascock DG, Tew AR (January 2005). "Antimuscarinic intoxication resulting from the ingestion of moonflower seeds". The Annals of Pharmacotherapy. 39 (1): 173–176. doi:10.1345/aph.1D536. PMID 15572604. S2CID 36465515.
- ^ Vetter J (January 1998). "Toxins of Amanita phalloides". Toxicon. 36 (1): 13–24. doi:10.1016/S0041-0101(97)00074-3. PMID 9604278.
- ^ Holmes CF, Maynes JT, Perreault KR, Dawson JF, James MN (November 2002). "Molecular enzymology underlying regulation of protein phosphatase-1 by natural toxins". Current Medicinal Chemistry. 9 (22): 1981–1989. doi:10.2174/0929867023368827. PMID 12369866.
- ^ Bischoff K (October 2001). "The toxicology of microcystin-LR: occurrence, toxicokinetics, toxicodynamics, diagnosis and treatment". Veterinary and Human Toxicology. 43 (5): 294–297. PMID 11577938.
- ^ Savage GP, Morrison SC (2003). "Trypsin inhibitors.". In Caballero B (ed.). Encyclopedia of Food Sciences and Nutrition (Second ed.). Academic Press. pp. 5878–5884. doi:10.1016/B0-12-227055-X/00934-2. ISBN 978-0-12-227055-0.
- ^ Polito L, Bortolotti M, Battelli MG, Calafato G, Bolognesi A (June 2019). "Ricin: An Ancient Story for a Timeless Plant Toxin". Toxins. 11 (6): 324. doi:10.3390/toxins11060324. PMC 6628454. PMID 31174319.
- ^ Sowa-Rogozińska N, Sominka H, Nowakowska-Gołacka J, Sandvig K, Słomińska-Wojewódzka M (June 2019). "Intracellular Transport and Cytotoxicity of the Protein Toxin Ricin". Toxins. 11 (6): 350. doi:10.3390/toxins11060350. PMC 6628406. PMID 31216687.
- ^ Hartley MR, Lord JM (September 2004). "Cytotoxic ribosome-inactivating lectins from plants". Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1701 (1–2): 1–14. doi:10.1016/j.bbapap.2004.06.004. PMID 15450171.
- ^ a b Finkel R, Cubeddu LX, Clark MA (2009). "Chapter 41: Antiinfammatory Drugs". Pharmacology (4th ed.). Philadelphia: Lippincott Williams & Wilkins. pp. 499–518 (502). ISBN 978-0-7817-7155-9. Archived from the original on 28 March 2023. Retrieved 14 April 2022.
- ^ a b Santos R, Ursu O, Gaulton A, Bento AP, Donadi RS, Bologa CG, Karlsson A, Al-Lazikani B, Hersey A, Oprea TI, Overington JP (January 2017). "A comprehensive map of molecular drug targets". Nature Reviews. Drug Discovery. 16 (1): 19–34 (Figure 1C). doi:10.1038/nrd.2016.230. PMC 6314433. PMID 27910877.
Figure 1C: Clinical success of privileged protein family classes (% approved drugs targeting each target class): Reductase 7.62, Kinase 5.94, Protease 3.35, Hydrolase 2.76, NPTase 2.09, Transferase 1.92, Lyase 1.59, Isomerase 1.51, Phosphodiesterase 1.50, Cytochrome p450 0.84, Epigenetic eraser 0.33, Total enzyme targets of approved drugs = 29.45%
- ^ Kannaiyan R, Mahadevan D (December 2018). "A comprehensive review of protein kinase inhibitors for cancer therapy". Expert Review of Anticancer Therapy. 18 (12): 1249–1270. doi:10.1080/14737140.2018.1527688. PMC 6322661. PMID 30259761.
- ^ McLornan DP, Pope JE, Gotlib J, Harrison CN (August 2021). "Current and future status of JAK inhibitors". Lancet. 398 (10302): 803–816. doi:10.1016/S0140-6736(21)00438-4. PMID 34454676. S2CID 237311419.
- ^ McGuire JJ (2003). "Anticancer antifolates: current status and future directions". Current Pharmaceutical Design. 9 (31): 2593–2613. doi:10.2174/1381612033453712. PMID 14529544.
- ^ Goldstein I, Burnett AL, Rosen RC, Park PW, Stecher VJ (January 2019). "The Serendipitous Story of Sildenafil: An Unexpected Oral Therapy for Erectile Dysfunction". Sexual Medicine Reviews. 7 (1): 115–128. doi:10.1016/j.sxmr.2018.06.005. PMID 30301707. S2CID 52945888.
- ^ Maggi M, Filippi S, Ledda F, Magini A, Forti G (August 2000). "Erectile dysfunction: from biochemical pharmacology to advances in medical therapy". European Journal of Endocrinology. 143 (2): 143–154. doi:10.1530/eje.0.1430143. PMID 10913932.
- ^ Cochrane SA, Lohans CT (May 2020). "Breaking down the cell wall: Strategies for antibiotic discovery targeting bacterial transpeptidases". European Journal of Medicinal Chemistry. 194: 112262. doi:10.1016/j.ejmech.2020.112262. PMID 32248005. S2CID 214809706. Archived from the original on 28 March 2023. Retrieved 20 July 2022.
- ^ Buynak JD (September 2007). "Cutting and stitching: the cross-linking of peptidoglycan in the assembly of the bacterial cell wall". ACS Chemical Biology. 2 (9): 602–5. doi:10.1021/cb700182u. PMID 17894443.
- ^ Dalhoff A (February 2021). "Selective toxicity of antibacterial agents-still a valid concept or do we miss chances and ignore risks?". Infection. 49 (1): 29–56. doi:10.1007/s15010-020-01536-y. PMC 7851017. PMID 33367978.
- ^ Mobley H (13 March 2006). "How do antibiotics kill bacterial cells but not human cells?". Scientific American. 294 (6): 98. PMID 16711368. Archived from the original on 9 April 2022. Retrieved 9 April 2022.
- ^ Zhang L, He J, Bai L, Ruan S, Yang T, Luo Y (July 2021). "Ribosome-targeting antibacterial agents: Advances, challenges, and opportunities". Medicinal Research Reviews. 41 (4): 1855–1889. doi:10.1002/med.21780. PMID 33501747. S2CID 231761270.
- ^ Yao J, Rock CO (November 2017). "Bacterial fatty acid metabolism in modern antibiotic discovery". Biochimica et Biophysica Acta (BBA) - Molecular and Cell Biology of Lipids. 1862 (11): 1300–1309. doi:10.1016/j.bbalip.2016.09.014. PMC 5364071. PMID 27668701.
- ^ Li G, Jing X, Pan, Zhang P, De Clercq E (2021). "Antiviral Classification". In Bamford D, Zuckerman MA (eds.). Encyclopedia of virology. Vol. 5. Amsterdam. pp. 129–130. doi:10.1016/B978-0-12-814515-9.00126-0. ISBN 978-0-12-814516-6. OCLC 1240584737.
{{cite book}}: CS1 유지 관리: 위치 누락 게시자(링크) - ^ Voshavar C (2019). "Protease Inhibitors for the Treatment of HIV/AIDS: Recent Advances and Future Challenges". Current Topics in Medicinal Chemistry. 19 (18): 1571–1598. doi:10.2174/1568026619666190619115243. PMID 31237209. S2CID 195356119.
- ^ de Leuw P, Stephan C (April 2018). "Protease inhibitor therapy for hepatitis C virus-infection". Expert Opinion on Pharmacotherapy. 19 (6): 577–587. doi:10.1080/14656566.2018.1454428. PMID 29595065. S2CID 4489039.
- ^ Li G, Wang Y, De Clercq E (April 2022). "Approved HIV reverse transcriptase inhibitors in the past decade". Acta Pharmaceutica Sinica B. 12 (4): 1567–1590. doi:10.1016/j.apsb.2021.11.009. PMC 9279714. PMID 35847492.
- ^ Gubareva L, Mohan T (January 2022). "Antivirals Targeting the Neuraminidase". Cold Spring Harbor Perspectives in Medicine. 12 (1): a038455. doi:10.1101/cshperspect.a038455. PMC 8725622. PMID 32152244. S2CID 212651676.
- ^ Gentry BG, Bogner E, Drach JC (January 2019). "Targeting the terminase: An important step forward in the treatment and prophylaxis of human cytomegalovirus infections". Antiviral Research. 161: 116–124. doi:10.1016/j.antiviral.2018.11.005. PMID 30472161. S2CID 53763831.
- ^ Kuhr RJ, Dorough HW (1976). Carbamate Insecticides: Chemistry, Biochemistry, and Toxicology. Cleveland: CRC Press. ISBN 978-0-87819-052-2.
- ^ Stone TW (October 2020). CNS Neurotransmitters and Neuromodulators: Acetylcholine (1st ed.). Boca Raton: CRC Press. ISBN 978-1-00-009898-3.
- ^ Gupta RC (2006). "Classification and Uses of Organophosphates and Carbamates". In Gupta RC (ed.). Toxicology of Organophosphate and Carbamate Compounds. Amsterdam: Elsevier Academic Press. pp. 5–24. ISBN 978-0-08-054310-9. Archived from the original on 21 July 2022. Retrieved 21 July 2022.
- ^ Farmakidis C, Pasnoor M, Dimachkie MM, Barohn RJ (May 2018). "Treatment of Myasthenia Gravis". Neurologic Clinics. 36 (2): 311–337. doi:10.1016/j.ncl.2018.01.011. PMC 6690491. PMID 29655452.
- ^ Butterworth JF, IV, Mackey DC, Wasnick JD, eds. (2013). "Chapter 12. Cholinesterase Inhibitors & Other Pharmacologic Antagonists to Neuromuscular Blocking Agents.". Morgan & Mikhail's Clinical Anesthesiology (5th ed.). McGraw Hill. Archived from the original on 30 June 2022. Retrieved 9 April 2022.
- ^ Thapa S, Lv M, Xu H (2017). "Acetylcholinesterase: A Primary Target for Drugs and Insecticides". Mini Reviews in Medicinal Chemistry. 17 (17): 1665–1676. doi:10.2174/1389557517666170120153930. PMID 28117022.
- ^ Tan S, Evans R, Singh B (March 2006). "Herbicidal inhibitors of amino acid biosynthesis and herbicide-tolerant crops". Amino Acids. 30 (2): 195–204. doi:10.1007/s00726-005-0254-1. PMID 16547651. S2CID 2358278.
- ^ Hirai K, Uchida A, Ohno R (2012). "Major Synthetic Routes for Modern Herbicide Classes and Agrochemical Characteristics". In Böger P, Wakabayashi K, Hirai K (eds.). Herbicide Classes in Development. Springer Science & Business Media. pp. 179–195. doi:10.1007/978-3-642-59416-8_10. ISBN 978-3-642-59416-8. Archived from the original on 27 June 2022. Retrieved 27 June 2022.
Chapter 10.2.1: Sulfonylurea acetolactate synthase inhibitors
- ^ Duke SO (July 1990). "Overview of herbicide mechanisms of action". Environmental Health Perspectives. 87: 263–271. doi:10.2307/3431034. JSTOR 3431034. PMC 1567841. PMID 1980104.
- ^ Ganguly AK, Alluri SS (12 September 2021). "Chapter 2: Enzyme Inhibitors". Medicinal Chemistry: A Look at How Drugs Are Discovered. Boca Raton, Florida: CRC Press. pp. 29–68. ISBN 978-1-00-043721-8.
- ^ Rufer AC (April 2021). "Drug discovery for enzymes". Drug Discovery Today. 26 (4): 875–886. doi:10.1016/j.drudis.2021.01.006. PMID 33454380. S2CID 231633284.
- ^ Lindquist RN (October 2013). "The design of enzyme inhibitors: Transition state analogues.". In Ariëns EJ (ed.). Drug Design: Medicinal Chemistry: A Series of Monographs. Vol. 5. Elsevier. pp. 24–80.
- ^ Scapin G (2006). "Structural biology and drug discovery". Current Pharmaceutical Design. 12 (17): 2087–2097. doi:10.2174/138161206777585201. PMID 16796557.
- ^ Gohlke H, Klebe G (August 2002). "Approaches to the description and prediction of the binding affinity of small-molecule ligands to macromolecular receptors". Angewandte Chemie. 41 (15): 2644–2676. doi:10.1002/1521-3773(20020802)41:15<2644::AID-ANIE2644>3.0.CO;2-O. PMID 12203463.
- ^ Koppitz M, Eis K (June 2006). "Automated medicinal chemistry". Drug Discovery Today. 11 (11–12): 561–568. doi:10.1016/j.drudis.2006.04.005. PMID 16713909.
- ^ Ciulli A, Abell C (December 2007). "Fragment-based approaches to enzyme inhibition". Current Opinion in Biotechnology. 18 (6): 489–96. doi:10.1016/j.copbio.2007.09.003. PMC 4441723. PMID 17959370.
- ^ Satz AL, Kuai L, Peng X (May 2021). "Selections and screenings of DNA-encoded chemical libraries against enzyme and cellular targets". Bioorganic & Medicinal Chemistry Letters. 39: 127851. doi:10.1016/j.bmcl.2021.127851. PMID 33631371. S2CID 232059044.
- ^ Perez O, Pena J, Fernandez-Vega V, Scampavia L, Spicer T (2019). "Chapter 4: High Throughput Screening". Drug Discovery and Development. Boca Raton, Florida: CRC Press. pp. 47–69. ISBN 978-1-315-11347-0.
- ^ Scarpino A, Ferenczy GG, Keserű GM (2020). "Covalent Docking in Drug Discovery: Scope and Limitations". Current Pharmaceutical Design. 26 (44): 5684–5699. doi:10.2174/1381612824999201105164942. PMID 33155894. S2CID 226271153.
- ^ Elsässer B, Goettig P (March 2021). "Mechanisms of Proteolytic Enzymes and Their Inhibition in QM/MM Studies". International Journal of Molecular Sciences. 22 (6): 3232. doi:10.3390/ijms22063232. PMC 8004986. PMID 33810118.
외부 링크
- "BRENDA". Archived from the original on 1 April 2022.각 항목에 대해 알려진 억제제 목록을 제공하는 "BRENDA". Archived from the original on 1 April 2022.효소의 데이터베이스
- "PubChem". National Center for Biotechnology Information. National Library of Medicine. 의약품 및 효소억제제 데이터베이스
- "Symbolism and Terminology in Enzyme Kinetics". Archived from the original on 20 June 2006. 효소 억제 용어에 대한 국제생화학연합(NC-IUB) 명명위원회의 권고사항
![{\displaystyle {\begin{aligned}{\cfrac {V_{\max }}{1+{\cfrac {\ce {[I]}}{K_{i}}}}}&={V_{\max }}\left({\cfrac {K_{i}}{K_{i}+[{\ce {I}}]}}\right)&&{\text{multiply by }}{\cfrac {K_{i}}{K_{i}}}=1\\&={V_{\max }}\left({\cfrac {K_{i}+[{\ce {I}}]-[{\ce {I}}]}{K_{i}+[{\ce {I}}]}}\right)&&{\text{add }}[{\ce {I}}]-[{\ce {I}}]=0{\text{ to numerator}}\\&={V_{\max }}\left(1-{\cfrac {[{\ce {I}}]}{K_{i}+[{\ce {I}}]}}\right)&&{\text{simplify }}{\cfrac {K_{i}+[{\ce {I}}]}{K_{i}+[{\ce {I}}]}}=1\\&=V_{\max }-V_{\max }{\cfrac {\ce {[I]}}{K_{i}+[{\ce {I}}]}}&&{\text{multiply out by }}V_{\max }\end{aligned}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/37eda4dec307f8acfca89b2d8f4811474ea764ec)
![{\displaystyle {\cfrac {\ce {[S]}}{[{\ce {S}}]+K_{m}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/eb08dd139085a394e6e7370f47ebfa255f1ad685)
![{\displaystyle {\cfrac {\ce {[I]}}{[{\ce {I}}]+K_{i}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/9ed50a1f7a5f2c52f406b52263916ab48b268e07)
![{\displaystyle V_{\max }-\Delta V_{\max }{\cfrac {\ce {[I]}}{[{\ce {I}}]+K_{i}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/7dff424ec79284c3a1cea14f0f82b0eaace53c69)
![{\displaystyle V_{\max 1}-(V_{\max 1}-V_{\max 2}){\cfrac {\ce {[I]}}{[{\ce {I}}]+K_{i}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/f3874623edd9524ba2741fe448927bf5cf0ab257)
![{\displaystyle V_{\max 1}-(V_{\max 1}-V_{\max 2}){\cfrac {\ce {[X]}}{[{\ce {X}}]+K_{x}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/306d44733a89308883053e3b8372a8cf9ce0239b)
![{\displaystyle K_{m1}-(K_{m1}-K_{m2}){\cfrac {\ce {[X]}}{[{\ce {X}}]+K_{x}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/cb4e0de216e1e625bb803ee725bf85c9989a15f5)
![V={\frac {V_{max}[S]}{\alpha K_{m}+\alpha ^{\prime }[S]}}={\frac {(1/\alpha ^{\prime })V_{max}[S]}{(\alpha /\alpha ^{\prime })K_{m}+[S]}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/4a8f0a9dda1d308de7f090f99c2833f944f11a09)
![\alpha =1+{\frac {[I]}{K_{i}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/57fcf54938a9784f9313437681b220079ff43ee5)
![\alpha ^{\prime }=1+{\frac {[I]}{K_{i}^{\prime }}}.](https://wikimedia.org/api/rest_v1/media/math/render/svg/65bf16742482cae7b0743781f47c327ddcf537e3)