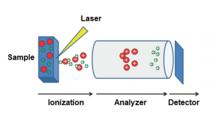
매트릭스 지원 레이저 탈리/이온화
Matrix-assisted laser desorption/ionization질량분석학에서 MALDI(Matrix-Assisted Laser Desolution/Ionization)는 레이저 에너지 흡수 매트릭스를 사용하여 최소한의 [1]조각으로 큰 분자에서 이온을 생성하는 이온화 기술이다.그것은 생체분자(DNA, 단백질, 펩타이드 및 탄수화물 등의 생체고분자)와 다양한 유기분자(폴리머, 덴드리머 및 기타 고분자)의 분석에 적용되어 왔으며, 보다 전통적인 이온화 방법에 의해 이온화되면 부서지고 조각화되는 경향이 있다.MALDI는 일반적으로 다중 전하 이온을 훨씬 적게 발생시키지만, 두 기술 모두 기체 단계에서 큰 분자의 이온을 얻는 비교적 부드러운(낮은 조각화) 방식이라는 점에서 일렉트로스프레이 이온화(ESI)와 유사합니다.
MALDI 방법론은 3단계 프로세스입니다.우선 시료를 적절한 매트릭스 재료와 혼합하여 금속판에 도포한다.둘째, 펄스 레이저가 시료를 조사하여 시료 및 매트릭스 재료의 절제 및 탈착을 트리거한다.마지막으로 분석물 분자를 압출가스의 열융기 내에서 양성자화 또는 탈양성자화함으로써 이온화시킨 후 [2]이를 분석하기 위해 사용하는 질량분석계 중 하나로 가속할 수 있다.
역사
매트릭스 지원 레이저 탈착 이온화(MALDI)라는 용어는 1985년 Franz Hillenkamp, Michael Karas 및 그 [3]동료들에 의해 만들어졌습니다.이 연구원들은 아미노산 알라닌이 아미노산 트립토판과 섞이고 펄스 266 nm 레이저를 조사하면 더 쉽게 이온화될 수 있다는 것을 발견했다.트립토판은 레이저 에너지를 흡수하고 비흡수성 알라닌을 이온화하는 것을 도왔다.2843 Da 펩타이드 멜리틴까지의 펩타이드는 이러한 종류의 "매트릭스"[4]와 혼합될 때 이온화될 수 있었다.대분자 레이저 탈착 이온화의 돌파구는 1987년 시마즈사의 다나카 고이치(田中光一)와 그의 동료들이 글리세롤의 30nm 코발트 입자와 337nm 질소 레이저를 이온화를 [5]위해 결합하는 이른바 "초미세 금속 플러스 액체 매트릭스법"을 사용하면서 이루어졌다.이 레이저와 매트릭스의 조합을 이용해 다나카는 34,472Da 단백질 카르복시펩티드가수분해효소 A와 같은 크기의 생체분자를 이온화할 수 있었다.다나카는 레이저 파장과 매트릭스의 적절한 조합으로 단백질을 [6]이온화할 수 있다는 것을 증명한 공로로 2002년 노벨 화학상의 4분의 1을 수상했다.이후 Karas와 Hillenkamp는 니코틴산 매트릭스와 266 nm [7]레이저를 사용하여 67 kDa 단백질 알부민을 이온화할 수 있었다.355 nm 레이저와 계피산 유도체인 페룰산, 카페산 및 시나핀산을 [8]매트릭스로 사용함으로써 한층 더 개선되었다.337 nm 파장에서 작동하는 작고 비교적 저렴한 질소 레이저의 가용성과 1990년대 초에 도입된 첫 번째 상용 장비는 MALDI를 더 많은 수의 [9]연구자들에게 가져다주었다.오늘날 MALDI 질량 분석에는 대부분 유기 매트릭스가 사용됩니다.
매트릭스
| 컴파운드 | 기타 이름 | 용제 | 파장(nm) | 적용들 |
|---|---|---|---|---|
| 2,5-디히드록시벤조산(젠티스산)[10] | DHB, 겐티신산 | 아세토니트릴, 물, 메탄올, 아세톤, 클로로포름 | 337, 355, 266 | 펩타이드, 뉴클레오티드, 올리고뉴클레오티드, 올리고당 |
| 3,5-디메톡시-4-히드록시신나민산[8][11] | 시나핀산 | 아세토니트릴, 물, 아세톤, 클로로포름 | 337, 355, 266 | 펩타이드, 단백질, 지질 |
| 4-히드록시-3-메톡시신나민산[8][11] | 페룰산 | 아세토니트릴, 물, 프로판올 | 337, 355, 266 | 단백질 |
| α-시아노-4-히드록시신나민산[12] | CHCA | 아세토니트릴, 물, 에탄올, 아세톤 | 337, 355 | 펩타이드, 지질, 뉴클레오티드 |
| 피콜린산[13] | PA | 에탄올 | 266 | 올리고뉴클레오티드 |
| 3-히드록시피콜린산[14] | HPA | 에탄올 | 337, 355 | 올리고뉴클레오티드 |
매트릭스는 결정화된 분자로 구성되며, 그 중 가장 일반적으로 사용되는 세 가지는 시나핀산, α-시아노-4-히드록시신나민산(α-CHCA, α-시아노 또는 α-매트릭스) 및 2,5-디히드록시 안식향산(DHB)[15]이다.이들 분자 중 하나의 용액은 종종 고도로 정제된 물과 아세토니트릴(ACN) 또는 에탄올과 같은 유기용매의 혼합물로 만들어진다.보통 트리플루오로아세트산(TFA) 등의 역이온원을 첨가하여 [M+H]이온을 생성한다.매트릭스 용액의 좋은 예는 ACN의 20mg/mL 시나핀산: 물:TFA(50:50:0.1).
적절한 매트릭스 화합물의 식별은 시행착오를 통해 어느 정도 결정되지만, 그것들은 몇 가지 특정한 분자 설계 고려사항에 기초한다.분자량이 상당히 낮지만(증발이 용이하지만), 샘플 준비 중 또는 질량 분석계에 서 있는 동안 증발하지 않을 만큼 충분히 크다(증기 압력이 낮다).그것들은 종종 산성이기 때문에 분석물질의 이온화를 촉진하는 양성자 공급원으로 작용한다.기본 매트릭스도 [16]보고되었다.UV 또는 IR [17]범위에서 강한 광학적 흡수를 가지고 있기 때문에 레이저 조사를 빠르고 효율적으로 흡수할 수 있습니다.이러한 효율은 계피산의 구조에서 볼 수 있듯이 여러 개의 켤레 이중 결합을 포함하는 화학 구조와 일반적으로 관련이 있다.극성기로 기능하여 수용액에서 사용할 수 있습니다.그것들은 전형적으로 발색단체를 포함하고 있다.
매트릭스 용액은 분석물과 혼합된다(예: 단백질 샘플).물과 유기 용제의 혼합물은 소수성 및 수용성(친수성) 분자를 용액에 녹일 수 있게 한다.이 용액은 MALDI 플레이트(일반적으로 이 용도를 위해 설계된 금속 플레이트)에 부착되어 있습니다.용제는 증발하여 재결정화된 매트릭스만 남지만 이제는 MALDI 결정에 분석 물질 분자가 박혀 있습니다.매트릭스와 분석물은 공결정화된다고 한다.공결정화는 관심 있는 분석물질의 양질의 질량 스펙트럼을 얻기 위해 적절한 매트릭스를 선택할 때 중요한 문제이다.
생물계 분석에서는 단백질 추출물의 일부인 무기염이 이온화 과정을 방해한다.소금은 고상 추출 또는 건조된 MALDI 스팟을 [18]찬물로 세척하여 제거할 수 있습니다.두 방법 모두 샘플에서 다른 물질을 제거할 수 있습니다.매트릭스-단백질 혼합물은 동일하지 않습니다. 왜냐하면 극성 차이로 인해 공결정화 중에 두 물질이 분리되기 때문입니다.타깃의 스폿 직경은 레이저의 스폿 직경보다 훨씬 크기 때문에 타깃의 다른 장소에서 많은 레이저를 쏴야 타깃 스팟 내의 물질 농도의 통계적 평균을 얻을 수 있다.
매트릭스를 사용하여 다양한 방법으로 검체를 이온화하도록 기기를 조정할 수 있습니다.앞에서 설명한 바와 같이 시료를 이온화하기 위해 산염기상반응을 이용하는 경우가 많지만, 나프탈렌상화합물 등 복합파이계를 가진 분자는 전자수용체로서도 기능할 수 있어 MALDI/[19]TOF의 매트릭스로서도 사용할 수 있다.이것은 켤레 파이 시스템을 [20]가지고 있는 분자를 연구하는데 특히 유용하다.이러한 매트릭스에 가장 널리 사용되는 것은 클로로필과 같은 포르피린 유사 화합물을 연구하는 것입니다.이들 매트릭스는 홀수 파편화 패턴이나 사이드 [21]체인의 완전한 손실을 초래하지 않는 더 나은 이온화 패턴을 가지고 있는 것으로 나타났습니다.또한 복합 포르피린과 같은 분자가 매트릭스 역할을 할 수 있고 별도의 매트릭스 [22]화합물의 필요성을 없애면서 스스로를 절단할 수 있다는 것이 제안되었다.
인스트루먼트
MALDI 기술에는 여러 가지 변형이 있으며, 오늘날에는 매우 다른 목적을 위해 유사한 계측기가 생산되고 있습니다.보다 학술적이고 분석적인 것에서 보다 산업적이고 높은 스루풋으로.질량 분석 분야는 FT-ICR 기구와[23][24] 같은 초고해상도 질량 분석뿐만 아니라 더 많은 처리량 [25]기구도 요구하는 분야로 확장되었다.많은 MALDI MS 기기들이 교환 가능한 이온화 소스(전기 프라이 이온화, MALDI, 대기압 이온화 등)와 함께 구입될 수 있기 때문에, 이러한 기술들은 종종 중복되며, 여러 번 어떠한 소프트 이온화 방식도 잠재적으로 사용될 수 있습니다.소프트 이온화 방법에 대한 자세한 내용은 소프트 레이저 탈착 또는 이온 소스를 참조하십시오.
레이저
MALDI 기술은 일반적으로 질소 레이저(337 nm) 및 주파수 3배 및 4배 Nd:YAG 레이저(각각 [26]355 nm 및 266 nm).
적외선 MALDI에 사용되는 적외선 레이저 파장은 2.94μm Er:YAG 레이저, 미드 IR 광학 파라미터 발진기 및 10.6μm 이산화탄소 레이저.적외선 레이저는 일반적으로는 아니지만 이온화 [27]모드가 부드러우므로 사용됩니다.IR-MALDI는 또한 생물학적 샘플에 유용한 더 큰 재료 제거, 낮은 질량 간섭 및 매트릭스 프리 레이저 탈착 질량 분석 방법과의 호환성이 있다는 장점이 있습니다.
비행시간
MALDI와 함께 가장 널리 사용되는 질량 분석계의 유형은 주로 질량 범위가 크기 때문에 비행 시간 분석계(TOF)입니다.TOF 측정 절차는 펄스 레이저가 연속 작동에서 작동하지 않고 개별 '샷'을 찍기 때문에 MALDI 이온화 프로세스에도 이상적입니다.MALDI-TOF 계측기는 종종 전계를 이용하여 이온을 반사하는 반사체('이온 미러')를 갖추고 있습니다.이로 인해 이온 비행 경로가 증가하여 m/z가 다른 이온 간의 비행 시간이 증가하고 분해능이 높아집니다.최신 상용 리플렉트론 TOF 계측기는 50,000 FWHM(전폭 하프 최대, 피크 높이의 50%에서 피크 폭으로 정의됨) 이상의 [28]분해능 m/δm에 도달합니다.
MALDI는 인산화 및 비인산화 [29][30]펩타이드를 식별하기 위해 IMS-TOF MS와 결합되었다.
MALDI-FT-ICR MS는 고해상도 MALDI-MS 측정이 [31]필요한 경우 유용한 기술로 입증되었습니다.
기압
대기압(AP) 매트릭스 지원 레이저 탈착/이온화(MALDI)는 진공 MALDI와 대조적으로 정상적인 [32]대기 환경에서 작동하는 이온화 기술(이온원)입니다.진공 MALDI와 AP-MALDI의 주요 차이점은 이온이 생성되는 압력입니다.진공 MALDI에서는 일반적으로 10 mTorr 이하에서 이온이 생성되며 AP-MALDI에서는 대기압에서 이온이 생성된다.기존 진공 MALDI에 비해 AP-MALDI 기법의 주요 단점은 감도가 제한적이라는 것이지만, 이온이 질량 분석계에 고효율로 전달될 수 있다는 점 및 아토몰 검출 한계가 [33]보고되었습니다.AP-MALDI는 단백질학에서 약물 발견까지 다양한 응용 분야에서 질량 분석(MS)에 사용됩니다.AP-MALDI 질량 분석으로 다루어지는 인기 있는 주제에는 단백질학, DNA, RNA, PNA, 지질, 올리고당, 포스포펩타이드, 박테리아, 소분자 및 합성 폴리머의 질량 분석, 진공 MALDI 기구에도 유사한 응용이 포함된다.AP-MALDI 이온원은 이온 트랩 질량 분석계[34] 또는 Electrospray Ionization(ESI) 또는 nanoESI 소스를 갖춘 다른 MS 시스템에 쉽게 결합됩니다.
에어로졸
에어로졸 질량 분석에서 이온화 기술 중 하나는 개별 물방울에 레이저를 발사하는 것입니다.이러한 시스템을 단일 입자 질량 분석계(SPMS)[35]라고 합니다.에어로졸화 전에 샘플과 MALDI 매트릭스를 혼합할 수도 있습니다.
이온화 메커니즘
건조된 액체 스폿의 매트릭스 결정을 향해 레이저가 발사됩니다.매트릭스는 레이저 에너지를 흡수하며, 주로 이 이벤트에 의해 매트릭스가 탈착 및 이온화된다고 생각됩니다(양성자 추가).절제 과정에서 생성되는 열융기는 중성화 및 이온화 매트릭스 분자, 양성자화 및 탈양성자 매트릭스 분자, 매트릭스 클러스터 및 나노드로플릿 등 많은 종을 포함합니다.MALDI의 메커니즘은 여전히 논의되고 있지만, Ablation 종은 분석 물질의 이온화에 참여할 수 있다.매트릭스는 양자를 분석 물질 분자(예: 단백질 분자)로 전달하여 분석 [36]물질을 충전하는 것으로 생각됩니다.이 과정 후에 관찰된 이온은 이온이 첨가 또는 제거된 초기 중성 분자 [M]로 구성됩니다.이를 준분자 이온이라고 하는데, 예를 들어 첨가된 양성자의 경우 [M++H], 첨가된 나트륨 이온의 경우 [M+Na],+ 제거된 양성자의 경우 [M-H]−라고 한다.MALDI는 매트릭스의 특성, 레이저 강도 및/또는 사용되는 전압에 따라 단일 전하 이온을 생성하거나 [M+nH]n+를 증배할 수 있습니다.이것들은 모두 짝수 전자종입니다.예를 들어 매트릭스 분자와 다른 유기 분자의 경우 래디칼 양이온(광전자화 분자)의 이온 신호를 관찰할 수 있다.
UV 레이저 MALDI의 결합 물리 화학 역학([37]CPCD) 모델로 구현된 기상 양성자 전달 [2]모델은 이온화를 [38]유도하는 1차 및 2차 프로세스를 가정합니다.1차 공정은 매트릭스에 의한 광자의 흡수를 통한 초기 전하 분리와 매트릭스 이온 쌍을 형성하기 위한 에너지 풀을 포함한다.1차 이온 형성은 UV 광자의 흡수를 통해 일어난다.
- S0 + hµ → S1
- S1 + S1 → S0 + Sn
- S1 + Sn → M+ + M−
여기서0 S는 지면 전자 상태, S는1 첫 번째 전자 들뜸 상태,[37] S는n 더 높은 전자 들뜸 상태입니다.생성물 이온은 위의 M과− M에서 나타내는+ 양성자 전달 또는 전자 전달 이온 쌍이 될 수 있습니다.2차 과정은 이온 분자 반응을 통해 분석물 이온을 형성합니다.
행운의 생존자 모델(클러스터 이온화 메커니즘[2])은 분석물 분자가 [39][40]용액에서 전하 상태를 유지하는 매트릭스에 통합된다고 가정합니다.이온 형성은 레이저 압출 클러스터 [2]파편화 시 전하 분리에 의해 발생합니다.광전자나 역이온과의 재결합으로 중화되지 않은 이온은 이른바 행운의 생존자다.
열모델은 고온으로 인해 용해된 매트릭스 [41]액체에서 매트릭스와 분석물 간의 양성자 전달이 용이하다고 가정합니다.이온 대 중성비는 이론 모델을 정당화하기 위한 중요한 파라미터이며, 이온 대 중성 비율을 잘못 인용하면 이온화 메커니즘을 [42]잘못 판단할 수 있습니다.이 모델은 분석물질의 농도와 양성자 친화력의 함수로서 총 이온 강도의 증가를, 레이저 [43][44]플루언스의 함수로서 이온 대 중성비의 증가를 정량적으로 예측한다.이 모델은 또한 금속 이온 부가물(예: [M+Na]+ 또는 [M+K])+이 주로 [45]소금의 열 유도 용해에서 발생함을 시사한다.
매트릭스 보조 이온화(MAI) 방법은 MALDI와 유사한 매트릭스 제제를 사용하지만 휘발성 또는 비휘발성 화합물의 분석물 이온을 생성하기 [46]위해 레이저 절제가 필요하지 않습니다.분석 물질로 매트릭스를 질량 분석계의 진공에 노출시키는 것만으로 전기로스프레이 이온화와 [47]거의 동일한 전하 상태의 이온이 생성됩니다.이 프로세스와 [40]MALDI 사이에는 기계적 공통성이 있을 수 있습니다.
이온 수율은 일반적으로 10에서−4−7 [48]10까지로 추정되며, 일부 실험에서는 수율이 [49]10까지−9 낮아지는 것으로 나타났습니다.저이온 수율 문제는 이미 MALDI 도입 직후 두 번째 [50]레이저를 이용한 포스트 이온화를 포함한 다양한 시도를 통해 해결되었다.이러한 시도는 대부분 제한적인 성공만을 보였으며, 신호 증가는 낮았습니다.이는 10 - 10의−6 소스−5 영역에서 압력으로 작동하는 축방향 비행 시간 계측기가 사용되었기 때문에 최대 1000m/[51]s의 입자 속도로 빠른 플룸 팽창이 발생한다.2015년에는 직교 비행시간 질량 분석기와 결합된 약 3mbar의 높은 압력으로 작동하는 변형 MALDI 선원을 사용하고, 매트릭스 사용의 2광자 이온화 임계값 이하인 260nm ~ 280nm 파장에서 작동하는 파장 조정 가능한 포스트 이온화 레이저를 사용하여 레이저 포스트 이온화에 성공한 것으로 보고되었다.d, 여러 지질과 작은 분자의 이온 수율을 최대 [52]3단계까지 증가시켰다.두 번째 레이저와 두 번째 MALDI 유사 이온화 과정 때문에 MALDI-2라고 불리는 이 접근방식은 이후 낮은 mbar [53][54]범위에서 작동하는 선원을 갖춘 다른 질량 분석기에 채택되었다.
적용들
생화학
프로테오믹스에서 MALDI는 겔 전기영동을 이용하여 분리된 단백질의 신속한 동정(SDS-PAGE, 사이즈 제외 크로마토그래피, 어피니티 크로마토그래피, 강약 이온 교환, 동위원소 코드 단백질 라벨링(ICPL) 및 2차원 겔 전기영동)에 사용된다.펩타이드 질량 지문은 MALDI-TOF 질량 분석기의 가장 인기 있는 분석 응용 프로그램입니다.MALDI TOF/TOF 질량분석계는 선원 붕괴 또는 고에너지 충돌 유도 해리를 이용하여 펩타이드의 아미노산 배열을 밝히는데 사용된다(자세한 용도는 질량분석 참조).
MALDI-TOF는 번역 후 수정을 특징짓기 위해 사용되어 왔다.예를 들어 단백질 메틸화 및 탈메틸화 [55][56]연구에 널리 사용되어 왔다.그러나 MALDI-TOF에 의한 번역 후 수정을 연구할 때는 주의해야 한다.예를 들어 디히드록시벤조산(DHB)이 글리코실화펩타이드의 MALDI MS 분석 매트릭스로 사용되었을 때 시알산의 손실이 논문에서 확인되었다고 보고되었다.S. Martin은 시나핀산, 4-HCCA 및 DHB를 매트릭스로 사용하여 선형 모드 및 반사기 [57]모드에서 MALDI/TOF의 준안정적 붕괴에 의한 글리코실화 펩타이드 내 시알산 손실을 연구하였다.시마즈사의 한 그룹은 검출 감도를[58] 향상시키는 방법으로 아미드화 반응에 의해 시알산을 유도하고, 시알릴화 [59]올리고당의 MALDI/TOF MS 분석 중에 이온 액체 매트릭스가 시알산 손실을 감소시킨다는 것을 증명했다.THAP,[60] DHAP [61]및 2-aza-2-티오티민과 페닐히드라진의[62] 혼합물은 글리코실화 펩타이드의 MALDI MS 분석 중 시알산 손실을 최소화하는 데 사용될 수 있는 매트릭스로 확인되었다.UV MALDI[63] 대신 IR MALDI를 사용하면 일부 번역 후 수정 손실 감소가 달성될 수 있는 것으로 보고되었습니다.
단백질 외에 [64]지질 연구에도 MALDI-TOF가 적용되고 있다.예를 들어, 포스포리파아제들의 [65][66]촉매 반응을 연구하기 위해 적용되었다.지질 외에도 올리고뉴클레오티드는 MALDI-TOF에 의해 특징지어졌다.예를 들어 분자생물학에서는 5-메톡시살리실산과 스펠민의 혼합물을 예를 들어 올리고뉴클레오티드 합성 후 [67]MALDI 질량분석의 올리고뉴클레오티드 분석 매트릭스로 사용할 수 있다.
유기화학
카테난과 로탁산과 같은 일부 합성 고분자, 덴드리머와 하이퍼브랜치 폴리머, 그리고 다른 집합체들은 분자량이 수천에서 수만까지 확장되는데, 대부분의 이온화 기술은 분자 이온을 생성하는데 어려움을 겪습니다.MALDI는 화학자가 이러한 합성 결과를 신속하게 분석하고 [citation needed]검증할 수 있는 간단하고 빠른 분석 방법입니다.
폴리머
폴리머 화학에서 MALDI는 몰 질량 [68]분포를 결정하기 위해 사용될 수 있다.다분산성이 1.2 이상인 폴리머는 높은 질량의 [69][70][71]올리고머에 대한 신호 강도 판별 때문에 MALDI로 특성화하기가 어렵다.
폴리머의 좋은 매트릭스는 디트라놀[72] 또는 AgTFA이다.[73]먼저 시료를 디트라놀과 혼합한 후 AgTFA를 첨가해야 합니다. 그렇지 않으면 시료가 용액 밖으로 침전됩니다.
미생물학
MALDI-TOF 스펙트럼은 박테리아나 곰팡이와 같은 미생물의 식별에 자주 사용된다.대상 미생물의 콜로니 일부를 시료 타깃 위에 올려 매트릭스와 겹친다.생성된 발현 단백질의 질량 스펙트럼은 전용 소프트웨어에 의해 분석되고 생물형식으로 알려진 종 결정을 위한 저장된 프로파일과 비교된다.그것은 다른 면역학적 또는 생화학적 절차에 이점을 제공하며 임상 미생물학 실험실에서 [74][75]종 식별을 위한 일반적인 방법이 되었다.푸리에 변환 이온 사이클로트론 공명 질량 분석법(FT-MS로도 알려져 있음)에 대해 수행된 고해상도 MALDI-MS의 이점은 단백질형이라고 알려진 단일 이온 검출을 통해 바이러스를 타이핑 및 서브타이핑하는 데 있어 특히 인플루엔자 [76]바이러스에 초점을 맞춘 것으로 입증되었습니다.
다른 미생물학적 식별방법에 비해 중요한 장점 중 하나는 다양한 미생물을 저비용으로 분리하는 데 사용되는 선택 배지에서 직접 빠르고 안정적으로 식별할 수 있다는 것이다.용의자 또는 "추정[77]" 식민지를 정화시킬 필요가 없기 때문에 훨씬 더 빠른 처리 시간이 허용된다.예를 들어, MALDI-TOF는 혈액 [78]배양에서 직접 박테리아를 검출하는 데 사용될 수 있다는 것이 입증되었습니다.
또 다른 장점은 세균의 항생제 감수성을 예측할 수 있는 잠재력이다.단일 질량 스펙트럼 피크는 황색포도상구균[79] MALDI의 메티실린 내성을 예측할 수 있으며, 아시네토박터 바우마니[81] [80][82]및 클렙시엘라 폐렴을 포함한 카르바페넴 내성 장내박테리아과의 카르바페네마아제를 검출할 수도 있다.그러나 항생제 내성을 매개하는 대부분의 단백질은 단백질 피크 해석 시 MALDI-TOF의 2000~20,000Da 범위보다 크며, 2011년 NIH에서 발생한 Klebsiella pneumne carbapenemase(KPC) 발생과 같이 간혹 피크와 저항 전달 단백질 간의 상관관계를 [83]만들 수 있다.
기생충학
MALDI-TOF 스펙트럼은 트라이파노소마이드,[84] 라이슈마니아 및 플라스모듐과 [86]같은 다양한 기생충의 검출 및 식별에 사용되었다.이들 단세포 기생충에 더해 MALDI/TOF를 트레마토드의 [88]자유수영 단계인 이나[87] 세카리아 등의 기생 곤충의 동정에도 사용할 수 있다.
약
MALDI-TOF 스펙트럼은 질병 진단에 다른 분석 및 스펙트럼 분석 기법과 함께 사용되는 경우가 많다.MALDI/TOF는 X선 결정학에서 결정 구조를 해결하는 [citation needed]데 필요한 기술이나 시간이나 비용이나 연산 능력 없이 단백질의 신속한 식별과 단백질로의 변화를 가능하게 하기 때문에 많은 잠재력을 가진 진단 도구이다.
이것의 한 예는 괴사성 장염인데, 이것은 미숙아의 장에 영향을 미치는 파괴적인 질병이다.NEC의 증상은 패혈증과 매우 유사하며 많은 유아들이 진단과 치료를 기다리며 사망한다.MALDI/TOF는 NEC 양성 유아의 분변 물질에 존재하는 박테리아를 식별하기 위해 사용되었다.이 연구는 NEC와 관련된 분변 미생물군의 특성에 초점을 맞췄으며 질병의 메커니즘을 다루지 않았다.유사한 기술을 시퀀싱이 [89]필요 없는 빠른 진단 도구로 사용할 수 있을 것으로 기대하고 있습니다.
MALDI/TOF의 진단력의 또 다른 예는 암 영역이다.췌장암은 여전히 가장 치명적이고 암 진단이 어려운 암 [90]중 하나이다.막 단백질의 돌연변이로 인한 세포 신호 장애는 오랫동안 췌장암에 [91]기여하는 것으로 의심되어 왔다.MALDI/TOF는 췌장암과 관련된 막단백질을 식별하기 위해 사용되어 왔으며, 어느 시점에서는 조기 발견 [92][non-primary source needed]기술로 사용될 수도 있다.
MALDI/TOF는 잠재적으로 치료 및 진단을 지시하는 데 사용될 수 있습니다.MALDI/TOF는 박테리아, 특히 β-락탐(페니실린 패밀리)에 대한 약제 내성을 결정하는 방법으로서 기능한다.MALDI/TOF는 표준 항생제에 대한 약물 내성을 나타내는 카르바페네마아제의 존재를 감지합니다.이는 불과 3시간 안에 약물에 내성이 있는 박테리아를 식별하는 방법이 될 수 있을 것으로 예측된다.이 기술은 의사들이 초기에 [93]보다 공격적인 항생제를 처방할지 여부를 결정하는데 도움을 줄 수 있다.
단백질 복합체 검출
일부 펩타이드-펩타이드 복합체가 MALDI 퇴적 및 [94]이온화에서 생존할 수 있다는 초기 관측에 따라 MALDI-MS를 이용한 대규모 단백질 복합체에 대한 연구가 [95][96]보고되었다.
「 」를 참조해 주세요.
레퍼런스
- ^ Hillenkamp, Franz; Karas, Michael; Beavis, Ronald C.; Chait, Brian T. (1991). "Matrix-assisted laser desorption/ionization mass spectrometry of biopolymers". Analytical Chemistry. 63 (24): 1193A–1203A. doi:10.1021/ac00024a002. ISSN 0003-2700. PMID 1789447.
- ^ a b c d Karas, Michael; Krüger, Ralf (2003). "Ion Formation in MALDI: The Cluster Ionization Mechanism". Chemical Reviews. 103 (2): 427–440. doi:10.1021/cr010376a. ISSN 0009-2665. PMID 12580637.
- ^ Karas, M.; Bachmann, D.; Hillenkamp, F. (1985). "Influence of the Wavelength in High-Irradiance Ultraviolet Laser Desorption Mass Spectrometry of Organic Molecules". Analytical Chemistry. 57 (14): 2935–9. doi:10.1021/ac00291a042.
- ^ Karas, M.; Bachmann, D.; Bahr, U.; Hillenkamp, F. (1987). "Matrix-Assisted Ultraviolet Laser Desorption of Non-Volatile Compounds". International Journal of Mass Spectrometry and Ion Processes. 78: 53–68. Bibcode:1987IJMSI..78...53K. doi:10.1016/0168-1176(87)87041-6.
- ^ Tanaka, K.; Waki, H.; Ido, Y.; Akita, S.; Yoshida, Y.; Yoshida, T.; Matsuo, T. (1988). "Protein and Polymer Analyses up to m/z 100 000 by Laser Ionization Time-of flight Mass Spectrometry". Rapid Communications in Mass Spectrometry. 2 (20): 151–3. Bibcode:1988RCMS....2..151T. doi:10.1002/rcm.1290020802.
- ^ Markides, K.; Gräslund, A. (9 October 2002). "Advanced information on the Nobel Prize in Chemistry 2002" (PDF). The Royal Swedish Academy of Sciences. pp. 1–13. Retrieved 2013-08-28.
- ^ Karas, M.; Hillenkamp, F. (1988). "Laser desorption ionization of proteins with molecular masses exceeding 10,000 daltons". Analytical Chemistry. 60 (20): 2299–301. doi:10.1021/ac00171a028. PMID 3239801.
- ^ a b c Beavis, R. C.; Chait, B. T.; Standing, K. G. (1989). "Matrix-assisted laser-desorption mass spectrometry using 355 nm radiation". Rapid Communications in Mass Spectrometry. 3 (12): 436–9. Bibcode:1989RCMS....3..436B. doi:10.1002/rcm.1290031208. PMID 2520224.
- ^ Karas, M.; Bahr, U. (1990). "Laser Desorption Ionization Mass Spectrometry of Large Biomolecules". Trends in Analytical Chemistry. 9 (10): 321–5. doi:10.1016/0165-9936(90)85065-F.
- ^ Strupat, K.; Karas, M.; Hillenkamp, F. (1991). "2,5-Dihidroxybenzoic acid: A new matrix for laser desorption—ionization mass spectrometry". International Journal of Mass Spectrometry and Ion Processes. 72 (111): 89–102. Bibcode:1991IJMSI.111...89S. doi:10.1016/0168-1176(91)85050-V.
- ^ a b Beavis, R. C.; Chait, B. T.; Fales, H. M. (1989). "Cinnamic acid derivatives as matrices for ultraviolet laser desorption mass spectrometry of proteins". Rapid Communications in Mass Spectrometry. 3 (12): 432–5. Bibcode:1989RCMS....3..432B. doi:10.1002/rcm.1290031207. PMID 2520223.
- ^ Beavis, R. C.; Chaudhary, T.; Chait, B. T. (1992). "α-Cyano-4-hydroxycinnamic acid as a matrix for matrix-assisted laser desorption mass spectrometry". Organic Mass Spectrometry. 27 (2): 156–8. doi:10.1002/oms.1210270217.
- ^ Tang, K.; Taranenko, N. I.; Allman, S. L.; Cháng, L. Y.; Chen, C. H.; Lubman, D. M. (1994). "Detection of 500-nucleotide DNA by laser desorption mass spectrometry". Rapid Communications in Mass Spectrometry. 8 (9): 727–30. Bibcode:1994RCMS....8..727T. doi:10.1002/rcm.1290080913. PMID 7949335.
- ^ Wu, K. J.; Steding, A.; Becker, C. H. (1993). "Matrix-assisted laser desorption time-of-flight mass spectrometry of oligonucleotides using 3-hydroxypicolinic acid as an ultraviolet-sensitive matrix". Rapid Communications in Mass Spectrometry. 7 (2): 142–6. Bibcode:1993RCMS....7..142W. doi:10.1002/rcm.1290070206. PMID 8457722.
- ^ Korfmacher, Walter A. (2009). Using Mass Spectrometry for Drug Metabolism Studies. CRC Press. p. 342. ISBN 9781420092219.
- ^ Fitzgerald, M. C.; Parr, G. R.; Smith, L. M. (1993). "Basic matrixes for the matrix-assisted laser desorption/ionization mass spectrometry of proteins and oligonucleotides". Analytical Chemistry. 65 (22): 3204–11. doi:10.1021/ac00070a007. PMID 8291672.
- ^ Zenobi, R.; Knochenmuss, R. (1998). "Ion formation in MALDI mass spectrometry". Mass Spectrometry Reviews. 17 (5): 337–366. Bibcode:1998MSRv...17..337Z. doi:10.1002/(SICI)1098-2787(1998)17:5<337::AID-MAS2>3.0.CO;2-S.
- ^ Xu, Yingda; Bruening, Merlin L.; Watson, J. Throck (2003). "Non-specific, on-probe cleanup methods for MALDI-MS samples". Mass Spectrometry Reviews. 22 (6): 429–440. Bibcode:2003MSRv...22..429X. doi:10.1002/mas.10064. ISSN 0277-7037. PMID 14528495.
- ^ Nazim Boutaghou, M.; Cole, R. B. (2012). "9,10-Diphenylanthracene as a matrix for MALDI-MS electron transfer secondary reactions". J. Mass Spectrom. 47 (8): 995–1003. Bibcode:2012JMSp...47..995N. doi:10.1002/jms.3027. PMID 22899508.
- ^ Suzuki, T.; Midonoya, H.; Shioi, Y. (2009). "Analysis of chlorophylls and their derivatives by matrix-assisted laser desorption/ionization–time-of-flight mass spectrometry". Anal. Biochem. 390 (1): 57–62. doi:10.1016/j.ab.2009.04.005. hdl:10297/4147. PMID 19364490. S2CID 2634483.
- ^ Wei, J.; Li, H.; Barrow, M. P.; O'Connor, P. B. (2013). "Structural characterization of chlorophyll-a by high resolution tandem mass spectrometry". J. Am. Soc. Mass Spectrom. 24 (5): 753–760. Bibcode:2013JASMS..24..753W. doi:10.1007/s13361-013-0577-1. PMID 23504642. S2CID 43158125.
- ^ Srinivasan, N.; Haney, C. A.; Lindsey, J. S.; Zhang, W.; Chait, B. T. (1999). "Investigation of MALDI-TOF mass spectrometry of diverse synthetic metalloporphyrins, phthalocyanines and multiporphyrin arrays". Journal of Porphyrins and Phthalocyanines. 3 (4): 283–291. doi:10.1002/(SICI)1099-1409(199904)3:4<283::AID-JPP132>3.0.CO;2-F.
- ^ "Talking About a Revolution: FT-ICR Mass Spectrometry Offers High Resolution and Mass Accuracy for Pr".
- ^ Schmitt-Kopplin, P; Hertkorn, N (2007). "Ultrahigh resolution mass spectrometry". Anal Bioanal Chem. 389 (5): 1309–1310. doi:10.1007/s00216-007-1589-0. PMC 2129108.
- ^ Ghyselinck, Jonas; Van Hoorde, Koenraad; Hoste, Bart; Heylen, Kim; De Vos, Paul (September 2011). "Evaluation of MALDI-TOF MS as a tool for high-throughput dereplication". Journal of Microbiological Methods. 86 (3): 327–336. doi:10.1016/j.mimet.2011.06.004. PMID 21699925.
- ^ Dreisewerd, Klaus (2014). "Recent methodological advances in MALDI mass spectrometry". Analytical and Bioanalytical Chemistry. 406 (9–10): 2261–2278. doi:10.1007/s00216-014-7646-6. ISSN 1618-2642. PMID 24652146. S2CID 5467069.
- ^ Murray, K (2006). "Chapter 9 Desorption by Photons: Laser Desorption and Matridx-Assisted Laser Desorption Ionization (MALDI) – Infrared Matrix-Assisted Laser Desorption Ionization". In Gross, Michael L.; Caprioli, Richard M. (eds.). The Encyclopedia of Mass Spectrometry. Vol. 6: Ionization Methods. Elsevier Science. ISBN 9780080438016. Retrieved 2017-04-05.
- ^ Xian, Feng; Hendrickson, Christopher L.; Marshall, Alan G. (2012-01-17). "High Resolution Mass Spectrometry". Analytical Chemistry. 84 (2): 708–719. doi:10.1021/ac203191t. ISSN 0003-2700. PMID 22263633.
- ^ Ruotolo, B. T.; Gillig, K. J.; Woods, A. S.; Egan, T. F.; Ugarov, M. V.; Schultz, J. A.; Russell, D. H. (2004). "Analysis of Phosphorylated Peptides by Ion Mobility-Mass Spectrometry". Analytical Chemistry. 76 (22): 6727–6733. doi:10.1021/ac0498009. PMID 15538797.(설명 필요)
- ^ Ruotolo, B. T.; Verbeck, G. F.; Thomson, L. M.; Woods, A. S.; Gillig, K. J.; Russell, D. H. (2002). "Distinguishing between Phosphorylated and Nonphosphorylated Peptides with Ion Mobility−Mass Spectrometry". Journal of Proteome Research. 1 (4): 303–306. doi:10.1021/pr025516r. PMID 12645885.(설명 필요)
- ^ Pasa-Tolic, L.; Huang, Y.; Guan, S.; Kim, H. S.; Marshall, A. G. (1995). "Ultrahigh-resolution matrix-assisted laser desorption/ionization Fourier transform ion cyclotron resonance mass spectra of peptides". Journal of Mass Spectrometry. 30 (6): 825–833. Bibcode:1995JMSp...30..825P. doi:10.1002/jms.1190300607.(설명 필요)
- ^ Laiko, V. V.; Baldwin, M. A.; Burlingame, A. L. (2000). "Atmospheric pressure matrix-assisted laser desorption/ionization mass spectrometry". Analytical Chemistry. 72 (4): 652–7. doi:10.1021/ac990998k. PMID 10701247.
- ^ Strupat, K.; Scheibner, O.; Arrey, T.; Bromirski, M. (2011). "Biological Applications of AP MALDI with Thermo Scientific Exactive Orbitrap MS" (PDF). 443-539-1710. Thermo Scientific. Retrieved 17 June 2011.
- ^ Laiko, V. V.; Moyer, S. C.; Cotter, R. J. (2000). "Atmospheric pressure MALDI/ion trap mass spectrometry". Analytical Chemistry. 72 (21): 5239–43. doi:10.1021/ac000530d. PMID 11080870.
- ^ Li, Yong Jie; Sun, Yele; Zhang, Qi; Li, Xue; Li, Mei; Zhou, Zhen; Chan, Chak K. (June 2017). "Real-time chemical characterization of atmospheric particulate matter in China: A review". Atmospheric Environment. 158: 270–304. Bibcode:2017AtmEn.158..270L. doi:10.1016/j.atmosenv.2017.02.027.
- ^ Knochenmuss, R (2006). "Ion formation mechanisms in UV-MALDI". Analyst. 131 (9): 966–986. Bibcode:2006Ana...131..966K. doi:10.1039/b605646f. PMID 17047796.
- ^ a b Knochenmuss, Richard (2016). "The Coupled Chemical and Physical Dynamics Model of MALDI". Annual Review of Analytical Chemistry. 9 (1): 365–385. Bibcode:2016ARAC....9..365K. doi:10.1146/annurev-anchem-071015-041750. ISSN 1936-1327. PMID 27070182.
- ^ Knochenmuss, Richard (2006). "Ion formation mechanisms in UV-MALDI". The Analyst. 131 (9): 966–86. Bibcode:2006Ana...131..966K. doi:10.1039/b605646f. ISSN 0003-2654. PMID 17047796.
- ^ Karas, Michael; Glückmann, Matthias; Schäfer, Jürgen (2000). "Ionization in matrix-assisted laser desorption/ionization: singly charged molecular ions are the lucky survivors". Journal of Mass Spectrometry. 35 (1): 1–12. Bibcode:2000JMSp...35....1K. doi:10.1002/(SICI)1096-9888(200001)35:1<1::AID-JMS904>3.0.CO;2-0. ISSN 1076-5174. PMID 10633229.
- ^ a b McEwen, Charles N.; Larsen, Barbara S. (2015). "Fifty years of desorption ionization of nonvolatile compounds". International Journal of Mass Spectrometry. 377: 515–531. Bibcode:2015IJMSp.377..515M. doi:10.1016/j.ijms.2014.07.018. ISSN 1387-3806.
- ^ Lu, I.-Chung; Lee, Chuping; Lee, Yuan-Tseh; Ni, Chi-Kung (2015). "Ionization Mechanism of Matrix-Assisted Laser Desorption/Ionization". Annual Review of Analytical Chemistry. 8: 21–39. Bibcode:2015ARAC....8...21L. doi:10.1146/annurev-anchem-071114-040315. PMID 26132345.
- ^ Tsai, Ming-Tsang; Lee, Sheng; Lu, I-Chung; Chu, Kuan Yu; Liang, Chi-Wei; Lee, Chih Hao; Lee, Yuan T.; Ni, Chi-Kung (2013-05-15). "Ion-to-neutral ratio of 2,5-dihydroxybenzoic acid in matrix-assisted laser desorption/ionization". Rapid Communications in Mass Spectrometry. 27 (9): 955–963. Bibcode:2013RCMS...27..955T. doi:10.1002/rcm.6534. ISSN 1097-0231. PMID 23592197.
- ^ Lu, I-Chung; Lee, Chuping; Chen, Hui-Yuan; Lin, Hou-Yu; Hung, Sheng-Wei; Dyakov, Yuri A.; Hsu, Kuo-Tung; Liao, Chih-Yu; Lee, Yin-Yu (2014). "Ion Intensity and Thermal Proton Transfer in Ultraviolet Matrix-Assisted Laser Desorption/Ionization". The Journal of Physical Chemistry B. 118 (15): 4132–4139. doi:10.1021/jp5008076. ISSN 1520-6106. PMID 24707818.
- ^ Lu, I.-Chung; Chu, Kuan Yu; Lin, Chih-Yuan; Wu, Shang-Yun; Dyakov, Yuri A.; Chen, Jien-Lian; Gray-Weale, Angus; Lee, Yuan-Tseh; Ni, Chi-Kung (2015). "Ion-to-Neutral Ratios and Thermal Proton Transfer in Matrix-Assisted Laser Desorption/Ionization". Journal of the American Society for Mass Spectrometry. 26 (7): 1242–1251. Bibcode:2015JASMS..26.1242L. doi:10.1007/s13361-015-1112-3. ISSN 1044-0305. PMID 25851654. S2CID 24085468.
- ^ Lee, Chuping; Lu, I.-Chung; Hsu, Hsu Chen; Lin, Hou-Yu; Liang, Sheng-Ping; Lee, Yuan-Tseh; Ni, Chi-Kung (2016). "Formation of Metal-Related Ions in Matrix-Assisted Laser Desorption Ionization". Journal of the American Society for Mass Spectrometry. 27 (9): 1491–1498. Bibcode:2016JASMS..27.1491L. doi:10.1007/s13361-016-1424-y. ISSN 1044-0305. PMID 27306427. S2CID 2139197.
- ^ Trimpin, Sarah (2015). ""Magic" Ionization Mass Spectrometry". Journal of the American Society for Mass Spectrometry. 27 (1): 4–21. Bibcode:2016JASMS..27....4T. doi:10.1007/s13361-015-1253-4. ISSN 1044-0305. PMC 4686549. PMID 26486514.
- ^ Trimpin, S, Inutan, ED (2013). "Matrix Assisted Ionization in Vacuum, a Sensitive and Widely Applicable Ionization Method for Mass Spectrometry". J. Am. Soc. Mass Spectrom. 24 (5): 722–732. Bibcode:2013JASMS..24..722T. doi:10.1007/s13361-012-0571-z. PMID 23526166. S2CID 29978978.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Lu, I-Chung; Chu, Kuan Yu; Lin, Chih-Yuan; Wu, Shang-Yun; Dyakov, Yuri A.; Chen, Jien-Lian; Gray-Weale, Angus; Lee, Yuan-Tseh; Ni, Chi-Kung (July 2015). "Ion-to-Neutral Ratios and Thermal Proton Transfer in Matrix-Assisted Laser Desorption/Ionization". Journal of the American Society for Mass Spectrometry. 26 (7): 1242–1251. Bibcode:2015JASMS..26.1242L. doi:10.1007/s13361-015-1112-3. ISSN 1044-0305. PMID 25851654. S2CID 24085468.
- ^ Robinson, Kenneth N.; Steven, Rory T.; Race, Alan M.; Bunch, Josephine (July 2019). "The Influence of MS Imaging Parameters on UV-MALDI Desorption and Ion Yield". Journal of the American Society for Mass Spectrometry. 30 (7): 1284–1293. Bibcode:2019JASMS..30.1284R. doi:10.1007/s13361-019-02193-8. ISSN 1044-0305. PMID 30949969. S2CID 96435099.
- ^ Spengler, B.; Bahr, U.; Karas, M.; Hillenkamp, F. (January 1988). "Postionization of Laser-Desorbed Organic and Inorganic Compounds in a Time of Flight Mass Spectrometer". Instrumentation Science & Technology. 17 (1–2): 173–193. Bibcode:1988IS&T...17..173S. doi:10.1080/10739148808543672. ISSN 1073-9149.
- ^ Dreisewerd, Klaus (February 2003). "The Desorption Process in MALDI". Chemical Reviews. 103 (2): 395–426. doi:10.1021/cr010375i. ISSN 0009-2665. PMID 12580636.
- ^ Soltwisch, J.; Kettling, H.; Vens-Cappell, S.; Wiegelmann, M.; Muthing, J.; Dreisewerd, K. (2015-04-10). "Mass spectrometry imaging with laser-induced postionization". Science. 348 (6231): 211–215. Bibcode:2015Sci...348..211S. doi:10.1126/science.aaa1051. ISSN 0036-8075. PMID 25745064. S2CID 206632790.
- ^ Soltwisch, Jens; Heijs, Bram; Koch, Annika; Vens-Cappell, Simeon; Höhndorf, Jens; Dreisewerd, Klaus (2020-07-07). "MALDI-2 on a Trapped Ion Mobility Quadrupole Time-of-Flight Instrument for Rapid Mass Spectrometry Imaging and Ion Mobility Separation of Complex Lipid Profiles". Analytical Chemistry. 92 (13): 8697–8703. doi:10.1021/acs.analchem.0c01747. ISSN 0003-2700. PMID 32449347. S2CID 218874538.
- ^ Ellis, S. R.; Soltwisch, J.; Paine, M. R. L.; Dreisewerd, K.; Heeren, R. M. A. (2017). "Laser post-ionisation combined with a high resolving power orbitrap mass spectrometer for enhanced MALDI-MS imaging of lipids". Chemical Communications. 53 (53): 7246–7249. doi:10.1039/C7CC02325A. ISSN 1359-7345. PMID 28573274.
- ^ Stacie L. Richardson, Pahul Hanjra, Gang Zhang, Brianna D. Mackie, Darrell L. Peterson, Rong Huang (2015). "A direct, ratiometric, and quantitative MALDI–MS assay for protein methyltransferases and acetyltransferases". Anal. Biochem. 478: 59–64. doi:10.1016/j.ab.2015.03.007. PMC 4855292. PMID 25778392.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Karine Guitot, Thierry Drujon, Fabienne Burlina, Sandrine Sagan, Sandra Beaupierre, Olivier Pamlard, Robert H Dodd, Catherine Guillou, Gérard Bolbach, Emmanuelle Sachon, Dominique Guianvarc'h (2017). "A direct label-free MALDI-TOF mass spectrometry based assay for the characterization of inhibitors of protein lysine methyltransferases". Anal. Bioanal. Chem. 409 (15): 3767–3777. doi:10.1007/s00216-017-0319-5. PMID 28389916. S2CID 4021309.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Huberty, M. C.; Vath, J. E.; Yu, W.; Martin, S. A. (1993). "Site-specific carbohydrate identification in recombinant proteins using MALD-TOF MS". Analytical Chemistry. 65 (20): 2791–2800. doi:10.1021/ac00068a015. PMID 8250262.
- ^ Sekiya, S.; Wada, Y.; Tanaka, K. (2005). "Derivatization for Stabilizing Sialic Acids in MALDI-MS". Analytical Chemistry. 77 (15): 4962–4968. doi:10.1021/ac050287o. PMID 16053310.
- ^ Fukuyama, Y.; Nakaya, S.; Yamazaki, Y.; Tanaka, K. (2008). "Ionic Liquid Matrixes Optimized for MALDI-MS of Sulfated/Sialylated/Neutral Oligosaccharides and Glycopeptides". Analytical Chemistry. 80 (6): 2171–2179. doi:10.1021/ac7021986. PMID 18275166.
- ^ Papac, D. I.; Wong, A.; Jones, A. J. S. (1996). "Analysis of Acidic Oligosaccharides and Glycopeptides by Matrix-Assisted Laser Desorption/Ionization Time-of-Flight Mass Spectrometry". Analytical Chemistry. 68 (18): 3215–3223. doi:10.1021/ac960324z. PMID 8797382.
- ^ Harvey, D. J. (1999). "Matrix-assisted laser desorption/ionization mass spectrometry of carbohydrates". Mass Spectrometry Reviews. 18 (6): 349–451. Bibcode:1999MSRv...18..349H. CiteSeerX 10.1.1.1012.1931. doi:10.1002/(SICI)1098-2787(1999)18:6<349::AID-MAS1>3.0.CO;2-H. PMID 10639030.
- ^ Lattova, E.; Chen, V. C.; Varma, S.; Bezabeh, T.; Perreault, H. (2007). "Matrix-assisted laser desorption/ionization on-target method for the investigation of oligosaccharides and glycosylation sites in glycopeptides and glycoproteins". Rapid Communications in Mass Spectrometry. 21 (10): 1644–1650. Bibcode:2007RCMS...21.1644L. doi:10.1002/rcm.3007. PMID 17465012.
- ^ Tajiri, M.; Takeuchi, T.; Wada, Y. (2009). "Distinct Features of Matrix-Assisted 6 μm Infrared Laser Desorption/Ionization Mass Spectrometry in Biomolecular Analysis". Analytical Chemistry. 81 (16): 6750–6755. doi:10.1021/ac900695q. PMID 19627133.
- ^ T. W. Jaskolla, K. Onischke, J. Schiller (2014). "2,5‐Dihydroxybenzoic acid salts for matrix‐assisted laser desorption/ionization time‐of‐flight mass spectrometric lipid analysis: Simplified spectra interpretation and insights into gas‐phase fragmentation". Rapid Commun. Mass Spectrom. 28 (12): 1353–1363. doi:10.1002/rcm.6910. PMID 24797946.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ J. Lee, Y.-K. Kim, D.-H. Min (2010). "Laser Desorption/Ionization Mass Spectrometric Assay for Phospholipase Activity Based on Graphene Oxide/Carbon Nanotube Double-Layer Films". J. Am. Chem. Soc. 132 (42): 14714–14717. doi:10.1021/ja106276j. PMID 20886850.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Nabangshu Sharma, Ries J. Langley, Chatchakorn Eurtivong, Euphemia Leung, Ryan Joseph Dixon, Emily K. Paulin, Shaun W. P. Rees, Lisa I Pilkington, David Barker, Johannes Reynisson, Ivanhoe K. H. Leung (2021). "An optimised MALDI-TOF assay for phosphatidylcholine-specific phospholipase C". Analytical Methods. 13 (4): 491–496. doi:10.1039/D0AY02208J. PMID 33432952. S2CID 231584395.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Distler, A. M.; Allison, J. (2001). "5-Methoxysalicylic acid and spermine: A new matrix for the matrix-assisted laser desorption/ionization mass spectrometry analysis of oligonucleotides". Journal of the American Society for Mass Spectrometry. 12 (4): 456–62. doi:10.1016/S1044-0305(01)00212-4. PMID 11322192. S2CID 18280663.
- ^ Schrepp, W.; Pasch, H. (2003). MALDI-TOF Mass Spectrometry of Synthetic Polymers. Springer-Verlag. ISBN 978-3-540-44259-2.
- ^ Nielen, M. W. F.; Malucha, S. (1997). "Characterization of polydisperse synthetic polymers by size-exclusion chromatography/matrix-assisted laser desorption/ionization time-of-flight mass spectrometry". Rapid Communications in Mass Spectrometry. 11 (11): 1194–1204. Bibcode:1997RCMS...11.1194N. doi:10.1002/(SICI)1097-0231(199707)11:11<1194::AID-RCM935>3.0.CO;2-L.
- ^ Wu, K. J.; Odom, R. W. (1998). "Characterizing synthetic polymers by MALDI MS". Analytical Chemistry. 70 (13): 456A–461A. doi:10.1021/ac981910q. PMID 9666717.
- ^ Schriemer, D. C.; Li, L. (1997). "Mass Discrimination in the Analysis of Polydisperse Polymers by MALDI Time-of-Flight Mass Spectrometry. 2. Instrumental Issues". Analytical Chemistry. 69 (20): 4176–4183. doi:10.1021/ac9707794.
- ^ Schaiberger, Audrey M.; Moss, Jason A. (2008). "Optimized sample preparation for MALDI mass spectrometry analysis of protected synthetic peptides". Journal of the American Society for Mass Spectrometry. 19 (4): 614–619. doi:10.1016/j.jasms.2008.01.010. ISSN 1044-0305. PMID 18295503. S2CID 5305444.
- ^ Bahr, Ute; Deppe, Andreas; Karas, Michael; Hillenkamp, Franz; Giessmann, Ulrich (1992). "Mass spectrometry of synthetic polymers by UV-matrix-assisted laser desorption/ionization". Analytical Chemistry. 64 (22): 2866–2869. doi:10.1021/ac00046a036. ISSN 0003-2700.
- ^ Seng, P.; Drancourt, M.; Gouriet, F.; La Scola, B.; Fournier, P. E.; Rolain, J. M.; Raoult, D. (2009). "Ongoing revolution in bacteriology: routine identification of bacteria by matrix-assisted laser desorption ionization time-of-flight mass spectrometry". Clinical Infectious Diseases. 49 (4): 552–3. doi:10.1086/600885. PMID 19583519.
- ^ Sandrin, Todd R.; Goldstein, Jason E.; Schumaker, Stephanie (2013). "MALDI TOF MS profiling of bacteria at the strain level: A review". Mass Spectrometry Reviews. 32 (3): 188–217. Bibcode:2013MSRv...32..188S. doi:10.1002/mas.21359. ISSN 0277-7037. PMID 22996584.
- ^ Downard, Kevin M. (2013). "Proteotyping for the rapid identification of influenza virus and other biopathogens". Chemical Society Reviews. 42 (22): 8584–8595. doi:10.1039/c3cs60081e. ISSN 1460-4744. PMID 23632861.
- ^ Capocefalo M., Ridley E. V., Tranfield E. Y. & Thompson K. C. (2015). "Ch. 9 - MALDI-TOF: A rapid microbiological confirmation technique for food and water analysis". In Cook N.; D'Agostino M.; Thompson K. C. (eds.). Molecular Microbial Diagnostic Methods - Pathways to Implementation for the Food and Water Industries. Elsevier. ISBN 978-0-12-416999-9.
{{cite book}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Seon Young Kim, Jeong Su Park, Yun Ji Hong, Taek Soo Kim, Kiho Hong, Kyoung-Ho Song, Hyunju Lee, Eu Suk Kim, Hong Bin Kim, Kyoung Un Park, Junghan Song, Sun Hoe Koo, Eui-Chong Kim (2019). "Microarray-Based Nucleic Acid Assay and MALDI-TOF MS Analysis for the Detection of Gram-Negative Bacteria in Direct Blood Cultures". Am. J. Clin. Pathol. 151 (2): 143–153. doi:10.1093/AJCP/AQY118. PMID 30383194.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Rhoads, DD (2016). "The presence of a single MALDI-TOF mass spectral peak predicts methicillin resistance in staphylococci. et al.". Diagn Microbiol Infect Dis. 86 (3): 257–261. doi:10.1016/j.diagmicrobio.2016.08.001. PMID 27568365.
- ^ Vogne, C.; et al. (2014). ""A simple, robust and rapid approach to detect carbapenemases in Gram-negative isolates by MALDI-TOF mass spectrometry " validation with triple quadripole tandem mass spectrometry, microarray and PCR". Clin Microbiol Infect. 20 (12): O1106-12. doi:10.1111/1469-0691.12715. PMID 24930405.
- ^ Abouseada, N; Raouf, M; El-Attar, E; Moez, P (2017). "Matrix-assisted laser desorption ionisation time-of-flight mass spectrometry rapid detection of carbapenamase activity in Acinetobacter baumannii isolates". Indian J Med Microbiol. 35 (1): 85–89. doi:10.4103/0255-0857.202335. PMID 28303824. S2CID 20480725.
- ^ Sakarikou, C; Ciotti, M; Dolfa, C; Angeletti, S; Favalli, C (2017). "Rapid detection of carbapenemase-producing Klebsiella pneumoniae strains derived from blood cultures by Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry (MALDI-TOF MS)". BMC Microbiol. 17 (1): 54. doi:10.1186/s12866-017-0952-3. PMC 5343375. PMID 28274205.
- ^ Lau, A.; et al. (August 2014). "A Rapid Matrix-Assisted Laser Desorption Ionization–Time of Flight Mass Spectrometry-Based Method for Single-Plasmid Tracking in an Outbreak of Carbapenem-Resistant Enterobacteriaceae". Journal of Clinical Microbiology. 52 (8): 2804–2812. doi:10.1128/JCM.00694-14. PMC 4136129. PMID 4136129. S2CID 43633405.
- ^ Avila, C. C.; Almeida, F. G.; Palmisano, G. (2016). "Direct identification of trypanosomatids by matrix-assisted laser desorption ionization-time of flight mass spectrometry (DIT MALDI-TOF MS)". Journal of Mass Spectrometry. 51 (8): 549–557. Bibcode:2016JMSp...51..549A. doi:10.1002/jms.3763. ISSN 1076-5174. PMID 27659938.
- ^ Lachaud, Laurence; Fernández-Arévalo, Anna; Normand, Anne-Cécile; Lami, Patrick; Nabet, Cécile; Donnadieu, Jean Luc; Piarroux, Martine; Djenad, Farid; Cassagne, Carole; Ravel, Christophe; Tebar, Silvia; Llovet, Teresa; Blanchet, Denis; Demar, Magalie; Harrat, Zoubir; Aoun, Karim; Bastien, Patrick; Muñoz, Carmen; Gállego, Montserrat; Piarroux, Renaud; Loeffelholz, Michael J. (2017). "Identification of Leishmania by Matrix-Assisted Laser Desorption Ionization–Time of Flight (MALDI-TOF) Mass Spectrometry Using a Free Web-Based Application and a Dedicated Mass-Spectral Library". Journal of Clinical Microbiology. 55 (10): 2924–2933. doi:10.1128/JCM.00845-17. ISSN 0095-1137. PMC 5625378. PMID 28724559.
- ^ Laroche, Maureen; Almeras, Lionel; Pecchi, Emilie; Bechah, Yassina; Raoult, Didier; Viola, Angèle; Parola, Philippe (2017). "MALDI-TOF MS as an innovative tool for detection of Plasmodium parasites in Anopheles mosquitoes". Malaria Journal. 16 (1): 5. doi:10.1186/s12936-016-1657-z. ISSN 1475-2875. PMC 5209920. PMID 28049524.
- ^ Ouarti, Basma; Laroche, Maureen; Righi, Souad; Meguini, Mohamed Nadir; Benakhla, Ahmed; Raoult, Didier; Parola, Philippe (2020). "Development of MALDI-TOF mass spectrometry for the identification of lice isolated from farm animals". Parasite. 27: 28. doi:10.1051/parasite/2020026. ISSN 1776-1042. PMC 7191974. PMID 32351208.

- ^ Huguenin, Antoine; Depaquit, Jérôme; Villena, Isabelle; Ferté, Hubert (2019). "MALDI-TOF mass spectrometry: a new tool for rapid identification of cercariae (Trematoda, Digenea)". Parasite. 26: 11. doi:10.1051/parasite/2019011. ISSN 1776-1042. PMC 6402365. PMID 30838972.

- ^ 심, K, 쇼, P. 랜델, M. J. 콕스, 맥클루어, Z.E.; Li. M. S.; Haddad., M. R., Langford, R., Cookson, M. F., M. M. M. F., M. M. M. M. F., M. M. M. M. M. M. M. M. F., Croll, J. S. K. S. 미숙아에서 괴사성 장염으로 인한 괴사성 장염이 매우 미숙아에게 괴사.클리닉. 감염.2014년 디스
- ^ Grantzdorffer, I; Carl-McGrath, S; Ebert, MP; Rocken, C (2008). "Proteomics of pancreatic cancer". Pancreas. 36 (4): 329–36. doi:10.1097/MPA.0b013e31815cc452. PMID 18437077. S2CID 29118712.
- ^ Dhillon, AS; Hagan, S; Rath, O; Kolch, W (2007). "MAP kinase signalling pathways in cancer". Oncogene. 26 (22): 3279–90. doi:10.1038/sj.onc.1210421. PMID 17496922. S2CID 17464318.
- ^ Zhung, N.; Cui, Y.; Zhou, X.; Li, T.; Han, J. 2D-MALDI-TOF-MS Bi/Tumorol과 결합된 수성 2상 칸막이 시스템을 사용하여 인간 췌장암에서 잠재적인 예측 바이오마커로서의 금지인 1의 동정.2014.
- ^ Hrabak, Jaroslav (2015)."매트릭스 보조 레이저 탈착 이온화 비행 시간 분석(MALDI-TOF MS) Meropenem 가수 분해 분석을 이용한 카르바페네마아제 검출"분자생물학의 방법(1064-3745), 1237, 페이지 91.
- ^ Woods, AS; Buchsbaum, JS; Worrall, TA; Berg, JM; Cotter, RJ (1995). "Matrix-Assisted Laser Desorption/Ionization of Non-covalently Bound Compounds". Anal. Chem. 67 (24): 4462–4465. doi:10.1021/ac00120a005.
- ^ Kiselar, JJG; Downard, KM (2000). "Preservation and Detection of Specific Antibody-Peptide Complexes by Matrix-Assisted Laser Desorption/Ionization Mass Spectrometry". J. Am. Soc. Mass Spectrom. 11 (8): 746–750. doi:10.1016/S1044-0305(00)00144-6. PMID 10937798. S2CID 33248551.
- ^ Downard, KM (2006). "2". Softly, Softly—Detection of Protein Complexes by Matrix‐Assisted Laser Desorption Ionization Mass Spectrometry. Wiley. pp. 25–43. doi:10.1002/9780470146330.ch2. ISBN 9780470146330.
참고 문헌
- Ragoussis, J.; Elvidge, G. P.; Kaur, K.; Colella, S. (2006). "Matrix-Assisted Laser Desorption/Ionisation, Time-of-Flight Mass Spectrometry in Genomics Research". PLOS Genetics. 2 (7): e100. doi:10.1371/journal.pgen.0020100. PMC 1523240. PMID 16895448.
- Hardouin, J. (2007). "Protein sequence information by matrix-assisted laser desorption/ionization in-source decay mass spectrometry". Mass Spectrometry Reviews. 26 (5): 672–82. Bibcode:2007MSRv...26..672H. doi:10.1002/mas.20142. PMID 17492750.
- Peter-Katalinic, J.; Hillenkamp, F. (2007). MALDI MS: A Practical Guide to Instrumentation, Methods and Applications. Wiley-VCH. ISBN 978-3-527-31440-9.
- Schrepp, W.; Pasch, H. (2003). MALDI-TOF Mass Spectrometry of Synthetic Polymers. Springer-Verlag. ISBN 978-3-540-44259-2.
외부 링크
- 매트릭스 지원 레이저 탈리 이온화(MALDI) 국립고자기장연구소 입문서