DnaG
DnaG| DNA프리마아제 | |||||||
|---|---|---|---|---|---|---|---|
| 식별자 | |||||||
| 유기체 | |||||||
| 기호. | dnaG | ||||||
| Alt. | dnaP | ||||||
| 엔트레즈 | 947570 | ||||||
| PDB | 1D0Q, 1DD9, 1DDE, 1EQ9, 2R6A, 2R6C | ||||||
| RefSeq(프로트) | NP_417538 | ||||||
| 유니프로트 | P0ABS5 | ||||||
| 기타 데이터 | |||||||
| EC 번호 | 2.7.7.7 | ||||||
| 염색체 | 염색체: 3.21~3.21Mb | ||||||
| |||||||
DnaG는 박테리아 DNA 프리마아제이며 dnaG 유전자에 의해 암호화된다.효소 DnaG와 다른 DNA 프리마아제는 DNA 복제 중에 올리고뉴클레오티드로 알려진 RNA의 짧은 가닥을 합성합니다.이러한 올리고뉴클레오티드는 DNA 합성의 시작점으로 작용하기 때문에 프라이머로 알려져 있다.DnaG는 10-60 뉴클레오티드(DNA와 RNA의 기본 단위) 길이의 올리고뉴클레오티드의 합성을 촉매하지만, 합성되는 올리고뉴클레오티드의 대부분은 11 뉴클레오티드이다.[1]이러한 RNA 올리고뉴클레오티드는 박테리아 DNA 중합효소 III(Pol III)에 의한 DNA 합성의 프라이머 또는 시작점 역할을 한다.DNA 중합효소는 DNA 가닥의 합성을 시작할 수 없고 기존의 [2]가닥에만 뉴클레오티드를 첨가할 수 있기 때문에 DnaG는 세균 DNA 복제에서 중요하다.DnaG는 복제의 원점에서 단일 RNA 프라이머를 합성합니다.이 프라이머는 선도적인 가닥 DNA 합성에 기여합니다.다른 부모 가닥인 지연 가닥의 경우, DnaG는 몇 킬로베이스(kb)마다 RNA 프라이머를 합성한다.이들 프라이머는 오카자키 [3]단편을 합성하는 기판 역할을 한다.
대장균 DnaG는 박테리아 복제 헬리케이스 DnaB와 비공유 상호작용을 통해 프라이머 활동을 수행하며, 3개의 DnaG 프라이머 단백질은 각 DnaB 헬리케이스와 관련지어 프라이머를 [4]형성한다.프라이머제는 단일 가닥 DNA(sssDNA) 템플릿에서 특정 세 개의 뉴클레오티드 배열에서 합성을 시작하는 경향이 있으며 대장균 DnaG의 경우 배열은 5'-[1]CTG-3'이다.
DnaG는 아연 결합 도메인, RNA 중합효소 도메인 및 DnaB 헬리케이스 결합 도메인의 세 가지 단백질 도메인을 포함한다.DNA 프리마아제 DnaG를 사용하는 여러 박테리아가 있다.DNA 프라이머아제로서 DNAG를 가지는 생물로는 대장균(E. coli), 스테아로더모필루스(Bacillus Stearothermophilus), 결핵균(MTB) 등이 있다.대장균 DnaG는 60킬로달톤(kDa)의 분자량을 가지고 있으며 581개의 아미노산을 함유하고 있습니다.
기능.
DnaG는 템플릿 결합, 뉴클레오사이드 삼인산(NTP) 결합, 개시, 프라이머를 형성하기 위한 확장 및 DNA 중합효소 [1]III로의 프라이머 전달의 다섯 가지 개별 단계에서 올리고뉴클레오티드의 합성을 촉매한다.DnaG는 DNA 복제 중에 DnaB 헬리케이스에 의해 형성된 복제 포크 근처에서 이러한 촉매 작용을 수행합니다.DnaG는 올리고뉴클레오티드 프라이머의 [1]형성을 촉매하기 위해 DnaB와 복합되어야 한다.
프라이머에 의한 프라이머 합성을 위한 메커니즘은 프라이머 단백질(DnaG)[5]의 2개의 NTP 결합 부위를 포함한다.NTP가 RNA 프라이머를 형성하기 위해 결합하기 전에 ssDNA 템플릿 배열은 DnaG에 결합합니다.ssDNA는 Watson-Crick 염기쌍에 [1]기초한 NTP를 모집하는 3개의 뉴클레오티드 인식 시퀀스를 포함한다.DNA 결합 후 DnaG는 효소-DNA-NTP-NTP 4차 복합체를 생성하기 위해 2개의 NTP를 결합해야 합니다.NTP 의 Michaelis 상수(km)는, 프라이머스와 [6]템플릿에 의해서 다릅니다.DnaG의 두 NTP 결합 부위를 시작 부위 및 신장 부위라고 합니다.개시 부위는 프라이머의 5' 말단에 통합될 NTP가 결합하는 부위이다.신장 부위는 프라이머의 3' 끝에 추가된 NTP를 결합합니다.
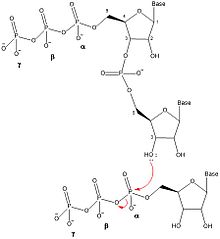
일단 2개의 뉴클레오티드가 프라이머에 결합되면 DnaG는 시작부위 뉴클레오티드의 3' 수산화기와 신장부위 뉴클레오티드의 α-인산 사이에 탈수합성을 통해 포스포디에스테르 결합을 형성함으로써 디뉴클레오티드의 형성을 촉매한다.이 반응은 디뉴클레오티드를 생성하고 α와 β 인 사이의 결합을 파괴하여 피로인산을 방출한다.생성된 피로인산은 무기인산염 [7]효소에 의해 두 개의 무기인산염 분자로 가수분해되기 때문에 이러한 반응은 돌이킬 수 없다.이 디뉴클레오티드 합성 반응은 DNA 또는 RNA(DNA 중합효소, RNA 중합효소)의 형성을 촉매하는 다른 효소와 동일한 반응이므로, DnaG는 항상 5'에서 3' 방향으로 올리고뉴클레오티드를 합성해야 한다.대장균에서 프라이머는 삼인산 아데닌-구아닌(ppp)으로 시작한다.AG) 5' 말단의 디뉴클레오티드.
디뉴클레오티드의 추가 연장이 발생하기 위해서는 3' NTP가 신장부위에서 개시부위로 전달되도록 올리고뉴클레오티드를 이동시켜 다른 NTP가 신장부위에 결합하고 올리고뉴클레오티드의 3' 하이드록시에 부착되도록 해야 한다.프라이머 합성의 신장공정에서 적당한 길이의 올리고뉴클레오티드가 합성되면 DnaG는 새로 합성된 프라이머를 DNA 중합효소 III로 이행시켜 DNA 리딩 스트랜드 또는 오카자키 [1]단편을 합성한다.프라이머 합성의 속도 제한 단계는 NTP 결합 후 디뉴클레오티드 [6]합성 전 또는 도중에 발생한다.
구조.
단백질 분해 연구에 따르면 대장균 DNAG 프리마아제는 세 가지 기능 도메인을 가진 581개의 잔류 단량체 단백질이다.하나의 히스티딘과 세 개의 시스테인 잔기 사이에 아연 이온이 사방정배위되는 N 말단 아연 결합 도메인(잔기 1~110)이 있으며, 이는 배열 특이적 DNA 결합 부위를 인식하는 역할을 한다.중앙 도메인(잔류 111~433)은 RNA 중합효소 활성을 나타내며, RNA 프라이머 합성의 현장이다.C 말단 도메인(잔류 434–581)은 DnaB 헬리케이스 [8]단백질에 대한 DnaG의 비공유 결합을 담당한다.
아연 결합 도메인

배열 특이적 DNA 결합 부위를 인식하는 역할을 하는 도메인인 아연 결합 도메인은 모든 바이러스, 박테리오파지,[9] 원핵생물 및 진핵생물 DNA 프라이머스에 걸쳐 보존됩니다.프리마아제 아연결합 도메인은 아연 리본으로 알려진 아연결합 도메인의 하위 패밀리의 일부이다.아연 리본 도메인은 아연 결합 도메인을 형성하는 2개의 β-헤어핀 루프를 특징으로 한다.일반적으로 아연 리본 도메인은 다른 아연 결합 도메인과 구별되는 알파-헬리시스가 결여된 것으로 생각된다.그러나 2000년 DnaG의 아연결합 도메인은 바실루스 스테아로테르모필러스로부터 결정화되었으며,[9] 도메인은 4개의 α 나선형 옆에 인접한 5개의 가닥이 있는 역평행 β 시트와 도메인의 c-말단 단부에 3개의10 나선형으로 구성되어 있었다.
B. 스테아더모필러스의 아연 결합 부위는 Cys40, Cys61, Cys64의 세 가지 시스테인 잔기와 히스티딘 잔기 His43으로 구성되어 있다.Cys40과 His43은 두 번째와 세 번째 [9]β 시트 사이의 β 헤어핀에 위치한다.Cys61은 5번째 β 시트에 위치하고 Cys64는 4번째와 5번째 β 시트 사이의 β-헤어핀에 위치한다.이들 4개의 잔류물은 아연 이온을 사방정면으로 배위시킨다.아연 이온은 두 번째와 세 번째 β 시트 및 네 번째와 다섯 번째 β 시트 사이의 루프를 안정화시키는 것으로 생각된다.도메인은 β 시트의 소수성 내부 표면 사이의 소수성 상호작용에 의해 더욱 안정화되며, β 시트는 두 번째 및 세 번째 α 나선형에 대해 채워진다.또한 β 시트의 외부 표면에는 보존된 소수성 및 염기성 잔류물이 많다.이러한 잔류물은 Lys30, Arg34, Lys46, Pro48, Lys56, Ile58, His60 및 Phe62입니다.[9]
DNA 결합
아연 결합 도메인의 기능은 배열 특이적 DNA 인식을 위한 것으로 생각된다.DNA 프라이머는 DNA 합성에 사용되는 RNA 프라이머를 만든다.RNA 프라이머의 배치는 무작위적이지 않으며, 특정 DNA 배열에 배치되어 있음을 나타냅니다.실제로, 다른 DNA 프라이머제들은 트리플렛 배열을 인식하는 것으로 나타났다; 스테아더모필러스에 의해 인식된 특정 배열은 [9]아직 확인되지 않았다.아연 이온을 배위하는 시스틴 잔류물이 변이되면 DNA 프리마아제 기능이 정지되는 것으로 나타났다.이것은 아연 결합 도메인이 시퀀스 인식에 역할을 한다는 것을 나타냅니다.또한 β 시트의 소수성 표면과 시트의 한쪽 가장자리에 주로 집적된 염기성 잔류물은 단일 가닥 DNA를 끌어들이는 역할을 하여 DNA [9]결합을 더욱 용이하게 한다.
DNA 프라이머스에 의한 DNA 결합에 대한 이전의 연구에 기초하여, DNA는 β 시트의 표면을 가로질러 아연 결합 도메인에 결합하고, 세 개의 뉴클레오티드가 β [9]시트의 세 가닥에 결합하는 것으로 생각된다.시트의 양전하 잔류물은 인산염과 접촉할 수 있고 방향족 잔류물은 염기와 쌓이는 상호작용을 형성할 수 있습니다.이것은 복제 단백질 A(RPA)[9]의 ssDNA 결합 도메인에 의한 DNA 결합 모델이다.RPA에서 DNA 결합에 중요한 잔류물이 B. stearothermophilus의 구조적으로 동등한 위치에서 발생하기 때문에 B. stearothermophilus의 [9]아연 결합 도메인이 유사한 방식으로 DNA를 결합한다고 가정하는 것이 논리적이다.
RNA중합효소 도메인

이름에서 알 수 있듯이, DnaG의 RNA 중합효소 도메인(RNAP)은 단일 가닥 DNA에서 RNA 프라이머를 합성하는 역할을 합니다.인비보(in-vivo)에서 DnaG는 최대 60개의 뉴클레오티드의 프라이머 조각을 합성할 수 있지만, 인비보 [10]프라이머 조각은 약 11개의 뉴클레오티드로 제한된다.지연된 가닥의 합성 중에 DnaG는 [10]초당 하나의 프라이머 속도로 2000~3000개의 프라이머를 합성한다.
DnaG의 RNAP 도메인은 혼합α 및 β 접힘을 갖는 N말단 도메인, 5개의 가닥이 있는 β 시트와 6개의 α 헬리크로 이루어진 중심 도메인, 그리고 마지막으로 3개의 역평행α 헬리크로 이루어진 나선 다발로 이루어진 C말단 도메인 등 3개의 서브 도메인을 가진다.중심 도메인은 많은 금속 결합 포스포트랜스포트 단백질에서 관찰된 폴드인 토프림 폴드의 일부로 구성됩니다.중심 도메인과 N-말단 도메인은 DnaG에서 RNA 사슬 신장 활성 부위를 구성하는 [10]얕은 틈을 형성한다.균열의 개구부에는 보존성이 높은 염기성 잔류물이 여러 개 있습니다.Arg146, Arg221 및 Lys229.이러한 잔류물은 N 말단 서브도메인의 정전 양성의 리지의 일부입니다.ssDNA와 상호작용하여 중앙 서브도메인에서 토프림 모티브의 금속 결합 중심과 N [10]말단 도메인의 보존된 프라이마아제 모티브로 구성된 구개열로 유도하는 것을 돕는 것이 이 능선이다.토프림 도메인의 금속 결합 부위는 프라이머가 합성되는 곳이다.그런 다음 RNA:DNA 듀플렉스는 또 다른 기본 우울증을 통해 빠져나갑니다.
C 터미널 도메인

아연 결합 도메인 및 RNA 중합효소 도메인과 달리, DNA 프라이머레이스의 C 말단 도메인은 보존되지 않는다.원핵생물 프라이머리에서, 이 도메인의 알려진 유일한 기능은 헬리케이스,[1] DnaB와 상호작용하는 것입니다.따라서 이 도메인을 헬리케이스 결합 도메인(HBD)이라고 한다.DnaG의 HBD는 2개의 서브도메인,[4][11] 즉 나선다발 C1 서브도메인과 나선헤어핀 C2 서브도메인으로 구성됩니다.DnaB 헥사머를 결합하는 2~3개의 DnaG 분자 각각에 대해 HBD의 C1 서브도메인은 헥사머 고리 내면의 N 말단 도메인에서 DnaB와 상호작용하고, C2 서브도메인은 헥사머 외면의 N 말단 도메인과 상호작용한다.

B. 스테아로모필루스 DnaB의 세 가지 잔류물은 DnaB, DnaG 계면 형성에 중요한 것으로 확인되었다.그 잔류물들은 Tyr88, Ile119,[4] Ile125를 포함합니다.Tyr88은 DnaG의 HBD와 가깝지만 접촉하지는 않습니다.Tyr88의 돌연변이는 DnaB의 N 말단 도메인 헬리컬 다발의 형성을 억제하여 DnaG의 [4]HBD와의 접촉을 방해한다.DnaB의 6진수 구조는 사실 이합체 삼량체이다.Ile119 및 Ile125는 모두 DnaB의 N말단 도메인 이합체 계면에 매설되어 있으며, 이들 잔류물의 돌연변이는 육중합체 구조의 형성을 저해하여 [4]DnaG와의 상호작용을 저해한다.DnaB와 DnaG의 상호작용에서 중요한 역할을 하는 것으로 확인된 다른 한 가지 잔류물은 Glu15이다.Glu15의 돌연변이는 DnaB, DnaG 복합체의 형성을 방해하지 않고 대신 DnaG에 [4]의해 합성되는 프라이머의 길이를 조절하는 역할을 한다.
DnaG 억제
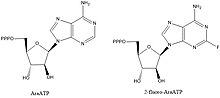
DNA 프라이머제의 억제제는 생화학적 경로와 주요 상호작용의 해명에 귀중한 화합물이지만, 박테리아 질병에 대한 약물을 설계하기 위한 납 화합물로서도 관심이 있다.프라이머 효소를 억제하는 것으로 알려진 대부분의 화합물은 AraATP(비다라빈 참조)와 2-플루오로-아라와 같은 뉴클레오티드 유사체이다.ATP. 이 화합물들은 종종 프리마아제에 의해 기질로 사용되지만, 일단 합성이나 연장은 더 이상 일어날 수 없습니다.예를 들어 대장균 DnaG는 기질로 2', 3'-디데옥시뉴클레오시드 5'-트리포스페이트(ddNTPs)를 사용한다.이들은 3'의 수산기가 부족하여 다음 [1]뉴클레오티드와 포스포디에스테르 결합을 형성하기 위해 사슬 종단자로 작용한다.
상대적으로 적은 수의 프리마아제 억제제는 효소에 대한 잠재적 결합 부위의 부족보다는 프리마아제 측정의 내재적 어려움을 반영한다.합성된 제품의 길이가 짧고 다른 복제 효소에 비해 효소의 속도가 일반적으로 느리기 때문에 HTS(High-Throughput Screening) 접근법을 개발하는 것이 [6]더 어렵습니다.어려움에도 불구하고 NTP 유사체가 아닌 DnaG의 몇 가지 알려진 억제제가 있습니다.독소루비신과 수라민은 모두 결핵균 DnaG의 [12]DNA 및 NTP 경쟁 억제제이다.수라민은 또한 GTP와 경쟁함으로써 진핵생물 DNA 프리마아제를 억제하는 것으로 알려져 있기 때문에 수라민은 유사한 [1]메커니즘을 통해 DNAG를 억제할 가능성이 있다.
외부 링크
- 미국 국립 의학 도서관 의학 주제 제목(MeSH)의 dnaG+단백질,+E+coli
- 미국 국립 의학 도서관(MeSH)의 DnaG+(Primase)
레퍼런스
- ^ a b c d e f g h i Frick DN, Richardson CC (2001). "DNA primase". Annual Review of Biochemistry. 70: 39–80. doi:10.1146/annurev.biochem.70.1.39. PMID 11395402. S2CID 33197061.
- ^ Russell P (2009). iGenetics: A Molecular Approach (3rd ed.). Benjamin Cummings. pp. 42–43. ISBN 978-0321772886.
- ^ Nelson D, Cox M (2008). Lehninger Principles of Biochemistry (5th ed.). New York: W.H. Freeman and Company. pp. 986–989. ISBN 978-0716771081.
- ^ a b c d e f Bailey S, Eliason WK, Steitz TA (19 October 2007). "Structure of Hexameric DnaB Helicase and Its complex with a Domain of DnaG Primase". Science. 318 (5849): 459–63. doi:10.1126/science.1147353. PMID 17947583.
- ^ Frick DN, Kumar S, Richardson CC (10 December 1999). "Interaction of ribonucleoside triphosphates with the gene 4 primase of bacteriphage T7". Journal of Biological Chemistry. 274 (50): 35899–907. doi:10.1074/jbc.274.50.35899. PMID 10585475.
- ^ a b c Kuchta RD, Stengel G (May 2010). "Mechanism and evolution of DNA primases". Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1804 (5): 1180–9. doi:10.1016/j.bbapap.2009.06.011. PMC 2846230. PMID 19540940.
- ^ Bruice PY (2007). Organic Chemistry (5th ed.). Pearson Education, Inc. pp. 1202–1203. ISBN 978-0-13-199631-1.
- ^ Voet, Donald (2010). Biochemistry (4th ed.). New York: J. Wiley & Sons. p. 1189. ISBN 978-0-470-57095-1.
- ^ a b c d e f g h i Pan H, Wigley DB (15 March 2000). "Structure of the zinc-binding domain of Bacillus stearothermophilus DNA primase". Structure. 8 (3): 231–9. doi:10.1016/S0969-2126(00)00101-5. PMID 10745010.
- ^ a b c d Keck JA, Roche DD, Lynch AS, Berger JM (31 March 2000). "Structure of the RNA Polymerase Domain of E. coli Primase". Science. 287 (5462): 2482–6. doi:10.1126/science.287.5462.2482. PMID 10741967. S2CID 27005599.
- ^ Oakley AJ, Loscha KV, Schaeffer PM, Liepinsh E, Pintacuda G, Wilce MCJ, Otting G, Dixon NE (15 January 2005). "Crystal and Solution Structures of the Helicase-binding Domain of Escherichia coli Primase". The Journal of Biological Chemistry. 280 (12): 11495–11504. doi:10.1074/jbc.M412645200. PMID 15649896.
- ^ Biswas T, Resto-Roldan E, Sawyer SK, Artsimovitch I, Tsodikov OV (December 2012). "A novel non-radioactive primase-pyrophosphatase activity assay and its application to the discovery of inhibitors of Mycobacterium tuberculosis primase DnaG". Nucleic Acids Research. 41 (4): e56. doi:10.1093/nar/gks1292. PMC 3575809. PMID 23267008.