DNA중합효소I
DNA polymerase I| DNA중합효소I | |||||||
|---|---|---|---|---|---|---|---|
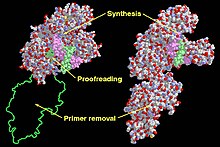 Klenow fragment(왼쪽) 및 DNA 중합효소 I(오른쪽)의 기능 영역. | |||||||
| 식별자 | |||||||
| 유기체 | |||||||
| 기호. | 폴라 | ||||||
| 엔트레즈 | 948356 | ||||||
| PDB | 1DPI | ||||||
| RefSeq(프로트) | NP_418300.1 | ||||||
| 유니프로트 | P00582 | ||||||
| 기타 데이터 | |||||||
| EC 번호 | 2.7.7.7 | ||||||
| 염색체 | 게놈: 4.04~4.05 Mb | ||||||
| |||||||
DNA 중합효소 I(또는 Pol I)는 원핵생물 DNA 복제 과정에 관여하는 효소이다.1956년 [1]Arthur Kornberg에 의해 발견된 그것은 최초의 알려진 DNA 중합효소이다.그것은 처음에 대장균에 특징지어졌고 원핵생물 어디에나 존재한다.대장균과 다른 많은 박테리아에서, Pol I을 코드하는 유전자는 polA로 알려져 있다.대장균 Pol I 효소는 928개의 아미노산으로 구성되어 있으며, 진행 효소의 한 예로서 단일 가닥 템플릿을 [2]방출하지 않고 순차적으로 다중 중합 단계를 촉매할 수 있습니다.Pol I의 생리 기능은 주로 손상된 DNA의 복구를 지원하는 것이지만 RNA 프라이머를 삭제하고 리보뉴클레오티드를 DNA로 치환함으로써 오카자키 단편을 연결하는 데에도 기여한다.
검출
1956년 Arthur Kornberg와 동료들은 대장균(E. coli) 추출물을 사용하여 DNA 합성 분석을 개발함으로써 Pol I을 발견했다.과학자들은 RNA가 아닌 DNA의 방사성 고분자가 추출될 수 있도록 C 라벨 티미딘을 첨가했다.DNA 중합효소의 정화를 시작하기 위해, 연구원들은 대장균 추출물에 황산 스트렙토마이신을 첨가했다.이를 통해 추출물이 핵산 무첨가물(S-fraction)과 핵산 함유 침전물(P-fraction)로 분리되었다.P-분할은 또한 DNA 합성 반응에 필수적인 Pol I과 열안정 인자를 포함했다.이러한 인자는 핵산의 구성 요소인 뉴클레오시드 트리포스페이트로 확인되었다.S-fraction에는 다중 디옥시뉴클레오시드 키나아제([3]kinase)1959년, 노벨 생리의학상은 "리보핵산과 [4]디옥시리보핵산의 생물학적 합성에 관련된 메커니즘을 발견한" Arthur Kornberg와 Severo Ochoa에게 수여되었다.
구조 및 기능
일반구조
폴리 I은 손상된 DNA를 복구하는 일을 주로 한다.구조적으로, Pol I은 알파/베타 단백질 슈퍼패밀리의 일원으로, α-헬리체와 β-스트랜드가 불규칙한 배열로 발생하는 단백질을 포함한다.대장균 DNA Pol I은 세 가지 다른 효소 활성을 가진 다중 도메인으로 구성됩니다.종종 엄지, 손가락, 손바닥 도메인으로 불리는 세 개의 도메인이 DNA 중합효소 [5]활동을 유지하기 위해 함께 작동합니다.팜 도메인 옆의 네 번째 도메인은 교정이라고 알려진 프로세스에서 잘못 삽입된 뉴클레오티드를 3~5' 방향으로 제거하는 핵산가수분해효소 활성 부위를 포함한다.5번 도메인은 5'~3' 방향으로 DNA 또는 RNA를 제거하는 또 다른 엑소핵산가수분해효소 활성부위를 포함하고 있으며, DNA 복제 중 RNA 프라이머 제거 또는 DNA 복구 과정 중 DNA 제거에 필수적이다.
대장균은 5가지 다른 DNA 중합효소를 생성한다: DNA Pol I, DNA Pol II, DNA Pol III, DNA Pol IV, 그리고 DNA Pol V.진핵세포는 α, β, β, β, β, γ, [6]β의 5가지 다른 DNA 중합효소를 포함하고 있다.진핵 DNA 중합효소 β는 복제보다는 DNA 수복과 관련이 있기 때문에 대장균 DNA Pol I과 가장 유사하다.DNA중합효소β는 염기절제수복 [7]및 뉴클레오티드절제수복에 주로 사용된다.총 15개의 인간 DNA 중합효소가 확인되었다.[8]
다른 중합효소와의 구조 및 기능적 유사성
DNA 복제에서는 선두 DNA 가닥이 복제 포크 이동 방향으로 연속적으로 늘어나는 반면, DNA 지연 가닥은 오카자키 [9]단편으로서 반대 방향으로 불연속적으로 주행한다.DNA 중합효소는 또한 DNA 사슬을 시작할 수 없기 때문에 프라이머로 [5]알려진 짧은 RNA 또는 DNA 세그먼트에 의해 시작되어야 한다.DNA 중합이 이루어지기 위해서는 두 가지 요건이 충족되어야 한다.우선, 모든 DNA 중합효소는 템플릿 스트랜드와 프라이머 스트랜드를 모두 가져야 한다.RNA와 달리, DNA 중합효소는 템플릿 가닥에서 DNA를 합성할 수 없습니다.합성은 5' ~ 3' 방향으로 프리마아제에 의해 합성된 RNA 프라이머로 알려진 짧은 RNA 세그먼트에 의해 시작되어야 한다.DNA 합성은 기존 DNA 가닥 또는 RNA 프라이머의 말단에서 3' 히드록실기에 dNTP를 첨가함으로써 일어난다.둘째, DNA 중합효소는 수소 [6]결합을 통해서만 기존 가닥에 새로운 뉴클레오티드를 첨가할 수 있다.모든 DNA 중합효소는 유사한 구조를 가지고 있기 때문에, 모두 2-금속 이온 촉매 중합효소 메커니즘을 공유합니다.금속 이온 중 하나는 프라이머 3' 하이드록실기를 활성화하고, 이 프라이머 3' 하이드록실기는 dNTP의 1차 5'인산염기를 활성화한다.두 번째 금속 이온은 남은 산소의 음전하를 안정화시키고, 이어서 두 개의 기존 인산염기를 [10]킬레이트합니다.
DNA 중합효소의 중합효소 도메인의 X선 결정 구조는 인간의 오른손과 유사하게 설명됩니다.모든 DNA 중합효소는 3개의 도메인을 포함한다.첫 번째 도메인은 "핑거 도메인"으로 알려져 있으며 dNTP 및 쌍으로 구성된 템플릿 기반과 상호 작용합니다.또한 "핑거 도메인"은 템플릿과 상호 작용하여 활성 [11]사이트에 올바르게 배치합니다.팜 도메인으로 알려진 두 번째 도메인은 포스포릴 그룹의 전달 반응을 촉매한다.마지막으로, "엄지 도메인"으로 알려진 세 번째 도메인은 이중 가닥 [12]DNA와 상호작용합니다.엑소핵산가수분해효소 도메인은 자체 촉매 사이트를 포함하고 잘못 짝을 이룬 염기를 제거한다.7개의 다른 DNA 중합효소 패밀리 중 "팜 도메인"은 이들 패밀리 중 5개에서 보존됩니다."[11]finger domain"과 "thumb domain"은 서로 다른 시퀀스의 2차 구조 요소가 다르기 때문에 각 패밀리에서 일관성이 없습니다.
기능.
폴리 I은 네 가지 효소 활성을 가지고 있다.
- 3' 프라이머 부위와 템플릿 스트랜드가 필요한 5'→3' DNA 의존성 DNA 중합효소 활성
- 교정을 매개하는 3'→5'(166)핵산가수분해효소 활성
- DNA 수복 중 닉 변환을 매개하는 5'→3'(전방) 핵산가수분해효소 활성.
- A 5'→3' (전방) RNA 의존성 DNA 중합효소 활성.Pol I은 DNA 템플릿보다 상당히 낮은 효율성(0.1~0.4%)을 가진 RNA 템플릿에서 작동하며, 이 활성은 아마도 생물학적 의미가 [13]제한적일 것이다.
Pol I이 주로 DNA 복제에 사용되는지 또는 DNA 손상 복구에 사용되는지를 결정하기 위해, 결손된 대장균의 Pol I 돌연변이 변종으로 실험을 수행했다.폴리 I이 결핍된 돌연변이 균주는 격리되었고 돌연변이 물질로 치료되었다.돌연변이 변종은 정상적으로 계속 성장하며 또한 Pol I이 결여된 박테리아 군락을 발달시켰다.이를 통해 Pol I이 DNA 복제에 필요하지 않음을 확인했습니다.그러나 돌연변이 변종은 또한 자외선과 같이 DNA를 손상시키는 특정 요인에 극도로 민감하게 반응하는 특징을 보였다.따라서, 이것은 폴 1이 DNA [6]복제보다는 DNA 손상을 복구하는 데 더 많이 관여할 가능성이 있다는 것을 재확인했다.
메커니즘
복제 공정에서 RNAase H는 후행하는 가닥에서 RNA 프라이머(프리마아제 생성)를 제거하고, 그 후 중합효소 I은 오카자키 조각 사이에 필요한 뉴클레오티드를 5'→3' 방향으로 채우고, 오차를 교정한다.템플릿 의존형 효소입니다. 템플릿 역할을 하는 기존 DNA 가닥과 정확하게 염기쌍을 이루는 뉴클레오티드를 첨가할 뿐입니다.DNA 연결효소가 다양한 조각들을 연속적인 DNA 가닥으로 결합할 수 있도록 이러한 뉴클레오티드가 DNA 템플릿 가닥과 염기쌍을 이루는 적절한 방향과 기하학적 구조를 갖는 것이 중요하다.중합효소 I에 대한 연구는 다른 dNTP가 중합효소 I의 동일한 활성 부위에 결합할 수 있다는 것을 확인했다.중합효소 I은 구조변화를 거친 후에야 다른 dNTP를 능동적으로 구별할 수 있다.이 변경이 발생하면 Pol I은 바인딩된 dNTP와 템플릿 스트랜드 상의 일치하는 베이스 사이에 형성된 베이스 쌍의 적절한 지오메트리와 정렬을 확인합니다.활성 부위에 들어갈 수 있는 것은 A=T 및 G-C 베이스 쌍의 올바른 형상뿐입니다.하지만, 10에서4 10개의5 뉴클레오티드 중 1개가 잘못 첨가된다는 것을 아는 것이 중요하다.그럼에도 불구하고, Pol I은 능동 [5]판별이라는 선택적 방법을 사용하여 DNA 복제에서 이 오류를 고칠 수 있다.
초기 특성화에도 불구하고 중합효소 I이 대부분의 DNA 합성에 관여하는 효소가 아니라는 것이 금방 분명해졌다.대장균의 DNA 복제는 약 1,000 뉴클레오티드/초로 진행되며, 중합효소 I에 의한 염기쌍 합성 속도는 평균 10~20 뉴클레오티드/초에 불과하다.또한 세포당 약 400분자의 세포 풍부성은 일반적으로 대장균에 복제 포크가 두 개뿐이라는 사실과 상관관계가 없었다.또한 25~50개의 뉴클레오티드를 함유한 후 떨어지기 때문에 전체 게놈을 복사하기에는 처리능력이 불충분하다.복제에서의 그것의 역할은 1969년 존 케언스가 중합효소 [14]활성이 결여된 생존 가능한 중합효소 I 돌연변이를 분리했을 때 증명되었다.케언스의 연구실 조수인 폴라 드 루시아는 대장균 군락에서 수천 개의 무세포 추출물을 만들어 DNA 중합효소 활성도를 측정했다.3,478번째 복제품에는 폴라 돌연변이가 들어있었고 케언스는 폴라([15]Paula)의 이름을 따서 명명했다.DNA 중합효소 III의 발견이 있은 후에야 주요 복제 DNA 중합효소가 마침내 확인되었다.
조사 어플리케이션

대장균으로부터 얻은 DNA중합효소 I은 분자생물학 연구에 광범위하게 이용된다.그러나 5'→3' 핵산가수분해효소 활성은 많은 용도에 적합하지 않다.이 바람직하지 않은 효소 활동은 분자생물학에서 널리 사용되는 클레노프 조각이라고 불리는 유용한 분자를 남기기 위해 홀로엔자임에서 간단히 제거될 수 있습니다.실제로 클레노프 조각은 1976년 [17]내열성 Taq 중합효소 I의 원천인 테르무스 아쿠아티쿠스가 발견될 때까지 중합효소 연쇄반응(PCR) 증폭의 첫 프로토콜에 사용되었다.단백질분해효소 서브틸리신에 대한 DNA 중합효소 I의 노출은 분자를 더 작은 조각으로 쪼개 DNA 중합효소 및 교정 활동만 유지한다.
「 」를 참조해 주세요.
레퍼런스
- ^ Lehman IR, Bessman MJ, Simms ES, Kornberg A (July 1958). "Enzymatic synthesis of deoxyribonucleic acid. I. Preparation of substrates and partial purification of an enzyme from Escherichia coli". The Journal of Biological Chemistry. 233 (1): 163–70. doi:10.1016/S0021-9258(19)68048-8. PMID 13563462.
- ^ Voet D, Voet JG, Pratt CW (1999). Fundamentals of Biochemistry. New York: Wiley.[페이지 필요]
- ^ Lehman IR (September 2003). "Discovery of DNA polymerase". The Journal of Biological Chemistry. 278 (37): 34733–8. doi:10.1074/jbc.X300002200. PMID 12791679.
- ^ "The Nobel Prize in Physiology or Medicine 1959". www.nobelprize.org. Retrieved 2016-11-08.
- ^ a b c Cox MM, Doudna J (2015). Molecular Biology (2nd ed.). New York: W.H. Freeman.[페이지 필요]
- ^ a b c Cooper, Geoffrey M. Geoffrey (2000-01-01). "DNA Replication".
{{cite journal}}:Cite 저널 요구 사항journal=(도움말) - ^ Wood RD, Shivji MK (April 1997). "Which DNA polymerases are used for DNA-repair in eukaryotes?". Carcinogenesis. 18 (4): 605–10. doi:10.1093/carcin/18.4.605. PMID 9111189.
- ^ Biertümpfel C, Zhao Y, Kondo Y, Ramón-Maiques S, Gregory M, Lee JY, Masutani C, Lehmann AR, Hanaoka F, Yang W (June 2010). "Structure and mechanism of human DNA polymerase eta". Nature. 465 (7301): 1044–8. Bibcode:2010Natur.465.1044B. doi:10.1038/nature09196. PMC 2899710. PMID 20577208.
- ^ Hübscher U, Spadari S, Villani G, Maga G (2010). DNA Polymerases. doi:10.1142/7667. ISBN 978-981-4299-16-9.[페이지 필요]
- ^ "DNA Polymerase I: Enzymatic Reactions".
- ^ a b "MBIO.4.14.5". bioscience.jbpub.com. Retrieved 2017-05-14.
- ^ Loeb LA, Monnat RJ (August 2008). "DNA polymerases and human disease". Nature Reviews Genetics. 9 (8): 594–604. doi:10.1038/nrg2345. PMID 18626473. S2CID 3344014.
- ^ Ricchetti M, Buc H (February 1993). "E. coli DNA polymerase I as a reverse transcriptase". The EMBO Journal. 12 (2): 387–96. doi:10.1002/j.1460-2075.1993.tb05670.x. PMC 413221. PMID 7679988.
- ^ De Lucia P, Cairns J (December 1969). "Isolation of an E. coli strain with a mutation affecting DNA polymerase". Nature. 224 (5225): 1164–6. Bibcode:1969Natur.224.1164D. doi:10.1038/2241164a0. PMID 4902142. S2CID 4182917.
- ^ Friedberg EC (February 2006). "The eureka enzyme: the discovery of DNA polymerase". Nature Reviews Molecular Cell Biology. 7 (2): 143–7. doi:10.1038/nrm1787. PMID 16493419. S2CID 39605644.
- ^ EMBL-EBI. "EMBL European Bioinformatics Institute". www.ebi.ac.uk. Retrieved 2016-11-08.
- ^ van Pelt-Verkuil E, van Belkum A, Hays JP (2008). "Taq and Other Thermostable DNA Polymerases". Principles and Technical Aspects of PCR Amplification. pp. 103–18. doi:10.1007/978-1-4020-6241-4_7. ISBN 978-1-4020-6240-7.



