생체 분자 구조
Biomolecular structure이 기사는 독자들에게 혼란스럽거나 불분명할 수 있다.(2016년 2월 (이 및 ) |
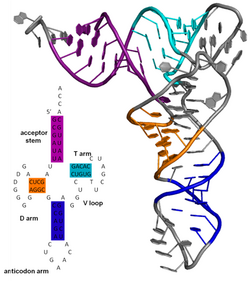
생체 분자 구조는 단백질, DNA, 또는 RNA 분자에 의해 형성되는 복잡한 접힌 3차원 형태이며, 그것은 그 기능에 중요하다.이러한 분자의 구조는 개별 원자의 수준에서 전체 단백질 소단위 사이의 관계에 이르는 여러 길이 척도 중 하나로 고려될 수 있다.척도의 이러한 유용한 차이는 종종 분자 구조를 4단계로 분해하는 것으로 표현된다: 1차, 2차, 3차, 4차.분자의 이 다중 스케일 구성을 위한 골격은 2차 수준에서 발생하는데, 여기서 기본적인 구조적 요소는 분자의 다양한 수소 결합이다.이는 단백질에 대한 알파 헬리크스 및 베타 시트, 핵산에 대한 헤어핀 루프, 팽대부 및 내부 루프와 같은 2차 구조 특징을 포함하여 단백질 구조와 핵산 구조의 몇 가지 인지 가능한 도메인으로 이어진다.1차, 2차, 3차 및 4차 구조라는 용어는 Kaj Ulrik Linderström-Lang이 1951년 스탠포드 대학의 Lane Medical 강의에서 소개한 것입니다.
일차 구조
생체 고분자의 1차 구조는 원자 조성과 이들 원자를 연결하는 화학적 결합의 정확한 사양이다.전형적인 비분기, 비교차 생체고분자(예를 들어 전형적인 세포내 단백질의 분자, 또는 DNA 또는 RNA)의 경우, 1차 구조는 아미노산 또는 뉴클레오티드와 같은 단량체 서브유닛의 배열을 지정하는 것과 같다.
단백질의 1차 구조는 아미노 N 말단에서 카르복실 C 말단으로 보고되며, DNA 또는 RNA 분자의 1차 구조는 5' 말단에서 3' 말단으로 보고되는 핵산 배열로 알려져 있다.핵산 배열은 전체 분자를 구성하는 뉴클레오티드의 정확한 배열이다.종종 일차 구조는 기능적으로 중요한 시퀀스 모티브를 인코딩합니다.그러한 모티브의 예로는 snoRNA의 C/D[1] 및 H/ACA 박스[2], U1, U2, U4, U5, U6, U12 및 U3, 샤인-달가르노 배열,[3] Kozak III 중합체 배열 및 합의[4] RNA가 있다.
이차 구조
단백질의 2차 구조는 생체 고분자 내 수소 결합 패턴이다.이것들은 생체 고분자의 국소적인 부분의 일반적인 3차원 형태를 결정하지만, 3차 구조로 간주되는 3차원 공간에서의 특정 원자 위치의 전역 구조를 설명하지는 않는다.2차 구조는 원자 분해능 구조에서 관찰된 바와 같이 생체 고분자의 수소 결합에 의해 정식으로 정의된다.단백질에서 2차 구조는 백본 아민과 카르복실기 사이의 수소 결합 패턴에 의해 정의되며(사이드체인-메인쇄 및 사이드체인-사이드체인 수소 결합은 무관하다). 여기서 수소 결합의 DSSP 정의가 사용된다.
핵산의 2차 구조는 질소 염기 사이의 수소 결합에 의해 정의된다.
그러나 단백질의 경우, 수소 결합은 다른 구조적 특징과 관련이 있으며, 이는 2차 구조의 덜 형식적인 정의를 낳았다.예를 들어, 나선형은 라마찬드란 플롯의 일부 영역에서 골격 이면각을 채택할 수 있다. 따라서 이러한 이면각을 가진 잔류물의 세그먼트는 올바른 수소 결합을 가지고 있는지 여부에 관계없이 종종 나선이라고 불린다.곡률 및 비틀림과 같은 곡선의 미분 기하학에서 개념을 적용하는 형식적이지 않은 정의들이 많이 제안되었다.새로운 원자 분해능 구조를 해결하는 구조 생물학자들은 때로 눈으로 2차 구조를 할당하고 해당 단백질 데이터 뱅크(PDB) 파일에 그들의 할당을 기록한다.
핵산 분자의 2차 구조는 하나의 분자 또는 일련의 상호작용하는 분자 내에서 염기쌍의 상호작용을 말한다.생물학적 RNA의 2차 구조는 종종 줄기와 고리로 독특하게 분해될 수 있다.종종 이러한 요소나 요소들의 조합은 테트라루프, 의사 노트 및 스템 루프와 같이 더욱 분류될 수 있다.생물학적 RNA에 기능적으로 중요한 많은 2차 구조 요소가 있습니다. 유명한 예로는 Rho 독립형 터미네이터 스템 루프와 전이 RNA(tRNA) 클로버 잎이 있습니다.RNA 분자의 2차 구조를 알아내려는 연구자들의 소규모 산업이 있다.접근법에는 실험 및 계산 방법이 모두 포함됩니다(RNA 구조 예측 소프트웨어 목록 참조).
3차 구조
단백질이나 다른 고분자의 3차 구조는 원자 [6]좌표에 의해 정의된 3차원 구조이다.단백질과 핵산은 복잡한 3차원 구조로 접혀져 분자의 기능을 일으킨다.그러한 구조는 다양하고 복잡하지만, 종종 반복적이고 인식할 수 있는 3차 구조 모티브와 분자 구성 요소 역할을 하는 도메인으로 구성됩니다.3차 구조는 주로 생체 분자의 1차 구조(아미노산 또는 뉴클레오티드의 배열)에 의해 결정되는 것으로 간주된다.
4차 구조
단백질 4차 구조는 다중 서브유닛 복합체 내 여러 단백질 분자의 수와 배치를 말한다.
핵산의 경우, 이 용어는 덜 흔하지만, 히스톤과의 상호작용을 포함한 염색질에서 [7]DNA의 높은 수준의 조직 또는 리보솜[8][9] 또는 스플라이세오솜에서 분리된 RNA 단위 사이의 상호작용을 나타낼 수 있다.
구조 결정
구조 탐침은 생체 분자 [10]구조를 결정하기 위해 생화학 기술을 사용하는 과정이다.이 분석은 분자 구조를 추론하기 위해 사용될 수 있는 패턴, 분자 구조와 기능의 실험 분석, 그리고 추가적인 생물학적 [11]연구를 위한 더 작은 분자의 개발에 대한 이해를 정의하는데 사용될 수 있습니다.구조 프로브 분석은 화학 프로브, 수산기 래디칼 프로브, 뉴클레오티드 아날로그 간섭 매핑(NAIM) 및 인라인 [10]프로브 등 다양한 방법을 통해 수행될 수 있습니다.
단백질 및 핵산 구조는 핵자기공명분광학(NMR) 또는 X선 결정학 또는 단립자 저온전자현미경학(cryoEM) 중 하나를 사용하여 결정할 수 있다.A-DNA X선 회절 패턴의 DNA에 대한 첫 번째 보고서(1953년 로잘린드 프랭클린과 레이먼드 고슬링에 의해)와 B-DNA는 [12][13]종아리 흉선에서 분리된 배향 DNA 섬유에 대해 제한된 양의 구조 정보만 제공하는 패터슨 함수 변환에 기초한 분석을 사용했다.그런 다음 1953년 윌킨스 외 연구진이 B-DNA X선 회절 및 B-DNA 박테리아 지향 DNA 섬유와 송어 정자 헤드의 산란 패턴에 대한 대체 분석을 제안했다.[14]B-DNA 형태'는 [15]세포에서 발견되는 조건 하에서 가장 흔하지만, 그것은 잘 정의된 배치가 아니라 다양한 살아있는 [16]세포에서 존재하는 높은 수화 수준에서 발생하는 DNA 배합의 패밀리 또는 퍼지 세트이다. 대응하는 X선 회절 및 산란 패턴은 현저한 무질서(20%[17][18] 이상)를 가진 분자 파라크리스탈의 특징이며, 표준 분석만으로는 구조를 추적할 수 없다.
이와는 대조적으로, 베셀[19] 함수와 DNA 분자 모델의 푸리에 변환만을 포함하는 표준 분석은 여전히 A-DNA 및 Z-DNA X선 [20]회절 패턴을 분석하는 데 일상적으로 사용된다.
구조 예측

생체 분자 구조 예측은 아미노산 배열로부터 단백질의 3차원 구조를 예측하거나 핵산(염기) 배열로부터 핵산을 예측하는 것이다.즉, 1차 구조로부터의 2차 및 3차 구조의 예측이다.구조 예측은 합리적인 설계, 단백질 설계, 핵산 설계 및 생체 분자 공학에서와 같이 생체 분자 설계의 반대입니다.
단백질 구조 예측은 생물정보학 및 이론화학이 추구하는 가장 중요한 목표 중 하나이다.단백질 구조 예측은 의학(예: 의약품 설계)과 생명공학(예: 새로운 효소 설계)에서 매우 중요하다.2년마다 단백질 구조 예측의 임계 평가(CASP) 실험에서 현재 방법의 성과를 평가합니다.
RNA 구조 예측 문제에 대한 상당한 양의 생체 정보학 연구도 있었다.RNA로 작업하는 연구자들에게 공통적인 문제는 핵산 배열만 주어진 분자의 3차원 구조를 결정하는 것이다.그러나 RNA의 경우 최종 구조의 대부분은 분자의 2차 구조 또는 분자 내 염기쌍화 상호작용에 의해 결정된다.이것은 다양한 종에 걸친 염기쌍의 높은 보존성으로 나타난다.
작은 핵산 분자의 2차 구조는 주로 수소 결합과 염기 적층 같은 강한 국소적 상호작용에 의해 결정된다.그러한 상호작용에 대한 자유 에너지의 합계는 보통 가장 가까운 이웃 방법을 사용하여 주어진 [21]구조의 안정성에 대한 근사치를 제공한다.가장 낮은 자유 에너지 구조를 찾는 가장 간단한 방법은 가능한 모든 구조를 생성하고 그것들을 위한 자유 에너지를 계산하는 것이지만, 일련의 가능한 구조의 수는 [22]분자의 길이에 따라 기하급수적으로 증가한다.더 긴 분자의 경우, 가능한 2차 구조의 수는 [21]방대합니다.
배열 공변화 방법은 관련이 있지만 유사하지 않은 배열과 함께 여러 개의 상동 RNA 배열로 구성된 데이터 세트의 존재에 의존합니다.이러한 방법들은 진화의 개별 염기 부위의 공변화를 분석한다. 염기쌍을 이루는 뉴클레오티드 쌍의 넓게 분리된 두 부위의 유지는 이들 위치 사이에 구조적으로 필요한 수소 결합의 존재를 나타낸다.pseudoknot 예측의 일반적인 문제는 [23]NP-complete인 것으로 나타났습니다.
설계.
생체 분자 설계는 구조 예측의 역순으로 간주될 수 있다.구조예측에서 구조는 기존의 배열로부터 결정되며, 단백질 또는 핵산 설계에서는 원하는 구조를 형성하는 배열이 생성된다.
기타 생체 분자
다당류, 폴리페놀 및 지질과 같은 다른 생체 분자들도 생물학적 결과의 고차 구조를 가질 수 있다.
「 」를 참조해 주세요.
메모들
레퍼런스
- ^ Samarsky DA, Fournier MJ, Singer RH, Bertrand E (July 1998). "The snoRNA box C/D motif directs nucleolar targeting and also couples snoRNA synthesis and localization". The EMBO Journal. 17 (13): 3747–57. doi:10.1093/emboj/17.13.3747. PMC 1170710. PMID 9649444.
- ^ Ganot P, Caizergues-Ferrer M, Kiss T (April 1997). "The family of box ACA small nucleolar RNAs is defined by an evolutionarily conserved secondary structure and ubiquitous sequence elements essential for RNA accumulation". Genes & Development. 11 (7): 941–56. doi:10.1101/gad.11.7.941. PMID 9106664.
- ^ Shine J, Dalgarno L (March 1975). "Determinant of cistron specificity in bacterial ribosomes". Nature. 254 (5495): 34–38. Bibcode:1975Natur.254...34S. doi:10.1038/254034a0. PMID 803646. S2CID 4162567.
- ^ Kozak M (October 1987). "An analysis of 5'-noncoding sequences from 699 vertebrate messenger RNAs". Nucleic Acids Research. 15 (20): 8125–48. doi:10.1093/nar/15.20.8125. PMC 306349. PMID 3313277.
- ^ Bogenhagen DF, Brown DD (April 1981). "Nucleotide sequences in Xenopus 5S DNA required for transcription termination". Cell. 24 (1): 261–70. doi:10.1016/0092-8674(81)90522-5. PMID 6263489. S2CID 9982829.
- ^ IUPAC, 화학 용어집, 제2판('골드북') (1997).온라인 수정판: (2006–) "teriary structure".doi:10.1351/goldbook.T06282
- ^ Sipski ML, Wagner TE (March 1977). "Probing DNA quaternary ordering with circular dichroism spectroscopy: studies of equine sperm chromosomal fibers". Biopolymers. 16 (3): 573–82. doi:10.1002/bip.1977.360160308. PMID 843604. S2CID 35930758.
- ^ Noller HF (1984). "Structure of ribosomal RNA". Annual Review of Biochemistry. 53: 119–62. doi:10.1146/annurev.bi.53.070184.001003. PMID 6206780.
- ^ Nissen P, Ippolito JA, Ban N, Moore PB, Steitz TA (April 2001). "RNA tertiary interactions in the large ribosomal subunit: the A-minor motif". Proceedings of the National Academy of Sciences of the United States of America. 98 (9): 4899–903. Bibcode:2001PNAS...98.4899N. doi:10.1073/pnas.081082398. PMC 33135. PMID 11296253.
- ^ a b Teunissen, A. W. M. (1979). RNA Structure Probing: Biochemical structure analysis of autoimmune-related RNA molecules. pp. 1–27. ISBN 978-90-901323-4-1.
- ^ Pace NR, Thomas BC, Woese CR (1999). Probing RNA Structure, Function, and History by Comparative Analysis. Cold Spring Harbor Laboratory Press. pp. 113–17. ISBN 978-0-87969-589-7.
- ^ Franklin RE, Gosling RG (6 March 1953). "The Structure of Sodium Thymonucleate Fibres (I. The Influence of Water Content, and II. The Cylindrically Symmetrical Patterson Function)" (PDF). Acta Crystallogr. 6 (8): 673–78. doi:10.1107/s0365110x53001939.
- ^ Franklin RE, Gosling RG (April 1953). "Molecular configuration in sodium thymonucleate". Nature. 171 (4356): 740–41. Bibcode:1953Natur.171..740F. doi:10.1038/171740a0. PMID 13054694. S2CID 4268222.
- ^ Wilkins MH, Stokes AR, Wilson HR (April 1953). "Molecular structure of deoxypentose nucleic acids". Nature. 171 (4356): 738–40. Bibcode:1953Natur.171..738W. doi:10.1038/171738a0. PMID 13054693. S2CID 4280080.
- ^ Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL (October 1980). "Polymorphism of DNA double helices". Journal of Molecular Biology. 143 (1): 49–72. doi:10.1016/0022-2836(80)90124-2. PMID 7441761.
- ^ Baianu, I. C. (1980). "Structural Order and Partial Disorder in Biological systems". Bull. Math. Biol. 42 (1): 137–41. doi:10.1007/BF02462372. S2CID 189888972.
- ^ Hosemann R, Bagchi RN (1962). Direct analysis of diffraction by matter. Amsterdam/New York: North-Holland.
- ^ Baianu IC (1978). "X-ray scattering by partially disordered membrane systems". Acta Crystallogr. A. 34 (5): 751–53. Bibcode:1978AcCrA..34..751B. doi:10.1107/s0567739478001540.
- ^ "Bessel functions and diffraction by helical structures". planetphysics.org.[영구 데드링크]
- ^ "X-Ray Diffraction Patterns of Double-Helical Deoxyribonucleic Acid (DNA) Crystals". planetphysics.org. Archived from the original on 24 July 2009.
- ^ a b Mathews DH (June 2006). "Revolutions in RNA secondary structure prediction". Journal of Molecular Biology. 359 (3): 526–32. doi:10.1016/j.jmb.2006.01.067. PMID 16500677.
- ^ Zuker M, Sankoff D (1984). "RNA secondary structures and their prediction". Bull. Math. Biol. 46 (4): 591–621. doi:10.1007/BF02459506. S2CID 189885784.
- ^ Lyngsø RB, Pedersen CN (2000). "RNA pseudoknot prediction in energy-based models". Journal of Computational Biology. 7 (3–4): 409–27. CiteSeerX 10.1.1.34.4044. doi:10.1089/106652700750050862. PMID 11108471.