POLD1
POLD1| POLD1 | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||||||||||||||||||
| 별칭 | POLD1, CDC2, CRCS10, MDPL, POLD, 폴리머아제(DNA) 델타 1, 촉매 서브 유닛, DNA 폴리머아제 델타 1, 촉매 서브 유닛 | ||||||||||||||||||||||||
| 외부 ID | OMIM: 174761 MGI: 97741 호몰로진: 2014 GeneCard: POLD1 | ||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| 직교체 | |||||||||||||||||||||||||
| 종 | 인간 | 마우스 | |||||||||||||||||||||||
| 엔트레스 | |||||||||||||||||||||||||
| 앙상블 | |||||||||||||||||||||||||
| 유니프로트 | |||||||||||||||||||||||||
| RefSeq(mRNA) | |||||||||||||||||||||||||
| RefSeq(단백질) | |||||||||||||||||||||||||
| 위치(UCSC) | Cr 19: 50.38 – 50.42Mb | Cr 7: 44.18 – 44.2Mb | |||||||||||||||||||||||
| PubMed 검색 | [3] | [4] | |||||||||||||||||||||||
| 위키다타 | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
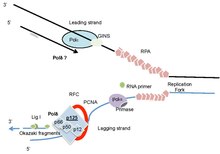
유전자 중합효소 델타 1(POLD1)은 DNA 중합효소 델타(PolΔ) 복합체의 촉매 소단위인 큰 POLD1/p125를 인코딩한다.[5][6]PolΔ 효소는 DNA의 후행 가닥을 합성하는 역할을 하며, 선행 가닥의 일부 활동에도 관여했다(그림 1).POLD1/p125 서브유닛은 DNA 중합 및 엑소누클레이저 영역을 모두 인코딩하며, 이는 DNA 합성 중 복제 정확성을 보장하기 위한 교정 및 DNA 손상 후 여러 종류의 복제 관련 DNA 수리에 있어 단백질을 중요한 제2의 기능을 제공한다.
POLD1의 활동을 손상시키는 세균선 돌연변이는 몇몇 산발적인 암의 유전성 암과 조산성 노화의 발달 증후군인 만디블라시아, 청각장애, 프로게로이드 특징과 Lipodystrophysis(MDPL/DP 증후군)에 관련되어 있다.POLD1의 연구는 종양기세증을 제한하기 위해 유전학적 안정성을 유지하는 것의 중요성을 강조한다.현재 POLD1 결함과 연관된 강화된 종양이 기저 대체물의 증가의 결과인지, 포크가 붕괴되고 DNA 이중 가닥 절단(DSB)의 생산에 의한 것인지는 불분명하다.[6][7]최근의 리뷰는 POLD1과 PolΔ의 중요한 기능을 다루었다.[6][7]
디스커버리
최초의 DNA 중합효소인 DNA 중합효소 I는 1956년 아서 코른베르크와 그의 동료들에 의해 발견되어 검토되었다.[8][9]1976년, Byrnes 외 연구진은 포유류 세포에서 중합효소 델타(Δ)라고 불리는 세 번째 DNA 중합효소 활동을 발견했다.[10]토끼 홍반성 과대증소성 골수에서 정제되어 내인성 3'에서 5'의 엑소뉴클레아제 활성을 가진 DNA 중합효소로 묘사되었다.DNA 중합체(대장균)에 대한 3'-5'의 엑소누클레스 교정 기능은 4년 전에 코른베르크와 브루틀랙에 의해 처음으로 설명되었다.[11][12]인간의 DNA PolΔ는 이성애자 입니다.4개의 하위 유니트는 (POLD1/p125), (POLD3/p66), (POLD2/p50) 및 (POLD4/p12)이며, 대체 이름은 킬로달톤(kDa)으로 표현된 분자량을 반영한다.폴리머아제 촉매 서브유닛은 1991년 활동 얼룩에 의해 125 kDa 폴리펩타이드로 식별되었다.[13]여러 그룹이 인간과 머린 POLD1 cDNA를 독립적으로 복제했다.[5][14][15]종아리 흉선, 인간 태반, 그리고 헤라 세포를 포함한 다양한 원천으로부터 정화되면서,[16][17][18][19][20] 그것의 활동은 DNA 수리에 관여하게 되었다.[21][22]
유전자
Polymerase (DNA) delta 1, catalytic subunit and POLD1 are the name and gene symbol approved by the Human Genome Organization (HUGO) Gene Nomenclature Committee (HGNC).[23] POLD1 is also known as CDC2, MDPL, POLD, and CRCS10), is ~34 kb long and its cytogenetic location is chromosome 19[24] q13.33.[25]GRCh38.2 조립체에서 정확한 위치는 염기쌍 50,384,290부터 염기쌍 50,418,018까지 19번 염색체에 있다.[26]생쥐 맞춤법은 생쥐 7번 염색체에 매핑된다.[27]인간의 경우 주요 POLD1 대본(NM_002691.3)은 27개의 엑손(exon)을 포함하고 있으며 p125 또는 A 서브유닛의 1107개의 아미노산으로 번역된다.아미노산 592(NP_001295561.1) 이후 26개의 아미노산 인프레임 삽입으로 더 긴 ISO 형식이 보고되었다.6번 염색체의 긴 팔에서 유사 유전자(LOC100422453)가 보고되었다.[26]표 1은 인간, 생쥐, 싹트기 효모(S. cerebisiae), 핵분열 효모(S. pombe)의 다양한 하위 단위에 대한 유전자 이름과 염색체 위치를 제공한다.
POLD1 유전자 촉진자는 세포주기 기계를 통해 규제되며, POLD1의 mRNA 발현이 DNA 복제 중 G1/S 후반기에 최고조에 도달한다.[28]POLD1 프로모터는 G/C가 풍부하며 TATA 박스가 없다.이 GC 박스 포함 프로모터의 전사는 Sp1 및 Sp3와 같은 Sp1 관련 전사 인자에 의해 규제되며, 이들의 결합은 11-bp 반복 결합 시퀀스를 통해 매개된다.[29][30]POLD1 프로모터는 주요 전사 시작 사이트 근처에 위치한 E2F와 유사한 시퀀스를 포함한다.[30]또 다른 규제 요소인 세포주기 요소/세포주기 유전자 호몰로지 영역(CDE/CHR)은 E2F1과 p21 단백질에 의한 G2/M상에서의 POLD1 전사에 중요하다.[31][32]P53은 p53-p21-DREAM-CDE/CHR 경로의 간접 종속적 활성화에 의해 POLD1 전사를 규제한다.[33]한 연구는 p53 종양 억제기 단백질이 POLD1 추진자와 결합하기 위해 Sp1과 경쟁한다는 것을 보고했다.[29]miR(miR) miR-155인 마이크로RNA는 POLD1 추진자(RTMAAYA; response element)에 배치 바인딩 사이트가 있는 [34]전사 계수 FOXO3a를 억제하여 POLD1을 간접적으로 하향 조정한다.[35]
단백질
POLD1/p125는 다른 DNA 중합체(Polα 및 ε)[37]와 유사한 B-패밀리 접힘이 있다.인간 POLD1/p125는 N-단자 끝(resides 4-19)에 배치 핵 국산화 신호가 있다.[24]잔류물 304-533은 exonuclease 영역(그림 2)을 포함하고, 잔류물 579-974는 중합효소 영역을 포함한다.exonuclease 도메인은 B-DNA 중합효소 계열에 공통되는 DEDDy형 dnaQ 같은 도메인이다.[38]이 영역은 뉴클레오타이드 오인시 중합효소와 엑소누클리스 활성부위 간 전환을 도와주는 베타 헤어핀 구조를 가지고 있다.
폴리머아제 영역 중 가장 많이 보존된 모티브 A와 C.이들에는 활성 부위에서 칼슘을 결합하는 A(DXXLYPS, D602)와 C(DTDS, D757)라는 두 개의 촉매 아스파테이트가 있다.모티프 A는 뉴클레오티드 결합과 인광 결합 형성에 중요한 11개의 아미노산을 가지고 있다.
티로신 Y701은 리보뉴클레오티드 결합을 방지하는 설탕 강직 게이트로서 RB69 박테리오파지 직교에서 티로신 Y567과 유사하게 기능한다.[39]LXCXE 모티브(711 ~ 715)는 셀 주기의 G1 단계 동안 pRB에 대한 바인딩을 매개한다.[40]또한 중합효소 영역은 결합 및 촉매 기능에 중요한 KKRY 모티브(잔여 806~809)가 보존되어 있다.[41]POLD1은 단백질 부호화 영역 전체에 분산된 작은 시퀀스 모티브로 대표되는 핵극억제 시퀀스(NoDS) 모티브를 통해 산성화 시 핵물질을 대상으로 할 수 있다.[42][43][44]C-단자 영역은 세포핵항원증식(PCNA) 결합과 부속품 서브유닛의 모집에 필요한 두 개의 보존된 사이스틴이 풍부한 금속 바인딩 모티브(CysA와 CysB)(1012년과 1083년)를 가지고 있다.[45]CysB는 미토콘드리아 철황 클러스터(ISC) 조립 기계의 기능이 필요한 시토솔릭 철황 단백질 조립체(CIA)를 통해 추가된 [4Fe-4S] 클러스터를 조정한다.[46]성숙 과정은 특정 Fe-S 클러스터 삽입을 보장하기 위해 아포프로테인과 상호작용하는 복합 CIA1-CIA2B/FAM96B-MMS19를 대상으로 하는 핵심 표적형 CIA1-CIA2B/FAM96B-MMS19에 의해 매개된다.[47][48]

결사 Binding 연구는 POLD2 단단히 POLD1과 관련된;각각의 그리고 POLD1과 POLD2.[51][52]Polδ heterotetramer 부분 Sf9 세포에서 coexpression에 의해 다시 구성되고 POLD4 상호 작용 속성 Polδ에 송아지 흉선에서 정제한 유사하다고 했던 또 다른과 POLD3과 POLD2 상호 작용하고, 완전한 holoenz을 보여 주었다.yme다PCNA에 의해 매우 강한 자극을 받았다.[53]많은 연구에서 POLD1이 중합효소와 3'-5'의 exonuclease 교정 활동을 모두 가지고 있지만, 다른 하위 단위는 이러한 활동, DNA 결합 능력, 그리고 PCNA 및 그 클램프 로더 복제 인자 C(RFC)와의 기능적으로 중요한 상호작용을 증가시킨다는 것을 보여주었다.DNA PolΔ 홀로엔자임(Holoenzyme)은 중합효소 복합체의 4개 서브유닛뿐만 아니라 PCNA와 RFC를 포함하는 것으로 종종 간주된다(그림 1).
많은 다른 연구와 화면은 DNA 복제 및 복구 기능과 관련된 추가적인 상호 작용 파트너를 식별했다.그림 3은 및 를 통해[54] 추가적으로 액세스할 수 있는 복제 및 수리 중 확립된 및 투입적 상호작용의 매트릭스를 보여준다.[55]밴더빌트 대학교의 한 웹사이트는 복잡하고 직접적인 물리적 상호작용, 규제관계, 공동표현 등의 기준에 기초하여 중요한 POLD1 단백질 구조와 다양한 등급의 유전자와 단백질 상호작용에 대한 추가적인 상호작용을 제공한다.[56]
| 중합효소 델타 서브유닛 | 단백질명 인간으로서 | 호모 사피엔스 | 무스 무스쿨루스 | 사카로마이오스 세레비시아아과 | 정신분열체 포메 |
|---|---|---|---|---|---|
| A(촉매) | p125 | POLD1-Chr 19q13.3 | Pold1-Chr 7B4 | POL3-Chr IV | cdc6-Chr II |
| B(액세서리) | p50 | POLD2-Chr 7p13 | Pold2-Chr 11A2 | POL31-Chr X | cdc1-Chr I |
| C(액세서리) | p66 | POLD3-Chr 11q14 | Pold3-Chr 7F1 | POL32-Chr X | cdc27-Chr II |
| D(액세서리) | p12 | POLD4-Chr 11q13 | 폴드4-크르 19A | - | cdm1-Chr II |
| 표 1: 인간, 생쥐, 싹트기 및 핵분열 효모에 있는 중합효소 델타의 다양한 아유닛에 대한 유전자 이름 및 염색체 위치. | |||||
표현 및 규정

POLD1/P125 단백질은 심장과 폐조직의 높은 수준을 가진 인간 조직의 패널에 걸쳐 보편적으로 표현된다.[60]POLD1/p125의 아세포 국산화(subcellular localization)는 주로 핵 및 핵종에 있다.[61]
POLD1/p125의 감소는 노년기 인간의 피부 섬유조직과 노년기 인구의 림프구에서 관찰되었다.[62][63]POLD1/p125 표현은 DNA 손상에 대응하여 후생적으로 규제된다.[64]다른 연구들도 POLD1/p125 식이 miR-155, [34]p53[29] 및 긴 비코딩 RNA, PVT1에 의해 조절된다는 것을 보여주었다.[65]DNA 손상 또는 복제 응력(UV등, 메틸메탄설폰산염, 히드록시우라 또는 아프리디콜린)이 존재할 경우 POLD4/p12 서브유닛은 급속히 저하된다.p125의 촉매 활성도는 헤테로테트라머(PolΔ4, p12[66][67] 포함) 또는 헤테로트리머(PolΔ3, p12 미포함)에 있는지 여부가 다르다.[68]헤테로트리머의 생산은 DSB의 수리와 가능한 동질 재조합(HR)에 관련된 단백질인 E3 리거아제 RNF8에 의한 p12 저하에 의존한다.[69]또한 E3 ligase CRL4는Cdt2 정상적인 DNA 복제 과정과 DNA 손상이 있는 상태에서 POLD4/p12를 저하시킬 수 있다.[70]또한 POLD4/p12는 칼슘 유발 사멸과 관련된 프로테아제 µ-calpain에 의해 분해될 수 있다.[71][72]
POLD1/p125에는 산증에 반응하여 뉴클레오스로의 전송을 조절하는 NoDS 도메인이 있다.[44]핵분열 수송은 p50 서브유닛과 WRN 단백질 사이의 직접적인 상호작용이 필요하다.[73]DNA 손상 반응 동안 WRN은 뉴클레오루스 밖으로 이동하여 폴Δ를 방출한다.[74][75]POLD1/p125는 PDIP46/SKAR[76] 및 LMO2와도 상호작용하는 것으로 나타났다.[77][78]
함수
DNA 복제
DNA 복제는 여러 개의 DNA 중합체를 포함한 많은 효소와 단백질을 포함하는 고도로 조직화된 과정이다.세포주기의 S상에서의 주요 복제활동은 중합효소 알파(Polα), 중합효소 델타(PolΔ), 중합효소 엡실론(Pollarase 엡실론)의 세 가지 DNA 중합효소에 의존한다.Polα에 의한 DNA 합성을 시작한 후, PolΔ 또는 Polε는 각각 후행 및 선행 Strand 합성을 실행한다.[79]이러한 중합체는 매우 높은 충실도를 유지하며, 왓슨-크릭 베이스 페어링과 3' exhonuclease(또는 교정) 활동에 의해 보장된다.[80]최근의 연구는 폴Δ가 선두의 가닥을 합성할 수 있다고 주장했다.[80][81][82][83][84]이러한 중합체가 복제와 관련된 다른 요소들과 어떻게 기능하는가는 그들이 결함이 있을 때 만들어내는 변이적 풍경을 설명하기 때문에 매우 흥미롭다.복제 충실도의 유지란 폴리머아제 Δ와 ε에 의한 고유 오차,[85] 교정과 MMR의 평형, 두 가닥 사이의 리보뉴클레오티드 처리의 구별을 미세하게 균형잡힌 것이다.[36]효모모델에 대한 광범위한 연구는 PolΔ와 Polε homologies의 exonuclease 영역에서의 돌연변이가 검토된 돌연변이 표현형을 유발할 수 있다는 것을 보여주었다.[86]지연된 스트랜드 합성 동안 합성된 단일 좌초(ss) DNA는 APOBEC 돌연변이를 위한 선택적 표적일 뿐만 아니라 ss-DNA 손상 작용에 의해 표적이 될 수 있다.[87]복제 후 빠르게 다시 결합하는 DNA 결합 단백질은 폴Δ가 성숙한 후행 가닥에서 폴α에 의해 생성된 오류를 복구하는 것을 막는다.[88]효모 연구는 PolΔ가 선행 스트랜드의 Pol proof 오류를 교정할 수 있다는 것을 보여주었다.[89]
DNA 수리
POLD1 활동은 MMR(Matches repair), TLS(Translesion resolation, TLS), BEER(Base exection repair), NER(Neucleotide excision repair, Double-Strand break, DSB) 수리를 포함하여 여러 가지 진화적으로 보존된 DNA 수리 프로세스에 기여한다.[6]POLD1은 BER, NER, MMR의 사후 처리 단계를 중재한다.[6] PolΔ는 새로 합성된 DNA의 복제 후 교정을 지원하기 위해 MMR 기계와 상호작용한다.[90] POLD1과 MMR 구성요소를 불활성화하는 돌연변이를 가진 세포와 함께 돌연변이율이 상승한다.[91][92]위에서 언급한 바와 같이 PolΔ 헤테로트리머(PolΔ3)는 POLD1의 지배적 과두형태가 되어 DNA 손상이 존재하는 동안 활동한다.폴Δ3는 (PolΔ4)보다 오류 발생률이 낮으며, 더 나은 교정 활동과 관련하여 일치하지 않는 쌍을 더 잘 구별할 수 있다. 그러나, 일부 기본 병변을 우회할 수 있는 능력이 감소되었다.[74][93]대신, Pol pol 촉매 서브 유닛인 p353의 p125를 대체하여 더 나은 우회 활동을 허용하기 때문에 TLS는 전문 폴리머아제 제타(Polζ)로 전환하는 것이 중요하다.[6]이 프로세스에서 POLD1/p125의 보존도가 높은 C-단자 도메인(CTD)은 Pol of의 CTD 도메인과 상호작용하며, 각 CTD 내의 철 클러스터는 TLS 동안 중합효소 전환을 허용하는 POLD2에 바인딩을 포함하는 상호작용을 중재한다.[94]최근 일부 연구에서는 PolΔ에서 Poll 람다(Poll 람다)로의 전환도 7,8-Dihydro-8-oxoguanine 병변과 같은 산화 DNA 손상의 복구와 TLS를 지원한다고 제안하고 있다.[95]
POLD1의 고갈은 인간 세포에서 G1과 G2/M 단계의 세포 주기를 정지시킬 수 있다.[96]이러한 단계의 세포 주기 블록은 일반적으로 DNA 손상의 존재와 DNA 손상 체크포인트의 활성화를 나타낸다.POLD1이 고갈된 세포는 DNA 손상 체크포인트 키나아제 ATR과 CHK1의 억제에 민감하다.[97]S. 퐁베에서 HR 메커니즘은 PolΔ Strand 합성 활동을 활용하여 정지된 복제 포크를 재시작할 수 있지만, 그러한 비알렐릭 HR 매개 재시작은 유전학적 불안정성을 증가시킬 가능성이 있는 오류 발생 가능성이 매우 높다.[98]폴Δ는 구조적으로 기능적으로 WRN 단백질과 상호작용하며, WRN은 뉴클레오루스에 폴Δ를 채용한다.[73]WRN 유전자는 베르너 증후군(자체 열성 장애)에서 돌연변이를 일으켜 노화를 가속화하고 유전적 불안정성을 증가시킨다.WRN과의 상호작용은 PCNA 독립적인 방식으로 PolΔ의 공정성을 증가시킨다.[99]이러한 상호작용을 통해 WRN은 DNA 복제-복제에 직접 영향을 미치며 폴Δ 매개 합성에서 도움을 준다.
임상적 유의성
암
DNA 수리 단백질은 암을 포함한 인간의 질병에서 중요한 것으로 밝혀졌다.예를 들어, MMR(MSH2, MLH1, MSH6, PMS2)에 관련된 DNA 수리 단백질(MSH2, MLH1, MSH6, PMS2)의 세균선 돌연변이가 린치증후군(LS)에서 설명되어 왔으며, 이는 마이크로위성 불안정성(MSI)이 존재하는 것이 특징이다.[100]보다 최근에는 폴로의 촉매 소단위인 POLD1과 PLAUC의 엑소누클리스 도메인에서 생식선 돌연변이가 보고되고 있다.이러한 돌연변이는 올리고성 다종증, 초기 대장암(CRC), 자궁내막암(EDMC), 유방암, 뇌종양과 관련이 있다.[101][102][103][104][105] (검토[7] 결과) 암과 연관된 보고된 POLD1 돌연변이의 대부분은 엑소누클리스 영역에 존재한다.[7][101][102][106][107][108]LS와 대조적으로 POLD1 돌연변이는 마이크로 위성 안정적이다.일부 데이터는 POLD1 종양이 APC와 KRAS를 포함한 유전자의 운전자 돌연변이와 연관되어 있다는 생각을 제시한다.[101]POLD1은 돌연변이 p를 잘못 이해한다.S478N은 exonuclease 영역에 손상 및 병원성으로 확인되었다.[101]기타 POLD1 변형은 임상적으로 확인되었으며, 손상될 것으로 예측되었으며 현재 추가 조사 중이다(예: D316H, 페이지 D316G, 페이지 R409W, 페이지 L474P, 페이지 P327L).[102][103][104]
소아과 환자의 경우 POLD1 또는 PARE에서 이중 적중 돌연변이와 bMMRD(biallelic match repair) 결핍증(bmMRD)이 극초단파 종양 표현형으로 이어진다.[109][110][111]비록 이 POLD1.[112][113][114][115][116][117]Bouffet는 POLE(P436H번째, S461P은 다른)에 체세포 돌연변이가bMMRD- 교아 종 multiforme고 알. 보고서 두 형제 자매를 위한 직접 평가할 필요가 있ultra-hypermutation로 종양에서의 그러한 표현형, s개발에 최근의 암 치료법에 더 좋은 반응을 나타낼 수 있다.a에 Howed은 항구적인 반응항균 사망 1 억제제 니볼루맵을 이용한 임상시험POLD1 돌연변이는 셀라인과[118][119][120][121] 마우스 모델에서 연구되었다.예를 들어 효소 기능을 교란시키는 생쥐의 동질성 폴Δ 돌연변이는 암 발생률이 매우 높아지게 된다.[122]
MDPL
POLD1의 유해한 돌연변이는 또한 하악성 저공포증, 청각장애, 프로게로이드 특징으로 알려진 지방질회생증후군(MDPL/MDP) 증후군(Online Mendelian Assistance in Man(OMIM) 데이터베이스의 #615381) 환자들에서도 관찰되었다.[60][123][124]이것은 매우 드문 증후군이며, 돌연변이를 설명하는 연구는 거의 보고되지 않았다.관찰된 돌연변이는 exonuclease 영역과 중합효소 영역에 영향을 미치는 영역이다.[60][123]동일한 이질 변종인 c.1812_1814delCTC p를 사용하여 관련 없는 5개의 사례가 설명되었다.Ser605del (RS398122386)S605는 고분자 활성 부위의 보존도가 높은 모티브 A에 있다.이 변종은 DNA 결합 활동을 억제하지 않고 촉매에 영향을 미친다.또 다른 변종이 별도의 환자(p)에 보고되었다.R507C).[123] 이 변종은 보존도가 높은 엑소III 영역에 위치하며, 아직 완전히 특징지어지지는 않았다.
POLD1 Ser605del 및 R507C 변종도 비정형 베르너 증후군(AWS) 환자의 하위 집합에서 확인되었다.분자검사 후 이 환자들은 MDPL/MDP 환자로 재분류되었다.MDPL/DP, AWS, 베르너 증후군은 모두 프로게리아와 함께 존재한다.[125]세균선 전이의 첫 번째 예는 Ser605del 돌연변이를 가진 어머니와 아들에게서 관찰되었다.[126]최근 두 개의 독립적인 연구에서 Polε의 촉매 서브 유닛인 PARE1에서 동일한 동질성 스플라이스 변형을 가진 환자를 확인했다.그 중 하나는 얼굴 이상형, 면역 결핍증, 산모, 단신형(FILS 증후군이라고도 한다)[127]의 표현형을 보여준다.두 번째 증상은 더 심한 증상을 보였다.[128]이러한 경우는 중합효소 유전자의 기능을 목표로 하는 유전적 돌연변이와 관련된 발달상의 결함의 수가 증가하고 있다.
POLD1의 연령에 따른 하향규제가 관찰되었다.[63]비록 임상적으로 어떤 의미도 아직 이 표현형과 연관되어 있지 않지만.이러한 병리학이나 돌연변이 사이에 관계가 있는지, 그리고 암에 걸리기 쉬운 성향이 있는지를 파악하기 위한 연구도 진행 중이다.현재 POLD1 결함이 병원성으로 제안된 메커니즘은 유전학적 불안정성과 체크포인트 활성화로 이어지는 복제 결함에 초점을 맞추고 있으며, 궁극적으로 세포사망이나 세포 노화를 초래한다.또는 폴Δ는 G1/S 체포 또는 초기 S 단계에서 라민 및 핵 봉투와 연관된다; 라민의 돌연변이는 MDPL/DP 및 Werner 증후군과 유사한 표현형을 가진 핵 봉투 관련 지질동물을 유발한다.[129]
암 위험도 평가 및 상업적 테스트
세습되는 직장암(회사)POLD1과 POLE의 교정 능력에 변화와 관련된 때때로“중합 효소 교정 폴립증 관련된”(PPAP),(비록 적어도 하나의 연구 POLD1 돌연변이non-polyposis 순환 중복 검사와 관련된 확인해 왔다)로 조건이 주어진다.[101][102][104][106][107]POLD1 돌연변이 또한 증가된 ca.와 연관 되어 왔던자궁내막암의 [101][104][105]소인최근의 한 연구는 1) 20-100 아데노마 발생과 2) 대장암 및 자궁내막암에 대한 암스테르담 II 기준을 충족하는 가족력을 포함하는 POLD1 돌연변이에 대한 유전자 검사에 대한 지침을 제안했다.[103]POLD1/POLE에 돌연변이가 있는 가족을 위한 현재 임상시험 지침에는 대장경련(매 1~2년), 조기(20~25년), 뇌종양 및 자궁내막암 검사 가능성(20~25년), 자궁내막암 검사(여성 보균자의 경우 40년부터 시작) 등이 있다.[103]현재 특정 POLD1 돌연변이로 인한 정확한 암 위험을 결정하기 위한 연구가 진행 중이다.현재 데이터에 따르면 이 유전자의 돌연변이는 침투성이 매우 높다.또 다른 최근의 연구는 PolΔ와 Polε 돌연변이에 영향을 미치는 돌연변이가 MMR 돌연변이와 함께 발생할 수 있다는 것을 보여주었다.[110]이는 패널 유전자 검사가 MSI 환자에서도 MMR과 Pol 유전자를 포함해야 함을 시사한다.
POLD1의 돌연변이에 대한 상업적 진단 테스트에는 몇 가지 옵션이 있다.[130] 유전자 테스트에는 일반적으로 POLD1 코딩 exon(26)과 인접한 비 코딩 영역에 최소 20개의 베이스가 포함된다.돌연변이가 알려진 가정의 경우 돌연변이의 존재를 확인하기 위한 단일 사이트 테스트도 이용할 수 있다.[130]이러한 유전자 테스트의 이용가능성은 이전에 유전적으로 정의되지 않은 대장암이나 대장암 타입 "X"[105]로 분류되었던 암에 대한 새로운 가능성을 열어주었다.MDPL/MDP를 위한 임상시험 자원도 개발되었다.[131]
메모들
이 글의 2016년 버전은 외부 전문가가 이중 출판 모델로 업데이트했다.해당 학술적 동료 검토 기사는 진에 게재되었으며, 다음과 같이 인용할 수 있다. Emmanuelle Nicolas; Erica A Golemis; Sanjeevani Arora (16 June 2016). "POLD1: Central mediator of DNA replication and repair, and implication in cancer and other pathologies". Gene. Gene Wiki Review Series. 590 (1): 128–141. doi:10.1016/J.GENE.2016.06.031. ISSN 0378-1119. PMC 4969162. PMID 27320729. Wikidata Q38870074. |
참조
- ^ a b c GRCh38: 앙상블 릴리스 89: ENSG000062822 - 앙상블, 2017년 5월
- ^ a b c GRCm38: 앙상블 릴리스 89: ENSMUSG000038644 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ a b Chung DW, Zhang JA, Tan CK, Davie EW, So AG, Downey KM (December 1991). "Primary structure of the catalytic subunit of human DNA polymerase delta and chromosomal location of the gene". Proceedings of the National Academy of Sciences of the United States of America. 88 (24): 11197–201. Bibcode:1991PNAS...8811197C. doi:10.1073/pnas.88.24.11197. PMC 53101. PMID 1722322.
- ^ a b c d e f Prindle MJ, Loeb LA (December 2012). "DNA polymerase delta in DNA replication and genome maintenance". Environmental and Molecular Mutagenesis. 53 (9): 666–82. doi:10.1002/em.21745. PMC 3694620. PMID 23065663.
- ^ a b c d Rayner E, van Gool IC, Palles C, Kearsey SE, Bosse T, Tomlinson I, Church DN (January 2016). "A panoply of errors: polymerase proofreading domain mutations in cancer". Nature Reviews. Cancer. 16 (2): 71–81. doi:10.1038/nrc.2015.12. PMID 26822575. S2CID 9359891.
- ^ Kornberg A, Kornberg SR, Simms ES (April 1956). "Metaphosphate synthesis by an enzyme from Escherichia coli". Biochimica et Biophysica Acta. 20 (1): 215–27. doi:10.1016/0006-3002(56)90280-3. PMID 13315368.
- ^ Friedberg EC (February 2006). "The eureka enzyme: the discovery of DNA polymerase". Nature Reviews Molecular Cell Biology. 7 (2): 143–7. doi:10.1038/nrm1787. PMID 16493419. S2CID 39605644.
- ^ Byrnes JJ, Downey KM, Black VL, So AG (June 1976). "A new mammalian DNA polymerase with 3' to 5' exonuclease activity: DNA polymerase delta". Biochemistry. 15 (13): 2817–23. doi:10.1021/bi00658a018. PMID 949478.
- ^ "Enzymatic synthesis of deoxyribonucleic acid. XXXVI. A proofreading function for the 3 5′ exonuclease activity in deoxyribonucleic acid polymerase". ResearchGate. Retrieved 2016-04-25.
- ^ Reha-Krantz LJ (May 2010). "DNA polymerase proofreading: Multiple roles maintain genome stability". Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1804 (5): 1049–63. doi:10.1016/j.bbapap.2009.06.012. PMID 19545649.
- ^ Lee MY, Jiang YQ, Zhang SJ, Toomey NL (February 1991). "Characterization of human DNA polymerase delta and its immunochemical relationships with DNA polymerase alpha and epsilon". The Journal of Biological Chemistry. 266 (4): 2423–9. doi:10.1016/S0021-9258(18)52261-4. PMID 1703528.
- ^ Yang CL, Chang LS, Zhang P, Hao H, Zhu L, Toomey NL, Lee MY (February 1992). "Molecular cloning of the cDNA for the catalytic subunit of human DNA polymerase delta". Nucleic Acids Research. 20 (4): 735–45. doi:10.1093/nar/20.4.735. PMC 312012. PMID 1542570.
- ^ Cullmann G, Hindges R, Berchtold MW, Hübscher U (December 1993). "Cloning of a mouse cDNA encoding DNA polymerase delta: refinement of the homology boxes". Gene. 134 (2): 191–200. doi:10.1016/0378-1119(93)90093-i. PMID 8262377.
- ^ Lee MY, Tan CK, So AG, Downey KM (May 1980). "Purification of deoxyribonucleic acid polymerase delta from calf thymus: partial characterization of physical properties". Biochemistry. 19 (10): 2096–101. doi:10.1021/bi00551a015. PMID 7378348.
- ^ Lee MY, Tan CK, Downey KM, So AG (April 1984). "Further studies on calf thymus DNA polymerase delta purified to homogeneity by a new procedure". Biochemistry. 23 (9): 1906–13. doi:10.1021/bi00304a003. PMID 6426510.
- ^ Crute JJ, Wahl AF, Bambara RA (January 1986). "Purification and characterization of two new high molecular weight forms of DNA polymerase delta". Biochemistry. 25 (1): 26–36. doi:10.1021/bi00349a005. PMID 3954990.
- ^ Wahl AF, Crute JJ, Sabatino RD, Bodner JB, Marraccino RL, Harwell LW, Lord EM, Bambara RA (December 1986). "Properties of two forms of DNA polymerase delta from calf thymus". Biochemistry. 25 (24): 7821–7. doi:10.1021/bi00372a006. PMID 3099836.
- ^ Lee MY, Toomey NL (February 1987). "Human placental DNA polymerase delta: identification of a 170-kilodalton polypeptide by activity staining and immunoblotting". Biochemistry. 26 (4): 1076–85. doi:10.1021/bi00378a014. PMID 2436659.
- ^ Dresler SL, Kimbro KS (May 1987). "2',3'-Dideoxythymidine 5'-triphosphate inhibition of DNA replication and ultraviolet-induced DNA repair synthesis in human cells: evidence for involvement of DNA polymerase delta". Biochemistry. 26 (10): 2664–8. doi:10.1021/bi00384a002. PMID 3606985.
- ^ Nishida C, Reinhard P, Linn S (January 1988). "DNA repair synthesis in human fibroblasts requires DNA polymerase delta". The Journal of Biological Chemistry. 263 (1): 501–10. doi:10.1016/S0021-9258(19)57421-X. PMID 3335506.
- ^ "HGNC database of human gene names HUGO Gene Nomenclature Committee". www.genenames.org. Retrieved 2016-04-25.
- ^ a b Chung DW, Zhang JA, Tan CK, Davie EW, So AG, Downey KM (December 1991). "Primary structure of the catalytic subunit of human DNA polymerase delta and chromosomal location of the gene". Proceedings of the National Academy of Sciences of the United States of America. 88 (24): 11197–201. Bibcode:1991PNAS...8811197C. doi:10.1073/pnas.88.24.11197. PMC 53101. PMID 1722322.
- ^ Kemper RR, Ahn ER, Zhang P, Lee MY, Rabin M (September 1992). "Human DNA polymerase delta gene maps to region 19q13.3-q13.4 by in situ hybridization". Genomics. 14 (1): 205–6. doi:10.1016/s0888-7543(05)80311-8. PMID 1427831.
- ^ a b "POLD1 polymerase (DNA) delta 1, catalytic subunit [Homo sapiens (human)] - Gene - NCBI". www.ncbi.nlm.nih.gov. Retrieved 2016-04-25.
- ^ Goldsby RE, Singh M, Preston BD (January 1998). "Mouse DNA polymerase delta gene (Pold1) maps to chromosome 7". Mammalian Genome. 9 (1): 92–3. doi:10.1007/s003359900693. PMID 9434960. S2CID 42967770.
- ^ Mjelle R, Hegre SA, Aas PA, Slupphaug G, Drabløs F, Saetrom P, Krokan HE (June 2015). "Cell cycle regulation of human DNA repair and chromatin remodeling genes". DNA Repair. 30: 53–67. doi:10.1016/j.dnarep.2015.03.007. PMID 25881042.
- ^ a b c Li B, Lee MY (August 2001). "Transcriptional regulation of the human DNA polymerase delta catalytic subunit gene POLD1 by p53 tumor suppressor and Sp1". The Journal of Biological Chemistry. 276 (32): 29729–39. doi:10.1074/jbc.M101167200. PMID 11375983.
- ^ a b Zhao L, Chang LS (February 1997). "The human POLD1 gene. Identification of an upstream activator sequence, activation by Sp1 and Sp3, and cell cycle regulation". The Journal of Biological Chemistry. 272 (8): 4869–82. doi:10.1074/jbc.272.8.4869. PMID 9030545.
- ^ Müller GA, Wintsche A, Stangner K, Prohaska SJ, Stadler PF, Engeland K (2014-01-01). "The CHR site: definition and genome-wide identification of a cell cycle transcriptional element". Nucleic Acids Research. 42 (16): 10331–50. doi:10.1093/nar/gku696. PMC 4176359. PMID 25106871.
- ^ Song N, Zhu X, Shi L, An J, Wu Y, Sang J (June 2009). "Identification and functional analysis of a CDE/CHR element in the POLD1 promoter". Science in China Series C: Life Sciences. 52 (6): 551–9. doi:10.1007/s11427-009-0077-5. PMID 19557333. S2CID 19278457.
- ^ Fischer M, Quaas M, Steiner L, Engeland K (January 2016). "The p53-p21-DREAM-CDE/CHR pathway regulates G2/M cell cycle genes". Nucleic Acids Research. 44 (1): 164–74. doi:10.1093/nar/gkv927. PMC 4705690. PMID 26384566.
- ^ a b Czochor JR, Sulkowski P, Glazer PM (April 2016). "miR-155 Overexpression Promotes Genomic Instability by Reducing High-fidelity Polymerase Delta Expression and Activating Error-Prone DSB Repair". Molecular Cancer Research. 14 (4): 363–73. doi:10.1158/1541-7786.MCR-15-0399. PMC 5021065. PMID 26850462.
- ^ Chen X, Ji Z, Webber A, Sharrocks AD (February 2016). "Genome-wide binding studies reveal DNA binding specificity mechanisms and functional interplay amongst Forkhead transcription factors". Nucleic Acids Research. 44 (4): 1566–78. doi:10.1093/nar/gkv1120. PMC 4770209. PMID 26578569.
- ^ a b Lujan SA, Williams JS, Kunkel TA (2016-02-01). "Eukaryotic genome instability in light of asymmetric DNA replication". Critical Reviews in Biochemistry and Molecular Biology. 51 (1): 43–52. doi:10.3109/10409238.2015.1117055. PMC 4922258. PMID 26822554.
- ^ Doublié S, Zahn KE (2014-01-01). "Structural insights into eukaryotic DNA replication". Frontiers in Microbiology. 5: 444. doi:10.3389/fmicb.2014.00444. PMC 4142720. PMID 25202305.
- ^ "NCBI CDD Conserved Protein Domain DNA_polB_delta_exo". www.ncbi.nlm.nih.gov. Retrieved 2016-04-25.
- ^ Brown JA, Suo Z (February 2011). "Unlocking the sugar "steric gate" of DNA polymerases". Biochemistry. 50 (7): 1135–42. doi:10.1021/bi101915z. PMC 3040255. PMID 21226515.
- ^ Krucher NA, Zygmunt A, Mazloum N, Tamrakar S, Ludlow JW, Lee MY (November 2000). "Interaction of the retinoblastoma protein (pRb) with the catalytic subunit of DNA polymerase delta (p125)". Oncogene. 19 (48): 5464–70. doi:10.1038/sj.onc.1203930. PMID 11114723.
- ^ Hogg M, Aller P, Konigsberg W, Wallace SS, Doublié S (January 2007). "Structural and biochemical investigation of the role in proofreading of a beta hairpin loop found in the exonuclease domain of a replicative DNA polymerase of the B family". The Journal of Biological Chemistry. 282 (2): 1432–44. doi:10.1074/jbc.M605675200. PMID 17098747.
- ^ Lam YW, Trinkle-Mulcahy L (2015-01-01). "New insights into nucleolar structure and function". F1000Prime Reports. 7: 48. doi:10.12703/P7-48. PMC 4447046. PMID 26097721.
- ^ Mekhail K, Rivero-Lopez L, Al-Masri A, Brandon C, Khacho M, Lee S (October 2007). "Identification of a common subnuclear localization signal". Molecular Biology of the Cell. 18 (10): 3966–77. doi:10.1091/mbc.E07-03-0295. PMC 1995723. PMID 17652456.
- ^ a b Audas TE, Jacob MD, Lee S (January 2012). "Immobilization of proteins in the nucleolus by ribosomal intergenic spacer noncoding RNA". Molecular Cell. 45 (2): 147–57. doi:10.1016/j.molcel.2011.12.012. PMID 22284675.
- ^ Netz DJ, Stith CM, Stümpfig M, Köpf G, Vogel D, Genau HM, Stodola JL, Lill R, Burgers PM, Pierik AJ (January 2012). "Eukaryotic DNA polymerases require an iron-sulfur cluster for the formation of active complexes". Nature Chemical Biology. 8 (1): 125–32. doi:10.1038/nchembio.721. PMC 3241888. PMID 22119860.
- ^ Paul VD, Lill R (June 2015). "Biogenesis of cytosolic and nuclear iron-sulfur proteins and their role in genome stability". Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1853 (6): 1528–39. doi:10.1016/j.bbamcr.2014.12.018. PMID 25583461.
- ^ Gari K, León Ortiz AM, Borel V, Flynn H, Skehel JM, Boulton SJ (July 2012). "MMS19 links cytoplasmic iron-sulfur cluster assembly to DNA metabolism". Science. 337 (6091): 243–5. Bibcode:2012Sci...337..243G. doi:10.1126/science.1219664. PMID 22678361. S2CID 26605576.
- ^ Stehling O, Vashisht AA, Mascarenhas J, Jonsson ZO, Sharma T, Netz DJ, Pierik AJ, Wohlschlegel JA, Lill R (July 2012). "MMS19 assembles iron-sulfur proteins required for DNA metabolism and genomic integrity". Science. 337 (6091): 195–9. Bibcode:2012Sci...337..195S. doi:10.1126/science.1219723. PMC 3420340. PMID 22678362.
- ^ Hansen MF, Johansen J, Bjørnevoll I, Sylvander AE, Steinsbekk KS, Sætrom P, Sandvik AK, Drabløs F, Sjursen W (September 2015). "A novel POLE mutation associated with cancers of colon, pancreas, ovaries and small intestine". Familial Cancer. 14 (3): 437–48. doi:10.1007/s10689-015-9803-2. PMC 4559173. PMID 25860647.
- ^ Zuo Y, Deutscher MP (March 2001). "Exoribonuclease superfamilies: structural analysis and phylogenetic distribution". Nucleic Acids Research. 29 (5): 1017–26. doi:10.1093/nar/29.5.1017. PMC 56904. PMID 11222749.
- ^ Simon M, Giot L, Faye G (August 1991). "The 3' to 5' exonuclease activity located in the DNA polymerase delta subunit of Saccharomyces cerevisiae is required for accurate replication". The EMBO Journal. 10 (8): 2165–70. doi:10.1002/j.1460-2075.1991.tb07751.x. PMC 452904. PMID 1648480.
- ^ Makarova KS, Krupovic M, Koonin EV (2014-01-01). "Evolution of replicative DNA polymerases in archaea and their contributions to the eukaryotic replication machinery". Frontiers in Microbiology. 5: 354. doi:10.3389/fmicb.2014.00354. PMC 4104785. PMID 25101062.
- ^ Xie B, Mazloum N, Liu L, Rahmeh A, Li H, Lee MY (November 2002). "Reconstitution and characterization of the human DNA polymerase delta four-subunit holoenzyme". Biochemistry. 41 (44): 13133–42. doi:10.1021/bi0262707. PMID 12403614.
- ^ Lab, Mike Tyers. "Database of Protein, Chemical, and Genetic Interactions BioGRID". thebiogrid.org. Retrieved 2016-04-25.
- ^ "POLD1 protein (Homo sapiens) - STRING network view". string-db.org. Retrieved 2016-04-25.
- ^ "Cancer Cell Metabolism Database ~~ Bioinformatics and Systems Medicine Laboratory ~~". bioinfo.mc.vanderbilt.edu. Archived from the original on 2016-04-26. Retrieved 2016-04-25.
- ^ "STRING: functional protein association networks". string-db.org. Retrieved 2016-04-25.
- ^ Ono, Keiichiro. "Cytoscape: An Open Source Platform for Complex Network Analysis and Visualization". www.cytoscape.org. Retrieved 2016-04-25.
- ^ "POLD1 protein (Homo sapiens) - STRING network view". string-db.org. Retrieved 2016-04-25.
- ^ a b c Weedon MN, Ellard S, Prindle MJ, Caswell R, Lango Allen H, Oram R, Godbole K, Yajnik CS, Sbraccia P, Novelli G, Turnpenny P, McCann E, Goh KJ, Wang Y, Fulford J, McCulloch LJ, Savage DB, O'Rahilly S, Kos K, Loeb LA, Semple RK, Hattersley AT (August 2013). "An in-frame deletion at the polymerase active site of POLD1 causes a multisystem disorder with lipodystrophy". Nature Genetics. 45 (8): 947–50. doi:10.1038/ng.2670. PMC 3785143. PMID 23770608.
- ^ "Genatlas sheet". genatlas.medecine.univ-paris5.fr. Retrieved 2016-04-25.
- ^ Takahashi Y, Moriwaki S, Sugiyama Y, Endo Y, Yamazaki K, Mori T, Takigawa M, Inoue S (February 2005). "Decreased gene expression responsible for post-ultraviolet DNA repair synthesis in aging: a possible mechanism of age-related reduction in DNA repair capacity". The Journal of Investigative Dermatology. 124 (2): 435–42. doi:10.1111/j.0022-202X.2004.23591.x. PMID 15675965.
- ^ a b Wang JL, Guo HL, Wang PC, Liu CG (December 2012). "Age-dependent down-regulation of DNA polymerase δ1 in human lymphocytes". Molecular and Cellular Biochemistry. 371 (1–2): 157–63. doi:10.1007/s11010-012-1432-6. PMID 22915169. S2CID 15443915.
- ^ Karkhanis V, Wang L, Tae S, Hu YJ, Imbalzano AN, Sif S (August 2012). "Protein arginine methyltransferase 7 regulates cellular response to DNA damage by methylating promoter histones H2A and H4 of the polymerase δ catalytic subunit gene, POLD1". The Journal of Biological Chemistry. 287 (35): 29801–14. doi:10.1074/jbc.M112.378281. PMC 3436169. PMID 22761421.
- ^ Cui M, You L, Ren X, Zhao W, Liao Q, Zhao Y (February 2016). "Long non-coding RNA PVT1 and cancer". Biochemical and Biophysical Research Communications. 471 (1): 10–4. doi:10.1016/j.bbrc.2015.12.101. PMID 26850852.
- ^ Li H, Xie B, Zhou Y, Rahmeh A, Trusa S, Zhang S, Gao Y, Lee EY, Lee MY (May 2006). "Functional roles of p12, the fourth subunit of human DNA polymerase delta". The Journal of Biological Chemistry. 281 (21): 14748–55. doi:10.1074/jbc.M600322200. PMID 16510448.
- ^ Podust VN, Chang LS, Ott R, Dianov GL, Fanning E (February 2002). "Reconstitution of human DNA polymerase delta using recombinant baculoviruses: the p12 subunit potentiates DNA polymerizing activity of the four-subunit enzyme". The Journal of Biological Chemistry. 277 (6): 3894–901. doi:10.1074/jbc.M109684200. PMID 11711545.
- ^ Zhang S, Zhou Y, Trusa S, Meng X, Lee EY, Lee MY (May 2007). "A novel DNA damage response: rapid degradation of the p12 subunit of dna polymerase delta". The Journal of Biological Chemistry. 282 (21): 15330–40. doi:10.1074/jbc.M610356200. PMID 17317665.
- ^ Lee MY, Zhang S, Lin SH, Wang X, Darzynkiewicz Z, Zhang Z, Lee EY (2014-01-01). "The tail that wags the dog: p12, the smallest subunit of DNA polymerase δ, is degraded by ubiquitin ligases in response to DNA damage and during cell cycle progression". Cell Cycle. 13 (1): 23–31. doi:10.4161/cc.27407. PMC 3925730. PMID 24300032.
- ^ Zhang S, Zhao H, Darzynkiewicz Z, Zhou P, Zhang Z, Lee EY, Lee MY (October 2013). "A novel function of CRL4(Cdt2): regulation of the subunit structure of DNA polymerase δ in response to DNA damage and during the S phase". The Journal of Biological Chemistry. 288 (41): 29550–61. doi:10.1074/jbc.M113.490466. PMC 3795253. PMID 23913683.
- ^ Fan X, Zhang Q, You C, Qian Y, Gao J, Liu P, Chen H, Song H, Chen Y, Chen K, Zhou Y (2014-01-01). "Proteolysis of the human DNA polymerase delta smallest subunit p12 by μ-calpain in calcium-triggered apoptotic HeLa cells". PLOS ONE. 9 (4): e93642. Bibcode:2014PLoSO...993642F. doi:10.1371/journal.pone.0093642. PMC 3972206. PMID 24691096.
- ^ Zhang Q, Zhang Q, Chen H, Chen Y, Zhou Y (February 2016). "Multiple forms of human DNA polymerase delta sub-assembling in cellular DNA transactions". Current Protein & Peptide Science. 17 (8): 746–755. doi:10.2174/1389203717666160226145006. PMID 26916162.
- ^ a b Szekely AM, Chen YH, Zhang C, Oshima J, Weissman SM (October 2000). "Werner protein recruits DNA polymerase delta to the nucleolus". Proceedings of the National Academy of Sciences of the United States of America. 97 (21): 11365–70. Bibcode:2000PNAS...9711365S. doi:10.1073/pnas.97.21.11365. PMC 17206. PMID 11027336.
- ^ a b Karmakar P, Bohr VA (November 2005). "Cellular dynamics and modulation of WRN protein is DNA damage specific". Mechanisms of Ageing and Development. 126 (11): 1146–58. doi:10.1016/j.mad.2005.06.004. PMID 16087220. S2CID 6128022.
- ^ Lee SY, Lee H, Kim ES, Park S, Lee J, Ahn B (April 2015). "WRN translocation from nucleolus to nucleoplasm is regulated by SIRT1 and required for DNA repair and the development of chemoresistance". Mutation Research. 774: 40–8. doi:10.1016/j.mrfmmm.2015.03.001. PMID 25801465.
- ^ Wang X, Zhang S, Zheng R, Yue F, Lin SH, Rahmeh AA, Lee EY, Zhang Z, Lee MY (February 2016). "PDIP46 (DNA polymerase δ interacting protein 46) is an activating factor for human DNA polymerase δ". Oncotarget. 7 (5): 6294–313. doi:10.18632/oncotarget.7034. PMC 4868757. PMID 26819372.
- ^ Boyer AS, Walter D, Sørensen CS (January 2016). "DNA replication and cancer: From dysfunctional replication origin activities to therapeutic opportunities". Seminars in Cancer Biology. 37–38: 16–25. doi:10.1016/j.semcancer.2016.01.001. PMID 26805514.
- ^ Sincennes MC, Humbert M, Grondin B, Lisi V, Veiga DF, Haman A, Cazaux C, Mashtalir N, Affar el B, Verreault A, Hoang T (February 2016). "The LMO2 oncogene regulates DNA replication in hematopoietic cells". Proceedings of the National Academy of Sciences of the United States of America. 113 (5): 1393–8. Bibcode:2016PNAS..113.1393S. doi:10.1073/pnas.1515071113. PMC 4747768. PMID 26764384.
- ^ Nick McElhinny SA, Gordenin DA, Stith CM, Burgers PM, Kunkel TA (April 2008). "Division of labor at the eukaryotic replication fork". Molecular Cell. 30 (2): 137–44. doi:10.1016/j.molcel.2008.02.022. PMC 2654179. PMID 18439893.
- ^ a b Johnson RE, Klassen R, Prakash L, Prakash S (July 2015). "A Major Role of DNA Polymerase δ in Replication of Both the Leading and Lagging DNA Strands". Molecular Cell. 59 (2): 163–75. doi:10.1016/j.molcel.2015.05.038. PMC 4517859. PMID 26145172.
- ^ Daigaku Y, Keszthelyi A, Müller CA, Miyabe I, Brooks T, Retkute R, Hubank M, Nieduszynski CA, Carr AM (March 2015). "A global profile of replicative polymerase usage". Nature Structural & Molecular Biology. 22 (3): 192–8. doi:10.1038/nsmb.2962. PMC 4789492. PMID 25664722.
- ^ Pavlov YI, Shcherbakova PV (March 2010). "DNA polymerases at the eukaryotic fork-20 years later". Mutation Research. 685 (1–2): 45–53. doi:10.1016/j.mrfmmm.2009.08.002. PMC 2822129. PMID 19682465.
- ^ Stillman B (July 2015). "Reconsidering DNA Polymerases at the Replication Fork in Eukaryotes". Molecular Cell. 59 (2): 139–41. doi:10.1016/j.molcel.2015.07.004. PMC 4636199. PMID 26186286.
- ^ Burgers PM, Gordenin D, Kunkel TA (February 2016). "Who Is Leading the Replication Fork, Pol ε or Pol δ?". Molecular Cell. 61 (4): 492–3. doi:10.1016/j.molcel.2016.01.017. PMC 4838066. PMID 26895421.
- ^ Korona DA, Lecompte KG, Pursell ZF (March 2011). "The high fidelity and unique error signature of human DNA polymerase epsilon". Nucleic Acids Research. 39 (5): 1763–73. doi:10.1093/nar/gkq1034. PMC 3061053. PMID 21036870.
- ^ Skoneczna A, Kaniak A, Skoneczny M (November 2015). "Genetic instability in budding and fission yeast-sources and mechanisms". FEMS Microbiology Reviews. 39 (6): 917–67. doi:10.1093/femsre/fuv028. PMC 4608483. PMID 26109598.
- ^ Hoopes JI, Cortez LM, Mertz TM, Malc EP, Mieczkowski PA, Roberts SA (February 2016). "APOBEC3A and APOBEC3B Preferentially Deaminate the Lagging Strand Template during DNA Replication". Cell Reports. 14 (6): 1273–82. doi:10.1016/j.celrep.2016.01.021. PMC 4758883. PMID 26832400.
- ^ Reijns MA, Kemp H, Ding J, de Procé SM, Jackson AP, Taylor MS (February 2015). "Lagging-strand replication shapes the mutational landscape of the genome". Nature. 518 (7540): 502–6. Bibcode:2015Natur.518..502R. doi:10.1038/nature14183. PMC 4374164. PMID 25624100.
- ^ Flood CL, Rodriguez GP, Bao G, Shockley AH, Kow YW, Crouse GF (March 2015). "Replicative DNA polymerase δ but not ε proofreads errors in Cis and in Trans". PLOS Genetics. 11 (3): e1005049. doi:10.1371/journal.pgen.1005049. PMC 4351087. PMID 25742645.
- ^ Herr AJ, Kennedy SR, Knowels GM, Schultz EM, Preston BD (March 2014). "DNA replication error-induced extinction of diploid yeast". Genetics. 196 (3): 677–91. doi:10.1534/genetics.113.160960. PMC 3948800. PMID 24388879.
- ^ Morrison A, Johnson AL, Johnston LH, Sugino A (April 1993). "Pathway correcting DNA replication errors in Saccharomyces cerevisiae". The EMBO Journal. 12 (4): 1467–73. doi:10.1002/j.1460-2075.1993.tb05790.x. PMC 413358. PMID 8385605.
- ^ Li L, Murphy KM, Kanevets U, Reha-Krantz LJ (June 2005). "Sensitivity to phosphonoacetic acid: a new phenotype to probe DNA polymerase delta in Saccharomyces cerevisiae". Genetics. 170 (2): 569–80. doi:10.1534/genetics.104.040295. PMC 1450396. PMID 15802517.
- ^ Meng X, Zhou Y, Zhang S, Lee EY, Frick DN, Lee MY (February 2009). "DNA damage alters DNA polymerase delta to a form that exhibits increased discrimination against modified template bases and mismatched primers". Nucleic Acids Research. 37 (2): 647–57. doi:10.1093/nar/gkn1000. PMC 2632934. PMID 19074196.
- ^ Baranovskiy AG, Lada AG, Siebler HM, Zhang Y, Pavlov YI, Tahirov TH (May 2012). "DNA polymerase δ and ζ switch by sharing accessory subunits of DNA polymerase δ". The Journal of Biological Chemistry. 287 (21): 17281–7. doi:10.1074/jbc.M112.351122. PMC 3366816. PMID 22465957.
- ^ Markkanen E, Castrec B, Villani G, Hübscher U (December 2012). "A switch between DNA polymerases δ and λ promotes error-free bypass of 8-oxo-G lesions". Proceedings of the National Academy of Sciences of the United States of America. 109 (50): 20401–6. Bibcode:2012PNAS..10920401M. doi:10.1073/pnas.1211532109. PMC 3528542. PMID 23175785.
- ^ Song J, Hong P, Liu C, Zhang Y, Wang J, Wang P (2015-01-01). "Human POLD1 modulates cell cycle progression and DNA damage repair". BMC Biochemistry. 16: 14. doi:10.1186/s12858-015-0044-7. PMC 4471906. PMID 26087769.
- ^ Hocke S, Guo Y, Job A, Orth M, Ziesch A, Lauber K, De Toni EN, Gress TM, Herbst A, Göke B, Gallmeier E (February 2016). "A synthetic lethal screen identifies ATR-inhibition as a novel therapeutic approach for POLD1-deficient cancers". Oncotarget. 7 (6): 7080–95. doi:10.18632/oncotarget.6857. PMC 4872770. PMID 26755646.
- ^ Miyabe I, Mizuno K, Keszthelyi A, Daigaku Y, Skouteri M, Mohebi S, Kunkel TA, Murray JM, Carr AM (November 2015). "Polymerase δ replicates both strands after homologous recombination-dependent fork restart". Nature Structural & Molecular Biology. 22 (11): 932–8. doi:10.1038/nsmb.3100. PMC 4655445. PMID 26436826.
- ^ Kamath-Loeb AS, Shen JC, Schmitt MW, Loeb LA (April 2012). "The Werner syndrome exonuclease facilitates DNA degradation and high fidelity DNA polymerization by human DNA polymerase δ". The Journal of Biological Chemistry. 287 (15): 12480–90. doi:10.1074/jbc.M111.332577. PMC 3320997. PMID 22351772.
- ^ Jansen AM, van Wezel T, van den Akker BE, Ventayol Garcia M, Ruano D, Tops CM, Wagner A, Letteboer TG, Gómez-García EB, Devilee P, Wijnen JT, Hes FJ, Morreau H (December 2015). "Combined mismatch repair and POLE/POLD1 defects explain unresolved suspected Lynch syndrome cancers". European Journal of Human Genetics. 24 (7): 1089–1092. doi:10.1038/ejhg.2015.252. PMC 5070903. PMID 26648449.
- ^ a b c d e f Palles C, Cazier JB, Howarth KM, Domingo E, Jones AM, Broderick P, et al. (February 2013). "Germline mutations affecting the proofreading domains of POLE and POLD1 predispose to colorectal adenomas and carcinomas". Nature Genetics. 45 (2): 136–44. doi:10.1038/ng.2503. PMC 3785128. PMID 23263490.
- ^ a b c d Valle L, Hernández-Illán E, Bellido F, Aiza G, Castillejo A, Castillejo MI, et al. (July 2014). "New insights into POLE and POLD1 germline mutations in familial colorectal cancer and polyposis". Human Molecular Genetics. 23 (13): 3506–12. doi:10.1093/hmg/ddu058. PMID 24501277.
- ^ a b c d Bellido F, Pineda M, Aiza G, Valdés-Mas R, Navarro M, Puente DA, Pons T, González S, Iglesias S, Darder E, Piñol V, Soto JL, Valencia A, Blanco I, Urioste M, Brunet J, Lázaro C, Capellá G, Puente XS, Valle L (April 2016). "POLE and POLD1 mutations in 529 kindred with familial colorectal cancer and/or polyposis: review of reported cases and recommendations for genetic testing and surveillance". Genetics in Medicine. 18 (4): 325–32. doi:10.1038/gim.2015.75. PMC 4823640. PMID 26133394.
- ^ a b c d Briggs S, Tomlinson I (June 2013). "Germline and somatic polymerase ε and δ mutations define a new class of hypermutated colorectal and endometrial cancers". The Journal of Pathology. 230 (2): 148–53. doi:10.1002/path.4185. PMC 3709119. PMID 23447401.
- ^ a b c Church DN, Briggs SE, Palles C, Domingo E, Kearsey SJ, Grimes JM, Gorman M, Martin L, Howarth KM, Hodgson SV, Kaur K, Taylor J, Tomlinson IP (July 2013). "DNA polymerase ε and δ exonuclease domain mutations in endometrial cancer". Human Molecular Genetics. 22 (14): 2820–8. doi:10.1093/hmg/ddt131. PMC 3690967. PMID 23528559.
- ^ a b Heitzer E, Tomlinson I (February 2014). "Replicative DNA polymerase mutations in cancer". Current Opinion in Genetics & Development. 24: 107–13. doi:10.1016/j.gde.2013.12.005. PMC 4003352. PMID 24583393.
- ^ a b Shinbrot E, Henninger EE, Weinhold N, Covington KR, Göksenin AY, Schultz N, Chao H, Doddapaneni H, Muzny DM, Gibbs RA, Sander C, Pursell ZF, Wheeler DA (November 2014). "Exonuclease mutations in DNA polymerase epsilon reveal replication strand specific mutation patterns and human origins of replication". Genome Research. 24 (11): 1740–50. doi:10.1101/gr.174789.114. PMC 4216916. PMID 25228659.
- ^ Arora S, Yan H, Cho I, Fan HY, Luo B, Gai X, Bodian DL, Vockley JG, Zhou Y, Handorf EA, Egleston BL, Andrake MD, Nicolas E, Serebriiskii IG, Yen TJ, Hall MJ, Golemis EA, Enders GH (December 2015). "Genetic Variants That Predispose to DNA Double-Strand Breaks in Lymphocytes From a Subset of Patients With Familial Colorectal Carcinomas". Gastroenterology. 149 (7): 1872–1883.e9. doi:10.1053/j.gastro.2015.08.052. PMC 4663158. PMID 26344056.
- ^ Waterfall JJ, Meltzer PS (March 2015). "Avalanching mutations in biallelic mismatch repair deficiency syndrome". Nature Genetics. 47 (3): 194–6. doi:10.1038/ng.3227. PMID 25711864. S2CID 28165945.
- ^ a b Schlesner M, Eils R (2015-01-01). "Hypermutation takes the driver's seat". Genome Medicine. 7 (1): 31. doi:10.1186/s13073-015-0159-x. PMC 4376156. PMID 25821521.
- ^ Shlien A, Campbell BB, de Borja R, Alexandrov LB, Merico D, Wedge D, et al. (March 2015). "Combined hereditary and somatic mutations of replication error repair genes result in rapid onset of ultra-hypermutated cancers". Nature Genetics. 47 (3): 257–62. doi:10.1038/ng.3202. PMID 25642631. S2CID 5338516.
- ^ Bouffet E, Larouche V, Campbell BB, Merico D, de Borja R, Aronson M, et al. (March 2016). "Immune Checkpoint Inhibition for Hypermutant Glioblastoma Multiforme Resulting From Germline Biallelic Mismatch Repair Deficiency". Journal of Clinical Oncology. 34 (19): 2206–2211. doi:10.1200/JCO.2016.66.6552. PMID 27001570.
- ^ Howitt BE, Shukla SA, Sholl LM, Ritterhouse LL, Watkins JC, Rodig S, Stover E, Strickland KC, D'Andrea AD, Wu CJ, Matulonis UA, Konstantinopoulos PA (December 2015). "Association of Polymerase e-Mutated and Microsatellite-Instable Endometrial Cancers With Neoantigen Load, Number of Tumor-Infiltrating Lymphocytes, and Expression of PD-1 and PD-L1" (PDF). JAMA Oncology. 1 (9): 1319–23. doi:10.1001/jamaoncol.2015.2151. PMID 26181000.
- ^ van Gool IC, Eggink FA, Freeman-Mills L, Stelloo E, Marchi E, de Bruyn M, Palles C, Nout RA, de Kroon CD, Osse EM, Klenerman P, Creutzberg CL, Tomlinson IP, Smit VT, Nijman HW, Bosse T, Church DN (July 2015). "POLE Proofreading Mutations Elicit an Antitumor Immune Response in Endometrial Cancer". Clinical Cancer Research. 21 (14): 3347–55. doi:10.1158/1078-0432.CCR-15-0057. PMC 4627582. PMID 25878334.
- ^ Khanna A (June 2015). "DNA damage in cancer therapeutics: a boon or a curse?". Cancer Research. 75 (11): 2133–8. doi:10.1158/0008-5472.CAN-14-3247. PMID 25931285.
- ^ Roberts SA, Gordenin DA (December 2014). "Hypermutation in human cancer genomes: footprints and mechanisms". Nature Reviews. Cancer. 14 (12): 786–800. doi:10.1038/nrc3816. PMC 4280484. PMID 25568919.
- ^ Roos WP, Thomas AD, Kaina B (January 2016). "DNA damage and the balance between survival and death in cancer biology" (PDF). Nature Reviews. Cancer. 16 (1): 20–33. doi:10.1038/nrc.2015.2. PMID 26678314. S2CID 10159855.
- ^ da Costa LT, Liu B, el-Deiry W, Hamilton SR, Kinzler KW, Vogelstein B, Markowitz S, Willson JK, de la Chapelle A, Downey KM (January 1995). "Polymerase delta variants in RER colorectal tumours". Nature Genetics. 9 (1): 10–1. doi:10.1038/ng0195-10. PMID 7704014. S2CID 19545401.
- ^ Flohr T, Dai JC, Büttner J, Popanda O, Hagmüller E, Thielmann HW (March 1999). "Detection of mutations in the DNA polymerase delta gene of human sporadic colorectal cancers and colon cancer cell lines". International Journal of Cancer. 80 (6): 919–29. doi:10.1002/(sici)1097-0215(19990315)80:6<919::aid-ijc19>3.0.co;2-u. PMID 10074927.
- ^ Preston BD, Albertson TM, Herr AJ (October 2010). "DNA replication fidelity and cancer". Seminars in Cancer Biology. 20 (5): 281–93. doi:10.1016/j.semcancer.2010.10.009. PMC 2993855. PMID 20951805.
- ^ Popanda O, Flohr T, Fox G, Thielmann HW (November 1999). "A mutation detected in DNA polymerase delta cDNA from Novikoff hepatoma cells correlates with abnormal catalytic properties of the enzyme". Journal of Cancer Research and Clinical Oncology. 125 (11): 598–608. doi:10.1007/s004320050322. PMID 10541966. S2CID 11582153.
- ^ Venkatesan RN, Treuting PM, Fuller ED, Goldsby RE, Norwood TH, Gooley TA, Ladiges WC, Preston BD, Loeb LA (November 2007). "Mutation at the polymerase active site of mouse DNA polymerase delta increases genomic instability and accelerates tumorigenesis". Molecular and Cellular Biology. 27 (21): 7669–82. doi:10.1128/MCB.00002-07. PMC 2169052. PMID 17785453.
- ^ a b c Pelosini C, Martinelli S, Ceccarini G, Magno S, Barone I, Basolo A, Fierabracci P, Vitti P, Maffei M, Santini F (November 2014). "Identification of a novel mutation in the polymerase delta 1 (POLD1) gene in a lipodystrophic patient affected by mandibular hypoplasia, deafness, progeroid features (MDPL) syndrome". Metabolism. 63 (11): 1385–9. doi:10.1016/j.metabol.2014.07.010. PMID 25131834.
- ^ Reinier F, Zoledziewska M, Hanna D, Smith JD, Valentini M, Zara I, Berutti R, Sanna S, Oppo M, Cusano R, Satta R, Montesu MA, Jones C, Cerimele D, Nickerson DA, Angius A, Cucca F, Cottoni F, Crisponi L (November 2015). "Mandibular hypoplasia, deafness, progeroid features and lipodystrophy (MDPL) syndrome in the context of inherited lipodystrophies". Metabolism. 64 (11): 1530–40. doi:10.1016/j.metabol.2015.07.022. PMID 26350127.
- ^ Oshima J, Sidorova JM, Monnat RJ (March 2016). "Werner syndrome: Clinical features, pathogenesis and potential therapeutic interventions". Ageing Research Reviews. 33: 105–114. doi:10.1016/j.arr.2016.03.002. PMC 5025328. PMID 26993153.
- ^ Lessel D, Hisama FM, Szakszon K, Saha B, Sanjuanelo AB, Salbert BA, Steele PD, Baldwin J, Brown WT, Piussan C, Plauchu H, Szilvássy J, Horkay E, Högel J, Martin GM, Herr AJ, Oshima J, Kubisch C (November 2015). "POLD1 Germline Mutations in Patients Initially Diagnosed with Werner Syndrome". Human Mutation. 36 (11): 1070–9. doi:10.1002/humu.22833. PMC 4684254. PMID 26172944.
- ^ Pachlopnik Schmid J, Lemoine R, Nehme N, Cormier-Daire V, Revy P, Debeurme F, Debré M, Nitschke P, Bole-Feysot C, Legeai-Mallet L, Lim A, de Villartay JP, Picard C, Durandy A, Fischer A, de Saint Basile G (December 2012). "Polymerase ε1 mutation in a human syndrome with facial dysmorphism, immunodeficiency, livedo, and short stature ("FILS syndrome")". The Journal of Experimental Medicine. 209 (13): 2323–30. doi:10.1084/jem.20121303. PMC 3526359. PMID 23230001.
- ^ Thiffault I, Saunders C, Jenkins J, Raje N, Canty K, Sharma M, Grote L, Welsh HI, Farrow E, Twist G, Miller N, Zwick D, Zellmer L, Kingsmore SF, Safina NP (2015-01-01). "A patient with polymerase E1 deficiency (POLE1): clinical features and overlap with DNA breakage/instability syndromes". BMC Medical Genetics. 16: 31. doi:10.1186/s12881-015-0177-y. PMC 4630961. PMID 25948378.
- ^ Guénantin AC, Briand N, Bidault G, Afonso P, Béréziat V, Vatier C, Lascols O, Caron-Debarle M, Capeau J, Vigouroux C (May 2014). "Nuclear envelope-related lipodystrophies". Seminars in Cell & Developmental Biology. 29: 148–57. doi:10.1016/j.semcdb.2013.12.015. PMID 24384368.
- ^ a b "GeneTests.org". GeneTests.org. Retrieved 2016-04-25.
- ^ "MDP syndrome caused by a change in the POLD1 gene".







