진핵 DNA 복제
Eukaryotic DNA replication진핵 DNA 복제는 DNA 복제를 세포 주기당 1회로 제한하는 보존 메커니즘이다.염색체 DNA의 진핵 DNA 복제는 세포의 중복을 위한 중심이며 진핵 게놈의 유지에 필요하다.
DNA 복제는 DNA 중합체가 원래 템플릿 가닥과 보완되는 DNA 가닥을 합성하는 작용이다.DNA를 합성하기 위해 이중 가닥 DNA는 중합체보다 앞서 DNA 나선형으로 분해되어 두 개의 단일 가닥 템플릿이 들어 있는 복제 포크를 형성한다.복제 과정은 하나의 DNA 이중나선을 두 개의 DNA 나선형으로 복사하는 것을 허용하는데, 이것은 유사시 딸세포로 나뉜다.복제포크에서 수행되는 주요 효소 함수는 원핵생물에서 진핵생물까지 잘 보존되어 있지만 진핵 DNA 복제의 복제 기계는 훨씬 더 큰 복합체로서 복제 현장에서 많은 단백질을 조정하여 재생체를 형성한다.[1]
복제는 각각의 증식세포의 유전체 DNA 전체를 복제하는 역할을 한다.이 과정은 부모 세포에서 딸 세포로 유전적/유전적 정보를 고도로 전달하는 것을 허용하며 따라서 모든 유기체에 필수적이다.세포 주기의 대부분은 DNA 복제가 오류 없이 이루어지도록 하는 것에 기초한다.[1]
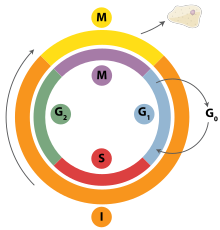
세포주기의 G단계에서는1 많은 DNA 복제 규제 프로세스가 개시된다.진핵생물에서 DNA 합성의 대부분은 세포주기의 S단계에서 발생하며, 전체 게놈을 분해하여 복제하여 두 개의 딸복사를 형성해야 한다.G2 중에 손상된 DNA나 복제 오류를 수정한다.마지막으로, 게놈의 한 부본이 유사분열 또는 M 단계에서 각 딸 세포로 분리된다.[2]이 딸 카피들은 각각 부모의 이중 DNA에서 나온 한 가닥과 초기 대퇴골 가닥 한 가닥을 포함하고 있다.
이 메커니즘은 원핵생물에서 진핵생물까지 보존되며 반원성 DNA 복제라고 알려져 있다.DNA 복제 부위의 반원복제 과정은 포크와 같은 DNA 구조인 복제포크로, DNA 나선이 열려 있거나 분리되지 않은 DNA 뉴클레오티드가 이중 가닥 DNA에 결합하기 위한 인식과 염기쌍을 위해 노출된다.[3]
입문
진핵 DNA 복제의 시작은 DNA 이중나선이 풀리고 DNA 중합효소 α에 의한 초기 프라이밍 이벤트가 선행 스트랜드에서 발생하는 DNA 합성 1단계다.후행 스트랜드의 프라이밍 이벤트는 복제 포크를 설정한다.DNA 나선의 프라이밍은 DNA 중합효소 α에 의한 DNA 합성이 가능하도록 RNA 프라이머의 합성으로 구성된다.프리밍은 선행 가닥의 원점과 후행 가닥의 각 오카자키 파편이 시작될 때 한 번 발생한다.
DNA 복제는 복제의 기원이라고 불리는 특정 시퀀스에서 시작되며 진핵 세포는 복수의 복제 기원을 가지고 있다.DNA 복제를 시작하기 위해, 다중 복제 단백질은 이러한 복제 기원에 결합되어 분리된다.[4]아래에 기술된 개별 요소는 복제 개시 과정의 핵심 매개체인 사전 복제 복합체(pre-RC)의 형성을 지시하기 위해 함께 작용한다.
복제기원을 가진 원산지인식복합체(ORC) 협회는 세포분할주기 6단백질(Cdc6)을 모집해 염색체 허가 및 DNA 복제인자 1단백질(Cdt1)에 의해 촉진된 미니크로모솜 유지(Mcm 2-7) 복합단백질을 적재하는 플랫폼을 형성한다.ORC, Cdc6, Cdt1은 세포주기의 G상1 동안 복제기원을 가진 Mcm2-7 단지의 안정적인 연계를 위해 함께 필요하다.[5]
사전복제복합체
복제의 진핵 기원은 두 개의 양방향 DNA 복제 포크의 조립으로 이어지는 다수의 단백질 복합체의 형성을 제어한다.이러한 이벤트는 복제의 기원에 사전 복제 콤플렉스(pre-RC)의 형성에 의해 시작된다.이 과정은 세포 주기의 G 단계에서1 일어난다.사전RC 형성은 원산지 인식 콤플렉스(ORC), Cdc6 단백질, Cdt1 단백질, 미니크로모솜 유지 단백질(Mcm2-7) 등 많은 복제인자를 순서대로 조립하는 것을 포함한다.[6][7]일단 프리RC가 형성되면, DNA 복제가 시작되기 전에 프리RC를 개시단지로 전환하는 것을 돕는 사이클린 의존키나제2(CDK)와 Dbf4 의존키나제(DDK)의 두 키나제에 의해 복합체 활성화가 촉발된다.이러한 전환은 DNA를 풀고 다중의 진핵 DNA 중합체를 DNA 주위에 축적하기 위한 추가 복제 인자의 순서화된 결합을 포함한다.복제 원점에서 양방향 복제 포크(fork)가 어떻게 구축되는가에 대한 문제의 중심은 ORC가 복제 원점마다 두 개의 정면대면 Mcm2-7 단지를 모집해 사전 복제 콤플렉스를 형성하는 메커니즘이다.[8][9][10]
원산지인식복합체
복제 전 복합체(pre-RC)의 조립의 첫 번째 단계는 복제 원점에 대한 원본 인식 복합체(ORC)의 결합이다.후기 유사분열에서 Cdc6 단백질은 바운드 ORC에 결합한 후 Cdt1-Mcm2-7 복합체를 결합한다.[11]ORC, Cdc6, Cdt1은 모두 6개의 단백질 미니크롬 정비(Mcm 2-7) 복합체를 DNA에 탑재하기 위해 필요하다.ORC는 복제 개시를 위해 DNA에 복제 원점 사이트를 선택하는 6개 서브 유닛 Orc1p-6 단백질 복합체로서, 염색체에 대한 ORC 결합은 세포 주기를 통해 규제된다.[6][12]일반적으로 ORC 서브유닛의 기능과 크기는 여러 진핵 게놈에 걸쳐 보존되며, 차이점은 분리된 DNA 결합 부위라는 것이다.
가장 널리 연구되고 있는 원산지 인식 콤플렉스는 자가복제 시퀀스(ARS)에 결합하는 것으로 알려진 사카로마이오스 세레비시아 또는 효모다.[13]S. 세레비시아 ORC는 특히 복제의 효모 기원 A 및 B1 원소와 상호작용하며, 30개의 기본 쌍의 영역에 걸쳐 있다.[14]이러한 시퀀스에 바인딩하려면 ATP가 필요하다.[6][14]
ARS DNA에 묶인 S. 세레비시아 ORC의 원자 구조가 결정되었다.[14]Orc1, Orc2, Orc3, Orc4 및 Orc5는 A 요소에서 DNA를 구부리는 두 가지 유형의 상호작용(기본 비특정 및 염기특정 교호작용)을 통해 A 요소를 둘러싸는다.5개의 서브유닛은 모두 A 원소의 여러 지점에서 설탕 인산염 백본과 접촉하여 베이스 특이성 없이 팽팽한 그립을 형성한다.Orc1과 Orc2는 A 요소의 작은 홈에 접촉하는 반면, Orc4의 날개 나선 영역은 삽입 나선(IH)을 통해 A 요소의 주요 홈에 있는 불변 Ts의 메틸 그룹에 접촉한다.메타조언에서[14] 이 IH의 부재는 인간 ORC에서의 시퀀스 특수성의 결여를 설명한다.[15][16]또한 ARS DNA는 Orc2, Orc5, Orc6와의 상호작용을 통해 B1 원소에서 구부러진다.[14]ORC에 의한 원점 DNA의 휨은 진화적으로 보존된 것으로 보이며 이는 Mcm2-7 복합 하중 메커니즘에 필요할 수 있음을 시사한다.[14][17]
ORC가 복제 원점에서 DNA에 결합할 때, 그것은 복제 전 복합체의 다른 주요 시작 요소들의 조립을 위한 발판 역할을 한다.[18]세포주기의 G단계1 중 이 사전복제 복합체 어셈블리는 S단계 중 DNA 복제가 활성화되기 전에 필요하다.[19]은유효소 염색체에서 복합체(Orc1)의 최소 일부를 제거하는 것은 은유효소가 완성되기 전에 재생성 이전의 복합체 형성이 제거되도록 하기 위한 포유류 ORC 규제의 일환이다.[20]
Cdc6단백질
세포분할주기 6(Cdc6) 단백질을 원점인식복합체(ORC)에 결합하는 것은 복제 원점에서 사전복제복합체(pre-RC)를 조립하는 데 필수적인 단계다.Cdc6는 ATP 의존적인 방식으로 DNA의 ORC에 바인딩되며, 이는 Orc1 ATPase를 필요로 하는 원산지 바인딩 패턴의 변화를 유도한다.[21]Cdc6는 염색질과 연결하기 위해 ORC를 요구하고 있으며, 차례로 Cdt1-Mcm2-7 헵타머가[11] 염색질에 결합해야 한다.[22]ORC-Cdc6 복합체는 고리 모양의 구조를 형성하며 다른 ATP 의존 단백질 기계와 유사하다.Cdc6의 수준과 활동은 셀 주기 동안 복제의 기원이 활용되는 빈도를 조절한다.
Cdt1단백질
DNA 복제를 위한 염색체 허가에는 염색체 허가 및 DNA 복제인자 1(Cdt1) 단백질이 필요하다.[23][24]S. 세레비시아에서 Cdt1은 Mcm2-7 단일 헥사머의 왼손 오픈링 구조를 안정화하여 Mcm2-7 복합체를 염색체에 한 번에 하나씩 적재하는 것을 용이하게 한다.[11][25][26]Cdt1은 Chromatin에 대한 Mcm 단백질의 연결을 협력적으로 촉진하기 위해 Cdc6의 C 종단부와 연관되어 있는 것으로 나타났다.[27]ORCM(ORC-Cdc6-Cdt1-MCM) 복합체의 극저온 전자파 구조는 Cdt1-CTD가 Mcm6-WHD와 상호 작용한다는 것을 보여준다.[28]메타조안스의 경우, 세포 주기 중의 Cdt1 활성도는 단백질 제미닌과의 연관성에 의해 엄격하게 규제되는데, 둘 다 DNA의 재복제를 막기 위해 S단계 중 Cdt1 활동을 억제하고 편재화 및 후속 단백질 분해를 방지한다.[29]
미니크로모솜유지단백질복합체
미니크로모솜 유지(Mcm) 단백질은 ARS 고유의 방식으로 플라스미드 안정성에 영향을 미치는 S. 세뇌에서 DNA 복제 개시 돌연변이를 위한 유전자 검사에서 이름을 따왔다.[30]mcm2, mcm3, mcm4, mcm5, mc6, mcm7은 mcm2와 mcm5의 틈새로 오픈링 구조를 가진 헥사메리카 콤플렉스를 형성한다.[11]염색질에 mcm 단백질을 조립하려면 원점 인식 콤플렉스(ORC), Cdc6, Cdt1의 조정된 기능이 필요하다.[31]일단 mcm 단백질이 염색체에 탑재되면 ORC와 Cdc6는 후속 DNA 복제를 막지 않고 염색체에서 제거할 수 있다.이러한 관찰은 사전 복제 콤플렉스의 1차적 역할이 맥름 단백질을 올바르게 적재하는 것임을 시사한다.[32]

염색질 위의 mcm 단백질은 두 개의 고리가 약간 기울어지고 비틀어지고 중심을 벗어난 상태에서 머리와 머리 사이의 이중 육각체를 형성하여 두 고리의 인터페이스에서 결합 DNA가 포착되는 중앙 채널에 꼬임(kink)을 만든다.[33][34]각 육각형 Mcm2-7 링은 먼저 환지체의 조립을 위한 비계 역할을 한 다음 환지체의 주성분인 촉매 CMG(Cdc45-MCM-GINS) 헬리코아제의 핵심 역할을 한다.각 Mcm 단백질은 다른 모든 단백질과 높은 연관성이 있지만, 각각의 서브유닛 유형을 구별하는 고유한 시퀀스는 eukaryotes에 걸쳐 보존된다.모든 eukaryote는 정확히 6개의 Mcm 단백질 아날로그를 가지고 있으며 각각은 기존 등급(Mcm2-7) 중 하나에 해당하며, 이는 각 mcm 단백질이 독특하고 중요한 기능을 가지고 있음을 나타낸다.[35][9]
DNA 헬리코아제 활성을 위해서는 미니크로모솜 유지단백질이 필요하다.S상 동안 6개의 Mcm 단백질 중 하나를 불활성화하면 헬리코아제를 재활용할 수 없으며 복제 원점에서 조립해야 한다는 복제 포크의 추가 진행을 방해한다.[36]이 단지는 미니크롬 유지보수 단백질 복합 헬리코아제 활동과 함께 ATP아제 활동도 함께 한다.[37]6개의 Mcm 단백질 중 하나에서 돌연변이가 발생하면 보존된 ATP 결합 부위가 감소하는데, 이는 ATP 가수분해가 Mcm 복합체의 6개 서브유닛 모두를 포함하는 조정된 사건임을 나타낸다.[38]연구는 Mcm 단백질 복합체 내에 ATP 가수분해를 조정하기 위해 함께 기능하는 Mcm 단백질의 특정 촉매 쌍이 있다는 것을 보여주었다.예를 들어 Mcm3는 활성화할 수 있지만 Mcm6는 활성화할 수 없다.맥m2-7 단지의 크라이오-EM 구조로 확인된 이들 연구는 맥m단지가 맥m7 옆에 맥m3, 맥m6 옆에 맥m2, 맥m5 옆에 맥m4가 있는 헥사머임을 시사한다.[11][33]촉매 쌍의 두 구성원은 모두 ATP 결합과 가수분해를 가능하게 하는 순응에 기여하며, 활성 및 비활성 서브유닛의 혼합은 Mcm 단백질 복합체가 전체적으로 ATP 결합과 가수분해를 완료할 수 있는 조정된 ATPase 활동을 생성한다.[39]
미니크로모솜 유지단백질의 핵 국산화 효모세포는 싹이 트는 효모세포에서 조절된다.[40][41]mcm단백질은 세포주기의1 G단계와 S단계에서 핵에 존재하지만2 G단계와 M단계에서는 세포질로 수출된다.세포핵에 들어가려면 완전하고 온전한 6개의 서브유닛 맥 콤플렉스가 필요하다.[42]S. 세레비시아에서 핵 수출은 사이클린 의존적 키나제(CDK) 활동에 의해 촉진된다.크로마틴과 연관된 mcm 단백질은 CDK에 대한 접근성이 떨어져 CDK 수출 기계로부터 보호된다.[43]
개시 콤플렉스
세포주기의 G단계에서는1 복제개시인자, 원산지인식복합체(ORC), Cdc6, Cdt1, mcm(Minicromosome maintenance) 단백질 복합체가 DNA에 순차적으로 결합해 사전복제복합체(pre-RC)를 형성한다.G단계가1 셀주기의 S단위로 전환될 때 S상별 사이클린 의존 단백질키나아제(CDK)와 Cdc7/Dbf4키나제(DDK)는 프리RC를 활성 복제포크로 변환한다.이 변환 중에 사전 RC는 Cdc6의 상실로 분해되어 개시 콤플렉스가 생성된다.mcm 단백질의 결합 외에도 세포분열 주기 45(Cdc45) 단백질이 DNA 복제를 시작하는데 필수적이다.[44][45]연구는 Mcm이 Chromatin에 Cdc45를 로드하는 데 매우 중요하며, Mcm과 Cdc45를 모두 포함하는 이 복합체는 세포 주기의 S상 시작에 형성된다는 것을 보여주었다.[46][47]Cdc45는 세포주기의 G단계1 중 복제 원점에서 사전RC의 성분으로 염색질에 탑재된 mcm단백질 복합체를 대상으로 한다.[48]
Cdc45단백질
세포분할주기 45 (Cdc45) 단백질은 재생전 복합체를 개시 복합체로 전환하는 데 중요한 성분이다.Cdc45 단백질은 시작 전 복제 원점에서 조립되며, 복제가 새카로미세스 세레비시아에서 시작되기 위해 필요하며, 신장하는 동안 필수적인 역할을 한다.따라서 Cdc45는 염색체 DNA 복제의 시작 단계와 연장 단계 모두에서 중심적인 역할을 한다.[49]
Cdc45는 G 후반1 단계에서 시작 후 그리고 셀 주기의 S 단계 동안에 염색질과 연관된다.Cdc45는 mcm5와 물리적으로 연관되어 있으며, mcm유전자과 ORC2유전자의 6개 멤버 중 5개 멤버와 유전적 상호작용을 나타낸다.[50][48]Chromatin에 Cdc45를 탑재하는 것은 DNA 중합효소 α, DNA 중합효소 ε, 복제단백질 A(RPA) 및 세포핵항원(PCNA)을 포함한 다른 다양한 복제단백질을 크로마틴에 탑재하는 데 매우 중요하다.[47][51][52][53]제노푸스 핵이 없는 시스템 내에서 플라스미드 DNA를 풀려면 Cdc45가 필요하다는 것이 입증되었다.[53]Xenopus 핵 없는 시스템은 또한 DNA가 벗겨지고 염색질에 대한 엄격한 RPA 결합이 Cdc45의 존재에서만 발생한다는 것을 보여준다.[47]
Chromatin에 대한 Cdc45의 바인딩은 Clb-Cdc28 키나제 활동뿐만 아니라 기능 Cdc6 및 Mcm2에 따라 달라지는데, 이는 Cdc45가 S상 사이클린 의존적 키나제(CDK) 활성화 후 사전 RC와 연관됨을 시사한다.시기와 CDK 의존성에서 알 수 있듯이, Cdc45를 염색질에 결합하는 것은 DNA 복제의 시작에 대한 약속에 매우 중요하다.S상 동안, Cdc45는 물리적으로 염색질의 Mcm 단백질과 상호작용을 하지만, 염색질과 Cdc45의 분리는 Mcm의 그것보다 더 느리다. 이것은 단백질이 다른 메커니즘에 의해 방출된다는 것을 나타낸다.[35]
진스
6개의 미니크로모솜 유지 단백질과 Cdc45는 복제 포크의 이동과 DNA의 상쇄를 위해 시작과 연장 동안에 필수적이다. GINS는 시작 동안에 복제의 기원에서 Mcm과 Cdc45의 상호작용을 위해 필수적이다. 그리고 다시 복제의 진행에 따라 DNA 복제 포크에서 필수적이다.[54][55]GINS 콤플렉스는 Sld5(Cdc105), Psf1(Cdc101), Psf2(Cdc102), Psf3(Cdc103) 4개의 소단백질로 이루어져 있으며, GINS는 일본어로 '5, 1, 2, 3'을 뜻하는 '고, 이치, 니, 산'을 나타낸다.[56]Cdc45, Mcm2-7, GINS가 함께 환지체의 복제 헬리코아제인 [57]CMG 헬리코아제를 형성한다.Mcm2-7 단지만 해도 헬리케아제 활성도가 약하지만, 강력한 헬리케아제 활성을[59][60] 위해서는 Cdc45와 GINS가 필요하다.
맥름10
Mcm10은 염색체 복제에 필수적이며 DNA 복제 원점에서 비활성 형태로 적재된 2-7 헬리코아제 유지와 상호작용한다.[61] [62] 또한 Mcm10은 촉매 DNA 중합효소 α를 보호하며 복제 포크에서 중합효소를 안정화하도록 돕는다.[63][64]
DDK 및 CDK 키나제스
S상 시작 시, 복제 원점에서 개시 콤플렉스를 형성하기 위해서는 사전 복제 콤플렉스가 두 개의 S상 특정 키나아스에 의해 활성화되어야 한다.하나의 키나제는 Dbf4 의존 키나제(DDK)라고 불리는 Cdc7-Dbf4 키나제(kinase)이고, 다른 하나는 사이클린 의존 키나제(CDK)이다.[65]효모와 Xenopus에 있는 Cdc45의 염색체 결합 분석은 CDK 작용의 다운스트림 사건이 Cdc45를 염색체에 로딩하는 것을 보여주었다.[45][46]Cdc6와 CDK의 연관성과 CDC6의 CDK 의존인인산화 때문에 CDC6가 CDK 액션의 대상으로 추측되어 왔다.Cdc6의 CDK 의존 인산화술은 S상 진입에 필요한 것으로 간주되어 왔다.[66]
DDK, Cdc7의 촉매 서브유닛과 활성제 단백질 Dbf4는 모두 진핵생물에 보존되어 있으며, 셀 주기의 S상 시작에 필요하다.[67][68]DDK와 Cdc7 모두 복제의 염색체 기원에 Cdc45를 로딩하는 데 필요하다.DDK kinase의 바인딩 대상은 mcm 복합체일 가능성이 있는 mcm2이다.[69][67]DDK는 Mcm 콤플렉스를 대상으로 하며, 그 인산화 작용은 Mcm 헬리코아제 활성을 가능하게 한다.[70]
Dpb11, Sld3, Sld2 단백질
Sld3, Sld2, Dpb11은 많은 복제 단백질과 상호작용한다.Sld3와 Cdc45는 G11 단계에서도 복제의 초기 원점에서 사전 RC와 상호 의존적인 방식으로 S 단계의 후기 복제 원점과 연관된 콤플렉스를 형성한다.[71][72]Dpb11과 Sld2는 폴리머아제 ɛ과 상호작용하고 교차링크 실험에서는 Dpb11과 폴리머아제 ɛ이 S 단계에서 상호호환되며 복제 원인과 연관됨을 밝혀냈다.[73][74]
Sld3와 Sld2는 CDK에 의해 인산염화 되어 있어 두 개의 복제 단백질이 Dpb11에 결합할 수 있다.Dpb11은 두 쌍의 BRCA1 C Terminus(BRCT) 도메인을 가지고 있었는데, 이 도메인은 인산염 결합 도메인으로 알려져 있다.[75]BRCT 도메인의 N-단자 쌍은 인산염화된 Sld3에 바인딩되고, C-단자 쌍은 인산염화된 Sld2에 바인딩된다.이 두 상호작용 모두 효모에서 DNA 싹트기의 CDK 의존적 활성화에 필수적이다.[76]
Dpb11은 또한 GINS와 상호작용하며 염색체 DNA 복제의 시작 및 연장 단계에 참여한다.[55][77][78]GINS는 복제 포크에서 발견되는 복제 단백질 중 하나로 Cdc45와 Mcm으로 콤플렉스를 형성한다.
Dpb11, Sld2, Sld3 사이의 이러한 인산화 의존적 상호작용은 DNA 복제의 CDK 의존적 활성화에 필수적이며, 일부 실험에서 교차 링크 시약을 사용함으로써 프리로드 콤플렉스(pre-loading complex, pre-LC)라고 불리는 취약한 복합체를 확인하였다.이 복합체에는 Pol ,, GINS, Sld2, Dpb11이 포함되어 있다.사전 LC는 CDK 의존적 및 DDK 의존적 방식으로 기원과 어떤 연관성보다 먼저 형성되는 것으로 확인되며, CDK 활동은 사전 LC 형성을 통한 DNA 복제의 시작을 규제한다.[79]
길쭉

복제 전 복합체(pre-RC)의 형성은 DNA 복제 개시를 위한 잠재적 장소를 나타낸다.이중 좌초된 DNA를 둘러싸는 미니크로모솜 정비단지와 일치하여, 사전 RC의 형성은 원산지 DNA의 즉각적인 풀림이나 DNA 중합체 모집으로 이어지지 않는다.대신 세포주기의 G 중에1 형성된 사전RC는 세포주기의 G상에서1 S상으로 세포가 전달된 후에야 DNA를 풀고 복제를 시작하도록 활성화된다.[2]
일단 개시 콤플렉스가 형성되고 세포가 S 단계로 넘어가면, 그 콤플렉스는 그 후 다시 복제가 된다.진핵 이식 콤플렉스는 DNA 복제를 조정하는 역할을 한다.선행 및 후행 가닥에 대한 복제는 DNA 중합효소 ε과 DNA 중합효소 Δ에 의해 수행된다.Claspin, And1, 복제 인자 C 클램프 로더 및 포크 보호 콤플렉스를 포함한 많은 재생 인자는 중합효소 기능을 조절하고 Cdc45-Mcm-GINS 콤플렉스에 의한 템플릿 스트랜드의 풀림과 DNA 합성을 조정하는 역할을 한다.DNA가 풀리면 트위스트 수가 줄어든다.이를 보완하기 위해 쓰기 횟수가 증가하여 DNA에 양의 슈퍼코일을 도입한다.이러한 슈퍼코일은 제거되지 않으면 DNA 복제가 중단될 것이다.Topoisomeras는 복제 포크 이전에 이러한 슈퍼코일을 제거하는 책임을 진다.
복제는 각각의 증식세포의 유전체 DNA 전체를 복제하는 역할을 한다.딸나선을 형성하는 염기쌍성과 체인형성반응은 DNA 중합효소에 의해 촉매된다.[80]이러한 효소는 단일 가닥의 DNA를 따라 이동하며, 템플릿 가닥을 "읽고" 적절한 퓨린 핵분열, 아데닌과 구아닌, 피리미딘 핵분열, 티민, 시토신 등의 결합을 허용함으로써 초기 DNA 가닥의 확장을 허용한다.활성 자유 디옥시리보뉴클레오티드는 디옥시리보뉴클레오티드 트리인산염(dNTPs)으로 세포 내에 존재한다.이러한 자유 뉴클레오티드는 마지막으로 통합된 뉴클레오티드의 노출된 3'-히드록실 그룹에 추가된다.이 반응에서, 파이로인산염은 자유 dNTP에서 방출되어 중합반응을 위한 에너지를 생성하고 5'의 모노인산염을 노출시켜 3'의 산소에 공동 결합된다.또한 잘못 삽입된 뉴클레오티드는 정력적으로 유리한 반응으로 올바른 뉴클레오티드로 대체될 수 있다.이 속성은 DNA 복제 중에 발생하는 오류의 적절한 교정 및 수리에 필수적이다.
복제 포크
복제 포크는 선행과 후행으로 알려진 새로 분리된 템플릿 가닥과 이중 좌초된 DNA 사이의 연결점이다.이중 DNA는 대위선이기 때문에 복제포크의 두 새 가닥 사이에 반대 방향으로 DNA 복제가 발생하지만, 모든 DNA 중합체는 새로 합성된 가닥에 대해 5에서 3의 방향으로 DNA를 합성한다.DNA 복제 시 추가적인 조정이 필요하다.두 개의 복제 중합체는 반대 방향으로 DNA를 합성한다.중합효소 ε은 '선도' DNA 가닥에 DNA를 연속적으로 합성하는데, 이는 재생체에 의해 풀리는 DNA와 같은 방향을 가리키고 있다.반대로 중합효소 Δ는 DNA 템플릿 스트랜드의 반대편인 "래깅" 스트랜드에 있는 DNA를 단편적이거나 불연속적인 방식으로 합성한다.
뒤떨어지는 가닥에 있는 DNA 복제 제품의 불연속적인 스트레칭은 오카자키 파편이라고 알려져 있으며 진핵 복제 포크의 길이는 약 100~200 베이스다.뒤처진 가닥은 대개 단일 가닥의 결합 단백질로 코팅된 단일 가닥 DNA가 더 길게 늘어져 있어 2차 구조 형성을 막아 단일 가닥의 템플릿을 안정시키는 데 도움이 된다.진핵생물에서 이러한 단일 가닥 결합 단백질은 복제 단백질 A(RPA)로 알려진 이질 복합체다.[81]
각각의 오카자키 파편에는 RNA 프라이머가 선행되는데, 합성을 하는 동안 다음 오카자키 파편의 행렬에 의해 이 파편이 옮겨진다.RNase H는 DNA를 인식한다.RNA 프라이머를 사용하여 생성되며, 프라이머:템플릿 접합부를 남겨두고 복제된 스트랜드에서 이러한 프리머를 제거하는 역할을 하는 RNA 하이브리드.DNA 중합효소 α는 이러한 부위를 인식하고 프라이머 제거로 남은 틈을 길게 한다.진핵 세포에서는 RNA 프라이머 바로 상류로 올라가는 소량의 DNA 세그먼트도 변위되어 플랩 구조를 만든다.그리고 나서 이 플랩은 내핵에 의해 갈라진다.복제포크에서는 플랩 제거 후 DNA의 간극이 DNA 리가세 I에 의해 봉합되어 새로 합성된 스트랜드의 3'-OH와 5'인산염 사이에 남아 있는 틈새를 수리한다.[82]진핵 오카자키 파편의 비교적 짧은 특성 때문에, 뒤처진 가닥에서 불연속적으로 발생하는 DNA 복제 합성은 선도-스트랜드 합성보다 효율성이 떨어지고 시간이 많이 소요된다.모든 RNA 프라이머를 제거하고 흠집을 고치면 DNA 합성이 완성된다.
리드 스트랜드
DNA 복제 중에, 복식자는 부모의 이중 DNA를 5에서 3 방향으로 두 개의 단일 가닥의 DNA 템플릿 복제 포크로 풀 것이다.선행 Strand는 복제 포크의 이동과 같은 방향으로 복제되고 있는 템플릿 Strand이다.이를 통해 복제포크의 이동과 동일한 방향으로 원래의 가닥을 보완하여 새로 합성된 가닥을 5'에서 3'까지 합성할 수 있다.[83]
일단 RNA 프라이머가 선행 스트랜드의 3의 끝에 프리마아제에 의해 첨가되면, DNA 합성은 중단 없이 선행 스트랜드에 대해 3'-5'의 방향으로 계속될 것이다.DNA 중합효소 ε은 템플릿 스트랜드에 뉴클레오티드를 지속적으로 첨가하므로 선행 스트랜드 합성을 위해서는 단 하나의 프라이머만 필요하며 중단 없는 DNA 중합효소 활성도가 있다.[84]
렉터 스트랜드
뒤처진 가닥의 DNA 복제는 불연속적이다.후행 스트랜드 합성에서는 복제 포크의 반대 방향으로 DNA 중합효소가 이동하려면 복수의 RNA 프라이머를 사용해야 한다.DNA 중합효소는 오카자키 파편이라고 불리는 DNA의 짧은 파편을 합성할 것이며, 이 파편은 프라이머의 3번째 끝에 추가될 것이다.이 파편들은 진핵생물에서 100~400개의 뉴클레오티드 사이에 있을 수 있다.[85]
오카자키 조각 합성이 끝나면 DNA 중합효소 Δ가 이전의 오카자키 조각으로 흘러들어와 RNA 프라이머와 DNA의 작은 부분을 포함하는 그것의 5'자 끝을 치환한다.이렇게 하면 RNA-DNA 단일 가닥 플랩이 생성되는데, 이 플랩은 반드시 쪼개져야 하며, 두 오카자키 파편 사이의 틈은 DNA 리가세 1로 봉해져야 한다.이 과정은 오카자키 단편적 성숙으로 알려져 있으며, 한 메커니즘은 짧은 플랩을 처리하는 반면 다른 프로세스는 긴 플랩을 처리하는 두 가지 방법으로 처리할 수 있다.[86]DNA 중합효소 Δ는 중합에 앞서 DNA나 RNA의 뉴클레오티드를 2~3개까지 대체할 수 있어 Fen1을 위한 짧은 '플랩' 기질을 생성해 한 번에 하나의 뉴클레오티드인 플랩에서 뉴클레오티드를 제거할 수 있다.
이 과정의 주기를 반복함으로써 DNA 중합효소 Δ와 Fen1은 RNA 프라이머의 제거를 조정할 수 있으며, 후행 스트랜드에 DNA의 흠집을 남길 수 있다.[87]이 반복적인 과정은 세포를 엄격하게 규제하고 배설할 필요가 있는 큰 플랩을 생성하지 않기 때문에 셀보다 더 바람직하다는 제안이 나왔다.[88]규제완화 Fen1/DNA 중합효소 Δ 활성의 경우, 셀은 대체 메커니즘을 사용하여 헬리코아제와 누클리스 활동을 모두 갖는 dna2를 이용하여 긴 플랩을 생성, 가공한다.[89]이러한 긴 플랩을 제거하기 위해 Na2의 누클리스 활성이 필요하며 Fen1이 더 짧은 플랩을 처리하도록 한다.전자 현미경 연구는 뒤처진 가닥에 대한 뉴클레오솜 하중이 합성 부위에서 매우 가깝게 발생한다는 것을 보여준다.[90]따라서 오카자키 단편적 성숙은 초기 DNA가 합성된 직후에 일어나는 효율적인 과정이다.
복제 DNA 중합체
두 개의 단일 가닥 DNA 템플릿이 노출되면서 복제 헬리케아제가 부모 DNA 이중화 현상을 푼 후, 부모 게놈의 두 복사본을 생성하기 위해 복제 중합체가 필요하다.DNA 중합효소 함수는 고도로 전문화되어 있으며, 특정 템플릿과 좁은 로컬라이제이션에서 복제가 가능하다.진핵 복제 포크에는 DNA 복제에 기여하는 세 가지 뚜렷한 복제 중합효소 복합체가 있다.폴리머아제 α, 폴리머아제 Δ, 폴리머아제 ε.이 세 가지 중합체는 세포의 생존을 위해 필수적이다.[91]
DNA 중합체는 DNA 합성을 시작하기 위한 프라이머를 필요로 하기 때문에, 중합효소 α(Pol α)는 복제 프리마아제 역할을 한다.폴 α는 RNA 프리마아제와 연관되어 있으며, 이 복합체는 RNA의 짧은 10개 뉴클레오티드 스트레칭이 포함된 프라이머를 합성하여 10개에서 20개의 DNA 베이스로 프리밍 작업을 수행한다.[3]중요한 것은, 이러한 프라이밍 작업은 선도-스트랜드 합성을 시작하기 위한 기원에서의 복제 시작과, 또한 후행 가닥의 각 오카자키 조각의 5' 끝부분에서도 일어난다.
그러나 폴 α는 DNA 복제를 계속할 수 없으며 DNA 합성을 계속하려면 다른 중합효소로 대체해야 한다.[92]폴리머아제 전환은 클램프 로더를 필요로 하며 정상적인 DNA 복제는 세 가지 DNA 중합체의 조정된 작용을 필요로 한다는 것이 입증되었다.프라이밍 합성의 경우 Pol α, 선행 스트랜드 복제의 경우 Pol α, 그리고 지속적으로 로드되는 Pol Δ는 후행 스트랜드 합성의 경우 오카자키 파편을 생성하기 위해 사용된다.[93]
- 폴리머아제 α(극 α):소형 촉매 서브 유닛(PriS)과 대형 비 촉매 서브 유닛(PriL)으로 콤플렉스를 형성한다.[94]첫째, RNA 프라이머의 합성은 DNA 중합효소 알파에 의한 DNA 합성을 가능하게 한다.선두 가닥의 기원과 후행 가닥의 각 오카자키 파편 시작점에 한 번 발생한다.프리 서브유닛은 RNA 프라이머를 합성하는 프리마제 역할을 한다.DNA Pol α는 DNA 뉴클레오티드로 새로 형성된 프라이머를 길게 한다.약 20개의 뉴클레오티드 후 연장은 선두 스트랜드의 폴 ε과 후행 스트랜드의 폴 Δ에 의해 인수된다.[95]
- 폴리머아제 Δ(극 Δ):3'->5' exonuclease 활동.체내에서는 후행 스트랜드와 선행 스트랜드 합성에 모두 관여하는 주요 중합효소다.[96]
- 중합효소 ε (극 ε):3'->5' exonuclease 활동.pol Δ와 높은 관련성이 있으며, 생체내에서는 pol Δ의 오류 확인에 주로 기능한다.[96]
Cdc45-Mcm-GINS 헬리캐아제 복합체
DNA 헬리캐아제와 중합체는 복제 포크에서 밀접하게 접촉해야 한다.합성에 앞서 너무 많이 풀리면 단일 가닥 DNA의 큰 전단이 노출된다.이를 통해 DNA 손상 신호를 활성화하거나 DNA 수리 과정을 유도할 수 있다.이러한 문제를 막기 위해 진핵세포는 복제포크에 앞서 헬리코아제 활동을 조절하도록 고안된 특화된 단백질을 함유하고 있다.또한 이 단백질은 헬리캐아제와 중합체 사이의 물리적 상호작용을 위한 도킹 부지를 제공하므로 이중 언로딩이 DNA 합성과 결합되도록 보장한다.[97]
DNA 중합체가 기능하려면 복선형 DNA 나선은 복제를 위한 단일선 DNA 템플릿 2개를 노출하기 위해 분리해야 한다.DNA 헬리캐이즈는 염색체 복제 시 이중 가닥 DNA를 풀어주는 역할을 한다.진핵 세포의 헬리카아제는 현저하게 복잡하다.[98]헬리코아제의 촉매 코어는 6개의 미니크로모솜 유지(Mcm2-7) 단백질로 구성되어 있으며, 육각 링을 형성하고 있다.DNA에서 떨어져서, Mcm2-7 단백질은 단일 헤테로헥사머를 형성하고 이중 가닥의 DNA 주위에 머리부터 머리까지 이중 육각체로 DNA 복제의 기원에 따라 비활성 형태로 적재된다.[98][99]Mcm 단백질은 복제 기원에 채용되었다가 S단계 동안 유전체 DNA 전체에 재분배되어 복제 포크에 대한 지역화를 나타낸다.[48]
Mcm 단백질의 하중은 세포주기의 G1 중에만 발생할 수 있으며, 그 후 Cdc45 단백질과 GINS 콤플렉스를 모집하여 DNA 복제 포크에서 활성 Cdc45–Mcm–GINS(CMG) 헬리코아제를 형성하여 S단계에서 로드 콤플렉스가 활성화된다.[100][101]DNA 복제를 위해 S단계 전체에 걸쳐 맥엠 활동이 필요하다.[36][102]다양한 규제 요인들이 CMG 헬리코아제를 중심으로 모여 DNA 중합체와 연계하여 진핵 재식물을 형성하는 '복제 진행 콤플렉스'를 생산하는데, 그 구조는 박테리아에 비해 아직 상당히 미흡하게 정의되어 있다.[54][103]
격리된 CMG 헬리코아제와 리디솜 진행 콤플렉스는 단일 Mcm 단백질 링 콤플렉스를 포함하고 있어, 두 복제 시 각 Mcm 단백질 복합 링이 CMG 헬리코아제의 핵심을 형성하며, 시작 과정의 일부로 Mcm 단백질의 로드된 이중 육각체를 두 개의 단일 육각 링으로 나눌 수 있음을 시사한다.각 기원에서 확립된 [54][100]포크DNA를 풀려면 완전한 CMG 콤플렉스가 필요하며, CDC45-Mcm-GINS의 콤플렉스는 진핵세포의 기능적 DNA 헬리코아제다.[104]
Ctf4 및 And1 단백질
CMG 복합체는 Ctf4 및 And1 단백질과의 상호작용을 통해 환지체와 상호작용한다.Ctf4/And1 단백질은 CMG 복합체와 DNA 중합효소 α 모두와 상호작용한다.[105]Ctf4는 중합효소 α 액세서리 계수로 복제 원점에 대한 중합효소 α의 모집에 필요하다.[106]
Mrc1과 Claspin 단백질
Mrc1/클라스핀 단백질은 CMG 복합 헬리코아제 활성과 납-스트랜드를 결합한다.mrc1은 mcm 단백질뿐만 아니라 중합효소 ε과 상호작용한다.[107]헬리코아제와 선행 스트랜드 폴리머아제 사이의 이러한 직접 연결의 중요성은 배양된 인간 세포의 결과로 강조되며, 여기서 효율적인 복제 포크 진행을 위해 Mrc1/Claspin이 필요하다.[108]이 결과들은 효율적인 DNA 복제가 헬리케이스와 리드 스트랜드 합성의 결합을 필요로 한다는 것을 암시한다...
증식세포핵항원
DNA 중합체는 DNA 복제를 지원하기 위한 추가 요소를 필요로 한다.DNA 중합체에는 세미락으로 된 '손' 구조가 있어 중합효소가 DNA에 적재되어 번역을 시작할 수 있다.이 구조는 DNA 중합효소가 단일 가닥 DNA 템플릿을 보유할 수 있게 하고 활성 부위에서 dNTP를 통합하며 새로 형성된 이중 가닥 DNA를 방출할 수 있게 한다.그러나 DNA 중합체의 구조는 템플릿 DNA와 지속적인 안정적인 상호작용을 허용하지 않는다.[1]
중합효소와 템플릿 DNA 사이의 상호작용을 강화하기 위해 DNA 슬라이딩 클램프는 중합효소와 연계하여 복제 중합효소의 공정성을 촉진한다.진핵생물에서 슬라이딩 클램프는 증식 세포핵항원(PCNA)으로 알려진 호모트리머 고리 구조물이다.PCNA 링은 DNA 중합체와 상호작용하는 표면과 극성을 가지고 있으며 DNA 템플릿에 단단히 고정시킨다.DNA 중합체의 PCNA 의존적 안정화는 PCNA가 최대 1,000배까지 중합효소 공정성을 향상시킬 수 있기 때문에 DNA 복제에 큰 영향을 미친다.[109][110]PCNA는 필수적인 공작용제로서 복제포크의 여러 공정을 수용하기 위한 재환형에서 가장 보편적인 상호작용 플랫폼 중 하나라는 구별을 가지고 있어, PCNA는 DNA 중합효소에 대한 규제적 공작용제로도 간주되고 있다.[111]
복제 팩터 C
PCNA는 DNA 템플릿 가닥을 완전히 감싸고 복제 포크의 DNA에 로딩해야 한다.선행 Strand에서 PCNA 로딩은 드물게 진행되는데, 선행 Strand의 DNA 복제는 복제가 종료될 때까지 계속되기 때문이다.단, 후행 가닥에서는 각 오카자키 파편 시작점에 DNA 중합효소 Δ를 지속적으로 탑재할 필요가 있다.오카자키 조각 합성의 이러한 끊임없는 시작은 효율적인 DNA 복제를 위해 PCNA 로딩을 반복적으로 필요로 한다.
PCNA 로딩은 복제 팩터 C(RFC) 콤플렉스에 의해 이루어진다.RFC 단지는 Rfc1, Rfc2, Rfc3, Rfc4, Rfc5의 5개의 ATPAS로 구성되어 있다.[112]RFC는 프라이머-템플릿 접합부를 인식하고 이러한 현장에서 PCNA를 로드한다.[113][114]PCNA 호모트리머는 ATP 가수 분해에 의해 RFC에 의해 개방된 후 중합효소와의 연결을 용이하게 하기 위해 적절한 방향으로 DNA에 적재된다.[115][116]클램프 로더는 또한 DNA에서 PCNA를 추출할 수 있다. 복제가 종료되어야 할 때 필요한 메커니즘이다.[116]
중단된 복제 포크
복제 포크의 DNA 복제는 디옥시뉴클레오티드 트리인산염(dNTP) 부족이나 DNA 손상으로 인해 중단되어 복제 스트레스를 유발할 수 있다.[117]이러한 복제 중지는 중단된 복제 포크로 설명된다.단백질의 포크 보호 복합체는 DNA 손상이나 다른 복제 문제가 해결될 때까지 복제 포크를 안정화시킨다.[117]복제 포크 지연이 길어지면 DNA 손상이 더 커질 수 있다.복제 포크의 원인이 되는 문제가 해결되면 정지 신호는 비활성화된다.[117]
종료
진핵 DNA 복제의 종식은 염색체가 원형인지 선형인지에 따라 다른 과정을 필요로 한다.선형 분자와 달리 원형 염색체는 분자 전체를 복제할 수 있다.그러나 두 개의 DNA 분자는 서로 연결되어 있을 것이다.이 문제는 타입 II topoisomerase에 의한 두 개의 DNA 분자의 디카틴화에 의해 처리된다.또한 타입 II topoisomeras는 세포 내의 뉴클레오솜으로 복잡하게 접히면서 선형 가닥을 분리하는데 사용된다.
앞서 언급했듯이, 선형 염색체는 원형 DNA 복제에서 볼 수 없는 또 다른 문제에 직면한다.DNA 합성 개시를 위해서는 RNA 프라이머가 필요하기 때문에, 뒤처진 가닥은 전체 염색체를 복제하는 데 있어서 불리하다.선두 스트랜드가 하나의 RNA 프라이머를 사용하여 복제 DNA 스트랜드의 5' 종말을 연장할 수 있지만, 복수의 RNA 프라이머가 후행 스트랜드 합성을 담당하여 오카자키 파편을 생성한다.이것은 DNA 중합효소가 오직 DNA 가닥의 3의 끝에만 첨가될 수 있기 때문에 문제로 이어진다.3'-5'의 모 가닥을 따라 작용하는 DNA 중합효소는 오카자키 파편이 수리되었을 때 모 가닥의 3의 끝 부분에 짧은 단일 가닥 DNA(ssDNA) 영역을 남긴다.모 염색체의 반대쪽 끝에서 반대 방향으로 복제가 일어나기 때문에 각 가닥은 한쪽 끝에서 뒤처지는 가닥이다.시간이 지남에 따라 이것은 두 딸 염색체의 점진적인 단축을 초래할 것이다.이것은 최종 복제 문제로 알려져 있다.[1]
최종 복제 문제는 말단소세포 지역과 말단소세포에 의해 처리된다.텔로미어는 부모 염색체의 3의 끝을 딸 가닥의 5의 끝 이상으로 확장시킨다.이 단일 가닥의 DNA 구조는 텔로머레이즈를 채용하는 복제의 기원으로 작용할 수 있다.텔로머라아제는 다수의 단백질 서브유닛과 RNA 성분으로 구성된 전문 DNA 중합효소다.텔로머아제 안네의 RNA 성분은 템플릿 DNA의 단일한 좌초된 3의 끝에 있으며, 1.5개의 텔로머릭 시퀀스를 포함하고 있다.[85]텔로머레이즈는 텔로머레이즈 역분해효소 또는 TERT라고 불리는 역분해효소인 단백질 서브단위를 함유하고 있으며, TERT는 텔로머레이즈 RNA 템플릿이 끝날 때까지 DNA를 합성해 분리한다.[85]이 과정은 부모의 DNA 분자의 3의 끝의 연장선으로 필요한 만큼 반복될 수 있다.이 3' 추가는 지연된 가닥 DNA 합성에 의한 딸 가닥의 5' 끝의 확장을 위한 템플릿을 제공한다.텔로머레이즈 활성의 조절은 텔로메레 결합 단백질에 의해 처리된다.
복제 포크 장벽
원핵 DNA 복제는 양방향이다. 복제 원점 내에서 복제 원점의 각 끝에 복식 콤플렉스가 생성되고 복식 콤플렉스는 초기 시작점에서 서로 멀어진다.원핵생물에서 양방향 복제는 원형 염색체에서 하나의 복제 원점에서 시작하여 원점의 초기 시작부터 반대되는 사이트에서 종료된다.[118]이러한 종단 부위는 Ter 사이트라고 알려진 DNA 서열을 가지고 있다.이 테르 사이트들은 투스 단백질로 묶여 있다.Ter-Tus 콤플렉스는 헬리케아제 활동을 중지할 수 있어 복제가 종료된다.[119]
진핵 세포에서 복제의 종식은 보통 두 개의 활성 복제 기원 사이에 두 개의 복제 포크가 충돌하여 발생한다.충돌 위치는 발화 시점에 따라 다르다.이런 식으로 복제포크가 특정 사이트에서 정체되거나 붕괴될 경우 반대 방향으로 이동하는 환지기가 지역 복사를 완료했을 때 사이트 복제를 구할 수 있다.게놈 전체에는 RFB 단백질로 묶인 프로그램된 복제 포크 장벽(RFB)이 있어 복제 포크(RFB)를 종료하거나 일시 중지할 수 있어 재생기의 진행을 멈출 수 있다.[118]
복제 공장
복제는 세포핵에서 국소화된 방식으로 일어나는 것으로 밝혀졌다.정체된 DNA를 따라 복제 포크를 이동시키는 기존 관점과 달리 복제 공장의 개념이 등장했는데, 복제 포크는 템플릿 DNA 가닥이 컨베이어 벨트처럼 지나가는 일부 고정된 '공장' 지역으로 집중된다는 뜻이다.[120]
세포주기조절
DNA 복제는 세포 주기의 맥락 안에서 제어되는 긴밀하게 조정된 과정이다.세포주기와 DNA 복제를 통한 진행은 사이클린 의존형 키나제(Cdks, CDKs)의 활성화와 비활성화를 통해 달성되는 사전복제복합체(사전RC)의 형성과 활성화에 의해 엄격하게 규제된다.특히1 G상에서 S상으로의 전환을 책임지는 것은 사이클린과 사이클린 의존 키나제의 상호작용이다.
셀 주기의 G1 페이즈 동안 CDK 활동의 낮은 레벨이 있다.이러한 낮은 수준의 CDK 활동은 새로운 사전 RC 단지의 형성을 허용하지만 새롭게 형성된 사전 RC에 의해 DNA 복제가 시작되기에 충분하지 않다.셀 주기의 나머지 단계 중에는 CDK 활동의 레벨이 상승한다.이 높은 수준의 CDK 활동은 DNA 복제를 시작할 뿐만 아니라 새로운 사전 RC 복합체 형성을 억제하는 역할을 한다.[2]일단 DNA 복제가 시작되면 사전 RC 콤플렉스는 분해된다.셀 사이클의 S상, G상2, M상 동안 CDK 레벨이 높게 유지된다는 사실 때문에 새로운 사전RC 콤플렉스는 형성될 수 없다.이 모든 것은 세포분열이 완료될 때까지 어떤 개시도 일어나지 않도록 하는 데 도움이 된다.
사이클린 의존적 키네즈 외에도 새로운 복제 라운드가 Cdt1의 다운규정을 통해 방지되는 것으로 생각된다.이것은 Cdt1의 분해뿐만 아니라 게미닌이라고 알려진 단백질의 억제 작용을 통해 달성된다.제미닌은 Cdt1에 단단히 묶여 있으며, 재복제의 주요 억제제라고 생각된다.[2]게미닌은 S상(S phase)에 처음 등장하며, 은유-아나파제 전환 시, 아마도 아나파제 추진 복합체(APC)에 의한 유비쿼터스화를 통해 퇴화된다.[121]
셀이 완전히 분열을 통해 진행될지 여부를 결정하는 셀 사이클 과정 내내 다양한 셀 사이클 체크포인트가 존재한다.중요하게도 복제하는 데-G1, 또는 제한, 검문소인지 아닌지 복제 개시 여부를 세포는 휴식 단계 G0으로 알려져에 배치될 것이다 시작할 것을 결정하게 만든다.때문에 꼬마 염색체 유지 단백질은 세포 주기의 G0 무대의 세포들은 복제의 둥근 시작할 수 없습니다.표현되지 않은S상으로의 전환은 복제가 시작되었음을 나타낸다.
복제 체크포인트 단백질
세포분열 중 유전정보를 보존하기 위해서는 DNA 복제가 높은 충실도로 완료되어야 한다.이 과제를 달성하기 위해 진핵세포는 DNA 복제 과정에서 어떤 오류도 감지할 수 있고 게놈의 무결성을 보존할 수 있는 복제 과정의 특정 지점 동안 단백질을 가지고 있다.이러한 체크포인트 단백질은 DNA 수리를 위한 시간을 허용하기 위해 세포 주기가 유사분열로 들어가는 것을 막을 수 있다.체크포인트 단백질은 복제포크의 구조를 안정화시켜 추가 손상을 방지하는 동시에 일부 DNA 복구 경로에도 관여한다.이러한 체크포인트 단백질은 돌연변이나 다른 염색체 이상을 자손에게 전하지 않기 위해 필수적이다.
진핵 체크포인트 단백질은 잘 보존되어 있고 두 개의 인산염리노시톨 3-키나제 관련 키나제(PIKKs), ATR, ATM을 포함한다.ATR과 ATM 모두 표적 인산화 시퀀스인 SQ/TQ 모티브를 공유하지만 세포에서 각각의 역할은 다르다.[122]
ATR은 DNA 이중 가닥 균열에 대응하여 세포 주기를 잡는 데 관여한다.ATR은 필수 체크포인트 파트너인 ATR-인터랙션-단백질(ATRIP)을 가지고 있으며, 이 두 단백질은 복제 단백질 A(RPA)에 의해 코팅된 단일 변형 DNA의 증설에 반응한다.[123]단일 가닥의 DNA 형성은 복제 스트레스 동안 더 자주 발생한다.ATR-ATRIP은 게놈 무결성을 보존하기 위해 세포 주기를 막을 수 있다.ATR은 S상 중 염색체에서 발견되는데, RPA 및 쇄신과 유사하다.[124]
단일 가닥의 DNA 트레이의 생성은 복제 손상의 다운스트림 체크포인트 경로를 시작하는 데 중요하다.단일 가닥의 DNA가 충분히 길어지면 RPA로 코팅된 단일 가닥의 DNA는 ATR-ATRIP을 모집할 수 있다.[125]ATR 키나아제는 완전히 활성화되기 위해 체크포인트 단백질이 DNA 복제 응력의 유효한 부위에 국부화되는지 여부를 감지하는 센서 단백질에 의존한다.RAD9-HUS1-Rad1 (9-1-1) 이형성 클램프와 그 클램프 로더 RFC는Rad17 걸프하거나 흠집이 난 DNA를 인식할 수 있다.RFCRad17 클램프 로더는 손상된 DNA에 9-1-1을 적재한다.[126]DNA에 9-1-1이 존재하면 TOPBP1과 Mrc1/claspin과 같이 ATR-ATRIP와 체크포인트 매개체라고 불리는 단백질 그룹 간의 상호작용을 용이하게 할 수 있다.TOPBP1은 9-1-1의 인산화 Rad9 성분과 상호 작용하여 채용하며 인산화 Chk1인 ATR-ATRIP을 결합한다.[127]또한 Mrc1/Claspin은 주요 다운스트림 체크포인트 이펙터 키나제인 Chk1을 인산화하여 ATR-ATRIP의 완전한 활성화를 위해 필요하다.[128]클라스핀은 재생체의 구성요소로서 Chk1과 도킹하기 위한 도메인을 포함하고 있어 DNA 복제 중 클라스핀의 특정 기능인 즉, 재생기에서 체크포인트 신호의 추진이 드러난다.[129]
Chk1 신호는 세포 주기를 고정시키고 불완전한 DNA 복제나 DNA 손상으로 세포가 유사분열로 들어가는 것을 막는데 필수적이다.Chk1 의존성 Cdk 억제는 ATR-Chk1 체크포인트의 기능과 세포 주기를 구속하고 DNA 수리 메커니즘의 완성을 위한 충분한 시간을 허용하는 데 중요하며, 이는 결국 손상된 DNA의 상속을 방지한다.또한 Chk1 의존성 Cdk 억제는 S상 중 원점 사격을 억제하는 데 중요한 역할을 한다.이 메커니즘은 지속적인 DNA 합성을 방지하며 복제 스트레스와 잠재적인 유전독성 조건의 존재에서 게놈의 보호를 위해 필요하다.[130]따라서 ATR-Chk1 활동은 셀 주기 구속을 유지하는 신호 캐스케이드가 꺼질 때까지 게놈 전체에 걸친 복제 시작을 금지함으로써 단일 복제 기원 수준에서 잠재적인 복제 문제를 더욱 방지한다.
뉴클레오솜을 통한 복제
진핵 DNA는 핵의 제한된 공간에 맞도록 단단히 압축되어야 한다.염색체는 히스톤 단백질의 옥타머에 147개의 뉴클레오티드를 감싸 핵물질을 형성함으로써 포장된다.뉴클레오솜 옥타머는 각 히스톤 H2A, H2B, H3 및 H4의 2개의 복사본을 포함한다.히스톤 단백질의 DNA와의 긴밀한 연관성 때문에 진핵 세포는 재생체의 원활한 진행을 위해 복제 포크보다 앞서 히스톤을 개조하도록 설계된 단백질을 갖고 있다.[131]또한 복제 포크 뒤에 있는 히스톤을 재조립하는 데 관여하는 단백질도 있다.[132]
복제 후 뉴클레오솜 조립에 관여하는 것으로 알려진 히스톤 샤페론이 여러 개 있다.팩트 콤플렉스는 DNA 중합효소 α-primase 복합체와 상호작용하는 것으로 밝혀졌으며, 팩트 콤플렉스의 서브유닛은 복제 인자와 유전적으로 상호작용했다.[133][134]팩트 콤플렉스는 ATP를 가수 분해하지 않고 뉴클레오솜에서 히스톤의 '루즈닝'을 용이하게 할 수 있는 헤테로디메이터지만, 어떻게 팩트 콤플렉스가 DNA 제거를 위한 히스톤의 긴밀한 연관성을 해소할 수 있는지는 여전히 풀리지 않고 있다.[135]
는 replisome과 교제하고 또 다른 히스톤 보호자는 Asf1는 히스톤 dimers에 H3-H4.[136]Asf1은 복제 분기점 뒤에 침전 요인과 이러한 활동은 H3-H4 히스톤 dimers 히스톤 침전의 사이트만 afte에 가능하도록 한다에 새로운 합성 H3-H4 2합체에 합격할 수 있는 Mcm 복잡한 피 부양자와 상호 작용합니다.r렙리케이션[137]또한 Asf1(및 그 파트너 Rtt109)은 S상 동안 복제된 유전자로부터 유전자 발현을 억제하는 일에 연루되어 왔다.[138]
헤테로테리릭 샤페론 크로마틴 조립 인자 1(CAF-1)은 새로 복제된 양쪽 DNA 가닥에 히스톤을 침전시켜 염색질을 형성하는 데 관여하는 염색체 형성 단백질이다.[139]CAF-1은 PIP-box라고 불리는 PCNA 결합 모티브를 포함하고 있어 CAF-1이 PCNA를 통해 환지체와 연결될 수 있고 히스톤 H3-H4 다이머를 새로 합성된 DNA에 저장할 수 있다.[140][141]Rtt106 샤페론도 이 과정에 관여하며, 염색질 형성 중 CAF-1 및 H3-H4 다이머와 관련된다.[142]이 과정들은 새로 합성된 히스톤을 DNA에 로딩한다.
히스톤 H3-H4의 퇴적 후, 핵소체는 히스톤 H2A-H2B의 연관성에 의해 형성된다.이 과정은 PRAT 콤플렉스를 통해 발생한다고 생각되는데, 이는 이미 환지체와 연관되어 있고 무료 H2A-H2B를 바인딩할 수 있거나, 또 다른 H2A-H2B 샤페론인 Nap1의 가능성이 있기 때문이다.[143]전자 현미경 연구는 복제 포크 이후 핵소체가 단지 수백 개의 염기쌍을 형성하는 것을 관찰할 수 있기 때문에 이러한 현상이 매우 빨리 일어난다는 것을 보여준다.[144]따라서 새로운 뉴클레오솜을 형성하는 전 과정은 히스톤 샤페론의 결합으로 인해 복제 직후에 일어난다.
원핵과 진핵 DNA 복제의 비교
원핵 DNA 복제와 비교할 때 진핵 DNA 복제의 완료는 더 복잡하고 복제 단백질과 복제 단백질의 다중 기원을 포함한다.원핵 DNA는 원형 형태로 배열되며, 복제가 시작될 때 복제 원점이 하나만 있다.대조적으로 진핵 DNA는 선형이다.복제할 때 복제의 기원은 무려 천 개에 이른다.[145]
진핵 DNA는 양방향이다.여기서 양방향이라는 말의 의미는 다르다.진핵 선형 DNA는 많은 기원을 가지고 있다(O라고 불림), 종말(T라고 함)."T"는 "O"의 오른쪽에 있다.한 개의 "O"와 한 개의 "T"가 함께 하나의 리디콘을 형성한다.사전 개시 콤플렉스가 형성된 후 한 개의 리디콘이 신장하기 시작하면 두 번째 리디콘에서 개시가 시작된다.이제, 첫 번째 리디콘이 시계방향으로 움직이면, 두 번째 리디콘은 반시계방향으로 움직인다. 첫 번째 리디콘의 "T"에 도달할 때까지."T"에서는 두 복제본이 병합되어 복제 과정을 완료한다.한편 제2환지 역시 제3환지대와 만나기 위해 전진하고 있다.두 복제자의 이러한 시계방향 및 시계 반대방향 이동을 양방향 복제라고 한다.
진핵 DNA 복제는 세포가 분열할 때마다 전체 게놈을 복제하기 위해 모든 DNA 중합체와 관련 단백질의 정밀한 조율을 필요로 한다.이 과정은 주로 DNA와 MCM 헬리코아제의 연관성에 DNA 복제의 규제를 집중하면서 복제의 기원에 있는 일련의 단백질 조립 단계를 통해 달성된다.이러한 복제의 기원은 복제를 시작하기 위해 형성될 단백질 복합체의 수를 지시한다.원핵생물 DNA 복제 규정에서 DNA에 대한 DNA 이니시에이터 단백질의 결합에 초점을 맞추고 있으며, 한 세포 주기 동안 복제가 여러 번 시작된다.[85]원핵 및 진핵 DNA 모두 ATP 결합과 가수분해를 사용해 헬리코아제 하중을 직접 전달하며, 두 경우 모두 헬리코아제가 비활성 형태로 로드된다.그러나 진핵 헬리코아제는 이중으로 좌초된 DNA에 적재되는 이중 육각체인 반면 원핵 헬리코아제는 단일 좌초된 DNA에 적재된 단일 육각체다.[146]
염색체 분리는 원핵세포와 진핵세포의 또 다른 차이점이다.박테리아와 같이 빠르게 분열하는 세포는 종종 복제 과정에 있는 염색체를 분리하기 시작할 것이다.진핵세포에서 염색체를 딸세포로 분리하는 것은 모든 염색체에서 복제가 완료될 때까지 시작되지 않는다.[85]그러나 이러한 차이에도 불구하고 복제의 기본 과정은 친핵과 진핵 DNA 모두에서 유사하다.
| 원핵 DNA 복제 | 진핵 DNA 복제 |
|---|---|
| 세포질 내부에서 발생한다. | 핵 내부에서 발생한다. |
| DNA 분자당 복제의 출처는 단 하나 | 각 염색체마다 복제의 기원이 많다. |
| 복제의 기원은 약 100~200개 이상의 뉴클레오티드 길이 | 복제의 각 기원은 약 150개의 뉴클레오티드로 구성된다. |
| 각 염색체의 한 지점에서 복제가 발생함 | 각 염색체의 여러 지점에서 동시에 복제 발생 |
| 복제 원점이 하나만 있음 | 복제 원본을 여러 개 포함 |
| 시작은 단백질 DNAA와 DNAB에 의해 수행된다. | 오리진 인식 콤플렉스에 의해 개시 수행 |
| Topoisomerase 필요 | Topoisomerase 필요 |
| 복제 속도가 매우 빠름 | 복제 속도가 매우 느림 |
진핵 DNA 복제 단백질 목록
진핵 DNA 복제에 관련된 주요 단백질 목록:
| 단백질 | 진핵 DNA 복제의 기능 |
|---|---|
| AND1 | DNA 중합효소 α를 후행 가닥의 CMG 복합체와 함께 염색질에 적재한다.싹이 돋아나는 효모 Ctf4로도 알려져 있다. |
| Cdc45 | DNA 복제의 시작 및 연장 단계에 필요함.Mcm2-7 헬리카아제 복합체의 일부.시작과 연장을 위한 다양한 단백질의 하중을 위한 사전 RC 단계 이후 필요. |
| Cdc45-Mcm-GINS(CMG) 복합체 | 진핵세포 내 기능 DNA 헬리코아제 |
| Cdc6 | ORC에서 Cdt1과 함께 Mcm2-7 복합체 조립에 필요. |
| Cdc7-Dbf4 kinase 또는 Dbf4 의존 kinase(DDK) | DNA 복제를 시작하기 위해 필요한 단백질 키나아제는 아마도 미니크로모솜 유지 단백질의 인산화를 통해서입니다. |
| Cdt1 | 사전 RC/라이선싱 단계에서 ORC의 DNA에 Mcm2-7 복합체를 적재한다.제미닌에 의한 메타조엔 억제. |
| 클라스핀 | CMG 복합 헬리캐아제 활동과 결합된 선도-스트랜드 합성.Mrc1과 함께 작업 |
| Ctf4 | DNA 중합효소 α를 후행 가닥의 CMG 복합체와 함께 염색질에 적재한다.메타조인에서 호몰로그는 AND-1로 알려져 있다. |
| CDK(Cyclin-dependent kinase) | 복제 시작 및 기타 후속 단계에 필요한 사이클린 의존성 단백질 키나아제. |
| dna2 | 오카자키 파편 처리에 관여하는 5' 플랩 엔도누클리스와 헬리코아제. |
| DNA 리가아제 I | DNA 복제 중에 오카자키 파편과 결합한다.리가아제 활동도 DNA 수리와 재조합에 필요했다. |
| DNA 중합효소 α(극 α) | 선행 및 후행 Strand에 대한 DNA 합성을 시작하는 데 필요한 primase 활성도를 포함한다. |
| DNA 중합효소 Δ (극 Δ) | DNA 중합효소 α에 의해 시작된 후행 스트랜드에 오카자키 파편 합성을 완료하는 데 필요. |
| DNA 중합효소 ε (극 ε) | 선두 스트랜드 폴리머아제.복제 포크에서 DNA 합성Dbp11을 통해 초기 기원에서 결합되며 DNA 중합효소 α 로딩에 필요. |
| Dpb11 | DNA 복제 시작 단백질.DNA 중합효소 ε을 기원 전 복제 복합체에 적재한다. |
| 펜1 | 오카자키 파편 처리에 관여하는 5' 플랩 엔도뉴클레아제. |
| 제미닌 | 메타조아에서 발견되고 효모에는 없는 단백질.Cdt1에 바인딩 및 비활성화하여 사전 복제/초기화 복합 형성을 규제한다.또한 바인딩에 의한 사전 RC 형성을 촉진하여 Cdt1 열화를 방지할 것을 제안한다. |
| 진스 | Sld5, Psf1, Psf2, Psf3으로 구성된 4중 복합체.시작 시점에 사전 복제 콤플렉스와 연관되고 연장 단계 중에 복제 포크와 함께 이동한다.DNA 복제의 연장 단계 및 Mcm 헬리코아제 복합체의 일부에 필요. |
| 미니크로모솜 유지단백질(Mcm) | AAA+ ATPase 계열의 6개의 서로 다른 단백질로 용액에서 헥사머를 형성한다.이 헥사머는 ORC, Cdc6, Cdt1에 의해 모집되어 로딩되며 DNA를 중심으로 국소적으로 연결된 이중 헥사머를 형성하여 염분 내성 사전복제 복합체를 형성한다.복제를 시작할 때 Mcm2-7은 복제 포크를 사용하여 ORC에서 이동한다. |
| 맥름10 | DNA 복제의 시작 및 연장 단계에 필요함.Cdc45와 DNA 중합효소 α의 염색질 결합에 관여한다.또한 신생 효모 S. 세레비시아에 있는 DNA 중합효소 α 촉매 서브유닛의 안정성에도 필요하다. |
| mrc1 | CMG 복합 헬리캐아제 활동과 결합된 선도-스트랜드 합성.Metazoan homologo는 Claspin으로 알려져 있다. |
| 오리진 인식 콤플렉스(ORC) | Orc1–Orc6 단백질로 구성된 헤테로헥사메리카 복합체.DNA에 바인딩하고, Cdc6, Cdt1과 함께 Chromatin에 Mcm2-7 콤플렉스를 조립한다. |
| 증식세포핵항원(PCNA) | 고리 모양의 구조를 가진 트리머 단백질은 DNA 중합효소의 분리를 막는 DNA를 감싸고 있다.중합체 Δ 및 ε의 슬라이딩 클램프 역할을 하여 복제 중합체의 공정성을 개선한다. |
| 복제 팩터 C(RFC) | PCNA를 프라이밍된 템플릿에 적재하고 DNA 중합효소 a와 복제 중합효소 Δ와 ε 사이의 전환에 관여한다. |
| 복제 포크 장벽(RFB) | 게놈 전체에 걸쳐 다양한 위치에서 RFB 단백질에 의해 결합된다.복제 포크를 종료하거나 일시 중지할 수 있으며, 복제가 진행되지 않음. |
| 복제 단백질 A(RPA) | 이질성 단일 가닥 결합 단백질.복제 포크에서 단일 가닥 DNA 안정화 |
| R나세 H | RNA를 DNA에 혼합하여 소화하는 리보누클레이저.오카자키 단편 가공에 관여했다. |
| Sld2 | 복제 시작 시 함수.CDK의 핵심 기질인 인산화 작용은 Dpb11과의 상호작용을 촉진한다.복제를 시작하는 데 필요함. |
| Sld3 | 복제 시작 시 함수.CDK의 핵심 기질인 인산화 작용은 Dpb11과의 상호작용을 촉진한다.복제를 시작하는 데 필요함. |
| 텔로머레이즈 | DNA 서열을 첨가한 리보핵단백질 "TTAGGG"는 텔로미어의 DNA 가닥의 3의 끝에 반복된다. |
| 토포아소메라즈 | DNA의 오버 바인딩 또는 언더인딩 조절 |
참고 항목
참조
- ^ a b c d Leman AR, Noguchi E (March 2013). "The replication fork: understanding the eukaryotic replication machinery and the challenges to genome duplication". Genes. 4 (1): 1–32. doi:10.3390/genes4010001. PMC 3627427. PMID 23599899.
- ^ a b c d Blow JJ, Dutta A (June 2005). "Preventing re-replication of chromosomal DNA". Nature Reviews Molecular Cell Biology. 6 (6): 476–86. doi:10.1038/nrm1663. PMC 2688777. PMID 15928711.
- ^ a b Fisher PA, Wang TS, Korn D (July 1979). "Enzymological characterization of DNA polymerase alpha. Basic catalytic properties processivity, and gap utilization of the homogeneous enzyme from human KB cells". The Journal of Biological Chemistry. 254 (13): 6128–37. doi:10.1016/S0021-9258(18)50528-7. PMID 447699.
- ^ Araki H (2011). "Initiation of chromosomal DNA replication in eukaryotic cells; contribution of yeast genetics to the elucidation". Genes & Genetic Systems. 86 (3): 141–9. doi:10.1266/ggs.86.141. PMID 21952204.
- ^ Maiorano D, Moreau J, Méchali M (April 2000). "XCDT1 is required for the assembly of pre-replicative complexes in Xenopus laevis". Nature. 404 (6778): 622–5. Bibcode:2000Natur.404..622M. doi:10.1038/35007104. PMID 10766247. S2CID 4416138.
- ^ a b c Bell SP, Dutta A (2002). "DNA replication in eukaryotic cells". Annual Review of Biochemistry. 71: 333–74. doi:10.1146/annurev.biochem.71.110601.135425. PMID 12045100.
- ^ Tye BK (1999). "MCM proteins in DNA replication". Annual Review of Biochemistry. 68 (1): 649–86. doi:10.1146/annurev.biochem.68.1.649. PMID 10872463.
- ^ Ticau S, Friedman LJ, Ivica NA, Gelles J, Bell SP (April 2015). "Single-molecule studies of origin licensing reveal mechanisms ensuring bidirectional helicase loading". Cell. 161 (3): 513–525. doi:10.1016/j.cell.2015.03.012. PMC 4445235. PMID 25892223.
- ^ a b Zhai Y, Li N, Jiang H, Huang X, Gao N, Tye BK (July 2017). "Unique Roles of the Non-identical MCM Subunits in DNA Replication Licensing". Molecular Cell. 67 (2): 168–179. doi:10.1016/j.molcel.2017.06.016. PMID 28732205.
- ^ Coster G, Diffley JF (July 2017). "Bidirectional eukaryotic DNA replication is established by quasi-symmetrical helicase loading". Science. 357 (6348): 314–318. Bibcode:2017Sci...357..314C. doi:10.1126/science.aan0063. PMC 5608077. PMID 28729513.
- ^ a b c d e Zhai Y, Cheng E, Wu H, Li N, Yung PY, Gao N, Tye BK (March 2017). "Open-ringed structure of the Cdt1-Mcm2-7 complex as a precursor of the MCM double hexamer". Nature Structural & Molecular Biology. 24 (3): 300–308. doi:10.1038/nsmb.3374. PMID 28191894. S2CID 3929807.
- ^ Bell SP (March 2002). "The origin recognition complex: from simple origins to complex functions". Genes & Development. 16 (6): 659–72. doi:10.1101/gad.969602. PMID 11914271.
- ^ Bell, S. P.; Stillman, B. (1992-05-14). "ATP-dependent recognition of eukaryotic origins of DNA replication by a multiprotein complex". Nature. 357 (6374): 128–134. Bibcode:1992Natur.357..128B. doi:10.1038/357128a0. ISSN 0028-0836. PMID 1579162. S2CID 4346767.
- ^ a b c d e f Li N, Lam WH, Zhai Y, Cheng J, Cheng E, Zhao Y, Gao N, Tye BK (July 2018). "Structure of the origin recognition complex bound to DNA replication origin". Nature. 559 (7713): 217–222. Bibcode:2018Natur.559..217L. doi:10.1038/s41586-018-0293-x. PMID 29973722. S2CID 49577101.
- ^ Miotto B, Ji Z, Struhl K (August 2016). "Selectivity of ORC binding sites and the relation to replication timing, fragile sites, and deletions in cancers". Proceedings of the National Academy of Sciences of the United States of America. 113 (33): E4810-9. doi:10.1073/pnas.1609060113. PMC 4995967. PMID 27436900.
- ^ Lee, Clare S. K.; Cheung, Ming Fung; Li, Jinsen; Zhao, Yongqian; Lam, Wai Hei; Ho, Vincy; Rohs, Remo; Zhai, Yuanliang; Leung, Danny; Tye, Bik-Kwoon (2021-01-04). "Humanizing the yeast origin recognition complex". Nature Communications. 12 (1): 33. Bibcode:2021NatCo..12...33L. doi:10.1038/s41467-020-20277-y. ISSN 2041-1723. PMC 7782691. PMID 33397927.
- ^ Bleichert F, Leitner A, Aebersold R, Botchan MR, Berger JM (June 2018). "Conformational control and DNA-binding mechanism of the metazoan origin recognition complex". Proceedings of the National Academy of Sciences of the United States of America. 115 (26): E5906–E5915. doi:10.1073/pnas.1806315115. PMC 6042147. PMID 29899147.
- ^ Chesnokov IN (2007). "Multiple functions of the origin recognition complex". International Review of Cytology. 256: 69–109. doi:10.1016/S0074-7696(07)56003-1. ISBN 9780123737007. PMID 17241905.
- ^ Matsuda K, Makise M, Sueyasu Y, Takehara M, Asano T, Mizushima T (December 2007). "Yeast two-hybrid analysis of the origin recognition complex of Saccharomyces cerevisiae: interaction between subunits and identification of binding proteins". FEMS Yeast Research. 7 (8): 1263–9. doi:10.1111/j.1567-1364.2007.00298.x. PMID 17825065.
- ^ Kreitz S, Ritzi M, Baack M, Knippers R (March 2001). "The human origin recognition complex protein 1 dissociates from chromatin during S phase in HeLa cells". The Journal of Biological Chemistry. 276 (9): 6337–42. doi:10.1074/jbc.M009473200. PMID 11102449.
- ^ Speck C, Chen Z, Li H, Stillman B (November 2005). "ATPase-dependent cooperative binding of ORC and Cdc6 to origin DNA". Nature Structural & Molecular Biology. 12 (11): 965–71. doi:10.1038/nsmb1002. PMC 2952294. PMID 16228006.
- ^ Coleman TR, Carpenter PB, Dunphy WG (October 1996). "The Xenopus Cdc6 protein is essential for the initiation of a single round of DNA replication in cell-free extracts". Cell. 87 (1): 53–63. doi:10.1016/S0092-8674(00)81322-7. PMID 8858148. S2CID 16897247.
- ^ Rialland M, Sola F, Santocanale C (April 2002). "Essential role of human CDT1 in DNA replication and chromatin licensing". Journal of Cell Science. 115 (Pt 7): 1435–40. doi:10.1242/jcs.115.7.1435. PMID 11896191.
- ^ Tanaka S, Diffley JF (March 2002). "Interdependent nuclear accumulation of budding yeast Cdt1 and Mcm2-7 during G1 phase". Nature Cell Biology. 4 (3): 198–207. doi:10.1038/ncb757. PMID 11836525. S2CID 45861829.
- ^ Frigola J, He J, Kinkelin K, Pye VE, Renault L, Douglas ME, Remus D, Cherepanov P, Costa A, Diffley JF (June 2017). "Cdt1 stabilizes an open MCM ring for helicase loading". Nature Communications. 8: 15720. Bibcode:2017NatCo...815720F. doi:10.1038/ncomms15720. PMC 5490006. PMID 28643783.
- ^ Ticau S, Friedman LJ, Champasa K, Corrêa IR, Gelles J, Bell SP (March 2017). "Mechanism and timing of Mcm2-7 ring closure during DNA replication origin licensing". Nature Structural & Molecular Biology. 24 (3): 309–315. doi:10.1038/nsmb.3375. PMC 5336523. PMID 28191892.
- ^ Nishitani H, Lygerou Z, Nishimoto T, Nurse P (April 2000). "The Cdt1 protein is required to license DNA for replication in fission yeast". Nature. 404 (6778): 625–8. Bibcode:2000Natur.404..625N. doi:10.1038/35007110. PMID 10766248. S2CID 205005540.
- ^ Yuan Z, Riera A, Bai L, Sun J, Nandi S, Spanos C, Chen ZA, Barbon M, Rappsilber J, Stillman B, Speck C, Li H (March 2017). "Structural basis of Mcm2-7 replicative helicase loading by ORC-Cdc6 and Cdt1". Nature Structural & Molecular Biology. 24 (3): 316–324. doi:10.1038/nsmb.3372. PMC 5503505. PMID 28191893.
- ^ Wohlschlegel JA, Dwyer BT, Dhar SK, Cvetic C, Walter JC, Dutta A (December 2000). "Inhibition of eukaryotic DNA replication by geminin binding to Cdt1". Science. 290 (5500): 2309–12. Bibcode:2000Sci...290.2309W. doi:10.1126/science.290.5500.2309. PMID 11125146.
- ^ Maine GT, Sinha P, Tye BK (March 1984). "Mutants of S. cerevisiae defective in the maintenance of minichromosomes". Genetics. 106 (3): 365–85. doi:10.1093/genetics/106.3.365. PMC 1224244. PMID 6323245.
- ^ Hua XH, Newport J (January 1998). "Identification of a preinitiation step in DNA replication that is independent of origin recognition complex and cdc6, but dependent on cdk2". The Journal of Cell Biology. 140 (2): 271–81. doi:10.1083/jcb.140.2.271. PMC 2132576. PMID 9442103.
- ^ Rowles A, Tada S, Blow JJ (June 1999). "Changes in association of the Xenopus origin recognition complex with chromatin on licensing of replication origins". Journal of Cell Science. 112 (12): 2011–8. doi:10.1242/jcs.112.12.2011. PMC 3605702. PMID 10341218.
- ^ a b c Li N, Zhai Y, Zhang Y, Li W, Yang M, Lei J, Tye BK, Gao N (August 2015). "Structure of the eukaryotic MCM complex at 3.8 Å". Nature. 524 (7564): 186–91. Bibcode:2015Natur.524..186L. doi:10.1038/nature14685. PMID 26222030. S2CID 4468690.
- ^ Noguchi Y, Yuan Z, Bai L, Schneider S, Zhao G, Stillman B, Speck C, Li H (November 2017). "Cryo-EM structure of Mcm2-7 double hexamer on DNA suggests a lagging-strand DNA extrusion model". Proceedings of the National Academy of Sciences of the United States of America. 114 (45): E9529–E9538. doi:10.1073/pnas.1712537114. PMC 5692578. PMID 29078375.
- ^ a b Dutta A, Bell SP (1997). "Initiation of DNA replication in eukaryotic cells". Annual Review of Cell and Developmental Biology. 13: 293–332. doi:10.1146/annurev.cellbio.13.1.293. PMID 9442876.
- ^ a b Labib K, Tercero JA, Diffley JF (June 2000). "Uninterrupted MCM2-7 function required for DNA replication fork progression". Science. 288 (5471): 1643–7. Bibcode:2000Sci...288.1643L. doi:10.1126/science.288.5471.1643. PMID 10834843.
- ^ Schwacha A, Bell SP (November 2001). "Interactions between two catalytically distinct MCM subgroups are essential for coordinated ATP hydrolysis and DNA replication". Molecular Cell. 8 (5): 1093–104. doi:10.1016/S1097-2765(01)00389-6. PMID 11741544.
- ^ Boyer PD (January 1993). "The binding change mechanism for ATP synthase--some probabilities and possibilities". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1140 (3): 215–50. doi:10.1016/0005-2728(93)90063-l. PMID 8417777.
- ^ Hingorani MM, Washington MT, Moore KC, Patel SS (May 1997). "The dTTPase mechanism of T7 DNA helicase resembles the binding change mechanism of the F1-ATPase". Proceedings of the National Academy of Sciences of the United States of America. 94 (10): 5012–7. Bibcode:1997PNAS...94.5012H. doi:10.1073/pnas.94.10.5012. PMC 24622. PMID 9144181.
- ^ Yan H, Merchant AM, Tye BK (November 1993). "Cell cycle-regulated nuclear localization of MCM2 and MCM3, which are required for the initiation of DNA synthesis at chromosomal replication origins in yeast". Genes & Development. 7 (11): 2149–60. doi:10.1101/gad.7.11.2149. PMID 8224843.
- ^ Young MR, Suzuki K, Yan H, Gibson S, Tye BK (October 1997). "Nuclear accumulation of Saccharomyces cerevisiae Mcm3 is dependent on its nuclear localization sequence". Genes to Cells. 2 (10): 631–43. doi:10.1046/j.1365-2443.1997.1510349.x. PMID 9427284.
- ^ Labib K, Diffley JF, Kearsey SE (November 1999). "G1-phase and B-type cyclins exclude the DNA-replication factor Mcm4 from the nucleus". Nature Cell Biology. 1 (7): 415–22. doi:10.1038/15649. PMID 10559985. S2CID 23407351.
- ^ Lei M, Tye BK (April 2001). "Initiating DNA synthesis: from recruiting to activating the MCM complex". Journal of Cell Science. 114 (Pt 8): 1447–54. doi:10.1242/jcs.114.8.1447. PMID 11282021.
- ^ Leatherwood J (December 1998). "Emerging mechanisms of eukaryotic DNA replication initiation". Current Opinion in Cell Biology. 10 (6): 742–8. doi:10.1016/S0955-0674(98)80117-8. PMID 9914182.
- ^ a b Mimura S, Takisawa H (October 1998). "Xenopus Cdc45-dependent loading of DNA polymerase alpha onto chromatin under the control of S-phase Cdk". The EMBO Journal. 17 (19): 5699–707. doi:10.1093/emboj/17.19.5699. PMC 1170898. PMID 9755170.
- ^ a b Zou L, Stillman B (April 1998). "Formation of a preinitiation complex by S-phase cyclin CDK-dependent loading of Cdc45p onto chromatin". Science. 280 (5363): 593–6. Bibcode:1998Sci...280..593Z. doi:10.1126/science.280.5363.593. PMID 9554851.
- ^ a b c Mimura S, Masuda T, Matsui T, Takisawa H (June 2000). "Central role for cdc45 in establishing an initiation complex of DNA replication in Xenopus egg extracts". Genes to Cells. 5 (6): 439–52. doi:10.1046/j.1365-2443.2000.00340.x. PMID 10886370.
- ^ a b c Aparicio OM, Weinstein DM, Bell SP (October 1997). "Components and dynamics of DNA replication complexes in S. cerevisiae: redistribution of MCM proteins and Cdc45p during S phase". Cell. 91 (1): 59–69. doi:10.1016/S0092-8674(01)80009-X. PMID 9335335. S2CID 10353164.
- ^ Tercero JA, Labib K, Diffley JF (May 2000). "DNA synthesis at individual replication forks requires the essential initiation factor Cdc45p". The EMBO Journal. 19 (9): 2082–93. doi:10.1093/emboj/19.9.2082. PMC 305696. PMID 10790374.
- ^ Hennessy KM, Lee A, Chen E, Botstein D (June 1991). "A group of interacting yeast DNA replication genes". Genes & Development. 5 (6): 958–69. doi:10.1101/gad.5.6.958. PMID 2044962.
- ^ Aparicio OM, Stout AM, Bell SP (August 1999). "Differential assembly of Cdc45p and DNA polymerases at early and late origins of DNA replication". Proceedings of the National Academy of Sciences of the United States of America. 96 (16): 9130–5. Bibcode:1999PNAS...96.9130A. doi:10.1073/pnas.96.16.9130. PMC 17744. PMID 10430907.
- ^ Zou L, Stillman B (May 2000). "Assembly of a complex containing Cdc45p, replication protein A, and Mcm2p at replication origins controlled by S-phase cyclin-dependent kinases and Cdc7p-Dbf4p kinase". Molecular and Cellular Biology. 20 (9): 3086–96. doi:10.1128/mcb.20.9.3086-3096.2000. PMC 85601. PMID 10757793.
- ^ a b Walter J, Newport J (April 2000). "Initiation of eukaryotic DNA replication: origin unwinding and sequential chromatin association of Cdc45, RPA, and DNA polymerase alpha". Molecular Cell. 5 (4): 617–27. doi:10.1016/S1097-2765(00)80241-5. PMID 10882098.
- ^ a b c Gambus A, Jones RC, Sanchez-Diaz A, Kanemaki M, van Deursen F, Edmondson RD, Labib K (April 2006). "GINS maintains association of Cdc45 with MCM in replisome progression complexes at eukaryotic DNA replication forks". Nature Cell Biology. 8 (4): 358–66. doi:10.1038/ncb1382. PMID 16531994. S2CID 21543095.
- ^ a b Kanemaki M, Sanchez-Diaz A, Gambus A, Labib K (June 2003). "Functional proteomic identification of DNA replication proteins by induced proteolysis in vivo". Nature. 423 (6941): 720–4. Bibcode:2003Natur.423..720K. doi:10.1038/nature01692. PMID 12768207. S2CID 4345091.
- ^ Makarova KS, Wolf YI, Mekhedov SL, Mirkin BG, Koonin EV (2005). "Ancestral paralogs and pseudoparalogs and their role in the emergence of the eukaryotic cell". Nucleic Acids Research. 33 (14): 4626–38. doi:10.1093/nar/gki775. PMC 1187821. PMID 16106042.
- ^ Moyer SE, Lewis PW, Botchan MR (July 2006). "Isolation of the Cdc45/Mcm2-7/GINS (CMG) complex, a candidate for the eukaryotic DNA replication fork helicase". Proceedings of the National Academy of Sciences of the United States of America. 103 (27): 10236–10241. Bibcode:2006PNAS..10310236M. doi:10.1073/pnas.0602400103. PMC 1482467. PMID 16798881.
- ^ Bochman ML, Schwacha A (July 2008). "The Mcm2-7 complex has in vitro helicase activity". Molecular Cell. 31 (2): 287–93. doi:10.1016/j.molcel.2008.05.020. PMID 18657510.
- ^ Costa A, Ilves I, Tamberg N, Petojevic T, Nogales E, Botchan MR, Berger JM (April 2011). "The structural basis for MCM2-7 helicase activation by GINS and Cdc45". Nature Structural & Molecular Biology. 18 (4): 471–7. doi:10.1038/nsmb.2004. PMC 4184033. PMID 21378962.
- ^ Yuan Z, Bai L, Sun J, Georgescu R, Liu J, O'Donnell ME, Li H (March 2016). "Structure of the eukaryotic replicative CMG helicase suggests a pumpjack motion for translocation". Nature Structural & Molecular Biology. 23 (3): 217–24. doi:10.1038/nsmb.3170. PMC 4812828. PMID 26854665.
- ^ Homesley L, Lei M, Kawasaki Y, Sawyer S, Christensen T, Tye BK (April 2000). "Mcm10 and the MCM2-7 complex interact to initiate DNA synthesis and to release replication factors from origins". Genes & Development. 14 (8): 913–26. doi:10.1101/gad.14.8.913. PMC 316538. PMID 10783164.
- ^ Christensen TW, Tye BK (June 2003). "Drosophila MCM10 interacts with members of the prereplication complex and is required for proper chromosome condensation". Molecular Biology of the Cell. 14 (6): 2206–15. doi:10.1091/mbc.e02-11-0706. PMC 194871. PMID 12808023.
- ^ Lee C, Liachko I, Bouten R, Kelman Z, Tye BK (January 2010). "Alternative mechanisms for coordinating polymerase alpha and MCM helicase". Molecular and Cellular Biology. 30 (2): 423–35. doi:10.1128/MCB.01240-09. PMC 2798462. PMID 19917723.
- ^ van Deursen F, Sengupta S, De Piccoli G, Sanchez-Diaz A, Labib K (May 2012). "Mcm10 associates with the loaded DNA helicase at replication origins and defines a novel step in its activation". The EMBO Journal. 31 (9): 2195–206. doi:10.1038/emboj.2012.69. PMC 3343467. PMID 22433841.
- ^ Masai H, Sato N, Takeda T, Arai K (December 1999). "CDC7 kinase complex as a molecular switch for DNA replication". Frontiers in Bioscience. 4 (1–3): D834-40. doi:10.2741/masai. PMID 10577390.
- ^ Jiang W, Wells NJ, Hunter T (May 1999). "Multistep regulation of DNA replication by Cdk phosphorylation of HsCdc6". Proceedings of the National Academy of Sciences of the United States of America. 96 (11): 6193–8. Bibcode:1999PNAS...96.6193J. doi:10.1073/pnas.96.11.6193. PMC 26858. PMID 10339564.
- ^ a b Jiang W, McDonald D, Hope TJ, Hunter T (October 1999). "Mammalian Cdc7-Dbf4 protein kinase complex is essential for initiation of DNA replication". The EMBO Journal. 18 (20): 5703–13. doi:10.1093/emboj/18.20.5703. PMC 1171637. PMID 10523313.
- ^ Kumagai H, Sato N, Yamada M, Mahony D, Seghezzi W, Lees E, Arai K, Masai H (July 1999). "A novel growth- and cell cycle-regulated protein, ASK, activates human Cdc7-related kinase and is essential for G1/S transition in mammalian cells". Molecular and Cellular Biology. 19 (7): 5083–95. doi:10.1128/MCB.19.7.5083. PMC 84351. PMID 10373557.
- ^ Lei M, Kawasaki Y, Young MR, Kihara M, Sugino A, Tye BK (December 1997). "Mcm2 is a target of regulation by Cdc7-Dbf4 during the initiation of DNA synthesis". Genes & Development. 11 (24): 3365–74. doi:10.1101/gad.11.24.3365. PMC 316824. PMID 9407029.
- ^ Hardy CF, Dryga O, Seematter S, Pahl PM, Sclafani RA (April 1997). "mcm5/cdc46-bob1 bypasses the requirement for the S phase activator Cdc7p". Proceedings of the National Academy of Sciences of the United States of America. 94 (7): 3151–5. Bibcode:1997PNAS...94.3151H. doi:10.1073/pnas.94.7.3151. PMC 20337. PMID 9096361.
- ^ Kamimura Y, Tak YS, Sugino A, Araki H (April 2001). "Sld3, which interacts with Cdc45 (Sld4), functions for chromosomal DNA replication in Saccharomyces cerevisiae". The EMBO Journal. 20 (8): 2097–107. doi:10.1093/emboj/20.8.2097. PMC 125422. PMID 11296242.
- ^ Kanemaki M, Labib K (April 2006). "Distinct roles for Sld3 and GINS during establishment and progression of eukaryotic DNA replication forks". The EMBO Journal. 25 (8): 1753–63. doi:10.1038/sj.emboj.7601063. PMC 1440835. PMID 16601689.
- ^ Masumoto H, Sugino A, Araki H (April 2000). "Dpb11 controls the association between DNA polymerases alpha and epsilon and the autonomously replicating sequence region of budding yeast". Molecular and Cellular Biology. 20 (8): 2809–17. doi:10.1128/mcb.20.8.2809-2817.2000. PMC 85497. PMID 10733584.
- ^ Araki H, Leem SH, Phongdara A, Sugino A (December 1995). "Dpb11, which interacts with DNA polymerase II(epsilon) in Saccharomyces cerevisiae, has a dual role in S-phase progression and at a cell cycle checkpoint". Proceedings of the National Academy of Sciences of the United States of America. 92 (25): 11791–5. doi:10.1073/pnas.92.25.11791. PMC 40488. PMID 8524850.
- ^ Glover JN, Williams RS, Lee MS (November 2004). "Interactions between BRCT repeats and phosphoproteins: tangled up in two". Trends in Biochemical Sciences. 29 (11): 579–85. doi:10.1016/j.tibs.2004.09.010. PMID 15501676.
- ^ Masumoto H, Muramatsu S, Kamimura Y, Araki H (February 2002). "S-Cdk-dependent phosphorylation of Sld2 essential for chromosomal DNA replication in budding yeast". Nature. 415 (6872): 651–5. Bibcode:2002Natur.415..651M. doi:10.1038/nature713. PMID 11807498. S2CID 4300863.
- ^ Takayama Y, Kamimura Y, Okawa M, Muramatsu S, Sugino A, Araki H (May 2003). "GINS, a novel multiprotein complex required for chromosomal DNA replication in budding yeast". Genes & Development. 17 (9): 1153–65. doi:10.1101/gad.1065903. PMC 196052. PMID 12730134.
- ^ Labib K, Gambus A (June 2007). "A key role for the GINS complex at DNA replication forks". Trends in Cell Biology. 17 (6): 271–8. doi:10.1016/j.tcb.2007.04.002. PMID 17467990.
- ^ Muramatsu S, Hirai K, Tak YS, Kamimura Y, Araki H (March 2010). "CDK-dependent complex formation between replication proteins Dpb11, Sld2, Pol (epsilon}, and GINS in budding yeast". Genes & Development. 24 (6): 602–12. doi:10.1101/gad.1883410. PMC 2841337. PMID 20231317.
- ^ Lehman IR, Bessman MJ, Simms ES, Kornberg A (July 1958). "Enzymatic synthesis of deoxyribonucleic acid. I. Preparation of substrates and partial purification of an enzyme from Escherichia coli". The Journal of Biological Chemistry. 233 (1): 163–70. doi:10.1016/S0021-9258(19)68048-8. PMID 13563462.
- ^ Alani E, Thresher R, Griffith JD, Kolodner RD (September 1992). "Characterization of DNA-binding and strand-exchange stimulation properties of y-RPA, a yeast single-strand-DNA-binding protein". Journal of Molecular Biology. 227 (1): 54–71. doi:10.1016/0022-2836(92)90681-9. PMID 1522601.
- ^ Goulian M, Richards SH, Heard CJ, Bigsby BM (October 1990). "Discontinuous DNA synthesis by purified mammalian proteins". The Journal of Biological Chemistry. 265 (30): 18461–71. doi:10.1016/S0021-9258(17)44775-2. PMID 2170412.
- ^ McCulloch SD, Kunkel TA (January 2008). "The fidelity of DNA synthesis by eukaryotic replicative and translesion synthesis polymerases". Cell Research. 18 (1): 148–61. doi:10.1038/cr.2008.4. PMC 3639319. PMID 18166979.
- ^ Pursell ZF, Isoz I, Lundström EB, Johansson E, Kunkel TA (July 2007). "Yeast DNA polymerase epsilon participates in leading-strand DNA replication". Science. 317 (5834): 127–30. Bibcode:2007Sci...317..127P. doi:10.1126/science.1144067. PMC 2233713. PMID 17615360.
- ^ a b c d e Watson J, Baker T, Bell S, Gann A, Levine M, Losick R, Molecular Biology of the Gene 6 Edition, Pearson Education Inc. 2008.ISBN 080539592X
- ^ Waga S, Bauer G, Stillman B (April 1994). "Reconstitution of complete SV40 DNA replication with purified replication factors". The Journal of Biological Chemistry. 269 (14): 10923–34. doi:10.1016/S0021-9258(17)34146-7. PMID 8144677.
- ^ Garg P, Stith CM, Sabouri N, Johansson E, Burgers PM (November 2004). "Idling by DNA polymerase delta maintains a ligatable nick during lagging-strand DNA replication". Genes & Development. 18 (22): 2764–73. doi:10.1101/gad.1252304. PMC 528896. PMID 15520275.
- ^ Burgers PM (February 2009). "Polymerase dynamics at the eukaryotic DNA replication fork". The Journal of Biological Chemistry. 284 (7): 4041–5. doi:10.1074/jbc.R800062200. PMC 2640984. PMID 18835809.
- ^ Jin YH, Ayyagari R, Resnick MA, Gordenin DA, Burgers PM (January 2003). "Okazaki fragment maturation in yeast. II. Cooperation between the polymerase and 3'-5'-exonuclease activities of Pol delta in the creation of a ligatable nick". The Journal of Biological Chemistry. 278 (3): 1626–33. doi:10.1074/jbc.M209803200. PMID 12424237.
- ^ Sogo JM, Lopes M, Foiani M (July 2002). "Fork reversal and ssDNA accumulation at stalled replication forks owing to checkpoint defects". Science. 297 (5581): 599–602. Bibcode:2002Sci...297..599S. doi:10.1126/science.1074023. PMID 12142537. S2CID 33502697.
- ^ Morrison A, Araki H, Clark AB, Hamatake RK, Sugino A (September 1990). "A third essential DNA polymerase in S. cerevisiae". Cell. 62 (6): 1143–51. doi:10.1016/0092-8674(90)90391-Q. PMID 2169349. S2CID 29672985.
- ^ Waga S, Stillman B (May 1994). "Anatomy of a DNA replication fork revealed by reconstitution of SV40 DNA replication in vitro". Nature. 369 (6477): 207–12. Bibcode:1994Natur.369..207W. doi:10.1038/369207a0. PMID 7910375. S2CID 4351628.
- ^ Tsurimoto T, Stillman B (January 1991). "Replication factors required for SV40 DNA replication in vitro. II. Switching of DNA polymerase alpha and delta during initiation of leading and lagging strand synthesis". The Journal of Biological Chemistry. 266 (3): 1961–8. doi:10.1016/S0021-9258(18)52386-3. PMID 1671046.
- ^ Barry ER, Bell SD (December 2006). "DNA replication in the archaea". Microbiology and Molecular Biology Reviews. 70 (4): 876–87. doi:10.1128/MMBR.00029-06. PMC 1698513. PMID 17158702.
- ^ Berg JM, Tymoczko JL, Stryer L (2003). Biochemie. Heidelberg/Berlin: Springer.
- ^ a b Johnson RE, Klassen R, Prakash L, Prakash S (July 2015). "A Major Role of DNA Polymerase δ in Replication of Both the Leading and Lagging DNA Strands". Molecular Cell. 59 (2): 163–175. doi:10.1016/j.molcel.2015.05.038. PMC 4517859. PMID 26145172.
- ^ Byun TS, Pacek M, Yee MC, Walter JC, Cimprich KA (May 2005). "Functional uncoupling of MCM helicase and DNA polymerase activities activates the ATR-dependent checkpoint". Genes & Development. 19 (9): 1040–52. doi:10.1101/gad.1301205. PMC 1091739. PMID 15833913.
- ^ a b Remus D, Beuron F, Tolun G, Griffith JD, Morris EP, Diffley JF (November 2009). "Concerted loading of Mcm2-7 double hexamers around DNA during DNA replication origin licensing". Cell. 139 (4): 719–30. doi:10.1016/j.cell.2009.10.015. PMC 2804858. PMID 19896182.
- ^ Evrin C, Clarke P, Zech J, Lurz R, Sun J, Uhle S, Li H, Stillman B, Speck C (December 2009). "A double-hexameric MCM2-7 complex is loaded onto origin DNA during licensing of eukaryotic DNA replication". Proceedings of the National Academy of Sciences of the United States of America. 106 (48): 20240–5. Bibcode:2009PNAS..10620240E. doi:10.1073/pnas.0911500106. PMC 2787165. PMID 19910535.
- ^ a b Moyer SE, Lewis PW, Botchan MR (July 2006). "Isolation of the Cdc45/Mcm2-7/GINS (CMG) complex, a candidate for the eukaryotic DNA replication fork helicase". Proceedings of the National Academy of Sciences of the United States of America. 103 (27): 10236–10241. Bibcode:2006PNAS..10310236M. doi:10.1073/pnas.0602400103. PMC 1482467. PMID 16798881.
- ^ Labib K (June 2010). "How do Cdc7 and cyclin-dependent kinases trigger the initiation of chromosome replication in eukaryotic cells?". Genes & Development. 24 (12): 1208–19. doi:10.1101/gad.1933010. PMC 2885657. PMID 20551170.
- ^ Pacek M, Walter JC (September 2004). "A requirement for MCM7 and Cdc45 in chromosome unwinding during eukaryotic DNA replication". The EMBO Journal. 23 (18): 3667–76. doi:10.1038/sj.emboj.7600369. PMC 517609. PMID 15329670.
- ^ Yao NY, O'Donnell M (June 2010). "SnapShot: The replisome". Cell. 141 (6): 1088–1088.e1. doi:10.1016/j.cell.2010.05.042. PMC 4007198. PMID 20550941.
- ^ Costa A, Ilves I, Tamberg N, Petojevic T, Nogales E, Botchan MR, Berger JM (April 2011). "The structural basis for MCM2-7 helicase activation by GINS and Cdc45". Nature Structural & Molecular Biology. 18 (4): 471–7. doi:10.1038/nsmb.2004. PMC 4184033. PMID 21378962.
- ^ Bermudez VP, Farina A, Tappin I, Hurwitz J (March 2010). "Influence of the human cohesion establishment factor Ctf4/AND-1 on DNA replication". The Journal of Biological Chemistry. 285 (13): 9493–505. doi:10.1074/jbc.M109.093609. PMC 2843200. PMID 20089864.
- ^ Zhu W, Ukomadu C, Jha S, Senga T, Dhar SK, Wohlschlegel JA, Nutt LK, Kornbluth S, Dutta A (September 2007). "Mcm10 and And-1/CTF4 recruit DNA polymerase alpha to chromatin for initiation of DNA replication". Genes & Development. 21 (18): 2288–99. doi:10.1101/gad.1585607. PMC 1973143. PMID 17761813.
- ^ Lou H, Komata M, Katou Y, Guan Z, Reis CC, Budd M, Shirahige K, Campbell JL (October 2008). "Mrc1 and DNA polymerase epsilon function together in linking DNA replication and the S phase checkpoint". Molecular Cell. 32 (1): 106–17. doi:10.1016/j.molcel.2008.08.020. PMC 2699584. PMID 18851837.
- ^ Petermann E, Helleday T, Caldecott KW (June 2008). "Claspin promotes normal replication fork rates in human cells". Molecular Biology of the Cell. 19 (6): 2373–8. doi:10.1091/mbc.E07-10-1035. PMC 2397295. PMID 18353973.
- ^ Bravo R, Frank R, Blundell PA, Macdonald-Bravo H (1987). "Cyclin/PCNA is the auxiliary protein of DNA polymerase-delta". Nature. 326 (6112): 515–7. Bibcode:1987Natur.326..515B. doi:10.1038/326515a0. PMID 2882423. S2CID 4344147.
- ^ Prelich G, Tan CK, Kostura M, Mathews MB, So AG, Downey KM, Stillman B (1987). "Functional identity of proliferating cell nuclear antigen and a DNA polymerase-delta auxiliary protein". Nature. 326 (6112): 517–20. Bibcode:1987Natur.326..517P. doi:10.1038/326517a0. PMID 2882424. S2CID 4345171.
- ^ Moldovan GL, Pfander B, Jentsch S (May 2007). "PCNA, the maestro of the replication fork". Cell. 129 (4): 665–79. doi:10.1016/j.cell.2007.05.003. PMID 17512402. S2CID 3547069.
- ^ Cai J, Yao N, Gibbs E, Finkelstein J, Phillips B, O'Donnell M, Hurwitz J (September 1998). "ATP hydrolysis catalyzed by human replication factor C requires participation of multiple subunits". Proceedings of the National Academy of Sciences of the United States of America. 95 (20): 11607–12. Bibcode:1998PNAS...9511607C. doi:10.1073/pnas.95.20.11607. PMC 21688. PMID 9751713.
- ^ Tsurimoto T, Stillman B (January 1991). "Replication factors required for SV40 DNA replication in vitro. I. DNA structure-specific recognition of a primer-template junction by eukaryotic DNA polymerases and their accessory proteins". The Journal of Biological Chemistry. 266 (3): 1950–60. doi:10.1016/S0021-9258(18)52385-1. PMID 1671045.
- ^ Podust LM, Podust VN, Sogo JM, Hübscher U (June 1995). "Mammalian DNA polymerase auxiliary proteins: analysis of replication factor C-catalyzed proliferating cell nuclear antigen loading onto circular double-stranded DNA". Molecular and Cellular Biology. 15 (6): 3072–81. doi:10.1128/MCB.15.6.3072. PMC 230538. PMID 7760803.
- ^ Zhang G, Gibbs E, Kelman Z, O'Donnell M, Hurwitz J (March 1999). "Studies on the interactions between human replication factor C and human proliferating cell nuclear antigen". Proceedings of the National Academy of Sciences of the United States of America. 96 (5): 1869–74. Bibcode:1999PNAS...96.1869Z. doi:10.1073/pnas.96.5.1869. PMC 26703. PMID 10051561.
- ^ a b Yao NY, Johnson A, Bowman GD, Kuriyan J, O'Donnell M (June 2006). "Mechanism of proliferating cell nuclear antigen clamp opening by replication factor C". The Journal of Biological Chemistry. 281 (25): 17528–39. doi:10.1074/jbc.M601273200. PMID 16608854.
- ^ a b c Sabatinos, SA (2010). "Recovering a Stalled Replication Fork". Scitable Nature Education. 3 (9): 31.
- ^ a b Rothstein R, Michel B, Gangloff S (January 2000). "Replication fork pausing and recombination or "gimme a break"". Genes & Development. 14 (1): 1–10. doi:10.1101/gad.14.1.1. PMID 10640269. S2CID 40751623.
- ^ Bastia D, Zzaman S, Krings G, Saxena M, Peng X, Greenberg MM (September 2008). "Replication termination mechanism as revealed by Tus-mediated polar arrest of a sliding helicase". Proceedings of the National Academy of Sciences of the United States of America. 105 (35): 12831–6. Bibcode:2008PNAS..10512831B. doi:10.1073/pnas.0805898105. PMC 2529109. PMID 18708526.
- ^ 복제 공장, PavelHozak, Peter R.Cook, Cell Biology 4권의 동향 2호; Scovendirect, DOI
- ^ McGarry TJ, Kirschner MW (June 1998). "Geminin, an inhibitor of DNA replication, is degraded during mitosis". Cell. 93 (6): 1043–53. doi:10.1016/S0092-8674(00)81209-X. PMID 9635433. S2CID 235485.
- ^ de Klein A, Muijtjens M, van Os R, Verhoeven Y, Smit B, Carr AM, Lehmann AR, Hoeijmakers JH (April 2000). "Targeted disruption of the cell-cycle checkpoint gene ATR leads to early embryonic lethality in mice". Current Biology. 10 (8): 479–82. doi:10.1016/S0960-9822(00)00447-4. PMID 10801416. S2CID 15401622.
- ^ Cortez D, Guntuku S, Qin J, Elledge SJ (November 2001). "ATR and ATRIP: partners in checkpoint signaling". Science. 294 (5547): 1713–6. Bibcode:2001Sci...294.1713C. doi:10.1126/science.1065521. PMID 11721054. S2CID 32810119.
- ^ Dart DA, Adams KE, Akerman I, Lakin ND (April 2004). "Recruitment of the cell cycle checkpoint kinase ATR to chromatin during S-phase". The Journal of Biological Chemistry. 279 (16): 16433–40. doi:10.1074/jbc.M314212200. PMID 14871897.
- ^ Ball HL, Myers JS, Cortez D (May 2005). "ATRIP binding to replication protein A-single-stranded DNA promotes ATR-ATRIP localization but is dispensable for Chk1 phosphorylation". Molecular Biology of the Cell. 16 (5): 2372–81. doi:10.1091/mbc.E04-11-1006. PMC 1087242. PMID 15743907.
- ^ Bermudez VP, Lindsey-Boltz LA, Cesare AJ, Maniwa Y, Griffith JD, Hurwitz J, Sancar A (February 2003). "Loading of the human 9-1-1 checkpoint complex onto DNA by the checkpoint clamp loader hRad17-replication factor C complex in vitro". Proceedings of the National Academy of Sciences of the United States of America. 100 (4): 1633–8. Bibcode:2003PNAS..100.1633B. doi:10.1073/pnas.0437927100. PMC 149884. PMID 12578958.
- ^ Zou L, Cortez D, Elledge SJ (January 2002). "Regulation of ATR substrate selection by Rad17-dependent loading of Rad9 complexes onto chromatin". Genes & Development. 16 (2): 198–208. doi:10.1101/gad.950302. PMC 155323. PMID 11799063.
- ^ Kumagai A, Dunphy WG (October 2000). "Claspin, a novel protein required for the activation of Chk1 during a DNA replication checkpoint response in Xenopus egg extracts". Molecular Cell. 6 (4): 839–49. doi:10.1016/S1097-2765(05)00092-4. PMID 11090622.
- ^ Kumagai A, Dunphy WG (February 2003). "Repeated phosphopeptide motifs in Claspin mediate the regulated binding of Chk1" (PDF). Nature Cell Biology. 5 (2): 161–5. doi:10.1038/ncb921. PMID 12545175. S2CID 29331203.
- ^ Miao H, Seiler JA, Burhans WC (February 2003). "Regulation of cellular and SV40 virus origins of replication by Chk1-dependent intrinsic and UVC radiation-induced checkpoints". The Journal of Biological Chemistry. 278 (6): 4295–304. doi:10.1074/jbc.M204264200. PMID 12424256.
- ^ Bar-Ziv R, Voichek Y, Barkai N (September 2016). "Chromatin dynamics during DNA replication". Genome Research. 26 (9): 1245–56. doi:10.1101/gr.201244.115. PMC 5052047. PMID 27225843.
- ^ Luger K, Mäder AW, Richmond RK, Sargent DF, Richmond TJ (September 1997). "Crystal structure of the nucleosome core particle at 2.8 A resolution". Nature. 389 (6648): 251–60. Bibcode:1997Natur.389..251L. doi:10.1038/38444. PMID 9305837. S2CID 4328827.
- ^ Wittmeyer J, Joss L, Formosa T (July 1999). "Spt16 and Pob3 of Saccharomyces cerevisiae form an essential, abundant heterodimer that is nuclear, chromatin-associated, and copurifies with DNA polymerase alpha". Biochemistry. 38 (28): 8961–71. doi:10.1021/bi982851d. PMID 10413469.
- ^ Wittmeyer J, Formosa T (July 1997). "The Saccharomyces cerevisiae DNA polymerase alpha catalytic subunit interacts with Cdc68/Spt16 and with Pob3, a protein similar to an HMG1-like protein". Molecular and Cellular Biology. 17 (7): 4178–90. doi:10.1128/MCB.17.7.4178. PMC 232271. PMID 9199353.
- ^ Xin H, Takahata S, Blanksma M, McCullough L, Stillman DJ, Formosa T (August 2009). "yFACT induces global accessibility of nucleosomal DNA without H2A-H2B displacement". Molecular Cell. 35 (3): 365–76. doi:10.1016/j.molcel.2009.06.024. PMC 2748400. PMID 19683499.
- ^ Groth A, Corpet A, Cook AJ, Roche D, Bartek J, Lukas J, Almouzni G (December 2007). "Regulation of replication fork progression through histone supply and demand". Science. 318 (5858): 1928–31. Bibcode:2007Sci...318.1928G. doi:10.1126/science.1148992. hdl:1885/53159. PMID 18096807. S2CID 22350778.
- ^ Daganzo SM, Erzberger JP, Lam WM, Skordalakes E, Zhang R, Franco AA, Brill SJ, Adams PD, Berger JM, Kaufman PD (December 2003). "Structure and function of the conserved core of histone deposition protein Asf1". Current Biology. 13 (24): 2148–58. doi:10.1016/j.cub.2003.11.027. PMID 14680630. S2CID 15164132.
- ^ Voichek Y, Bar-Ziv R, Barkai N (March 2016). "Expression homeostasis during DNA replication". Science. 351 (6277): 1087–90. Bibcode:2016Sci...351.1087V. doi:10.1126/science.aad1162. PMID 26941319. S2CID 32751800.
- ^ Stillman B (May 1986). "Chromatin assembly during SV40 DNA replication in vitro". Cell. 45 (4): 555–65. doi:10.1016/0092-8674(86)90287-4. PMID 3011272. S2CID 21212160.
- ^ Shibahara K, Stillman B (February 1999). "Replication-dependent marking of DNA by PCNA facilitates CAF-1-coupled inheritance of chromatin". Cell. 96 (4): 575–85. doi:10.1016/S0092-8674(00)80661-3. PMID 10052459. S2CID 13912324.
- ^ Rolef Ben-Shahar T, Castillo AG, Osborne MJ, Borden KL, Kornblatt J, Verreault A (December 2009). "Two fundamentally distinct PCNA interaction peptides contribute to chromatin assembly factor 1 function". Molecular and Cellular Biology. 29 (24): 6353–65. doi:10.1128/MCB.01051-09. PMC 2786881. PMID 19822659.
- ^ Huang S, Zhou H, Katzmann D, Hochstrasser M, Atanasova E, Zhang Z (September 2005). "Rtt106p is a histone chaperone involved in heterochromatin-mediated silencing". Proceedings of the National Academy of Sciences of the United States of America. 102 (38): 13410–5. Bibcode:2005PNAS..10213410H. doi:10.1073/pnas.0506176102. PMC 1224646. PMID 16157874.
- ^ Ito T, Tyler JK, Bulger M, Kobayashi R, Kadonaga JT (October 1996). "ATP-facilitated chromatin assembly with a nucleoplasmin-like protein from Drosophila melanogaster". The Journal of Biological Chemistry. 271 (40): 25041–8. doi:10.1074/jbc.271.40.25041. PMID 8798787.
- ^ Gasser R, Koller T, Sogo JM (May 1996). "The stability of nucleosomes at the replication fork". Journal of Molecular Biology. 258 (2): 224–39. doi:10.1006/jmbi.1996.0245. PMID 8627621.
- ^ Meselson M, Stahl FW (July 1958). "The replication of DNA in Escherichia coli". Proceedings of the National Academy of Sciences of the United States of America. 44 (7): 671–82. Bibcode:1958PNAS...44..671M. doi:10.1073/pnas.44.7.671. PMC 528642. PMID 16590258.
- ^ Bell SP, Kaguni JM (June 2013). "Helicase loading at chromosomal origins of replication". Cold Spring Harbor Perspectives in Biology. 5 (6): a010124. doi:10.1101/cshperspect.a010124. PMC 3660832. PMID 23613349.