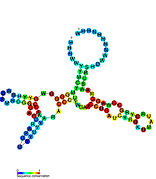
리보스위치
Riboswitch분자생물학에서 리보스위치는 작은 분자와 [1][2][3][4]결합하는 메신저 RNA 분자의 조절 세그먼트이며, mRNA에 의해 코드된 단백질의 생산 변화를 일으킨다.따라서 리보스위치를 포함하는 mRNA는 이펙터 분자의 농도에 반응하여 자신의 활성을 조절하는 데 직접적으로 관여한다.현대의 유기체들이 작은 분자들을 결합하고, 밀접하게 연관된 유사체들을 구별하기 위해 RNA를 사용한다는 발견은, RNA의 알려진 자연적인 능력을 단백질을 코드화하고, 반응을 촉매하고, 또는 다른 RNA나 단백질 고분자를 결합하는 능력 이상으로 확장시켰다.
"리보스위치"라는 용어의 원래 정의는 소분자 대사물의 [5]농도를 직접 감지하도록 명시했습니다.이 정의는 여전히 일반적으로 사용되고 있지만, 일부 생물학자들은 다른 시스 조절 RNA를 포함하는 더 넓은 정의를 사용해 왔다.그러나 이 문서에서는 대사물 결합 리보스위치만 설명합니다.
대부분의 알려진 리보스위치는 박테리아에서 발생하지만, 한 종류의 기능성 리보스위치(TPP 리보스위치)는 고세균, 식물 및 특정 균류에서 발견되었습니다.TPP 리보스위치는 고세균에서도 [6]예측되었지만 실험적으로 테스트되지는 않았다.
이력 및 검출
리보스위치가 발견되기 전에는 여러 대사 경로에 관여하는 일부 유전자가 조절되는 메커니즘이 미스터리로 남아 있었다.축적된 증거는 관련된 mRNA가 대사물을 직접 결합하여 자신의 조절에 영향을 미칠 수 있다는 당시 전례 없는 생각을 점점 더 암시했다.이러한 데이터에는 관련 유전자의 미번역 영역(UTR)에서 종종 발견되는 보존된 RNA 2차 구조와 [7][8][9][10][11]압타머라고 불리는 인공 소분자 결합 RNA를 만드는 절차의 성공이 포함되었다.2002년에 단백질 없는 결합 분석을 포함한 여러 종류의 리보스위치에 대한 최초의 포괄적인 증거가 발표되었고, 유전자 [5][12][13][14]조절의 새로운 메커니즘으로 대사물 결합 리보스위치가 확립되었다.
초기 riboswitches 발견되어 지는 것의 많은 여러분에게 순서에 주고받"모티브"(패턴)에 5'UTRs는 등장에 일치하는 것 잘 구성된 RNA. 예를 들면, 예를 들어, 비교 분석 상류 지역의 몇몇 유전자를 것으로 보일 co-regulated에 관한 설명 S-box[15](현재 SAM-I riboswitch), THI-box[9](.한 regioTPP 리보스위치 내 n), RFN 요소[8](현 FMN 리보스위치) 및 B-박스12[16](코발라민 리보스위치의 일부), 그리고 경우에 따라서는 그들이 알려지지 않은 메커니즘을 통해 유전자 조절에 관여했다는 실험 시연.생물정보학은 최근의 발견에서 기본적인 비교게노믹스 전략의 자동화를 증가시키면서 역할을 해왔다.배릭 등(2004)[17]는 BLAST를 사용하여 Bacillus subtilis의 모든 UTR과 상동하는 UTRs를 찾아냈다.이러한 상동성 세트 중 일부는 보존 구조를 검사하여 10개의 RNA 유사 모티브를 얻었다.이들 중 3개는 나중에 glmS, 글리신 및 PreQ1-I 리보스위치(아래 참조)로 실험적으로 확인되었다.박테리아의 추가 분류군과 개선된 컴퓨터 알고리즘을 사용한 후속 비교 유전체 연구는 [18][19][20]리보스위치로서 기능하는 것으로 가정된 보존된 RNA 구조와 함께 실험적으로 확인된 추가적인 리보스위치를 식별했다.
메커니즘
리보스위치는 개념적으로 압타머와 표현 플랫폼의 두 부분으로 구분되는 경우가 많습니다.압타머는 작은 분자를 직접 결합하고 발현 플랫폼은 압타머의 변화에 반응하여 구조적 변화를 겪습니다.발현 플랫폼은 유전자 발현을 조절하는 것이다.
발현 플랫폼은 일반적으로 작은 분자에 반응하여 유전자 발현을 끄지만 일부는 그것을 활성화시킨다.다음과 같은 리보스위치 메커니즘이 실험적으로 입증되었습니다.
- 리보스위치 제어에 의한 Rho비의존성 전사종단 헤어핀의 형성은 조기 전사종단으로 이어진다.
- 리보솜 결합 부위를 리보스위치 매개 접이식으로 격리하여 번역을 저해한다.
- 리보스위치는 충분한 농도의 대사물 존재 하에서 스스로를 분해하는 리보자임이다.
- 리보스위치 대체 구조는 사전 mRNA의 스플라이싱에 영향을 줍니다.
- 클로스트리디움 아세트뷰틸리쿰의 리보스위치는 동일한 mRNA 전사체의 일부가 아닌 인접 유전자를 조절한다.이 조절에서 리보스위치는 유전자의 전사를 방해한다.메커니즘은 불확실하지만 동일한 [24]DNA를 동시에 전사하기 때문에 두 RNA 중합효소 단위 간의 충돌로 인해 발생할 수 있습니다.
- 리스테리아 모노사이토제네스의 리보스위치는 하류 유전자의 발현을 조절한다.그러나 리보스위치 전사는 이후 [25]게놈의 다른 곳에 위치한 유전자의 발현을 조절한다.이 전달 조절은 말단 유전자의 mRNA에 염기쌍을 통해 일어난다.박테리아의 온도가 올라가면 리보스위치가 녹아서 전사가 가능해진다.발표되지 않은 학부 연구는 리스테리아 모노사이토제네스 [26]배열의 무작위 돌연변이를 통해 유사한 리보스위치 또는 '서모센서'를 생성했다.
종류들
다음은 실험적으로 검증된 리보스위치의 리간드별로 정리한 목록입니다.
- 코발라민 리보스위치(또한 B-요소12)는 아데노실코발라민(비타민12 B의 조효소 형태) 또는 아쿠코발라민을 결합하여 코발라민 생합성 및 코발라민 및 유사한 대사물 및 기타 유전자의 수송을 조절한다.
- 고리형 AMP-GMP 리보스위치는 시그널링 분자 고리형 AMP-GMP와 결합합니다.이러한 리보스위치는 고리형 di-GMP-I 리보스위치와 구조적으로 관련되어 있습니다(아래의 「고리형 di-GMP」도 참조).
- 고리형 di-AMP 리보스위치(ydaO/yuA라고도 함)는 신호 분자 고리형 di-AMP와 결합합니다.
- 사이클릭 di-GMP 리보스위치는 시그널링 분자 사이클릭 di-GMP와 결합하여 이 두 번째 메신저에 의해 제어되는 다양한 유전자를 조절합니다.사이클릭 di-GMP 리보스위치에는 사이클릭 di-GMP-I 리보스위치와 사이클릭 di-GMP-II 리보스위치의 2가지 클래스가 있습니다.이들 클래스는 구조적으로 관련이 없는 것으로 보입니다.
- 플루오르화 리보스위치는 플루오르화 이온을 감지하고 높은 수준의 플루오르화물에서도 살아남는 기능을 합니다.
- FMN 리보스위치(FMN-element)는 플라빈 모노뉴클레오티드(FMN)와 결합하여 리보플라빈 생합성 및 수송을 조절합니다.
- glmS 리보스위치, 글루코사민-6-인산 농도가 충분할 때 스스로 분해되는 리보자임입니다.
- 글루타민 리보스위치는 글루타민과 질소 대사에 관여하는 유전자 및 알려지지 않은 기능의 짧은 펩타이드를 조절하기 위해 글루타민과 결합합니다.글루타민 리보스위치에는 glnA RNA 모티브와 다운스트림-펩타이드 모티브의 두 가지 클래스가 알려져 있습니다.이러한 클래스는 구조적으로 관련이 있는 것으로 생각됩니다(이러한 기사의 논의 참조).
- 글리신 리보스위치는 글리신을 결합시켜 글리신 대사 유전자를 에너지원으로서 사용하는 것을 포함하여 글리신 대사 유전자를 조절합니다.2012년 이전에는 이 리보스위치가 연속된 이중 압타머를 포함하고 있기 때문에 협동 결합을 나타내는 유일한 것으로 생각되었다.더 이상 협조적인 것으로 보이지는 않지만, 이중 압타머의 원인은 여전히 [27]모호하다.
- 리신 리보스위치(L-box도 L-box)는 리신을 결합시켜 리신 생합성, 이화 및 수송을 조절합니다.
- 망간 리보스위치는 망간 이온을 결합시킨다.
- NiCo 리보스위치는 니켈과 코발트 금속 이온을 결합시킵니다.
- PreQ1 리보스위치는 프리큐오신을1 결합시켜 이 전구체를 큐오신으로의 합성 또는 운반에 관여하는 유전자를 조절한다.PreQ1 리보스위치에는 PreQ1-I 리보스위치, PreQ1-II 리보스위치 및 PreQ1-II 리보스위치의 3가지 클래스가 있습니다.PreQ1-I 리보스위치의 바인딩 도메인은 자연적으로 발생하는 리보스위치 중에서 비정상적으로 작습니다.Streptoccus와 Lactoccus의 특정 종에서만 볼 수 있는 PreQ1-II 리보스위치는 완전히 다른 구조를 가지고 있으며 PreQ1-II 리보스위치와 마찬가지로 더 크다.
- 푸린 리보스위치는 푸린과 결합하여 푸린 대사와 수송을 조절합니다.퓨린 리보스위치의 다른 형태는 구아닌 또는 아데닌과 결합합니다.구아닌 또는 아데닌의 특이성은 위치 Y74의 리보스위치에서 단일 피리미딘과의 왓슨-크릭 상호작용에 완전히 의존한다.구아닌 리보스위치에서는 이 잔류물이 항상 시토신(C74), 아데닌 잔류물에서는 항상 유라실(U74)이다.퓨린 리보스위치의 상동 유형은 디옥시구아노신을 결합하지만 단일 뉴클레오티드 돌연변이보다 더 큰 차이를 가지고 있다.
- SAH 리보스위치는 S-아데노실호모시스테인과 결합해 S-아데노실메티오닌이 메틸화 반응에 사용될 때 생성되는 이 대사물을 재활용하는 데 관여하는 유전자를 조절한다.
- SAM 리보스위치는 S-아데노실 메티오닌(SAM)을 결합하여 메티오닌과 SAM의 생합성 및 수송을 조절한다.SAM 리보스위치에는 SAM-I(원래는 S박스), SAM-II 및 S박스MK 리보스위치의 3가지가 있습니다.SAM-I는 박테리아에 널리 분포하지만 SAM-II는 알파, 베타 및 일부 감마프로테오박테리아에서만 발견된다.SMK 상자 리보스위치는 Lactobacillales(락토바실랄레스) 순서로만 제공됩니다.이 세 가지 종류의 리보스위치는 순서나 구조 면에서 뚜렷한 유사점이 없습니다.네 번째 변종인 SAM-IV 리보스위치는 SAM-I 리보스위치의 리간드 결합 코어와 유사한 리간드 결합 코어를 가진 것으로 보이지만, 별개의 골격의 맥락이다.
- SAM-SAH 리보스위치는 SAM과 SAH를 모두 유사한 어피니티로 바인드합니다.그들은 항상 메티오닌 아데노실전달효소를 코드하는 유전자를 조절하는 위치에 있기 때문에, SAM에 대한 결합만이 생리적으로 관련이 있다고 제안되었다.
- 테트라하이드로폴산 리보스위치는 테트라하이드로폴산염과 결합하여 합성과 운반 유전자를 조절합니다.
- TPP 리보스위치(THI-box)는 티아민 피로인산염(TPP)을 결합하여 티아민 생합성 및 운반은 물론 유사한 대사물의 운반을 조절합니다.그것은 지금까지 진핵생물에서 [28]발견된 유일한 리보스위치이다.
- ZMP/ZTP 리보스위치는 10-포르밀테트라히드로폴레이트 수치가 낮을 때 데노보 푸린 대사의 부산물인 ZMP와 ZTP를 감지한다.
추정 리보스위치:
- Moco RNA 모티브는 몰리브덴 보조인자를 결합하는 것으로 추정되며, 이 보조인자의 생합성 및 운반에 관여하는 유전자 및 보조인자로 그것 또는 그 유도체를 사용하는 효소를 조절한다.
후보 대사물 결합 리보스위치는 생물 정보학을 사용하여 확인되었으며, 이러한 특징들은 작은 분자를 특이적으로 결합해야 하는 리보스위치의 전형적이기 때문에 중간 정도로 복잡한 2차 구조와 여러 고도로 보존된 뉴클레오티드 위치를 가지고 있다.리보스위치 후보는 또한 단백질 코드 유전자의 5' UTR에 일관되게 위치하고 있으며, 이들 유전자는 또한 대부분의 알려진 리보스위치의 특징이기 때문에 대사물 결합을 암시한다.상기 기준과 매우 일치하는 가설상의 리보스위치 후보는 crcB RNA 모티브, manA RNA 모티브, pfl RNA 모티브, ydaO/yuA 리더, yjdF RNA 모티브, ykkC-yxkD 리더(및 관련된 ykkC-III RNA 모티브) 및 yyP-y 리더이다.이러한 가상의 리보스위치의 기능은 아직 알려지지 않았다.
계산 모델
리보스위치도 실리코 [29][30][31]내 접근방식을 사용하여 조사되었습니다.특히 리보스위치 예측 솔루션은 두 가지 범주로 나눌 수 있습니다.
- 리보스위치 유전자 검출기, 즉 유전자 검사를 통해 리보스위치를 발견하는 것을 목표로 하는 시스템, 주로 모티브 변형 메커니즘을 기반으로 한다.이 그룹에는 Rfam 데이터베이스의 [32]기초 컴포넌트인 Infernal과 RibEx나 [34]RiboSW와 같은[33] 보다 구체적인 툴이 포함됩니다.
- 구성 스위치 예측 변수, 즉 paRNAss,[35] RNAshapes[36] 및 RNAbor와 [37]같은 대체 구조의 구조적 분류에 기초한 방법.또한 ON/OFF 구조 예측을 위한 가족별 접근법도 [38]제안되었다.
SwiSpot[39] 도구는 구조 예측을 사용하여 리보스위치의 존재를 평가하기 때문에 두 그룹을 모두 커버합니다.
RNA 세계 가설
리보스위치는 자연적으로 발생하는 RNA가 작은 분자들을 특이하게 결합할 수 있다는 것을 보여주는데, 이것은 많은 사람들이 이전에 단백질의 영역이라고 믿었던 능력이나 압타머라고 불리는 인공적으로 만들어진 RNA의 능력이다.따라서 생명체의 모든 영역에서 리보스위치의 존재는 원래 RNA만을 사용하여 생명체가 존재했고, 단백질이 나중에 나왔다는 RNA 세계 가설에 약간의 지지를 더합니다; 이 가설은 단백질에 의해 수행되는 모든 중요한 기능이 RNA에 의해 수행될 수 있어야 합니다.일부 리보스위치는 고대 조절 시스템 또는 결합 도메인이 [13][18][40]보존된 RNA 세계 리보자임의 잔여물을 나타낼 수 있다고 제안되어 왔다.
항생제 표적으로
리보스위치는 새로운 항생제의 표적이 될 수 있다.실제로, 수십 년 동안 작용 메커니즘이 알려지지 않았던 일부 항생제는 리보스위치를 [41]목표로 하는 것으로 나타났다.예를 들어 항생제 피리티아민이 세포에 들어가면 피리티아민 피로인산염으로 대사된다.피리티아민 피로인산은 TPP 리보스위치를 결합하고 활성화하여 세포가 TPP의 합성과 수입을 중단시키는 것으로 나타났다.피리티아민 피로인산염은 조효소로서 TPP를 치환하지 않기 때문에 세포는 사멸한다.
엔지니어링된 리보스위치
리보스위치는 자연 유기체의 유전자 발현을 제어하는 효과적인 방법이기 때문에 유전자 [45][46]치료와 같은 산업 및 의료 응용을 위한 인공 리보스위치에[42][43][44] 대한 공학적인 관심이 있었다.
「 」를 참조해 주세요.
- RNA 온도계 - 온도 변동에 따라 구조를 변화시켜 리보솜 결합 부위를 노출 또는 폐쇄하는 또 다른 종류의 mRNA 조절 세그먼트.
레퍼런스
- ^ Nudler E, Mironov AS (January 2004). "The riboswitch control of bacterial metabolism". Trends in Biochemical Sciences. 29 (1): 11–17. doi:10.1016/j.tibs.2003.11.004. PMID 14729327.
- ^ Tucker BJ, Breaker RR (June 2005). "Riboswitches as versatile gene control elements". Current Opinion in Structural Biology. 15 (3): 342–348. doi:10.1016/j.sbi.2005.05.003. PMID 15919195.
- ^ Vitreschak AG, Rodionov DA, Mironov AA, Gelfand MS (January 2004). "Riboswitches: the oldest mechanism for the regulation of gene expression?". Trends in Genetics. 20 (1): 44–50. CiteSeerX 10.1.1.312.9100. doi:10.1016/j.tig.2003.11.008. PMID 14698618.
- ^ Batey RT (June 2006). "Structures of regulatory elements in mRNAs". Current Opinion in Structural Biology. 16 (3): 299–306. doi:10.1016/j.sbi.2006.05.001. PMID 16707260.
- ^ a b Nahvi A, Sudarsan N, Ebert MS, Zou X, Brown KL, Breaker RR (September 2002). "Genetic control by a metabolite binding mRNA". Chemistry & Biology. 9 (9): 1043–1049. doi:10.1016/S1074-5521(02)00224-7. PMID 12323379.
- ^ Sudarsan N, Barrick JE, Breaker RR (June 2003). "Metabolite-binding RNA domains are present in the genes of eukaryotes". RNA. 9 (6): 644–647. doi:10.1261/rna.5090103. PMC 1370431. PMID 12756322.
- ^ Nou X, Kadner RJ (June 2000). "Adenosylcobalamin inhibits ribosome binding to btuB RNA". Proceedings of the National Academy of Sciences of the United States of America. 97 (13): 7190–7195. Bibcode:2000PNAS...97.7190N. doi:10.1073/pnas.130013897. PMC 16521. PMID 10852957.
- ^ a b Gelfand MS, Mironov AA, Jomantas J, Kozlov YI, Perumov DA (November 1999). "A conserved RNA structure element involved in the regulation of bacterial riboflavin synthesis genes". Trends in Genetics. 15 (11): 439–442. doi:10.1016/S0168-9525(99)01856-9. PMID 10529804.
- ^ a b Miranda-Ríos J, Navarro M, Soberón M (August 2001). "A conserved RNA structure (thi box) is involved in regulation of thiamin biosynthetic gene expression in bacteria". Proceedings of the National Academy of Sciences of the United States of America. 98 (17): 9736–9741. doi:10.1073/pnas.161168098. PMC 55522. PMID 11470904.
- ^ Stormo GD, Ji Y (August 2001). "Do mRNAs act as direct sensors of small molecules to control their expression?". Proceedings of the National Academy of Sciences of the United States of America. 98 (17): 9465–9467. Bibcode:2001PNAS...98.9465S. doi:10.1073/pnas.181334498. PMC 55472. PMID 11504932.
- ^ Gold L, Brown D, He Y, Shtatland T, Singer BS, Wu Y (January 1997). "From oligonucleotide shapes to genomic SELEX: novel biological regulatory loops". Proceedings of the National Academy of Sciences of the United States of America. 94 (1): 59–64. Bibcode:1997PNAS...94...59G. doi:10.1073/pnas.94.1.59. PMC 19236. PMID 8990161.
- ^ Mironov AS, Gusarov I, Rafikov R, Lopez LE, Shatalin K, Kreneva RA, Perumov DA, Nudler E (November 2002). "Sensing small molecules by nascent RNA: a mechanism to control transcription in bacteria". Cell. 111 (5): 747–756. doi:10.1016/S0092-8674(02)01134-0. PMID 12464185.
- ^ a b Winkler W, Nahvi A, Breaker RR (October 2002). "Thiamine derivatives bind messenger RNAs directly to regulate bacterial gene expression". Nature. 419 (6910): 952–956. Bibcode:2002Natur.419..952W. doi:10.1038/nature01145. PMID 12410317. S2CID 4408592.
- ^ Winkler WC, Cohen-Chalamish S, Breaker RR (December 2002). "An mRNA structure that controls gene expression by binding FMN". Proceedings of the National Academy of Sciences of the United States of America. 99 (25): 15908–15913. Bibcode:2002PNAS...9915908W. doi:10.1073/pnas.212628899. PMC 138538. PMID 12456892.
- ^ Grundy FJ, Henkin TM (November 1998). "The S box regulon: a new global transcription termination control system for methionine and cysteine biosynthesis genes in gram-positive bacteria". Molecular Microbiology. 30 (4): 737–749. doi:10.1046/j.1365-2958.1998.01105.x. PMID 10094622.
- ^ Franklund CV, Kadner RJ (June 1997). "Multiple transcribed elements control expression of the Escherichia coli btuB gene". Journal of Bacteriology. 179 (12): 4039–4042. doi:10.1128/jb.179.12.4039-4042.1997. PMC 179215. PMID 9190822.
- ^ Barrick JE, Corbino KA, Winkler WC, Nahvi A, Mandal M, Collins J, Lee M, Roth A, Sudarsan N, Jona I, Wickiser JK, Breaker RR (April 2004). "New RNA motifs suggest an expanded scope for riboswitches in bacterial genetic control". Proceedings of the National Academy of Sciences of the United States of America. 101 (17): 6421–6426. Bibcode:2004PNAS..101.6421B. doi:10.1073/pnas.0308014101. PMC 404060. PMID 15096624.
- ^ a b Corbino KA, Barrick JE, Lim J, Welz R, Tucker BJ, Puskarz I, Mandal M, Rudnick ND, Breaker RR (2005). "Evidence for a second class of S-adenosylmethionine riboswitches and other regulatory RNA motifs in alpha-proteobacteria". Genome Biology. 6 (8): R70. doi:10.1186/gb-2005-6-8-r70. PMC 1273637. PMID 16086852.
- ^ Weinberg Z, Barrick JE, Yao Z, Roth A, Kim JN, Gore J, Wang JX, Lee ER, Block KF, Sudarsan N, Neph S, Tompa M, Ruzzo WL, Breaker RR (2007). "Identification of 22 candidate structured RNAs in bacteria using the CMfinder comparative genomics pipeline". Nucleic Acids Research. 35 (14): 4809–4819. doi:10.1093/nar/gkm487. PMC 1950547. PMID 17621584.
- ^ Weinberg Z, Wang JX, Bogue J, Yang J, Corbino K, Moy RH, Breaker RR (March 2010). "Comparative genomics reveals 104 candidate structured RNAs from bacteria, archaea, and their metagenomes". Genome Biology. 11 (3): R31. doi:10.1186/gb-2010-11-3-r31. PMC 2864571. PMID 20230605.
- ^ Cheah MT, Wachter A, Sudarsan N, Breaker RR (May 2007). "Control of alternative RNA splicing and gene expression by eukaryotic riboswitches". Nature. 447 (7143): 497–500. Bibcode:2007Natur.447..497C. doi:10.1038/nature05769. PMID 17468745. S2CID 4393918.
- ^ Wachter A, Tunc-Ozdemir M, Grove BC, Green PJ, Shintani DK, Breaker RR (November 2007). "Riboswitch control of gene expression in plants by splicing and alternative 3' end processing of mRNAs". The Plant Cell. 19 (11): 3437–3450. doi:10.1105/tpc.107.053645. PMC 2174889. PMID 17993623.
- ^ Bocobza S, Adato A, Mandel T, Shapira M, Nudler E, Aharoni A (November 2007). "Riboswitch-dependent gene regulation and its evolution in the plant kingdom". Genes & Development. 21 (22): 2874–2879. doi:10.1101/gad.443907. PMC 2049190. PMID 18006684.
- ^ André G, Even S, Putzer H, Burguière P, Croux C, Danchin A, Martin-Verstraete I, Soutourina O (October 2008). "S-box and T-box riboswitches and antisense RNA control a sulfur metabolic operon of Clostridium acetobutylicum". Nucleic Acids Research. 36 (18): 5955–5969. doi:10.1093/nar/gkn601. PMC 2566862. PMID 18812398.
- ^ Loh E, Dussurget O, Gripenland J, Vaitkevicius K, Tiensuu T, Mandin P, Repoila F, Buchrieser C, Cossart P, Johansson J (November 2009). "A trans-acting riboswitch controls expression of the virulence regulator PrfA in Listeria monocytogenes". Cell. 139 (4): 770–779. doi:10.1016/j.cell.2009.08.046. PMID 19914169.
- ^ http://2011.igem.org/Team:BYU_Provo/Results
- ^ Sherman EM, Esquiaqui J, Elsayed G, Ye JD (March 2012). "An energetically beneficial leader-linker interaction abolishes ligand-binding cooperativity in glycine riboswitches". RNA. 18 (3): 496–507. doi:10.1261/rna.031286.111. PMC 3285937. PMID 22279151.
- ^ Bocobza SE, Aharoni A (October 2008). "Switching the light on plant riboswitches". Trends Plant Sci. 13 (10): 526–33. doi:10.1016/j.tplants.2008.07.004. PMID 18778966.
- ^ Barrick JE (2009). "Predicting riboswitch regulation on a genomic scale". Riboswitches. Methods in Molecular Biology. Vol. 540. pp. 1–13. doi:10.1007/978-1-59745-558-9_1. ISBN 978-1-934115-88-6. PMID 19381548.
- ^ Barash D, Gabdank I (January 2010). "Energy minimization applied to riboswitches: a perspective and challenges". RNA Biology. 7 (1): 90–97. doi:10.4161/rna.7.1.10657. PMID 20061789.
- ^ Chen, Shi-Jie; Burke, Donald H; Adamiak, R W (2015). Computational methods for understanding riboswitches / Methods in Enzymology, vol 553. Academic Press. ISBN 978-0-12-801618-3.
- ^ Nawrocki EP, Kolbe DL, Eddy SR (May 2009). "Infernal 1.0: inference of RNA alignments". Bioinformatics. 25 (10): 1335–1337. doi:10.1093/bioinformatics/btp157. PMC 2732312. PMID 19307242.
- ^ Abreu-Goodger C, Merino E (July 2005). "RibEx: a web server for locating riboswitches and other conserved bacterial regulatory elements". Nucleic Acids Research. 33 (Web Server issue): W690-2. doi:10.1093/nar/gki445. PMC 1160206. PMID 15980564.
- ^ Chang TH, Huang HD, Wu LC, Yeh CT, Liu BJ, Horng JT (July 2009). "Computational identification of riboswitches based on RNA conserved functional sequences and conformations". RNA. 15 (7): 1426–1430. doi:10.1261/rna.1623809. PMC 2704089. PMID 19460868.
- ^ Voss B, Meyer C, Giegerich R (July 2004). "Evaluating the predictability of conformational switching in RNA". Bioinformatics. 20 (10): 1573–1582. doi:10.1093/bioinformatics/bth129. PMID 14962925.
- ^ Janssen S, Giegerich R (February 2015). "The RNA shapes studio". Bioinformatics. 31 (3): 423–425. doi:10.1093/bioinformatics/btu649. PMC 4308662. PMID 25273103.
- ^ Freyhult E, Moulton V, Clote P (August 2007). "Boltzmann probability of RNA structural neighbors and riboswitch detection". Bioinformatics. 23 (16): 2054–2062. doi:10.1093/bioinformatics/btm314. PMID 17573364.
- ^ Clote P, Lou F, Lorenz WA (April 2012). "Maximum expected accuracy structural neighbors of an RNA secondary structure". BMC Bioinformatics. 13 Suppl 5 (Suppl 5): S6. doi:10.1186/1471-2105-13-S5-S6. PMC 3358666. PMID 22537010.
- ^ Barsacchi M, Novoa EM, Kellis M, Bechini A (November 2016). "SwiSpot: modeling riboswitches by spotting out switching sequences". Bioinformatics. 32 (21): 3252–3259. doi:10.1093/bioinformatics/btw401. PMID 27378291.
- ^ Cochrane JC, Strobel SA (June 2008). "Riboswitch effectors as protein enzyme cofactors". RNA. 14 (6): 993–1002. doi:10.1261/rna.908408. PMC 2390802. PMID 18430893.
- ^ Blount KF, Breaker RR (December 2006). "Riboswitches as antibacterial drug targets". Nature Biotechnology. 24 (12): 1558–1564. doi:10.1038/nbt1268. PMID 17160062. S2CID 34398395.
- ^ Bauer G, Suess B (June 2006). "Engineered riboswitches as novel tools in molecular biology". Journal of Biotechnology. 124 (1): 4–11. doi:10.1016/j.jbiotec.2005.12.006. PMID 16442180.
- ^ Dixon N, Duncan JN, Geerlings T, Dunstan MS, McCarthy JE, Leys D, Micklefield J (February 2010). "Reengineering orthogonally selective riboswitches". Proceedings of the National Academy of Sciences of the United States of America. 107 (7): 2830–2835. Bibcode:2010PNAS..107.2830D. doi:10.1073/pnas.0911209107. PMC 2840279. PMID 20133756.
- ^ Verhounig A, Karcher D, Bock R (April 2010). "Inducible gene expression from the plastid genome by a synthetic riboswitch". Proceedings of the National Academy of Sciences of the United States of America. 107 (14): 6204–6209. Bibcode:2010PNAS..107.6204V. doi:10.1073/pnas.0914423107. PMC 2852001. PMID 20308585.
- ^ Ketzer P, Kaufmann JK, Engelhardt S, Bossow S, von Kalle C, Hartig JS, Ungerechts G, Nettelbeck DM (February 2014). "Artificial riboswitches for gene expression and replication control of DNA and RNA viruses". Proceedings of the National Academy of Sciences of the United States of America. 111 (5): E554–562. Bibcode:2014PNAS..111E.554K. doi:10.1073/pnas.1318563111. PMC 3918795. PMID 24449891.
- ^ Strobel B, Klauser B, Hartig JS, Lamla T, Gantner F, Kreuz S (October 2015). "Riboswitch-mediated Attenuation of Transgene Cytotoxicity Increases Adeno-associated Virus Vector Yields in HEK-293 Cells". Molecular Therapy. 23 (10): 1582–1591. doi:10.1038/mt.2015.123. PMC 4817922. PMID 26137851.
추가 정보
- Ferré-D'Amaré, Adrian R.; Winkler, Wade C. (2011). "Chapter 5. The Roles of Metal Ions in Regulation by Riboswitches". In Astrid Sigel, Helmut Sigel and Roland K. O. Sigel (ed.). Structural and catalytic roles of metal ions in RNA. Metal Ions in Life Sciences. Vol. 9. Cambridge, U.K.: RSC Publishing. pp. 141–173. doi:10.1039/9781849732512-00141. ISBN 978-1-84973-094-5. PMC 3454353. PMID 22010271.