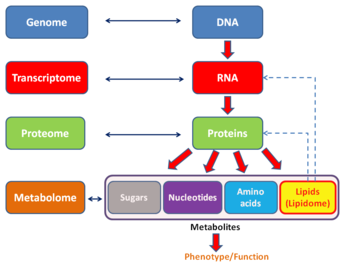
문자 변환기
Transcriptome트랜스크립텀은 세포 집단 또는 개인에서 코딩과 비코딩을 포함한 모든 RNA 전사의 집합이다.이 용어는 특정 실험에 따라 모든 RNA 또는 mRNA를 지칭하는 데 사용될 수도 있습니다.transcriptome이라는 용어는 transcript와 genome의 합성어이다. 이것은 전사의 생물학적 과정에서의 transcript 생성 과정과 관련이 있다.
트랜스크립텀 주석의 초기 단계는 1980년대에 발표된 cDNA 라이브러리에서 시작되었다.그 후, 높은 처리량 기술의 등장으로 트랜스크립텀에 대한 데이터를 보다 빠르고 효율적으로 얻을 수 있게 되었습니다.두 가지 생물학적 기술, 즉 교배 기반 기술인 DNA 마이크로어레이와 배열 기반 [1]접근인 RNA-seq를 연구하기 위해 사용된다.RNA-seq는 선호되는 방법이며 2010년대부터 지배적인 전사체학 기술이다.단세포 전사체학에서는 개별 세포 내에서 시간에 따른 전사체 변화를 추적할 수 있습니다.
전사체로부터 얻은 데이터는 세포분화, 발암, 전사조절 및 바이오마커 발견과 같은 과정에 대한 통찰력을 얻기 위해 연구에 사용된다.또한 트랜스스크립트옴 보유 데이터는 진화 과정 중 및 시험관 내 수정에서 계통학적 관계를 확립하는 데에도 적용된다.트랜스크립텀은 다른 -ome 기반 생물학적 연구 분야와 밀접하게 관련되어 있으며, 프로테옴과 대사체를 보완하고 특정 유형의 RNA 트랜스크립트를 연구하는 오메필드로 볼 수 있는 트랜스라톰, 엑소좀, 마이옴 및 타나토트랜스크립텀을 포함합니다.는 Transcriptome과 다른 -om 사이의 정량화 및 보존 관계이며, Transcriptomics 데이터는 대사물과 [2]같은 다른 분자 종을 예측하는 데 효과적으로 사용될 수 있습니다.공개적으로 이용할 수 있는 수많은 트랜스크립텀 데이터베이스가 있습니다.
어원과 역사
transcriptome이라는 단어는 transcript와 genome의 합성어이다.그것은 접미사 -ome과 -omics를 사용하여 형성된 다른 신조어와 함께 나타나 생명과학 및 기술 분야에서 게놈 전체에 걸쳐 수행된 모든 연구를 의미했다.이처럼, 트랜스크립트옴과 트랜스크립트옴은 게놈,[3] 프로테옴과 함께 등장한 최초의 단어 중 하나였다.누에나방 mRNA를 위한 cDNA 라이브러리 컬렉션의 사례를 제시한 첫 번째 [4]연구는 1979년에 발표되었다.유기체의 트랜스크립텀을 언급하고 조사한 최초의 정설 연구는 1997년에 발표되었으며, 그것은 유전자 발현의 연속 분석을 사용하여 60,633개의 트랜스크립트를 S.[5] cerevisiae로 표현하였다.높은 처리량 기술 및 생물 정보학의 발전과 그에 따른 계산 능력의 증가로 인해 엄청난 양의 데이터를 [3]더욱 효율적이고 쉽게 특성화하고 분석할 수 있게 되었습니다.1980년대 자동화된 DNA 염기서열 분석의 등장으로 인해 트랜스크립텀의 특성을 분석하려는 시도가 더욱 두드러졌다.[6]1990년대에는 발현된 배열 태그 시퀀싱이 유전자와 그 [7]단편들을 식별하기 위해 사용되었다.이어 유전자 발현 직렬 분석(SAGE), 유전자 발현 캡 분석(CAGE), 대량 병렬 시그니처 배열 분석(MPSS) 등의 기술이 이어졌다.
문자 변환
transcriptome은 주어진 생물 또는 실험 [8]샘플에 존재하는 모든 리보핵산(RNA) transcript를 포함합니다.RNA는 DNA를 유기체의 표현형으로 바꾸는 과정을 담당하는 유전 정보의 주요 운반체이다.유전자는 전사라고 알려진 분자 과정을 통해 단일 가닥 메신저 RNA를 발생시킬 수 있다; 이 mRNA는 그것이 [6]유래한 DNA 가닥에 상보적이다.RNA 중합효소 II는 템플릿 DNA 가닥에 부착되고 mRNA [9]전사체의 성장 배열의 3' 말단에 리보뉴클레오티드의 첨가를 촉매한다.
그 기능을 시작하기 위해 RNA 중합효소 II는 유전자의 상류(5')에 위치한 프로모터 배열을 인식할 필요가 있다.진핵생물에서 이 과정은 TATA 박스를 인식하고 적절한 시작 부위에 RNA 중합효소를 배치하는 데 도움이 되는 전사인자 II D(TFIID)에 의해 매개된다.RNA 전사체의 생산을 완료하기 위해, 종단은 보통 종단 배열로부터 수백 개의 핵핵 코타이드에서 일어나며, [9]분열이 일어난다.이 과정은 세포질로 옮겨지기 전에 안정성을 높이기 위해 mRNA 분자의 캡, 스플라이스 및 폴리아데닐화를 통해 이루어지는 RNA 처리와 함께 세포의 핵에서 발생합니다.mRNA는 리보솜에서 일어나는 번역 과정을 통해 단백질을 발생시킨다.
RNA전사의 종류
분자생물학의 중심 교의에 따라, 트랜스크립텀은 처음에 단백질 코드 mRNA 트랜스크립트만을 포함했다.그럼에도 불구하고, 뚜렷한 기능을 가진 몇 가지 RNA 아형이 존재한다.많은 RNA 전사는 단백질을 코드하지 않거나 유전자 전사와 번역 과정에서 다른 조절 기능을 가지고 있다.분자생물학의 중심 교의 범위에 속하지 않는 RNA 유형은 긴 비부호화 RNA와 짧은 비부호화 RNA의 두 그룹으로 나눌 수 있는 비부호화 RNA이다.
긴 비부호화 RNA는 200 뉴클레오티드보다 긴 모든 비부호화 RNA 전사를 포함한다.이 그룹의 구성원은 코드화되지 않은 문자 중 가장 큰 부분을 구성합니다.짧은 비코드 RNA에는 다음과 같은 구성 요소가 포함됩니다.
연구 범위
인간 게놈에서는 모든 유전자의 약 5%가 [8]RNA로 전사된다.전사체는 전체의 약 1~4%를 차지하는 코드화 mRNA와 나머지 게놈을 구성하며 단백질을 [11][12]생성하지 않는 비코드화 RNA로 구성됩니다.비단백질 코드 배열의 수는 더 복잡한 [13]유기체에서 증가한다.
몇몇 요소들이 대본의 내용을 확립하기 어렵게 만든다.여기에는 대체 스플라이싱,[13] RNA 편집 및 대체 전사가 포함됩니다.또, 트랜스크립텀의 함유량은 [6]분화중에 변화할 수 있지만, 트랜스크립텀 기술은 특정 시점에서 샘플에서 발생하는 전사를 포착할 수 있다.전사체학(transcriptomics)의 주요 목적은 다음과 같다. "mRNA, 비부호화 RNA 및 작은 RNA를 포함한 모든 전사종을 카탈로그화한다. 시작 부위, 5' 및 3' 말단, 스플라이싱 패턴 및 기타 전사 후 수정의 관점에서 유전자의 전사 구조를 결정하고, 발현 수준의 변화를 정량화한다.다른 조건 하에서 개발 중에 각 사본의 내용을 확인합니다."[1]
이 용어는 특정 유기체의 총 전사 세트 또는 특정 세포 유형에 존재하는 전사 서브셋에 적용할 수 있습니다.특정 세포주(돌연변이 제외)에 대해 대략 고정된 게놈과 달리, 트랜스크립텀은 외부 환경 조건에 따라 달라질 수 있다.그것은 세포 내의 모든 mRNA 전사물을 포함하기 때문에, 전사체는 전사 감쇠와 같은 mRNA 분해 현상을 제외하고, 주어진 시간에 활발하게 발현되는 유전자를 반영한다.(발현 프로파일링, 스플라이스 변형 분석 등을 포함하는) 전사체 연구는 종종 mRNA에 초점을 맞추고 때로는 tRNA와 sRNA와 같은 다른 것을 포함하는 주어진 세포 집단에서 RNA의 발현 수준을 조사한다.
공법
전사체학은 개체(게놈의 전사체)에 문자열(읽기) 목록을 할당하는 것을 포괄하는 정량 과학이다.표현 강도를 계산하기 위해 각 오브젝트에 대응하는 판독 밀도를 [14]카운트한다.처음에는 발현 배열 태그 라이브러리와 유전자 발현 직렬 및 캡 분석(SAGE)을 사용하여 전사체를 분석 및 연구했다.
현재 두 가지 주요 전사체 기술은 DNA 마이크로어레이와 RNA-Seq를 포함한다.두 기술 모두 RNA 추출 기술을 통한 RNA 분리를 필요로 하며, 다른 세포 성분으로부터의 분리 및 mRNA의 [15][16]농축이 뒤따른다.
트랜스크립텀 시퀀스를 추론하는 두 가지 일반적인 방법이 있습니다.하나의 접근법은 염기서열을 유기체 자체(transcriptome이 연구되고 있는) 또는 밀접하게 관련된 종의 참조 게놈으로 읽는다.또 다른 접근법인 de novo transcriptome assembly는 짧은 염기서열 판독으로부터 직접 전사물을 추론하는 소프트웨어를 사용하며 염기서열이 [17]없는 게놈을 가진 유기체에 사용된다.
DNA마이크로어레이
최초의 전사체 연구는 마이크로어레이 기술 (DNA 칩으로도 알려져 있음)에 기초했다.마이크로어레이는 "프로브"로 알려진 올리고뉴클레오티드가 배열된 점들이 있는 얇은 유리층으로 구성되어 있습니다; 각각의 점들은 알려진 DNA [18]서열을 포함합니다.
마이크로 어레이 분석을 수행할 때 mRNA는 대조군과 실험 샘플에서 수집되며, 실험 샘플은 보통 질병을 나타낸다.관심 RNA는 안정성을 높이기 위해 cDNA로 변환되고 두 그룹에 대해 보통 녹색과 빨간색의 두 가지 색상의 형광체로 표시됩니다.cDNA는 마이크로 어레이의 표면에 퍼져 칩의 올리고뉴클레오티드와 교배하고 레이저를 사용하여 스캔합니다.마이크로어레이의 각 지점에서의 형광 강도는 유전자 발현 수준에 해당하며, 선택된 형광구의 색상에 따라 샘플 중 관심 있는 [7]mRNA의 높은 수준을 나타내는 것을 결정할 수 있다.
하나의 마이크로 어레이는 일반적으로 모든 알려진 유전자를 나타내기에 충분한 올리고뉴클레오티드를 포함하고 있다; 그러나 마이크로어레이를 사용하여 얻은 데이터는 알려지지 않은 유전자에 대한 정보를 제공하지 않는다.2010년대에 마이크로어레이는 DNA 염기서열 분석을 기반으로 하는 차세대 기술로 거의 완전히 대체되었다.
RNA순서결정
RNA 배열 기술은 차세대 배열 기술입니다. 따라서 소량의 RNA만 필요로 하며 [3]게놈에 대한 사전 지식은 없습니다.이는 RNA 전사물의 정성적 및 정량적 분석을 모두 가능하게 하며, 전자는 새로운 전사물의 발견을 가능하게 하고 후자는 [10]샘플에서 전사물에 대한 상대적인 양의 측정을 가능하게 한다.
생물학적 샘플의 전사체를 배열하는 세 가지 주요 단계는 RNA 정제, RNA 또는 cDNA 라이브러리의 합성 및 라이브러리 [10]배열 지정입니다.RNA 정제 과정은 짧은 RNA와 [10]긴 RNA가 다르다.이 단계는 보통 DNA와 같은 오염물질이나 샘플 처리와 관련된 기술적 오염물질을 피하기 위해 RNA 품질 평가가 뒤따릅니다.RNA 품질은 260 [19]nm의 흡광도 피크를 가진 UV 분광법을 사용하여 측정됩니다.RNA 무결성 번호([19]RIN) 점수에 보고된 28S RNA 대 18S RNA의 비율과 강도를 정량적으로 비교할 수도 있습니다.mRNA는 관심종이며 전체 함량의 3%에 불과하므로, RNA 샘플은 rRNA 및 tRNA 및 조직 특이적 RNA [19]전사물을 제거하기 위해 처리되어야 한다.
짧은 cDNA 단편을 생성하는 것을 목적으로 하는 라이브러리 준비 단계는 50에서 300개의 염기쌍 사이의 길이의 전사물에 대한 RNA 조각화에서 시작합니다.단편화는 효소적(RNA 엔도핵산가수분해효소), 화학적(트리마그네슘 소금 완충제, 화학적 가수분해) 또는 기계적(소음화, 분무)[20]일 수 있다.역전사는 RNA 템플릿을 cDNA로 변환하기 위해 사용되며, 이를 달성하기 위해 랜덤 프라이머를 사용하거나 특수 어댑터 올리고를 결합하는 세 가지 프라이밍 방법을 사용할 수 있습니다.
단세포 전사체학
전사는 또한 단세포 전사체학에 의해 개별 세포 수준에서 연구될 수 있다.단세포 RNA 배열 분석(scRNA-seq)은 단세포의 전사체 분석을 가능하게 하는 최근 개발된 기술이다.단세포 전사체학에서는 관심 조직을 구성하는 세포 유형의 하위 집단도 [21]고려된다.이 접근방식은 실험 샘플의 변화가 증식과 반대로 표현형 세포 변화에 기인하는지 여부를 확인할 수 있으며,[22] 이 경우 표본에서 특정 세포 유형이 과도하게 발현될 수 있다.또한 분화를 통한 세포진행을 평가할 때 평균발현 프로파일은 세포가 발달하는 단계가 아닌 시간에 의해서만 세포를 정렬할 수 있으며, 결과적으로 특정 [23]단계에 특정한 유전자 발현 수준의 추세를 나타낼 수 없다.단세포 트란스크립토믹 기술은 순환하는 종양 세포, 고형 종양의 암 줄기세포, 포유류의 [24]배반포의 배아줄기세포(ESC)와 같은 희귀 세포군을 특징짓기 위해 사용되어 왔다.
단세포 전사체학을 위한 표준화된 기술은 없지만, 몇 가지 단계를 수행해야 합니다.첫 번째 단계에는 셀 분리가 포함됩니다.셀 분리는 저스루풋 및 고스루풋 기술을 사용하여 실행할 수 있습니다.이어서 qPCR 단계를 거쳐 관심 RNA가 cDNA로 변환되는 단세포 RNAseq를 수행합니다.단세포 전사체학의 새로운 개발은 얇은 조직 조각의 저온 절단 및 각 조각의 전사체 배열 분석을 통해 조직 및 세포 내 위치 파악을 가능하게 한다.또 다른 기술은 현미경으로 단일 트랜스크립트를 시각화할 수 있도록 하며,[24] 그것들이 발현되는 각 개별 세포의 공간 정보를 보존합니다.
분석.
다른 세포 집단에서 다르게 발현되는 유전자의 식별을 돕기 위해 많은 유기체 특이적 전사체 데이터베이스가 구성되고 주석을 달았다.
RNA-seq는 DNA 마이크로어레이의 오래된 기술이 여전히 [1]사용되고 있지만(2013년) 유기체의 전사체 측정을 위한 선택 방법으로 떠오르고 있다.RNA-seq는 긴 RNA를 cDNA 단편 라이브러리로 변환함으로써 특정 유전자의 전사를 측정합니다.그런 다음 cDNA 조각은 높은 처리량 배열 기술을 사용하여 배열되고 [1]유전자의 발현 프로파일을 만들기 위해 사용되는 참조 게놈 또는 전사체에 정렬됩니다.
적용들
포유동물
줄기세포와 암세포의 전사체는 세포 분화와 발암 과정을 이해하려는 연구자들에게 특히 흥미롭다.RNA-seq 또는 유전자 배열 데이터를 이용한 파이프라인은 줄기세포 및 전구세포에서 발생하는 유전자 변화를 추적하기 위해 사용될 수 있으며, 전세포형 및 성숙한 [25]세포로부터 적어도 3개의 독립된 유전자 발현 데이터를 필요로 한다.
인간 난모세포와 배아의 전사체 분석은 초기 배아 발달을 제어하는 분자 메커니즘과 신호 경로를 이해하기 위해 사용되며, 이론적으로 체외 [citation needed]수정에서 적절한 배아 선택을 하는 데 강력한 도구가 될 수 있다.시험관내 수정 및 배아 전이(IVT-ET)에서 임신 첫 3개월 태반의 전사체 함량 분석 결과, 더 높은 빈도의 신생아 주변 결과와 관련된 유전자 발현 차이가 밝혀졌다.이러한 통찰력은 [26]관행을 최적화하는 데 사용될 수 있습니다.또한 트랜스크립텀 분석을 사용하여 [27]프로세스와 관련된 손상을 감소시킴으로써 난모세포의 저온 보존을 최적화할 수 있다.
트랜스스크립토믹스는 의약품의 안전성 평가 또는 화학적 위험 평가에 사용되는 바이오마커 [28]발견에서 부상하고 지속적으로 성장하는 분야이다.
트랜스스크립텀은 또한 개인 간의 계통발생적 관계를 추론하거나 트랜스스크립텀 [29]보존의 진화적 패턴을 검출하기 위해 사용될 수 있다.
전사체 분석은 안티센스 전사의 발생률, 주변 유전자와의 상호작용을 통한 유전자 발현에서의 그들의 역할, 그리고 [30]다른 염색체에서의 풍부함을 발견하기 위해 사용되었다.RNA-seq는 또한 같은 유전자에서 유래했지만 구조가 다른 RNA 동질체가 제한된 [17]게놈에서 복잡한 표현형을 어떻게 생산할 수 있는지를 보여주기 위해 사용되었다.
식물
트랜스크립텀 분석은 식물 종의 진화와 다양화 과정을 연구하는데 사용되어 왔다.2014년에는 비리디플랜트과, 글라우코피타과, 진달래과의 1,124종의 식물의 전사체 배열을 분석하는 '1000식물 게놈 프로젝트'가 완료되었다.단백질 코딩 시퀀스는 이후 식물 사이의 계통발생적 관계를 추론하고 [31]진화 과정에서 다양화 시기를 특징짓기 위해 비교되었다.성숙한 꽃가루의 유전자 발현을 특징짓고 정량화하기 위해 트랜스크립텀 연구가 사용되어 왔다.세포벽 대사 및 세포골격에 관여하는 유전자가 과도하게 발현된 것으로 밝혀졌다.트랜스크립텀 접근법은 또한 미세 포자에서 성숙한 꽃가루 알갱이에 이르는 꽃가루의 다양한 발달 단계를 통해 유전자 발현의 변화를 추적할 수 있게 해주었다; 추가적으로 그러한 단계는 아라비도시스, 쌀 그리고 [32]담배를 포함한 다른 식물의 종에 걸쳐 비교될 수 있다.
다른 OME 필드와의 관계
다른 -ome 기반 기술과 마찬가지로, transcriptome의 분석은 가설을 실험적으로 검증할 때 편향되지 않은 접근을 가능하게 한다.이 접근방식은 신호 [14]경로에서 새로운 매개자를 발견할 수도 있습니다.다른 -omics 기반 기술과 마찬가지로, Transcriptome은 다중체학 접근의 범위 내에서 분석될 수 있습니다.이는 대사체학과는 상호보완적이지만 단백질체학과는 달리 전사물과 대사물 사이의 직접적인 연관성을 확립할 수 없다.
문자 변환기의 하위 범주로 볼 수 있는 여러 개의 -ome 필드가 있습니다.엑솜은 특정 세포 집단에서 발견되는 RNA 분자만을 포함한다는 점에서 전사체와 다르며, 보통 분자 정체성 외에 각 RNA 분자의 양이나 농도를 포함합니다.또한 트랜스크리티폼은 번역 중인 RNA의 집합인 트랜스크리티폼과 다르다.
마이옴이라는 용어는 기능 유전체학에서 [33]감수분열 과정에서 생성된 감수성 전사체 또는 RNA 전사체 세트를 설명하기 위해 사용된다.감수분열은 진핵생물의 성생식의 주요 특징이며 상동 염색체, 시냅스, 재조합을 포함한다.대부분의 생물에서 감수분열은 단기간에 발생하기 때문에 감수생물 전사 프로파일링은 감수생물 세포(마이오사이트)의 분리(또는 농축)의 도전으로 인해 어렵다.전사체 분석과 마찬가지로 마이옴은 대규모 전사체 [34]기술을 사용하여 전체 유전자 수준에서 연구될 수 있다.마이옴은 포유류와 효모계에서 잘 특징지어지고 식물에서는 [35]다소 덜 광범위하게 특징지어진다.
타나토트랜스크립텀은 사망 후 24시간에서 48시간 동안 사체의 내부 장기에서 계속 발현되거나 다시 발현되기 시작하는 모든 RNA 전사체로 구성됩니다.일부 유전자는 태아 발달 후 억제되는 유전자를 포함한다.만약 타나토트랜스포트롬이 프로그램된 세포사멸(아포토시스) 과정과 관련되어 있다면, 그것은 아포토시스 타나토트랜스포트롬으로 언급될 수 있다.타나토트랜스포트롬의 분석은 [36]법의학에서 사용된다.
eQTL 매핑은 유전체학을 전사체학으로 보완하기 위해 사용될 수 있습니다. DNA 수준의 유전자 변형 및 RNA [37]수준의 유전자 발현 측정입니다.
프로테옴과의 관계
트랜스크립텀은 단백질의 서브셋, 즉 게놈에 의해 발현되는 단백질의 전체 세트로 볼 수 있습니다.
그러나 상대적으로 mRNA 발현의 작은 변화가 세포 내에 존재하는 해당 단백질의 총량에 큰 변화를 일으킬 수 있다는 사실에 의해 상대적인 mRNA 발현 수준의 분석은 복잡해질 수 있다.유전자 세트 농축 분석으로 알려진 한 분석 방법은 다른 세포 [1]집단에서 업 또는 다운 조절되는 개별 유전자보다는 코어 조절된 유전자 네트워크를 식별합니다.
미세 배열 연구는 세포에서 다른 mRNA의 상대적 양을 밝힐 수 있지만, mRNA의 수준은 그들이 [38]코드화하는 단백질의 발현 수준에 직접적으로 비례하지 않습니다.주어진 mRNA 분자를 템플릿으로 사용하여 합성되는 단백질 분자의 수는 mRNA 배열의 번역 개시 특성에 크게 의존하며, 특히 번역 개시 배열의 능력은 단백질 번역을 위한 리보솜의 모집에 있어 중요한 결정 요인이다.
트랜스크립트옴 데이터베이스
「 」를 참조해 주세요.
메모들
- ^ a b c d Wang, Zhong; Gerstein, Mark; Snyder, Michael (January 2009). "RNA-Seq: a revolutionary tool for transcriptomics". Nature Reviews Genetics. 10 (1): 57–63. doi:10.1038/nrg2484. PMC 2949280. PMID 19015660.
- ^ Cavicchioli, Maria Vittoria; Santorsola, Mariangela; Balboni, Nicola; Mercatelli, Daniele; Giorgi, Federico Manuel (January 2022). "Prediction of Metabolic Profiles from Transcriptomics Data in Human Cancer Cell Lines". International Journal of Molecular Sciences. 23 (7): 3867. doi:10.3390/ijms23073867. ISSN 1422-0067. PMID 35409231.
- ^ a b c d e Jiménez-Chillarón, Josep C.; Díaz, Rubén; Ramón-Krauel, Marta (2014). "Chapter 4 - Omics Tools for the Genome-Wide Analysis of Methylation and Histone Modifications". Comprehensive Analytical Chemistry. 64: 81–110. doi:10.1016/B978-0-444-62651-6.00004-0. ISBN 9780444626516. Retrieved 25 April 2020.
- ^ GK, Sim; FC, Kafatos; CW, Jones; MD, Koehler; A, Efstratiadis; T., Maniatis (December 1979). "Use of a cDNA library for studies on evolution and developmental expression of the chorion multigene families". Cell. 8 (4): 1303–16. doi:10.1016/0092-8674(79)90241-1. PMID 519770.
- ^ E Velculescu, Victor; Zhang, Lin; Zhou, Wei; Vogelstein, Jacob; A Basrai, Munira; E Bassett Jr., Douglas; Hieter, Phil; Vogelstein, Bert; W Kinzler, Kenneth (1997). "Characterization of the Yeast Transcriptome". Cell. 2 (88): 243–51. doi:10.1016/S0092-8674(00)81845-0. PMID 9008165. S2CID 11430660.
- ^ a b c Peralta, Mihaela (2012). "The Human Transcriptome: An Unfinished Story". Genes. 3 (3): 344–360. doi:10.3390/genes3030344. PMC 3422666. PMID 22916334.
- ^ a b Govindarajan, Rajeshwar; Duraiyan, Jeyapradha; Kaliyappan, Karunakaran; Palanisamy, Murugesan (2012). "Microarray and its applications". Journal of Pharmacy and Bioallied Sciences. 4 (6): S310-2. doi:10.4103/0975-7406.100283. PMC 3467903. PMID 23066278.
- ^ a b C Frith, Martin; Pheasant, Michael; S Mattick, John (2005). "Genomics: The amazing complexity of the human transcriptome". European Journal of Human Genetics. 13 (8): 894–897. doi:10.1038/sj.ejhg.5201459. PMID 15970949. S2CID 2836126.
- ^ a b Clancy, Suzanne (2008). "DNA Transcription". Nature Education. 1 (11): 41.
- ^ a b c d Celerino & Sanguanini 2018, 12페이지
- ^ Berg JMTJ, Stryer L.생화학.뉴욕: W H Freeman, 2002
- ^ 매틱 JS, 마쿠닌 4세비부호화 RNA.Hum Mol Genet 2006;15 사양 No 1:R17–29
- ^ a b U. Adams, Jill (2008). "Transcriptome: Connecting the Genome to Gene Function". Nature Education. 1 (1): 195.
- ^ a b Cellerino & Sanguanini 2018, 서문 페이지
- ^ Bryant S, Manning DL (1998). "Isolation of messenger RNA". RNA Isolation and Characterization Protocols. Methods in Molecular Biology. Vol. 86. pp. 61–4. doi:10.1385/0-89603-494-1:61. ISBN 978-0-89603-494-5. PMID 9664454.
- ^ Chomczynski P, Sacchi N (April 1987). "Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction". Analytical Biochemistry. 162 (1): 156–9. doi:10.1016/0003-2697(87)90021-2. PMID 2440339.
- ^ a b Tachibana, Chris (31 July 2015). "Transcriptomics today: Microarrays, RNA-seq, and more". Science Magazine. 349 (6247): 544. Bibcode:2015Sci...349..544T. Retrieved 2 May 2020.
- ^ Schena, M.; Shalon, D.; Davis, R. W.; Brown, P. O. (20 October 1995). "Quantitative monitoring of gene expression patterns with a complementary DNA microarray". Science. New York, N.Y.). 270 (5235): 467–470. Bibcode:1995Sci...270..467S. doi:10.1126/science.270.5235.467. ISSN 0036-8075. PMID 7569999. S2CID 6720459.
- ^ a b c Celerino & Sanguanini 2018, 13페이지
- ^ Celerino & Sanguanini 2018, 18페이지
- ^ Kanter, Itamar; Kalisky, Tomer (10 March 2015). "Single Cell Transcriptomics: Methods and Applications". Frontiers in Oncology. 5: 53. doi:10.3389/fonc.2015.00053. ISSN 2234-943X. PMC 4354386. PMID 25806353.
- ^ Stegle, Oliver; A. Teichmann, Sarah; C. Marioni, John (2015). "Computational and analytical challenges in single-cell transcriptomics". Nature Reviews Genetics. 16 (3): 133–45. doi:10.1038/nrg3833. PMID 25628217. S2CID 205486032.
- ^ Trapnell, Cole (1 October 2015). "Defining cell types and states with single-cell genomics". Genome Research. 25 (10): 1491–1498. doi:10.1101/gr.190595.115. ISSN 1088-9051. PMC 4579334. PMID 26430159.
- ^ a b Kanter, Itamar; Kalisky, Tomer (2015). "Single Cell Transcriptomics: Methods and Applications". Frontiers in Oncology. 5 (13): 53. doi:10.3389/fonc.2015.00053. PMC 4354386. PMID 25806353.
- ^ Godoy, Patricio; Schmidt-Heck, Wolfgang; Hellwig, Birte; Nell, Patrick; Feuerborn, David; Rahnenführer, Jörg; Kattler, Kathrin; Walter, Jörn; Blüthgen, Nils; G. Hengstler, Jan (5 July 2018). "Assessment of stem cell differentiation based on genome-wide expression profiles". Philosophical Transactions of the Royal Society B. 373 (1750): 20170221. doi:10.1098/rstb.2017.0221. PMC 5974444. PMID 29786556.
- ^ Zhao, L; Zheng, X; Liu, J; Zheng, R; Yang, R; Wang, Y; Sun, L (1 July 2019). "The placental transcriptome of the first-trimester placenta is affected by in vitro fertilization and embryo transfer". Reproductive Biology and Endocrinology. 17 (1): 50. doi:10.1186/s12958-019-0494-7. PMC 6604150. PMID 31262321.
- ^ Eroglu, Binnur; A. Szurek, Edyta; Schall, Peter; E. Latham, Keith; Eroglu, Ali (6 April 2020). "Probing lasting cryoinjuries to oocyte-embryo transcriptome". PLOS ONE. 15 (4): e0231108. Bibcode:2020PLoSO..1531108E. doi:10.1371/journal.pone.0231108. PMC 7135251. PMID 32251418.
- ^ Szabo, David (2014). "Transcriptomic biomarkers in safety and risk assessment of chemicals". Transcriptomic biomarkers in safety and risk assessment of chemicals. In Ramesh Gupta, editors:Gupta - Biomarkers in Toxicology, Oxford:Academic Press. pp. 1033–1038. doi:10.1016/B978-0-12-404630-6.00062-2. ISBN 978-0-12-404630-6.
- ^ Drost, Hajk-Georg; Gabel, Alexander; Grosse, Ivo; Quint, Marcel; Grosse, Ivo (2018-05-01). "myTAI: evolutionary transcriptomics with R". Bioinformatics. 34 (9): 1589–1590. doi:10.1093/molbev/msv012. ISSN 0737-4038. PMC 5925770. PMID 29309527.
- ^ S, Katayama; et al. (2005). "Antisense Transcription in the Mammalian Transcriptome". Science. 309 (5740): 1564–6. Bibcode:2005Sci...309.1564R. doi:10.1126/science.1112009. PMID 16141073. S2CID 34559885.
- ^ One Thousand Plant Transcriptomes Initiative (23 October 2019). "One thousand plant transcriptomes and the phylogenomics of green plants". Nature. 574 (7780): 679–685. doi:10.1038/s41586-019-1693-2. PMC 6872490. PMID 31645766.
- ^ Rutley, Nicholas; Twell, David (12 March 2015). "A decade of pollen transcriptomics". Plant Reproduction. 28 (2): 73–89. doi:10.1007/s00497-015-0261-7. PMC 4432081. PMID 25761645.
- ^ Crismani, Wayne; Baumann, Ute; Sutton, Tim; Shirley, Neil; Webster, Tracie; Spangenberg, German; Langridge, Peter; A Able, Jason (2006). "Microarray expression analysis of meiosis and microsporogenesis in hexaploid bread wheat". BMC Genomics. 7 (267): 267. doi:10.1186/1471-2164-7-267. PMC 1647286. PMID 17052357.
- ^ D. Bovill, William; Deveshwar, Priyanka; Kapoor, Sanjay; A. Able, Jason (2009). "Whole genome approaches to identify early meiotic gene candidates in cereals". Functional & Integrative Genomics. 9 (2): 219–29. doi:10.1007/s10142-008-0097-4. PMID 18836753. S2CID 22854431.
- ^ Deveshwar, Priyanka; D Bovill, William; Sharma, Rita; A Able, Jason; Kapoor, Sanjay (9 May 2011). "Analysis of anther transcriptomes to identify genes contributing to meiosis and male gametophyte development in rice". BMC Plant Biology. 11 (78): 78. doi:10.1186/1471-2229-11-78. PMC 3112077. PMID 21554676.
- ^ Javan, G. T.; Can, I.; Finley, S. J.; Soni, S (2015). "The apoptotic thanatotranscriptome associated with the liver of cadavers". Forensic Science, Medicine, and Pathology. 11 (4): 509–516. doi:10.1007/s12024-015-9704-6. PMID 26318598. S2CID 21583165.
- ^ Manzoni, Claudia; A Kia, Demis; Vandrovcova, Jana; Hardy, John; W Wood, Nicholas; A Lewis, Patrick; Ferrari, Raffaele (March 2018). "Genome, transcriptome and proteome: the rise of omics data and their integration in biomedical sciences". Briefings in Bioinformatics. 19 (2): 286–302. doi:10.1093/bib/bbw114. PMC 6018996. PMID 27881428.
- ^ Schwanhäusser, Björn; et al. (May 2011). "Global quantification of mammalian gene expression control" (PDF). Nature. 473 (7347): 337–342. Bibcode:2011Natur.473..337S. doi:10.1038/nature10098. PMID 21593866. S2CID 205224972.
레퍼런스
- Cellerino, A; Sanguanini, M (2018), Transcriptome Analysis: Introduction and Examples from the Neurosciences, doi:10.1007/978-88-7642-642-1, ISBN 978-88-7642-641-4
추가 정보
- ^ Subramanian A, Tamayo P, Mootha VK, Mukherjee S, Ebert BL, Gillete MA, Paulovich A, Pomeroy SL, Golub TR, Lander ES, Messirov JP. (2005)유전자 세트 농축 분석: 게놈 전체의 발현 프로파일을 해석하기 위한 지식 기반 접근법.Proc Natl Acad Sci USA 102 (43) : 15545-50.
- ^ Laule O, Hirsch-Hoffmann M, Hruz T, Gruissm W, P Zimmermann.(2006) Genevestigator를 사용한 마우스 트랜스크립텀의 웹 기반 분석.BMC 생물정보학 7:311
- ^ Assou, S.; Boumela, I.; Haouzi, D.; Anahory, T.; Dechaud, H.; De Vos, J.; Hamamah, S. (2010). "Dynamic changes in gene expression during human early embryo development: From fundamental aspects to clinical applications". Human Reproduction Update. 17 (2): 272–290. doi:10.1093/humupd/dmq036. PMC 3189516. PMID 20716614.
- ^ Ogorodnikov, A; Kargapolova, Y; Danckwardt, S. (2016). "Processing and transcriptome expansion at the mRNA 3′ end in health and disease: finding the right end". Eur J Physiol. 468 (6): 993–1012. doi:10.1007/s00424-016-1828-3. PMC 4893057. PMID 27220521.