전달 전달 RNA
Transfer-messenger RNA| 전달 메신저 RNA | |
|---|---|
 | |
| 식별자 | |
| 기호. | 전송하다 |
| Rfam | RF00023 |
| 기타 데이터 | |
| RNA형 | 유전자 |
| PDB 구조 | PDBe |
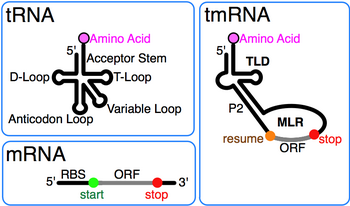
Transfer-Messenger RNA(약칭 tmRNA, 10Sa RNA 및 그 유전명 SsrA)는 이중 tRNA 유사 및 전달 RNA 유사 특성을 가진 박테리아 RNA 분자입니다.tmRNA는 소단백질 B(SmpB), 신장인자 Tu(EF-Tu), 리보솜 단백질 S1과 함께 리보핵단백질 복합체(tmRNP)를 형성한다.번역에서 tmRNA 및 그 관련 단백질은 예를 들어 정지 코돈을 잃은 메신저 RNA의 말단에 도달했을 때 단백질 생합성 도중에 정지된 세균 리보솜에 결합한다.tmRNA는 매우 다용도적이며, 정지된 리보솜을 재활용하고, 미완성 폴리펩타이드에 단백질 분해 유도 태그를 추가하며, 이상 [1]메신저 RNA의 분해를 촉진합니다.대부분의 박테리아에서 이러한 기능은 표준 원피스 tmRNA에 의해 수행됩니다.다른 세균종에서는 치환된 srA 유전자가 2개의 개별 RNA 사슬이 염기쌍으로 결합되는 2피스 tmRNA를 생성한다.
발견 및 초기 작업
tmRNA는 대장균 RNA의 혼합 "10S" 전기영동 분율이 tmRNA와 유사한 크기의 RNase P RNA(10Sb)[2]로 추가로 분해된 후 처음 10Sa RNA로 지정되었다.혼합된 10S RNA에서 의사우리딘의 존재는 tmRNA가 tRNA에서도 발견되는 염기를 변형했음을 암시했다.tmRNA의 3' 말단에서 tRNA의 T 스템루프와 유사성은 Mycobacterium [3]결핵으로부터 srA를 배열할 때 처음 확인되었다.후속 배열 비교 결과 알라닌-tRNA 연결효소에 [4]의한 아미노아실화를 촉진하는 알라닌 tRNA와 같은 요소를 가진 수용체 스템을 포함하여 tmRNA의 5' 및 3' 말단에 의해 형성된 전체 tRNA 유사 도메인(TLD)이 밝혀졌다.또한 tRNA와의 차이점도 밝혀졌습니다. 즉, tmRNA에는 안티코돈 암이 없고 D 암 영역은 베이스 쌍이 없는 루프입니다.
구조.
표준 원피스 tmRNA의 2차 구조
전체 대장균 tmRNA 2차 구조는 비교 배열 분석 및 구조 [5][6]탐사에 의해 설명되었다.Watson-Crick 및 G-U 염기쌍은 수동 정렬 [7][8]절차와 조합하여 자동화된 계산 방법을 사용하여 세균의 tmRNA 염기서열을 비교함으로써 식별되었다.첨부된 그림은 계통학적으로 지지된 12개의 나선형(P1 - P12 쌍이라고도 함)으로 구성된 이 시제품 tmRNA의 기본 쌍 패턴을 보여줍니다. 일부는 나선형 세그먼트로 나뉩니다.
각 tmRNA의 두드러진 특징은 헬리클 1, 12 및 2a(각각 tRNA 수용체 스템, T-스템 및 가변 스템의 아날로그)로 구성되며, 5' 일인산 및 알라닐화 가능 3' CCA 말단을 포함하는 보존된 tRNA 유사 도메인(TLD)이다.mRNA-like region(MLR; mRNA 유사영역)은 표준 tmRNA에 있으며, 재개 코돈과 정지 코돈으로 표시된 태그 펩타이드의 의사 노트와 부호화 시퀀스(CDS)를 포함하는 큰 루프이다.코드화된 태그 펩타이드(대장균의 ANDENYALAA)는 사용 [9]가능한 단백질 분해효소 및 어댑터 세트에 따라 박테리아에 따라 다르다.
tmRNA에는 일반적으로 태그펩타이드 CDS의 업스트림(pk1)과 CDS의 다운스트림(pk2~pk4)의 4개의 의사 노트가 포함됩니다.pseudoknot 영역은 일반적으로 보존되지만 진화적으로 플라스틱입니다.예를 들어 시아노박테리아의 (원피스) tmRNA에서 pk4는 2개의 탠덤으로 배치된 작은 의사 노트로 치환된다.이는 TLD 외부에서 tmRNA 접힘이 중요할 수 있지만, 의사 노트는 보존된 잔류물이 부족하며, srA 배열이 플라스티드와 엔도심비언트 라인에서 분리될 때 가장 먼저 손실되는 구조 중 하나임을 시사한다.대장균 tmRNA의 3의사성 영역에서의 염기쌍은 번역 [7][10]중에 파괴된다.
투피스 TMNA
Circularly permuted ssrA 3대 혈통에:i)alphaproteobacteria과jakobid 원생 생물의 원시적인 미토콘드리아, ii)cyanobacteria(Gloeobacter고 clade 프로 클로로 코커스를 포함하고 많은 시네 코코 커스)의 두 차갑그룹, 그리고 iii)betaproteobacteria(Cupriavidus과 몇몇 Rhodocyclales)의 일부 의원들이 보도되었다.[11][12]모두 판독 프레임의 다운스트림에서 절단된 표준 형태와 동일한 전체 2피스(수용체 및 코딩 조각) 형태를 생성합니다.표준 tmRNA의 4개(또는 그 이상)에 비해 2개 이상의 의사노트를 유지하는 것은 없습니다.
알파프로테오박테리아는 일반적인 T루프 배열 TaCRANY를 GGCRGUA로 치환하는 것과 3' 말단 의사 노트의 큰 루프에서의 배열 AACAGAA의 두 가지 시그니처 배열을 가진다.미토콘드리아에서는 MLR이 상실되어 미토콘드리아 srA의 현저한 재분열로 야코바 리베라에서는 [13]작은 원피스 제품이 탄생한다.
시아노박테리아는 다른 Synechoccus 균주에서 발생하는 두 가지 유전자 유형 사이의 현저한 배열 유사성 때문에 표준 유전자에서 치환된 유전자의 진화에 가장 그럴듯한 사례를 제공합니다.
tmRNA 처리
대부분의 tmRNA는 tRNA처럼 처리되는 더 큰 전구체로 전사된다.리보핵산가수분해효소 [4]P에 의해 5' 끝의 분열이 이루어진다.RNase T와 RNase PH가 가장 [14][15]효과적이지만 다중 엑소뉴클라아제들은 tmRNA의 3' 말단 처리에 참여할 수 있다.세균종에 따라 3'-CCA는 tRNA 뉴클레오티딜전달효소에 의해 암호화되거나 첨가된다.
치환된 전구체 tmRNA의 내부 부위에서의 유사한 처리는 물리적으로 두 조각으로 분할되는 것을 설명한다.2피스 전송 RNA에는 처리를 고려해야 하는 두 개의 추가 단부가 있습니다.알파프로테오박테리아의 경우,[16] 5분의 1은 전사의 미처리 시작 지점이다.경우에 따라서는 맨 끝 3'이 rho에 의존하지 않는 종료의 결과일 수 있습니다.
입체 구조물
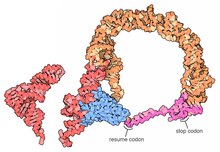
완전한 tmRNA 분자의 고해상도 구조는 현재 이용할 수 없으며 MLR의 고유 유연성 때문에 얻기 어려울 수 있으며, 2007년에는 SmpB 단백질에 결합된 Thermus thermophilus TLD의 결정 구조를 3Ω 분해능으로 얻었다.이 구조는 SmpB가 표준 tRNA의 D 스템과 항코돈을 모방하는 반면, tmRNA의 헬리컬 섹션 2a는 tRNA의 [18]가변팔에 해당하며, 트랜스 번역의 초기 단계에서 TMRNA에 대한 극저온전자현미경연구는 리보솜과 TMRNA 결합(TMNP)의 공간적 관계를 나타낸다.TLD는 50S 리보솜 서브유닛의 GTPase 관련 중심 부근에 위치하며, 나선 5와 의사노트 pk2 ~ pk4는 30S 리보솜 서브유닛의 [19]부리 주위에 호를 형성한다.
번역
tmRNA에 의한 코딩은 1995년[20] Simpson과 동료들이 대장균에서 마우스 사이토카인 IL-6을 과다 투여하여 동일한 11-아미노산 잔기 확장(A)ANDENYALAA를 가진 카르복실 흰개미에서 각각 태그된 여러 개의 잘린 사이토카인 유래 펩타이드를 발견했을 때 발견되었다.tmRNA 자체의 3' 말단에서 나오는 N-말단 알라닌을 제외하고, 이 태그 시퀀스는 대장균 tmRNA에서 짧은 개방 판독 프레임으로 추적되었다.태그 펩타이드가 단백질 분해를 촉진한다는 것을 인식하여 tmRNA 작용에 대한 트랜스 번역 모델을 제안했다.[21]
변환 메커니즘에 대한 자세한 내용은 조사 중이지만, 일반적으로 tmRNA가 먼저 정지된 리보솜의 빈 A 부위를 차지한다는 데 동의합니다.이어서 리보솜은 잘린 메신저 RNA의 3' 말단에서 MLR의 레줌 코돈으로 이동하고, 이어서 정상적으로 번역이 계속된 후 프레임 내 tmRNA 정지 코돈에 도달할 때까지 미끄러지기 쉬운 단계를 거친다.일부 박테리아 종에서는 번역이 필수적인 반면, 다른 박테리아는 스트레스를 받는 성장 [22]조건에 노출될 때 살아남기 위해 tmRNA를 필요로 합니다.tmRNA는 항생제에 [23]의해 정지된 리보솜을 구조함으로써 항생제 내성을 가진 세포를 도울 수 있다고 믿어진다.태그펩타이드는 유기체에 따라 다양한 단백질분해효소 또는 단백질분해효소 [9]어댑터에 의해 인식될 수 있다.
이동성 유전요소 및 tmRNA 유전자
srA는 일부 모바일 DNA의 표적이기도 하고 다른 DNA의 표적이 되기도 합니다.세 가지 유형의 모바일 요소에 의해 중단된 것으로 확인되었습니다.다른 전략으로 유전자 기능을 교란시키는 것은 없다.그룹 I의 침입자는 자가 분열에 의해 스스로 제거되고, 무해한 부위에 리케티알 회문체 요소(RPE)가 삽입되며, 인테그라아제 인코딩 게놈 섬이 표적 srA를 분할하지만 분할 부분을 [24][25][26][27]복구한다.
비염색체 srA는 마이코박터리오파지의 게놈 조사(파지의 10%에서)[28]에서 처음 검출되었다.플라스미드와 게놈 섬을 포함한 다른 이동 원소는 SSRA를 가지고 있는 것으로 밝혀졌다.한 가지 흥미로운 사례는 Rhodobacter sphaeroides ATCC 17025로, 유전자의 tmRNA 유전자가 유전자에 의해 파괴되었다; tmRNA 유전자의 다른 모든 게놈 섬들과는 달리, 이 섬은 복원 없이 토종 표적 유전자를 불활성화시켰지만, 자신의 tmRNA 유전자를 운반함으로써 보상한다.용해성 마이코박터리오파지 DS6A에서 srA의 매우 특이한 친척이 발견되어 TLD보다 조금 더 많이 암호화된다.
미토콘드리아전달RNA(srA유전자)
미토콘드리아로 인코딩된 구조적으로 환원된 tmRNA(mt-tmRNA)가 자코비드 편모충류 Reclinomonas [11]americana에 대해 최초로 가정되었다.그 후, tmRNA를 코드하는 미토콘드리아 유전자(ssrA)의 존재와 전사 부위 및 RNA 처리 부위의 존재가 자코비드의 [29][13]한 멤버를 제외한 모든 멤버에 대해 확인되었다.기능적 증거, 즉 알라닌에 의한 mt-tmRNA 아미노실화는 야코바 리베라에 [13]이용 가능하다.최근에는 난모균의 미토콘드리아 게놈에서도 srA가 확인되었다.[30]α-프로테오박테리아(미토콘드리아의 조상)와 마찬가지로 mt-tmRNA는 원피스 tmRNA [13]구조를 코드하는 Jakoba libera를 제외하고 원형으로 배열된 2피스 RNA 분자이다.
미토콘드리아 게놈에서 srA의 동정
미토콘드리아 tmRNA 유전자는 초기에 자코브류 사이에 보존되고 독특한 tRNA와 같은 2차 구조로 접힐 수 있는 짧은 배열로 인식되었다.9개의 완전한 jakobid mtDNA [29]배열의 가용성과 현저하게 개선된 공분산 검색 도구(Infernal;)[31][32][33]를 통해, 공분산 모델은 oomycete에서도 미토콘드리아 srA 유전자를 식별한 jakobid 미토콘드리아 tmRNA를 기반으로 개발되었다.현재 알부고, 브레미아, 피토프토라, 피티오페로노스포라, 피티움, 사프로레그니아 등 6개 속에 걸쳐 총 34종의 난균 mt-tmRNA가 검출됐다.Jakobid 및 oomycete 시퀀스로 작성된 공분산 모델은 이제 Rfam에서 'mt-tmRNA'[30]라는 이름으로 제공됩니다.
mt-tmRNA 구조
표준 세균 전달 RNA는 tRNA(Ala) 유사 도메인(종료 부호화가 결여된 mRNA에 비암호화 알라닌을 추가할 수 있음)과 단백질 용해를 위해 폴리펩타이드를 대상으로 하는 단백질 태그를 코드하는 mRNA 유사 도메인으로 구성됩니다.mRNA 유사 도메인이 mt-tmRNA로 손실되었습니다.비교 시퀀스 분석 결과 mt-tmRNA에서 [30]일반적인 특징이 나타납니다.가장 보존된 것은 아미노산 수용체 줄기의 1차 배열이다.분자의 이 부분은 식별자 위치에 불변 A 잔기가 있고 위치 3에 G-U 쌍이 있다(G-C 쌍이 있는 Seculamonas acquadoriensis 제외). 이 위치는 알라닐 tRNA 합성효소의 인식 부위이다.P2는 가변 길이(3~10개의 염기쌍)의 나선형이며, tRNA의 안티코돈 스템에 해당하지만 안티코돈 루프가 없다(tmRNA 기능에 필요하지 않음).P2는 tRNA 유사 구조를 안정화시키지만, 난균류와 자코비드에 걸쳐 불변한 4개의 뉴클레오티드는 현재 확인되지 않은 추가적인 기능을 암시한다.P3는 5개의 염기쌍을 가지며 tRNA의 T-암에 해당하지만 쌍체 영역과 루프 모두에서 서로 다른 컨센서스 뉴클레오티드를 가진다.T-루프 시퀀스는 난균류와 자코비드에 걸쳐 보존되며, 편차는 거의 없다(예: 사프롤레그니아 페락스).마지막으로 세균의 tmRNA에 대해 짧은 3뉴클레오티드 D-루프 특성을 가진 tRNA 유사 D-줄기 대신 미토콘드리아 대응물은 5~14nt의 매우 가변적인 긴 루프를 가진다.2피스 mt-tmRNA의 중간 시퀀스(Int.)는 A+U 농후하고 길이가 불규칙합니다(4-34nt).원피스 및 투피스 mt-tmRNA의 2차 구조 모델은 그림 1을 참조하십시오.
mt-tmRNA 처리 및 표현
Phytophthora sojae의 RNA-Seq 데이터는 인접한 미토콘드리아 tRNA와 유사한 발현 수준을 나타내며, 4대 가공 부위에서 성숙한 mt-tmRNA의 [30]예측 말단을 확인하였다.tmRNA 전구체 분자는 RNase P와 tRNA 3' 처리 핵산가수분해효소(그림 2 참조)에 의해 처리될 가능성이 높으며, 후자의 활성은 간섭 배열을 제거하는 것으로 가정한다.3' 판별기 뉴클레오티드에 CCA를 첨가한 후 알라닐-tRNA 합성효소에 의해 알라닌을 충전할 수 있다.
「 」를 참조해 주세요.
레퍼런스
- ^ Keiler KC (2008). "Biology of trans-translation". Annual Review of Microbiology. 62: 133–51. doi:10.1146/annurev.micro.62.081307.162948. PMID 18557701.
- ^ Ray BK, Apirion D (July 1979). "Characterization of 10S RNA: a new stable rna molecule from Escherichia coli". Molecular & General Genetics. 174 (1): 25–32. doi:10.1007/BF00433301. PMID 384159. S2CID 22699560.
- ^ Tyagi JS, Kinger AK (January 1992). "Identification of the 10Sa RNA structural gene of Mycobacterium tuberculosis". Nucleic Acids Research. 20 (1): 138. doi:10.1093/nar/20.1.138. PMC 310338. PMID 1371186.
- ^ a b Komine Y, Kitabatake M, Yokogawa T, Nishikawa K, Inokuchi H (September 1994). "A tRNA-like structure is present in 10Sa RNA, a small stable RNA from Escherichia coli". Proceedings of the National Academy of Sciences of the United States of America. 91 (20): 9223–7. doi:10.1073/pnas.91.20.9223. PMC 44784. PMID 7524073.
- ^ Williams KP, Bartel DP (December 1996). "Phylogenetic analysis of tmRNA secondary structure". RNA. 2 (12): 1306–10. PMC 1369456. PMID 8972778.
- ^ Felden B, Himeno H, Muto A, McCutcheon JP, Atkins JF, Gesteland RF (January 1997). "Probing the structure of the Escherichia coli 10Sa RNA (tmRNA)". RNA. 3 (1): 89–103. PMC 1369465. PMID 8990402.
- ^ a b Zwieb C, Wower I, Wower J (May 1999). "Comparative sequence analysis of tmRNA". Nucleic Acids Research. 27 (10): 2063–71. doi:10.1093/nar/27.10.2063. PMC 148424. PMID 10219077.
- ^ Andersen ES, Lind-Thomsen A, Knudsen B, Kristensen SE, Havgaard JH, Torarinsson E, Larsen N, Zwieb C, Sestoft P, Kjems J, Gorodkin J (November 2007). "Semiautomated improvement of RNA alignments". RNA. 13 (11): 1850–9. doi:10.1261/rna.215407. PMC 2040093. PMID 17804647.
- ^ a b Gur E, Sauer RT (October 2008). "Evolution of the ssrA degradation tag in Mycoplasma: specificity switch to a different protease". Proceedings of the National Academy of Sciences of the United States of America. 105 (42): 16113–8. doi:10.1073/pnas.0808802105. PMC 2570983. PMID 18852454.
- ^ Wower IK, Zwieb C, Wower J (May 2005). "Transfer-messenger RNA unfolds as it transits the ribosome". RNA. 11 (5): 668–73. doi:10.1261/rna.7269305. PMC 1370753. PMID 15811920.
- ^ a b Keiler KC, Shapiro L, Williams KP (July 2000). "tmRNAs that encode proteolysis-inducing tags are found in all known bacterial genomes: A two-piece tmRNA functions in Caulobacter". Proceedings of the National Academy of Sciences of the United States of America. 97 (14): 7778–83. doi:10.1073/pnas.97.14.7778. PMC 16621. PMID 10884408.
- ^ Sharkady SM, Williams KP (2004). "A third lineage with two-piece tmRNA". Nucleic Acids Research. 32 (15): 4531–8. doi:10.1093/nar/gkh795. PMC 516066. PMID 15326226.
- ^ a b c d Jacob Y, Seif E, Paquet PO, Lang BF (April 2004). "Loss of the mRNA-like region in mitochondrial tmRNAs of jakobids". RNA. 10 (4): 605–14. doi:10.1261/rna.5227904. PMC 1370551. PMID 15037770.
- ^ Srivastava RA, Srivastava N, Apirion D (May 1992). "Characterization of the RNA processing enzyme RNase III from wild type and overexpressing Escherichia coli cells in processing natural RNA substrates". The International Journal of Biochemistry. 24 (5): 737–49. doi:10.1016/0020-711X(92)90007-N. PMID 1375563.
- ^ Li Z, Pandit S, Deutscher MP (March 1998). "3' exoribonucleolytic trimming is a common feature of the maturation of small, stable RNAs in Escherichia coli". Proceedings of the National Academy of Sciences of the United States of America. 95 (6): 2856–61. doi:10.1073/pnas.95.6.2856. PMC 19659. PMID 9501180.
- ^ Mao C, Bhardwaj K, Sharkady SM, Fish RI, Driscoll T, Wower J, Zwieb C, Sobral BW, Williams KP (2009). "Variations on the tmRNA gene". RNA Biology. 6 (4): 355–61. doi:10.4161/rna.6.4.9172. PMID 19617710.
- ^ Someya T, Nameki N, Hosoi H, Suzuki S, Hatanaka H, Fujii M, Terada T, Shirouzu M, Inoue Y, Shibata T, Kuramitsu S, Yokoyama S, Kawai G (January 2003). "Solution structure of a tmRNA-binding protein, SmpB, from Thermus thermophilus". FEBS Letters. 535 (1–3): 94–100. doi:10.1016/S0014-5793(02)03880-2. PMID 12560085.
- ^ a b Bessho Y, Shibata R, Sekine S, Murayama K, Higashijima K, Hori-Takemoto C, Shirouzu M, Kuramitsu S, Yokoyama S (May 2007). "Structural basis for functional mimicry of long-variable-arm tRNA by transfer-messenger RNA". Proceedings of the National Academy of Sciences of the United States of America. 104 (20): 8293–8. doi:10.1073/pnas.0700402104. PMC 1895943. PMID 17488812.
- ^ Valle M, Gillet R, Kaur S, Henne A, Ramakrishnan V, Frank J (April 2003). "Visualizing tmRNA entry into a stalled ribosome". Science. 300 (5616): 127–30. doi:10.1126/science.1081798. PMID 12677067. S2CID 28845151.
- ^ Tu GF, Reid GE, Zhang JG, Moritz RL, Simpson RJ (April 1995). "C-terminal extension of truncated recombinant proteins in Escherichia coli with a 10Sa RNA decapeptide". The Journal of Biological Chemistry. 270 (16): 9322–6. doi:10.1074/jbc.270.16.9322. PMID 7536743.
- ^ Keiler KC, Waller PR, Sauer RT (February 1996). "Role of a peptide tagging system in degradation of proteins synthesized from damaged messenger RNA". Science. 271 (5251): 990–3. doi:10.1126/science.271.5251.990. PMID 8584937. S2CID 29254050.
- ^ Thibonnier M, Thiberge JM, De Reuse H (2008). Ahmed N (ed.). "Trans-translation in Helicobacter pylori: essentiality of ribosome rescue and requirement of protein tagging for stress resistance and competence". PLOS ONE. 3 (11): e3810. doi:10.1371/journal.pone.0003810. PMC 2584231. PMID 19043582.
- ^ Müller, Claudia; Crowe-McAuliffe, Caillan; Wilson, Daniel N. (2021-03-18). "Ribosome Rescue Pathways in Bacteria". Frontiers in Microbiology. 12: 652980. doi:10.3389/fmicb.2021.652980. ISSN 1664-302X. PMC 8012679. PMID 33815344.
- ^ Kirby JE, Trempy JE, Gottesman S (April 1994). "Excision of a P4-like cryptic prophage leads to Alp protease expression in Escherichia coli". Journal of Bacteriology. 176 (7): 2068–81. doi:10.1128/jb.176.7.2068-2081.1994. PMC 205313. PMID 7511583.
- ^ Williams KP (January 2002). "The tmRNA Website: invasion by an intron". Nucleic Acids Research. 30 (1): 179–82. doi:10.1093/nar/30.1.179. PMC 99078. PMID 11752287.
- ^ Dwyer DS (January 2001). "Selfish DNA and the origin of genes". Science. 291 (5502): 252–3. doi:10.1126/science.291.5502.252. PMID 11253208. S2CID 5369275.
- ^ Williams KP (February 2003). "Traffic at the tmRNA gene". Journal of Bacteriology. 185 (3): 1059–70. doi:10.1128/JB.185.3.1059-1070.2003. PMC 142792. PMID 12533482.
- ^ Hatfull GF, Pedulla ML, Jacobs-Sera D, Cichon PM, Foley A, Ford ME, Gonda RM, Houtz JM, Hryckowian AJ, Kelchner VA, Namburi S, Pajcini KV, Popovich MG, Schleicher DT, Simanek BZ, Smith AL, Zdanowicz GM, Kumar V, Peebles CL, Jacobs WR, Lawrence JG, Hendrix RW (June 2006). "Exploring the mycobacteriophage metaproteome: phage genomics as an educational platform". PLOS Genetics. 2 (6): e92. doi:10.1371/journal.pgen.0020092. PMC 1475703. PMID 16789831.
- ^ a b Burger G, Gray MW, Forget L, Lang BF (2013). "Strikingly bacteria-like and gene-rich mitochondrial genomes throughout jakobid protists". Genome Biology and Evolution. 5 (2): 418–38. doi:10.1093/gbe/evt008. PMC 3590771. PMID 23335123.
- ^ a b c d Hafez M, Burger G, Steinberg SV, Lang BF (July 2013). "A second eukaryotic group with mitochondrion-encoded tmRNA: in silico identification and experimental confirmation". RNA Biology. 10 (7): 1117–24. doi:10.4161/rna.25376. PMC 3849159. PMID 23823571. Archived from the original on 2014-02-21. Retrieved 2014-02-13.
- ^ Eddy, S. "Infernal website". Retrieved 14 August 2016.
- ^ Eddy SR, Durbin R (June 1994). "RNA sequence analysis using covariance models". Nucleic Acids Research. 22 (11): 2079–88. doi:10.1093/nar/22.11.2079. PMC 308124. PMID 8029015.
- ^ Nawrocki EP, Kolbe DL, Eddy SR (May 2009). "Infernal 1.0: inference of RNA alignments". Bioinformatics. 25 (10): 1335–7. doi:10.1093/bioinformatics/btp157. PMC 2732312. PMID 19307242.
추가 정보
- Hong SJ, Tran QA, Keiler KC (July 2005). "Cell cycle-regulated degradation of tmRNA is controlled by RNase R and SmpB". Molecular Microbiology. 57 (2): 565–75. doi:10.1111/j.1365-2958.2005.04709.x. PMC 3776457. PMID 15978085.