시스 천연 안티센스 전사물
Cis-natural antisense transcript자연 안티센스 트랜스크립트(NATs)는 다른 RNA [1]트랜스크립트에 대한 트랜스크립트 상보성을 가진 세포 내에서 암호화된 RNA의 그룹입니다.그들은 사람, 쥐, 효모, 아라비도시스 [2]탈리아나를 포함한 여러 진핵생물에서 확인되었다.이 종류의 RNA는 단백질 코드화 RNA와 비코드화 RNA를 [3]모두 포함한다.현재 증거는 RNA 간섭(RNAi), 대체 스플라이싱, 게놈 임프린트 및 X-염색체 불활성화 [4]등 NAT에 대한 다양한 조절 역할을 제안하고 있습니다.NAT은 cis 또는 [5]trans에서 동작하는지에 따라 크게2개의 카테고리로 분류됩니다.트랜스 NAT 는, 타겟과는 다른 로케이션으로부터 변환되어 통상,[6] 몇개의 불일치가 있는 복수의 트랜스크립트에 상보성이 있습니다.MicroRNA(miRNA)는,[6] 몇개의 미스매치가 있는 복수의 트랜스크립트를 대상으로 할 수 있는 트랜스 NAT 의 예입니다.한편, 시스 천연 안티센스 전사물(cis-NATs)은, 타겟과 같은 게놈 궤적으로부터 전사되지만, 반대쪽 DNA 사슬로부터 전사되어 완벽한 [7]쌍을 형성한다.
오리엔테이션
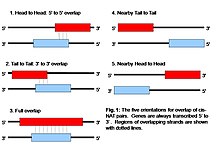
Cis-NAT에는 다양한 방향이 있으며 [7]쌍 간에 겹치는 길이가 다릅니다.지금까지 [8]cis-NAT에 대해 확인된 오리엔테이션은 5개입니다.가장 일반적인 방향은 머리부터 머리까지로, 양쪽 스크립트의 5' 끝부분이 [3]함께 정렬됩니다.만약 전사 충돌이 전사 [1]억제의 이유라면, 이러한 배향은 유전자 발현의 가장 큰 붕괴를 초래할 것이다.단, 가장 일반적인 NAT [1]쌍이 테일 투 테일 방향이라는 연구 결과가 있습니다.꼬리 대 꼬리, 겹침, 머리 대 머리 근처 및 꼬리 대 꼬리 근처와 같은 다른 것들은 [1]덜 자주 발생한다.완전히 중복되는 NAT은 서로 [3]완전히 겹쳐진 안티센스 유전자와 관련이 있습니다.머리부터 머리까지의 방향과 꼬리부터 꼬리까지의 방향은 물리적으로 서로 분리되어 있지만 서로 [1]매우 가까운 위치에 있습니다.현재의 증거는 촉매 [3]활성을 가진 유전자에서 NAT 쌍의 과잉 표현이 있음을 시사한다.특히 이 유전자에는 이런 종류의 조절에 더 잘 걸리는 무언가가 있을 수 있다.
식별 접근법
전체 게놈에서 NAT의 식별은 여러 유기체에서 사용할 수 있는 배열 데이터 수집이 많기 때문에 가능하다.silico 검출 방법에서는 시퀀스 정보의 [7]소스에 따라서는, NAT 에 몇개의 단점이 있습니다.mRNA를 사용하는 연구에는 방향이 알려진 시퀀스가 있지만 사용 가능한 mRNA 시퀀스 정보의 양은 [3]적다.유전자를 찾기 위해 훈련된 알고리즘을 사용하여 예측된 유전자 모델은 확인된 [7]유전자에 대한 신뢰의 대가로 게놈에 대한 더 많은 범위를 제공합니다.또 다른 자원은 광범위한 표현 시퀀스 태그(EST) 라이브러리입니다만,[3] 이러한 작은 시퀀스는 유용한 정보를 추출하기 전에 먼저 방향을 할당해야 합니다.일부 연구에서는 EST의 폴리(A) 신호,[1] 폴리(A) 테일 및 스플라이싱 부위와 같은 특수 시퀀스 정보를 사용하여 EST를 필터링하고 올바른 전사 방향을 제공했습니다.다른 시퀀스 소스의 조합은 커버리지를 최대화하고 데이터의 무결성을 유지하려고 합니다.
NAT 쌍은 오버랩클러스터를 형성할 때 식별됩니다.서로 다른 연구에서 사용되는 컷오프 값에는 가변성이 있지만 일반적으로 20개의 염기서열 중첩 뉴클레오티드가 고려되어야 할 전사물과 중복 [1]클러스터에 대한 최소값으로 간주됩니다.또한 트랜스크립트가 NAT [1][7]쌍으로 간주되기 위해서는 다른 하나의 mRNA 분자에만 매핑되어야 합니다.현재 안티센스 쌍을 찾기 위해 사용할 수 있는 다양한 웹 및 소프트웨어 리소스가 있습니다.NATsdb 또는 Natural Antisense Transcript 데이터베이스는 여러 유기체에서 안티센스 쌍을 검색할 수 있는 풍부한 도구입니다.
메커니즘
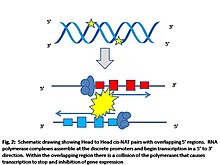
cis-NATs의 조절 역할 뒤에 있는 분자 메커니즘은 현재 [3]잘 알려져 있지 않다.cis-NAT가 유전자 발현에 미치는 조절 효과를 설명하기 위해 세 가지 모델이 제안되었다.cis-NAT와 그 보완적 전사물 사이의 기본 쌍이 되는 첫 번째 모델 속성은 mRNA [9]표현의 녹다운을 초래한다.이 모델의 가정은 이중 가닥 [1]RNA를 만들기 위해 cis-NAT 쌍 사이에 최소 6개의 염기쌍이 정밀하게 정렬된다는 것이다. DNA 메틸화와 코어 히스톤의 번역 후 수정과 같은 후생성 변형이 두 번째 [1]모델의 기초를 형성한다.아직 명확하게 이해되지 않았지만 역전사가 메틸화 착체 및/또는 히스톤 수식 착체를 감각전사의 프로모터 영역으로 유도하여 [1]유전자로부터의 발현 억제를 일으킨다고 생각된다.현재 cis-NATs의 어떤 속성이 [1]후생적 조절 모델에 중요한지는 알려지지 않았다.최근 실험 증거로 인해 인기를 얻고 있는 최종 제안 모델은 전사 충돌 모델이다.cis-NAT의 전사 과정에서 전사 복합체는 유전자의 프로모터 영역에 모인다.RNA 중합효소는 뉴클레오티드를 5'에서 3' 방향으로 [6]내려놓는 전사 개시 부위에서 유전자 전사를 시작할 것이다.시스-NAT 간의 중첩 영역에서 RNA 중합효소는 충돌하고 충돌 [1]지점에서 멈춥니다.RNA 중합효소가 조기에 정지하고 불완전한 전사가 [10]저하되기 때문에 전사가 억제됩니다.
중요성
발달, 신진대사와 같은 많은 생물학적 과정의 조절은 많은 다른 유전자들 사이의 세심한 조정을 필요로 합니다; 이것은 보통 유전자 조절 네트워크라고 불립니다.유전자 조절 네트워크에 대한 관심은 여러 유기체의 배열된 게놈의 출현으로 촉발되었다.다음 단계는 이 정보를 사용하여 유전자가 고립되어 있는 것이 아니라 어떻게 함께 작용하는지 알아내는 것이다.포유류의 발달 과정에서 암컷에서 여분의 X염색체가 불활성화된다.Xist와 Tsix라고 불리는 NAT 쌍이 염색체의 [11]과메틸화에 관여하는 것으로 나타났다.포유류 유전자의 20-30%가 miRNA의 표적이 되는 것으로 나타났으며, 이는 다양한 [12]유전자에서 이러한 분자의 조절제로서의 중요성을 강조한다.유전자의 조절을 위해 RNA를 이용하는 진화적 이유는 [1]세포에 의해 필요하지 않은 단백질을 합성하는 것보다 비용이 적게 들고 빠르기 때문일 수 있다.이것은 이러한 유형의 전사 조절을 통해 초기 진핵 생물의 선택적인 이점을 가질 수 있었다.
질병
안티센스 전사는 이상 안티센스 [4]전사를 생성하는 염색체 변화를 통해 질병에 기여할 수 있다.인간질환에 관여하는 시스NAT의 증례는 시스NAT의 [4]작용을 통해 헤모글로빈α-2 유전자의 침묵이 존재하는 유전형태의 α-thalassemia에서 유래한다.전이성 요소가 활성화된 악성 암세포에서는 많은 양의 전사 [4]노이즈가 발생하는 것으로 생각된다.이 전사 잡음으로부터 발생하는 이상 안티센스 RNA 전사물은 종양유전자와 [4]종양억제유전자와 관련된 CpG섬의 확률적 메틸화를 일으킬 수 있다.이 억제 작용은 세포들이 주요 [4]조절 유전자를 잃기 때문에 세포의 악성 종양을 더욱 진행시킬 것이다.종양 세포에서 상향 조절된 안티센스 전달물을 보면, 연구자들은 더 많은 후보 종양 억제 유전자를 [4]찾을 수 있다.또한 이상 시스NAT는 파킨슨병과 [4]같은 신경계 질환에 관여하고 있다.
레퍼런스
- ^ a b c d e f g h i j k l m n Osato N, Suzuki Y, Ikeo K, Gojobori T (2007). "Transcriptional Interferences in cis Natural Antisense Transcripts of Humans and Mice". Genetics. 176 (12): 1299–1306. doi:10.1534/genetics.106.069484. PMC 1894591. PMID 17409075.
- ^ Vanhée-Brossollet C, Vaquero C (1998). "Do natural antisense transcripts make sense in eukaryotes?". Gene. 211 (1): 1–9. doi:10.1016/S0378-1119(98)00093-6. PMID 9573333.
- ^ a b c d e f g Lavorgna G, Dahary D, Lehner B, Sorek R, Sanderson CM, Casari G (2004). "In search of antisense". Trends Biochem. Sci. 29 (2): 88–94. doi:10.1016/j.tibs.2003.12.002. PMID 15102435.
- ^ a b c d e f g h Zhang Y, Liu XS, Liu QR, Wei L (2006). "Genome-wide in silico identification and analysis of cis natural antisense transcripts (cis-NATs) in ten species". Nucleic Acids Research. 34 (12): 3465–3475. doi:10.1093/nar/gkl473. PMC 1524920. PMID 16849434.
- ^ Chen J, Sun M, Kent WJ, Huang X, Xie H, Wang W, Zhou G, Shi RZ, Rowley JD (2004). "Over 20% of human transcripts might form sense–antisense pairs". Nucleic Acids Res. 32 (16): 4812–4820. doi:10.1093/nar/gkh818. PMC 519112. PMID 15356298.
- ^ a b c Carmichael GG (2003). "Antisense starts making more sense". Nat Biotechnol. 21 (4): 371–372. doi:10.1038/nbt0403-371. PMID 12665819.
- ^ a b c d e Wang XJ, Gaasterland T, Chua NH (2005). "Genome-wide prediction and identification of cis-natural antisense transcripts in Arabidopsis thaliana". Genome Biol. 6 (4): R30. doi:10.1186/gb-2005-6-4-r30. PMC 1088958. PMID 15833117.
- ^ Fahey,M.E.; Moore,T.F. Higgins,D.G. (2002). "Overlapping Antisense Transcription in the Human Genome". Comparative and Functional Genomics. 3 (3): 244–253. doi:10.1002/cfg.173. PMC 2447278. PMID 18628857.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Borsani O, Zhu J, Verslues PE, Sunkar R, Zhu JK (2005). "Endogenous siRNAs Derived from a Pair of Natural cis-Antisense Transcripts Regulate Salt Tolerance in Arabidopsis". Cell. 123 (7): 1279–1291. doi:10.1016/j.cell.2005.11.035. PMC 3137516. PMID 16377568.
- ^ Røsok O, Sioud M (2005). "Systematic search for natural antisense transcripts in eukaryotes (review)". Int J Mol Med. 15 (2): 197–203. doi:10.3892/ijmm.15.2.197. PMID 15647831.
- ^ Li YY, Qin L, Guo ZM, Liu L, Xu H, Hao P, Su J, Shi Y, He WZ, Li YX (2006). "In silico discovery of human natural antisense transcripts". BMC Bioinformatics. 7: 18. doi:10.1186/1471-2105-7-18. PMC 1369008. PMID 16409644.
- ^ Lehner B, Williams G, Campbell RD, Sanderson CM (2002). "Antisense transcripts in the human genome". Trends Genet. 18 (2): 63–65. doi:10.1016/S0168-9525(02)02598-2. PMID 11818131.