분자생물학
Molecular biology| 시리즈의 일부 |
| 생물학 |
|---|
분자생물학 /məlkkjʊlər/는 분자합성, 변형, 메커니즘 및 [1][2][3]상호작용을 포함한 세포와 세포 사이의 생물학적 활동의 분자적 기초를 이해하는 생물학 분야이다.생물학적 고분자의 화학적, 물리적 구조에 대한 연구는 분자생물학으로 [4]알려져 있다.
분자생물학은 처음에 생물학적 현상의 기초에 초점을 맞춘 접근법으로 설명되었다 - 생물 분자의 구조와 상호작용, 그리고 이러한 상호작용이 고전 생물학의 [5]관찰을 어떻게 설명하는지.
1945년에 분자생물학이라는 용어는 물리학자 윌리엄 애스트버리에 의해 사용되었다.분자생물학 분야에서의 발전은 매우 늦게 일어났고, 복잡한 시스템이나 유리한 접근법이 박테리아와 박테리오파지를 사용함으로써 이해의 간단한 방법으로 만들어질 것이라는 것을 이해하게 되었다. 이 유기체는 동물 세포보다 더 쉽게 기본적인 생물학적 과정에 대한 정보를 생산한다.1953년, 그리고 나서 프란시스 크릭과 제임스 왓슨이라는 이름의 두 젊은이가 캠브리지의 캐번디시 연구소(현재의 MRC 분자생물학 연구소)에서 일하면서 로잘린드 프랭클린에 의해 수행된 이전 연구에 기초해 그들이 제안한 DNA 구조를 바꾼 DNA의 이중나선 모형을 만들었다.그리고 나서 모리스 윌킨스는 연구를 통해 다른 미생물, 식물,[4] 동물에서 DNA 물질을 발견하게 되었다.
분자생물학은 단순히 생물학적 분자와 그들의 상호작용에 대한 연구가 아니다; 오히려, 그것은 또한 과학자들이 분자 [6]과정에 대해 배울 수 있게 해준 그 분야의 창시 이래 개발된 기술들의 집합이다.이 분야에 혁명을 일으킨 한 가지 주목할 만한 기술은 [6]1983년에 개발된 중합효소 연쇄 반응이다.PCR은 소량의 DNA를 증폭시키는 반응으로,[7][8] 나중에 논의될 과학 분야 전반에 걸쳐 많은 응용 분야에서 사용됩니다.
분자생물학의 중심 교의는 DNA가 RNA로 전사되는 과정을 설명하고, RNA는 단백질로 [2][9]변환됩니다.
분자생물학은 또한 개별 세포 내의 구조, 기능, 그리고 내부 통제의 이해에 중요한 역할을 하는데, 이 모든 것들은 효과적으로 신약을 목표로 하고, 질병을 진단하고, 세포 [10]생리를 더 잘 이해하는데 사용될 수 있다.분자생물학에서 발생하는 일부 임상 연구와 치료법은 유전자 치료로 다루어지는 반면, 의학에서 분자생물학이나 분자세포생물학을 사용하는 것은 현재 분자 의학으로 언급되고 있다.
분자생물학의 역사
분자생물학은 생화학과 유전학의 교차점에 있다; 이러한 과학 분야가 20세기에 등장하고 진화하면서, 그들 둘 다 중요한 세포 [11]기능의 기초가 되는 분자 메커니즘을 결정하려고 노력했다는 것이 명백해졌다.분자생물학의 발전은 새로운 기술의 개발과 [12]그 최적화와 밀접한 관련이 있다.분자생물학은 많은 과학자들의 연구에 의해 설명되었고, 따라서 그 분야의 역사는 이 과학자들과 그들의 실험에 대한 이해에 달려있다.
이 모든 것은 박테리아에서 변형 현상으로 시작되는데, 1928년 프레드릭 그리피스는 한 박테리아에서 다른 박테리아로 변형되는 현상을 관찰했다.당시 그는 변혁의 현상을 설명할 수 없었다.이후 1944년, 세 명의 과학자 오스왈드 에이버리, 콜린 매클리드, 매클린은 박테리아에서 일어나는 모든 변형 현상을 증명했다.2년 후인 1930년에 분자생물학은 과학의 공식 분야로 확립되었다.하지만 "분자 생물학"이라는 용어는 1938년까지 만들어지지 않았고 그것은 록펠러 [4]재단의 자연과학 책임자로 일하던 과학자 워렌 위버가 만든 것입니다.
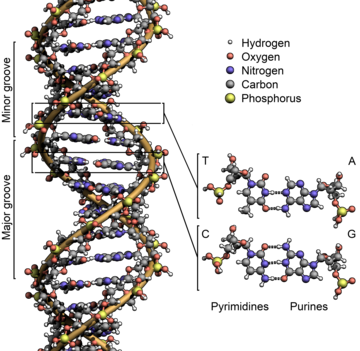
다음 실험에서 DNA가 유전자 변화를 일으킨 기본 유전 물질이라는 결론이 내려졌다.DNA의 기본 구성은 아데닌, 구아닌, 티민, 시토신으로 알려진 네 가지 염기를 포함하고 있는 것으로 알려졌다.모리스 윌킨스와 로잘린드 프랭클린이 한 화학조성과 X선 결정학의 기초 위에 DNA 구조가 제임스 왓슨과 프란시스 크릭에 의해 제안되었습니다.그러나 왓슨과 크릭이 DNA 구조를 제안하기 전, 1950년 오스트리아 태생의 과학자 에르빈 차르가프는 아데닌과 티민, 구아닌과 시토신의 수가 동일한 [4]비율이라는 이론과 규칙을 제안했다.
샤가프의 법칙
Chargaff의 법칙은 어떤 생물종의 DNA도 푸린과 피리미딘의 1:1 화학이론적 비율(즉, A+G=T+C)을 가져야 하며, 더 구체적으로 구아닌의 양은 시토신과 같아야 하며 아데닌의 양은 티민과 같아야 한다고 말했습니다. 이 패턴은 DNA의 양쪽 가닥에서 발견됩니다."[4]
유전학 분야는 유전의 분자 메커니즘과 유전자의 구조를 이해하기 위한 시도로 시작되었다.그레고르 멘델은 1866년 완두콩 [13]식물의 짝짓기 십자가에 대한 연구를 바탕으로 유전 유전법칙을 처음 썼을 때 이 작품을 개척했다.그러한 유전적 유전의 법칙 중 하나는 특정 유전자에 대해 두 개의 대립 유전자가 있는 이배체 개체는 그들의 [14]자손에게 이러한 대립 유전자들 중 하나를 전달한다는 분리 법칙이다.그의 중요한 연구 때문에, 유전 유전 연구는 일반적으로 멘델 유전학으로 언급된다.[15]
분자생물학의 중요한 이정표는 DNA 구조의 발견이었다.이 작업은 1869년 스위스의 생화학자인 프리드리히 미셰르에 의해 시작되었는데, 그는 우리가 현재 디옥시리보핵산, [16]즉 DNA로 알려진 핵이라고 불리는 구조를 처음으로 제안했다.그는 고름이 채워진 붕대의 성분을 연구하고 인을 [17]함유하는 물질의 고유한 특성에 주목함으로써 이 독특한 물질을 발견했다.DNA 모델의 또 다른 주목할 만한 기여자는 [18]효모에 대한 생화학 실험의 결과로 1919년 DNA의 "폴리뉴클레오티드 모델"을 제안한 피버스 레베네였다.1950년, Erwin Chargaff는 Levene의 연구를 확장하여 핵산의 몇 가지 중요한 특성을 설명했습니다. 첫째,[19] 핵산의 배열은 종에 따라 다릅니다.둘째, 푸린(아데닌 및 구아닌)의 총 농도는 항상 피리미딘(시스테인 및 티민)[16]의 총 농도와 동일합니다.이것은 이제 Chargaff의 법칙으로 알려져 있다.1953년 제임스 왓슨과 프란시스 크릭은 로잘린드 프랭클린과 모리스 윌킨스의 X선 결정학 연구를 이용하여 DNA의 [20]이중 나선 구조를 발표했다.왓슨과 크릭은 DNA의 구조를 설명했고 [20]DNA 복제의 가능한 메커니즘에 대한 이 독특한 구조의 함의에 대해 추측했다.
J.D. 왓슨과 F.H. C. 크릭은 DNA [4]구조의 모형을 제안한 공로로 1962년 모리스 윌켄스와 함께 노벨상을 받았다.
시간이 지남에 따라 1964년에 K.A.Marcker와 Frederick Sanger는 E.coli에서 N-formyl-methionyl – tRNA라고 불리는 독특한 아미노아실-tRNA를 발견했고 이 분자가 사슬 연장의 특별한 메커니즘에 역할을 한다고 설명했다.그는 F' 174 박테리오파지의 [4]단일 가닥 DNA 5,400개의 완전한 염기서열을 발견한 공로로 두 번째 노벨상을 받았다.
1961년에, 유전자가 단백질을 코드할 때, 유전자의 DNA의 세 개의 순차적인 염기가 [21]단백질의 각각의 연속된 아미노산을 지정한다는 것이 증명되었다.따라서 유전자 코드는 트리플렛 코드이며, 여기서 각 트리플렛은 특정 아미노산을 지정한다.또한 단백질을 코드하는 DNA 배열에서 코돈이 서로 겹치지 않고 각 배열을 일정한 시작점에서 읽어내는 것으로 나타났다.
1962-1964년 동안, [22]박테리아 바이러스의 조건부 치사 돌연변이를 사용함으로써, DNA 복제, DNA 복구, DNA 재조합 및 분자 구조의 조립에 사용되는 단백질의 기능과 상호작용에 대한 우리의 이해에 근본적인 발전이 이루어졌다.
F.그리피스 실험
1928년 프레드릭 그리피스는 폐렴구균 박테리아에서 독성을 발견했는데, 그것은 실험용 쥐를 죽이고 있었다.당시 유행했던 멘델에 따르면 유전자 전달은 부모 세포에서 딸 세포로만 일어날 수 있었다.그리피스는 같은 세대의 구성원에게서 일어나는 유전자 전달을 수평 유전자 전달(HGT)이라고 하며 또 다른 이론을 발전시켰다.이 현상은 현재 유전자 변형이라고 불린다.
그리피스는 두 가지 다른 변종인 스트렙토코커스 폐렴균에 대해 언급했는데 하나는 독성이 있고 매끄럽고 다른 하나는 독성이 있고 거칠다.매끄러운 균주는 특정 다당류(포도당과 글루쿠론산 캡슐의 중합체)의 존재로 인해 반짝이는 모습을 보였다.이 다당류 박테리아의 층 때문에 숙주의 면역 체계는 박테리아를 인식하지 못하고 숙주를 죽인다.다른, 독성이 강한 거친 품종은 이 다당류 캡슐이 없고, 둔하고 거친 외관을 가지고 있다.
변종 내 캡슐의 유무는 유전적으로 결정되는 것으로 알려져 있다.매끄러운 균주와 거친 균주는 각각 S-I, S-II, S-II 등 여러 가지 다른 유형으로 발생한다.S와 R 박테리아의 모든 아형은 그들이 생성하는 [4]항원 유형에서 서로 다르다.
허쉬와 체이스 실험
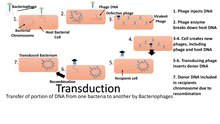
허쉬와 체이스의 실험에서 DNA가 감염의 원인이 되는 유전 물질이라는 것이 확인되었다.그들은 실험을 위해 E.coli와 박테리오파지를 사용했다.이 실험은 또한 믹서 실험으로 알려져 있는데, 주방 믹서가 주요 기기로 사용되었기 때문이다.Alfred Hershey와 Martha Chase는 파지 입자에 의해 박테리아에 주입된 DNA가 자손 파지 입자를 합성하는데 필요한 모든 정보를 포함하고 있다는 것을 증명했다.그들은 방사능을 이용하여 박테리오파지의 단백질 외피에는 방사성 황을, DNA에는 방사성 인을 각각 두 개의 다른 시험관에 부착했다.시험관에 박테리오파지와 E.coli를 혼합한 후, 파지가 E.coli 세포의 유전 물질을 변형시키는 잠복기가 시작된다.그런 다음 혼합물을 혼합하거나 교반하여 E.coli 세포에서 파지를 분리합니다.전체 혼합물을 원심분리하여 E.coli 세포를 포함하는 펠릿을 검사하여 상등액을 폐기하였다.E.coli 세포는 방사성 인을 보여주었는데, 이것은 변형된 물질이 단백질 피막이 아닌 DNA임을 나타냈다.
변형된 DNA는 E.coli의 DNA에 부착되고 방사능은 박테리오파지의 DNA에서만 보입니다.이 변이된 DNA는 다음 세대에 전해질 수 있고 변환이론이 생겨났다.도입은 박테리아 DNA가 박테리오파지의 파편을 운반해 다음 세대에 전달하는 과정이다.이것은 또한 수평적 유전자 [4]이동의 한 종류이다.
현대 분자생물학
20대 중반으로 접어들면서 분자생물학은 수직적, 수평적 기술 발전에 의해 정의되는 황금시대로 접어들고 있다.수직적으로, 새로운 기술은 원자 [23]수준에서 생물학적 과정을 실시간으로 감시할 수 있게 해준다.오늘날 분자생물학자들은 점점 더 높은 깊이에서 점점 더 저렴한 배열 데이터에 접근할 수 있으며, 새로운 비모델 유기체에서의 새로운 유전자 조작 방법의 개발을 촉진합니다.마찬가지로, 합성 분자 생물학자들은 다양한 원핵 및 진핵 세포주에서의 [24]외인성 대사 경로의 도입을 통해 작은 분자와 매크로 분자의 산업 생산을 촉진할 것이다.
수평적으로, 데이터 시퀀싱이 보다 저렴해지고 다양한 과학 분야에서 활용되고 있습니다.이것은 개발도상국의 산업 발전을 촉진하고 연구자 개개인의 접근성을 높일 것이다.마찬가지로, CRISPR-Cas9 유전자 편집 실험은 이제 개인에 의해 10,000달러 이하의 새로운 유기체에 의해 구상되고 실행될 수 있으며, 이것은 산업 및 의료 애플리케이션의 개발을 촉진할 것이다.
다른 생물과학과의 관계
아래 목록은 분자생물학과 다른 관련 [26]분야 사이의 학문 간 관계에 대한 관점을 설명한다.
- 분자생물학은 분자합성, 변형, 메커니즘 및 상호작용에 초점을 맞춘 생물학적 현상의 분자적 기초에 대한 연구이다.
- 생화학은 살아있는 유기체에서 일어나는 화학 물질과 중요한 과정을 연구하는 학문이다.생화학자는 단백질, 지질, 탄수화물, 핵산과 [27]같은 생체 분자의 역할, 기능, 구조에 집중한다.
- 유전학은 유전적 차이가 유기체에 어떻게 영향을 미치는지에 대한 연구이다.유전학은 돌연변이, 개별 유전자, 유전자 상호작용이 표현형의[28] 발현에 어떻게 영향을 미칠 수 있는지를 예측하려고 시도한다.
연구자들이 분자 생물학에 특화된 기술을 연습하는 동안, 이것들을 유전학과 생화학에서 나온 방법들과 결합하는 것은 흔한 일이다.분자생물학의 대부분은 양적인 것이고, 최근에는 생물정보학이나 계산생물학과 같은 컴퓨터 과학 기술을 사용하여 상당한 양의 작업이 수행되었다.유전자 구조와 기능에 대한 연구인 분자 유전학은 2000년대 초부터 분자생물학의 가장 두드러진 하위 분야 중 하나였다.생물학의 다른 부문은 세포생물학과 발달생물학과 같이 분자 자체의 상호작용을 직접 연구하거나 간접적으로 분자기술이 모집단이나 종의 역사적 속성을 추론하는 데 사용됨으로써 분자생물학에 의해 알려진다.유전학과 계통학입니다.생물물리학에서는 [29]생체분자를 처음부터, 혹은 분자적으로 연구하는 오랜 전통이 있다.
분자생물학 기술
분자 복제
분자 복제는 관심 있는 DNA 서열을 분리한 후 플라스미드 [30]벡터에 전달하기 위해 사용된다.이 재조합 DNA 기술은 1960년대에 [31]처음 개발되었습니다.본 발명은 중합효소사슬반응(PCR) 및/또는 제한효소를 사용하여 관심단백질을 코드하는 DNA배열을 플라스미드(발현벡터)로 복제한다.플라스미드 벡터는 보통 복제의 기원, 다중 복제 사이트(MCS) 및 선택적 마커(일반적으로 항생제 내성)의 적어도 세 가지 뚜렷한 특징을 가지고 있습니다.또한 MCS의 상류에는 프로모터 영역과 복제 유전자의 발현을 조절하는 전사 개시 부위가 있다.
이 플라스미드는 박테리아나 동물 세포에 삽입될 수 있다.박테리아 세포에 DNA를 도입하는 것은 벌거벗은 DNA의 흡수를 통한 형질전환, 세포-세포 접촉을 통한 결합 또는 바이러스 벡터를 통한 형질전환에 의해 이루어질 수 있다.물리적 또는 화학적 수단으로 동물 세포와 같은 진핵 세포에 DNA를 도입하는 것을 트랜스펙션이라고 한다.인산칼슘 트랜스펙션, 일렉트로포레이션, 미세주입 및 리포좀 트랜스펙션과 같은 몇 가지 다른 트랜스펙션 기술을 사용할 수 있습니다.플라스미드는 게놈에 통합되어 안정된 트랜스펙션을 일으키거나 게놈으로부터 독립하여 일시적으로 발현되는 과도 [32][33]트랜스펙션이라고 할 수 있습니다.
관심 단백질을 코드하는 DNA는 이제 세포 안에 있고, 그 단백질은 이제 발현될 수 있다.유도성 촉진제 및 특정 세포 신호 인자와 같은 다양한 시스템을 사용하여 관심 단백질을 높은 수준으로 발현할 수 있습니다.그 후 세균 또는 진핵세포에서 다량의 단백질을 추출할 수 있다.단백질은 다양한 상황에서 효소 활성을 시험할 수 있고, 단백질은 결정화되어 3차 구조를 연구할 수 있으며, 제약업계에서 단백질에 대한 신약 활성을 [34]연구할 수 있다.
중합효소 연쇄반응
중합효소 연쇄반응(PCR)은 DNA를 복제하기 위한 매우 다용도 기술이다.간단히 말해 PCR은 특정 DNA 배열을 미리 정해진 방법으로 복사하거나 수정할 수 있도록 한다.이 반응은 매우 강력하며, 완벽한 조건에서는 한 개의 DNA 분자를 증폭시켜 2시간 이내에 10억 7천만 개의 분자가 될 수 있다.PCR은 유전자 발현 연구, 병원성 미생물 검출, 유전자 돌연변이 검출, [35]DNA로의 돌연변이 도입 등 많은 응용 분야를 가지고 있다.PCR 기술은 제한 효소 부위를 DNA 분자의 끝에 도입하거나 DNA의 특정 염기를 돌연변이시키기 위해 사용될 수 있으며, 후자는 부위 지향적 돌연변이 유발이라고 불리는 방법이다.PCR은 또한 특정 DNA 조각이 cDNA 라이브러리에서 발견되는지 여부를 결정하기 위해 사용될 수 있습니다.PCR에는 RNA의 증폭을 위한 역전사 PCR(RT-PCR)과 보다 최근에는 DNA 또는 RNA [36][37]분자의 정량적 측정을 가능하게 하는 정량적 PCR과 같은 많은 변형이 있다.
겔 전기영동
겔 전기영동은 아가로스 또는 폴리아크릴아미드 [38]겔을 사용하여 분자를 크기에 따라 분리하는 기술이다.이 기술은 분자생물학의 주요 도구 중 하나이다.기본적인 원리는 DNA 조각이 겔 전체에 전류를 가함으로써 분리될 수 있다는 것이다. DNA 골격은 음전하를 띤 인산기를 포함하고 있기 때문에, DNA는 아가로스 겔을 통해 [38]전류의 양의 끝을 향해 이동할 것이다.또한 단백질은 SDS-PAGE 겔을 사용한 사이즈 또는 2D [39]겔 전기영동으로 알려진 것을 사용하여 크기와 전하를 기준으로 분리될 수 있다.
브래드포드 어세이
Bradford Assay는 Coomassie Brilliant Blue [40]G-250이라고 불리는 염료의 독특한 특성을 이용하여 단백질 분자의 빠르고 정확한 정량화를 가능하게 하는 분자생물학 기술이다.쿠마시 블루는 [40]단백질에 결합하면 적갈색에서 밝은 파란색으로 가시적인 색 변화를 겪습니다.쿠마시 블루는 불안정한 양이온 상태에서 배경 파장이 465nm이고 적갈색을 [41]발산한다.쿠마시 블루가 산성 용액에서 단백질과 결합하면 배경 파장이 595 nm로 바뀌고 염료가 밝은 파란색을 [41]발산합니다.이 분석에서 단백질은 쿠마시 블루와 약 2분 만에 결합하고 단백질-염색 복합체는 반응 개시 [40]후 5분에서 20분 이내에 흡광도를 측정하는 것이 권장되지만 약 1시간 동안 안정적입니다.Bradford 분석의 단백질 농도는 가시광선 분광 광도계를 사용하여 측정할 수 있으므로 광범위한 [41]장비가 필요하지 않습니다.
이 방법은 Marion M. Bradford에 의해 1975년에 개발되었으며 이전의 방법인 Lowry 시술과 biuret [40]assay에 비해 상당히 빠르고 정확한 단백질 정량화를 가능하게 했다.이전 방법과는 달리 Bradford 검사는 에탄올, 염화나트륨 및 [40]염화마그네슘을 포함한 여러 비단백질 분자의 간섭을 받지 않습니다.그러나 황산나트륨(SDS)[40]과 같은 강한 알칼리성 완충제의 영향을 받기 쉽다.
고분자 블롯 및 프로브
노던, 웨스턴, 이스턴 블롯이라는 용어는 에드윈 서던이 블롯된 DNA의 잡종을 위해 기술한 후에 서던 블롯이라는 용어를 사용한 분자생물학적 농담에서 유래했다.e term.[42]
서던 블롯
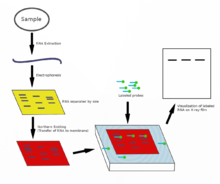
발명자인 생물학자인 에드윈 서던의 이름을 딴 서던 블롯은 DNA 샘플 내의 특정 DNA 배열의 존재를 조사하는 방법이다.제한효소(제한핵산가수분해효소) 소화 전후의 DNA 샘플을 겔 전기영동에 의해 분리한 후 모세관 작용을 통해 블롯에 의해 막으로 이행한다.그런 다음 해당 [43]막은 관심 DNA의 염기서열을 보완하는 염기서열을 가진 라벨이 부착된 DNA 프로브에 노출됩니다.서던 블롯은 DNA 샘플에서 특정 DNA 서열을 검출하는 PCR과 같은 다른 기술의 능력 때문에 실험실 과학에서 덜 일반적으로 사용됩니다.그러나 이러한 블롯은 트랜스제닉 마우스 또는 유전자 녹아웃 배아줄기세포주의 [29]엔지니어링에서 트랜스제닉 복제 수를 측정하는 것과 같은 일부 응용 분야에 여전히 사용됩니다.
노던 블롯
노던 블롯은 RNA의 서로 다른 샘플 세트 간의 상대적 비교로서 특정 RNA 분자의 존재를 연구하는 데 사용됩니다.이것은 본질적으로 변성 RNA 겔 전기영동과 블롯의 조합이다.이 과정에서 RNA는 크기에 따라 분리되고 이어서 관심 시퀀스의 라벨이 부착된 보체로 프로빙되는 막으로 옮겨진다.결과는 사용된 라벨에 따라 다양한 방법으로 시각화할 수 있지만, 대부분 샘플에서 검출된 RNA의 크기를 나타내는 밴드가 노출됩니다.이러한 대역의 강도는 분석된 검체의 표적 RNA 양과 관련이 있습니다.이 절차는 전사 후 조절이 일어나지 않고 mRNA의 수준이 생산되는 단백질의 비례적 수준을 반영한다고 가정할 때 다양한 샘플에 얼마나 많은 RNA가 존재하는지 측정하여 유전자 발현을 언제, 얼마나 발생하는지 연구하는 데 일반적으로 사용됩니다.그것은 언제, 어떤 조건에서, 특정 유전자가 살아있는 조직에서 [44][45]발현되는지를 결정하는 가장 기본적인 도구 중 하나이다.
웨스턴 블롯
웨스턴 블롯은 [46]단백질의 혼합물에서 특정 단백질을 검출할 수 있는 기술이다.웨스턴 블롯은 분리된 단백질의 크기를 결정하고 그들의 발현을 [47]정량화하는 데 사용될 수 있다.웨스턴 블롯팅에서 단백질은 SDS-PAGE로 알려진 기술로 두 개의 유리판 사이에 끼워진 얇은 겔에서 크기별로 먼저 분리된다.그리고 나서 겔 내의 단백질은 폴리불화비닐리덴(PVDF), 니트로셀룰로오스, 나일론 또는 다른 지지막으로 옮겨집니다.그리고 나서 이 막은 항체 용액으로 탐사될 수 있다.관심 단백질에 특이하게 결합하는 항체는 착색 제품, 화학 발광 또는 자동 방사선 촬영을 포함한 다양한 기술로 시각화할 수 있습니다.종종, 항체들은 효소로 라벨이 붙여진다.화학 발광 기질이 효소에 노출되면 검출이 가능합니다.웨스턴 블로팅 기술을 사용하면 검출뿐만 아니라 정량 분석도 가능합니다.웨스턴 블로팅과 유사한 방법은 살아있는 세포 또는 [46][48]조직 부분에서 특정 단백질을 직접 염색하는 데 사용될 수 있다.
이스턴 블롯팅
이스턴 블로팅 기술은 단백질의 번역 후 변형을 검출하기 위해 사용된다.PVDF 또는 니트로셀룰로오스 막에 블롯된 단백질은 특정 [49]기질을 사용하여 변형을 위해 프로빙된다.
마이크로어레이
DNA 마이크로어레이는 현미경 슬라이드 등의 고체 지지체에 부착된 점의 집합으로, 각 점은 하나 이상의 단가닥 DNA 올리고뉴클레오티드 단편을 포함한다.어레이를 사용하면, 1개의 슬라이드에 다량의 매우 작은(직경 100 마이크로미터) 스팟을 배치할 수 있습니다.각 점에는 단일 DNA 염기서열을 보완하는 DNA 조각 분자가 있습니다.이 기술의 변형은 발달의 특정 단계에서 유기체의 유전자 발현을 검증할 수 있게 한다(발현 프로파일링).이 기술에서 조직의 RNA는 분리되고 레이블이 부착된 보완 DNA(cDNA)로 변환됩니다.이 cDNA는 어레이상의 fragment에 하이브리드화되어 하이브리드화를 시각화할 수 있습니다.복수의 배열은 정확히 동일한 조각의 위치로 만들어질 수 있기 때문에, 그것들은 건강한 조직과 암 조직과 같은 두 개의 다른 조직의 유전자 발현을 비교하는데 특히 유용하다.또한, 어떤 유전자가 발현되는지, 그리고 그 표현이 시간이나 다른 요인들과 함께 어떻게 변화하는지 측정할 수 있다.마이크로어레이를 제작하는 방법에는 여러 가지가 있습니다.가장 일반적인 것은 실리콘 칩, 직경 100마이크로미터 이하의 현미경 슬라이드, 커스텀 어레이, 다공질막(매크로어레이)에 큰 반점이 있는 어레이입니다.특정 어레이에는 100개에서 10,000개 이상의 스팟이 존재할 수 있습니다.배열은 [50][51][52][53]DNA 이외의 분자로도 만들 수 있다.
알레알 특이적 올리고뉴클레오티드
알레알 특정 올리고뉴클레오티드(ASO)는 PCR이나 겔 전기영동 없이도 단일 염기 돌연변이를 검출할 수 있는 기술이다.짧은 길이(길이 20-25 뉴클레오티드)로 라벨이 부착된 탐침은 비분할 표적 DNA에 노출되며, 탐침의 길이가 짧기 때문에 높은 특이성으로 교배되고 단일 염기 변화도 교배를 방해한다.그런 다음 표적 DNA를 세척하고 잡종화되지 않은 표지 탐침은 제거된다.그런 다음 대상 DNA가 방사능 또는 형광을 통해 프로브의 존재를 분석합니다.이 실험에서는 대부분의 분자생물학 기술과 마찬가지로 성공적인 [54][55]실험을 보장하기 위해 제어가 사용되어야 한다.
분자생물학에서는 절차와 기술이 지속적으로 개발되고 있으며 오래된 기술은 폐기되고 있습니다.예를 들어, DNA 겔 전기영동(아가로스 또는 폴리아크릴아미드)이 등장하기 전에는 DNA 분자의 크기가 일반적으로 수크로스 구배에서의 속도 침전에 의해 결정되었으며, 수크로스 구배 전에는 점도가 사용되었다.그들의 역사적 관심과는 별개로,[56] 새로운 기술이 부적절한 또 다른 새로운 문제를 해결하는 것이 때때로 유용하기 때문에 오래된 기술에 대해 알 가치가 있는 경우가 많습니다.
「 」를 참조해 주세요.
레퍼런스
- ^ Alberts B, Johnson A, Lewis J, Morgan D, Raff M, Roberts K, Walter P (2014). Molecular Biology of the Cell, Sixth Edition. Garland Science. pp. 1–10. ISBN 978-1-317-56375-4.
- ^ a b Gannon F (February 2002). "Molecular biology--what's in a name?". EMBO Reports. 3 (2): 101. doi:10.1093/embo-reports/kvf039. PMC 1083977. PMID 11839687.
- ^ "Molecular biology - Latest research and news Nature". www.nature.com. Retrieved 2021-11-07.
- ^ a b c d e f g h i S., Verma, P. (2004). Cell biology, genetics, molecular biology, evolution and ecology. S Chand and Company. ISBN 81-219-2442-1. OCLC 1045495545.
- ^ Astbury, W. T. (June 1961). "Molecular Biology or Ultrastructural Biology ?". Nature. 190 (4781): 1124. Bibcode:1961Natur.190.1124A. doi:10.1038/1901124a0. ISSN 1476-4687. PMID 13684868. S2CID 4172248.
- ^ a b Morange, Michel (2016-02-15), "History of Molecular Biology", eLS, Chichester, UK: John Wiley & Sons, Ltd, pp. 1–8, doi:10.1002/9780470015902.a0003079.pub3, ISBN 9780470016176, retrieved 2021-11-07
- ^ "Polymerase Chain Reaction (PCR)", Definitions, Qeios, 2019-11-26, doi:10.32388/167113, S2CID 94561339, retrieved 2021-11-07
- ^ "Smithsonian Institution Archives". siarchives.si.edu. Retrieved 2021-11-07.
- ^ Cox, Michael M. (2015-03-16). Molecular biology: principles and practice. Doudna, Jennifer A.,, O'Donnell, Michael (Biochemist) (Second ed.). New York. ISBN 978-1-4641-2614-7. OCLC 905380069.
- ^ Bello, Elizabeth A.; Schwinn, Debra A. (1996-12-01). "Molecular Biology and Medicine: A Primer for the Clinician". Anesthesiology. 85 (6): 1462–1478. doi:10.1097/00000542-199612000-00029. ISSN 0003-3022. PMID 8968195. S2CID 29581630.
- ^ Morange, Michel (June 2021). A history of biology. ISBN 978-0-691-18878-2. OCLC 1184123419.
- ^ Fields, Stanley (2001-08-28). "The interplay of biology and technology". Proceedings of the National Academy of Sciences. 98 (18): 10051–10054. doi:10.1073/pnas.191380098. ISSN 0027-8424. PMC 56913. PMID 11517346.
- ^ Ellis, T. H. Noel; Hofer, Julie M. I.; Timmerman-Vaughan, Gail M.; Coyne, Clarice J.; Hellens, Roger P. (2011-11-01). "Mendel, 150 years on". Trends in Plant Science. 16 (11): 590–596. doi:10.1016/j.tplants.2011.06.006. ISSN 1360-1385. PMID 21775188.
- ^ "12.3C: Mendel's Law of Segregation". Biology LibreTexts. 2018-07-12. Retrieved 2021-11-18.
- ^ "Mendelian Inheritance". Genome.gov. Retrieved 2021-11-18.
- ^ a b "Discovery of DNA Double Helix: Watson and Crick Learn Science at Scitable". www.nature.com. Retrieved 2021-11-25.
- ^ George., Wolf (2003). Friedrich Miescher: the man who discovered DNA. OCLC 907773747.
- ^ Levene, P.A. (1919). "Structure of Yeast Nucleic Acid". Journal of Biological Chemistry. 43 (2): 379–382. doi:10.1016/s0021-9258(18)86289-5. ISSN 0021-9258.
- ^ Chargaff, Erwin (1950). "Chemical specificity of nucleic acids and mechanism of their enzymatic degradation". Experientia. 6 (6): 201–209. doi:10.1007/bf02173653. ISSN 0014-4754. PMID 15421335. S2CID 2522535.
- ^ a b Watson, J. D.; Crick, F. H. C. (April 1953). "Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid". Nature. 171 (4356): 737–738. Bibcode:1953Natur.171..737W. doi:10.1038/171737a0. ISSN 1476-4687. PMID 13054692. S2CID 4253007.
- ^ Crick, F. H. C.; Barnett, Leslie; Brenner, S.; Watts-Tobin, R. J. (1961). "General Nature of the Genetic Code for Proteins". Nature. Springer Science and Business Media LLC. 192 (4809): 1227–1232. Bibcode:1961Natur.192.1227C. doi:10.1038/1921227a0. ISSN 0028-0836. PMID 13882203. S2CID 4276146.
- ^ Epstein, R. H.; Bolle, A.; Steinberg, C. M.; Kellenberger, E.; Boy de la Tour, E.; et al. (1963-01-01). "Physiological Studies of Conditional Lethal Mutants of Bacteriophage T4D". Cold Spring Harbor Symposia on Quantitative Biology. Cold Spring Harbor Laboratory. 28: 375–394. doi:10.1101/sqb.1963.028.01.053. ISSN 0091-7451.
- ^ Mojiri, Soheil; Isbaner, Sebastian; Mühle, Steffen; Jang, Hongje; Bae, Albert Johann; Gregor, Ingo; Gholami, Azam; Gholami, Azam; Enderlein, Jörg (2021-06-01). "Rapid multi-plane phase-contrast microscopy reveals torsional dynamics in flagellar motion". Biomedical Optics Express. 12 (6): 3169–3180. doi:10.1364/BOE.419099. ISSN 2156-7085. PMC 8221972. PMID 34221652.
- ^ van Warmerdam, T. "Molecular Biology Laboratory Resource". Yourbiohelper.com.
{{cite web}}: CS1 maint :url-status (링크) - ^ van Warmerdam, T. "Molecular biology laboratory resource". Yourbiohelper.com.
{{cite web}}: CS1 maint :url-status (링크) - ^ Lodish H, Berk A, Zipursky SL, Matsudaira P, Baltimore D, Darnell J (2000). Molecular cell biology (4th ed.). New York: Scientific American Books. ISBN 978-0-7167-3136-8.
- ^ Berg, Jeremy (2002). Biochemistry. Tymoczko, John L.; Stryer, Lubert (5th ed.). New York: W.H. Freeman. ISBN 0-7167-3051-0. OCLC 48055706.
- ^ Reference, Genetics Home. "Help Me Understand Genetics". Genetics Home Reference. Retrieved 31 December 2016.
- ^ a b Tian J, ed. (2013). Molecular Imaging: Fundamentals and Applications. Springer-Verlag Berlin & Heidelberg GmbH & Co. K. p. 542. ISBN 9783642343032. Retrieved 2019-07-08.
- ^ "Foundations of Molecular Cloning - Past, Present and Future NEB". www.neb.com. Retrieved 2021-11-25.
- ^ "Foundations of Molecular Cloning - Past, Present and Future NEB". www.neb.com. Retrieved 2021-11-04.
- ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P. Isolating, Cloning, and Sequencing DNA. Retrieved 31 December 2016.
- ^ Lessard, Juliane C. (1 January 2013). "Molecular cloning". Laboratory Methods in Enzymology: DNA. Methods in Enzymology. Vol. 529. pp. 85–98. doi:10.1016/B978-0-12-418687-3.00007-0. ISBN 978-0-12-418687-3. ISSN 1557-7988. PMID 24011038.
- ^ Kokate C, Jalalpure SS, Hurakadle PJ (2016). Textbook of Pharmaceutical Biotechnology. Expression Cloning. Elsevier. p. 125. ISBN 9788131239872. Retrieved 2019-07-08.
- ^ Lenstra, J. A. (July 1995). "The applications of the polymerase chain reaction in the life sciences". Cellular and Molecular Biology (Noisy-Le-Grand, France). 41 (5): 603–614. ISSN 0145-5680. PMID 7580841.
- ^ "Polymerase Chain Reaction (PCR)". National Center for Biotechnology Information. U.S. National Library of Medicine. Retrieved 31 December 2016.
- ^ "Polymerase Chain Reaction (PCR) Fact Sheet". National Human Genome Research Institute (NHGRI). Retrieved 31 December 2016.
- ^ a b Lee, Pei Yun; Costumbrado, John; Hsu, Chih-Yuan; Kim, Yong Hoon (2012-04-20). "Agarose Gel Electrophoresis for the Separation of DNA Fragments". Journal of Visualized Experiments (62): 3923. doi:10.3791/3923. ISSN 1940-087X. PMC 4846332. PMID 22546956.
- ^ Lee PY, Costumbrado J, Hsu CY, Kim YH (April 2012). "Agarose gel electrophoresis for the separation of DNA fragments". Journal of Visualized Experiments (62). doi:10.3791/3923. PMC 4846332. PMID 22546956.
- ^ a b c d e f Bradford, Marion M. (1976-05-07). "A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding". Analytical Biochemistry. 72 (1): 248–254. doi:10.1016/0003-2697(76)90527-3. ISSN 0003-2697. PMID 942051.
- ^ a b c "Protein determination by the Bradford method". www.ruf.rice.edu. Retrieved 2021-11-08.
- ^ Thomas PS (September 1980). "Hybridization of denatured RNA and small DNA fragments transferred to nitrocellulose". Proceedings of the National Academy of Sciences of the United States of America. 77 (9): 5201–5. Bibcode:1980PNAS...77.5201T. doi:10.1073/pnas.77.9.5201. PMC 350025. PMID 6159641.
- ^ Brown T (May 2001). "Southern blotting". Current Protocols in Immunology. Chapter 10: Unit 10.6A. doi:10.1002/0471142735.im1006as06. ISBN 978-0-471-14273-7. PMID 18432697. S2CID 20686993.
- ^ Josefsen K, Nielsen H (2011). Nielsen H (ed.). RNA methods and protocols. Methods in Molecular Biology. Vol. 703. New York: Humana Press. pp. 87–105. doi:10.1007/978-1-59745-248-9_7. ISBN 978-1-59745-248-9. PMID 21125485.
- ^ He SL, Green R (1 January 2013). "Northern blotting". Methods in Enzymology. 530: 75–87. doi:10.1016/B978-0-12-420037-1.00003-8. ISBN 978-0-12-420037-1. PMC 4287216. PMID 24034315.
- ^ a b Mahmood T, Yang PC (September 2012). "Western blot: technique, theory, and trouble shooting". North American Journal of Medical Sciences. 4 (9): 429–34. doi:10.4103/1947-2714.100998. PMC 3456489. PMID 23050259.
- ^ "Western blot Learn Science at Scitable". www.nature.com. Retrieved 2021-11-25.
- ^ Kurien BT, Scofield RH (April 2006). "Western blotting". Methods. 38 (4): 283–93. doi:10.1016/j.ymeth.2005.11.007. PMID 16483794. – Science Direct 경유 (서브스크립션이 필요하거나 라이브러리에서 콘텐츠를 이용할 수 있습니다.)
- ^ Thomas S, Thirumalapura N, Crossley EC, Ismail N, Walker DH (June 2009). "Antigenic protein modifications in Ehrlichia". Parasite Immunology. 31 (6): 296–303. doi:10.1111/j.1365-3024.2009.01099.x. PMC 2731653. PMID 19493209.
- ^ "Microarrays". National Center for Biotechnology Information. U.S. National Library of Medicine. Retrieved 31 December 2016.
- ^ Bumgarner R (January 2013). Frederick M. Ausubel, et al. (eds.). "Overview of DNA microarrays: types, applications, and their future". Current Protocols in Molecular Biology. Chapter 22: Unit 22.1. doi:10.1002/0471142727.mb2201s101. ISBN 978-0-471-14272-0. PMC 4011503. PMID 23288464.
- ^ Govindarajan R, Duraiyan J, Kaliyappan K, Palanisamy M (August 2012). "Microarray and its applications". Journal of Pharmacy & Bioallied Sciences. 4 (Suppl 2): S310-2. doi:10.4103/0975-7406.100283. PMC 3467903. PMID 23066278.
- ^ Tarca AL, Romero R, Draghici S (August 2006). "Analysis of microarray experiments of gene expression profiling". American Journal of Obstetrics and Gynecology. 195 (2): 373–88. doi:10.1016/j.ajog.2006.07.001. PMC 2435252. PMID 16890548.
- ^ Cheng L, Zhang DY, eds. (2008). Molecular genetic pathology. Totowa, NJ: Humana. p. 96. ISBN 978-1-59745-405-6. Retrieved 31 December 2016.
- ^ Leonard DG (2016). Molecular Pathology in Clinical Practice. Springer. p. 31. ISBN 978-3-319-19674-9. Retrieved 31 December 2016.
- ^ Tian J, ed. (2013). Molecular Imaging: Fundamentals and Applications. Springer-Verlag Berlin & Heidelberg GmbH & Co.K. pp. 550, 552. ISBN 9783642343032. Retrieved 2019-07-08.
추가 정보
- Cohen SN, Chang AC, Boyer HW, Helling RB (November 1973). "Construction of biologically functional bacterial plasmids in vitro". Proceedings of the National Academy of Sciences of the United States of America. 70 (11): 3240–4. Bibcode:1973PNAS...70.3240C. doi:10.1073/pnas.70.11.3240. PMC 427208. PMID 4594039.
- Rodgers M (June 1975). "The Pandora's box congress". Rolling Stone. Vol. 189. pp. 37–77.
- Roberts K, Raff M, Alberts B, Walter P, Lewis J, Johnson A (2002). Molecular Biology of the Cell. Garland Science. ISBN 978-0-8153-3218-3.
외부 링크
| 라이브러리 리소스 정보 분자생물학 |