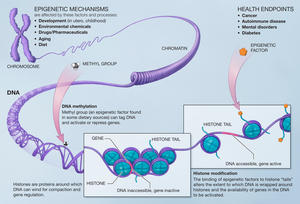
히스톤 메틸화
Histone methylation히스톤 메틸화는 메틸기가 뉴클레오솜을 구성하는 히스톤 단백질의 아미노산으로 전달되는 과정으로, DNA 이중 나선은 염색체를 형성하기 위해 감싸고 있다.히스톤의 메틸화는 히스톤의 어떤 아미노산이 메틸화되고 얼마나 많은 메틸기가 붙어 있는지에 따라 유전자의 전사를 증가시키거나 감소시킬 수 있다.히스톤 꼬리와 DNA 사이의 화학적 흡인력을 약화시키는 메틸화 사건은 DNA가 뉴클레오솜에서 풀릴 수 있게 하여 전사인자 단백질과 RNA 중합효소가 DNA에 접근할 수 있도록 하기 때문에 전사를 증가시킨다.이 과정은 다른 세포들이 다른 유전자를 발현하도록 하는 유전자 발현 조절에 매우 중요하다.
기능.
염색질 구조를 수정하는 메커니즘으로서의 히스톤 메틸화는 장기 기억 형성 및 [1]학습에 중요한 것으로 알려진 신경 경로의 자극과 관련된다.동물 모델은 메틸화 및 다른 후생유전학적 조절 메커니즘이 노화, 신경변성 질환, 지적[1] 장애와 관련이 있음을 보여 주었다(루빈스타인).–타이비 증후군, X-연계 지적 장애).[2]H3K4, H3K27, H4K20의 잘못된 조절은 [3]암과 관련이 있다.이 변형은 뉴클레오좀의 특성을 바꾸고 다른 단백질과의 상호작용, 특히 유전자 전사 과정에 영향을 미친다.
- 히스톤 메틸화는 전사 억제 또는 활성화와 관련될 수 있다.예를 들어 리신4(H3K4me3)에서 히스톤H3의 트리메틸화는 전사를 위한 활성 마크이며, 랫드에서 맥락적 공포조건화 후 1시간 후에 해마에서 상향 조절된다.그러나 공포조건화 또는 신규환경에 대한 [4]노광 후에 전사사일런트화 신호인 리신9(H3K9me2)에서의 히스톤H3의 디메틸화를 증가시킨다.
- 히스톤의 일부 리신(K) 및 아르기닌(R) 잔류물의 메틸화는 전사 활성화를 초래한다.예를 들어 히스톤3(H3K4me1)의 리신4의 메틸화 및 H3 및 H4의 아르기닌(R) 잔기를 들 수 있다.
- 히스톤 메틸전달효소에 의한 히스톤에 대한 메틸기의 첨가는 메틸화되는 아미노산 및 [5]근처에 다른 메틸기 또는 아세틸기의 존재에 따라 전사를 활성화하거나 더 억제할 수 있다.
메커니즘
뉴클레오솜이라고 불리는 염색질의 기본 단위는 단백질 옥타머에 감긴 DNA를 포함합니다.이 옥타머는 4개의 히스톤 단백질 각각 2개의 복사본으로 구성됩니다.H2A, H2B, H3, H4.이들 단백질 각각은 꼬리 연장선을 가지며, 이들 꼬리는 메틸화에 의한 뉴클레오솜 수식의 대상이다.DNA 활성화 또는 불활성화는 메틸화된 특정 꼬리 잔기와 메틸화 정도에 크게 좌우된다.히스톤은 리신(K) 및 아르기닌(R) 잔기에 대해서만 메틸화될 수 있지만, 메틸화는 히스톤 꼬리 H3 및 H4의 [6]리신 잔기에 가장 일반적으로 관찰된다.뉴클레오솜 코어에서 가장 멀리 있는 꼬리 끝은 N-말단이다(여기에는 이 끝부터 번호가 매겨진다).메틸화 반응 유전자 활성화와 관련된 일반 사이트들 유전자 불활성화에 H3K4, H3K48, H3K79. 일반적인 사이트 이런 사이트들의 H3K9과 H3K27.[7]연구에 따르면 히스톤 꼬리의 다른 잔류물에서 메틸화는 chrom을 규제하기 위해서 다양한 단백질이나 단백질 단지의 신입 사원 모집에 표지 역할을 하는 발견했다 등을 포함한다.에서활성화 또는 비활성화 시.
리신과 아르기닌 잔기는 모두 아미노기를 포함하고 있으며, 아미노기는 염기성 및 소수성 특성을 부여한다.리신은 NH3+ 그룹의 각 수소를 메틸기로 치환하여 모노, 디 또는 트리메틸화될 수 있다.유리 NH2 및 NH2+ 군에서 아르기닌은 모노메틸화 또는 디메틸화 될 수 있다.이 디메틸화는 NH2 그룹에서 비대칭적으로 발생하거나 각 [8]그룹에서 1개의 메틸화로 대칭적으로 발생할 수 있다.각 잔기에 메틸기를 추가할 때마다 다양한 기질 및 보조 인자를 가진 특정 단백질 효소 세트가 필요합니다.일반적으로 아르기닌 잔기의 메틸화는 단백질 아르기닌메틸전달효소(PRMT)를 포함한 복합체를 필요로 하며, 리신은 특정 히스톤메틸전달효소(HMT)를 필요로 하며, 보통 진화적으로 보존된 SET [9]도메인을 포함한다.
일반적으로 연구되는 H4K20 잔류물의 메틸화에서 예증된 바와 같이 잔류물의 메틸화 정도가 다르면 다른 기능을 부여할 수 있다.모노메틸화 H4K20(H4K20me1)은 크로마틴의 압축에 관여하여 전사 억제를 실시한다.그러나 H4K20me2는 손상된 DNA를 복구하는 데 매우 중요합니다.디메틸화되었을 때, 잔류물은 비호몰로지 말단 접합에 의한 이중사슬 DNA 파괴의 수복에 관여하는 단백질 53BP1의 결합을 위한 플랫폼을 제공한다.H4K20me3는 헤테로크로마틴에 농축되어 있으며, 이 트리메틸화의 감소는 암 진행에서 관찰된다.따라서 H4K20me3는 염색질 [9]억제에 추가적인 역할을 한다.염색질의 DNA 이중가닥절단 복구는 상동재조합에 의해서도 일어나며,[10] 손상 부위에 대한 복구효소의 접근을 용이하게 하기 위해 히스톤메틸화(H3K9me3)를 포함한다.
히스톤메틸전달효소
게놈은 염색질에 촘촘히 응축되어 있는데, 전사가 일어나려면 염색질이 느슨해져야 한다.유전자의 전사를 멈추기 위해서는 DNA가 더 강하게 감겨야 한다.이것은 메틸화에 의해 특정 부위의 히스톤을 수정함으로써 이루어질 수 있다.히스톤 메틸전달효소는 S-아데노실메티오닌(SAM)에서 H3 및 H4 히스톤의 리신 또는 아르기닌 잔기로 메틸기를 전달하는 효소이다.히스톤의 핵심 구상 도메인이 메틸화된 사례도 있다.
히스톤 메틸전달효소는 리신 또는 아르기닌에 특이적이다.리신 특이적 전달 효소는 SET 도메인이 있는지 또는 비 SET 도메인이 있는지 여부를 더욱 세분화한다.이러한 도메인은 효소가 SAM에서 전달 단백질로, 나아가 히스톤 [11]잔기로의 메틸 전달을 촉매하는 방법을 정확하게 지정합니다.메틸전달효소는 표적 잔류물에 1-3 메틸을 첨가할 수 있다.
히스톤에 첨가되는 이러한 메틸은 전사 인자에 대한 DNA 접근을 차단하거나 장려함으로써 전사를 조절하는 역할을 한다.이와 같이 게놈의 완전성과 유전자의 후생유전적 유전은 히스톤메틸전달효소의 작용에 의해 제어된다.히스톤 메틸화는 게놈의 무결성과 세포에 의해 발현되는 유전자를 구별하는 데 핵심적이며, 따라서 세포에 그들의 정체성을 부여한다.
메틸화 히스톤은 [11]전사를 억제하거나 활성화할 수 있다.예를 들어 H3K4me2, H3K4me3, H3K79me3는 일반적으로 전사 액티비티에 관련지어지지만 H3K9me2, H3K9me3, H3K27me2, H3K27me3, 및 H4K20me3은 전사 [12]액티비티에 관련지어집니다.
후생유전학
히스톤에 대한 수정은 세포 내에서 발현되는 유전자에 영향을 미치며, 이는 히스톤 메틸전달효소에 [13]의해 히스톤 잔기에 메틸이 첨가되는 경우이다.히스톤 메틸화는 헤테로크로마틴 메커니즘의 조립과 전사된 유전자와 그렇지 않은 유전자 사이의 유전자 경계 유지에 중요한 역할을 한다.이러한 변화는 자손에게 전해지고 세포가 영향을 받는 환경에 의해 영향을 받을 수 있다.후생유전학적 변화는 치료의 표적이 될 수 있다는 것을 의미한다.
히스톤 메틸전달효소의 활성은 히스톤 데메틸라아제 활성에 의해 상쇄된다.이것에 의해, 기존의 변경을 되돌리는 것으로써, 변환을 온/오프 할 수 있습니다.히스톤 메틸트랜스페라아제와 히스톤 데메틸라아제 모두의 활성을 엄격하게 조절해야 한다.둘 중 하나를 잘못 조절하면 유전자가 발현되어 질병에 걸리기 쉽다.많은 암들은 잘못 조절된 [14]메틸화의 부적절한 후생유전 효과에서 발생한다.그러나 이러한 과정은 때때로 되돌릴 수 있기 때문에 항암 [14]치료와 함께 그 활동을 활용하는 것에 관심이 있다.
X염색체 불활성화 시
여성 유기체에서는 X염색체를 포함한 정자가 난자를 수정해 X염색체를 두 개 복제한다.하지만, 여성들은 처음에는 X 염색체의 두 복사본이 필요하지 않습니다. 왜냐하면 X 염색체는 용량 보상의 가설에서 보여지는 것과 같이 전사된 단백질 생성물의 양만 두 배로 증가하기 때문입니다.아버지 X 염색체는 처음 몇 개의 [15]분할 동안 빠르게 불활성화된다.이 비활성 X 염색체 (Xi)는 [16]헤테로크로마틴이라고 불리는 믿을 수 없을 정도로 단단한 형태의 염색질에 채워져 있다.이 패킹은 다양한 히스톤 형성을 돕는 다양한 리신 잔기의 메틸화 때문에 발생합니다.인간에서 X 불활성화는 무작위 과정이며, 비부호화 RNA [17]XIST에 의해 매개된다.
리신 잔기의 메틸화는 많은 다른 히스톤에서 발생하지만, Xi의 가장 특징은 세 번째 히스톤(H3K9)의 아홉 번째 리신에서 발생한다.이 영역의 단일 메틸화는 유전자가 전사 [18]활성 상태로 유지되도록 하는 반면, 헤테로크로마틴에서는 종종 DNA 결합을 확실히 하기 위해 H3K9me2 또는 H3K9me3의 두 번 또는 세 번 메틸화된다.더 최근의 연구는 H3K27me3와 H4K20me1 또한 초기 배아에서 흔하다는 것을 보여주었다.DNA의 전사 활성 영역과 관련된 다른 메틸화 표시인 H3K4me2와 H3K4me3는 많은 아세틸화 표시와 함께 Xi 염색체에서 누락된다.특정 Xi히스톤 메틸화 표식이 종 간에 비교적 일정하게 유지되었다고 알려져 있지만, 최근 한 유기체 내의 다른 유기체와 심지어 다른 세포들이 그들의 X [19]불활성화에 대한 다른 표식을 가질 수 있다는 것이 발견되었다.히스톤 메틸화를 통해, 유전적인 각인이 있기 때문에, 염색체 복제와 세포 분열을 통해 동일한 X 호몰로지가 불활성화 상태를 유지합니다.
돌연변이
히스톤 메틸화가 유전자가 전사되는 것의 많은 부분을 조절한다는 사실 때문에, 메틸화 패턴에 약간의 변화도 생물에게 끔찍한 영향을 미칠 수 있습니다.메틸화를 증가시키거나 감소시키기 위해 발생하는 돌연변이는 유전자 조절에 큰 변화가 있는 반면, 메틸전달효소나 데메틸전달효소와 같은 효소에 대한 돌연변이는 주어진 세포에서 어떤 단백질이 전사되는지를 완전히 바꿀 수 있다.염색체의 과도한 메틸화는 정상적인 세포 기능에 필요한 특정 유전자를 비활성화 시킬 수 있다.특정 효모주에서는 제3히스톤의 3가지 리신 잔기인 H3K4, H3K36 및 H3K79를 메틸화시키는 돌연변이인 사카로미세스 세레비시아에 대해 이 진행에 필요한 많은 유전자가 비활성화되어 있는 만큼 유사분열 세포주기의 지연을 일으킨다.이 극단적인 돌연변이는 유기체의 죽음으로 이어진다.결국 히스톤 메틸전달효소의 생성을 허용하는 유전자의 결실은 리신 잔기가 [20]메틸화되지 않은 상태에서 이 유기체가 살 수 있게 한다는 것이 발견되었다.
최근 몇 년 동안 많은 종류의 암이 후생유전적 요인에 의해 발생한다는 것이 연구자들의 주목을 받고 있다.암은 히스톤의 다른 메틸화에 의해 다양한 방법으로 유발될 수 있다.종양 억제 유전자뿐만 아니라 종양 억제 유전자가 발견된 이후 암을 유발하고 억제하는 큰 요인이 우리 자신의 게놈 안에 있다는 것이 알려져 왔다.만약 종양유전자 주변이 메틸화되지 않는다면, 이 암을 유발하는 유전자들은 놀라운 속도로 전사될 수 있는 잠재력을 가지고 있다.이것의 반대는 종양 억제 유전자의 메틸화이다.이들 유전자 주변의 부위가 고도로 메틸화된 경우 종양 억제 유전자가 활성화되지 않아 암이 발생할 가능성이 높았다.메틸화 패턴의 이러한 변화는 종종 메틸전달효소 [21]및 데메틸전달효소의 돌연변이 때문이다.이소질산탈수소효소 1(IDH1)과 이소질산탈수소효소 2(IDH2)와 같은 단백질의 다른 유형의 돌연변이는 히스톤 데메틸전달효소의 불활성화를 야기할 수 있으며, 이는 돌연변이가 일어나는 [22]세포에 따라 다양한 암, 교종 및 백혈병을 야기할 수 있다.
1탄소 대사가 히스톤 메틸화를 변화시킨다.
1탄소 대사에서 아미노산 글리신 및 세린은 엽산 및 메티오닌 회로를 통해 뉴클레오티드 전구체 및 SAM으로 변환된다.포도당, 세린, 글리신, 트레오닌을 포함한 여러 영양소가 1탄소 대사를 촉진합니다.높은 수준의 메틸 공여 SAM은 히스톤 메틸화에 영향을 미치며, 이는 높은 수준의 SAM이 악성 변형을 [23]어떻게 예방하는지 설명할 수 있다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b Kramer, Jamie M. (February 2013). "Epigenetic regulation of memory: implications in human cognitive disorders". Biomolecular Concepts. 4 (1): 1–12. doi:10.1515/bmc-2012-0026. hdl:2066/118379. PMID 25436561. S2CID 6195513.
- ^ Lilja, Tobias; Nina Heldring; Ola Hermanson (February 2013). "Like a rolling histone: Epigenetic regulation of neural stem cells and brain development by factors controlling histone acetylation and methylation". Biochimica et Biophysica Acta (BBA) - General Subjects. 1830 (2): 2354–2360. doi:10.1016/j.bbagen.2012.08.011. PMID 22986149.
- ^ Hyun K, Jeon J, Park K, Kim J (2017). "Writing, erasing and reading histone lysine methylations". Experimental & Molecular Medicine. 49 (4): e324. doi:10.1038/emm.2017.11. PMC 6130214. PMID 28450737.
- ^ Gupta, Swati; Se Y. Kim; Sonja Artis; David L. Molfese; Armin Schumacher; J. David Sweatt; Richard E. Paylor; Farah D. Lubin (10 March 2010). "Histone Methylation Regulates Memory Formation". The Journal of Neuroscience. 30 (10): 3589–3599. doi:10.1523/JNEUROSCI.3732-09.2010. PMC 2859898. PMID 20219993.
- ^ Greer, Eric L.; Shi, Yang (2012). "Histone Methylation: A Dynamic Mark in Health, Disease and Inheritance". Nature Reviews Genetics. 13 (5): 343–57. doi:10.1038/nrg3173. PMC 4073795. PMID 22473383.
- ^ Jia, Songtao; Wang, Yu (2009). "Degrees make all the difference" (PDF). Epigenetics. 4 (5): 1–4.
- ^ 길버트, S.F. (2010년)발달생물학.(제9판).시나우어 어소시에이츠 주식회사 35-37
- ^ Blanc, Roméo S; Richard, Stéphane (5 January 2017). "Arginine Methylation: The Coming of Age". Molecular Cell. 65 (1): 8–24. doi:10.1016/j.molcel.2016.11.003. PMID 28061334.
- ^ a b Reinberg, Danny; Zhang, Yi (2001). "Transcription regulation by histone methylation: Interplay between different covalent modifications of the core histone tails". Genes & Development. 15 (18): 2343–2360. doi:10.1101/gad.927301. PMID 11562345.
- ^ Wei S, Li C, Yin Z, Wen J, Meng H, Xue L, Wang J (2018). "Histone methylation in DNA repair and clinical practice: new findings during the past 5-years". J Cancer. 9 (12): 2072–2081. doi:10.7150/jca.23427. PMC 6010677. PMID 29937925.
- ^ a b Rice, Judd C.; Briggs, Scott D.; Ueberheide, Beatrix; Barber, Cynthia M.; Shabanowitz, Jeffrey; Hunt, Donald F.; Shinkai, Yoichi; Allis, C.David (2003). "Histone Methyltransferases Direct Different Degrees of Methylation to Define Distinct Chromatin Domains". Molecular Cell. 12 (6): 1591–1598. doi:10.1016/S1097-2765(03)00479-9. PMID 14690610.
- ^ Kooistra, Susanne Marije; Helin, Kristian (2012-04-04). "Molecular mechanisms and potential functions of histone demethylases". Nature Reviews Molecular Cell Biology. 13 (5): 297–311. doi:10.1038/nrm3327. ISSN 1471-0072. PMID 22473470. S2CID 26181430.
- ^ Cheung, Peter; Lau, Priscilla (2005). "Epigenetic Regulation by Histone Methylation and Histone Variants". Molecular Endocrinology. 19 (3): 563–73. doi:10.1210/me.2004-0496. PMID 15677708.
- ^ a b Albert, Mareike; Helin, Kristian (2010). "Histone methyltransferases in cancer". Seminars in Cell & Developmental Biology. 21 (2): 209–220. doi:10.1016/j.semcdb.2009.10.007. PMID 19892027.
- ^ Akagi, Nobuo; Sasaki, Motomichi (1975). "Preferential Inactivation of the Paternally Derived X Chromosome in the Extraembryonic Membranes of the Mouse". Nature. 256 (5519): 640–42. Bibcode:1975Natur.256..640T. doi:10.1038/256640a0. PMID 1152998. S2CID 4190616.
- ^ Elgin, Sarah C.R.; Grewal, Shiv I.S. (2003). "Heterochromatin: Silence Is Golden". Current Biology. 13 (23): R895–898. doi:10.1016/j.cub.2003.11.006. PMID 14654010. S2CID 9728548.
- ^ Ahn, J.; Lee, J. (2008). "X chromosome: X inactivation". Nature Education. 1 (1): 24.
- ^ Barski, A.; Cuddapah, S.; Cui, K.; Roh, T.; Schones, D.; Wang, Z.; Wei, G.; Chepelev, I.; Zhao, K. (2007). "High-Resolution Profiling of Histone Methylations in the Human Genome". Cell. 129 (4): 823–37. doi:10.1016/j.cell.2007.05.009. PMID 17512414. S2CID 6326093.
- ^ Chow, Jennifer; Heard, Edith (2009). "X Inactivation and the Complexities of Silencing a Sex Chromosome". Current Opinion in Cell Biology. 21 (3): 359–66. doi:10.1016/j.ceb.2009.04.012. PMID 19477626.
- ^ Jin, Y.; Rodriguez, A. M.; Stanton, J. D.; Kitazono, A. A.; Wyrick, J. J. (2007). "Simultaneous Mutation of Methylated Lysine Residues in Histone H3 Causes Enhanced Gene Silencing, Cell Cycle Defects, and Cell Lethality in Saccharomyces Cerevisiae". Molecular and Cellular Biology. 27 (19): 6832–841. doi:10.1128/mcb.00745-07. PMC 2099221. PMID 17664279.
- ^ Esteller, M (2006). "Epigenetics Provides a New Generation of Oncogenes and Tumour-suppressor Genes". British Journal of Cancer. 94 (2): 179–83. doi:10.1038/sj.bjc.6602918. PMC 2361113. PMID 16404435.
- ^ Lu, Chao; Ward, Patrick S.; Kapoor, Gurpreet S.; Rohle, Dan; Turcan, Sevin; Abdel-Wahab, Omar; Edwards, Christopher R.; Khanin, Raya; Figueroa, Maria E.; Melnick, Ari; Wellen, Kathryn E.; O'Rourke, Donald M.; Berger, Shelley L.; Chan, Timothy A.; Levine, Ross L.; Mellinghoff, Ingo K.; Thompson, Craig B. (2012). "DH Mutation Impairs Histone Demethylation and Results in a Block to Cell Differentiation". Nature. 483 (7390): 474–78. Bibcode:2012Natur.483..474L. doi:10.1038/nature10860. PMC 3478770. PMID 22343901.
- ^ Wang YP, Lei QY (2018). "Metabolic recoding of epigenetics in cancer". Cancer Commun (Lond). 38 (1): 25. doi:10.1186/s40880-018-0302-3. PMC 5993135. PMID 29784032.
추가 정보
- 오루지, 엘리아스 & 우티칼, 요헨(2018).후생유전적으로 악성 흑색종에 대처: 히스톤 리신 메틸화.임상 후생유전학 2018 10:http://https://clinicalepigeneticsjournal.biomedcentral.com/articles/10.1186/s13148-018-0583-z
- 고자니·오·시·유(2014년).염색질 시그널링에서의 히스톤 메틸화.인: 염색질의 기초 (p. 213-256).스프링거 뉴욕: 10.1007/978-1-4614-8624-4_5
- Shi, Yang; Whetstine, Johnathan R. (January 12, 2007). "Dynamic Regulation of Histone Lysine Methylation by Demethylases". Molecular Cell. 25 (1): 1–14. doi:10.1016/j.molcel.2006.12.010. PMID 17218267.